Method Article
Hochauflösende 3D-Bildgebung der Tollwut-Virusinfektion im lösungsmittelfreien Hirngewebe
In diesem Artikel
Zusammenfassung
Neuartige, immunstaining-kompatible Gewebeclearing-Techniken wie die ultimative 3D-Bildgebung von lösungsmittelgereinigten Organen ermöglichen die 3D-Visualisierung von Tollwutvirus-Hirninfektionen und ihrer komplexen zellulären Umgebung. Dicke, antikörperbeschriftete Hirngewebescheiben werden optisch transparent gemacht, um die Bildtiefe zu erhöhen und eine 3D-Analyse durch konfokale Laserscanmikroskopie zu ermöglichen.
Zusammenfassung
Die Visualisierung von Infektionsprozessen in Geweben und Organen durch Immunlabeling ist eine Schlüsselmethode in der modernen Infektionsbiologie. Die Fähigkeit, die Verteilung, den Tropismus und die Häufigkeit von Krankheitserregern innerhalb des Organgewebes zu beobachten und zu untersuchen, liefert entscheidende Daten über die Entwicklung und das Fortschreiten von Krankheiten. Mit herkömmlichen Mikroskopieverfahren ist die Immunkennzeichnung meist auf dünne Abschnitte beschränkt, die aus paraffineingebetteten oder gefrorenen Proben gewonnen werden. Die begrenzte 2D-Bildebene dieser dünnen Abschnitte kann jedoch zum Verlust wichtiger Informationen über die komplexe Struktur eines infizierten Organs und den zellulären Kontext der Infektion führen. Moderne mehrfarbige, immunstaining-kompatible Gewebeclearing-Techniken bieten jetzt eine relativ schnelle und kostengünstige Möglichkeit, hochvolumige 3D-Bildstapel von vireninfiziertem Organgewebe zu untersuchen. Durch die Exposition des Gewebes gegenüber organischen Lösungsmitteln wird es optisch transparent. Dies entspricht den Brechungsindizes der Stichprobe und führt schließlich zu einer signifikanten Reduzierung der Lichtstreuung. So können in Kombination mit langen freien Arbeitswegobjektiven große Gewebeabschnitte bis zu einer Größe von 1 mm durch die konventionelle konfokale Laserscanmikroskopie (CLSM) mit hoher Auflösung abgebildet werden. Hier beschreiben wir ein Protokoll zur Anwendung von Tiefengewebe-Bildgebung nach Gewebeklärung, um die Verteilung von Tollwutviren in infizierten Gehirnen zu visualisieren, um Themen wie Viruspathogenese, Ausbreitung, Tropismus und Neuroinvasion zu untersuchen.
Einleitung
Herkömmliche Histologietechniken basieren meist auf dünnen Abschnitten von Organgeweben, die von Natur aus nur 2D-Einblicke in eine komplexe 3D-Umgebung liefern können. Obwohl grundsätzlich machbar, erfordert die 3D-Rekonstruktion aus seriellen dünner Abschnitte anspruchsvolle technische Rohrleitungen sowohl für das Schneiden als auch für die anschließende Silico-Ausrichtung der aufgenommenen Bilder1. Darüber hinaus ist eine nahtlose Rekonstruktion von Z-Volumes nach dem Mikrotom-Schneiden von entscheidender Bedeutung, da sowohl mechanische als auch rechnerische Artefakte aufgrund einer suboptimalen Bildregistrierung bleiben können, die durch nicht überlappende Bildebenen, Färbungsvariationen und physikalische Zerstörung von Gewebe z.B. durch die Mikrotomklinge. Im Gegensatz dazu ermöglicht das reine optische Schneiden intakter dicker Gewebeproben die Erfassung überlappender Bildebenen (Oversampling) und erleichtert damit die 3D-Rekonstruktion. Dies wiederum ist sehr vorteilhaft für die Analyse von Infektionsprozessen in komplexen Zellpopulationen (z.B. neuronale Netzwerke im Kontext der umgebenden Glia- und Immunzellen). Zu den inhärenten Hindernissen dicker Gewebeabschnitte gehören jedoch Lichtstreuung und ein begrenztes Eindringen von Antikörpern in das Gewebe. In den letzten Jahren wurde eine Vielzahl von Techniken entwickelt und optimiert, um diese Probleme zu überwinden2,3,4,5,6,7,8 , 9 , 10 , 11 , 12 , 13. Im Wesentlichen werden Zielgewebe durch Behandlung mit wässrigen2,3,4,5,6,7 optisch transparent gedreht ,8,9 oder organische lösemittelbasierte10,11,12,13 Lösungen. Die Einführung von 3DISCO (3D-Bildgebung von lösungsmittelgereinigten Organen)11,12 und seinem Nachfolger uDISCO (ultimative 3D-Bildgebung von lösungsmittelgereinigten Organen)13 bot ein relativ schnelles, einfaches und kostengünstiges Werkzeug mit hervorragende Clearing-Fähigkeiten. Die Hauptbestandteile des Clearingprotokolls sind die organischen Lösungsmittel tert-Butanol (TBA), Benzylalkohol (BA), Benzylbenzoat (BB) und Diphenylether (DPE). Die Entwicklung und Zugabe von iDISCO (immunolabeling-enabled 3D imaging of solvent-cleared organs)14, ein kompatibles Immunstaining-Protokoll, stellte einen weiteren Vorteil gegenüber bestehenden Methoden dar und ermöglichte die Tiefengewebekennzeichnung von Antigenen sowie die langfristige Lagerung von immunbefleckten Proben. So ermöglicht die Kombination von iDISCO14 und uDISCO13 die hochauflösende Bildgebung von antikörperbeschriftten Proteinen in großen Gewebeabschnitten (bis zu 1 mm) mit herkömmlichem CLSM.
Die Erhaltung der komplexen Struktur eines Organs in allen drei Dimensionen ist besonders wichtig für das Hirngewebe. Neuronen bestehen aus einer sehr heterogenen zellulären Subpopulation mit sehr unterschiedlichen 3D-Morphologien, die auf ihren Neuritenprojektionen basieren (rezensiert von Masland15). Darüber hinaus besteht das Gehirn aus einer Reihe von Kompartimenten und Unterabteilungen, die jeweils aus verschiedenen zellulären Subpopulationen und Verhältnissen davon bestehen, einschließlich Gliazellen und Neuronen (überprüft von Bartheld et al.16). Als neurotropes Virus infiziert das Tollwutvirus (RABV, überprüft von Fooks et al.17) in erster Linie Neuronen, indem es ihre Transportmaschinen nutzt, um in retrograde Richtung entlang von Axonen vom primären Infektionsort zum zentralnervösen System (ZNS) zu reisen. Das hier beschriebene Protokoll (Abbildung 1A) ermöglicht die immunstainierungsunterstützte Detektion und Visualisierung von RABV- und RABV-infizierten Zellen in großen, kohärenten Bildstapeln aus infiziertem Hirngewebe. Dies ermöglicht eine unvoreingenommene, hochauflösende 3D-Bewertung der Infektionsumgebung. Es gilt für Hirngewebe aus einer Vielzahl von Arten, kann unmittelbar nach der Fixierung oder nach der Langzeitlagerung von Proben in Paraformaldehyd (PFA) durchgeführt werden und ermöglicht die Lagerung und Reimaging von gefärbten und gereinigten Proben für Monate.
Protokoll
RABV-infiziertes, PFA-fixiertes archiviertes Hirnmaterial wurde verwendet. Die jeweiligen Tierversuchsstudien wurden von der zuständigen Tierpflege-, Einsatz- und Ethikkommission des Landesamtes für Landwirtschaft, Lebensmittelsicherheit und Fischerei in Mecklenburg-Vorpommern (LALFF M-V) ausgewertet und mit Genehmigungen genehmigt. 7221.3-2.1-002/11 (Mäuse) und 7221.3-1-068/16 (Ferrets). Die allgemeine Pflege und die in den Tierversuchen verwendeten Methoden wurden nach den genehmigten Richtlinien durchgeführt.
VORSICHT: Dieses Protokoll verwendet verschiedene toxische und/oder schädliche Substanzen, einschließlich PFA, Methanol (MeOH), Wasserstoffperoxid (H2O2 ), Natriumazid (NaN3), TBA, BA, BB und DPE. MeOH und TBA sind leicht entzündlich. Vermeiden Sie die Exposition, indem Sie geeignete persönliche Schutzausrüstung (Labormantel, Handschuhe und Augenschutz) tragen und Experimente in einer Dunstabzugshaube durchführen. Sammeln Sie Abfälle getrennt in geeigneten Behältern und entsorgen Sie sie gemäß den örtlichen Vorschriften. Das Tollwutvirus wird als Biosicherheits-Level (BSL)-2-Erreger eingestuft und kann daher in der Regel unter BSL-2-Bedingungen behandelt werden. Einige Aktivitäten, einschließlich Verfahren, die Aerosole erzeugen, mit hohen Viruskonzentrationen arbeiten oder mit neuartigen Lyssaviren arbeiten, können eine BSL-3-Klassifizierung erfordern. Die Präexpositionsprophylaxe wird für Hochrisikopersonal empfohlen, einschließlich Tierpfleger und Laborarbeiter18,19. Beziehen Sie sich auf die Vorschriften der lokalen Behörden.
1. Fixierung von Hirngewebe und Schnitt
- Fixieren Sie Hirnproben in einem geeigneten Volumen von 4% PFA in phosphatgepufferter Saline (PBS [pH 7.4]) für mindestens 48 h bei 4 °C (mit einem ungefähren Gewebe-zu-Fixativen Verhältnis von 1:10 [v/v]).
- Waschen Sie die Gewebeproben 3x in PBS für mindestens 30 min jeder Wäsche und lagern Sie sie in 0,02% NaN3/PBS bei 4 °C bis zur Verwendung.
- Schneiden Sie das Gewebe mit einem Vibratom in 1 mm dicke Abschnitte (Blattvorschubrate: 0,3–0,5 mm/s, Amplitude: 1 mm, Scheibendicke: 1.000 m).
- Um die richtige Slice-Sequenz beizubehalten, bewahren Sie jeden Gewebeabschnitt separat in einem Brunnen einer Multiwell-Zellkulturplatte auf. Fügen Sie 0,02% NaN3/PBS hinzu und lagern Sie die Gewebeabschnitte bei 4 °C bis zur Anwendung.
2. Probenvorbehandlung mit Methanol
HINWEIS: Führen Sie alle Inkubationsschritte mit sanfter Schwingung und, wenn nicht anders angegeben, bei Raumtemperatur durch. Schützen Sie die Proben vor Licht. Die Probenvorbehandlung dient dem allgemeinen Zweck der Verbesserung der Antikörperdiffusion und der Verringerungder Gewebeautofluoreszenz durch Exposition gegenüber MeOH bzw. H2 O2 , bzw.14.
- Bereiten Sie 20% (v/v), 40%, 60% und 80% MeOH-Lösungen in destilliertem Wasser vor. Zum Beispiel, für 20% MeOH, fügen Sie 10 ml von 100% MeOH zu 40 ml destilliertem Wasser in einem geeigneten verschließbaren Gefäß und mischen, indem Sie es invertieren.
- Übertragen Sie die Proben auf halbwegs große Gefäße (z. B. 5 ml Reaktionsrohre). Achten Sie darauf, Materialien zu verwenden, die chemisch beständig gegen die in diesem Protokoll verwendeten Reagenzien sind. Beachten Sie beispielsweise, dass Polypropylen zwar geeignet ist, Polystyrol jedoch nicht geeignet ist.
HINWEIS: Die Volumenspezifikationen in diesem Protokoll beziehen sich auf 5 ml Reaktionsrohre. Wenn ein anderes Gefäß verwendet wird, passen Sie die Volumina entsprechend an. - Inkubieren Sie die Proben in 4 ml jeder Konzentration der vorbereiteten Serie von MeOH-Lösungen in aufsteigender Reihenfolge für jeweils 1 h.
- Inkubieren Sie die Proben 2x für je 1 h in rein (100%) MeOH.
- Kühlen Sie die Proben auf 4 °C (z.B. in einem laborsicheren Kühlschrank).
- Bereiten Sie Bleichlösung (5% H2O2 in MeOH) vor, indem Sie z.B. eine 30% H2 O2-Lagerlösung bei 1:6 in rein (100%) verdünnen MeOH, und kühlen Sie es bei 4 °C.
- Entfernen Sie die 100% MeOH aus den gekühlten Proben und fügen Sie 4 ml vorgekühlte Bleichlösung hinzu (5% H2O2 in MeOH). Über Nacht bei 4 °C inkubieren.
- Tauschen Sie die Bleichlösung gegen 4 ml 80% MeOH und inkubieren Sie für 1 h. Verwenden Sie die vorbereitete Reihe von MeOH-Lösungen in absteigender Reihenfolge für jeweils 1 h, bis die Proben für 1 h in 4 ml von 20% MeOH inkubiert wurden.
- Waschen Sie die Proben 1x für 1 h mit 4 ml PBS.
3. Immunostaining
HINWEIS: Führen Sie alle Inkubationsschritte mit sanfter Schwingung und, wenn nicht anders angegeben, bei Raumtemperatur durch. Schützen Sie die Proben vor Licht. Um mikrobielles Wachstum zu verhindern, fügen Sie NaN3 zu einer Endkonzentration von 0,02% zu den Lösungen in diesem Abschnitt hinzu. Gewebeproben werden durch die Behandlung mit den nichtionischen Reinigungsmitteln Triton X-100 und Tween 20 weiter durchlässig. Normales Serum wird verwendet, um die unspezifische Antikörperbindung zu blockieren. Glycin und Heparin werden hinzugefügt, um den Immunlabeling-Hintergrund14zu reduzieren.
- Waschen Sie die Proben 2x für je 1 h in 4 ml 0,2% Triton X-100/PBS.
- Permeabilisieren Sie die Proben für 2 Tage bei 37 °C mit 4 ml 0,2% Triton X-100/20% DMSO/0,3 M Glycin/PBS.
- Blockieren Sie die unspezifische Bindung von Antikörpern, indem Sie die Proben 2 Tage lang bei 37 °C in 4 ml mit 0,2 % Triton X-100/10% DMSO/6% normales Serum/PBS inkubieren.
HINWEIS: Verwenden Sie normales Serum von derselben Spezies, in dem der sekundäre Antikörper aufgezogen wurde, um ideale Blockierungsergebnisse zu erzielen. - Inkubieren Sie die Proben in 2 ml Primärantikörperlösung (3% normales Serum/5% DMSO/PTwH [PBS-Tween 20 mit Heparin] + Primärantikörper/Antikörper) 5 Tage bei 37 °C. Erfrischen Sie die primäre Antikörperlösung nach 2,5 Tagen.
- Für PTwH das Heparin-Natriumsalz in destilliertem Wasser zu einer 10 mg/ml-Stammlösung rekonstituieren (diese Lösung, aliquoted, bei 4 °C lagern). Fügen Sie die Lagerlösung auf 0,2% Tween 20/PBS zu einer Endkonzentration von 10 g/ml hinzu.
HINWEIS: Die Auswahl der richtigen Antikörperverdünnung kann eine Optimierung erfordern. Im Allgemeinen sind Standard-Immunhistochemie-Konzentrationen ein guter Ausgangspunkt.
- Für PTwH das Heparin-Natriumsalz in destilliertem Wasser zu einer 10 mg/ml-Stammlösung rekonstituieren (diese Lösung, aliquoted, bei 4 °C lagern). Fügen Sie die Lagerlösung auf 0,2% Tween 20/PBS zu einer Endkonzentration von 10 g/ml hinzu.
- Waschen Sie die Proben für 1 Tag in 4 ml PTwH, tauschen Sie den Waschpuffer mindestens 4x–5x im Laufe des Tages und lassen Sie die letzte Wäsche über Nacht.
- Inkubieren Sie die Proben in 2 ml sekundärer Antikörperlösung (3% normales Serum/PTwH + Sekundärantikörper/Antikörper) für 5 Tage bei 37 °C. Erfrischen Sie die sekundäre Antikörperlösung nach 2,5 Tagen.
- Verdünnen Sie den sekundären Antikörper/Antikörper bei 1:500 in sekundärer Antikörperlösung (d. h. 4 l in 2 ml).
- Waschen Sie die Proben für 1 Tag, wie in Schritt 3.5 beschrieben, so dass die letzte Wäsche über Nacht eingeschaltet.
4. Nukleare Färbung
HINWEIS: Führen Sie alle Inkubationsschritte mit sanfter Schwingung und, wenn nicht anders angegeben, bei Raumtemperatur durch. Schützen Sie die Proben vor Licht. Wenn keine kerntechnische Färbung erforderlich ist oder die Anregungswellenlänge/Emissionsspektrum von TO-PRO-3 für die Anregung oder detektion eines anderen Fluorophors erforderlich ist, überspringen Sie diesen Schritt.
- Den Nukleinsäurefleck TO-PRO-3 bei 1:1.000 in PTwH verdünnen und die Proben in 4 ml Kernfärbungslösung für 5 h inkubieren.
- Waschen Sie die Proben für 1 Tag, wie in Schritt 3.5 beschrieben, so dass die letzte Wäsche über Nacht eingeschaltet.
HINWEIS: Nach den Wässern können die Proben bis zur optischen Räumung bei 4 °C in PBS gelagert werden.
5. Geweberäumung
HINWEIS: Führen Sie alle Inkubationsschritte mit sanfter Schwingung und, wenn nicht anders angegeben, bei Raumtemperatur durch. Schützen Sie die Proben vor Licht. Die Gewebeproben werden in einer abgestuften Reihe von TBA-Lösungen dehydriert. Da die Immunfärbung wässrige Lösungen erfordert, müssen alle Färbeverfahren vor der Geweberäumung abgeschlossen sein. Optische Clearance und Brechungsindex-Matching werden durch die Behandlung mit einer Mischung aus BA, BB und DPE erreicht. Die Clearing-Lösung wird mit DL-A-Tocopherol als Antioxidans13ergänzt.
- Bereiten Sie 30% (v/v), 50%, 70%, 80%, 90% und 96% TBA-Lösungen in destilliertem Wasser vor. Zum Beispiel, für 30% TBA, fügen Sie 15 ml von 100% TBA zu 35 ml destilliertem Wasser in einem geeigneten verschließbaren Gefäß und mischen durch Inverting.
HINWEIS: TBA hat einen Schmelzpunkt von 25–26 °C; daher neigt es dazu, bei Raumtemperatur fest zu sein. Um TBA-Lösungen vorzubereiten, erhitzen Sie die gut verschlossene Flasche bei 37 °C in einem Inkubator oder Wasserbad. - Dehydrieren Sie die Proben mit 4 ml jeder Konzentration der vorbereiteten Serie von TBA-Lösungen in aufsteigender Reihenfolge für jeweils 2 h. Lassen Sie die 96% TBA über Nacht.
- Dehydrieren Sie die Proben weiter in rein (100%) TBA für 2 h.
- Vorbereiten der Clearinglösung BABB-D15.
HINWEIS: BABB-D15 ist eine Kombination aus BA und BB (BABB), die mit DPE im Verhältnis x:1 gemischt wird, wobei x im Namen der Lösung angegeben ist, in diesem Fall 15.- Für BABB einteiliges BA mit zwei Teilen BB mischen.
- Mischen Sie BABB und DPE im Verhältnis 15:1.
- Fügen Sie 0,4 Vol% DL--Tocopherol (Vitamin E) hinzu.
HINWEIS: Zum Beispiel für 20 ml BABB-D15 6,25 ml BA mit 12,5 ml BB mischen. Fügen Sie 1,25 ml DPE hinzu und ergänzen Sie es mit 0,08 ml DL-A-Tocopherol.
- Löschen Sie die Proben in der Clearinglösung, bis sie optisch transparent sind (2-6 h).
- Die Proben können bei 4 °C in BABB-D15, vor Licht geschützt, bis zur Montage und Bildgebung gelagert werden.
6. Probenmontage
- Drucken Sie mit einem 3D-Drucker die Bildkammer und den Deckel (Material: copolyester [CPE], Düse: 0,25 mm, Schichthöhe: 0,06 mm, Wandstärke: 0,88 mm, Wandanzahl: 4, Füllung: 100%, keine Tragstruktur; die entsprechende . Die STL-Datei finden Sie in den Ergänzenden Materialien dieses Protokolls).
- Montieren Sie die Bildgebungskammer (Abbildung 2).
- Montieren Sie einen runden Abdecksch (Durchmesser: 30 mm) auf der Bildkammer mit RTV-1 (Einkomponenten Raum-Temperatur-vulkanisierender) Silikonkautschuk. Entfernen Sie den überschüssigen Silikonkautschuk mit einem wasserbenetzten Wattestäbchen und härten Sie ihn über Nacht aus.
- Montieren Sie einen runden Abdeckschlupf (Durchmesser: 22 mm) auf dem Deckel mit RTV-1 Silikonkautschuk. Entfernen Sie den überschüssigen Silikonkautschuk mit einem wasserbenetzten Wattestäbchen und härten Sie ihn über Nacht aus.
- Legen Sie die Probe in eine Bildkammer, fügen Sie ein kleines Volumen BABB-D15 hinzu, und legen Sie den Deckel ein. Füllen Sie die Kammer mit BABB-D15 durch den Einlass, mit einer hypodermischen Nadel (27 G x 3/4 Zoll [0,40 mm x 20 mm]).
- Stecken Sie den Einlass und versiegeln Sie die Bildkammer mit RTV-1 Silikonkautschuk. Über Nacht im Dunkeln heilen.
7. Bildgebung und Bildverarbeitung
- Richten Sie die Bildaufnahme ein, indem Sie die jeweiligen Laserlinien entsprechend den verwendeten Fluorophoren auswählen. Passen Sie die Erfassungsbereiche jedes Detektors an, um Signalüberlappungen zwischen Kanälen zu verhindern.
HINWEIS: Die Beispielreichweiten für Alexa Fluor 488, Alexa Fluor 568 und TO-PRO-3 betragen 500–550 nm, 590–620 nm bzw. 645–700 nm. - Wählen Sie die Erfassungsparameter aus, definieren Sie den oberen und unteren Rand des Z-Stacks, und erfassen Sie den Image-Stack.
HINWEIS: Beispielhafte Erfassungsparameter sind ein sequenzieller Scan mit einer Pixelgröße von 60-90 nm, einer Z-Schritt-Größe von 0,5 m, einem Liniendurchschnitt von 1, einer Scangeschwindigkeit von 400 Hz und einer Lochgröße von 1 Airy-Einheit. - Verarbeiten Sie den Bildstapel mit einer geeigneten Bildanalysesoftware (z. B. Fidschi), um 3D-Projektionen zu generieren oder detaillierte Analysen durchzuführen.
HINWEIS: Aufgrund der großen Größe der erworbenen Bilddateien ist in der Regel die Verwendung einer Arbeitsstation erforderlich.- Öffnen Sie die Aufnahme- oder Bilddatei(en) in Fidschi (Datei | Öffnen | Dateien auswählen).
- Wenn Sie z. B. . LIF-Dateien, wählen Oder deaktivieren Sie die Gewünschten Optionen im Dialogfenster Bio-Formats. Stapel mit Hyperstackanzeigen . Abgesehen davon sind keine spezifischen Auswahlen oder Ticks erforderlich. Drücken Sie OK.
- Wenn die Datei mehrere Bildstapel enthält, wählen Sie die zu analysierenden aus und bestätigen Sie, indem Sie OKdrücken.
- Bleichkorrektur durchführen, indem das zusammengeführte Bild in einzelne Kanäle aufgeteilt wird (Bild | Farbe | Channels Tool, wählen Sie dann Mehr | Split-Kanäle). Wählen Sie für jeden Kanal die Bleichkorrektur aus (Bild | Anpassen | Bleichkorrektur) und wählen Sie Simple Ratio (Hintergrundintensität: 0,0).
HINWEIS: In einigen Fällen, zum Beispiel, wenn es keinen linearen Zerfall des Signals oder das Signal ist zu schwach insgesamt, Simple Ratio kann fehlschlagen. Alternativ können Sie Exponential Fit ausprobieren oder die Bleichkorrektur überspringen. - Passen Sie die Helligkeit und den Kontrast für jeden Kanal mit den Schiebereglern (Bild | Anpassen | Helligkeit | Kontrast).
- Zusammenführen der Kanäle (Bild | Farbe | Merge-Kanäle), erstellen Sie einen Verbund (Bild | Farbe | Channels Tool, wählen Sie dann Mehr | Erstellen Sie Composite, und konvertieren Sie es in das RGB-Format (Bild | Farbe | Channels Tool, wählen Sie dann Mehr | Konvertieren in RGB).
- Ändern Sie bei Bedarf die Größe des Bildstapels, um die Berechnungszeit und Dateigröße zu reduzieren(Bild | Anpassen | Größe, beide Optionen angekreuzt plus bilineare Interpolation).
- Generieren einer 3D-Projektion (Bild | Stapel | 3D-Projekt). Wählen Sie Hellester Punkt als Projektionsmethode aus, und legen Sie den Slice-Abstand so fest, dass er der Z-Schritt-Größe des erworbenen Bildstapels entspricht. Legen Sie für maximale Qualität das Drehwinkelinkrement auf 1 fest, und aktivieren Sie die Interpolation. Ändern Sie die Gesamtrotation, Transparenzschwellenwerte und Deckkraft nach Bedarf.
- Stellen Sie bei Bedarf den Kontrast und die Helligkeit wieder ein, indem Sie die 3D-Projektion wieder in das 8-Bit-Format konvertieren (Bild | Typ | 8-Bit). Verwenden Sie die entsprechenden Schieberegler (Bild | Anpassen | Helligkeit | Kontrast) und konvertieren Sie den Bildstapel wie in Schritt 7.3.4 beschrieben in das RGB-Format.
- Speichern Sie die 3D-Projektion als . TIF-Datei (Bilddateiformat) und . AVI-Datei (Videodateiformat).
- Öffnen Sie die Aufnahme- oder Bilddatei(en) in Fidschi (Datei | Öffnen | Dateien auswählen).
Ergebnisse
Die Kombination von iDISCO14 und uDISCO13 in Verbindung mit hochauflösendem CLSM bietet tiefe Einblicke in die raumzeitliche Auflösung und Plastizität der RABV-Infektion von Hirngewebe und den umgebenden zellulären Kontext.
Mit Der Immunostainierung von RABV-Phosphoprotein (P) können komplexe Schichten infizierter neuronaler Zellen in dicken Abschnitten des Mausgehirns visualisiert werden (Abbildung 3). Anschließend können nahtlose 3D-Projektionen der erfassten Bildstapel rekonstruiert werden (Abbildung3A, B, rechte samfele; Animierte Abbildung 1). Bei der Verwendung von Primärantikörpern und sekundären Antikörpern gegen die Art, aus der das Organmaterial stammt, ist Vorsicht geboten. Die Verwendung von Anti-Maus-IgG-Antikörpern auf Maus-Hirngewebe führte zu einer deutlichen Färbung des Gefäßsystems (Abbildung3B, linkes Panel). Aufgrund der hohen Auflösung, mit der die Bildstapel erfasst werden, kann die Infektion bis zu einer Einzelzellebene beurteilt werden (Abbildung 4), so dass Behauptungen beispielsweise auf die Häufigkeit und Verteilung von Antigen innerhalb der Zelle (Abbildung4C) ).
Neben dem Gehirngewebe der Maus kann das Protokoll auch auf Hirngewebe anderer Tierarten (z. B. Frettchen) angewendet werden (Abbildung5; Animierte Abbildung 2). Abschnitte aus verschiedenen Fächern eines infizierten Frettchengehirns ergaben einen unterschiedlichen Grad der RABV-Infektion (Abbildung 5A-D).
Da das Gehirn viele verschiedene zelluläre Subpopulationen umfasst, ist die Differenzierung zwischen diesen Populationen von entscheidender Bedeutung. Mit Antikörpern gegen Zellmarker ist eine Bewertung der zellulären Identität infizierter und benachbarter Zellen möglich. Beispielsweise können Astrozyten durch die Expression von glialfibrillärem saurem Protein (GFAP) unterschieden werden (Abbildung 6A,C; Animierte Abbildung 3), während Neuriten speziell für mikrotubuli-assoziiertes Protein 2 (MAP2) gebeizt werden können (Abbildung 6B,D; Animierte Abbildung 4). Gleichzeitig können virale Proteine, in diesem Fall RABV-Nukleoprotein (N), costainiert werden, um die Beziehung zwischen infizierten Zellen und der hervorgehobenen zellulären Subpopulation zu bewerten.

Abbildung 1 : Grundprinzip und Workflow des Protokolls. (A) Grafische Darstellung des Workflows auf Basis der Protokolle von Renier et al.14 und Pan et al.13. (B) Zwei beispielhafte Frettchen-Gehirnscheiben, eine vor (linkes Panel) und eine nach der Behandlung mit organischen Lösungsmitteln (rechte sam). Das Clearing macht das Gewebe optisch transparent, wie es durch den dann lesbaren Text beobachtbar ist. Die gelöschte Hirnscheibe ist in eine 3D-gedruckte Bildgebungskammer (rechte sam) eingebettet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
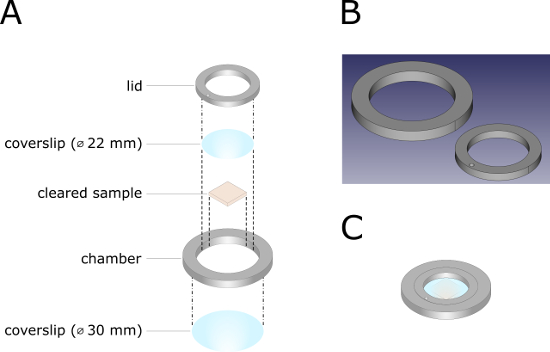
Abbildung 2 : Technische Darstellung der 3D-gedruckten Bildgebungskammer. (A) Explosionsansichtszeichnung der Bildkammer. Die punktgestrichten Linien markieren Komponenten, die mit RTV-1 Silikonkautschuk aufeinander montiert werden müssen. Die gestrichelten Linien stellen Anweisungen für die nachfolgende Montage der Kammer dar. (B) CAD-Datei (computergestütztes Design) der Bildkammer. Die entsprechende . STL-Datei zum Drucken der Bildkammer finden Sie in den Ergänzenden Materialien. (C) Vollständig montierte Bildgebungskammer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3 : Tiefengewebe-Bildgebung von RABV-infiziertem Maus-Hirngewebe. (A und B) Mäuse wurden mit einem rekombinanten Vulpine-Straßenvirus infiziert. RABV P Färbung (grün) zeigt infizierte neuronale Schichten mit großen, verschränkten Neuritenprojektionen im gereinigten Hirngewebe. Nuclei wurden mit TO-PRO-3 (blau) kontert. (B) Die Verwendung von fluorophor-markierten Anti-Maus-IgG-Sekundärantikörpern (rot) auf Mausgewebe führt zu einer deutlichen Kennzeichnung des Gefäßsystems. Die 3D-Rekonstruktion der erfassten Bildstapel ermöglicht die Beobachtung aus verschiedenen Betrachtungswinkeln (A und B,rechte Paneele). Skalenbalken = 60 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4 : Die hochauflösende Bildaufnahme ermöglicht komplexe Bewertungen bis zu einer Einzelzellenebene. Das Gehirn wurde von einer SAD L16-infizierten Maus seziert. (A) RABV P Färbung (grün) hebt ein individuell infiziertes Neuron hervor. (B und C) Detailbilder und Projektionen zeigen, dass eingehende Analysen der Antigenfülle, -verteilung und -lokalisierung durchgeführt werden können. Nuclei wurden mit TO-PRO-3 (blau) kontert. Skalenbalken = 20 m (A), 5 m (C). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
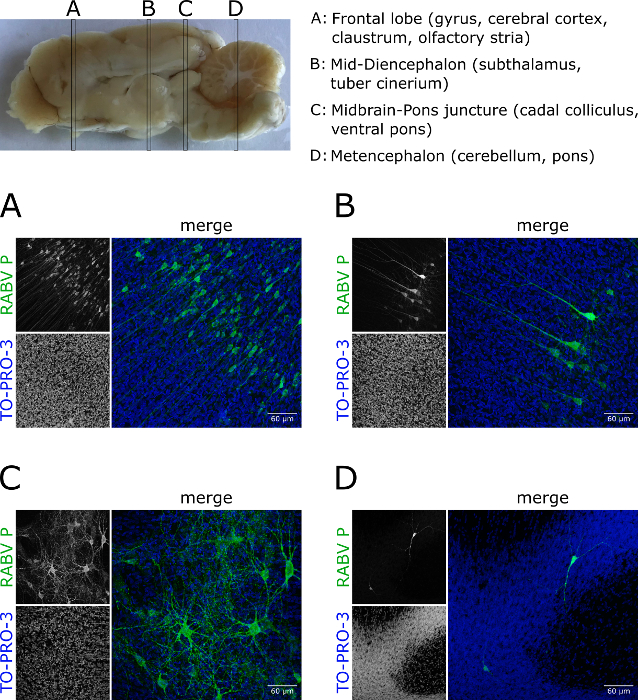
Abbildung 5 : Tiefengewebe-Bildgebung von RABV-infiziertem Frettchen-Hirngewebe. Frettchen wurden mit der Weinstraße RABV infiziert. (A–D) Scheiben aus bestimmten Bereichen des Gehirns wurden entnommen, immunstainiert für RABV P (grün) und optisch gelöscht. Projektionen zeigen, dass sich die infizierten Zellen in verschiedenen Teilen des Gehirns in Menge und Morphologie unterscheiden. Darüber hinaus unterstreichen sie die Anwendbarkeit des Protokolls auf andere Gewebe als mausabgeleitete Gewebe. Nuclei wurden mit TO-PRO-3 (blau) kontert. Skalenbalken = 60 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6 : Die multicolor-Immunfluoreszenz ermöglicht die Costainierung von Zellmarkern. Hirngewebescheiben von Frettchen, die mit der Canine Street RABV infiziert sind, wurden verwendet und für RABV N (rot) und entweder (A und C) GFAP (grün) oder (B und D) MAP2 (grün) koimmunstainiert. Während GFAP ein Astrozytenmarker ist, hebt MAP2 speziell Neuriten hervor. Nuclei wurden mit TO-PRO-3 (blau) kontert. (C und D) Am unteren Rand werden einzelne Slice-Extraktionen der aufgezählten Detailansichten aus den zusammengeführten Projektionen dargestellt. Skalenbalken = 15 m (A und B) 10 m (C und D). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Animierte Abbildung 1: 3D-Rekonstruktionen und Detailprojektionen von Bildstapeln von RABV-infizierten Maushirnen. Die Projektionen wurden aus den in Abbildung 3beschriebenen Bildstapeln generiert. (A und C) Animationen der gesamten z-Stacks. (B) Eine detaillierte Projektion einer 3D-Rekonstruktion eines Teils des Z-Stacks der am Anfang des Videos hervorgehobenen Bereiche. (D) Eine detaillierte Projektion eines Tomogramms eines Teils des Z-Stacks der am Anfang des Videos hervorgehobenen Bereiche. Grün = RABV P; rot = Maus IgG; blau = Kerne. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)

Animierte Abbildung 2: 3D-Rekonstruktionen von Bildstapeln, die von einem RABV-infizierten Frettchenhirn erworben wurden. Die Projektionen wurden aus den in Abbildung 5beschriebenen Bildstapeln generiert. (A–D) Die Anmerkungen beziehen sich auf die gleiche Figur und beschreiben die jeweiligen Bereiche des Gehirns, aus denen die Scheiben entnommen wurden. Grün = RABV P; blau = Kerne. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)

Animierte Abbildung 3: Schrittweise Projektion der verschiedenen Kanäle einer 3D-Rekonstruktion eines RABV-infizierten Frettchenhirns mit Astrozytenmarker GFAP. Projektionen wurden aus dem in Abbildung 6Abeschriebenen Bildstapel generiert. Die allmähliche Zugabe von Kanälen beginnt mit RABV N (rot), danach folgen die Zellkerne (blau) und schließlich GFAP (grün), während die Subtraktion zuerst RABV N, dann die Zellkerne und schließlich GFAP entfernt. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)

Animierte Abbildung 4: Schrittweise Projektion der verschiedenen Kanäle einer 3D-Rekonstruktion eines RABV-infizierten Frettchenhirns, das mit dem neuronalen Marker MAP2 gecostainiert ist. Projektionen wurden aus dem in Abbildung 6Bbeschriebenen Bildstapel generiert. Die allmähliche Addition von Kanälen beginnt mit RABV N (rot), danach folgen sie den Zellkernen (blau) und schließlich MAP2 (grün), während die Subtraktion zuerst RABV N, dann die Zellkerne und schließlich MAP2 entfernt. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)
Diskussion
Das Wiederaufleben und die Weiterentwicklung von Gewebeclearing-Techniken in den letzten Jahren2,3,4,5,6,7,8, 9 , 10 , 11 , 12 , 13 , 14 haben viele neue Möglichkeiten eröffnet, um großvolumige Bildstapel von Organgewebe zu erhalten. Dies bot ein unvergleichliches und leistungsfähiges Werkzeug, um zu studieren, unter vielen anderen Themen, Virusinfektion. Die anschließende 3D-Rekonstruktion dieser Bildstapel ermöglicht ausgeklügelte Behauptungen zum Beispiel über Virustropismus, Überfluss und den Zeitlichen der Infektion. Dieses Protokoll beschreibt die immunlabeling-unterstützte Visualisierung einer RABV-Infektion im lösungsmittelgereinigten Hirngewebe.
Es gibt mehrere kritische Schritte bei der Herstellung und Erfassung der Bildstapel von immunmarkiertem, lösungsmittelgereinigtem Gewebe. Längere Exposition gegenüber PFA kann Epitope maskieren und somitzu einer verminderten Antigenität 20,21,22führen. Daher ist es wichtig, die Fixierungszeiten auf das notwendige Minimum zu begrenzen und die Proben auf eine geeignete Lösung zu übertragen (z. B. PBS, ergänzt durch 0,02 % NaN3 für die Langzeitlagerung). Alle hier zur Verfügung gestellten repräsentativen Bilder und Projektionen wurden jedoch aus archivierten Hirnproben gewonnen, von denen einige wochenlang in PFA gelagert worden waren, was die Anwendbarkeit der Technik auf Organmaterial unterstreicht, das PFA für längere Zeiträume. Ähnliche Ergebnisse wurden für menschliche Gehirnprobengezeigt 13. Ein weiterer, wenn nicht der kritischste Schritt ist die Antikörperinkubation. Antikörperkonzentrationen müssen sorgfältig ausgewählt werden. Während in den meisten Fällen Standard-IHC-Konzentrationen ein guter Ausgangspunkt für die Kennzeichnung von Tiefgewebe-Antikörpern sind, können einige Antigene eine zusätzliche Optimierung der Antikörperkonzentration erfordern. Dieses Protokoll zeigt, dass sowohl RABV N als auch P durch die verwendeten Antikörper leicht nachweisbar sind. Während die MeOH-Vorbehandlung in der Regel die Immunkennzeichnung verbessert, sind einige Antigene mit dieser Behandlung nicht kompatibel. Für diese boten Renier und Kollegen14 eine alternative MeOH-freie Probenvorbehandlung an. Die Analyse der erfassten Bildstapel erfordert eine ausreichende Rechenleistung. Aufgrund der großen Dateigrößen von bis zu mehreren Gigabyte pro Stack werden leistungsstarke Computer benötigt, um die Images zu verarbeiten. Die Nachbearbeitung beinhaltet häufig die Subtraktion von Hintergrundgeräuschen und eine Bleichkorrektur, um akquisitionsassoziierte Bleicheffekte auszugleichen. Bei der Entfernungsmessung ist zu beachten, dass das Gewebe als Nebeneffekt während des Räumprozesses schrumpft.
Im Vergleich zu anderen Mikroskopieplattformen wie der Lichtbogenfluoreszenzmikroskopie (LSFM) oder der Zwei-Photonen-Laserscanmikroskopie (2PLSM) hat CLSM den begrenztsten Arbeitsabstand. Daher ist es notwendig, Organe bis 1 mm Dicke für die Bildgebung vorzuschneiden. Darüber hinaus hat CLSM aufgrund seiner hohen Bildauflösung die langsamste Erfassungsgeschwindigkeit. Der Einsatz eines Lichtbogenmikroskops würde eine schnellere Bildgebung größerer Volumina bis hin zur Bildgebung des gesamten Gehirns ermöglichen und gleichzeitig die Bildauflösung opfern. Eine weitere Einschränkung ist die inhärente Zunahme der Gewebe-Autofluoreszenz mit abnehmender Wellenlänge. Dies macht die Verwendung von Fluorophoren und Farbstoffen, die durch die Laserlinie bei 405 nm angeregt werden, einschließlich Hoechst-Farbstoffen, unpraktisch bis unmöglich.
Im Vergleich zu anderen Geweberäumtechniken vereint der Hybrid von iDISCO14 und uDISCO13 die positivsten Eigenschaften: Er ist hochkompatibel mit immunostainierenden, sehr vielseitigen, vergleichsweise schnellen und kostengünstigen hervorragende Räumfähigkeiten, und es ist mit einem Standard-Konfokalmikroskop machbar. Darüber hinaus beschränkt sich dieses Protokoll nicht auf die Immunfärbung und Reinigung von Hirngewebe, sondern kann auch auf eine Vielzahl anderer Weichteile und Krankheitserreger angewendet werden, sowohl neuroinvasorische als auch nicht-neuroinvasive. Zusammenfassend stellt das hier beschriebene Protokoll eine Pipeline für die hochauflösende 3D-Bildgebung von RABV-infiziertem Hirngewebe dar. Die 3D-Rekonstruktionen von infiziertem Hirngewebe können verwendet werden, um eine Vielzahl von Fragen bezüglich der RABV-Krankheitsprogression, Pathogenese und Neuroinvasion zu beantworten.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren danken Thomas C. Mettenleiter und Verena te Kamp für die kritische Lektüre des Manuskripts. Diese Arbeit wurde unterstützt durch die Bundesexzellenzinitiative Mecklenburg-Vorpommern und den Europäischen Sozialfonds (ESF) Zuschuss KoInfekt (ESF/14-BM-A55-0002/16) und ein intramurales Gemeinschaftsforschungsstipendium zu Lyssaviren an der Friedrich-Loeffler-Institut (Ri-0372).
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Benzyl alcohol | Alfa Aesar | 41218 | Clearing reagent |
| Benzyl benzoate | Sigma-Aldrich | BB6630-500ML | Clearing reagent |
| Dimethyl sulfoxide | Carl Roth | 4720.2 | Various buffers |
| Diphenyl ether | Sigma-Aldrich | 240834-100G | Clearing reagent |
| DL-α-Tocopherol | Alfa Aesar | A17039 | Antioxidant |
| Donkey serum | Bio-Rad | C06SBZ | Blocking reagent |
| Glycine | Carl Roth | 3908.2 | Background reduction |
| Goat serum | Merck | S26-100ML | Blocking reagent |
| Heparin sodium salt | Carl Roth | 7692.1 | Background reduction |
| Hydrogen peroxide solution (30 %) | Carl Roth | 8070.2 | Sample bleaching |
| Methanol | Carl Roth | 4627.4 | Sample pretreatment |
| Paraformaldehyde | Carl Roth | 0335.3 | Crystalline powder to make fixative solution |
| Sodium azide | Carl Roth | K305.1 | Prevention of microbial growth in stock solutions |
| tert-Butanol | Alfa Aesar | 33278 | Sample dehydration for tissue clearing |
| TO-PRO-3 | Thermo Fisher | T3605 | Nucleic acid stain |
| Triton X-100 | Carl Roth | 3051.2 | Detergent |
| Tween 20 | AppliChem | A4974,0500 | Detergent |
| Miscellaneous | |||
| 5 mL reaction tubes | Eppendorf | 0030119401 | Sample tubes |
| Coverslip, circular (diameter: 22 mm) | Marienfeld | 0111620 | Part of imaging chamber |
| Coverslip, circular (diameter: 30 mm) | Marienfeld | 0111700 | Part of imaging chamber |
| Hypodermic needle (27 G x ¾” [0.40 mm x 20 mm]) | B. Braun | 4657705 | Filling of the imaging chamber with clearing solution |
| RTV-1 silicone rubber | Wacker | Elastosil E43 | Adhesive for the assembly of the imaging chamber |
| Ultimaker CPE 2.85 mm transparent | Ultimaker | 8718836374869 | Copolyester filament for 3D printer to print parts of the imaging chamber |
| Technical equipment and software | |||
| 3D printer | Ultimaker | Ultimaker 2+ | Printing of imaging chamber |
| Automated water immersion system | Leica | 15640019 | Software-controlled water pump |
| Benchtop orbital shaker | Elmi | DOS-20M | Sample incubation at room temperature (~ 150 rpm) |
| Benchtop orbital shaker, heated | New Brunswick Scientific | G24 Environmental Shaker | Sample incubation at 37 °C (~ 150 rpm) |
| Confocal laser scanning microscope | Leica | DMI 6000 TCS SP5 | Inverted confocal microscope for sample imaging |
| Fiji | NIH (ImageJ) | open source software (v1.52h) | Image processing package based on ImageJ |
| Long working distance water immersion objective | Leica | 15506360 | HC PL APO 40x/1.10 W motCORR CS2 |
| Vibratome | Leica | VT1200S | Sample slicing |
| Workstation | Dell | Precision 7920 | CPU: Intel Xeon Gold 5118 GPU: Nvidia Quadro P5000 RAM: 128 GB 2666 MHz DDR4 SSD: 2 TB |
| Primary antibodies | |||
| Goat anti-RABV N | Friedrich-Loeffler-Institut | Monospecific polyclonal goat anti-RABV N serum, generated by goat immunization with baculovirus-expressed and His-tag-purified RABV nucleoprotein N Dilution: 1:400 | |
| Rabbit anti-GFAP | Dako | Z0334 | Polyclonal antibody (RRID:AB_10013382) Dilution: 1:100 |
| Rabbit anti-MAP2 | Abcam | ab32454 | Polyclonal antibody (RRID:AB_776174) Dilution: 1:250 |
| Rabbit anti-RABV P 160-5 | Friedrich-Loeffler-Institut | Monospecific polyclonal rabbit anti-RABV P serum, generated by rabbit immunization with baculovirus-expressed and His-tag-purified RABV phosphoprotein P (see reference 23: Orbanz et al., 2010) Dilution: 1:1,000 | |
| Secondary antibodies | |||
| Donkey anti-goat IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
Referenzen
- Pichat, J., Iglesias, J. E., Yousry, T., Ourselin, S., Modat, M. A Survey of Methods for 3D Histology Reconstruction. Medical Image Analysis. 46, 73-105 (2018).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Ke, M. T., Fujimoto, S., Imai, T. SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction. Nature Neuroscience. 16 (8), 1154-1161 (2013).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Susaki, E. A., et al. Whole-brain imaging with single-cell resolution using chemical cocktails and computational analysis. Cell. 157 (3), 726-739 (2014).
- Susaki, E. A., et al. Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging. Nature Protocols. 10 (11), 1709-1727 (2015).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Treweek, J. B., et al. Whole-body tissue stabilization and selective extractions via tissue-hydrogel hybrids for high-resolution intact circuit mapping and phenotyping. Nature Protocols. 10 (11), 1860-1896 (2015).
- Dodt, H. U., et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods. 4 (4), 331-336 (2007).
- Erturk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-171 (2011).
- Erturk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Pan, C., et al. Shrinkage-mediated imaging of entire organs and organisms using uDISCO. Nature Methods. 13 (10), 859-867 (2016).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2014).
- Masland, R. H. Neuronal cell types. Current Biology. 14 (13), 497-500 (2004).
- von Bartheld, C. S., Bahney, J., Herculano-Houzel, S. The search for true numbers of neurons and glial cells in the human brain: A review of 150 years of cell counting. The Journal of Comparative Neurology. 524 (18), 3865-3895 (2016).
- Fooks, A. R., et al. Rabies. Nature Reviews Disease Primers. 3, 17091 (2017).
- WHO. WHO Expert Consultation on Rabies, Third Report. WHO Technical Report Series. , (2018).
- CDC. . Biosafety in Microbiological and Biomedical Laboratories, 5th Edition. US Department of Health and Human Services. , (2009).
- Arnold, M. M., et al. Effects of fixation and tissue processing on immunohistochemical demonstration of specific antigens. Biotechnic & Histochemistry. 71 (5), 224-230 (1996).
- Webster, J. D., Miller, M. A., Dusold, D., Ramos-Vara, J. Effects of prolonged formalin fixation on diagnostic immunohistochemistry in domestic animals. Journal of Histochemistry and Cytochemistry. 57 (8), 753-761 (2009).
- Werner, M., Chott, A., Fabiano, A., Battifora, H. Effect of formalin tissue fixation and processing on immunohistochemistry. The American Journal of Surgical Pathology. 24 (7), 1016-1019 (2000).
- Orbanz, J., Finke, S. Generation of recombinant European bat lyssavirus type 1 and inter-genotypic compatibility of lyssavirus genotype 1 and 5 antigenome promoters. Archives of Virology. 155 (10), 1631-1641 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten