Method Article
Imágenes 3D de alta resolución de la infección por el virus de la rabia en el tejido cerebral con disolventes
En este artículo
Resumen
Las nuevas técnicas de limpieza de tejidos compatibles con la inmunomancha, como la imagen 3D definitiva de órganos con disolventes, permiten la visualización 3D de la infección cerebral del virus de la rabia y su complejo entorno celular. Las rebanadas gruesas de tejido cerebral con etiqueta de anticuerpos se hacen ópticamente transparentes para aumentar la profundidad de la imagen y permitir el análisis 3D mediante microscopía de escaneo láser confocal.
Resumen
La visualización de los procesos de infección en tejidos y órganos mediante inmunoetiquetado es un método clave en la biología moderna de la infección. La capacidad de observar y estudiar la distribución, el tropismo y la abundancia de patógenos dentro de los tejidos de órganos proporciona datos fundamentales sobre el desarrollo y la progresión de la enfermedad. Mediante métodos de microscopía convencionales, el inmunoetiquetado se limita principalmente a secciones delgadas obtenidas de muestras congeladas o incrustadas en parafina. Sin embargo, el plano de imagen 2D limitado de estas secciones delgadas puede conducir a la pérdida de información crucial sobre la estructura compleja de un órgano infectado y el contexto celular de la infección. Las modernas técnicas multicolores y compatibles con la inmunomancha ahora proporcionan una manera relativamente rápida y económica de estudiar pilas de imágenes 3D de gran volumen de tejido orgánico infectado por virus. Al exponer el tejido a disolventes orgánicos, se vuelve ópticamente transparente. Esto coincide con los índices de refracción de la muestra y, finalmente, conduce a una reducción significativa de la dispersión de la luz. Por lo tanto, en combinación con objetivos de larga distancia de trabajo libre, grandes secciones de tejido de hasta 1 mm de tamaño se pueden tomar imágenes mediante microscopía de escaneo láser confocal convencional (CLSM) a alta resolución. Aquí, describimos un protocolo para aplicar imágenes de tejido profundo después de la limpieza del tejido para visualizar la distribución del virus de la rabia en cerebros infectados con el fin de estudiar temas como la patogénesis del virus, la propagación, el tropismo y la neuroinvasión.
Introducción
Las técnicas de histología convencionales se basan principalmente en secciones delgadas de tejidos de órganos, que pueden proporcionar intrínsecamente sólo información 2D en un entorno 3D complejo. Aunque es factible en principio, la reconstrucción 3D a partir de secciones delgadas en serie requiere tuberías técnicas exigentes tanto para el corte como para posteriores en la alineación de silico de las imágenes adquiridas1. Además, la reconstrucción sin fisuras de los volúmenes z después del corte del microtomo es fundamental, ya que los artefactos mecánicos y computacionales pueden permanecer debido al registro de imágenes subóptimo causado por planos de imagen no superpuestos, variaciones de tinción y destrucción de tejido por, por ejemplo, la hoja del microtome. Por el contrario, el corte óptico puro de muestras de tejido grueso intactas permite la adquisición de planos de imagen superpuestos (sobremuestreo) y, por lo que, facilita la reconstrucción 3D. Esto, a su vez, es altamente beneficioso para el análisis de procesos de infección en poblaciones celulares complejas (por ejemplo, redes neuronales en el contexto de las células gliales e inmunitarias circundantes). Sin embargo, los obstáculos inherentes de las secciones de tejido grueso incluyen dispersión de la luz y penetración limitada de anticuerpos en el tejido. En los últimos años, se ha desarrollado y optimizado una variedad de técnicas para superar estos problemas2,3,4,5,6,7,8 , 9 , 10 , 11 , 12 , 13. Esencialmente, los tejidos diana se vuelven ópticamente transparentes por tratamiento con acuoso2,3,4,5,6,7 ,8,9 o soluciones a base de disolventes orgánicos10,11,12,13. La introducción de 3DISCO (imágenes 3D de órganos con disolventes)11,12 y su sucesor uDISCO (imágenes 3D finales de órganos con disolventes)13 proporcionaron una herramienta relativamente rápida, simple y barata con excelentes capacidades de compensación. Los principales componentes del protocolo de limpieza son los disolventes orgánicos tert-butanol (TBA), alcohol bencílico (BA), benzoato de bencilo (BB) y éter de difenilo (DPE). El desarrollo y adición de iDISCO (imágenes 3D habilitadas para inmunoetiquetado de órganos con disolventes)14, un protocolo de inmunomanchas compatible, constituyó otra ventaja sobre los métodos existentes y permitió el etiquetado de tejido profundo de antígenos de interés, así como el almacenamiento a largo plazo de muestras inmunomanchadas. Por lo tanto, la combinación de iDISCO14 y uDISCO13 permite la toma de imágenes de alta resolución de proteínas etiquetadas con anticuerpos en grandes secciones de tejido (hasta 1 mm) utilizando CLSM convencional.
La preservación de la estructura compleja de un órgano en las tres dimensiones es particularmente importante para el tejido cerebral. Las neuronas comprenden una subpoblación celular muy heterogénea con morfologías 3D muy diversas basadas en sus proyecciones de neurita (revisadas por Masland15). Además, el cerebro consta de una serie de compartimentos y subcompartimentos, cada uno compuesto por diferentes subpoblaciones celulares y proporciones de los mismos, incluyendo células gliales y neuronas (revisado por von Bartheld et al.16). Como virus neurotrópico, el virus de la rabia (RABV, revisado por Fooks et al.17) infecta principalmente a las neuronas, utilizando su maquinaria de transporte para viajar en dirección retrógrada a lo largo de los axons desde el sitio primario de la infección hasta el sistema nervioso central (SNC). El protocolo descrito aquí (Figura1A) permite la detección asistida por inmunomanchas y la visualización de células infectadas por RABV y RABV en pilas de imágenes grandes y coherentes obtenidas del tejido cerebral infectado. Esto permite una evaluación imparcial y en 3D de alta resolución del entorno de la infección. Es aplicable al tejido cerebral de una variedad de especies, se puede realizar inmediatamente después de la fijación o después del almacenamiento a largo plazo de muestras en paraformaldehído (PFA), y permite el almacenamiento y reimagen de muestras manchadas y despejadas durante meses.
Protocolo
Se utilizó material cerebral archivado fijado por la PFA infectado por RABV. Los respectivos estudios experimentales en animales fueron evaluados por el comité responsable de cuidado, uso y ética de los animales de la Oficina Estatal de Agricultura, Seguridad Alimentaria y Pesca en Mecklemburgo-Pomerania Occidental (LALFF M-V) y obtuvieron la aprobación con permisos 7221.3-2.1-002/11 (ratones) y 7221.3-1-068/16 (ferrets). Los cuidados generales y los métodos utilizados en los experimentos con animales se llevaron a cabo de acuerdo con las directrices aprobadas.
ADVERTENCIA: Este protocolo utiliza diversas sustancias tóxicas y/o nocivas, incluyendo PFA, metanol (MeOH), peróxido de hidrógeno (H2O2),azida sódica (NaN3),TBA, BA, BB y DPE. MeOH y TBA son altamente inflamables. Evite la exposición usando el equipo de protección personal adecuado (un abrigo de laboratorio, guantes y protección para los ojos) y realizando experimentos en una campana de humo. Recoger los residuos por separado en los contenedores apropiados y desecharlos de acuerdo con la normativa local. El virus de la rabia se clasifica como un patógeno de nivel de bioseguridad (BSL)-2 y, por lo tanto, generalmente puede ser manejado bajo condiciones BSL-2. Algunas actividades, incluidos los procedimientos que pueden generar aerosoles, trabajar con altas concentraciones de virus o trabajar con nuevos lissavirus, pueden requerir la clasificación BSL-3. Se recomienda la profilaxis previa a la exposición para el personal de alto riesgo, incluidos los cuidadores de animales y los trabajadores de laboratorio18,19. Consulte las regulaciones de las autoridades locales.
1. Fijación del tejido cerebral y seccionamiento
- Fijar muestras cerebrales en un volumen apropiado de 4% PFA en solución salina tamponada de fosfato (PBS [pH 7.4]) durante al menos 48 h a 4 oC (con una relación de tejido-fijador aproximada de 1:10 [v/v]).
- Lavar las muestras de tejido 3 veces en PBS durante al menos 30 minutos cada lavado y almacenarlas en 0.02% NaN3/PBS a 4 oC hasta su uso.
- Seccionar el tejido en secciones de 1 mm de espesor utilizando un vibratomo (velocidad de avance de la hoja: 0,3–0,5 mm/s, amplitud: 1 mm, espesor de la rebanada: 1.000 m).
- Para conservar la secuencia de rebanadas correcta, guarde cada sección de tejido por separado en un pozo de una placa de cultivo de células multipocillos. Añadir 0.02% NaN3/PBS y almacenar las secciones de tejido a 4 oC hasta su uso.
2. Pretratamiento de muestras con metanol
NOTA: Realice todos los pasos de incubación con una oscilación suave y, si no se indica lo contrario, a temperatura ambiente. Proteja las muestras de la luz. El pretratamiento de la muestra sirve para mejorar la difusión de anticuerpos y reducir la autofluorescencia tisular mediante la exposición a MeOH y H2O2, respectivamente14.
- Prepare soluciones MeOH al 20% (v/v), al 40%, al 60% y al 80% en agua destilada. Por ejemplo, para el 20% de MeOH, añadir 10 ml de 100% MeOH a 40 ml de agua destilada en un recipiente sellado relable adecuado y mezclar invirtiéndolo.
- Transfiera las muestras a recipientes de tamaño razonable (por ejemplo, tubos de reacción de 5 ml). Tenga cuidado de utilizar materiales químicamente resistentes a los reactivos utilizados en este protocolo. Por ejemplo, tenga en cuenta que, si bien el polipropileno es adecuado, el poliestireno no lo es.
NOTA: Las especificaciones de volumen de este protocolo se refieren a tubos de reacción de 5 ml. Si se utiliza un recipiente diferente, ajuste los volúmenes en consecuencia. - Incubar las muestras en 4 ml de cada concentración de la serie preparada de soluciones MeOH en orden ascendente durante 1 h cada una.
- Incubar las muestras 2x durante 1 h cada una en puro (100%) Meoh.
- Enfríe las muestras a 4 oC (p. ej., en un refrigerador seguro para laboratorio).
- Preparar la solución de blanqueo (5% H2O2 en MeOH) diluyendo, por ejemplo, una solución de material de 30% H2O2 a 1:6 en puro (100%) MeOH, y enfriarlo a 4 oC.
- Retire el 100% MeOH de las muestras refrigeradas y agregue 4 ml de solución de blanqueo pretalado (5% H2O2 en MeOH). Incubar durante la noche a 4oC.
- Cambiar la solución de blanqueo por 4 ml de 80% MeOH e incubar por 1 h. Continuar utilizando la serie preparada de soluciones MeOH en orden descendente durante 1 h cada una hasta que las muestras hayan sido incubadas durante 1 h en 4 mL de 20% MeOH.
- Lavar las muestras 1x durante 1 h con 4 ml de PBS.
3. Inmunomanchación
NOTA: Realice todos los pasos de incubación con una oscilación suave y, si no se indica lo contrario, a temperatura ambiente. Proteja las muestras de la luz. Para evitar el crecimiento microbiano, añada NaN3 a una concentración final del 0,02% a las soluciones de esta sección. Las muestras de tejido se permeabilizan aún más mediante el tratamiento con detergentes no iónicos Triton X-100 y Tween 20. El suero normal se utiliza para bloquear la unión inespecífica de anticuerpos. Se añaden glicina y heparina para reducir el fondo de inmunoetiquetado14.
- Lavar las muestras 2x durante 1 h cada una en 4 ml de 0,2% De Tritón X-100/PBS.
- Permeabilizar las muestras durante 2 días a 37oC con 4 mL de 0,2% de Tritón X-100/20% DMSO/0.3 M glicina/PBS.
- Bloquear la unión inespecífica de los anticuerpos incubando las muestras durante 2 días a 37 oC en 4 ml de 0,2% de Tritón X-100/10% DMSO/6% suero normal/PBS.
NOTA: Utilice suero normal de la misma especie en la que se elevó el anticuerpo secundario para lograr resultados de bloqueo ideales. - Incubar las muestras en 2 ml de solución primaria de anticuerpos (3% de suero normal/5% DMSO/PTwH [PBS-Tween 20 con heparina] + anticuerpos/anticuerpos primarios) durante 5 días a 37 oC. Actualice la solución de anticuerpos primarios después de 2,5 días.
- Para PTwH, reconstituir la sal sódica de heparina en agua destilada para hacer una solución de material de 10 mg/ml (almacenar esta solución, alícotada, a 4oC). Añadir la solución de stock a 0.2% Tween 20/PBS a una concentración final de 10 g/ml.
NOTA: La elección de la dilución correcta de anticuerpos puede requerir optimización. Generalmente, las concentraciones estándar de inmunohistoquímica son un buen punto de partida.
- Para PTwH, reconstituir la sal sódica de heparina en agua destilada para hacer una solución de material de 10 mg/ml (almacenar esta solución, alícotada, a 4oC). Añadir la solución de stock a 0.2% Tween 20/PBS a una concentración final de 10 g/ml.
- Lavar las muestras durante 1 día en 4 ml de PTwH, intercambiando el tampón de lavado al menos 4veces-5 veces durante el transcurso del día y dejando el lavado final durante la noche.
- Incubar las muestras en 2 ml de solución secundaria de anticuerpos (3% suero/PTwH normal + anticuerpos/anticuerpos secundarios) durante 5 días a 37 oC. Actualice la solución de anticuerpos secundarios después de 2,5 días.
- Diluir el anticuerpo/anticuerpo secundario a 1:500 en solución secundaria de anticuerpos (es decir, 4 l en 2 ml).
- Lave las muestras durante 1 día como se describe en el paso 3.5, dejando el lavado final durante la noche.
4. Tinción nuclear
NOTA: Realice todos los pasos de incubación con una oscilación suave y, si no se indica lo contrario, a temperatura ambiente. Proteja las muestras de la luz. Si no se requiere ninguna tinción nuclear o se requiere la longitud de onda de excitación/espectro de emisión de TO-PRO-3 para la excitación o detección de otro fluoróforo, omita este paso.
- Diluir la mancha de ácido nucleico TO-PRO-3 a 1:1.000 en PTwH e incubar las muestras en 4 ml de solución de tinción nuclear durante 5 h.
- Lave las muestras durante 1 día como se describe en el paso 3.5, dejando el lavado final durante la noche.
NOTA: Después de los lavados, las muestras se pueden almacenar en PBS a 4 oC hasta la limpieza óptica.
5. Limpieza de tejidos
NOTA: Realice todos los pasos de incubación con una oscilación suave y, si no se indica lo contrario, a temperatura ambiente. Proteja las muestras de la luz. Las muestras de tejido se deshidratan en una serie calificada de soluciones TBA. Como la inmunomancha requiere soluciones acuosas, todos los procedimientos de tinción deben terminarse antes de la limpieza del tejido. El aclaramiento óptico y la coincidencia del índice de refracción se logran mediante el tratamiento con una mezcla de BA, BB y DPE. La solución de limpieza se complementa con DL-o-tocoferol como antioxidante13.
- Preparar soluciones TBA al 30% (v/v), 50%, 70%, 80%, 90% y 96% en agua destilada. Por ejemplo, para 30% TBA, añadir 15 ml de 100% TBA a 35 ml de agua destilada en un recipiente sellado relable adecuado y mezclar invirtiendo.
NOTA: TBA tiene un punto de fusión de 25–26 oC; por lo tanto, tiende a ser sólido a temperatura ambiente. Para preparar las soluciones de TBA, caliente la botella bien sellada a 37 oC en una incubadora o baño de agua. - Deshidratar las muestras con 4 ml de cada concentración de la serie preparada de soluciones TBA en orden ascendente para 2 h cada una. Deje el 96% de TBA encendido durante la noche.
- Deshidratar las muestras aún más en puro (100%) TBA durante 2 h.
- Preparar la solución de compensación BABB-D15.
NOTA: BABB-D15 es una combinación de BA y BB (BABB) que se mezcla con DPE en una relación de x:1, donde x se especifica en el nombre de la solución, en este caso 15.- Para BABB, mezcle BA de una parte con dos partes BB.
- Mezclar BABB y DPE en una proporción de 15:1.
- Añadir 0,4 vol% DL-a-tocoferol (vitamina E).
NOTA: Por ejemplo, para 20 mL de BABB-D15, mezclar 6,25 mL de BA con 12,5 mL de BB. Añadir 1,25 ml de DPE y complementarlo con 0,08 ml de DL-o-tocoferol.
- Limpie las muestras en la solución de limpieza hasta que sean ópticamente transparentes (2-6 h).
- Las muestras se pueden almacenar a 4oC en BABB-D15, protegidas de la luz, hasta el montaje y la toma de imágenes.
6. Montaje de muestras
- Utilizando una impresora 3D, imprima la cámara de imágenes y la tapa (material: copoliéster [CPE], boquilla: 0,25 mm, altura de la capa: 0,06 mm, espesor de la pared: 0,88 mm, recuento de paredes: 4, relleno: 100%, sin estructura de soporte; el correspondiente . STL se puede encontrar en los Materiales Suplementarios de este protocolo).
- Montar la cámara de imágenes (Figura2).
- Monte un soporte redondo (diámetro: 30 mm) en la cámara de imágenes con goma de silicona RTV-1 (de un componente de temperatura ambiente-vulcanizadora). Retire el exceso de caucho de silicona con un hisopo de algodón humedecido en agua y cure durante la noche.
- Montar un cubreobjetos redondo (diámetro: 22 mm) en la tapa con goma de silicona RTV-1. Retire el exceso de caucho de silicona con un hisopo de algodón humedecido en agua y cure durante la noche.
- Coloque la muestra en una cámara de imágenes, agregue un pequeño volumen de BABB-D15 e inserte la tapa. Llene la cámara con BABB-D15 a través de la entrada, utilizando una aguja hipodérmica (27 G x 3/4 pulgadas [0,40 mm x 20 mm]).
- Enchufe la entrada y selle la cámara de imágenes con goma de silicona RTV-1. Curar durante la noche en la oscuridad.
7. Procesamiento de imágenes y imágenes
- Configure la adquisición de la imagen seleccionando las líneas láser respectivas para que coincidan con los fluoróforos utilizados. Ajuste los rangos de detección de cada detector para evitar la superposición de señal entre canales.
NOTA: Los rangos de detección ejemplares para Alexa Fluor 488, Alexa Fluor 568 y TO-PRO-3 son de 500 a 550 nm, 590–620 nm y 645–700 nm, respectivamente. - Elija los parámetros de adquisición, defina el borde superior e inferior de la pila z y adquiera la pila de imágenes.
NOTA: Los parámetros de adquisición ejemplares son un escaneo secuencial con un tamaño de píxel de 60-90 nm, un tamaño de paso z de 0,5 m, un promedio de línea de 1, una velocidad de escaneo de 400 Hz y un tamaño de agujero de 1 unidad Airy. - Procesar la pila de imágenes utilizando el software de análisis de imágenes adecuado (por ejemplo, Fiji) para generar proyecciones 3D o realizar análisis en profundidad.
NOTA: Debido al gran tamaño de los archivos de imagen adquiridos, normalmente es necesario el uso de una estación de trabajo.- Abra los archivos de adquisición o de imagen en Fiji (Archivo ? Abiertos ? Seleccione archivos).
- Si utiliza, por ejemplo, . LIF, seleccione o anule la selección de las opciones deseadas en la ventana de diálogo Formatos biológicos. Ver pila con Hyperstack. Aparte de eso, no se necesarian selecciones o ticks específicos. Pulse OK.
- Si el archivo contiene varias pilas de imágenes, seleccione las que desea analizar y confirme pulsando OK.
- Realice la corrección de lejía dividiendo la imagen fusionada en canales individuales (Imagen ? Color ( Color ) Herramienta Canalesy, a continuación, seleccione Más . Dividir canales). Para cada canal, seleccione corrección de lejía (Imagen ? Ajustar ? Corrección de lejía) y elegir Simple Ratio (intensidad de fondo: 0.0).
NOTA: En algunos casos, por ejemplo, cuando no hay decaimiento lineal de la señal o la señal es demasiado débil en general, Simple Ratio puede fallar. Alternativamente, pruebe Ajuste exponencial u omita la corrección de lejía. - Ajuste el brillo y el contraste de cada canal con los controles deslizantes (Imagen ? Ajustar ? Brillo ? Contraste).
- Combinar los canales (Imagen ? Color ( Color ) Fusionar canales), hacer un compuesto (Imagen ? Color ( Color ) Herramienta Canalesy, a continuación, seleccione Más . Crear compuesto) y convertirlo a formato RGB (Imagen ? Color ( Color ) Herramienta Canalesy, a continuación, seleccione Más . Convertir a RGB).
- Si es necesario, cambie el tamaño de la pila de imágenes para reducir el tiempo de cálculo y el tamaño del archivo (Imagen ? Ajustar ? Tamaño, ambas opciones marcadas más interpolación bilineal).
- Generar una proyección 3D (Imagen ? Pilas ? Proyecto 3D). Elija Punto más brillante como método de proyección y establezca el espaciado de sector para que coincida con el tamaño del paso z de la pila de imágenes adquirida. Para obtener la máxima calidad, establezca el incremento del ángulo de rotación en 1 y habilite la interpolación. Modifique la rotación total, los umbrales de transparencia y la opacidad según sea necesario.
- Si es necesario, reajuste el contraste y el brillo convirtiendo la proyección 3D a formato de 8 bits (Imagen ? Tipo de la clase de texto (Type 8 bits). Utilice los controles deslizantes correspondientes (Imagen ? Ajustar ? Brillo ? Contraste) y reconvertir la pila de imágenes al formato RGB como se describe en el paso 7.3.4.
- Guarde la proyección 3D como . TIF (formato de archivo de imagen) y . AVI (formato de archivo de vídeo).
- Abra los archivos de adquisición o de imagen en Fiji (Archivo ? Abiertos ? Seleccione archivos).
Resultados
La combinación de iDISCO14 y uDISCO13 junto con CLSM de alta resolución proporciona una visión profunda de la resolución espaciotemporal y la plasticidad de la infección RABV del tejido cerebral y el contexto celular circundante.
Mediante la inmunoutilización de la fosfoproteína RABV (P), se pueden visualizar capas complejas de células neuronales infectadas en secciones gruesas del cerebro del ratón (Figura3). Posteriormente, se pueden reconstruir las proyecciones 3D sin fisuras de las pilas de imágenes adquiridas (Figura3A,B, panel derecho; Figura animada 1). Se debe tener cuidado al usar anticuerpos primarios y anticuerpos secundarios contra la especie de la que proviene el material del órgano. El uso de anticuerpos IgG anti-ratón en el tejido cerebral del ratón dio lugar a una tinción distinta del sistema vascular (Figura3B,panel izquierdo). Debido a la alta resolución con la que se adquieren las pilas de imágenes, la infección se puede evaluar hasta un nivel de una sola célula (Figura4), lo que permite aserciones sobre, por ejemplo, la abundancia y distribución de antígeno dentro de la célula (Figura4C ).
Aparte del tejido cerebral del ratón, el protocolo también se puede aplicar al tejido cerebral de otras especies animales (por ejemplo, hurones) (Figura 5; Figura animada 2). Las secciones tomadas de diferentes compartimentos de un cerebro hurón infectado revelaron un grado variable de infección RABV (Figura 5A-D).
Como el cerebro comprende muchas subpoblaciones celulares diferentes, la diferenciación entre estas poblaciones es vital. Utilizando anticuerpos dirigidos contra marcadores celulares, es posible evaluar la identidad celular de las células infectadas y vecinas. Por ejemplo, los astrocitos se pueden diferenciar mediante la expresión de proteína ácida fibrilar glial (GFAP) (Figura6A,C; Figura animada 3), mientras que los neurófilos pueden ser teñidos específicamente para la proteína asociada al microtúbulo 2 (MAP2) (Figura6B,D; Figura animada 4). Simultáneamente, las proteínas virales, en este caso, la nucleoproteína RABV (N), se pueden cotejar para evaluar la relación entre las células infectadas y la subpoblación celular destacada.

Figura 1 : Principio básico y flujo de trabajo del protocolo. (A) Representación gráfica del flujo de trabajo basada en los protocolos de Renier et al.14 y Pan et al.13. (B) Dos rebanadas ejemplares de cerebro de hurón, una antes (panel izquierdo) y otra después del tratamiento con disolventes orgánicos (panel derecho). La limpieza hace que el tejido sea ópticamente transparente como observable por el texto legible. La rebanada cerebral despejada está incrustada en una cámara de imágenes impresa en 3D (panel derecho). Haga clic aquí para ver una versión más grande de esta figura.
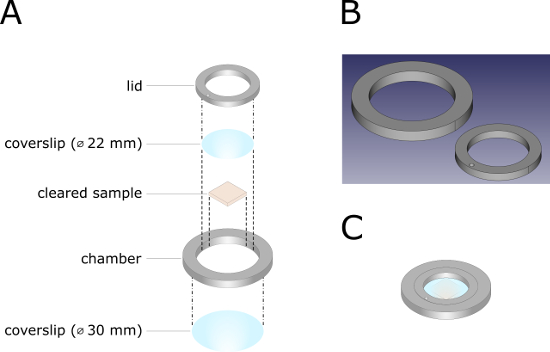
Figura 2 : Ilustración técnica de la cámara de imágenes impresa en 3D. (A) Dibujo de vista explosionada de la cámara de imágenes. Las líneas con puntas resaltan los componentes que deben montarse entre sí con caucho de silicona RTV-1. Las líneas discontinuas representan instrucciones para el montaje posterior de la cámara. (B) CAD (diseño asistido por ordenador) archivo de la cámara de imágenes. El archivo . STL para imprimir la cámara de imágenes se puede encontrar en Materiales Suplementarios. (C) Cámara de imágenes totalmente montada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Imágenes de tejido profundo del tejido cerebral del ratón infectado por RABV. (A y B) Los ratones se infectaron con un virus de la calle vulpina recombinante. La tinción RABV P (verde) revela capas neuronales infectadas con grandes proyecciones de neurita enredadas dentro del tejido cerebral despejado. Los núcleos fueron contramanchados con TO-PRO-3 (azul). (B) El uso de anticuerpos secundarios IgG antiratón etiquetados con fluoróforo (rojo) en el tejido del ratón da lugar a un etiquetado distinto del sistema vascular. La reconstrucción 3D de las pilas de imágenes adquiridas permite la observación desde diferentes ángulos de visión (A y B,paneles derecho). Barras de escala de 60 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : La adquisición de imágenes de alta resolución permite evaluaciones complejas hasta un nivel de una sola celda. El cerebro fue diseccionado de un ratón infectado por SAD L16. (A) La tinción RABV P (verde) resalta una neurona infectada individualmente. (B y C) Las imágenes y proyecciones detalladas demuestran que se pueden realizar análisis en profundidad de la abundancia, distribución y localización de antígenos. Los núcleos fueron contramanchados con TO-PRO-3 (azul). Barras de escala a20 m (A ), 5 m (C ). Haga clic aquí para ver una versión más grande de esta figura.
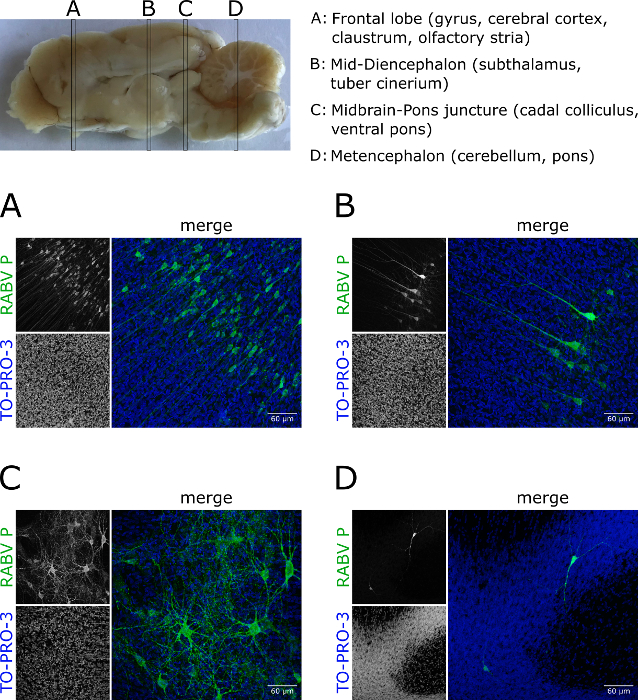
Figura 5 : Imágenes de tejido profundo del tejido cerebral de hurón infectado por RABV. Los hurones estaban infectados con la calle canina RABV. (A–D) Se tomaron cortes de áreas específicas del cerebro, inmunomanchados para RABV P (verde) y borrados ópticamente. Las proyecciones demuestran que las células infectadas en diferentes partes del cerebro difieren en cantidad y morfología. Además, destacan la aplicabilidad del protocolo a tejidos distintos de los derivados del ratón. Los núcleos fueron contramanchados con TO-PRO-3 (azul). Barras de escala de 60 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 6 : La inmunofluorescencia multicolor permite la costamiento de marcadores celulares. Se utilizaron y coinmunosparan las rebanadas de tejido cerebral de hurones infectados con la calle canina RABV para RABV N (rojo) y (A y C)GFAP (verde) o (B y D) MAP2 (verde). Mientras que GFAP es un marcador de astrocitos, MAP2 resalta específicamente los neuritas. Los núcleos fueron contramanchados con TO-PRO-3 (azul). (C y D) En la parte inferior, se representan las extracciones de un solo sector de las vistas de detalle enumeradas de las proyecciones combinadas. Barras de escala a 15 m (A y B) 10 m (C y D). Haga clic aquí para ver una versión más grande de esta figura.

Figura animada 1: Reconstrucciones 3D y proyecciones de detalle de pilas de imágenes de cerebros de ratón infectados por RABV. Las proyecciones se generaron a partir de las pilas de imágenes descritas en la Figura3. (A y C) Animaciones de todas las pilas z respectivas. (B) Una proyección detallada de una reconstrucción 3D de una parte de la pila z de las áreas resaltadas al principio del vídeo. (D) Una proyección detallada de un tomograma de una parte de la pila z de las áreas resaltadas al principio del vídeo. Verde - RABV P; rojo - ratón IgG; azules- núcleos. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)

Figura animada 2: Reconstrucciones 3D de pilas de imágenes adquiridas de un cerebro de hurón infectado por RABV. Las proyecciones se generaron a partir de las pilas de imágenes descritas en la Figura5. (A–D) Las anotaciones se refieren a la misma figura y describen las áreas respectivas del cerebro de las que se tomaron las rebanadas. Verde - RABV P; azules- núcleos. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)

Figura animada 3: Proyección gradual de los diferentes canales de una reconstrucción 3D de un cerebro de hurón infectado por RABV cocine con el marcador de astrocitoGF. Las proyecciones se generaron a partir de la pila de imágenes descrita en la Figura 6A. La adición gradual de canales comienza con RABV N (rojo), después de lo cual sigue los núcleos celulares (azul) y, finalmente, GFAP (verde), mientras que la resta primero elimina RABV N, luego los núcleos celulares, y finalmente GFAP. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)

Figura animada 4: Proyección gradual de los diferentes canales de una reconstrucción 3D de un cerebro de hurón infectado por RABV cocine con el marcador neuronal MAP2. Las proyecciones se generaron a partir de la pila de imágenes descrita en la Figura 6B. La adición gradual de canales comienza con RABV N (rojo), después de lo cual sigue los núcleos celulares (azul) y, finalmente, MAP2 (verde), mientras que la resta primero elimina RABV N, luego los núcleos celulares, y finalmente MAP2. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)
Discusión
El resurgimiento y desarrollo de técnicas de limpieza de tejidos en los últimos años2,3,4,5,6,7,8, 9 , 10 , 11 , 12 , 13 , 14 han abierto muchas nuevas posibilidades para obtener pilas de imágenes de gran volumen de tejido orgánico. Esto proporcionó una herramienta sin igual y potente para estudiar, entre muchos otros temas, la infección por virus. La posterior reconstrucción 3D de estas pilas de imágenes permite aserciones sofisticadas en, por ejemplo, el tropismo de virus, la abundancia y el curso de tiempo de la infección. Este protocolo describe la visualización asistida por inmunoetiquetado de la infección RABV en tejido cerebral con disolvente.
Hay varios pasos críticos en la preparación y adquisición de las pilas de imágenes de tejido inmunoetiquetado y con disolvente. La exposición prolongada a PFA puede enmascarar epítopos y, por lo tanto, dar lugar a una disminución de la antigenicidad20,21,22. Por lo tanto, es importante limitar los tiempos de fijación al mínimo necesario y transferir las muestras a una solución adecuada (por ejemplo, PBS complementado con 0.02% NaN3 para almacenamiento a largo plazo). Sin embargo, todas las imágenes y proyecciones representativas proporcionadas aquí se han adquirido a partir de muestras cerebrales archivadas, algunas de las cuales se habían almacenado en PFA durante semanas, destacando la aplicabilidad de la técnica al material orgánico que ha estado expuesto a la PFA para períodos prolongados de tiempo. Se han demostrado resultados similares para muestras de cerebro humano13. Otro paso crítico, si no el más, es la incubación de anticuerpos. Las concentraciones de anticuerpos deben elegirse cuidadosamente. Mientras que en la mayoría de los casos, las concentraciones estándar de IHC son un buen punto de partida para el etiquetado de anticuerpos de tejido profundo, algunos antígenos pueden requerir una optimización adicional de la concentración de anticuerpos. Este protocolo demuestra que tanto RABV N como P son fácilmente detectables por los anticuerpos utilizados. Mientras que el pretratamiento de MeOH generalmente mejora el inmunoetiquetado, algunos antígenos son incompatibles con este tratamiento. Para ellos, Renier y sus colegas14 proporcionaron un pretratamiento alternativo de muestras sin MeOH. El análisis de las pilas de imágenes adquiridas requiere una potencia computacional adecuada. Debido a los tamaños de archivo grandes de hasta varios gigabytes por pila, se necesitan equipos potentes para procesar las imágenes. El postprocesamiento a menudo incluye la resta del ruido de fondo y una corrección de lejía para compensar los efectos de blanqueo asociados a la adquisición. Al medir las distancias, hay que tener en cuenta que, como efecto secundario, el tejido se está encogiendo durante el proceso de limpieza.
En comparación con otras plataformas de microscopía, como la microscopía de fluorescencia de láminas ligeras (LSFM) o la microscopía de escaneo láser de dos fotones (2PLSM), CLSM tiene la distancia de trabajo más limitada. Por lo tanto, es necesario precortar los órganos a 1 mm de espesor para la toma de imágenes. Además, CLSM tiene la velocidad de adquisición más lenta debido a su alta resolución de imágenes. El uso de un microscopio de hoja de luz permitiría obtener imágenes más rápidas de volúmenes más grandes hasta imágenes de todo el cerebro, al mismo tiempo que sacrificaría la resolución de la imagen. Otra limitación es el aumento inherente de la autofluorescencia tisular con longitud de onda decreciente. Esto hace que el uso de fluoróforos y diotos excitados por la línea láser a 405 nm, incluyendo los tendederos Hoechst, poco prácticos a imposibles.
Con respecto a otras técnicas de limpieza de tejidos, el híbrido de iDISCO14 y uDISCO13 combina los atributos más positivos: es altamente compatible con inmunostaining, altamente versátil, comparativamente rápido y barato, tiene excelentes capacidades de limpieza, y es factible con un microscopio confocal estándar. Además, este protocolo no se limita a la inmunoutilización y limpieza del tejido cerebral, sino que también se puede aplicar a una variedad de otros tejidos blandos y patógenos, tanto neuroinvasivos como no neuroinvasivos. En conclusión, el protocolo descrito aquí representa una canalización para la toma de imágenes 3D de alta resolución del tejido cerebral infectado por RABV. Las reconstrucciones 3D del tejido cerebral infectado se pueden utilizar para responder a una variedad de preguntas sobre la progresión de la enfermedad rabv, la patogénesis y la neuroinvasión.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen a Thomas C. Mettenleiter y Verena te Kamp por leer críticamente el manuscrito. Este trabajo fue apoyado por la Iniciativa Federal de Excelencia de Mecklemburgo Pomerania Occidental y el Fondo Social Europeo (FSE) Grant KoInfekt (ESF/14-BM-A55-0002/16) y una beca de investigación colaborativa intramuros sobre Lyssavirus Friedrich-Loeffler-Institute (Ri-0372).
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Benzyl alcohol | Alfa Aesar | 41218 | Clearing reagent |
| Benzyl benzoate | Sigma-Aldrich | BB6630-500ML | Clearing reagent |
| Dimethyl sulfoxide | Carl Roth | 4720.2 | Various buffers |
| Diphenyl ether | Sigma-Aldrich | 240834-100G | Clearing reagent |
| DL-α-Tocopherol | Alfa Aesar | A17039 | Antioxidant |
| Donkey serum | Bio-Rad | C06SBZ | Blocking reagent |
| Glycine | Carl Roth | 3908.2 | Background reduction |
| Goat serum | Merck | S26-100ML | Blocking reagent |
| Heparin sodium salt | Carl Roth | 7692.1 | Background reduction |
| Hydrogen peroxide solution (30 %) | Carl Roth | 8070.2 | Sample bleaching |
| Methanol | Carl Roth | 4627.4 | Sample pretreatment |
| Paraformaldehyde | Carl Roth | 0335.3 | Crystalline powder to make fixative solution |
| Sodium azide | Carl Roth | K305.1 | Prevention of microbial growth in stock solutions |
| tert-Butanol | Alfa Aesar | 33278 | Sample dehydration for tissue clearing |
| TO-PRO-3 | Thermo Fisher | T3605 | Nucleic acid stain |
| Triton X-100 | Carl Roth | 3051.2 | Detergent |
| Tween 20 | AppliChem | A4974,0500 | Detergent |
| Miscellaneous | |||
| 5 mL reaction tubes | Eppendorf | 0030119401 | Sample tubes |
| Coverslip, circular (diameter: 22 mm) | Marienfeld | 0111620 | Part of imaging chamber |
| Coverslip, circular (diameter: 30 mm) | Marienfeld | 0111700 | Part of imaging chamber |
| Hypodermic needle (27 G x ¾” [0.40 mm x 20 mm]) | B. Braun | 4657705 | Filling of the imaging chamber with clearing solution |
| RTV-1 silicone rubber | Wacker | Elastosil E43 | Adhesive for the assembly of the imaging chamber |
| Ultimaker CPE 2.85 mm transparent | Ultimaker | 8718836374869 | Copolyester filament for 3D printer to print parts of the imaging chamber |
| Technical equipment and software | |||
| 3D printer | Ultimaker | Ultimaker 2+ | Printing of imaging chamber |
| Automated water immersion system | Leica | 15640019 | Software-controlled water pump |
| Benchtop orbital shaker | Elmi | DOS-20M | Sample incubation at room temperature (~ 150 rpm) |
| Benchtop orbital shaker, heated | New Brunswick Scientific | G24 Environmental Shaker | Sample incubation at 37 °C (~ 150 rpm) |
| Confocal laser scanning microscope | Leica | DMI 6000 TCS SP5 | Inverted confocal microscope for sample imaging |
| Fiji | NIH (ImageJ) | open source software (v1.52h) | Image processing package based on ImageJ |
| Long working distance water immersion objective | Leica | 15506360 | HC PL APO 40x/1.10 W motCORR CS2 |
| Vibratome | Leica | VT1200S | Sample slicing |
| Workstation | Dell | Precision 7920 | CPU: Intel Xeon Gold 5118 GPU: Nvidia Quadro P5000 RAM: 128 GB 2666 MHz DDR4 SSD: 2 TB |
| Primary antibodies | |||
| Goat anti-RABV N | Friedrich-Loeffler-Institut | Monospecific polyclonal goat anti-RABV N serum, generated by goat immunization with baculovirus-expressed and His-tag-purified RABV nucleoprotein N Dilution: 1:400 | |
| Rabbit anti-GFAP | Dako | Z0334 | Polyclonal antibody (RRID:AB_10013382) Dilution: 1:100 |
| Rabbit anti-MAP2 | Abcam | ab32454 | Polyclonal antibody (RRID:AB_776174) Dilution: 1:250 |
| Rabbit anti-RABV P 160-5 | Friedrich-Loeffler-Institut | Monospecific polyclonal rabbit anti-RABV P serum, generated by rabbit immunization with baculovirus-expressed and His-tag-purified RABV phosphoprotein P (see reference 23: Orbanz et al., 2010) Dilution: 1:1,000 | |
| Secondary antibodies | |||
| Donkey anti-goat IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
Referencias
- Pichat, J., Iglesias, J. E., Yousry, T., Ourselin, S., Modat, M. A Survey of Methods for 3D Histology Reconstruction. Medical Image Analysis. 46, 73-105 (2018).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Ke, M. T., Fujimoto, S., Imai, T. SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction. Nature Neuroscience. 16 (8), 1154-1161 (2013).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Susaki, E. A., et al. Whole-brain imaging with single-cell resolution using chemical cocktails and computational analysis. Cell. 157 (3), 726-739 (2014).
- Susaki, E. A., et al. Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging. Nature Protocols. 10 (11), 1709-1727 (2015).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Treweek, J. B., et al. Whole-body tissue stabilization and selective extractions via tissue-hydrogel hybrids for high-resolution intact circuit mapping and phenotyping. Nature Protocols. 10 (11), 1860-1896 (2015).
- Dodt, H. U., et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods. 4 (4), 331-336 (2007).
- Erturk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-171 (2011).
- Erturk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Pan, C., et al. Shrinkage-mediated imaging of entire organs and organisms using uDISCO. Nature Methods. 13 (10), 859-867 (2016).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2014).
- Masland, R. H. Neuronal cell types. Current Biology. 14 (13), 497-500 (2004).
- von Bartheld, C. S., Bahney, J., Herculano-Houzel, S. The search for true numbers of neurons and glial cells in the human brain: A review of 150 years of cell counting. The Journal of Comparative Neurology. 524 (18), 3865-3895 (2016).
- Fooks, A. R., et al. Rabies. Nature Reviews Disease Primers. 3, 17091 (2017).
- WHO. WHO Expert Consultation on Rabies, Third Report. WHO Technical Report Series. , (2018).
- CDC. . Biosafety in Microbiological and Biomedical Laboratories, 5th Edition. US Department of Health and Human Services. , (2009).
- Arnold, M. M., et al. Effects of fixation and tissue processing on immunohistochemical demonstration of specific antigens. Biotechnic & Histochemistry. 71 (5), 224-230 (1996).
- Webster, J. D., Miller, M. A., Dusold, D., Ramos-Vara, J. Effects of prolonged formalin fixation on diagnostic immunohistochemistry in domestic animals. Journal of Histochemistry and Cytochemistry. 57 (8), 753-761 (2009).
- Werner, M., Chott, A., Fabiano, A., Battifora, H. Effect of formalin tissue fixation and processing on immunohistochemistry. The American Journal of Surgical Pathology. 24 (7), 1016-1019 (2000).
- Orbanz, J., Finke, S. Generation of recombinant European bat lyssavirus type 1 and inter-genotypic compatibility of lyssavirus genotype 1 and 5 antigenome promoters. Archives of Virology. 155 (10), 1631-1641 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados