Method Article
Phthalic Acid Ester-liaison ADN aptamère sélection, caractérisation et Application d’un Aptasensor électrochimique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un protocole de sélection de in vitro et la caractérisation de l’acide phtalique spécifique au groupe ester-liaison ADN aptamères est présenté. L’application de l’aptamère sélectionné dans un aptasensor électrochimique est également incluse.
Résumé
Phthalic acid esters (PAEs) areone des grands groupes de polluants organiques persistants. La détection de groupes spécifiques de PAEs est hautement souhaitée en raison de la croissance rapide des congénères. Aptamères ADN ont été appliquées de plus en plus en tant qu’éléments de reconnaissance sur les plates-formes de biocapteur, mais en sélectionnant aptamères pour atteindre les cibles hautement hydrophobes de petites molécules, telles que PAEs, sont rarement rapporté. Cet ouvrage décrit une méthode axée sur le talon conçue pour sélectionner les aptamères ADN spécifiques à un groupe de PAEs. Le groupement aminé fonctionnalisé phtalate de dibutyle (DBP-NH2) comme la cible de l’ancre a été synthétisée et immobilisée sur les perles de l’agarose époxy-activé, permettant d’afficher le groupe ester phtalique à la surface de la matrice de l’immobilisation, et par conséquent, le choix des liants spécifiques à un groupe. Nous avons déterminé les constantes de dissociation des candidats aptamère par réaction en chaîne de polymérisation quantitative couplée avec séparation magnétique. Les affinités relatives et la sélectivité des aptamères à autres PAEs ont été déterminées par les essais concurrentiels, où les candidats aptamère ont été préalablement délimitées à la DBP-NH2 attaché billes magnétiques et rejetés dans le surnageant après incubation avec la PAEs testée ou autres substances interférentes potentiels. L’analyse concurrentielle a été appliquée parce qu’il a fourni une comparaison facile affinité entre PAEs qui n’avait aucun groupes fonctionnels pour l’immobilisation de la surface. Enfin, nous avons démontré la fabrication d’un aptasensor électrochimique et utilisé pour détection ultrasensible et sélective de phtalate de 2-éthylhexyle. Ce protocole prévoit des idées pour la découverte d’aptamère d’autres petites molécules hydrophobes.
Introduction
Ainsi que le développement économique rapide, accélération de l’industrialisation et la construction urbaine, pollution de l’environnement est plus sévère que jamais. Les polluants environnementaux typiques comprennent les ions de métaux lourds, toxines, antibiotiques, pesticides, perturbateurs endocriniens et les polluants organiques persistants (POP). En plus des ions métalliques et des toxines, autres polluants sont de petites molécules qui sont souvent constituées d’une variété de congénères. Par exemple, la pop plus toxiques comprennent des hydrocarbures aromatiques polycycliques (HAP), les éthers biphényles polybromés (PBDE), des polychlorodibenzo-p-dioxines (PCDD), les biphényles polychlorés (BPC), dibenzofurannes polychlorés (PCDF) et phtalique esters de l’acide (PAEs)1,2, qui toutes se composent de plusieurs congénères. Détection de petites molécules a été principalement réalisée par chromatography/mass spectrometry sur des techniques en raison de la diversité des applications3,4,5,6. Pour la détection sur place, les méthodes axées sur les anticorps ont été récemment développés7,8,9. Toutefois, étant donné que ces méthodes sont hautement spécifiques pour certain un congénère, plusieurs essais sont effectués. Ce qui est plus grave, c’est que les nouveaux congénères grandissent si vite que leurs anticorps ne peuvent pas être générés dans le temps. Par conséquent, le développement de biocapteurs spécifique pour surveiller les niveaux totaux de tous les congénères lors d’un test peut fournir une métrique de précieuse pour évaluer l’état de la pollution de l’environnement.
Récemment, les aptamères nucléotidiques ont été largement appliquées comme éléments de reconnaissance dans les diverses plateformes de biodétection en raison de leur capacité à reconnaître une grande variété de cibles, d’ions et de petites molécules de protéines et cellules10,11 ,,12. Aptamères sont identifiés au moyen d’une méthode in vitro appelée évolution systématique des ligands par enrichissement exponentiel (SELEX)13,14. SELEX commence avec la bibliothèque d’oligonucléotides monocaténaires synthétique aléatoire, qui contient environ 10 séquences de15 14-10. La taille de la bibliothèque aléatoire assure la diversité des structures candidat ARN ou ADN. Le processus typique de SELEX se compose de plusieurs tours d’enrichissement jusqu'à ce que la bibliothèque est enrichie en séquences avec grande affinité et spécificité à la cible. La poule finale enrichie est ensuite séquencée, et les constantes de dissociation (Kd) et la sélectivité contre potentiel substances interférentes sont déterminées par différentes techniques telles que filtrent binding, chromatographie d’affinité, surface résonance plasmon (SPR), etc. 15
En raison de la solubilité dans l’eau est extrêmement faible et manque de groupes fonctionnels pour l’immobilisation de la surface, l’aptamère pop est théoriquement difficile. Des avancées significatives pour SELEX ont accéléré la découverte d’aptamères. Toutefois, la sélection des aptamères spécifiques à un groupe de pop n’a pas encore été signalée. Jusqu'à présent, seulement PCB-liaison ADN aptamères avec grande spécificité pour certain un congénère ont été identifiés16. PAEs sont principalement utilisés dans les matériaux de polychlorure de vinyle, changeant de polychlorure de vinyle plastique dur à un élastique plastique, agissant ainsi comme un plastifiant. Certains PAEs ont été identifiés comme des perturbateurs endocriniens, peut causer des dommages graves au foie et de la fonction rénale, réduire la motilité des spermatozoïdes mâles et peuvent entraîner la morphologie des spermatozoïdes anormaux et cancer du testicule,17. Le composé - ni aptamères PAE-liaison spécifique à un groupe ont été signalés.
L’objectif de ce travail est de fournir un protocole représentatif de sélection spécifique au groupe ADN aptamères à fortement hydrophobes petites molécules telles que PAEs, un groupe représentatif des polluants organiques persistants. Nous démontrons également l’application de l’aptamère sélectionné pour la détection de la pollution de l’environnement. Ce protocole fournit des conseils et des idées pour la découverte d’aptamère d’autres petites molécules hydrophobes.
Protocole
1. Bibliothèque et de conception d’amorce et de synthèse
-
Concevoir la bibliothèque initiale et les amorces.
Bibliothèque (piscine0) : 5'-TCCCACGCATTCTCCACATC-N40-CCTTTCTGTCCTTCCGTCAC-3'
Avant l’amorce (FP) : 5'-TCCCACGCATTCTCCACATC-3'
Phosphorylée apprêt inverse (PO4- RP) : 5'-PO4- GTGACGGAAGGACAGAAAGG-3' - Synthétiser la piscine0, FP et PO4- RP en utilisant le standard phosphoramidite chimie18,19,20et purifier tous les ADN par standard chromatographie liquide haute performance (CLHP)21.
- Reconstituer la piscine0, FP et PO4- RP en catégorie exempte de nucléase à 100 µM, aliquote, de l’eau et les stocker à -20 ou -80 ° C pendant un an.
Remarque : Il est fortement conseillé de stocker piscine0, FP et PO4- PR dans 10-20 µL d’extraits pour éviter la contamination croisée possible.
2. synthétiser la cible de l’ancre et son immobilisation sur époxy-activé d’Agarose perles
- Synthétiser le groupement aminé fonctionnalisé de phtalate de dibutyle (DBP-NH2) comme la cible de l’ancre.
Remarque : Les détails expérimentaux sur la synthèse et la caractérisation de la DBP-NH2 ont été décrits dans la littérature22. - Préparer l’ancre cible solutions dans un milieu approprié pour la solubilité de la cible. Pour cette étude, préparer 100 mM DBP-NH2 solution dans le diméthylsulfoxyde (DMSO) et conserver la solution à 4 ou -20 ° C pendant un an.
- Immobiliser le DBP-NH2 sur époxy-activé d’agarose perles selon un protocole modifié le fabricant.
- Peser à 0,1 g lyophilisé en poudre d’époxy-activé d’agarose perles et le suspendre dans l’eau distillée. Laver le milieu pendant 1 h à l’aide de 20 mL d’eau distillée ajoutée dans plusieurs parties aliquotes. Laver le milieu avec 0,2 M Na2CO3 (pH 12).
NOTE : Le support gonfle immédiatement après l’ajout de l’eau dedans. - DBP-NH2 (46,7 mg, 0,15 mmol), dissoudre dans les 500 µL de tampon de couplage (0,2 M mémoire tampon Na2CO3 ).
- Mélanger la solution de couplage contenant le ligand avec le milieu. Utiliser un agitateur à 5 tr/min pendant 48 h à température ambiante.
- Répéter le lavage du produit trois fois, dans l’ordre en utilisant un tampon acétate 0,1 M (0,5 M de NaCl, pH 4,5) et 0,2 M carbonate tampon (0,5 M de NaCl, pH 12) à chaque cycle de lavage. Laver et suspendre le produit dans l’eau pour faire le volume final de 500 µL.
- Stocker le produit à 4 ° C avant de l’utiliser.
- Confirmer le succès de l’immobilisation de la cible par analyse élémentaire.
- Peser à 0,1 g lyophilisé en poudre d’époxy-activé d’agarose perles et le suspendre dans l’eau distillée. Laver le milieu pendant 1 h à l’aide de 20 mL d’eau distillée ajoutée dans plusieurs parties aliquotes. Laver le milieu avec 0,2 M Na2CO3 (pH 12).
3. SELEX
- Préparer 500 mL de tampon de liaison PAE en eau ultra pure ou de l’eau exempte de nucléase grade avec 20 mM Tris· HCl, 100 mM NaCl, 2 mM MgCl2, 5 mM KCl, 1 mM CaCl2, 1 % polyoxyethy-lene monolaurate de sorbaitan (20) et 0,03 % agent de surface non ioniques (pH de 7,9). La mémoire tampon du filtre à travers un filtre de nitrocellulose de 0,22 µm stérile et conserver à une température supérieure à 20 ° C pour le mois.
Remarque : Il est essentiel de maintenir une bonne dispersion de PAEs dans le tampon de liaison PAE. Plusieurs types d’agents tensio-actifs sont nécessaires pour améliorer la solubilité du PAEs en solution aqueuse. Toutefois, le nombre d’agents tensio-actifs doit être au minimum pour éviter la formation de micelles de surfactant qui incorporerait des molécules PAE. Non ionique, agent de surface actif est facile à précipiter à des températures inférieures à 20 ° C, et donc la mémoire tampon doit être conservé à une température supérieure à 20 ° C. - Diluer 10 µL de 100 µM piscine0(nmol ~1.0 pour 10 séquences de15 14-10) pour 490 µL de tampon de liaison PAE. Faites chauffer les aliquotes de0 piscine (5 x 100 µL) dans les tubes à centrifuger de mince-mur dans le jeu de bain d’eau à 95 ° C pendant 10 min. Place les tubes sur la glace pendant 5 min et ensuite Incuber les tubes pendant 5 min à température ambiante (environ 25 ° C dans ce travail).
- Le premier tour de sélection : incubation de piscine0 et DBP−NH2−coated moyen.
- Laver 200 µL de milieu de −coated2DBP−NH 3 fois avec 500 µL de tampon de liaison PAE. Mix DBP−NH2−coated medium avec piscine0 et incuber le mélange à température ambiante pendant 1 h, sous légère secousse.
- Séparé le milieu de −coated de2DBP−NH lié avec aptamères de l’ADN non lié par ultrafiltration usingan ultrafiltration tube avec une coupure moléculaire de 100 kDa. Laver le milieu trois fois avec 500 µL de tampon de liaison PAE et centrifuger à 9 168 × g pendant 10 min à 4 ° C.
- Le premier tour de sélection : aptamère élution de DBP−NH2−coated moyen.
- Ajouter 50 µL de tampon de liaison PAE dans la moyennes lavées et chauffer le mélange à 90 ° C pendant 9 min en secouant.
- Récupérer le surnageant contenant les ADN éluées par ultrafiltration. Répétez ce processus d’élution trois fois pour récupérer plus liés DNAs.
- Le premier tour de sélection : PCR
-
Petite échelle PCR
- Effectuer une petite échelle PCR23 (4 × 20 µL d’extraits) à l’aide de ~ 15 à 30 cycles. Mettre en place une réaction de PCR24 comme suit : 6,5 µL d’eau exempte de RNase, 1 µL d’apprêt de 10 µM de chaque (FP, PO4- RP, 0,01 nmol de chaque), 1,5 µL éluée pool à partir de la section 3.4 (ou eau exempte de RNase comme contrôle négatif) et 10 µL de prémélangés contenant de mélange de réaction dNTPs, hot start polymérase et 2 × tampon de réaction PCR (20 mM tris· HCl, 100 mM KCl, 3 mM MgCl2, pH 8,3).
- Exécuter la PCR à l’aide d’un thermocycleur avec les paramètres suivants : 1 cycle de 95 ° C, 1 min ; 30 cycles de [95 ° C, 30 s ; 56 ° C, 30 s ; 72 ° C, 30 s] ; 1 cycle de 72 ° C, 2 min et maintenez à 4 ° C. Lorsque l’instrument est en cours d’exécution le 20ème s les étapes d’extension à la 15, 20, 25 et 30 cycles, appuyez sur la touche pause, ouvrir le couvercle de l’instrument et prendre un tube de sortie, puis appuyez sur la touche pause pour reprendre la PCR.
- Préparer 12 % dénaturé sur gel de polyacrylamide (PAGE) l’électrophorèse25,26. Mélanger 4.8 g d’urée, 6 mL d’eau ultra pure, 1 mL d’acide borique/tris de 10 × / acide éthylène diamine tétraacétique (TBE), 3 mL de 40 % d’acrylamide, 10 µL de tétraméthyléthylènediamine (TEMED) et 100 µL 10 % de persulfate d’ammonium (APS), dans cet ordre. Bien remuer et attendre pendant 1,5 à 2 h pour s’assurer que le gel se solidifie complètement.
- Analyser les résultats de l’optimisation du cycle : mélanger 1 µL de chaque produit PCR avec 5 µL de tampon de chargement RNA (62,5 % formamide, formaldéhyde 0,4 M, 1.25×3-(N-morpholino) propansulfonic tampon acide, 0,02 % de xylène cyanol FF, bleu de bromophénol 0,02 %) et 4 µL de Eau exempte de RNase ; chauffer le mélange à 95 ° C pendant 10 min et refroidir rapidement à 0 ° C sur la glace ; incuber 5 min à température ambiante ; charge dans 5 puits séparés de la 12 % dénaturé PAGE ; Exécutez l’électrophorèse par un système d’électrophorèse verticale en tampon TBE 1 × 170 V pendant 45 min.
- Après électrophorèse, tache le gel avec 3 µL de 1 × acide nucléique gel détachant dans 30 mL de tampon TBE × 1 pendant 10 min et prendre une image du gel.
-
Grande échelle PCR : sélectionnez le nombre de cycles pour produire la bande d’intensité plus élevée dans le marqueur de taille correcte sans sous-produits indésirables. Effectuer une grande échelle PCR (20 × 100 µL d’extraits) utilisant les conditions et le nombre de cycles déterminé au point 3.5.1.
Remarque : Le montant final de bibliothèque simple brin qui peut être obtenu est proportionnel au volume total de la PCR, pas la concentration du modèle. En règle générale, pour obtenir 1 nmol de bibliothèque, la piscine simple brin a besoin de 2 mL PCR. PCR est très sensible et facilement contaminés par l’ADN à l’extérieur. Mesures devraient être prises pour réduire les risques de contamination, comme des gants, utilisez des pointes stérilisées, ne pas cracher dans les tubes et ne pas ouvrir le bouchon du tube PCR avant la centrifugation.
-
Petite échelle PCR
- Le premier tour de sélection : purification du produit PCR par précipitation à l’éthanol.
- Combiner deux tubes de mélange PCR dans un tube (200 µL). Répétez cette opération pour tous les tubes.
- Ajouter 15 µL de solution d’acétate (3M, pH 5,2) de sodium, 4 µL de solution d’ADN/ARN précipitations transporteur et 2,5 fois le volume d’éthanol refroidi à-20 ° C dans chaque tube. Mélanger uniformément.
Remarque : Transporteur de précipitation de l’ADN/ARN est largement utilisé dans les expériences de précipitation d’éthanol pour améliorer significativement le pourcentage de récupération de l’ADN/ARN à de faibles concentrations. Il n’interfère pas avec les applications en aval telles que la PCR et séquençage. - Centrifuger le mélange à 20 627 × g pendant 20 min à 4 ° C et soigneusement éliminer le surnageant et laisser le précipité blanc.
- Ajouter 400 µL de solution d’éthanol à-20 ° C de préréfrigération dans chaque tube pour laver la précipitation de 70 %. Centrifuger le mélange à 20 627 × g pendant 5 min à 4 ° C et soigneusement éliminer le surnageant et laisser le précipité blanc. Répétez l’étape de lavage deux fois plus.
- Ouvrez le couvercle du tube ; piquer la membrane d’étanchéité ; Laissez l’éthanol se volatiliser à 40 ° C sur le bloc de chauffage jusqu'à ce que le précipité blanc devient transparent. Stocker le précipité à-20 ° C.
Remarque : Le produit PCR purifié peut être stocké à-20 ° C.
- Le premier tour de sélection : génération d’ADN simple brin (génération de poule enrichi1) en effectuant la réaction d’exonucléase λ pour digérer le brin arrière phosphorylé.
-
Réaction de petite échelle λ exonucléase
- Ajouter 100 µL d’eau exempte de RNase dans un tube contenant le produit PCR purifié du point 3.6. Vortex le tube pour dissoudre complètement le précipité dans le tube.
- Préparez cinq tubes à centrifuger et ajouter 5 µL de la solution ci-dessus, 11 µL de RNase - eau gratuite et 2 µL de tampon de réaction 10 × (670 mM glycine-KOH, 25 mM MgCl2 , 0,1 % (v/v) pH de Triton X-100 9,4) dans chaque tube.
- Ajouter 2 µL d’eau ou de solution d’exonucléase λ dilués contenant 2 U, U, 8 U, 5et 10 exonucléase λ U dans ces tubes, respectivement. Mélanger la solution bien par pipetage doucement. Le tampon de réaction × 10 est fourni avec λ exonucleasefrom du fournisseur.
- Incuber les tubes pendant 35 min à 37 ° C, 15 min à 80 ° C et de tenir à 4 ° C.
- Préparer le natif de 12 % PAGE. Mélanger 6 mL d’eau ultra pure, 1 mL de 10 × TBE, 3 mL de 100 µL de 10 %, 40 % d’acrylamide et 10 µL de TEMED APS, dans cet ordre. Bien remuer et attendre pendant 60 à 90 min pour s’assurer que le gel se solidifie complètement.
- Analyser les résultats des réactions en combinant 5 µL de chaque réaction avec 1 µL de chargement colorant et 4 µL d’eau exempte de RNase et le chargement de ces mélanges dans 5 puits séparés de 12 % indigène PAGE (150 V pendant 45 min).
Remarque : Une échelle d’ADN double-brin bp 20 peut également être chargée sur le gel, mais il ne peut pas être nécessaire étant donné que les bandes distinctes du produit de PCR et la bibliothèque de brin unique générée sur le gel.
Remarque : La réaction d’exonucléase λ a une excellente sélectivité structurale. Le produit PCR double-brin (le brin anti-sens est marqué avec un groupement phosphate à l’extrémité 5') peut être complètement digéré dans une bibliothèque simple brin en présence de l’exonucléase λ dans une gamme assez large de concentration (Figure 4). Le montant minimal de l’exonucléase λ qui peut générer complètement ADN simple brin dans la réaction d’exonucléase λ à petite échelle est également utilisé pour la réaction d’exonucléase λ à grande échelle. Une concentration plus élevée de l’exonucléase λ peut également être utilisée, car l’exonucléase λ fonctionne bien dans une gamme large de concentration sans perdre son rendement de sélectivité et produit de structure. La réaction d’exonucléase λ est inhibée par une forte concentration de sel. La digestion incomplète du produit PCR implique généralement l’existence de trop de sel dans le produit PCR. La purification des produits PCR par précipitation à l’éthanol (section 3.6) est généralement compatible avec cette étape, alors que le produit PCR purifié par précipitation d’isopropanol assez souvent contient trop de sel.
- Réaction de grande échelle λ exonucléase : Choisissez le montant minimal de l’exonucléase λ qui peut générer entièrement l’ADN simple brin pour la réaction d’exonucléase λ à grande échelle. La réaction est entartrée proportionnellement selon cette condition. Par exemple : si 2 U est le montant minimum de l’exonucléase λ nécessaire, mélangent 38 µL d’eau exempte de Rnase, 10 µL de mémoire tampon 10 x 50 µL d’ADN poule1 et 2 µL de 10 exonucléase λ U/µL.
-
Réaction de petite échelle λ exonucléase
- Le premier tour de sélection : purification de la simple brin poule1 par précipitation à l’éthanol.
- Suivez les instructions de l’étape 3.6.
- Déterminer la concentration de purifiée poule1 par absorption UV à 260 nm.
- Reconstituer le 90 % purifiée de la poule1 dans un volume suffisant de tampon de liaison PAE. S’il n’y a pas assez ADN obtenu pour la prochaine ronde de sélection, répétez les sections 3.5-3.8.2.
Remarque : Le volume de tampon de liaison PAE varie généralement de 200 à 500 µL selon le montant de la bibliothèque et la concentration finale souhaitée de bibliothèque dans la prochaine série de SELEX.
- La deuxième, troisième, quatrième et cinquième cycles de SELEX.
- Procéder à la deuxième, troisième, quatrième et cinquième cycles de SELEX par la suite suivant la même procédure décrite ci-dessus (section 3.2-3.8), sauf que la bibliothèque de ~ 300 pmol était entrée afin d’accroître la rigueur de la sélection.
- Afin de minimiser l’absorption non spécifique d’ADN sur le support, incuber la piscine avec un milieu non modifié sur un agitateur rotatif pendant 30 min dans le troisième et quatrième tour de sélection avant l’incubation avec le milieu de −coated de2DBP−NH.
Remarque : Le SELEX processus s’est terminé à la cinquième manche en raison de la grave réticulation entre ADN révélées par la grande quantité de produit de haut poids moléculaire qui ne migre dans le gel.
4. séquençage haut débit
- Préparer la poule4 de l’article 3.9.2 séquençage par amplification par PCR avec des amorces compatibles.
- Concevoir et synthétiser un apprêt avant indexé avec 6 nt à l’extrémité 5′ pour faciliter l’analyse de la séquence.
Indexés avant apprêt (FP-séquençage) : 5'-agtacgaTCCCACGCATTCTCCACATC-3' - Mettre en place une réaction de 1 000 µLPCR comme suit : 380 µL d’eau exempte de RNase, 50 µL de chaque amorce de 10 µM (FP-séquençage, PO4- RP, 0,5 nmol de chaque), 20 µL de piscine éluée de section 3.9.2 et 500 µL de prémélangée mélange réactionnel contenant des dNTPs, polymérase de démarrage à chaud et 2 × tampon de réaction PCR. Aliquoter le mélange dans 10 tubes PCR. Utilisez les mêmes conditions PCR comme décrit au paragraphe 3.5.1.2.
- Concevoir et synthétiser un apprêt avant indexé avec 6 nt à l’extrémité 5′ pour faciliter l’analyse de la séquence.
- Purifier le produit PCR pour répondre aux exigences de l’installation de séquençage. Par exemple, utiliser le kit de récupération PAGE-gel ADN selon les instructions du fabricant.
- Envoyer le produit PCR purifié (~ 2 µg) à une installation de séquençage dans les conditions d’expédition requis par l’installation de séquençage. Par exemple, expédition du produit PCR dans un tube à centrifuger avec bouchons soigneusement scellés avec packs de glace à la facilité de séquençage.
NOTE : A reçu les résultats qui s’est classé les séquences selon le nombre de copies de chaque séquence pour évaluer l’enrichissement de la piscine séquencée. La séquence supérieure a été nommée DBP-1 (5'-CTTTCTGTCCTTCCGTCACATCCCACGCATTCTCCACAT-3'). Son affinité et une sélectivité ont été mesurées par le protocole suivant (article 5 et 6). Son application dans une bio-détection électrochimique a été démontrée par la suite à l’article 7.
5. Kd détermination de sélectionné aptamère candidats à l’aide de Magnetic Bead-Based PCR Quantitative (qPCR)
- Synthétiser les amorces standard phosphoramidite chimie et DBP-1-qPCR et de le purifier par HPLC standard.
Aptamère candidat (DBP-1-qPCR) : 5'-ATACCAGCTTATTCAATTCTTTCTGTCCTTCCGTCACATC CCACGCATTCTCCACATAGATAGTAAGTGCAATCT-3'
Apprêt pour qPCR (FP-qPCR) en avant : 5'-ATACCAGCTTATTCAATT-3'
Inverser l’apprêt pour qPCR (RP-qPCR) : 5'-AGATTGCACTTACTATCT-3'
NOTE : Les régions de l’amorce de la séquence testée diffèrent de celles de piscine0 utilisés à l’article 3. Le but de changer les régions de l’apprêt est de tester l’affinité de la séquence de base seule, à l’exclusion des régions des amorces. - Immobiliser le DBP-NH2 sur billes magnétiques recouvertes d’acide carboxylique, suivant les instructions du fabricant.
- Laver les billes magnétiques acide carboxylique-couché deux fois avec 25 mM 2-(N-morpholino) ethanesulfonic acide (MES) (pH 5,0), en utilisant un volume égal de billes magnétiques (100 µL) éjectant hors de la cuvette, pendant 10 min avec le bon mélange (plus fin ou similaire).
- Immédiatement avant l’emploi, préparer 50 mg/mL de solution de chlorhydrate (EDC) de carbodiimide 1-ethyl-3-(3-dimethylaminopropyl) avec froid 25 mM MES (pH 5.0).
- Immédiatement avant l’emploi, préparer 50 mg/mL de solution de N-hydroxysuccinimide (NHS) avec froid 25 mM MES (pH 5.0).
- Mélanger 100 µL de solution d’EDC et 100 µL de solution de NHS avec les perles lavées et incuber 30 min à température ambiante avec une rotation lente inclinaison.
- Mettre le tube sur un aimant pour les 4 min après incubation, éliminer le surnageant et laver perles deux fois avec 100 µL de froid 25 mM MES (pH 5.0).
- Incuber 6 µL de la solution mère de 100 mM DBP-NH2 et 25 mM MES (pH 5.0, 290 µL) avec les perles activés sous rotation pendant au moins 30 min à température ambiante ou 2 h à 4 ° C, avec une rotation lente inclinaison à 5 t/mn.
- Mettre le tuyau sur l’aimant pendant 4 min après incubation et éliminer le surnageant. Laver les billes revêtues 4 fois et remettre en suspension les perles avec 200 µL de tampon de liaison PAE et conserver à 4 ° C jusqu'à l’utilisation.
-
Recueillir la courbe de titrage pour déterminer Kd.
- Préparer une série de solutions DBP-1 (500 µL) à des concentrations variées dans le tampon de liaison PAE de 13:00 à 300 nM.
- Ajouter 10 µL de DBP-NH2-enduit de billes magnétiques comme décrit dans section 5.2.7 à chaque solution DBP-1. Incuber pendant 1 heure à température ambiante sous rotation.
- Placer les tubes sur un aimant pour les 4 min et retirez le surnageant. Laver les perles 4 fois avec 200 µL de tampon de liaison PAE.
- Ajouter 60 µL de tampon de liaison PAE dans chaque tube. Incuber les tubes à 95 ° C pendant 15 min. à frais virés le surnageant contenant le DBP-1 éluées par séparation magnétique. Répétez ce processus d’élution pour récupérer plus lié DBP-1.
- Diluer la solution d’élution 100 fois et prendre 3 µL de qPCR en temps réel. Mettre en place une réaction de qPCR comme suit : 3 µL d’eau exempte de RNase, 2 µL d’apprêt de 10 µM de chaque (FP, PO4- RP, 0,01 nmol de chaque), 3 µL d’éluée et dilué DBP-1 et 10 µL de prémélangée mélange réactionnel contenant des dNTPs, polymérase et 2 tampon de réaction x PCR.
- Déterminer le nombre de DBP-1 dans chaque échantillon en exécutant qPCR utilisant un thermocycleur avec les paramètres suivants : 1 cycle de 95 ° C, 30 s ; 30 cycles de [95 ° C, 30 s ; 45 ° C, 30 s ; 72 ° C, 30 s] ; 1 cycle de 72 ° C, 30 s et maintenez à 4 ° C.
- Tracer la courbe de titrage et déterminer Kd par un montage non linéaire de la courbe en supposant un ratio de 1:1 liaison27.
6. relative affinité et spécificité Test par des analyses concurrentielles
- Ajouter 10 µL de DBP-NH2-enduit de billes magnétiques à DBP-1 solution (500 µL, 1 µM). Incuber pendant 1 heure à température ambiante sous rotation. Placer les tubes sur un aimant pour les 4 min et retirez le surnageant. Laver les perles 4 fois avec 200 µL de tampon de liaison PAE et il resuspendre dans 10 µL de tampon de liaison PAE.
- Ajouter 10 µL de DBP-NH2-enduit limite moyenne avec DBP-1 à 110 µL de chaque 10 µM testé échantillon (DBP-NH2, DBP, DEHP, butyl benzyl phthalate(BBP), ions de métaux lourds, etc..) Incuber à température ambiante pendant 1 h. recueillir le surnageant par séparation magnétique.
- Diluer le liquide surnageant 100 fois et prendre 3 µL de qPCR quantification. Mettre en place une réaction de qPCR comme suit : 3 µL d’eau exempte de RNase, 2 µL d’apprêt de 10 µM de chaque (FP, PO4- RP, 0,01 nmol de chaque), 3 µL d’éluée et dilué DBP-1 et 10 µL de prémélangée mélange réactionnel contenant des dNTPs, polymérase et 2 tampon de réaction x PCR.
- Déterminer le nombre de DBP-1 dans chaque échantillon en exécutant qPCR utilisant un thermocycleur avec les paramètres suivants : 1 cycle de 95 ° C, 30 s ; 30 cycles de [95 ° C, 30 s ; 45 ° C, 30 s ; 72 ° C, 30 s] ; 1 cycle de 72 ° C, 30 s et maintenez à 4 ° C.
- Calculer l’affinité relative en divisant le nombre de DBP-1 détache les perles en présence de l’échantillon par le nombre de sortie de perles dans le tampon de liaison PAE DBP-1 : affinité Relative =N/nmémoire tampon.
7. fabrication et mesures électrochimiques de DEHP biocapteurs électrochimiques
- Synthétiser la séquence centrale THIOLÉS (HS-DBP-1) et la sonde de signalisation (DBP-1-C-Fc) à l’aide de chimie phosphoramidite standard et de le purifier par HPLC standard.
HS-DBP-1 : 5' - HS-(CH2)6- CTTTCTGTCCTTCCGTCACATCCCACGCATTCTCCACAT -3 '
DBP-1-C-Fc : 5 '- GATGTGACGGAAGGTTTTTTTT-(CH2)6Fc -3'
Remarque : La conception rationnelle des sondes capteur a été décrit dans notre précédent pbulications18,22. - Reconstituer les HS-DBP-1 et DBP-1-C-Fc dans l’eau exempte de nucléase grade à 100 µM, aliquote et les conserver à -20 ou -80 ° C pendant un an.
- Polir l’électrode d’or avec soin à une surface réfléchissante avec 1, 0,3 et 0,05 µm Al2O3 en poudre sur une microcloth pendant 5 min et il soniquer dans de l’eau ultrapure pendant 5 min enlever les résidus Al2O3. Puis nettoyer l’électrode par polissage électrochimique avec 35 scans successifs voltampérométrie cyclique (CV) de 0,4 à 1,2 V (vs Hg-Hg2SO4) 0,5 M H2SO4 à 100 mV/s.
- Préparer un mélange de 0,5 µM HS-DBP-1 et 0,5 µM DBP-1-C-Fc dans 100 µL de tampon phosphate de 10 mM contenant 1 M NaCl, pH 7,4 (PBS). Chauffer le mélange dans le tube à centrifuger de mince-mur dans le jeu de bain d’eau à 95 ° C pendant 10 min et lentement refroidir le mélange à température ambiante.
- Ajouter 1 µL de chlorhydrate de-(2-carboxyethyl)-phosphine tris (TCEP, 10 mM, solution mère) dans le mélange. Conserver à température ambiante pendant 1 h.
- Plonger l’électrode d’or propre dans la solution ci-dessus pendant 12 h, ou toute la nuit à température ambiante.
- Rincer l’électrode à PBS et immerger l’électrode dans 1 mM [S (CH2)2(OCH2CH2)6OCH3]2 (HS-EG6- OMe) dans du PBS pendant 1 h. Rincer l’électrode soigneusement à l’aide de PAE liant le tampon et immerger l’électrode dans le tampon de liaison PAE.
- Prendre une analyse voltampérométrie (SWV) onde carrée en arrière-plan dans le tampon exempte de cible de liaison de PAE.
- Nettoyer tout le matériel expérimental : cellules électrolytiques, électrode de travail or, platine contre-électrode et électrode de référence au calomel saturée (ECS).
- Activer les logiciels installés ; Définissez les paramètres expérimentaux pour les mesures de SWV.
Remarque : Les paramètres expérimentaux suivants ont été utilisés pour les expériences SWV : aire de répartition potentielle : de -0,2 à 0,7 V ; modulation d’amplitude : 25 mV ; largeur d’impulsion : 5 ms ; taux de balayage : 50 mV/s ; potentiel de l’étape : 1 mV. - Connectez l’or travail électrode, électrode de platine compteur et SCE à un potentiostat et mettez ces trois électrodes dans une cuve d’électrolyse contenant le tampon de liaison PAE.
- Exécuter la mesure SWV.
Remarque : Une fois le SWV mesure commence, une courbe SWV sera affichée sur l’écran de l’ordinateur. L’analyse se répète jusqu'à ce que les scans sont constants et aucuns autres changements dans la forme ou la hauteur des pics ne sont observées.
- Plongez l’électrode de travail dans le tampon de liaison PAE contenant une certaine concentration de DEHP (p. ex. 22:00) pendant 30 min à température ambiante. Rincez l’électrode soigneusement à l’aide de tampon de liaison PAE et immerger l’électrode dans le tampon de liaison PAE avec la contre-électrode et CPE. Recueillir la courbe SWV dans les mêmes conditions, tel que décrit à l’article 7.8.
- Répétez l’étape 7.9, sauf que la concentration du DEHP (p. ex. 100 pM, 1 nM, 10 nM, 100 nM, 1 µM) a été augmentée de façon séquentielle. Tracer la courbe de titrage.
- Tests de spécificité : Répétez les étapes 7.3-7,9, sauf que le tampon de liaison PAE contenant du DEHP est remplacé par le tampon de liaison PAE contenant la substance interférente potentielle à la concentration désirée.
Résultats
Nous avons conçu et synthétisé le groupement aminé fonctionnalisé de phtalate de dibutyle (DBP-NH2) comme cible d’ancrage (Figure 1F). Ensuite, nous avons effectué la sélection d’aptamère ADN du PAEs à l’aide de DBP-NH2 comme la cible de l’ancre et en suivant la méthode axée sur l’immobilisation de cible classique (Figure 2). À chaque tour, un pilote PCR a été réalisée à l’aide de la PAGE dénaturée afin d’optimiser le nombre de cycles de PCR (Figure 3). La PAGE dénaturée, au lieu de la PAGE native, est fortement conseillée car les autres collègues et nous avons trouvé que les sous-produits suspects de haut poids moléculaire montrés sur les gels natifs dans le plus grand nombre de cycle sont effectivement des complexes de réticulation formées par séquences de la bonne taille. Ainsi, l’intensité de la bande de la PAGE dénaturée peut vraiment refléter le montant du bon produit. La génération de l’ADN brin unique est une autre étape critique, et plusieurs méthodes pour cette étape ont été déclarés28. La méthode de digestion exonucléase λ est la méthode moins chère. Le succès de la génération de l’ADN simple brin peut être commodément vérifié par PAGE native, où le produit PCR est présenté comme simple (premières plusieurs séries de SELEX) ou plusieurs bandes, alors que la seule bande apparaît après la digestion (Figure 4).
La SELEX a été arrêtée après le cinquième tour de la sélection en raison de la quantité importante d’ADN accumulé en haut de la PAGE, même après le traitement de la dénaturation (dans lequel l’ADN est chauffés à 95 ° C pendant 10 min dans 2 × TBE – contenant 8,3 M d’urée) , qui a suggéré la réticulation graves entre DNAs et a également indiqué que les séquences ont été enrichies. Par conséquent, la poule4 a été envoyé pour le séquençage à haut débit. Le résultat de haut-débit a montré que les 100 meilleurs survenant plus fréquemment des séquences ont été hautement conservée et étaient d’une même famille. La séquence supérieure DBP-1 affiche nanomolaires affinité (Figure 5) et bonne groupe-spécificité congénères PAE (DBP, BBP, DEHP) (Figure 6) via les dosages de qPCR commode, comme décrit dans la section protocole.
DBP-1 a été utilisé pour construire un brin basée sur le déplacement des aptasenor électrochimique en conséquence pour nos rapportées antérieurement travail29. Le capteur DEHP peut répondre avec sensibilité et sélectivement au DEHP (Figure 7). Les polluants environnementaux communs tels que les ions de métaux lourds, antibiotiques et petites molécules ayant des groupements fonctionnels similaires ont montré une réponse très faible à DBP-1.

Figure 1 : Structures chimiques de (A) PAEs, (B) BBP, DBP (C), (D) DEHP, e 4-OH-DEHP, (F) DBP−NH2. Ce chiffre a été réimprimé avec la permission de Han Y. et al. 22 S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : ADN spécifique au groupe aptamère procédure de sélection pour PAEs comme une cible d’ancrage à l’aide de DBP-NH2 . Ce chiffre a été réimprimé avec la permission de Han Y. et al. 22 S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : représentant résulte de l’optimisation de cycle PCR à l’aide de la PAGE dénaturée. De gauche à droite, représentent les voies : 20 bp ADN échelle standard, 15 cycles (aucun modèle d’ADN), 20 cycles (aucun modèle d’ADN), 25 cycles (aucun modèle d’ADN), 30 cycles (aucun modèle d’ADN), 15 cycles, cycles de 20, 25 cycles et 30 cycles. Conditions optimales ont été observées à 25 cycles, où le groupe produit unique était à haute intensité sans sous-produits indésirables, en outre, aucune bande n’a été observée chez le témoin correspondant. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : l’optimisation de la réaction exonucléase λ pour digérer le brin phosphorylé en utilisant la PAGE native. De gauche à droite, représentent les voies : 20 bp ADN échelle standard, négatifs (aucun exonucléase λ), 2U, 5U, 8U et 10U. Le montant minimal de l’enzyme a été observé à 2U, où le produit PCR double devient l’ADN simple brin. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
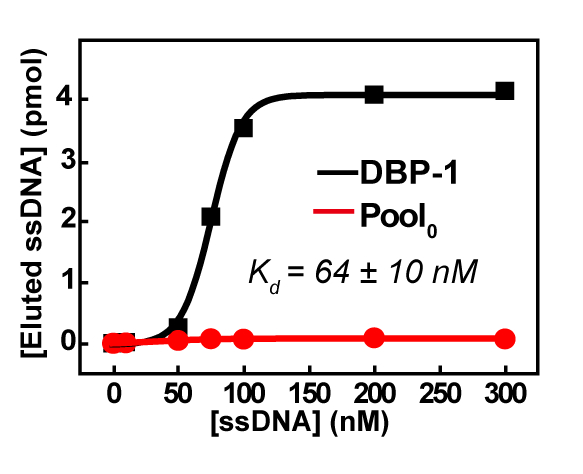
Figure 5 : mesures d’affinité du DBP-1 par analyses qPCR. L’erreur (écart-type, SD) de la Kd a été calculée à partir des trois mesures d’un même échantillon. Ce chiffre a été réimprimé avec la permission de Han Y. et al. 22

Figure 6 : mesures d’affinité Relative du DBP-1 pour libre DBP-NH2 (10 µM), DBP (10 µM), DEHP (10 µM), BBP (10 µM), l’acétate d’éthyle (10 µM), l’acide benzoïque (10 µM), l’acide phtalique (10 µM) et autres via des analyses de compétition. Autres : un mélange des interférences potentielles (glucose, kanamycine, ampicilline et éthanol) à 10 µM. Le rapport (l’affinité relative) a été calculé en divisant le nombre d’aptamères détache les perles en présence de l’échantillon par le nombre d’aptamères libérés des perles dans le tampon de liaison PAE. Les barres représentent la moyenne ± SD Le SDs ont été calculées à partir de trois mesures individuelles. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 7 : biocapteurs électrochimiques DEHP pour détection ultrasensible et ultraselective du DEHP : mécanisme (A), SWV courbe b, courbe d’étalonnage (C), et de sélectivité teste d. Les erreurs (SDs) ont été calculés de trois mesures individuelles. Ce chiffre a été réimprimé avec la permission de Han Y. et al. 22 S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Un des avantages exceptionnels d’aptamères sont qu’ils sont identifiés par le biais de la méthode in vitro SELEX, tandis que les anticorps sont générés via in vivo immunoreactions. Par conséquent, aptamères sont sélectionnables avec spécificité de cible souhaitée dans des conditions expérimentales bien conçues, alors que les anticorps se limitent aux conditions physiologiques.
Pour faciliter la séparation des séquences liées des séquences gratuits, SELEX modifié plusieurs ont récemment été rapportés, l’électrophorèse capillaire30, microfluidique31, perles magnétiques / acryliques / gel d’agarose perles14, etc., ont remplacé les filtres de nitrocellulose ou de colonnes d’affinité pour réaliser une séparation plus efficace. Parmi ces techniques, les méthodes axées sur les perles ont été plus largement utilisées pour la sélection aptamère de petites cibles moléculaires en raison de leur simple mise en place, facilité d’utilisation et son ouverture à des analyses de petites molécules.
Il existe deux groupes de méthodes qui sont couramment utilisés pour la sélection aptamère de petites cibles moléculaires : les cible13 et bibliothèque14 immobilisation méthodes. Dans le premier groupe de méthodes, les objectifs de petites molécules sont immobilisés sur la phase solide comme des billes magnétiques ou agarose perles par des réactions de couplage covalent. Les bibliothèques sont incubés avec perles enrobés de petites molécules et ces séquences ne pas lier ou lier faiblement à la cible sur la phase solide sont supprimés simplement en effectuant les étapes de séparation magnétique et la centrifugation ou la laver. Les séquences liés sont ensuite élués et amplifiés par PCR. Les cibles de petite molécule doivent avoir au moins un groupe fonctionnel disponible pour la réaction de couplage. Pour les autres groupes fonctionnels appropriés, le groupe fonctionnel doit être intégré à l’objectif initial par l’intermédiaire de synthèse organique. La synthèse de la cible étudiée pourrait comporter plusieurs étapes et est parfois assez difficile aussi bien. La modification structurale des cibles pourrait affecter fortement les accepteurs et affinité avec leurs aptamères. Pour éviter ces problèmes, bibliothèque axée sur l’immobilisation des méthodes ont été développées, où la bibliothèque est hybridée complémentaires à capture de l’ADN des sondes magnétiques, et les séquences de liaison tombent les billes lors de la liaison avec les objectifs. Dans ce groupe de méthodes, les cibles sont ajoutées dans le tampon de liaison, et aucun des groupes fonctionnels ne sont nécessaires. PAEs sont les dérivés d’esters de phtalate acide et un PAE typique se compose d’un groupe d’ester de l’acide de phtalate et une ou deux chaînes alkyles (Figure 1 a-D). PAEs n’ont aucun groupes fonctionnels disponibles pour l’immobilisation de la phase solide. Ainsi, les méthodes d’immobilisation base bibliothèque semblent plus attrayants pour la sélection aptamère de PAEs.
Un état de dispersion bien contrôlée est essentiel pour assurer l’enrichissement désiré de la bibliothèque par l’intermédiaire de SELEX. Cependant, PAEs sont extrêmement hydrophobes et facilement forment des agrégats en solution aqueuse à une concentration supérieure à plusieurs µM, même en présence de plusieurs agents tensio-actifs pour faciliter la dispersion. Ainsi, l’état de dispersion de PAEs est difficile à maîtriser, et il serait difficile de choisir les aptamères qui se lient spécifiquement à des molécules individuelles de PAE. Pour résoudre le problème de la solubilité, méthodes d’immobilisation-cible ont été choisis, dans laquelle PAEs ont été immobilisés sur les perles hydrophiles par liaison covalente. Grâce à cette stratégie d’immobilisation, les cibles devraient être principalement présents sur la surface de la perle dans un seul État moléculaire, au lieu d’agrégats.
La conception structurelle de la cible de l’ancre est également très critique pour la sélection réussie des aptamères propres à chaque groupe. Trois facteurs doivent être considérés pour la conception structurale. Le premier facteur est le but de la sélection aptamère. Compte tenu de l’objectif de sélection spécifique au groupe aptamères, il est essentiel d’exposer le groupe commun de PAEs pour la liaison aptamère et empêchent le reste des parties de participer à la liaison aptamère. Ainsi, le groupe fonctionnel doit être introduit sur le terminal de la chaîne latérale. Au début, nous conçu fonctionnalisés OH DEHP (4-OH−DEHP) (Figure 1E) comme la cible de l’ancre et il immobilisés sur l’acide carboxylique billes magnétiques16. Ainsi, les chaînes alkyles ont été exposés à la surface de la matrice. Nous avons essayé de choisir des billes magnétiques avec des groupes carboxyle comme la matrice solide. Aucune amélioration évidente affinité a été observée après cinq tours de sélection, et l’absorption non spécifique sur la matrice nue a été forte.
Par conséquent, DBP-NH2 a été plus tard conçu a basé sur les pensées suivantes : (1) le groupe de phtalate est plus susceptible d’interagir spécifiquement avec l’aptamère grâce à la pile de π-π et la liaison hydrogène de la chaîne alkyle ; (2) NHS-mediated carbodiimide réaction est douce et a une efficacité de couplage très efficace, donc -NH2 est introduit à la fin d’une chaîne alkyle de DBP ; (3) il est facile à utiliser avec les billes magnétiques comme une partition de la phase solide, mais l’adsorption non spécifique de la bibliothèque est forte. Bien que la perte des billes d’agarose pendant la séparation est plus élevée que les billes magnétiques, l’adsorption non spécifique de la bibliothèque est beaucoup plus faible. Ainsi, DBP-NH2 a été immobilisé sur les perles de l’agarose époxy-activé, et le groupe acidester phtalique a été exposé à la surface de la matrice.
Le choix de la mémoire tampon de sélection est important, surtout pour les petites cibles moléculaires dont la solubilité dans diverse. Le tampon de liaison doit être soigneusement préparé pour éviter l’agrégation et assurer un état de bonne dispersion des polluants organiques persistants au cours du processus de sélection et caractérisation d’aptamère ensemble. Dans notre étude, nous avons constaté que DEHP ne peut se dissoudre dans des mémoires tampons standard sans tensioactifs, montrant deux couches distinctes. La couche disparaît lors de l’addition de multiples agents tensio-actifs dans la quantité optimisée. Les solutions claires doivent être conservés à des températures supérieures à 20 ° C, pour conserver son statut. Détails sont fournis à l’étape de protocole 3.1.
Génération d’ADN simple brin de produits PCR double brin est une étape cruciale dans le processus SELEX. Différentes méthodes ont actuellement été décrits dans la littérature32,33, y compris les PCR asymétrique, digestion exonucléase λ, séparation magnétique avec perles streptavidine et séparation de taille en dénaturant gel de polyacrylamide-urée.
Différentes méthodes ont leurs propres forces et faiblesses. Actuellement, la méthode la plus couramment utilisée pour la production d’ADN simple brin est séparation magnétique avec la Streptavidine perles. Les remarquables avantages de cette méthode est son fonctionnement simple et de gagner du temps. L’inconvénient est son coût élevé par rapport aux autres méthodes. En revanche, la méthode axée sur la séparation de la taille, en utilisant des amorces inverses avec structure de tige-boucle riche en GC, est une des méthodes moins chers, tandis que le rendement de l’ADN monocaténaire est le plus faible parmi ces méthodes32. Dans ce protocole, nous décrit l’utilisation de la digestion exonucléase λ pour générer l’ADN simple brin, qui est une des méthodes moins chers. Nous avons constaté que le rendement de l’ADN monocaténaire est comparable avec les deux méthodes de séparation magnétique et streptavidine perles. En outre, nous avons constaté que la réaction de l’exonucléase est inhibée par la forte concentration de sel. La digestion incomplète du produit PCR rapporté dans la littérature33 est probablement due à beaucoup trop salée existant dans le produit PCR. En outre, l’exonucléase λ est très actif et peu coûteux (Figure 4).
La caractérisation et validation des aptamères est laborieux, long et incarne un goulot d’étranglement majeur dans l’aptamère découverte pipeline33. La plupart des techniques sont sensibles à la masse les méthodes qui fonctionnent bien pour les partenaires de liaison aptamère plus grands (> 10 000 amu), mais ne sont pas suffisamment sensibles pour mesurer les interactions avec les cibles de faible poids moléculaire (< 1 000 amu)15. La caractérisation et validation des aptamères hydrophobe petites molécules comme PAEs est encore plus difficile. Leur solubilité dans l’eau pauvres se traduit par l’insaturation de la courbe de titrage, qui empêche les Kddétermination en solution ou immobilise aptamères sur surface. Par conséquent, nous avons déterminé Kds des aptamères identifiés par séquençage à haut débit par DBP-NH2 sur les billes magnétiques hydrophiles pour éviter le problème de la solubilité de l’immobilisation. Les affinités relatives et la sélectivité des aptamères à autres PAEs ont été déterminée par les analyses concurrentielles, où les candidats aptamère ont été préalablement délimitées à la DBP-NH2 attaché billes magnétiques puis rejetés dans le surnageant lors de l’incubation avec la PAEs testée ou autres substances potentiellement interférentes. L’analyse concurrentielle a été appliquée parce qu’il a fourni une comparaison facile affinité entre PAEs qui n’avait aucun groupes fonctionnels pour l’immobilisation de la surface. En outre, analyses fluorescents magnétiques perle ne conviennent pas pour l’étude de l’affinité de petite molécule-aptamère interactions34. Cependant, nous avons constaté que les billes magnétiques provoquent parfois extinction de la fluorescence pour des raisons inconnues. Ainsi, les essais de qPCR ont été utilisés pour les mesures d’affinité.
Une astuce technique critique pour les biocapteurs électrochimiques décrites dans la présente étude est la passivation de surface de l' électrode35. En raison de l’hydrophobicité élevée de DEHP, il a une forte tendance à l’absorption non spécifique sur l’électrode d’or, conduisant à l’échec de la détection. Le plus communément utilisé agent de passivation de surface, 6-mercapto-1-hexanol (MCH)36,37, n’est pas suffisant pour empêcher l’absorption non spécifique du DEHP, alors que nous avons trouvé que HS-(CH2)2-[OCH2CH2 ]6- OCH3 a été assez efficace pour activer la détection sensible du DEHP38.
Cette procédure décrit un protocole de sélection des aptamères ADN spécifiques à un groupe de molécules petites hautement hydrophobes et une demande de l’aptamère sélectionné dans un Biocapteur électrochimique. Le protocole facilite la sélection d’autres petites molécules hydrophobes et donne un aperçu sur le développement de capteurs hautement hydrophobes petites molécules ainsi. Le processus de sélection aptamère appartient à la catégorie des méthodes axées sur l’immobilisation de la cible. Les limites de ce type de méthode existent aussi pour ce protocole, par exemple la nécessité pour la synthèse de complexe de cibles d’ancrage et les impacts de la phase solide sur la liaison aptamère. Les avantages attrayants des biocapteurs électrochimiques décrites dans le présent protocole sont leur conception simple et une grande sensibilité. L’inconvénient majeur est leur précision limitée en raison de leur plage dynamique extrêmement large. Par conséquent, les biocapteurs décrites ici sont plus adaptés pour dépistage, plutôt que des mesures quantitatives des cibles.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrente.
Remerciements
Nous sommes reconnaissants pour le soutien financier de la Fondation nationale des sciences naturelles (21675112), projet clé du plan sciences et technologies de la Commission de l’éducation de Pékin (KZ201710028027) et Yanjing jeune savant programme de Capital Normal University.
matériels
| Name | Company | Catalog Number | Comments |
| UV-2550 | Shimadzu,Japan | protocol, section 3.8.2 | |
| DNA Engnine Thermal cycler,PTC0200 | BIO-RAD | section 3.5.1.2 and 3.5.2 | |
| C1000 Touch | BIO-RAD | section 5.3.6 and 6.3 | |
| VMP3 multichannel potentiostat | Bio-Logic Science, Claix, France | section 7.4,7.8 and 7.11 | |
| Epoxy-activated Sepharose 6B | GE Healthcare (Piscataway, NJ, USA) | 10220020 | argarose beads, section 2.3 and 3.3 |
| Dynabeads M-270 carboxylic acid magnetic beads | Invitrogen, USA | 420420 | magnetic beads,section 5.2. and 5.3 |
| Premix Taq Hot Start Version | Takara,Dalian,China | R028A | polymerase, section 3.5.1.1 |
| PARAFILM Sealing Membrane | Bemis, USA | PM-996 | section 3.6.5 |
| Lambda Exonuclease | Invitrogen, USA | EN0561 | section3.7.1.2.The 10 × reaction buffer is provided along with λ exonuclease by the provider. |
| Dr. GenTLE Precipitation Carrier | Takara,Dalian,China | 9094 | section 3.6.2 and 3.8.1 |
| UNIQ-10 PAGE DNA recovery kit | Sangon Biotech (Shanghai) | B511135 | section 4.2 |
| SYBR Gold nucleic acid gel stain | Invitrogen, USA | 1811838 | nucelic acid stain dye, section 3.5.1.5 |
| SYBR Premix Ex Taq II | Takara,Dalian,China | RR820A | polymerase mix contaning polymerase and dNTPs, section 5.3.5 |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Sigma-Aldrich | CAS: 1132-61-2 | section 5.2.1 |
| 1-ethyl-3-(3-dimethylaminopropyl) carbodiimide hydrochloride (EDC) | Invitrogen, USA | CAS: 25952-53-8 | section 5.2.2 |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 6066-82-6 | section 5.2.3 |
| mercaptohexanol (MCH) | Sigma-Aldrich | CAS: 1633-78-9 | section 7.7 |
| Gold electrode | Shanghai Chenhua | CHI101 | section 7.4. - 7.11 |
| tris(2-carboxyethyl) phosphine hydrochloride (TCEP) | Sigma-Aldrich | CAS: 51805-45-9 | section 7.5 |
| O-(2-Mercaptoethyl)-O'-methyl-hexa-(ethylene glycol) | Sigma-Aldrich | CAS: 651042-82-9 | section 7.7 |
| diethylhexyl phthalate (DEHP) | National Institute of Metrology, China | CAS: 117-81-7 | section 7.11 |
| Tween 20 | Sigma-Aldrich | CAS: 9005-64-5 | polyoxyethy-lene(20) sorbaitan monolaurate |
| Triton X-100 | Sigma-Aldrich | CAS: 9002-93-1 | non-ionic surface active agent |
| PBS | Sigma-Aldrich | P5368 | 10 mM phosphate buffer containing 1 M NaCl, pH 7.4 |
Références
- Vorkamp, K., Riget, F. F. A review of new and current-use contaminants in the Arctic environment: Evidence of long-range transport and indications of bioaccumulation. Chemosphere. 111, 379-395 (2014).
- Net, S., Sempere, R., Delmont, A., Paluselli, A., Ouddane, B. Occurrence, fate, behavior and ecotoxicological state of phthalates in different environmental matrices. Environ Sci Technol. 49 (7), 4019-4035 (2015).
- Xie, Q. L., Liu, S. H., Fan, Y. Y., Sun, J. Z., Zhang, X. K. Determination of phthalate esters in edible oils by use of QuEChERS coupled with ionic-liquid-based dispersive liquid-liquid microextraction before high-performance liquid chromatography. Anal BioanalChem. 406 (18), 4563-4569 (2014).
- Ierapetritis, I., Lioupis, A., Lampi, E. Determination of phthalates into vegetable oils by isotopic dilution gas chromatography mass spectrometry. Food Anal Methods. 7 (7), 1451-1457 (2014).
- Sun, J. Z., He, H., Liu, S. H. Determination of phthalic acid esters in Chinese white spirit using dispersive liquid-liquid microextraction coupled with sweeping beta-cyclodextrin-modified micellar electrokinetic chromatography. J Sep Sci. 37 (13), 1679-1686 (2014).
- Yilmaz, P. K., Ertas, A., Kolak, U. Simultaneous determination of seven phthalic acid esters in beverages using ultrasound and vortex-assisted dispersive liquid-liquid microextraction followed by high-performance liquid chromatography. J Sep Sci. 37 (16), 2111-2117 (2014).
- Sun, R., Zhuang, H. An ultrasensitive gold nanoparticles improved real-time immuno-PCR assay for detecting diethyl phthalate in foodstuff samples. Anal Biochem. 480, 49-57 (2015).
- Sun, R. Y., Zhuang, H. S. A sensitive heterogeneous biotin-streptavidin enzyme-linked immunosorbent assay for the determination of di-(2-ethylhexyl)phthalate (DEHP) in beverages using a specific polyclonal antibody. Anal Methods. 6 (24), 9807-9815 (2014).
- Zhou, L., Lei, Y., Zhang, D., Ahmed, S., Chen, S. An ultra-sensitive monoclonal antibody-based enzyme-linked immunosobent assay for dibutyl phthalate in human urinary. Sci Total Environ. 541, 570-578 (2016).
- Shen, J., Li, Y., Gu, H., Xia, F., Zuo, X. Recent development of sandwich assay based on the nanobiotechnologies for proteins, nucleic Acids, small Molecules, and ions. Chem Rev. 114 (15), 7631-7677 (2014).
- Yin, X. -B. Functional nucleic acids for electrochemical and electrochemiluminescent sensing applications. TrAC, Trends Anal Chem. 33, 81-94 (2012).
- Nguyen, V. -T., Kwon, Y. S., Gu, M. B. Aptamer-based environmental biosensors for small molecule contaminants. Curr Opin Biotechnol. 45, 15-23 (2017).
- Groher, F., Suess, B. In vitro selection of antibiotic-binding aptamers. Methods. 106, 42-50 (2016).
- Yang, K. -A., Pei, R., Stojanovic, M. N. In vitro selection and amplification protocols for isolation of aptameric sensors for small molecules. Methods. 106, 58-65 (2016).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal. Chim. Acta. 686 (1-2), 9-18 (2011).
- Mehta, J., et al. Selection and characterization of PCB-binding DNA aptamers. Anal Chem. 84 (3), 1669-1676 (2012).
- Matsumoto, M., Hirata-Koizumi, M., Ema, M. Potential adverse effects of phthalic acid esters on human health: A review of recent studies on reproduction. Regul Toxicol Pharm. 50 (1), 37-49 (2008).
- Goodchild, J. Conjugates of oligonucleotides and modified oligonucleotides: a review of their synthesis and properties. Bioconjug Chem. 1 (3), 165-187 (1990).
- Brown, D. M. A brief history of oligonucleotide synthesis. Protocols for Oligonucleotides and Analogs: Synthesis and Properties. , 1-17 (1993).
- Reese, C. B. Oligo-and poly-nucleotides: 50 years of chemical synthesis. Org Biomol Chem. 3 (21), 3851-3868 (2005).
- Sproat, B., Colonna, F., Mullah, B., et al. An efficient method for the isolation and purification of oligoribonucleotides. Nucleos Nucleot Nucl. 14 (1-2), 255-273 (1995).
- Han, Y., et al. Selection of group-specific phthalic acid esters binding DNA aptamers via rationally designed target immobilization and applications for ultrasensitive and highly selective detection of phthalic acid esters. Anal Chem. 89 (10), 5270-5277 (2017).
- Bartlett, J. M. S., Stirling, D. A short history of the polymerase chain reaction. PCR protocols. , 3-6 (2003).
- Saiki, R. K., Scharf, S., Faloona, F., et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 230, 1350-1354 (1985).
- Albright, L. M., Slatko, B. E. Denaturing polyacrylamide gel electrophoresis. Current Protocols in Nucleic Acid Chemistry. , A. 3B 1-A. 3B 5 (2001).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). JoVE. (32), e1485(2009).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1), 9-18 (2011).
- Sharma, T. K., Bruno, J. G., Dhiman, A. ABCs of DNA aptamer and related assay development. Biotechnol Adv. 35 (2), 275-301 (2017).
- Liu, R., et al. Signaling-probe displacement electrochemical aptamer-based sensor (SD-EAB) for detection of nanomolar kanamycin A. Electrochim Acta. 182, 516-523 (2015).
- Mendonsa, S. D., Bowser, M. T. In vitro evolution of functional DNA using capillary electrophoresis. J Am Chem Soc. 126, 20-21 (2004).
- Lou, X. H., et al. Micromagnetic selection of aptamers in microfluidic channels. Proc Natl Acad Sci USA. 106 (9), 2989-2994 (2009).
- Cho, M., et al. Quantitative selection of DNA aptamers through microfluidic selection and high-throughput sequencing. Proc. Natl. Acad. Sci. U.S.A. 107, 15373-15378 (2010).
- Cho, M., et al. Quantitative selection and parallel characterization of aptamers. Proc Nat Acad Sci USA. 110 (46), 18460-18465 (2013).
- Song, K. -M., et al. Gold nanoparticle-based colorimetric detection of kanamycin using a DNA aptamer. Anal Biochem. 415 (2), 175-181 (2011).
- Yang, Z., Ding, X., Guo, Q., et al. Second generation of signaling-probe displacement electrochemical aptasensor for detection of picomolar ampicillin and sulfadimethoxine. Sens Actuators B. 253 (2017), 1129-1136 (2017).
- Lou, X., Zhao, T., Liu, R., Ma, J., Xiao, Y. Self-assembled DNA monolayer buffered dynamic ranges of mercuric electrochemical sensor. Anal Chem. 85 (15), 7574-7580 (2013).
- Zhao, T., et al. Nanoprobe-enhanced, split aptamer-based electrochemical sandwich assay for ultrasensitive detection of small molecules. Anal Chem. 87 (15), 7712-7719 (2015).
- Lou, X., He, L. A. Surface passivation using oligo(ethylene glycol) in ATRP-assisted DNA detection. Sens Actuators,B. 129 (1), 225-230 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon