Method Article
Phthalsäure Acid Ester-Bindung DNA-Aptamer Auswahl, Charakterisierung und Anwendung eines elektrochemischen Aptasensor
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Ein Protokoll für die in-vitro- Selektion und Charakterisierung von gruppenspezifischen Phthalsäure Acid Ester-Bindung DNA Aptameren wird vorgestellt. Die Anwendung von der ausgewählten Aptamer in einem elektrochemischen Aptasensor ist ebenfalls enthalten.
Zusammenfassung
Phthalsäure Acid Ester (PAEs) einer der wichtigsten Gruppen von persistenten organischen Schadstoffen. Die gruppenspezifische Erkennung von PAEs ist sehr erwünscht, aufgrund der schnellen Anbau von Artgenossen. DNA-Aptameren zunehmend als Erkennungselemente auf Biosensor Plattformen angewendet wurden, aber Auswahl Aptamere auf stark hydrophobe niedermolekularer Ziele, wie z. B. PAEs, wird selten berichtet. Diese Arbeit beschreibt eine Perle basierende Methode entwickelt, um gruppenspezifische DNA Aptameren, PAEs auswählen. Die Aminogruppe funktionalisiert Dibutyl Phthalat (DBP-NH2) wie das Anker-Ziel wurde synthetisiert und an den Epoxy-aktivierte Agarose-Perlen immobilisiert ermöglicht die Anzeige der phthalic Estergruppe an der Oberfläche der Immobilisierung Matrix und Daher ist die Auswahl der gruppenspezifischen Bindemittel. Wir entschlossen die Dissoziation Konstanten der Aptamer Kandidaten durch quantitative Polymerisation Kettenreaktion gepaart mit magnetische Trennung. Die relativen Affinitäten und Selektivität der Aptamere für andere PAEs wurden durch die wettbewerbsfähige Assays bestimmt, wo die Aptamer-Kandidaten der DBP-NH2 Pre begrenzt waren magnetische Beads angebracht und veröffentlicht, um den Überstand nach Inkubation mit die getesteten PAEs oder andere potenziellen störenden Substanzen. Die wettbewerbsfähigere Assay wurde angewendet, weil sie einen einfache Affinität Vergleich zwischen PAEs bereitgestellt, die keine funktionellen Gruppen für Oberfläche Immobilisierung hatte. Schließlich demonstriert die Herstellung eines elektrochemischen Aptasensor und benutzte es für Gromer und selektive Detektion von bis(2-ethylhexyl) Phthalat. Dieses Protokoll bietet Einblicke für die Aptamer-Entdeckung des anderen hydrophoben kleine Moleküle.
Einleitung
Zusammen mit der rasanten wirtschaftlichen Entwicklung ist die Beschleunigung der Industrialisierung und Städtebau, Umweltbelastungen strenger als je zuvor. Typischen Umweltschadstoffen gehören Schwermetall-Ionen, Toxine, Antibiotika, Pestizide, endokrine Disruptoren und persistente organische Schadstoffe (POPs). Neben der Metall-Ionen und Giftstoffe, andere Schadstoffe sind kleine Moleküle, dass ganz oft bestehen aus einer Vielzahl von Artgenossen. Die giftigsten POPs zählen beispielsweise polychlorierte Biphenyle (PCB), polychlorierten Dibenzofuran (PCDFs), polyzyklische aromatische Kohlenwasserstoffe (PAK), polybromierten APEO Ether (PBDE), polychlorierten Dibenzo-p-Dioxin (PCDDs) und phthalic saure Ester (PAEs)1,2, die alle viele Artgenossen aus. Kleines Molekül Erkennung wurde vor allem durch Chromatographie/Mass Spectrometry-basierten Techniken wegen ihrer Vielfalt von Anwendungen3,4,5,6durchgeführt. Für vor-Ort-Erkennungen wurden Antikörper-basierten Methoden kürzlich entwickelten7,8,9. Da diese Methoden sehr spezifisch für eine bestimmte gleichartig sind, müssen mehrere Tests durchgeführt werden. Gravierender ist, dass die neuartige Kongenere wachsen so schnell, dass ihre Antikörper in der Zeit erzeugt werden können. Daher kann die Entwicklung von zielgruppenspezifischen Biosensoren, die insgesamt Level alle Artgenossen in einem Test zu überwachen eine unschätzbare Metrik zur Bewertung von Umweltbelastungen Status bereitstellen.
Vor kurzem wurden Nukleinsäure-Aptamere weithin als Erkennungselemente in verschiedenen Biosensoren Plattformen durch ihre Fähigkeit, eine Vielzahl von Zielen, von Ionen und kleinen Molekülen an Proteine und Zellen10,11 erkennen angewendet ,12. Aptamere werden durch eine in-vitro- Methode namens systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX)13,14identifiziert. SELEX beginnt mit der zufällige synthetische Einzelstrang Oligonukleotid-Bibliothek, die ca. 1014-1015 Sequenzen enthält. Die Größe der zufälligen Bibliothek sorgt für die Vielfalt der RNA oder DNA-Kandidat Strukturen. Der typische SELEX-Prozess besteht aus mehrere Runden der Bereicherung bis die Bibliothek in Sequenzen mit hoher Affinität und Spezifität zum Ziel angereichert ist. Der endgültige angereicherte Pool dann sequenziert und die Dissoziation konstanten (K-d) und Selektivität gegen mögliche Störsubstanzen durch verschiedene Techniken wie z. B. bestimmt werden Filtern verbindlich, Affinitätschromatographie, Oberfläche Plasmon-Resonanz (SPR), etc. 15
Aufgrund der extrem schlechten Wasserlöslichkeit und Mangel an funktionellen Gruppen für Oberfläche Immobilisierung ist die Aptamer-Auswahl des POPs theoretisch schwierig. Die Entdeckung der Aptamere haben bedeutende Fortschritte für SELEX beschleunigt. Die Auswahl der gruppenspezifischen Aptamere für POPs wurde jedoch noch nicht gemeldet. Bisher wurden nur PCB-Bindung DNA Aptameren mit hoher Spezifität für eine bestimmte Artgenossen identifizierte16. PAEs werden hauptsächlich in Polyvinylchlorid Materialien, Wechsel Polyvinylchlorid aus einem harten Kunststoff zu einem elastischen Kunststoff, fungiert somit als Weichmacher eingesetzt. Einige PAEs als endokrine Disruptoren eingestuft wurden, können schwere Schäden verursachen, Leber- und Nierenfunktion, reduzieren die Beweglichkeit der männlichen Spermien und abnormen Spermien Morphologie und Hodenkrebs17führen kann. Verbindung- weder die gruppenspezifischen PAE-Bindung Aptamere sind berichtet worden.
Das Ziel dieser Arbeit ist es, eine repräsentative Protokoll bieten für die Auswahl von gruppenspezifischen DNA Aptameren, stark hydrophobe kleine Moleküle wie PAEs, eine repräsentative Gruppe von POPs. Wir zeigen auch die Anwendung von der ausgewählten Aptamer für Umweltverschmutzung Erkennung. Dieses Protokoll bietet Beratung und Einblicke für die Aptamer-Entdeckung des anderen hydrophoben kleine Moleküle.
Protokoll
(1) Bibliothek und besser gekleideteres Design und Synthese
-
Die erste Bibliothek und Primer Design.
Bibliothek (Pool0): 5'-TCCCACGCATTCTCCACATC-N40-CCTTTCTGTCCTTCCGTCAC-3 "
Grundierung (FP) weiterleiten: 5'-TCCCACGCATTCTCCACATC-3 "
Phosphoryliert rückwärts-Primer (PO4- RP): 5'-PO4- GTGACGGAAGGACAGAAAGG-3 " - Synthetisieren Pool0FP und PO4- RP mit standard Phosphoramidit Chemie18,19,20, und reinigen Sie die DNA durch standard Hochleistungs-Flüssigkeitschromatographie (HPLC)21.
- Rekonstituieren Pool0FP und PO4- RP in der Nuklease-freie Klasse Wasser bei 100 µM, Aliquoten, und speichern Sie diese-20 oder-80 ° C bis zu einem Jahr.
Hinweis: Es wird dringend empfohlen, zum Speichern von Pool0, FP und PO4- RP in 10-20 µL Aliquots möglich Kreuzkontamination zu vermeiden.
(2) synthetisieren Sie das Anker-Ziel und die Immobilisierung auf Epoxy-aktivierte Agarose Bead
- Die Aminogruppe funktionalisiert Dibutyl Phthalat (DBP-NH2) als Anker Ziel zu synthetisieren.
Hinweis: Die experimentelle Details auf die Synthese und Charakterisierung von DBP-NH2 wurden in der Literatur22beschrieben. - Bereiten Sie den Anker Ziel Stammlösungen in einem Medium für die Ziel-Löslichkeit geeignet. Für diese Studie bereiten 100 mM DBP-NH2 Stammlösung in Dimethyl Sulfoxid (DMSO) und die Lösung bei 4 lagern oder-20 ° C bis zu einem Jahr.
- DBP-NH2 auf Epoxy-aktivierte Agarose Korne nach einem modifizierten Protokoll vom Hersteller zu immobilisieren.
- 0,1 g gefriergetrocknete Pulver von Epoxy-aktivierte Agarose Perlen abzuwiegen Sie und hängen Sie es in destilliertem Wasser. Waschen Sie das Medium für 1 h mit 20 mL destilliertem Wasser in mehrere Aliquote hinzugefügt. Waschen Sie das Medium mit 0,2 M Na2CO3 (pH-Wert 12).
Hinweis: Die mittlere schwillt unmittelbar nach Zugabe von Wasser hinein. - DBP-NH2 (46,7 mg, 0,15 Mmol) in 500 µL Kupplung Puffer (0,2 M Na2CO3 Puffer) auflösen.
- Mischen Sie die Kupplung-Lösung mit dem Liganden mit dem Medium. Verwenden Sie einen Shaker 5 u/min, für 48 h bei Raumtemperatur.
- Wiederholen Sie waschen das Produkt drei Mal nacheinander mit 0,1 M Acetat-Puffer (0,5 M NaCl, pH 4,5) und 0,2 M Carbonat Puffer (0,5 M NaCl, pH 12) in jedem Waschgang. Waschen Sie und hängen Sie das Produkt in Wasser, damit das Endvolumen von 500 µL.
- Lagern Sie das Produkt bei 4 ° C vor dem Gebrauch.
- Bestätigen Sie den Erfolg der Ziel-Immobilisierung durch Elementaranalyse.
- 0,1 g gefriergetrocknete Pulver von Epoxy-aktivierte Agarose Perlen abzuwiegen Sie und hängen Sie es in destilliertem Wasser. Waschen Sie das Medium für 1 h mit 20 mL destilliertem Wasser in mehrere Aliquote hinzugefügt. Waschen Sie das Medium mit 0,2 M Na2CO3 (pH-Wert 12).
(3) SELEX
- Bereiten Sie 500 mL PAE Bindung Puffer in Reinstwasser oder Nuklease-freie Grade Wasser mit 20 mM Tris· HCl, 100 mM NaCl 2 mM MgCl2, 5 mM KCl, 1 mM CaCl2, 1 % Polyoxyethy-Lene (20) Sorbaitan Monolaurate und 0,03 % nichtionischen oberflächenaktiven Mittel (pH 7,9). Filtern Sie des Puffers durch einen Sterilfilter 0,22 µm Nitrozellulose und Monate bei einer Temperatur über 20 ° C lagern.
Hinweis: Es ist entscheidend für eine gute Dispersion Status des PAEs im PAE Bindung Puffer aufrecht zu erhalten. Mehrere Arten von Tensiden sind erforderlich, um die Löslichkeit von PAEs in wässriger Lösung zu verbessern. Allerdings sollte die Anzahl der Tenside gehalten werden, auf ein Minimum, die Bildung von Surfactant Micellen zu vermeiden, die PAE Moleküle einschließen würde. Nicht-ionische Oberfläche Wirkstoff ist einfach zu überstürzen sich bei Temperaturen unter 20 ° C, und daher sollte der Puffer gespeichert werden, bei einer Temperatur über 20 ° C. - Verdünnen Sie 10 µL 100 µM Pool0(~1.0 Nmol für 10-14-10-15 -Sequenzen) in 490 µL PAE Bindung Puffer. Erhitzen Sie die Rohre für 5 min auf Eis Pool0 Aliquote (5 × 100 µL) in die Dünnwand-Zentrifuge Röhrchen in der Wasser-Bad-Set bei 95 ° C für 10 min. Platz und inkubieren Sie anschließend die Rohre für 5 min bei Raumtemperatur (~ 25 ° C in diesem Werk).
- Die erste Runde der Auswahl: Inkubation von Pool-0 und DBP−NH2−coated Medium.
- Waschen Sie 200 µL des DBP−NH2−coated Mediums dreimal mit 500 µL PAE Bindung Puffer. Mix DBP−NH2−coated Medium mit0 zu bündeln und die Mischung bei Raumtemperatur 1 h unter Schütteln milde inkubieren.
- Separate gebunden DBP−NH2−coated Medium mit Aptamere aus ungebundenen DNAs durch Ultrafiltration einem Ultrafiltration Rohr mit einem molekularen Cut-off von 100 kDa. Waschen Sie das Medium dreimal mit 500 µL PAE Bindung Puffer und Zentrifuge 9.168 × g für 10 min bei 4 ° C.
- Die erste Runde der Auswahl: Aptamer Elution von DBP−NH2−coated Medium.
- Fügen Sie 50 µL PAE Bindung Puffer der gewaschenen Medium und Hitze die Mischung bei 90 ° C für 9 min unter schütteln.
- Den Überstand mit der eluierten DNAs durch Ultrafiltration zu sammeln. Wiederholen Sie Elution Vorgang dreimal, mehr gebundene DNA wiederherstellen.
- Die erste Runde der Auswahl: PCR
-
Kleinen Maßstab PCR
- Führen Sie einen kleinen Maßstab PCR23 (4 × 20 µL Aliquots) mit ~ 15-30 Zyklen. Richten Sie eine PCR Reaktion24 wie folgt: 6,5 µL RNase-freies Wasser, 1 µL 10 µM Grundierung (FP, PO4- RP, 0,01 Nmol), 1,5 µL eluiert Pool ab Abschnitt 3.4 (oder RNase-freies Wasser als Negativkontrolle) und 10 µL Komplettformel Reaktion Mischung mit dNTPs, hot start Polymerase und 2 × PCR Reaktion Puffer (20 mM tris· HCl, 100 mM KCl, 3 mM MgCl2, pH 8,3).
- PCR mit einem Thermocycler mit folgenden Parametern ausführen: 1 Zyklus von 95 ° C, 1 min; 30 Zyklen von [95 ° C, 30 s, 56 ° C, 30 s, 72 ° C, 30 s]; 1 Zyklus von 72 ° C, 2 min und halten bei 4 ° C. Wenn das Gerät läuft, der 20th -s der Erweiterung Schritte an die 15, 20, 25 und 30 Zyklen, die Pause-Taste drücken, öffnen Sie den Gerätedeckel und nehmen Sie eine Röhre heraus, dann drücken Sie die Pause-Taste PCR fortzusetzen.
- 12 % denaturiert Polyacrylamid-Gel-Elektrophorese (PAGE)25,26vorzubereiten. 4,8 g Harnstoff, 6 mL Reinstwasser, 1 mL 10 × Tris/Borsäure mischen/Ethylen Diamin Tetraacetic Säure (TBE), 3 mL 40 % Acrylamid, 10 µL Tetramethylethylenediamine (TEMED) und 100 µL 10 % Ammonium Bleichen (APS), in dieser Reihenfolge. Gut umrühren und warten auf 1,5-2 h um sicherzustellen, dass das Gel vollständig erstarrt.
- Analysieren Sie die Ergebnisse der Zyklus-Optimierung: 1 µL des PCR-Produktes mit 5 µL RNA laden Puffer (62,5 % Formamid, 0,4 M Formaldehyd, 1.25×3-(N-morpholino)-Propansulfonic-Säure-Puffer, 0,02 % Xylol Cyanol FF, 0,02 % Bromophenol blue) und 4 µL kombinieren RNase-freies Wasser; Erhitzen Sie die Mischung bei 95 ° C für 10 min, und lassen Sie schnell auf 0 ° C auf Eis abkühlen; 5 min bei Raumtemperatur inkubieren; Last in 5 separate Vertiefungen des 12 % denaturiert Seite; laufen Sie Elektrophorese von einem vertikalen Elektrophorese-System in 1 × TBE Puffer bei 170 V 45 min.
- Nach Elektrophorese, Fleck, das Gel mit 3 µL 1 × Nukleinsäure-Gel in 30 mL 1 × TBE-Puffer für 10 min. Färben, und nehmen Sie ein Bild des Gels.
-
Large scale PCR: wählen Sie die entsprechende Anzahl von Zyklen, die höchste Intensität-Band in der richtigen Größe Marker ohne unerwünschte Nebenprodukte zu produzieren. Führen Sie großflächig PCR (20 × 100 µL Aliquots) Nutzung der Bedingungen und die entsprechende Anzahl von Zyklen, die in Abschnitt 3.5.1 bestimmt.
Hinweis: Die endgültige Höhe der einzelsträngige Bibliothek, die erreicht werden kann ist proportional zum Gesamtvolumen der PCR, nicht die Konzentration der Vorlage. In der Regel um 1 Nmol Bibliothek zu erhalten, benötigt die einsträngige Pool 2 mL PCR. PCR ist sehr empfindlich und leicht verschmutzte von außen DNAs. Schritte sollten unternommen werden, um vermindern das Risiko von Verunreinigungen, z. B. mit Handschuhen mit sterilisierten Tipps, nicht spucken in den Rohren und die Kappe des PCR-Röhrchen vor der Zentrifugation nicht öffnen.
-
Kleinen Maßstab PCR
- Die erste Runde der Auswahl: Reinigung des PCR-Produktes durch Ethanol Fällung.
- Kombinieren Sie zwei Röhren von PCR Mischung in eine Röhre (200 µL). Wiederholen Sie diesen Vorgang für alle Rohre.
- Fügen Sie 15 µL Natrium Acetat (3 M, pH 5,2) Lösung, 4 µL DNA/RNA-Niederschlag-Träger-Lösung und 2,5-Mal das Volumen der vorgekühlte Ethanol bei-20 ° C in jedem Rohr. Mischen Sie sie gleichmäßig.
Hinweis: DNA/RNA Niederschlag Träger ist in Ethanol Niederschlag Experimente verbreitet, um den Erholung-Anteil der DNA/RNA in geringen Konzentrationen deutlich zu verbessern. Downstream-Anwendungen wie PCR und Sequenzierung nicht beeinträchtigt werden. - Die Mischung bei 20.627 × g für 20 min bei 4 ° C zentrifugiert und sorgfältig überstand verwerfen und weißen Niederschlag verlassen.
- Fügen Sie 400 µL 70 % Vorkühlung Ethanol-Lösung bei-20 ° C in jedem Rohr um den Niederschlag zu waschen. Die Mischung bei 20.627 × g für 5 min bei 4 ° C zentrifugiert und sorgfältig überstand verwerfen und weißen Niederschlag verlassen. Der Waschschritt zwei weitere Male zu wiederholen.
- Öffnen Sie die Abdeckung der Röhre; Stechen Sie die Abdichtung Membran; lassen Sie Ethanol bei 40 ° C auf dem Heizblock verdampfen, bis die weiße Niederschlag durchsichtig wird. Speichern des Niederschlags bei-20 ° C.
Hinweis: Die gereinigte PCR-Produkt kann bei-20 ° c aufbewahrt werden
- Die erste Runde der Auswahl: einsträngige DNA-Generation (Generation angereicherter Pool1) durch die Durchführung der λ Exonuclease Reaktion den phosphorylierten reverse Strang zu verdauen.
-
Kleinen Maßstab λ Exonuclease Reaktion
- Eine Röhre mit dem gereinigten PCR-Produkt aus Abschnitt 3.6 Fügen Sie 100 µL RNase-freies Wasser hinzu. Wirbel der Röhre des Niederschlags in das Rohr vollständig auflösen.
- Bereiten Sie fünf Zentrifuge Rohre und fügen Sie 5 µL die oben genannte Lösung, 11 µL RNase - freies Wasser und 2 µL 10 × Reaktion Puffer (670 mM Glycin-KOH, 25 mM MgCl2 , 0,1 % (V/V) Triton x-100 pH 9,4) in jedes Rohr.
- Fügen Sie 2 µL Wasser oder verdünnten λ Exonuclease Lösung mit 2 U, U 5 und 8 U 10 U λ Exonuclease in diese Rohre bzw. hinzu. Mischen Sie die Lösung gut durch sanft pipettieren. 10 × Reaktion Puffer ist zusammen mit λ Exonucleasefrom den Anbieter zur Verfügung gestellt.
- Die Rohre für 35 min bei 37 ° C, 15 min bei 80 ° C inkubieren und bei 4 ° c zu halten
- Bereiten Sie die 12 % Native Seite. Mischen Sie 6 mL Reinstwasser, 1 mL 10 × TBE, 3 mL 40 % Acrylamid, 10 µL TEMED und 100 µL 10 % APS, in dieser Reihenfolge. Umrühren Sie gut und warten Sie 60-90 min um sicherzustellen, dass das Gel vollständig erstarrt.
- Analysieren Sie die Ergebnisse der Reaktionen durch die Kombination von 5 µL jeder Reaktion mit 1 µL Farbstoff und 4 µL RNase-freies Wasser laden und laden diese Mischungen in 5 separate Brunnen von 12 % Native Seite (150 V 45 min).
Hinweis: Eine 20 bp doppelsträngigen DNA-Leiter kann auch auf das Gel geladen werden, aber es möglicherweise nicht notwendig, da die unterschiedlichen Banden von PCR-Produkt und die generierten gestrandeten Bibliothek auf dem Gel zeigen.
Hinweis: Die λ Exonuclease Reaktion hat eine ausgezeichnete strukturellen Selektivität. Der doppelsträngigen PCR-Produkt (Anti-Sinnstrang trägt eine Phosphatgruppe am 5'-Ende) kann in eine einzelsträngige Bibliothek im Beisein von λ Exonuclease in einem ziemlich breiten Konzentrationsbereich (Abbildung 4) vollständig verdaut werden. Die groß angelegte λ Exonuclease Reaktion dient auch der Mindestbetrag von λ Exonuclease, die völlig einzelsträngiger DNA in kleinem Maßstab λ Exonuclease Reaktion erzeugen kann. Eine höhere Konzentration von λ Exonuclease kann auch verwendet werden, da λ Exonuclease in einem breiten Konzentrationsbereich gut funktioniert, ohne seine Struktur Selektivität und Produkt-Ausbeute. Die λ-Exonuclease-Reaktion wird durch eine hohe Konzentration des Salzes gehemmt. Die unvollständige Verdauung des PCR Produktes impliziert in der Regel, dass zu viel Salz in das PCR-Produkt vorhanden ist. Die Reinigung der PCR Produkte von Ethanol Niederschlag (Abschnitt 3.6) ist in der Regel kompatibel mit diesem Schritt, während das PCR-Produkt gereinigt durch Isopropanol-Fällung oft zu viel Salz enthält.
- Groß angelegte λ Exonuclease Reaktion: Wählen Sie den Mindestbetrag von λ Exonuclease, die völlig einzelsträngiger DNA für die groß angelegte λ Exonuclease Reaktion erzeugen kann. Die Reaktion ist nach diesem Zustand proportional skaliert. Zum Beispiel: Wenn 2 U den Mindestbetrag von λ Exonuclease erforderlich ist, mischen Sie 38 µL Rnase-freies Wasser, 10 µL 10-fach Puffer, 50 µL DNA-Pool1 und 2 µL 10 U/µL λ Exonuclease.
-
Kleinen Maßstab λ Exonuclease Reaktion
- Der ersten Runde der Auswahl: Reinigung der einzelsträngigen Pool1 durch Ethanol Fällung.
- Folgen Sie den Anweisungen des Schrittes 3.6.
- Bestimmen die Konzentration von gereinigt Pool1 durch UV-Absorption bei 260 nm.
- Bereiten Sie die 90 % gereinigt Pool1 in einem entsprechenden Volumen von PAE Bindung Puffer. Wenn es nicht genügend DNA für die nächste Runde der Auswahl erhalten, wiederholen Sie Abschnitte 3.5-3.8.2.
Hinweis: Die Lautstärke des PAE Bindung Puffer variiert in der Regel von 200 bis 500 µL entsprechend dem Umfang der Bibliothek und die gewünschte Endkonzentration der Bibliothek in die nächste Runde von SELEX.
- Die zweite, dritte, vierte und fünfte Runden von SELEX.
- Führen die zweite dritte, vierte und fünfte Runden von SELEX anschließend nach dem gleichen Verfahren beschrieben (Abschnitt 3.2-3.8), außer dass die ~ 300 Pmol Bibliothek eingegeben wurde, um die strenge Auswahl zu erhöhen.
- Um die unspezifische Absorption von DNAs auf das Medium zu minimieren, brüten am Pool mit unveränderten Medium auf einen Dreh Shaker für 30 min in die dritte und vierte Runde der Auswahl vor der Inkubation mit dem DBP−NH2−coated Medium.
Hinweis: Die SELEX Prozess endete in der fünften Runde aufgrund der schweren Crosslink zwischen DNAs durch die große Menge an hochmolekularen Produkt, das in das Gel migrieren nicht enthüllt.
4. Hochdurchsatz-Sequenzierung
- Bereiten Sie Pool4 von Abschnitt 3.9.2 für die Sequenzierung durch PCR-Amplifikation mit kompatiblen Primern.
- Entwerfen und synthetisieren eine indizierte vorwärts Grundierung mit 6 nt am 5 '-Ende die Sequenzanalyse zu erleichtern.
Forward Primer (FP-Sequenzierung) indiziert: 5'-AgtacgaTCCCACGCATTCTCCACATC-3 " - Richten Sie eine 1.000 µLPCR Reaktion wie folgt: 380 µL RNase-freies Wasser, 50 µL jeder 10 µM Grundierung (FP-Sequenzierung, PO4- RP, 0,5 Nmol), 20 µL von Abschnitt 3.9.2 eluierten Pool und 500 µL Komplettformel Reaktionsgemisch mit dNTPs, Heißstart Polymerase , und 2 × PCR Reaktion Puffer. Aliquoten Mischung in 10 PCR-Röhrchen. Verwenden Sie die gleichen PCR-Bedingungen, wie im Abschnitt 3.5.1.2 beschrieben.
- Entwerfen und synthetisieren eine indizierte vorwärts Grundierung mit 6 nt am 5 '-Ende die Sequenzanalyse zu erleichtern.
- Reinigen Sie das PCR-Produkt die Sequenzierung Anlage Anforderungen angepasst. Z. B. das Seite-Gel DNA Recovery Kit gemäß den Anweisungen des Herstellers.
- Senden Sie die gereinigte PCR-Produkt (~ 2 µg) an eine Sequenzierung Anlage unter den Versandbedingungen erforderlich durch die Sequenzierung Anlage. Zum Beispiel das PCR-Produkt in ein Zentrifugenröhrchen mit Kappen gründlich mit Eisbeutel auf die Sequenzierung Anlage versiegelt ausgeliefert.
Hinweis: Haben die Ergebnisse, die die Sequenzen nach der Kopienzahl jeder Sequenz zur Bereicherung der sequenzierten Pool Bewertung geordnet. Die obere Sequenz hieß DBP-1 (5'-CTTTCTGTCCTTCCGTCACATCCCACGCATTCTCCACAT-3 "). Seine Affinität und Selektivität wurden durch das folgende Protokoll (Abschnitt 5 und 6) gemessen. Ihre Anwendung in einer elektrochemischen Bio-sensing zeigte sich anschließend im Abschnitt 7.
5. Kd Bestimmung der ausgewählten Aptamer Kandidaten mittels Magnetic Bead-Based Quantitative PCR (qPCR)
- DBP-1-qPCR und Primer verwenden standard Phosphoramidit Chemie zu synthetisieren und durch den standard HPLC zu reinigen.
Aptamer Kandidat (DBP-1-qPCR): 5'-ATACCAGCTTATTCAATTCTTTCTGTCCTTCCGTCACATC-CCACGCATTCTCCACATAGATAGTAAGTGCAATCT-3 "
Grundierung für qPCR (FP-qPCR) weiterleiten: 5'-ATACCAGCTTATTCAATT-3 "
Grundierung für qPCR (RP-qPCR) umkehren: 5'-AGATTGCACTTACTATCT-3 "
Hinweis: Die Grundierung Regionen der getestete Sequenz unterscheiden sich von denen der Pool0 verwendet in Abschnitt 3. Ändern der Grundierung Regionen soll die Affinität der Kern Sequenz allein, mit Ausnahme der Grundierung Regionen testen. - DBP-NH2 auf Carbonsäure-beschichtete magnetische Beads nach den Anweisungen des Herstellers zu immobilisieren.
- Waschen Sie die Carbonsäure-beschichtete magnetische Beads zweimal mit 25 mM 2-(N-Morpholino) Ethanesulfonic Säure (MES) (pH 5,0), mit ein gleiches Volumen des magnetischen Beads (100 µL) pipettiert aus der Durchstechflasche für 10 min mit gute Durchmischung (über durchgängige o.ä.).
- Bereiten Sie unmittelbar vor der Anwendung 50 mg/mL 1-ethyl-3-(3-dimethylaminopropyl) Carbodiimide Hydrochlorid (EDC) Lösung mit kalten 25 mM MES (pH 5,0).
- Bereiten Sie unmittelbar vor der Anwendung 50 mg/mL N-Hydroxysuccinimide (NHS) Lösung mit kalten 25 mM MES (pH 5,0).
- Mischen Sie 100 µL der EDC-Lösung und 100 µL der NHS-Lösung mit den gewaschenen Perlen und inkubieren Sie für 30 min bei Raumtemperatur mit langsamen Tilt Rotation.
- Habe das Rohr auf ein Magnet für 4 min nach der Inkubation, überstand verwerfen und Perlen zweimal mit 100 µL der kalten 25 mM MES (pH 5,0) zu waschen.
- Inkubieren Sie 6 µL der Stammlösung DBP-NH2 100 mM und 25 mM MES (pH 5,0, 290 µL) mit den aktivierten Perlen unter Drehung für mindestens 30 min bei Raumtemperatur oder 2 h bei 4 ° C, mit langsamen Tilt Rotation bei 5 u/min.
- Habe das Rohr auf den Magneten für 4 min nach der Inkubation und den Überstand verwerfen. Waschen der beschichteten Perlen 4 Mal und Aufschwemmen Perlen mit 200 µL PAE Bindung Puffer und Speicher bei 4 ° C bis zur Verwendung.
-
Sammeln Sie Titration Kurve um Kdzu bestimmen.
- Bereiten Sie eine Reihe von DBP-1 Lösungen (500 µL) bei unterschiedlichen Konzentrationen in PAE Bindung Puffer von 13:00 bis 300 nM.
- Fügen Sie 10 µL des DBP-NH2-magnetische Kügelchen beschichtet, wie im Abschnitt 5.2.7 zu jeder DBP-1-Lösung beschrieben. Inkubation für 1 h bei Raumtemperatur unter Drehung.
- Legen Sie die Röhren auf ein Magnet für 4 min und entfernen Sie den Überstand zu. Waschen Sie die Perlen 4 Mal mit 200 µL des PAE-Bindung-Puffers.
- Jedes Rohr 60 µL PAE Bindung Puffer hinzufügen. Inkubieren Sie die Rohre bei 95 ° C für 15 min. sammeln des Überstands mit der eluierten DBP-1 durch magnetische Trennung. Wiederholen Sie Elution um mehr gebundene DBP-1 wiederherzustellen.
- Die Elute Lösung 100fach verdünnen und 3 µL für Echtzeit-qPCR zu nehmen. Richten Sie eine qPCR Reaktion wie folgt: 3 µL RNase-freies Wasser, 2 µL 10 µM Grundierung (FP, PO4- RP, 0,01 Nmol), 3 µL eluiert und verdünnt DBP-1 und 10 µL Komplettformel Reaktionsgemisch, dNTPs, Polymerase und 2 X PCR Reaktion Puffer enthält.
- Bestimmen Sie die Nummern der DBP-1 in jeder Probe mit qPCR mit einem Thermocycler mit den folgenden Einstellungen: 1 Zyklus von 95 ° C, 30 s; 30 Zyklen von [95 ° C, 30 s; 45 ° C, 30 s, 72 ° C, 30 s]; 1 Zyklus von 72 ° C, 30 s und Halt bei 4 ° C.
- Die Titration Kurve des Grundstückes und Kd durch nichtlineare Anpassung der Kurve unter der Annahme einer Bindung 1:1 Verhältnis27bestimmen.
(6) relative Affinität und Spezifität Test von wettbewerbsfähigen Assays
- Fügen Sie 10 µL des DBP-NH2-magnetische Beads, DBP-1-Lösung (500 µL, 1 µM) beschichtet. Inkubation für 1 h bei Raumtemperatur unter Drehung. Legen Sie die Röhren auf ein Magnet für 4 min und entfernen Sie den Überstand zu. Waschen Sie die Perlen 4 Mal mit 200 µL PAE Bindung Puffer und in 10 µL PAE Bindung Puffer Aufschwemmen.
- Fügen Sie 10 µL des DBP-NH2-beschichtete mittlere gebunden mit DBP-1 110 µL jeder 10 µM getestet Probe (DBP-NH2, DBP, DEHP, Butyl Benzyl phthalate(BBP), Schwermetall-Ionen, etc..) Inkubation bei Raumtemperatur für 1 h sammeln der Überstand durch magnetische Trennung.
- Den Überstand 100fach verdünnen und 3 µL für qPCR Quantifizierung zu nehmen. Richten Sie eine qPCR Reaktion wie folgt: 3 µL RNase-freies Wasser, 2 µL 10 µM Grundierung (FP, PO4- RP, 0,01 Nmol), 3 µL eluiert und verdünnt DBP-1 und 10 µL Komplettformel Reaktionsgemisch, dNTPs, Polymerase und 2 X PCR Reaktion Puffer enthält.
- Bestimmen Sie die Nummern der DBP-1 in jeder Probe mit qPCR mit einem Thermocycler mit den folgenden Einstellungen: 1 Zyklus von 95 ° C, 30 s; 30 Zyklen von [95 ° C, 30 s; 45 ° C, 30 s, 72 ° C, 30 s]; 1 Zyklus von 72 ° C, 30 s und Halt bei 4 ° C.
- Die relative Affinität zu berechnen, indem die Anzahl der DBP-1 befreit die Perlen im Beisein der Probe durch die Anzahl der DBP-1 aus den Perlen im PAE Bindung Puffer freigegeben: Relative Affinität = NProbe/nPuffer.
7. Herstellung und elektrochemische Messungen von DEHP elektrochemische Biosensoren
- Thiolated Kern Sequenz (HS-DBP-1) und die Signalisierung Sonde (DBP-1-C-Fc) mit standard Phosphoramidit Chemie zu synthetisieren und durch den standard HPLC zu reinigen.
HS-DBP-1: 5' - HS-(CH2)6- CTTTCTGTCCTTCCGTCACATCCCACGCATTCTCCACAT -3 "
DBP-1-C-Fc: 5 '- GATGTGACGGAAGGTTTTTTTT-(CH2)6Fc-3"
Hinweis: Die rationale Gestaltung der Sensor Sonden beschrieb unserer bisherigen Pbulications18,22. - HS-DBP-1 und DBP-1-C-Fc in Nuklease-freie Grade Wasser bei 100 µM, Aliquoten, rekonstruieren und speichern Sie diese-20 oder-80 ° C bis zu einem Jahr.
- Polieren der gold Elektrode sorgfältig, um eine spiegelglatte Oberfläche mit 1, 0,3 und 0,05 µm Al2O3 Pulver auf eine Mikrotresse für 5 min und Reinstwasser für 5 min zum Rest Al2O3entfernen beschallen. Reinigen Sie die Elektrode durch elektrochemische Polieren mit 35 aufeinanderfolgenden zyklischer Voltammetrie (CV) Scans von 0,4 bis 1,2 V (vs. Hg-Hg2SO4) in 0,5 M H2SO4 bei 100 mV/s.
- Bereiten Sie eine Mischung von 0,5 µM HS-DBP-1 und 0,5 µM DBP-1-C-Fc in 100 µL 10 mM Phosphatpuffer mit 1 M NaCl, pH 7.4 (PBS). Erhitzen Sie die Mischung in die Dünnwand-Zentrifugenröhrchen in das Wasser-Bad-Set bei 95 ° C für 10 min, und kühlen Sie langsam das Gemisch auf Raumtemperatur ab.
- Fügen Sie 1 µL Tris--(2-carboxyethyl)-Phosphin-Hydrochlorid (DÄMMUND, 10 mM, Stammlösung) in die Mischung. Halten Sie bei Raumtemperatur für 1 h.
- Tauchen die saubere goldene Elektrode in die oben genannte Lösung für 12 h, oder über Nacht bei Raumtemperatur.
- Spülen Sie die Elektrode mit PBS und tauchen die Elektrode 1 mM [S (CH2)2(OCH2CH2)6OCH3]2 (HS-zB6- OMe) mit PBS-Puffer für 1 h spülen Sie gründlich mit PAE Elektrode verbindliche Puffer und tauchen die Elektrode im PAE Bindung Puffer.
- Ziel-freie PAE Bindung Puffer beteiligen Sie Voltammetrie (SWV) Hintergrundsuche Rechtecksignal.
- Reinigen Sie die experimentelle Ausstattung: Elektrolysezellen, gold Arbeitselektrode und Platin Gegenelektrode gesättigt Kalomel Bezugselektrode (SCE).
- Aktivieren Sie die installierte Software; die experimentellen Parameter für SWV Messungen festgelegt.
Hinweis: Die folgenden experimentellen Parameter dienten für die SWV-Experimente: Potentialbereich: von -0,2 bis 0,7 V; Amplitude Modulation: 25 mV; Puls-Breite: 5 ms; Abtastrate: 50 mV/s; Schritt Potenzial: 1 mV. - Anschließen der Gold arbeiten Elektrode, Platin Gegenelektrode und SCE zu einem Potentiostaten, und diese drei Elektroden in einer Elektrolysezelle, PAE Bindung Puffer enthält.
- SWV Messung ausgeführt.
Hinweis: Einmal die SWV Messung beginnt eine SWV-Kurve werden auf dem Bildschirm des Computers angezeigt. Der Scan wird wiederholt, bis die Scans konstant sind und keine weiteren Änderungen in Peakhöhe oder Form eingehalten werden.
- Tauchen die Arbeitselektrode im PAE Bindung Puffer mit einer bestimmten Konzentration von DEHP (z. B. 22:00) für 30 min bei Raumtemperatur. Spülen Sie gründlich mit PAE Bindung Puffer Elektrode und Tauchen Sie die Elektrode im PAE Bindung Puffer mit der Gegenelektrode und SCE. Sammeln der SWV-Kurve unter den gleichen Bedingungen wie in Abschnitt 7.8 beschrieben.
- Wiederholen Sie Schritt 7.9, außer dass die Konzentration von DEHP (z. B. 100 pM, 1 nM, 10 nM, 100 nM, 1 µM) konnte gegenüber dem Vorquartal gesteigert werden. Plotten Sie die Titration Kurve.
- Spezifität Tests: Wiederholen Sie die Schritte 7.3-7.9, außer dass die PAE-Bind-Puffer mit DEHP durch den PAE-Bindung-Puffer, die den möglichen störenden Stoff in der gewünschten Konzentration ersetzt wird.
Ergebnisse
Wir entwickelt und synthetisiert die Aminogruppe funktionalisiert Dibutyl Phthalat (DBP-NH2) als Anker Ziel (Abb. 1F). Wir spielten dann die DNA-Aptamer-Auswahl der PAEs mit DBP-NH2 als Anker Ziel und im Anschluss an das klassische Ziel Immobilisierung basierende Methode (Abbildung 2). In jeder Runde war eine pilot PCR durchgeführt mit der denaturierten Seite optimieren die Zykluszahl PCR (Abbildung 3). Die denaturierte Seite statt native Seite, wird dringend empfohlen, weil wir und andere Kollegen gefunden zu haben, dass die vermuteten hochmolekularen Nebenprodukte gezeigt auf die native Gele bei höheren Taktzahlen tatsächlich Vernetzung komplexe gebildet durch Sequenzen der richtigen Größe. Somit kann die Band Intensität der denaturierten Seite wirklich die Menge an das richtige Produkt reflektieren. Die einzige stranded DNA-Generation ist ein weiterer wichtiger Schritt, und viele Methoden für diesen Schritt gewesen gemeldeten28. Die λ Exonuclease Verdauung ist die billigste Methode. Der Erfolg der einzelsträngigen DNA-Generation kann bequem vom native Seite, wo das PCR-Produkt als angezeigt wird kontrolliert werden (erste mehrere Runden von SELEX) einzelne oder mehrere bands, während nur eine Band nach der Verdauung (Abb. 4) dargestellt ist.
Die SELEX wurde gestoppt, nachdem die fünfte Runde der Auswahl wegen der bedeutenden Menge von DNA gesammelt am oberen Rand der Seite, auch nach der Denaturierung Behandlung (wobei DNAs bei 95 ° C für 10 min in 2 × TBE – enthält 8,3 M Harnstoff erhitzt werden) , die ernsthafte Vernetzung zwischen DNAs vorgeschlagen und auch darauf hingewiesen, dass die Sequenzen angereichert wurden. Daher wurde der Pool4 für den Hochdurchsatz-Sequenzierung verschickt. Das Hochdurchsatz-Ergebnis zeigte, dass die Top 100, die am häufigsten auftretenden Sequenzen hoch konserviert wurden und von einer Familie waren. Die obere Sequenz DBP-1 nanomolaren Affinität (Abbildung 5) und gute Gruppe-Spezifität für PAE Kongenere (DBP, BBP, DEHP) angezeigt (Abbildung 6) über die bequeme qPCR-Assays, wie im Abschnitt Protokoll beschrieben.
DBP-1 wurde verwendet, um einen Strang Verschiebung-basierte elektrochemischen Aptasenor entsprechend konstruieren, unsere bereits berichtet arbeiten29. Die DEHP-Sensor kann empfindlich und selektiv auf DEHP (Abbildung 7) reagieren. Die gemeinsame Umweltschadstoffe wie Schwermetall-Ionen, Antibiotika und kleine Moleküle mit ähnlichen funktionellen Gruppen zeigten sehr schwachen Reaktion auf DBP-1.

Abbildung 1: Chemische Strukturen von (A) PAEs, (B) BBP, DBP (C), (D) DEHP, (E) 4-OH-DEHP, (F) DBP−NH2. Diese Zahl wurde mit freundlicher Genehmigung von Y. Han Et Al. nachgedruckt 22 Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Gruppe-spezifische DNA-Aptamer Auswahlverfahren für PAEs mit DBP-NH2 als Anker Ziel. Diese Zahl wurde mit freundlicher Genehmigung von Y. Han Et Al. nachgedruckt 22 Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Vertreter ergibt sich aus PCR-Zyklus-Optimierung mittels denaturierten Seite. Von links nach rechts, die Gassen vertreten: 20 bp DNA-Leiter Standard 15 Zyklen (keine DNA-Vorlage), 20 Zyklen (keine DNA-Vorlage), 25 Zyklen (keine DNA-Vorlage), 30 Zyklen (keine DNA-Vorlage), 15 Zyklen, 20 Zyklen, 25 Zyklen und 30 Zyklen. Optimale Bedingungen wurden beobachtet bei 25 Zyklen, wo war die Einzelprodukt Band mit hoher Intensität ohne unerwünschte Nebenprodukte, darüber hinaus keine Band in der entsprechenden Blindkontrolle beobachtet wurde. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: die Optimierung der λ Exonuclease Reaktion den phosphorylierten Strang mit native Seite verdauen. Von links nach rechts, die Gassen vertreten: 20 bp DNA-Leiter Standard, negativ (keine λ Exonuclease), 2U, 5U, 8U und 10U. Der Mindestbetrag des Enzyms wurde bei 2U, beobachtet wo wird das doppelte PCR-Produkt einzelsträngiger DNA. Bitte klicken Sie hier für eine größere Version dieser Figur.
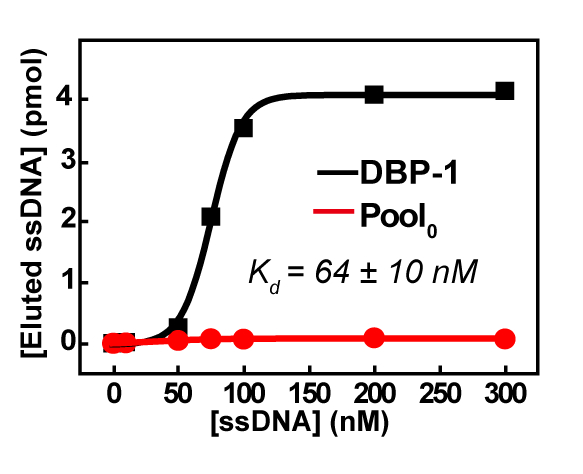
Abbildung 5: Affinität Messungen der DBP-1 von qPCR basierende Assays. Der Fehler (Standardabweichung, SD) von der K-d wurde aus drei Messungen der gleichen Probe berechnet. Diese Zahl wurde mit freundlicher Genehmigung von Y. Han Et Al. nachgedruckt 22

Abbildung 6: Relative Affinität Messungen der DBP-1 gratis DBP-NH2 (10 µM), DBP (10 µM), DEHP (10 µM), BBP (10 µM), Ethylacetat (10 µM), Benzoesäure (10 µM), Phthalsäure Säure (10 µM) und anderen über Wettbewerb Assays. Sonstiges: eine Mischung aus potenziellen Störungen (Glukose, Kanamycin, Ampicillin und Ethanol) jeweils 10 µM. Das Verhältnis (die relative Affinität) wurde berechnet, indem die Anzahl der Aptamere befreit die Perlen im Beisein der Probe durch die Anzahl der Aptamere aus Perlen im PAE Bindung Puffer freigegeben. Die Balken repräsentieren Mittelwert ± SD Die SDs wurden aus drei Einzelmessungen berechnet. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 7: DEHP elektrochemische Biosensor Gromer und ultraselective Nachweis von DEHP: Mechanismus (A), SWV Kurven (B), Eichkurve (C) und Selektivität testet (D). Die Fehler (SDs) wurden aus drei Einzelmessungen berechnet. Diese Zahl wurde mit freundlicher Genehmigung von Y. Han Et Al. nachgedruckt 22 Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Ein herausragender Vorteil von Aptameren ist, dass sie erkannt werden, durch die in-Vitro -Methode SELEX, während Antikörper über in Vivo Immunoreactions generiert werden. Daher können Aptamere mit gewünschten Spezifität durchdachte experimentellen Bedingungen ausgewählt werden, während Antikörper auf physiologischen Bedingungen beschränkt sind.
Um die Trennung der gebundenen Sequenzen von freien Sequenzen zu erleichtern, mehrere modifizierten SELEX vor kurzem berichtet wurde, in welche Kapillarelektrophorese30, Mikrofluidik31, magnetisch / Acryl Perlen / Agarose Perlen14, usw., ersetzt Nitrozellulosefilter oder Affinität Spalten um eine effizientere Trennung zu erzielen. Unter diesen Techniken, die Wulst-basierten Methoden am häufigsten werden seit der Aptamer-Auswahl der niedermolekularer Ziele durch ihre einfache Einrichtung, einfache Bedienung und sein kleines Molekül Analysen zugänglich.
Es gibt zwei Gruppen von Methoden, die üblicherweise für Aptamer Auswahl der niedermolekularer Ziele verwendet werden: das Ziel13 und Bibliothek14 Immobilisierung-basierten Methoden. In der ehemaligen Gruppe von Methoden sind die kleinen Molekül-Ziele auf der Festphase wie funktionalisierten magnetische Beads oder Agarose Perlen über kovalente Kupplungsreaktionen immobilisiert. Bibliotheken sind mit kleinen Molekül beschichtete Perlen inkubiert und die Sequenzen, die nicht binden oder binden Sie schwach an das Ziel an der festen Phase werden einfach durch Waschen und Zentrifugation oder magnetische Trennung Schritte entfernt. Die gebundenen Sequenzen sind anschliessend eluiert und durch PCR verstärkt. Das kleine Molekül-Ziele müssen mindestens eine funktionelle Gruppe für die Kupplung Reaktion zur Verfügung. Für diejenigen ohne geeignete funktionelle Gruppen hat die funktionelle Gruppe in das ursprüngliche Ziel durch organische Synthese eingearbeitet werden. Die Synthese aus den sorgfältig gestalteten Ziel könnte mehrere Schritte und manchmal auch recht anspruchsvoll ist. Die bauliche Veränderung der Ziele könnte auch stark die Bindungsstellen und Affinität zu ihren Aptamere beeinflussen. Zur Vermeidung dieser Probleme Bibliothek Immobilisierung-basierte Methoden entwickelt wurden, wo die Bibliothek zu ergänzenden hybridisiert ist DNA-Erfassung, Sonden auf magnetische Beads und Bindung Sequenzen Abfallen der Perlen auf Bindung mit den Zielen. In dieser Gruppe von Methoden die Ziele sind in der Bindung Puffer hinzugefügt und keine funktionellen Gruppen erforderlich sind. PAEs sind die Ester-Derivate von Phthalat-Säure, und eine typische PAE besteht aus einer Phthalat-Acid Ester-Gruppe und ein oder zwei Alkyl-Ketten (Abbildung 1A-D). PAEs haben keine funktionellen Gruppen für feste Phase Ruhigstellung zur Verfügung. So scheinen die Bibliothek Immobilisierung-basierte Methoden noch attraktiver für die Aptamer-Auswahl der PAEs.
Ein gut kontrolliertes Abstrahlverhalten-Status ist wichtig, um die gewünschte Bereicherung der Bibliothek per SELEX sicherzustellen. Allerdings sind extrem hydrophoben PAEs und leicht bilden Aggregate in wässrigen Lösungen mit einer Konzentration höher als einige µM, sogar in Anwesenheit von mehreren Tensiden Dispersion zu erleichtern. So, der Dispersion Status PAEs ist schwer zu kontrollieren, und es wäre schwierig, Aptamere auszuwählen, die speziell auf einzelne PAE-Moleküle binden. Um die Löslichkeit-Problem zu lösen, wurden Ziel-Immobilisierung Methoden ausgewählt, in denen PAEs auf der hydrophilen Perlen über kovalente Bindung immobilisiert wurden. Mithilfe dieser Immobilisierung Strategie sollte die Ziele auf der Oberfläche der Perle in einem molekularen begeben, anstatt Aggregate dominant vorhanden sein.
Der konstruktive Aufbau des Ziels Anker ist auch ganz entscheidend für die erfolgreiche Selektion von gruppenspezifischen Aptamere. Drei Faktoren für die Konstruktion berücksichtigt werden müssen. Der erste Faktor ist der Zweck der Aptamer-Auswahl. In Anbetracht der Zweck der Auswahl gruppenspezifische Aptamere unbedingt zu entlarven die gemeinsame Gruppe von PAEs für Aptamer Bindung und verhindern, dass der Rest der Teile an die Aptamer-Bindung. Daher sollte der funktionellen Gruppe am Terminal der Seitenkette eingeführt werden. Am Anfang, wir OH funktionalisiert DEHP (4-OH−DEHP) (Abbildung 1E) als Anker Ziel entwickelt und auf die Carbonsäure magnetische Beads16immobilisiert. So wurden die Alkyl-Ketten auf der Oberfläche der Matrix ausgesetzt. Wir haben versucht, magnetische Beads mit Carboxylgruppen als festen Matrix wählen. Keine offensichtlichen Affinität Verbesserung wurde nach fünf Runden der Auswahl beobachtet, und die unspezifische Absorption auf der nackten Matrix blieb stark.
Daher, DBP-NH2 wurde später entwickelt, basierend auf folgenden Gedanken: (1) die Phthalat-Gruppe ist eher zu interagieren, speziell mit dem Aptamer durch die π-π-Stapel und die Wasserstoff-Bindung als Alkyl Kette; (2) NHS-vermittelten Carbodiimide Reaktion ist mild und hat einen sehr effizienten Kupplung Wirkungsgrad, so dass am Ende ein Alkyl Kette von DBP -NH2 eingeführt wird; (3) Es ist einfach zu bedienen mit magnetischen Beads als Festphase Partition, aber die unspezifische Adsorption der Bibliothek ist stark. Obwohl der Verlust der Agarose Korne während Trennung höher ist als die magnetische Beads ist, ist die unspezifische Adsorption der Bibliothek wesentlich geringer. So, DBP-NH2 wurde auf den Epoxy-aktivierte Agarose-Perlen immobilisiert und phthalic Acidester Gruppe auf der Oberfläche der Matrix ausgesetzt war.
Die Wahl der Auswahlpuffer ist wichtig, besonders für kleine Molekül Ziele mit unterschiedlichen Löslichkeit. Die Bindung Puffer muss sorgfältig vorbereitet zu vermeiden Aggregation und gewährleisten einen gute Dispersion Zustand von POPs während des gesamten Aptamer Auswahl und Charakterisierung. In unserer Studie fanden wir, dass DEHP in reguläre Puffer ohne Tenside, auflösen kann nicht mit zwei unterschiedlichen Schichten. Die Schicht verschwindet bei Zugabe von mehreren Tenside in der optimalen Menge. Die klare Lösungen bei Temperaturen über 20 ° C auf ihren Status gespeichert werden müssen. Angaben im Protokoll Schritt 3.1.
Einzelsträngigen DNA-Generation von doppelsträngigen PCR-Produkte ist ein wichtiger Schritt in die SELEX-Verfahren. Verschiedene Methoden sind derzeit in der Literatur32,33, einschließlich asymmetrische PCR, λ Exonuclease Verdauung und magnetische Trennung mit Streptavidin beschichteten Perlen Größe Trennung durch Denaturierung beschrieben worden Harnstoff-Polyacrylamid-Gel.
Verschiedene Methoden haben ihre eigenen Stärken und Schwächen. Derzeit ist die am häufigsten verwendete Methode zur Erzeugung von SsDNA magnetische Trennung mit Streptavidin beschichteten Perlen. Der herausragende Vorteil dieser Methode ist die zeitsparende und einfache Bedienung. Der Nachteil ist sein hoher Preis im Vergleich zu anderen Methoden. Im Gegensatz dazu gehört die Größe Trennung-basierte Methode, bei der reverse Primer mit GC-reichen Stem-Loop-Struktur, die billigsten Methoden während der Ertrag von einzelsträngiger DNA die niedrigste unter diesen Methoden32ist. In diesem Protokoll beschrieben wir die Verwendung von λ Exonuclease Verdauung, einzelsträngigen DNA zu generieren ist eine der billigsten Methoden. Wir fanden, dass die Ausbeute von einzelsträngiger DNA vergleichbar mit den zwei Methoden der magnetische Trennung und Streptavidin beschichteten Perlen. Darüber hinaus fanden wir, dass die Exonuclease Reaktion durch die hohe Konzentration des Salzes gehemmt wurde. Die unvollständige Verdauung des PCR Produktes berichtet in der Literatur33 war wahrscheinlich wegen zu viel Salz in das PCR-Produkt vorhanden. Darüber hinaus ist der λ-Exonuclease sehr aktiv und kostengünstig (Abbildung 4).
Die Charakterisierung und Validierung von Aptameren ist aufwändig, zeitraubend und verkörpert einen größeren Engpass in der Aptamer-Entdeckung-Pipeline-33. Die meisten Techniken sind Masse-Sensitive Methoden, die auch für die größeren Aptamer Bindungspartner arbeiten (> 10.000 amu), aber sind nicht ausreichend empfindlich auf die Interaktionen mit niedrigem Molekulargewicht Ziele messen (< 1.000 amu)15. Die Charakterisierung und Validierung von Aptameren hydrophobe kleiner Moleküle wie PAEs ist sogar noch schwieriger. Ihre schlechte Wasserlöslichkeit ergibt die Doppelbindungen der Titration Kurve, die verhindert, dass die K-d-Bestimmung in Lösung oder Aptamere auf Oberfläche immobilisiert. Deshalb entschlossen wir Kds der identifizierten Aptamere von Hochdurchsatz-Sequenzierung von Immobilisierung DBP-NH2 auf der hydrophilen magnetische Beads um die Löslichkeit zu vermeiden. Die relativen Affinitäten und Selektivität der Aptamere für andere PAEs wurden dann durch die wettbewerbsfähige Assays bestimmt, wo die Aptamer-Kandidaten der DBP-NH2 Pre begrenzt waren angeschlossen magnetische Beads und veröffentlicht, um den Überstand nach Inkubation mit den getesteten PAEs oder anderen potentiell störenden Substanzen. Die wettbewerbsfähigere Assay wurde angewendet, weil sie einen einfache Affinität Vergleich zwischen PAEs bereitgestellt, die keine funktionellen Gruppen für Oberfläche Immobilisierung hatte. Darüber hinaus eignen sich magnetische Wulst-basierte fluoreszierende Assays für die Affinität Studie von kleinen Molekül-Aptamer Interaktionen34. Allerdings haben wir festgestellt, dass die magnetische Beads manchmal verursachen Fluoreszenzlöschung aus unbekannten Gründen. So wurden die qPCR-Assays für die Affinität Messungen verwendet.
Eine kritische Technik-Tipp für die elektrochemische Biosensor beschrieben in dieser Studie ist die Oberfläche Passivierung der Elektrode35. Aufgrund der hohen Hydrophobie von DEHP hat es eine starke Tendenz zur nonspecifically auf die gold Elektrode, führt zum Ausfall des Fundes aufgenommen werden. Am meisten allgemein verwendeten Oberflächen-Passivierung Agent, 6-Mercapto-1-Hexanol (MCH)36,37, ist nicht ausreichend, um zu verhindern, dass die unspezifische Absorption von DEHP, während wir, dass HS-(CH2)2-[OCH2CH2 fanden ]6- OCH3 war wirksam genug, um die empfindliche Detektion von DEHP38zu ermöglichen.
Dieses Verfahren beschreibt ein Protokoll für die Auswahl von gruppenspezifischen DNA Aptameren stark hydrophobe kleiner Moleküle und eine Anwendung von der ausgewählten Aptamer in eine elektrochemische Biosensor. Das Protokoll hilft bei der Auswahl der anderen hydrophoben kleine Moleküle und bietet Einblicke in Sensorentwicklung stark hydrophobe kleiner Moleküle sowie. Das Aptamer-Auswahlverfahren gehört zur Kategorie Ziel Immobilisierung-basierter Methoden. Die Grenzen dieser Art von Methode gibt es auch für dieses Protokoll, z. B. die Notwendigkeit für komplizierte Synthese von Anker-Ziele und die Auswirkungen der Festphase Aptamer-Bindung. Die attraktive elektrochemischen Biosensoren, die in diesem Protokoll beschriebenen Vorteile ihr schlichtes Design und hohe Empfindlichkeit. Der größte Nachteil ist die begrenzte Präzision durch ihre extrem breiten Dynamikbereich. Daher eignen sich die hier beschriebenen Biosensoren mehr für screening-Tests statt quantitative Messungen der Ziele.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Wir sind dankbar für die finanzielle Unterstützung von der National Natural Science Foundation (21675112), Key-Projekt von Wissenschaft und Technik Plan von Peking Education Commission (KZ201710028027) und Yanjing Young Scholar Program der Capital Normal University.
Materialien
| Name | Company | Catalog Number | Comments |
| UV-2550 | Shimadzu,Japan | protocol, section 3.8.2 | |
| DNA Engnine Thermal cycler,PTC0200 | BIO-RAD | section 3.5.1.2 and 3.5.2 | |
| C1000 Touch | BIO-RAD | section 5.3.6 and 6.3 | |
| VMP3 multichannel potentiostat | Bio-Logic Science, Claix, France | section 7.4,7.8 and 7.11 | |
| Epoxy-activated Sepharose 6B | GE Healthcare (Piscataway, NJ, USA) | 10220020 | argarose beads, section 2.3 and 3.3 |
| Dynabeads M-270 carboxylic acid magnetic beads | Invitrogen, USA | 420420 | magnetic beads,section 5.2. and 5.3 |
| Premix Taq Hot Start Version | Takara,Dalian,China | R028A | polymerase, section 3.5.1.1 |
| PARAFILM Sealing Membrane | Bemis, USA | PM-996 | section 3.6.5 |
| Lambda Exonuclease | Invitrogen, USA | EN0561 | section3.7.1.2.The 10 × reaction buffer is provided along with λ exonuclease by the provider. |
| Dr. GenTLE Precipitation Carrier | Takara,Dalian,China | 9094 | section 3.6.2 and 3.8.1 |
| UNIQ-10 PAGE DNA recovery kit | Sangon Biotech (Shanghai) | B511135 | section 4.2 |
| SYBR Gold nucleic acid gel stain | Invitrogen, USA | 1811838 | nucelic acid stain dye, section 3.5.1.5 |
| SYBR Premix Ex Taq II | Takara,Dalian,China | RR820A | polymerase mix contaning polymerase and dNTPs, section 5.3.5 |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Sigma-Aldrich | CAS: 1132-61-2 | section 5.2.1 |
| 1-ethyl-3-(3-dimethylaminopropyl) carbodiimide hydrochloride (EDC) | Invitrogen, USA | CAS: 25952-53-8 | section 5.2.2 |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 6066-82-6 | section 5.2.3 |
| mercaptohexanol (MCH) | Sigma-Aldrich | CAS: 1633-78-9 | section 7.7 |
| Gold electrode | Shanghai Chenhua | CHI101 | section 7.4. - 7.11 |
| tris(2-carboxyethyl) phosphine hydrochloride (TCEP) | Sigma-Aldrich | CAS: 51805-45-9 | section 7.5 |
| O-(2-Mercaptoethyl)-O'-methyl-hexa-(ethylene glycol) | Sigma-Aldrich | CAS: 651042-82-9 | section 7.7 |
| diethylhexyl phthalate (DEHP) | National Institute of Metrology, China | CAS: 117-81-7 | section 7.11 |
| Tween 20 | Sigma-Aldrich | CAS: 9005-64-5 | polyoxyethy-lene(20) sorbaitan monolaurate |
| Triton X-100 | Sigma-Aldrich | CAS: 9002-93-1 | non-ionic surface active agent |
| PBS | Sigma-Aldrich | P5368 | 10 mM phosphate buffer containing 1 M NaCl, pH 7.4 |
Referenzen
- Vorkamp, K., Riget, F. F. A review of new and current-use contaminants in the Arctic environment: Evidence of long-range transport and indications of bioaccumulation. Chemosphere. 111, 379-395 (2014).
- Net, S., Sempere, R., Delmont, A., Paluselli, A., Ouddane, B. Occurrence, fate, behavior and ecotoxicological state of phthalates in different environmental matrices. Environ Sci Technol. 49 (7), 4019-4035 (2015).
- Xie, Q. L., Liu, S. H., Fan, Y. Y., Sun, J. Z., Zhang, X. K. Determination of phthalate esters in edible oils by use of QuEChERS coupled with ionic-liquid-based dispersive liquid-liquid microextraction before high-performance liquid chromatography. Anal BioanalChem. 406 (18), 4563-4569 (2014).
- Ierapetritis, I., Lioupis, A., Lampi, E. Determination of phthalates into vegetable oils by isotopic dilution gas chromatography mass spectrometry. Food Anal Methods. 7 (7), 1451-1457 (2014).
- Sun, J. Z., He, H., Liu, S. H. Determination of phthalic acid esters in Chinese white spirit using dispersive liquid-liquid microextraction coupled with sweeping beta-cyclodextrin-modified micellar electrokinetic chromatography. J Sep Sci. 37 (13), 1679-1686 (2014).
- Yilmaz, P. K., Ertas, A., Kolak, U. Simultaneous determination of seven phthalic acid esters in beverages using ultrasound and vortex-assisted dispersive liquid-liquid microextraction followed by high-performance liquid chromatography. J Sep Sci. 37 (16), 2111-2117 (2014).
- Sun, R., Zhuang, H. An ultrasensitive gold nanoparticles improved real-time immuno-PCR assay for detecting diethyl phthalate in foodstuff samples. Anal Biochem. 480, 49-57 (2015).
- Sun, R. Y., Zhuang, H. S. A sensitive heterogeneous biotin-streptavidin enzyme-linked immunosorbent assay for the determination of di-(2-ethylhexyl)phthalate (DEHP) in beverages using a specific polyclonal antibody. Anal Methods. 6 (24), 9807-9815 (2014).
- Zhou, L., Lei, Y., Zhang, D., Ahmed, S., Chen, S. An ultra-sensitive monoclonal antibody-based enzyme-linked immunosobent assay for dibutyl phthalate in human urinary. Sci Total Environ. 541, 570-578 (2016).
- Shen, J., Li, Y., Gu, H., Xia, F., Zuo, X. Recent development of sandwich assay based on the nanobiotechnologies for proteins, nucleic Acids, small Molecules, and ions. Chem Rev. 114 (15), 7631-7677 (2014).
- Yin, X. -. B. Functional nucleic acids for electrochemical and electrochemiluminescent sensing applications. TrAC, Trends Anal Chem. 33, 81-94 (2012).
- Nguyen, V. -. T., Kwon, Y. S., Gu, M. B. Aptamer-based environmental biosensors for small molecule contaminants. Curr Opin Biotechnol. 45, 15-23 (2017).
- Groher, F., Suess, B. In vitro selection of antibiotic-binding aptamers. Methods. 106, 42-50 (2016).
- Yang, K. -. A., Pei, R., Stojanovic, M. N. In vitro selection and amplification protocols for isolation of aptameric sensors for small molecules. Methods. 106, 58-65 (2016).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal. Chim. Acta. 686 (1-2), 9-18 (2011).
- Mehta, J., et al. Selection and characterization of PCB-binding DNA aptamers. Anal Chem. 84 (3), 1669-1676 (2012).
- Matsumoto, M., Hirata-Koizumi, M., Ema, M. Potential adverse effects of phthalic acid esters on human health: A review of recent studies on reproduction. Regul Toxicol Pharm. 50 (1), 37-49 (2008).
- Goodchild, J. Conjugates of oligonucleotides and modified oligonucleotides: a review of their synthesis and properties. Bioconjug Chem. 1 (3), 165-187 (1990).
- Brown, D. M. A brief history of oligonucleotide synthesis. Protocols for Oligonucleotides and Analogs: Synthesis and Properties. , 1-17 (1993).
- Reese, C. B. Oligo-and poly-nucleotides: 50 years of chemical synthesis. Org Biomol Chem. 3 (21), 3851-3868 (2005).
- Sproat, B., Colonna, F., Mullah, B., et al. An efficient method for the isolation and purification of oligoribonucleotides. Nucleos Nucleot Nucl. 14 (1-2), 255-273 (1995).
- Han, Y., et al. Selection of group-specific phthalic acid esters binding DNA aptamers via rationally designed target immobilization and applications for ultrasensitive and highly selective detection of phthalic acid esters. Anal Chem. 89 (10), 5270-5277 (2017).
- Bartlett, J. M. S., Stirling, D. A short history of the polymerase chain reaction. PCR protocols. , 3-6 (2003).
- Saiki, R. K., Scharf, S., Faloona, F., et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 230, 1350-1354 (1985).
- Albright, L. M., Slatko, B. E. Denaturing polyacrylamide gel electrophoresis. Current Protocols in Nucleic Acid Chemistry. , A. 3B 1-A. 3B 5 (2001).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). JoVE. (32), e1485 (2009).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1), 9-18 (2011).
- Sharma, T. K., Bruno, J. G., Dhiman, A. ABCs of DNA aptamer and related assay development. Biotechnol Adv. 35 (2), 275-301 (2017).
- Liu, R., et al. Signaling-probe displacement electrochemical aptamer-based sensor (SD-EAB) for detection of nanomolar kanamycin A. Electrochim Acta. 182, 516-523 (2015).
- Mendonsa, S. D., Bowser, M. T. In vitro evolution of functional DNA using capillary electrophoresis. J Am Chem Soc. 126, 20-21 (2004).
- Lou, X. H., et al. Micromagnetic selection of aptamers in microfluidic channels. Proc Natl Acad Sci USA. 106 (9), 2989-2994 (2009).
- Cho, M., et al. Quantitative selection of DNA aptamers through microfluidic selection and high-throughput sequencing. Proc. Natl. Acad. Sci. U.S.A. 107, 15373-15378 (2010).
- Cho, M., et al. Quantitative selection and parallel characterization of aptamers. Proc Nat Acad Sci USA. 110 (46), 18460-18465 (2013).
- Song, K. -. M., et al. Gold nanoparticle-based colorimetric detection of kanamycin using a DNA aptamer. Anal Biochem. 415 (2), 175-181 (2011).
- Yang, Z., Ding, X., Guo, Q., et al. Second generation of signaling-probe displacement electrochemical aptasensor for detection of picomolar ampicillin and sulfadimethoxine. Sens Actuators B. 253 (2017), 1129-1136 (2017).
- Lou, X., Zhao, T., Liu, R., Ma, J., Xiao, Y. Self-assembled DNA monolayer buffered dynamic ranges of mercuric electrochemical sensor. Anal Chem. 85 (15), 7574-7580 (2013).
- Zhao, T., et al. Nanoprobe-enhanced, split aptamer-based electrochemical sandwich assay for ultrasensitive detection of small molecules. Anal Chem. 87 (15), 7712-7719 (2015).
- Lou, X., He, L. A. Surface passivation using oligo(ethylene glycol) in ATRP-assisted DNA detection. Sens Actuators,B. 129 (1), 225-230 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten