Method Article
Adaptation des semi-automatique de circulation des cellules tumorales (CTC) Essais pour les applications cliniques et précliniques de recherche
Dans cet article
Résumé
Cellules tumorales circulantes (CTC) sont pronostique dans plusieurs cancers métastatiques. Ce manuscrit décrit le système de l'étalon-or CellSearch (CSS) CTC plate-forme d'énumération et met en lumière les erreurs de classification communs. En outre, deux protocoles adaptés sont décrits pour défini par l'utilisateur marqueur caractérisation des CTC et CTC énumération dans des modèles murins précliniques de métastases utilisant cette technologie.
Résumé
La majorité des décès liés au cancer surviennent à la suite de l'apparition de la maladie métastatique. Ce stade de la maladie hautement létale est associée à la présence de cellules tumorales circulantes (CTC). Ces cellules rares ont été démontrés comme étant d'importance clinique dans le sein métastatique, de la prostate et les cancers colorectaux. L'étalon-or actuel dans la détection clinique de la CCT et le dénombrement est le système CellSearch approuvé par la FDA (CSS). Ce manuscrit décrit le protocole standard utilisé par cette plate-forme, ainsi que deux protocoles adaptés supplémentaires qui décrivent le processus détaillé de l'optimisation de marqueur définie par l'utilisateur pour la caractérisation des protéines de CTC patient et un protocole comparable pour CTC capture dans de très faibles volumes de sang, en utilisant la norme réactifs CSS, pour étudier dans des modèles murins précliniques in vivo de métastases. En outre, les différences de qualité entre les CTC sang de donneur sain dopés avec des cellules de culture de tissu par rapport à des patients samp de sangles sont mis en évidence. Enfin, plusieurs articles couramment discordants qui peuvent conduire à des erreurs de classification erronée de la CCT sont présentés. Pris ensemble, ces protocoles fourniront une ressource utile pour les utilisateurs de cette plate-forme intéressé dans la recherche préclinique et clinique relative à la métastase et CTC.
Introduction
En 2013, on estime que 580 350 personnes mourront du cancer et que 1.660.290 nouveaux cas de cette maladie sera diagnostiquée aux États-Unis seulement 1. La majorité de ces décès surviennent à la suite de l'apparition de la maladie métastatique 2. L'absence actuelle de traitements efficaces dans le traitement des métastases et une compréhension limitée de la cascade métastatique rend ce stade de la maladie souvent mortelle. La présence de cellules tumorales circulantes (CTC) dans le sang ont été démontrées en corrélation avec une maladie métastatique 3. Ces cellules sont extrêmement rares et leur détection est indicatif de la survie globale dans le sein métastatique 4, 5 prostate, colorectal et le cancer 6. Chez ces patients, la présence de ≥ 5 ≥ 3 (colorectal) CTC dans 7,5 ml de sang (sein et prostate) ou est indicatif de moins bon pronostic par rapport aux patients avec peu ou pas du CTC détectables dans le samle volume de courrier de sang. En outre, le changement du nombre de CTC pendant ou après une intervention thérapeutique a été démontrée comme étant utile en tant que prédicteur de la réponse au traitement, souvent plus rapidement que les techniques actuellement utilisées 7-10.
Il a été estimé que, chez les patients atteints d'un cancer métastatique, CTC se produisent à une fréquence d'environ 1 CTC par 10 5 à 10 7 cellules mononucléées du sang et chez les patients présentant une maladie localisée, cette fréquence peut être encore plus faible (~ 1 à 10 8). La rareté de ces cellules, il peut être difficile à détecter avec précision et de manière fiable et analyser CTC 11. Plusieurs méthodes (examen préalable 12-14) ont été utilisées pour enrichir et de détecter ces cellules en exploitant les propriétés qui les différencient des environs composants sanguins. En général, la CCT énumération est un processus en deux parties qui nécessite à la fois une étape d'enrichissement et une étape de détection. Traditionnellement, les étapes d'enrichissement s'appuient sur les différences dans l'éducation physiqueques des propriétés CTC (taille des cellules, la densité, la déformabilité) ou sur l'expression du marqueur de protéine (par exemple la molécule d'adhésion des cellules épithéliales [EpCAM], de cytokératine [CK]). À la suite de l'enrichissement, la détection de CTC peut être effectuée dans un certain nombre de façons différentes, dont les plus courantes sont les dosages à base d'acide nucléique et / ou des méthodes de cytométrie. Chacune de ces stratégies sont uniques, présentant des avantages et des inconvénients distincts, mais ils manquent tous de normalisation; une nécessité pour l'entrée dans le milieu clinique. Le système CellSearch (CSS) a donc été développé pour fournir une méthode normalisée pour la recherche et le dénombrement des CTC rares dans le sang humain en utilisant la microscopie par fluorescence et des techniques à base d'anticorps 4-6. Cette plate-forme est actuellement considéré comme l'étalon-or dans la CCT énumération et est la seule technique approuvé par la US Food and Drug Administration (FDA) pour une utilisation dans la clinique 15.
Le CSS est une plate-forme à deux composants consisting de, (1) le système CellTracks AutoPrep (ci-après dénommé l'instrument de préparation), qui automatise la préparation des échantillons de sang humain, et (2) la CellTracks Analyzer II (ci-après dénommé l'instrument d'analyse), qui numérise ces échantillons suivants préparation. Pour distinguer les CTC de leucocytes contaminants de l'instrument de préparation emploie un anticorps médiée, l'approche de séparation magnétique à base ferrofluide et différencié coloration fluorescente. Initialement, le système CTC étiquettes en utilisant des anticorps anti-EpCAM conjugués à des nanoparticules de fer. L'échantillon est ensuite incubé dans un champ magnétique, et toutes les cellules non marquées sont aspirés. Cellules tumorales sélectionnées sont remises en suspension, et on les incube dans un colorant de fluorescence différentielle, constitué d'anticorps marqués par fluorescence et un réactif de coloration nucléaire. Enfin, l'échantillon est transféré à une cartouche magnétique, appelé magnest (ci-après dénommé le dispositif magnétique), et scanned en utilisant l'instrument d'analyse.
L'instrument d'analyse est utilisé pour numériser les échantillons préparés en utilisant les filtres de fluorescence différents, chacun optimisé pour la particule fluorescente appropriée, en utilisant une lentille d'objectif 10X. CTC sont identifiées comme des cellules qui sont liées par des anticorps anti-EpCAM, l'anti-pan-CK-phycoérythrine (PE) (CK8, 18, et 19), et la coloration nucléaire 4 ',6-diamidino-2-phénylindole (DAPI). A l'inverse, les leucocytes contaminants sont identifiées comme des cellules qui sont liées par l'anti-CD45-allophycocyanine (APC) et DAPI. À la suite de l'analyse, les cellules tumorales possibles définis par l'ordinateur sont présentées à l'utilisateur. A partir de ces images, l'utilisateur doit employer une analyse qualitative en utilisant les paramètres définis et coloration différentielle décrits ci-dessus afin de déterminer quels événements sont CTC.
En plus de fournir une méthode standardisée de dénombrement CTC, le CSS permet la caractérisation moléculaire des CTC sur la base de marqueurs de protéines d'intérêt. Cette interrogation cun être effectuée au niveau de la cellule, à l'aide d'un isothiocyanate de fluorescéine (FITC) canal de fluorescence n'est pas nécessaire pour l'identification de la CCT 16. Bien que cette plate-forme fournit la capacité pour la caractérisation moléculaire, le processus détaillé de développement de protocole et l'optimisation n'est pas bien définie. Trois marqueurs disponibles dans le commerce ont été mis au point par le fabricant pour l'utilisation avec le CSS, incluant récepteur épidermique de facteur de croissance (EGFR), récepteur humain de croissance épidermique de facteur 2 (HER2), et le facteur de croissance analogue à l'insuline 1 receptor (IGF-1R). analyse de HER2, en combinaison avec le CSS, a été utilisé par plusieurs groupes pour illustrer le potentiel pour la caractérisation de la CCT pour éclairer la prise de décision clinique et de modifier potentiellement des directives de traitement existantes. Par exemple, Fehm et al. 17 démontré que près d'un tiers des patients atteints de cancer du sein avec des tumeurs HER2-primaire avait HER2 + CTC. En outre, Liu et al.18 récemment rapporté que jusqu'à 50% des patients atteints de SA + cancer du sein métastatique HER2 n'a pas eu + CTC. Herceptin, un récepteur HER2 interférer anticorps monoclonal démontré à bénéficier grandement les patients dont les tumeurs expriment des niveaux suffisants de HER2, est un traitement couramment utilisé pour les patients atteints de HER2 + tumeurs primaires 19-21. Cependant, ces études suggèrent que Herceptin peut être étant sous-utilisés de manière optimale et que la CCT caractérisation peut aider à prédire la réponse au traitement. En fin de compte, la caractérisation de la CCT peut avoir le potentiel d'améliorer les soins personnalisé.
Recherche de la CCT est unique en ce qu'il a largement utilisé une approche chevet à paillasse. Cette méthode, contrairement paillasse au chevet du patient recherche, qui peut souvent prendre des années à l'impact des soins aux patients, a permis CTC entrée rapide dans le cadre clinique. Toutefois, les médecins hésitent à utiliser les résultats de l'analyse de la CCT dans le traitement des patients prise de décisionment en raison d'un manque de compréhension de la biologie sous-jacente. Par conséquent modèles murins précliniques appropriées des techniques de métastases et d'analyse complémentaire CTC doivent être utilisés afin d'étudier ces questions en suspens. En général, il existe deux types de modèles précliniques utilisés pour étudier la cascade métastatique, (1) les modèles de métastase spontanée, qui permettent l'étude de toutes les étapes de la cascade métastatique, et (2) des modèles de métastases expérimentales qui ne permettent l'étude des étapes ultérieures du processus métastatique tels que extravasation et la formation secondaire de la tumeur 22. Des modèles de métastase spontanée, impliquent des injections de cellules tumorales dans des emplacements appropriés (par exemple orthotopique d'injection de cellules de cancer de la prostate dans la prostate pour l'étude du cancer de la prostate). Les cellules sont alors données de temps pour former des tumeurs primaires et des métastases spontanément à des sites secondaires, tels que l'os, le poumon et les ganglions lymphatiques. Dansrevanche, les modèles de métastases expérimentales impliquent l'injection directe de cellules tumorales dans la circulation sanguine (par exemple, via la veine caudale ou par injection intracardiaque de cellules cibles à des emplacements spécifiques) et donc sauter les étapes initiales de intravasation et la diffusion d'organes secondaires 22. Jusqu'à présent, la majorité de l'analyse CTC in vivo des systèmes modèles a été réalisée en utilisant soit 23 ou adaptés techniques CCT basée sur les droits, sur la base de cytométrie (p. ex AdnaTest) 24. Bien qu'utile, aucune de ces techniques reflètent adéquatement CTC énumération en utilisant le CSS de l'étalon-or. Sur la base de l'approbation clinique, la nature standardisée, et l'utilisation généralisée de la CSS, le développement d'une capture et la détection technique CTC pour la modélisation in vivo qui utilise la préparation de l'échantillon équivalent, le traitement et les critères d'identification serait avantageux que les résultats seraient comparables à ceux obtenu à partir d'échantillons de patients. Toutefois, en raison de la req de volumeuirements de l'instrument de préparation, il n'est pas possible de traiter des petits volumes de sang à l'aide de cette plate-forme automatisée. . Par ailleurs, des travaux antérieurs par Eliane et al 25 a démontré que la contamination des échantillons avec des cellules épithéliales de la souris (qui répondent aussi à la définition standard de la CCT [EpCAM + CK + DAPI + CD45 -]) peut conduire à des erreurs de classification des cellules épithéliales squameuses de la souris comme CTC. Pour répondre à ces questions une technique adaptée qui permet l'utilisation des réactifs du kit CSS CCT combinés avec un manuel de procédure d'isolement a été développé. L'addition d'un antigène de leucocyte humain marqué au FITC (HLA) de l'anticorps à l'essai permet de cellules tumorales humaines pour les distinguer des cellules épithéliales squameuses de souris.
Ce manuscrit décrit la norme, développée commercialement et protocole CSS optimisé pour le traitement des échantillons de sang des patients et les pièges courants qui peuvent être rencontrés, y compris discrepant des éléments qui peuvent conduire à des erreurs de classification de la CCT. Par ailleurs, la personnalisation de l'essai de CSS pour examiner les caractéristiques de protéines définies par l'utilisateur de CTC capturées et une technique comparable CSS adapté qui permet l'enrichissement et la détection de CTC à partir de petits volumes de sang chez les souris modèles précliniques de métastase sont décrits.
Protocole
Toutes les études humaines décrites dans ce manuscrit ont été réalisées selon des protocoles approuvés par le Comité d'éthique de la recherche humaine de l'Université Western. Toutes les études sur les animaux ont été effectuées en conformité avec les recommandations du Conseil canadien de protection des animaux, selon des protocoles approuvés par le Sous-Comité d'utilisation des animaux de l'Université Western.
Une. Norme CTC énumération des échantillons de sang de patients avec des CSS
Une. Sang humain Collection et préparation des échantillons pour le traitement de l'instrument Préparation
- En utilisant des techniques de saignée d'asepsie standard, tirer un minimum de 8,0 ml de sang humain dans un tube de 10 ml de CellSave (ci-après dénommé le tube de conservation CTC), qui contient de l'acide éthylène diamine (EDTA) et un conservateur cellulaire propriétaire. Inversez les 5x tube pour empêcher le sang de coaguler. Les échantillons peuvent être traités immédiatement ou conservés à température ambiante pendant jusqu'à 96 heures.
- REMOVréactifs e CSS du réfrigérateur et les laisser se réchauffer à température ambiante avant de l'utiliser.
- Utilisation d'une pipette jetable de 10 ml et pipette automatisée, collecter 7,5 ml de sang à partir du tube d'agent de conservation et distribuer CTC lentement sang dans un tube d'instrument préparation transformation marquée de manière appropriée.
- Ajouter 6,5 ml de tampon de dilution de chaque échantillon. Mélanger en retournant échantillon 5x. Extrait Centrifugeuse à 800 g pendant 10 min avec le frein en position "off". Suivez les instructions à l'écran sur l'instrument de préparation pour charger tous les échantillons de patients dans le système de traitement. Les échantillons doivent être traitées dans les 1 heure de préparation.
2. Préparation de contrôle pour le traitement de l'instrument Préparation
- Vortex doucement le flacon de contrôle et inverser 5x pour mélanger.
- Retirer le bouchon du flacon de contrôle et placer une préparation instrument tube de traitement inversé sur le dessus du flacon non bouché. Dans un mouvement rapide, inverser lacontrôler flacon, et verser le contenu dans le tube de traitement. Tandis inversé, effleurer doucement les côtés du flacon de commande pour libérer le contenu restant.
- Retirez délicatement le flacon de contrôle inversée à partir du tube de traitement, en veillant à ce qu'aucun liquide est perdu et mettre de côté. En utilisant une pipette de 1000 ul, recueillir le contenu restant de la cuvette et le couvercle et la pipette doucement dans le tube de traitement.
- Suivez les instructions à l'écran sur l'instrument de préparation à charger le contrôle sur le système de traitement.
3. Numérisation de l'échantillon sur l'instrument d'analyse
- Suivez les instructions à l'écran sur l'instrument de préparation pour décharger tous les échantillons du système. Librement plafonner chaque cartouche de dispositif magnétique et appuyez sur l'appareil magnétique à l'aide des mains ou paillasse pour libérer les bulles qui sont collées sur les bords de la cartouche. Une fois toutes les bulles ont été enlevés, Cap fermement la cartouche, mets le dispositif magnétique plat, et incubate dans l'obscurité pendant au moins 20 min à température ambiante. Les échantillons doivent être analysés dans les 24 heures de préparation.
- Allumer l'instrument d'analyse et d'initialiser la lampe. Une fois réchauffé (~ 15 min), de charger la cartouche de vérification du système sur l'instrument d'analyse et sélectionnez l'onglet Test QC. Suivez les instructions à l'écran pour effectuer les mesures de contrôle de qualité nécessaires.
- Chargez un échantillon sur l'instrument d'analyse et sélectionnez l'onglet Test patients. Toutes les informations enregistrées par l'instrument de préparation sera affiché. Cliquez sur Démarrer pour initialiser la numérisation de l'échantillon. Le système effectue une mise au point rapide et la détection de bord sur la cartouche de dispositif magnétique.
- Réglez tous les bords que nécessaire en utilisant les touches directionnelles. Sélectionnez Accepter. Le système effectue une mise au point fine et commencer la numérisation de l'échantillon.
- Après contrôle la numérisation des résultats devrait être validée en utilisant les critères définis pour les cellules enrichis à haute (CK + DAPI + CD45 - APC +) et bas (CK + DAPI + CD45 - FITC +) des concentrations. Après échantillon de patient balayage des résultats devrait être revu pour CTC capturées en utilisant les critères définis CCT (CK + DAPI + CD45 -).
2. Caractérisation de la CCT pour les marqueurs définis par l'utilisateur en utilisant le CSS
Une. Préparation des marqueurs définis par l'utilisateur et l'instrument d'initialisation
- Diluer l'anticorps d'intérêt en utilisant Bond Anticorps primaire diluant à la concentration désirée dans une cuvette de réactif de marquage à l'aide de la formule suivante, où la concentration de travail est la concentration de l'anticorps après addition de l'échantillon et la concentration des stocks est la concentration d'anticorps dans l' réactif tasse. Pour de multiples échantillons, ajuster les volumes anticorps tels que décrits dans le Tableau 1. Placer la cuvette dans le réactif marqueur dans une position de la cartouche de réactif et lOad la cartouche sur CSS.
Stock Concentration = (Concentration de travail x 850 pi) / 150 pi - Prélever le sang, la préparation des échantillons, et charger l'instrument de préparation comme décrit dans la norme ci-dessus CTC énumération des échantillons de sang des patients utilisant le protocole de CSS. Pour permettre plus de marqueur personnalisé, sélectionnez Défini par l'utilisateur test lorsque vous êtes invité par l'instrument de préparation. Entrez le nom du marqueur et sélectionnez Enregistrer. Comme les échantillons sont chargés sur l'instrument, l'opérateur sera invité à indiquer qui doit recevoir marqueur personnalisé en sélectionnant Oui ou Non, si nécessaire.
2. Numérisation de l'échantillon de marqueurs définis par l'utilisateur sur l'instrument d'analyse
- Allumez l'instrument d'analyse, initialiser la lampe, et effectuer un contrôle de la qualité et de la vérification du système comme décrit dans la section 3.2 de la Norme CTC énumération des échantillons de sang des patients utilisant le CSS protocole.
- Chargez un échantillon sur l'instrument d'analyse et sélectionnez l'onglet Configuration. Pour initialiser le canal FITC, sélectionnez CellSearch CTC comme l'ID du kit dans la section Protocoles de test. Dans ce menu, sélectionnez recherche de la CCT, cliquez sur le bouton Modifier et définir le temps d'exposition souhaitée. Il est recommandé une durée d'exposition de 1,0 sec pas être dépassée lors de l'utilisation du kit CSS CTC car cela peut augmenter purge par d'autres canaux fluorescents utilisés pour l'identification de la CCT.
3. Adaptation de la CSSProtocol standard pour une utilisation dans des modèles précliniques de souris
** Adapté de Veridex souris / rat CellCapture Kit (n'est plus disponible dans le commerce)
Une. Souris de prélèvement sanguin et de stockage
- Avant la collecte de sang, entrent ~ 30 ul d'EDTA 0,5 M et-vient à travers une aiguille 22 G, en laissant une petite quantité d'EDTA dans le moyeu.
- Recueillir un minimum de 50 pi de sang de souris de souris préalablement injectées avec des cellules tumorales humaines par des voies orthotopiques, veine de la queue, ou intracardiaque. Prélever le sang de la veine saphène (pour l'analyse de série CTC) ou par ponction cardiaque (pour l'analyse borne CTC). Retirer l'aiguille et distribuer le sang dans un tube EDTA Microtainer collecte de sang de 1 ml. Mélanger par inversion ou le tube doucement film pour empêcher le sang de coaguler. Le sang peut être traité immédiatement ou conservé à la température ambiante pendant jusqu'à 48 heures après l'addition d'un volume égal de CytoChex conservateur cellulaire.
2. CTC enrichissement
- Retirer réactifs CSS du réfrigérateur et laissez-les se réchauffer à température ambiante avant de l'utiliser.
- Transférer l'équivalent de 50 ul de sang entier à un débit de 12 mm x 75 mm cytométrie tube. Ajouter 500 pi de tampon de dilution de chaque échantillon, en faisant descendre le sang qui reste sur les parois du tube. Si nécessaire, une rotation de la centrifugeuse à courtpeut être utilisé pour recueillir du sang restant.
- Vortexer doucement le ferrofluide anti-EpCAM et ajouter 25 ul de chaque échantillon en plaçant la pointe de la pipette directement dans l'échantillon. Ajouter 25 ul de réactif de capture d'amélioration et vortex doucement pour mélanger. Incuber les échantillons à la température ambiante pendant 15 min.
- Placer les tubes échantillons dans l'aimant et incuber pendant 10 min. Alors que les tubes d'échantillons sont encore en l'aimant, utiliser une pipette en verre pour aspirer délicatement le liquide résiduel sans toucher la paroi du tube à côté de l'aimant et le jeter.
3. CTC coloration
- Retirer les tubes d'échantillon à partir de l'aimant et remettre en suspension dans 50 pl d'acide nucléique de colorant, 50 ul de réactif de coloration, 1,5 ul d'anticorps anti-souris CD45-APC, 5,0 ul d'anticorps anti-humain HLA-Alexa Fluor 488, et 100 ul de perméabilisation Réactif. Pour de multiples échantillons, ces réactifs peuvent être prémélangés et 206,5 pi de mélange peuvent être ajoutés à chaque tube. Vortex doucement pour mélangeret incuber pendant 20 min à température ambiante dans l'obscurité.
- Ajouter 500 ul de tampon de dilution, vortex doucement, tubes lieu de l'échantillon dans l'aimant, et incuber pendant 10 min. Alors que les tubes d'échantillons sont encore en l'aimant, utiliser une pipette en verre pour aspirer délicatement le liquide résiduel sans toucher la paroi du tube à côté de l'aimant et le jeter. Retirer les tubes d'échantillon à partir de l'aimant et remettre en suspension dans 350 ul de tampon de dilution. Vortex légèrement pour bien mélanger.
4. Dispositif magnétique Chargement en cours
- En utilisant une pointe de charge de gel, transfert soigneusement la totalité du volume du tube à échantillon dans une cartouche dans le dispositif magnétique. Commencez par le bas de la cartouche et retirer lentement la pointe de l'échantillon est distribué.
- Une fois l'ensemble de l'échantillon a été transféré, le capuchon lâche la cartouche de dispositif magnétique et appuyez sur l'appareil magnétique à l'aide des mains ou paillasse pour libérer les bulles qui sont collées sur les bords de la cartouche comme décrit dans Section 3.1 de la Norme CTC énumération des échantillons de sang des patients utilisant le CSS.
- Pop toutes les bulles en utilisant une aiguille 22 G stérile en les piégeant entre le biseau et le bord de la cartouche. Une fois toutes les bulles ont été enlevés, plafonner fermement la cartouche, fixer le dispositif magnétique plat, et incuber à l'obscurité pendant au moins 10 min. Les échantillons doivent être analysés dans les 24 heures de préparation.
5. Numérisation des échantillons manuellement séparés sur l'instrument d'analyse
- Allumez l'instrument d'analyse, initialiser la lampe, et effectuer un contrôle de la qualité et de la vérification du système comme décrit dans la section 3.2 de la Norme CTC énumération des échantillons de sang des patients utilisant le protocole de CSS.
- Chargez l'échantillon sur l'instrument d'analyse et sélectionnez l'onglet Configuration. Effacer toutes les données existantes sur le bouton de données de dispositif magnétique en cliquant sur le bouton Format de l'échantillon. Activer le canal FITCe régler le temps d'exposition à 1,0 sec, comme décrit à la section 2.2 de la caractérisation de la CCT pour les marqueurs définis par l'utilisateur à l'aide de la CSS.
- Cliquez sur l'onglet Test du patient et sélectionnez Modifier pour saisir l'information échantillon. Sélectionnez CellSearch CTC comme ID Kit et recherche de la CCT que le protocole de test. Entrez les informations nécessaires restant comme indiqué que l'astérisque. Sauvegarder l'information échantillon et cliquez sur Démarrer.
Résultats
Norme CTC énumération Assay
La sensibilité et la spécificité du CSS a été bien documenté dans la littérature. Toutefois, pour valider la récupération CTC équivalent, dopés (cellules de cancer de la prostate humain 1000 LNCaP) et non surchargés échantillons de sang humain provenant de donneurs volontaires en bonne santé ont été traitées sur le CSS en utilisant le protocole de CSSCTC standard. Comme prévu, les échantillons non dopés étaient libres de CTC, 0,00 ± 0,00%, et la récupération de la CCT a été démontrée pour être de 86,9 ± 4,71% pour les échantillons enrichis (figure 1A). Galerie CSS images obtenues à partir d'échantillons enrichis ont été d'une qualité optimale et CTC étaient faciles à distinguer des non-CTC. Cependant, lors du traitement des échantillons obtenus à partir de patients atteints de cancer, l'identification des CTC est un peu plus difficile, avec de nombreuses cellules apparaissant de plus petite taille et être moins faciles à distinguer des non-CTC (figure 1B). En outre, lors de l'examen des échantillons de patients 6 catégories d'événements étaient identificationed qui étaient souvent discordants entre les éléments critiques (figure 1C). Ces 6 catégories confondues, (1) les petits événements qui ne satisfont à l'exigence 4 de la taille de um pour la classification CTC; (2) éléments avec CK faible et / ou coloration DAPI; (3) justifié (doivent être comptés comme un CTC) par rapport injustifiée (ne doit pas être compté comme un CTC) saigner à travers dans le canal CD45-APC causée par lumineux coloration CK-PE; (4) FITC + événements; (5) images pixélisées dans la CK et / ou canaux de DAPI, et (6) les événements avec la coloration DAPI qui est plus grand que les images de la CK ou ceux avec la coloration DAPI qui ne se chevauchent pas> 50% avec l'image de la CK. Pour les catégories (2) et (5) critères spécifiques existent pour la classification CTC. Pour la catégorie (2), les articles avec CK dim / DAPI peuvent être classés comme CTC à condition que une membrane intacte peut être observée dans le canal de CK et unimage de taille appropriée DAPI est noté. Pour la catégorie (5), les articles avec CK pixélisé / DAPI ne peuvent pas être classés comme CTC cas échéant pixellisation est observée dans le canal de CK. Cependant, la pixellisation est acceptable dans le canal DAPI à condition que ce n'est pas trop grave (c.-à-image est entièrement blanc sur un fond, pas de zones grises - décrites par Janssen Diagnostics (anciennement Veridex) que la peinture blanche sur un fond noir) ou diffuse (doit apparaissent toujours de forme ovale et s'inscrire dans le CK).
User-Defined Marker Élaboration d'épreuves
Adaptation de la CSS pour caractériser CTC pour les marqueurs définis par l'utilisateur nécessite d'importants travaux avec des contrôles rigoureux et a été décrit précédemment 16. En règle générale, l'optimisation appropriée de tout marqueur défini par l'utilisateur nécessite que les contrôles négatifs être utilisés pour s'assurer que les résultats sont précis. Les meilleurs résultats sont obtenus lorsque les échantillons enrichis sont traitées à la foisun contrôle d'IgG non spécifique à la place de l'anticorps cible et avec le diluant d'anticorps seul, comme décrit précédemment. Différentes concentrations d'anticorps cible et le temps d'exposition devraient également être évalués et validés en utilisant des lignées cellulaires avec (négatifs) des densités élevées, basses, et d'absence de l'antigène. Conditions de protocole optimaux sont obtenus lorsque le test démontre à la fois une sensibilité élevée pour l'antigène cible et un faible bruit de fond non spécifique de la liaison 16.
Un exemple de ce traitement final à l'aide d'un marqueur de cellules souches du cancer, CD44, est présentée ici. Test initial avec ce marqueur a commencé à utiliser le kit standard CSS CTC (ci-après dénommé le kit traditionnelle CTC), qui utilise le canal FITC pour le développement de marqueur défini par l'utilisateur. En utilisant le kit traditionnelle CCT, il a été démontré que, après optimisation significative, le nombre maximum de CTC qui pourraient être classés comme CD44 + a été de 69,3 ± 2,67% en utilisant des échantillons enrichis avec 1000 MDA-MB-468 HUMAn cellules mammaires cancéreuses, connus pour démontrer une expression élevée de CD44 avec la majorité des cellules (98,4 ± 0,90%; comme déterminé par cytométrie en flux) exprimant cette protéine (Figure 2A). Sur la base de ces constatations, il a été émis l'hypothèse que le kit CSS CXC disponible dans le commerce peut produire de meilleurs résultats. Ce kit permet une meilleure visualisation des marqueurs avec une densité d'antigène inférieure (~ 50 000 antigènes / cellule) par rapport à la trousse traditionnel CTC (optimisé pour des marqueurs avec une densité de ~ 100 000 antigènes / cellule) en inversant le canal de fluorescence, dans lequel le CK8 / 18/19 (traditionnellement représenté dans le canal PE) et le marqueur de l'utilisateur d'intérêt (traditionnellement représenté dans le canal FITC) sont représentés (donc après le kit CXC sera dénommé kit CCT de la densité de l'antigène bas) 26. Après optimisation significative, il a été démontré que cette modification a permis d'améliorer la coloration de CD44, avec 98,8 ± 0,51% de CTC classés comme CD44 + </ Sup> en utilisant CD44-PE à une concentration de 1,0 pg / ml et un temps d'exposition de 0,6 sec (figure 2A). Optimisation appropriée de tout marqueur défini par l'utilisateur nécessite également la validation en utilisant la forte densité de l'antigène (MDA-MB-468), une faible densité de l'antigène (21 NT) et négatives (LNCaPs) des lignées cellulaires pour le marqueur d'intérêt (Figure 2B).
CTC analyse dans des modèles précliniques de souris
Afin de déterminer la sensibilité et la spécificité du protocole de CSS de souris adapté, dopés (1000 cellules cancéreuses de la prostate humaine LNCaP) et des échantillons de sang de souris non chargés ont été traitées manuellement et balayées sur l'instrument d'analyse et comparés aux résultats obtenus en utilisant la même lignée cellulaire transformée à l'aide de la norme automatisé protocole de CSS sur l'instrument de préparation (figure 3A). Comme prévu, les échantillons non dopés étaient libres de CTC à l'aide de deux essais, 0,00 ± 0,00% et la récupération de la CCT en utilisant le kit de souris adapté (90,8 ± 5,18%) n'était pas significantly différents des résultats obtenus à l'aide du système automatisé standard (86,9 ± 4,71%, p> 0,05). Les images obtenues en utilisant le protocole de souris adapté manuel ne différaient pas de celles observées en utilisant la méthode automatisée standard, à l'exception de l'addition du marqueur HLA-FITC. En outre, les cellules épithéliales squameuses de souris ne se colorent pas de façon positive pour le HLA-FITC (figure 3B). Pour confirmer que cette technique était aussi sensible que le protocole de CSS standard pour l'isolement d'un faible nombre de CTC, des dilutions en série ont été effectuées avec des échantillons de sang à pointes et la corrélation du nombre attendu de cellules par rapport au nombre récupéré de cellules ont été évaluées (figure 3C). Les résultats démontrent que les CTC pourrait effectivement être récupéré jusqu'à une sensibilité de 5 cellules/50 ul de sang total à l'aide de ce test. Ces valeurs corrélées avec les résultats escomptés avec un r 2 = 0,99.

Figure 1. CTC énumération et l'interprétation en utilisant le protocole standard CSS. (A) la récupération CTC mesurée comme un pourcentage du nombre de cellules à pointes. Les cellules ont été comptées par un hémocytomètre et ~ 1000 cellules cancéreuses de la prostate humaine LNCaP ont été mélangés dans 7,5 ml de sang humain. Des échantillons de sang humain non surchargés ont été utilisés comme témoin négatif (n = 3). Les données sont présentées sous forme de moyenne ± SEM. (B) représentatifs des images de la galerie CSS des différences de qualité CTC observé dans les échantillons de sang à pointes (c'est à dire de sang de donneur sain dopés avec des cellules tumorales de culture) par rapport à des échantillons prélevés sur les patients atteints de cancer. (C) représentant images de la galerie de CSS d'articles souvent contradictoires qui sont souvent mal classés. Carrés orange indiquent CTC acceptables, identifiés comme CK + / DAPI + / CD45-. Les images acquises au grossissement 10X objectif. Cliquez ici pour agrandir l'image.
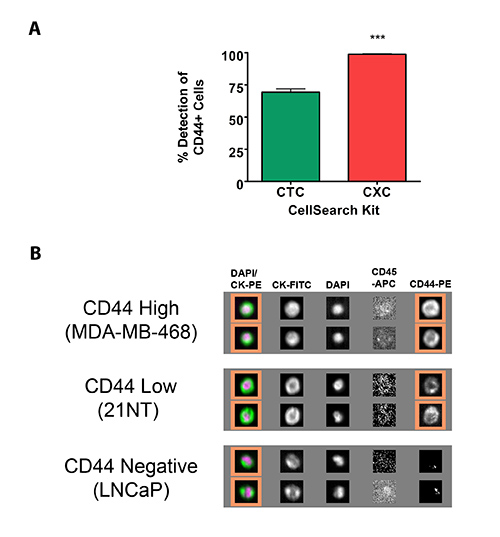
Figure 2. Caractérisation des CTC pour les marqueurs définis par l'utilisateur à l'aide du CSS. (A) Pourcentage de cellules CD44 + classés comme en utilisant les kits de CTC et CXC sur le CSS (n = 3). Les données sont présentées sous forme de moyenne ± SEM. *** = Significativement différent (P <0,0005). (B) représentatifs des images de la galerie CSS de sang provenant d'un donneur volontaire sain (7,5 ml), dopés avec ~ 1000 cellules de la lignée cellulaire identifiée, incubées avec 1,5 pg / ml d'anti -CD44-PE, et scanné à un temps d'exposition de 0,2 sec. Carrés orange indiquent CD44 + </ Sup> CTC, identifié comme CK + / DAPI + / CD45 - / CD44 -. PE + Cliquez ici pour agrandir l'image.
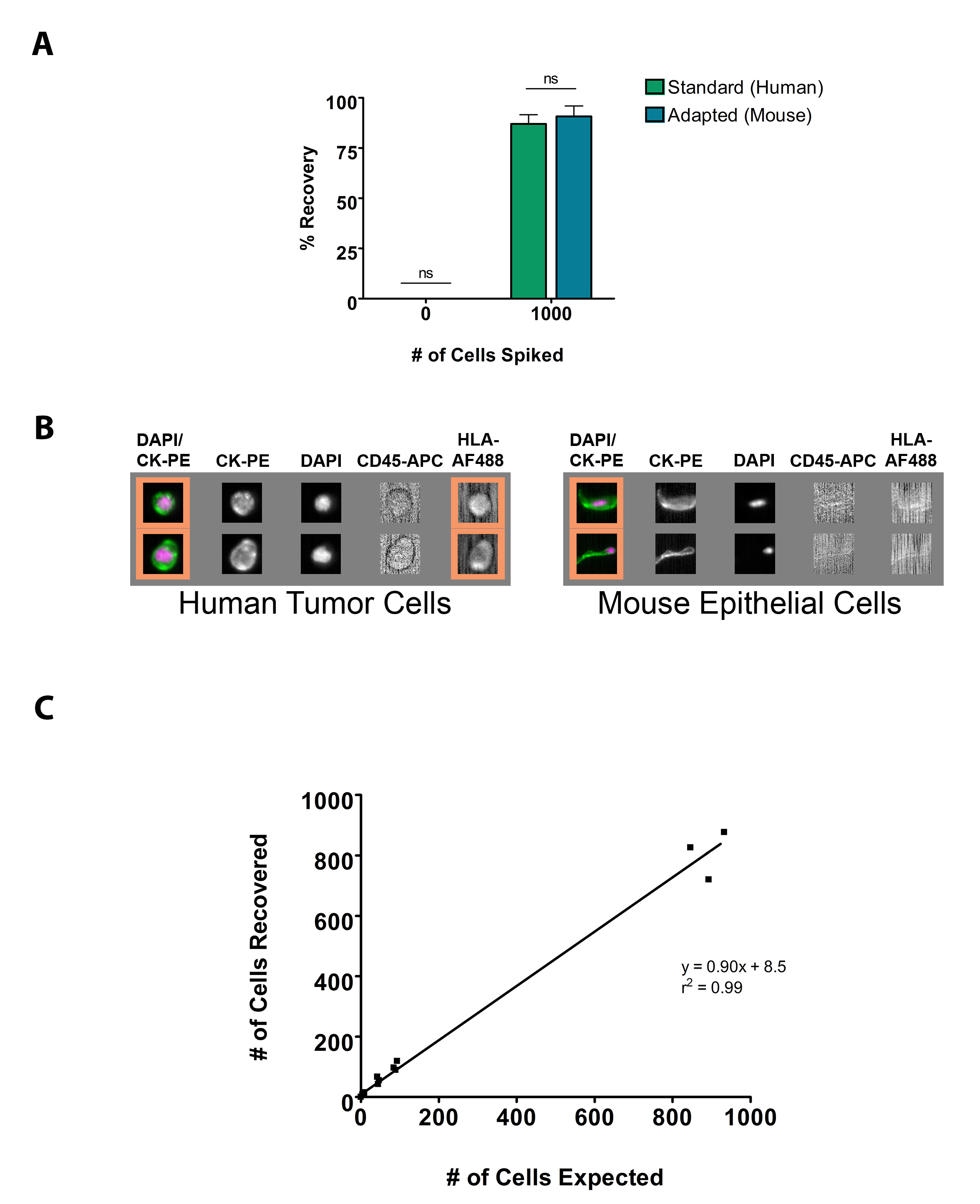
Figure 3. L'adaptation de la procédure de CSS pour une utilisation dans des modèles murins précliniques de métastases. (A) le recouvrement de CTC en utilisant le protocole de CSS de souris adapté mesuré en tant que pourcentage du nombre de cellules à pointes et comparés aux résultats obtenus en utilisant le protocole de CSS humain standard. Les cellules ont été comptées par un hémocytomètre et ~ 1000 cellules cancéreuses de la prostate humaine LNCaP ont été mélangés dans 7,5 ml de sang humain. Des échantillons de sang humain non surchargés ont été utilisés comme témoin négatif (n = 3). Les données sont présentées sous forme de moyenne ± SEM. ns = non significatif. (B) représentatifs images Galerie CSS de CTC capturé le protocole de CSS de la souris adaptée démontrer que HLA-AF488 est capable de distinguer humain à partir de cellules de souris. (C) Analyse de la corrélation et de régression linéaire de l'attendu et le nombre récupéré des cellules tumorales dopés à différentes concentrations . Des cellules de cancer de la prostate LNCaP humaines ont d'abord été comptées par un hémocytomètre et ensuite injectés dans le sang de souris à une concentration de ~ 1000 cellules/50 ul de tumeur du sang. Sang de souris Pointu a ensuite été dilué en série à une concentration de 5 tumeur ul cellules/50 et traitées en utilisant le protocole de la souris adaptée CSS (n = 3). Cliquez ici pour agrandir l'image.
| # D'échantillons avec User Defined Marker Ajouté | Total Volumoi d'ajouter à la Coupe du réactif (pl) |
| 1 | 450 |
| 2 | 600 |
| 3 | 750 |
| 4 | 900 |
| 5 | 1050 |
| 6 | 1200 |
| 7 | 1350 |
| 8 | 1500 |
Tableau 1. . Total des besoins de volume pour le CSS lors du traitement de différents nombres d'échantillons avec un marqueur défini par l'utilisateur ** Adapté de Livre blanc Veridex (disponible à l'adresse: http://www.veridex.com/pdf/CXC_Application_Guideline.PDF ).
Discussion
Malgré le développement de nombreuses technologies nouvelles de la CCT depuis l'introduction de la CSS en 2004, cette technique est encore la seule technologie cliniquement approuvé sur le marché aujourd'hui et il est donc considéré comme l'étalon-or actuel pour la détection et le dénombrement de la CCT. Ce manuscrit a démontré que, bien que le CSS a des normes de contrôle de qualité rigoureuses, il peut être sujet à des biais d'interprétation et que l'identification de la CCT dans les échantillons de patients est très différente de l'identification dans les échantillons enrichis. Six catégories d'articles souvent discordants ont été identifiées qui peuvent causer des erreurs de classification de la CCT de se produire. Ces éléments discordants soulignent la nécessité pour plusieurs réviseurs sur chaque échantillon de patient traitées sur cet instrument. En outre, les différences observées dans enrichi contre du patient obtenus CTC démontre qu'il ya une nécessité pour toutes les nouvelles technologies de la CCT pour être validés dans les deux échantillons enrichis et des patients. En outre, ces nouvelles technologies doivent be par rapport à la CSS de l'étalon-or en utilisant le test de l'échantillon scission des deux échantillons enrichis et de patients, comme la capture efficace de la CCT à partir d'échantillons enrichis seulement ne reflète pas nécessairement la CCT efficacité de capture dans les échantillons de patients.
Bien que le CSS a la capacité d'effectuer la caractérisation des CTC capturés, il est assez restreint en ce qui concerne l'optimisation hautement personnalisable. En général, les seuls paramètres qui peuvent être modifiés sur cet instrument pour l'optimisation de marqueurs définis par l'utilisateur sont la concentration d'anticorps et la durée pendant laquelle le fluorophore est exposée à la lampe à mercure. Cette capacité limitée pour l'optimisation peut présenter des problèmes lorsque vous travaillez-vous marqueurs définis par l'utilisateur sur le CSS. Une solution proposée dans ce manuscrit (décrite en détail précédemment 16) est l'utilisation de la trousse de tétrachlorure de carbone à faible densité de l'antigène qui commute le FITC et les canaux de fluorescence de PE qui permet pour une meilleure visualisation des marqueurs avec une densité de l'antigène faible. Peu importede kit qui est utilisé (densité traditionnelle ou à faible antigène kit CTC) il ya plusieurs étapes nécessaires qui doivent être prises pour assurer la sensibilité de marqueur approprié, la spécificité et l'optimisation. En premier lieu, la sensibilité du dosage doit être évaluée par rapport à une méthode bien validée, comme la cytométrie de flux, qui va permettre de déterminer le niveau de détection prévue (à savoir le pourcentage de cellules dans la population de cellules qui expriment le marqueur d'intérêt) de l'utilisateur, marqueur défini. Deuxièmement, le test doit être évalué pour sa capacité à détecter le marqueur d'intérêt dans des lignées cellulaires avec différents niveaux d'expression (c. densités antigène haut et bas) et sa spécificité doit être validé dans une lignée cellulaire qui est négatif pour le marqueur d'intérêt . Dans tous les cas, toutes les lignées cellulaires doivent être testés à l'aide d'une seule commande de cellules (pas d'anticorps ajouté), le contrôle appropriée des IgG, et l'anticorps d'intérêt à diverses concentrations et les durées d'exposition pour déterminer le most paramètres appropriés qui assureront l'optimisation du marqueur défini par l'utilisateur. Toutefois, il convient de noter que, bien que la caractérisation de CTC est disponible sur le CSS, actuellement seulement un marqueur défini par l'utilisateur d'intérêt peut être exploré dans chaque échantillon, et que le système est très limité en ce qui concerne les applications en aval en raison de la transformation dure des échantillons.
L'approche chevet à paillasse uniques utilisés dans la recherche de la CCT a permis une entrée rapide de ce test utile dans le cadre clinique. Cependant, il a donné lieu à une mauvaise compréhension de la biologie fondamentale de ces cellules rares. Par conséquent, le développement et l'optimisation des dosages qui permettent une évaluation préclinique de CTC dans des modèles murins in vivo de cancer sont nécessaires. Ce manuscrit décrit un protocole CSS adapté qui permet de CTC à évaluer dans des volumes de 50 ul de sang de souris, idéalement adapté pour la collection de série CTC modèles expérimentaux. Ce manuscrit demonstrates que le recensement de la CCT en utilisant ce protocole est comparable aux résultats obtenus en utilisant le CSS en combinaison avec le kit traditionnelle CTC, avec aucune différence significative dans la CCT efficacité de capture entre les techniques de séparation automatisées et manuelles. En outre, au cours de l'élaboration de ce test, il a été reconnu, comme précédemment décrit dans la littérature 25, qui épidermoïde contamination des cellules épithéliales de la souris peut effectuer l'identification précise des CTC plus difficile et parfois impossible. Par conséquent, pour lutter contre ce problème une définie par l'utilisateur HLA-Alexa Fluor 488 a été ajouté à ce protocole pour garantir que seules les cellules humaines (CK + DAPI + CD45 - HLA - Alexa Fluor 488 +) sont affectées de manière appropriée que les CTC. Il est important de noter que la lignée cellulaire LNCaP utilisée dans ce manuscrit est donc HLAlow et HLA-Alexa Fluor 488 peut avoir besoin d'être titrée pour les lignées cellulaires avec variant expression de HLA. Bien que nous avons ajouté un Fluo HLA-Alexar 488 à notre protocole pour assurer l'identification précise de CTC, il convient de noter que la grande majorité des cellules épithéliales squameuses de souris étaient facilement identifiables par la morphologie et ont été limités dans le nombre de cellules (0,33 ± 0,24 events/50 pi, n = 9). Le nombre de cellules plus élevées (jusqu'à 11 events/50 ul) n'ont été observés lorsque la collecte de sang (par ponction cardiaque) s'est avéré difficile et les tentatives répétées sont nécessaires. Par conséquent, nous proposons que, si on le souhaite, le système de caractérisation de CTC pourrait être réalisée en supprimant cette borne. En outre, bien que n'étant pas décrite ici, nous nous attendons à ce que la collecte des CTC et une caractérisation plus poussée en aval du CTC à l'aide d'autres méthodes pourraient être facilement réalisé comme précédemment démontré en utilisant le CSS 27,28.
Bien que le CSS a été utilisé en clinique pour détecter efficacement les CTC dans le sang du cancer du sein métastatique, de la prostate et le cancer colorectal 4,10,29, il ne ont plusieurs limitations. Dans jusqu'à 35% des patients atteints de divers cancers métastatiques, les CTC sont indétectables en dépit de la présence d'une maladie systémique généralisée 30. Cette absence de détection a été proposé pour être à la suite de la transition épithéliale-mésenchymateuse, un processus bien documenté connu pour améliorer l'invasion de cancer, les métastases, l'agressivité et l'ensemble 31. Cette transition a été associée à une réduction significative des marqueurs épithéliaux, tels que EpCAM, et une augmentation correspondante dans les marqueurs 32 mésenchymateuses. Plusieurs études ont récemment démontré que la présence de ces marqueurs mésenchymateuses dans les CTC sont indicatifs de moins bon pronostic et que beaucoup de ces cellules manquent expression de marqueurs épithéliaux qui seraient nécessaires à leur détection en utilisant le CSS 24,33-38. Ceci suggère que la définition de la norme CSS peut-être manquant un peu de CTC les plus agressives.
En dépit des limitations décrites, il est prévu èmeprotocoles e décrits dans ce manuscrit seront des outils importants pour l'amélioration de l'analyse de la CCT en utilisant le CSS, le développement de nouvelles technologies de la CCT, l'optimisation des marqueurs définis par l'utilisateur, et une meilleure compréhension de la biologie de la CCT en utilisant des modèles de souris précliniques in vivo. Pris ensemble, ces protocoles fourniront une ressource utile pour les utilisateurs de cette plate-forme intéressé dans la recherche préclinique et clinique relative à la métastase et CTC.
Déclarations de divulgation
L'auteur (ALA) a reçu un financement qui a été fourni par Janssen oncologie, dont la société mère Johnson & Johnson possède également Janssen Diagnostics LLC, qui produit des réactifs et des instruments utilisés dans cet article.
Remerciements
Ce travail a été soutenu par des subventions de l'Institut ontarien de recherche sur le cancer (# 08NOV230), la Fondation canadienne pour l'innovation (# 13199), Cancer de la Prostate Canada, Janssen-oncologie, le London Regional Cancer Program, et le soutien des bailleurs de fonds de John et Donna Bristol grâce la London Health Sciences Foundation (ALA). LIE est soutenu par une Frederick Banting et Charles Best canadienne des bourses d'études supérieures au doctorat. ALA est soutenu par une bourse de nouveau chercheur des IRSC et une bourse de nouveau chercheur du ministère de la Recherche et de l'Innovation de l'Ontario.
matériels
| Name | Company | Catalog Number | Comments |
| 0.5 M EDTA | |||
| Anti-human CD44-FITC | BD Pharmigen | 555478 | |
| Anti-human CD44-PE | BD Pharmigen | 555479 | |
| Anti-human HLA-Alexa Fluor 488 | BioLegend | 311415 | |
| Anti-mouse CD45-APC | eBioscience | 17-0451-82 | |
| Bond Primary Antibody Diluent | Leica | AR9352 | |
| CellSave Preservative Tubes | Veridex | 952820 (20 pack) 79100005 (100 pack) | |
| CellSearch CTC Control Kit | Veridex | 7900003 | |
| CellSearch CTC Kit | Veridex | 7900001 | |
| CellSearch CXC Control Kit | Veridex | 7900018RUO | |
| CellSearch CXC Kit | Veridex | 7900017RUO | |
| Instrument Buffer | Veridex | 7901003 | |
| Streck Cell Preservative (aka CytoChex) | Streck | 213350 | |
| 1 ml Syringe | |||
| 10 ml Serological pipette | |||
| 1,000 µl Pipette | |||
| 1,000 µl Pipette tips | |||
| 12 mm x 75 mm Flow tubes | |||
| 200 µl Gel loading tips | |||
| 200 µl Pipette | |||
| 22 G Needle | |||
| 5 3/4 in Disposible Pasteur pipet | VWR | 14672-200 | |
| 5 ml Serological pipette | |||
| Automated pipettor | |||
| Capillary Blood Collection Tube (EDTA) | BD Microtainer | 365974 | |
| CellSearch Analyzer II | Veridex | 9555 | Includes magnests and verification cartirdges |
| CellSearch AutoPrep System | Veridex | 9541 | |
| Centrifuge | |||
| MagCellect Magnet | R&D Systems | MAG997 | |
| Small Latex Bulb | VWR | 82024-550 | |
| Vortex |
Références
- Siegel, R., Naishadham, D., Jemal, A. Cancer statistics. 63 (1), 11-30 (2013).
- Chambers, A. F., Groom, A. C., MacDonald, I. C. Dissemination and growth of cancer cells in metastatic sites. Nat. Rev. Cancer. 2 (8), 563-572 (2002).
- Pantel, K., Brakenhoff, R. H. Dissecting the metastatic cascade. Nat. Rev. Cancer. 4 (6), 448-456 (2004).
- Cristofanilli, M., Budd, G. T., et al. Circulating tumor cells, disease progression, and survival in metastatic breast cancer. New Engl. J. Med. 351 (8), 781-791 (2004).
- De Bono, J. S., Scher, H. I., et al. Circulating tumor cells predict survival benefit from treatment in metastatic castration-resistant prostate cancer. Clin. Cancer Res. 14 (19), 6302-6309 (2008).
- Cohen, S. J., Ja Punt, C., et al. Prognostic significance of circulating tumor cells in patients with metastatic colorectal cancer. Ann. Oncol. 20 (7), 1223-1229 (2009).
- Hayes, D. F., Cristofanilli, M., et al. Circulating tumor cells at each follow-up time point during therapy of metastatic breast cancer patients predict progression-free and overall survival. Clin. Cancer Res. 12 (14), 4218-4224 (2006).
- Budd, G. T., Cristofanilli, M., et al. Circulating tumor cells versus imaging--predicting overall survival in metastatic breast cancer. Clin. Cancer Res. 12 (21), 6403-6409 (2006).
- Olmos, D., Arkenau, H. -. T., et al. Circulating tumour cell (CTC) counts as intermediate end points in castration-resistant prostate cancer (CRPC): a single-centre experience. Ann. Oncol. 20 (1), 27-33 (2009).
- De Bono, J. S., Scher, H. I., et al. Circulating tumor cells predict survival benefit from treatment in metastatic castration-resistant prostate cancer. Clin. Cancer Res. 14 (19), 6302-6309 (2008).
- Allan, A. L., Keeney, M. Circulating tumor cell analysis: technical and statistical considerations for application to the clinic. J. Oncol. 2010, 426218 (2010).
- Lowes, L. E., Goodale, D., Keeney, M., Allan, A. L. Image cytometry analysis of circulating tumor cells. Methods Cell Biol. 102, 261-290 (2011).
- Lianidou, E. S., Markou, A. Circulating tumor cells in breast cancer: detection systems, molecular characterization, and future challenges. Clin. Chem. 57 (9), 1242-1255 (2011).
- Alix-Panabières, C., Pantel, K. Circulating tumor cells: liquid biopsy of cancer. Clin. Chem. 59 (1), 110-118 (2013).
- Parkinson, D. R., Dracopoli, N., et al. Considerations in the development of circulating tumor cell technology for clinical use. J. Transl. Med. 10, 138 (2012).
- Lowes, L. E., Hedley, B. D., Keeney, M., Allan, A. L. User-defined protein marker assay development for characterization of circulating tumor cells using the CellSearch system. Cytomet. A. 81 (11), 983-995 (2012).
- Fehm, T., Müller, V., et al. HER2 status of circulating tumor cells in patients with metastatic breast cancer: a prospective, multicenter trial. Breast Cancer Res. Treat. 124 (2), 403-412 (2010).
- Liu, Y., Liu, Q., et al. Circulating tumor cells in HER2-positive metastatic breast cancer patients: a valuable prognostic and predictive biomarker. BMC Cancer. 13, 202 (2013).
- Slamon, D. J., Leyland-Jones, B., et al. Use of chemotherapy plus a monoclonal antibody against HER2 for metastatic breast cancer that overexpresses HER2. New Engl. J. Med. 344 (11), 783-792 (2001).
- Marty, M., Cognetti, F., et al. Randomized phase II trial of the efficacy and safety of trastuzumab combined with docetaxel in patients with human epidermal growth factor receptor 2-positive metastatic breast cancer administered as first-line treatment: the M77001 study group. J. Clin. Oncol. 23 (19), 4265-4274 (2005).
- Slamon, D., Eiermann, W., et al. Adjuvant trastuzumab in HER2-positive breast cancer. New Engl. J. Med. 365 (14), 1273-1283 (2011).
- Welch, D. R. Technical considerations for studying cancer metastasis in vivo. Clin. Exp. Metast. 15 (3), 272-306 (1997).
- Goodale, D., Phay, C., Postenka, C. O., Keeney, M., Allan, A. L. Characterization of tumor cell dissemination patterns in preclinical models of cancer metastasis using flow cytometry and laser scanning cytometry. Cytometry A. 75 (4), 344-355 (2009).
- Gorges, T. M., Tinhofer, I., et al. Circulating tumour cells escape from EpCAM-based detection due to epithelial-to-mesenchymal transition. BMC Cancer. 12, 178 (2012).
- Eliane, J. -. P., Repollet, M., et al. Monitoring serial changes in circulating human breast cancer cells in murine xenograft models. Cancer Res. 68 (14), 5529-5532 (2008).
- Flores, L. M., Kindelberger, D. W., et al. Improving the yield of circulating tumour cells facilitates molecular characterisation and recognition of discordant HER2 amplification in breast. 102 (10), 1495-1502 (2010).
- Sieuwerts, A. M., Kraan, J., et al. Molecular characterization of circulating tumor cells in large quantities of contaminating leukocytes by a multiplex real-time PCR. Breast Cancer Res. Treat. 118 (3), 455-468 (2009).
- Cohen, S. J., Ja Punt, C., et al. Relationship of circulating tumor cells to tumor response, progression-free survival, and overall survival in patients with metastatic colorectal cancer. J. Clin. Oncol. 26 (19), 3213-3221 (2008).
- Allard, W. J., Matera, J., et al. Tumor cells circulate in the peripheral blood of all major carcinomas but not in healthy subjects or patients with nonmalignant diseases. Clin. Cancer Res. 10 (20), 6897-6904 (2004).
- Thiery, J. P., Acloque, H., Huang, R. Y. J., Nieto, M. A. Epithelial-mesenchymal transitions in development and disease. Cell. 139 (5), 871-890 (2009).
- Yang, J., Weinberg, R. A Epithelial-mesenchymal transition: at the crossroads of development and tumor metastasis. Dev. Cell. 14 (6), 818-829 (2008).
- Gradilone, A., Raimondi, C., et al. Circulating tumour cells lacking cytokeratin in breast cancer: the importance of being mesenchymal. J. Cell. Mol. Med. 15 (5), 1066-1070 (2011).
- Königsberg, R., Obermayr, E., et al. Detection of EpCAM positive and negative circulating tumor cells in metastatic breast cancer patients. Acta Oncol. 50 (5), 700-710 (2011).
- Kasimir-Bauer, S., Hoffmann, O., Wallwiener, D., Kimmig, R., Fehm, T. Expression of stem cell and epithelial-mesenchymal transition markers in primary breast cancer patients with circulating tumor cells. Breast Cancer Res. 14 (1), (2012).
- Mego, M., Mani, S. A., et al. Expression of epithelial-mesenchymal transition-inducing transcription factors in primary breast cancer: The effect of neoadjuvant therapy. Int. J. Cancer. 130 (4), 808-816 (2012).
- Yu, M., Bardia, A., et al. Circulating breast tumor cells exhibit dynamic changes in epithelial and mesenchymal composition. Science. 339 (6119), 580-584 (2013).
- Armstrong, A. J., Marengo, M. S., et al. Circulating tumor cells from patients with advanced prostate and breast cancer display both epithelial and mesenchymal markers. Mol. Cancer Res. 9 (8), 997-1007 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon