RNA-Analyse von Umweltproben mittels RT-PCR
Überblick
Quelle: Labors von Dr. Ian Pfeffer und Dr. Charles Gerba - Arizona University
Demonstrierende Autor: Bradley Schmitz
Reverse Transkription-Polymerase-Kettenreaktion (RT-PCR) beinhaltet den gleichen Prozess wie konventionelle PCR-Radsport Temperatur um Nukleinsäuren. Jedoch während konventionelle PCR nur Deoxyribonucleic Säuren (DNA) verstärkt, ermöglicht RT-PCR die Verstärkung der Säuren Ribonukleinsäure (RNA) durch die Bildung von komplementären DNA (cDNA). Dies ermöglicht RNA-basierten Organismen gefunden in der Umgebung zu analysierten nutzt Methoden und Technologien, die für DNA bestimmt werden.
Viele in der Umgebung gefundenen Viren verwenden RNA als ihr genetisches Material. Verschiedene RNA-basierten virale Erreger, wie Norovirusund Indikator-Organismen, wie z. B. Paprika mild Mottle Virus (PMMoV), haben keine Kultur-basierte Nachweisverfahren für die Quantifizierung. Um auf das Vorhandensein dieser RNA-Viren in Umweltproben aus Erde, Wasser, Landwirtschaft, etc.zu erkennen, setzen molekulare Tests auf RT-PCR, RNA in DNA umzuwandeln. Ohne RT-PCR wäre Mikrobiologen nicht in der Lage, Tests und Forschung zahlreiche RNA-basierten Viren, die Risiken für die Gesundheit von Mensch und Umwelt darstellen.
RT-PCR kann auch als Werkzeug eingesetzt werden, um mikrobielle Aktivität in die Umwelt zu messen. Boten-RNA (mRNA) ist die einsträngige Vorlage für Protein Übersetzung, und messen die Höhe der verschiedenen mRNAs gibt an, welche Gene aus der Mikroben in der Umgebung zum Ausdruck gebracht werden. Analyse der Genexpression gibt Hinweise, welche biologischen Bahnen von Organismen verwendet werden, um unter verschiedenen Umweltbedingungen zu überleben. In einigen Fällen kann Genexpression genutzt werden, um festzustellen, welche Organismen können überleben unter widrigen Bedingungen am besten und haben Fähigkeiten für Bioremediation von kontaminiertem Erdreich oder Wasser.
Grundsätze
PCR basiert auf die Verstärkung von DNA-Templates, die seine Nutzung bei der Aufdeckung von RNA aus Organismen beschränkt. RT-PCR bietet jedoch ein Mittel für die Verwendung von RNA, um cDNA mit Hilfe von spezialisierten Enzymen, bekannt als Reverse Transkriptase (RT) zu produzieren. Diese cDNA kann dann als Ausgangspunkt Vorlage für spätere Verstärkung mit konventionellen PCR (Abbildung 1) verwendet werden.
Reversen Transkription kann gesteuert werden, verstärken nur die gewünschten Produkte oder eine ganze Gemeinschaft von Nukleinsäuren, die innerhalb einer ökologischen Probe je nach die Primer, die zur Vorlage der cDNA-Synthesis. Dies ist wichtig, da Boden- und Wasserproben sind oft mit verschiedenen Nukleinsäuren, die für spezifische Auswertungen gewünscht sind nicht gesättigt. Zufällige Zündkapseln, die RNA-Sequenzen gefunden in jeder Art von Mikroben binden können, können in RT-PCR verwendet werden, um die meisten RNA zu erkennen, so dass die Probe für das Vorhandensein und die relative Häufigkeit von mehreren Organismen in der Umwelt analysiert werden kann. Auf der anderen Seite initiiert Sequenz-spezifische Primer cDNA Synthese für exakte Sequenzen in nur einem oder wenigen Organismen gefunden. Dies ermöglicht eine ökologische Probe für einen bestimmten Zweck, wie die Bestimmung getestet werden, ob Norovirus, die Magen-Darm-Erkrankungen beim Menschen verursachen können, im Wasser vorhanden ist.

Abbildung 1. Schritt für Schritt Prozess der RT-PCR-Analyse von Umweltproben RNA.
Verfahren
(1) Entnahme von Proben: Bodenprobe
- Suchen Sie eine Probe Position über GPS-Koordinaten oder aus den Augen.
- Wählen Sie für zufällige Stichproben Zufallspunkte innerhalb eines Bereichs um eine allgemeine Volkszählung mikrobielle Habitate zu erhalten. Transekt Sampling sammelt von Punkten entlang einer geraden Linie, z.B.neben einem Bachbett. Raster-Proben sind systematisch Punkte in regelmäßigen Abständen entnommen und eignen sich für Zuordnung mikrobieller Gemeinschaften in einem Gebiet, unbekannt oder variabel.
- Probenahme in einem 3 bis 6 Zoll (7,5-15 cm) bietet tiefe Zugriff auf die am häufigsten vorkommende mikrobielle Aktivität, die in der Nähe, aber nicht in der Rhizosphäre (die schmale Region des Bodens direkt betroffen von Pflanzenwurzeln und ihre damit verbundenen Mikroorganismen) ist.
- Um die Bodenprobe zu erfassen, drücken Sie und drehen Sie eine Hand-Schnecke in den Boden auf die vorgegebene Tiefe.
- Heben Sie die Schnecke. Der Boden wird innerhalb der hohlen Stiel der Schnecke gefunden.
- Kratzen Sie den Boden an der Unterseite der Schnecke in eine Boden-Fangsack. Achten Sie darauf, nicht zu berühren oder den Boden zu verunreinigen.
- Beschriften Sie die Tasche richtig mit Standort, Name, Datum und Uhrzeit.
- Durchlaufen Sie im Labor den Boden eine 2-mm-Sieb um Kies und Felsen zu entfernen.
- Einen Teil des Bodens für Bodenfeuchte zu analysieren. Einzelheiten zu diesem Schritt siehe Jupiter Science Education video auf Bodenfeuchte.
2. Beispiel-Sammlung: Wasserprobe
- Finden Sie den Probe-Standort per GPS, Koordinaten oder aus den Augen.
- Sammeln Sie das Wasser in einer Vorratsflasche. Das Volumen des aufgefangenen Wassers zu erfassen; Wenn Mikroben in der Probe quantitativ untersucht werden, kann die mikrobielle Konzentration basierend auf den gesammelten Volumen ermittelt werden.
- Testen Sie sofort das Wasser für alle Parameter erforderlich für das Experiment (Temperatur, pH-Wert, Leitfähigkeit, Salzgehalt, Stickstoff und Phosphor Inhalt, etc.) mit der entsprechenden elektronischen Sonden.
- Stellen Sie die Flasche mit der Wasserprobe in eine Kühlbox mit Eis. Übertragen Sie den Kühler an das Labor.
(3) RNA-Extraktion
- Zu sammeln und Mikroorganismen aus der Umwelt Probe zu konzentrieren, entnehmen Sie bitte dem Jupiter Science Education video-on-Gemeinschaft Nukleinsäure-Extraktion.
- Viren mit einem kommerziellen Extraktion-Kit nach Herstellerangaben extrahieren Sie RNA.
- Kurz, zunächst die Proben mit Lyse Puffer ergänzt mit Ethanol mischen, dann die Proben in Spin Spalten hinzufügen.
- Zentrifugieren Sie die Spalten bei ca. 12.000 x g, verwerfen Bestrahlungskammer. Die Spalten Waschpuffer hinzufügen und erneut zentrifugieren.
- RNase-freies Wasser hinzufügen, um die Spalten und die Zentrifuge für 30 s an RNA in sterilen 1,5 mL niedrigem Kraftschlußbeiwert Microfuge Röhren eluieren.
(4) reverse Transkription - PCR
- Die folgenden Reagenzien von-20 ° C-Gefrierschrank abrufen: dNTP, konzentriert (z.B. 10 X) reverse Transkription Puffer, Primer (in diesem Beispiel, zufällige Primer). Tauen Sie diese auf Eis oder bei Raumtemperatur innerhalb eine cleane Motorhaube, und halten sie auf dem Eis einmal aufgetaut. Auch abgerufen Sie die Reverse Transkriptase und RNase-Inhibitor und halten sie auf dem Eis.
- Die Reagenz Volumina erforderlich, um eine "master-Mix", die alle Reagenzien verbindet konstant unter jede Reaktion zu berechnen (siehe Tabelle 1 für eine Probe-Reaktion). Bereiten Sie genügend master-Mix für jede Probe sowie eine Positivkontrolle (mit einer bekannten Protokoll-Vorlage) und negative (z.B.ohne Reverse Transkriptase, mit nur Wasser als Vorlage, etc.) Reaktionen zu kontrollieren. Enthalten Sie 10 % extra Volumen.
- Montieren Sie den master-Mix in 1,5 mL niedrigem Kraftschlußbeiwert Microfuge Röhren. Dadurch wird die Bindung von Reagenz-Molekülen, die Rohre Kunststoffwände minimiert.
- Wenn Reagenzien aufgetaut sind, fügen Sie berechnete Volumen das Microfuge Rohr hinzu. Sanft Wirbel und Zentrifugieren jedes Röhrchen vor der Addition. Achten Sie darauf, die Pipette Spitzenwechsel zwischen dem Hinzufügen jedes Reagens um Verunreinigungen zu verhindern.
- Nachdem alle Reagenzien sind hinzugekommen, Wirbel und Zentrifuge der master-Mix um eine homogene Mischung zu gewährleisten. Reagenzien in Lagerung bei-20 ° c zurückgestellt
- Bereiten Sie vor und beschriften Sie 8-Röhre PCR-Streifen, eine Röhre für jede Probe oder Kontrolle Reaktion benannt.
- Aliquoten ein gleiches Volumen des master-Mix in jedes Rohr. Fügen Sie dann die Reaktion-spezifische Komponenten, wie z. B. die RNA-Extrakte.
- Legen Sie die Streifen Kappe sicher auf den PCR-Streifen und Zentrifugieren in einem Minicentrifuge mit einem Streifen-Rohr-Adapter um sicherzustellen, alle Flüssigkeit am unteren Rand jedes Rohr (Abbildung 2) gesammelt werden.
- Ort-PCR-Streifen sicher im Thermocycler. Drücken Sie, um sicherzustellen, dass die Rohre befestigt sind.
- Festlegen der Thermocycler zum Ausführen des Programms für die RT verwendet wird (siehe Tabelle 2 für ein Probe-Protokoll). Wenn das Programm abgeschlossen ist, werden die Rohre cDNA Produkte enthalten, die dann PCR Verstärkung unterworfen werden können. Bitte Einzelheiten Sie zu den Jupiter Science Education Videos auf PCR und quantitative PCR. CDNA bei-20 ° C bis zu seiner Verwendung zu speichern.

Abbildung 2. Angeschnittene Ärmel 8-Röhre Streifen mit master-Mix und Extrakt.
| Reagenz | Volumen pro 1 Reaktion (μL) |
| 10 x RT Buffer | 2.0 |
| 25 x dNTPs | 0,8 |
| 10 x zufällige Primer | 2.0 |
| Multiscribe | 1.0 |
| RNase-Inhibitor | 1.0 |
| Molekularen Klasse H2O | 3.2 |
| Gesamtvolumen | 10 |
Tabelle 1. RT-Master Mix Zutaten.
| Schritt 1 | Schritt 2 | Schritt 3 | Schritt 4 |
| 25 ° C, 10 min | 37 ° C, 120 min. | 85 ° C, 5 min | 4 ° C, ∞ |
Tabelle 2. RT-Reaktion-Thermocycler-Programm.
Ergebnisse
Wenn die RT - PCR abgeschlossen ist, können einige das PCR-Produkt getrennt und visualisiert auf einer Agarosegel (Abbildung 3). In diesem Beispiel wurde eine Gen-spezifische Primer verwendet, um auf das Vorhandensein eines RNA-Virus zu erkennen. Bands die erwartete Größe werden von zwei der Proben und die Positivkontrolle Reaktion, aber nicht von der negativen Kontrolle, auf das Vorhandensein des Virus in zwei der getesteten Wasserproben gewonnen.
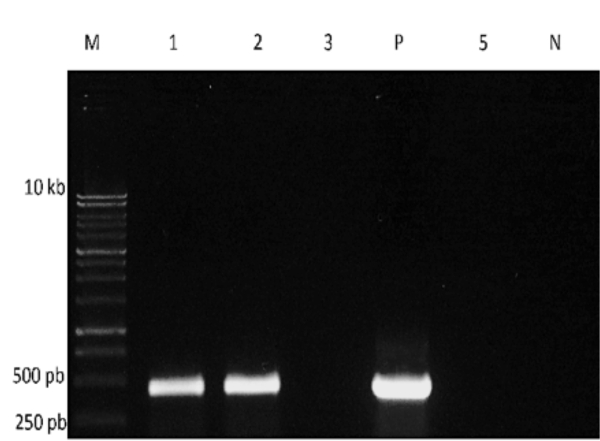
Abbildung 3. Gelelektrophorese von RT-PCR-Produkten. M: DNA Größe Marker; P: Positivkontrolle; N: Negativkontrolle. Reaktionen mit RNA aus vier Wasserproben wurden in Bahnen 1, 2, 3 und 5 laufen.
Anwendung und Zusammenfassung
RT-PCR ist notwendig für die Erstellung von cDNA aus einer RNA-Vorlage. Dies ermöglicht RNA-basierten Mikroorganismen analysierten Nutzung molekularer Assays für DNA entwickelt werden. Sobald die cDNA synthetisiert wird, bestimmen das Vorhandensein oder Fehlen von RNA-basierten Mikroorganismen innerhalb einer ökologischen Probe PCR-Assays. Dies ermöglicht weitere nachgelagerte Analyse zur Bestimmung der mikrobiellen Ökologie, Gesundheitsrisiken und Umweltrisiken.
RT-PCR kann auch genutzt werden, um assay mRNA als Mittel zu beobachten, welche Gene in einer Umgebung zum Ausdruck gebracht werden. Dies gibt Aufschluss darüber, welche Proteine und Wege Mikroben auf verlassen, um insbesondere Umweltbedingungen zu überleben. Genexpressionsanalysen können mikrobielle Wege dieser Aufschlüsselung Umweltkontaminanten wie Kohlenwasserstoffe oder chlorierten identifizieren und Mikroben mit diese Wege können für Bioremediation nutzbar gemacht werden.
Risikobewertung beinhaltet RT-PCR um Risiken für die Gesundheit von Mensch und Umwelt zu analysieren. Kombination von RT mit quantitativer PCR ermöglicht RNA-Viren innerhalb von Proben, aufgelistet werden sollen, so dass Exposition von Mensch und Umwelt für die Zwecke der quantitativen mikrobielle Risiko Bewertung (QMRA) berechnet werden kann.
pringen zu...
Videos aus dieser Sammlung:

Now Playing
RNA-Analyse von Umweltproben mittels RT-PCR
Environmental Microbiology
40.4K Ansichten

Bestimmung des Feuchtigkeitsgehalts im Boden
Environmental Microbiology
359.7K Ansichten

Aseptische Techniken in den Umweltwissenschaften
Environmental Microbiology
126.5K Ansichten

Gramfärbung von Bakterien aus Umweltquellen
Environmental Microbiology
100.4K Ansichten

Visualisierung von Bodenmikroorganismen mit dem Contact Slide Assay und Mikroskopie
Environmental Microbiology
42.4K Ansichten

Filamentöse Pilze
Environmental Microbiology
57.4K Ansichten

Extraktion von DNA aus Bakterienkolonien
Environmental Microbiology
28.9K Ansichten

Erkennen von ökologischen Mikroorganismen mit der Polymerase-Kettenreaktion und Gelelektrophorese
Environmental Microbiology
44.6K Ansichten

Quantifizierung der ökologischen Mikroorganismen und Viren mit qPCR
Environmental Microbiology
47.9K Ansichten

Wasserqualitätsanalyse über Indikatororganismen
Environmental Microbiology
29.6K Ansichten

Isolierung von fäkalen Bakterien aus Wasserproben durch Filtration
Environmental Microbiology
39.4K Ansichten

Erkennung von Bakteriophagen in Umweltproben
Environmental Microbiology
40.8K Ansichten

Kultivierung und Auflisten von Bakterien aus Bodenproben
Environmental Microbiology
184.7K Ansichten

Analyse der bakteriellen Wachstumskurve und ihre Umweltanwendungen
Environmental Microbiology
296.2K Ansichten

Algenzählung mittels kultivierbarer Methodik
Environmental Microbiology
13.8K Ansichten
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten