Method Article
Chemogenetische Regulation in reprogrammierten Stammzell-abgeleiteten Vorläuferzellen bei der Behandlung neurodegenerativer Erkrankungen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll für die Entwicklung chemisch umprogrammierter Stammzellen, um eine präzise neuronale Modulation zu erreichen, indem diese Zellen in dopaminerge Vorläuferzellen differenziert, in Mausmodelle der Parkinson-Krankheit transplantiert und verhaltensbezogene und elektrophysiologische Ergebnisse ausgewertet werden, um die erfolgreiche Integration und funktionelle Wirksamkeit der transplantierten Zellen zu bestätigen.
Zusammenfassung
Die Integration von Designer-Rezeptoren, die ausschließlich durch Designerdrogen aktiviert werden (DREADD), mit stammzellbasierten Therapien stellt eine fortschrittliche Strategie zur präzisen neuronalen Modulation dar. Hier verwendeten wir CRISPR-technisch veränderte humane Stammzellen, die exzitatorische (hM3Dq) oder inhibitorische (hM4Di) DREADD-Rezeptoren exprimieren, um die funktionelle Integration und Modulation von transplantierten dopaminergen Vorläufern in einem Mausmodell der Parkinson-Krankheit (PD) zu bewerten. Zu den wichtigsten Schritten gehörten die Generierung von nicht-fusionellen DREADD-exprimierenden Stammzelllinien, deren Differenzierung in dopaminerge Vorläufer des Mittelhirns und die Transplantation dieser Zellen in das Striatum von 6-Hydroxydopamin (6-OHDA)-läsionierten Mäusen. Wir führten Verhaltensbewertungen und elektrophysiologische Aufzeichnungen durch, um die Auswirkungen der transplantierten Zellen zu analysieren. Verhaltenstests, wie z. B. der Zylindertest, zeigten eine signifikante Modulation der motorischen Funktion nach Verabreichung von Clozapin-N-oxid (CNO). Insbesondere reduzierte die Aktivierung von hM4Di die kontralaterale Bewegung der Vordergliedmaßen, während die Aktivierung von hM3Dq mit einem verbesserten motorischen Verhalten verbunden war. Elektrophysiologische Ableitungen zeigten deutliche synaptische Reaktionen. Die hM4Di-Aktivierung erhöhte die Ereignisintervalle und verringerte die Spitzenamplituden spontaner exzitatorischer postsynaptischer Ströme (sEPSCs), während die hM3Dq-Aktivierung die Ereignisintervalle verringerte und die Spitzenamplituden erhöhte, was eine verstärkte exzitatorische Signalübertragung widerspiegelt. Zusammenfassend lässt sich sagen, dass die Integration von Verhaltens- und elektrophysiologischen Bewertungen den präzisen funktionellen Einbau von chemisch umprogrammierten Stammzellen in die neuronalen Schaltkreise des Wirts validiert.
Einleitung
Designer-Rezeptoren, die ausschließlich durch Designerdrogen (DREADD) aktiviert werden, sind technisch hergestellte G-Protein-gekoppelte Rezeptoren, die selektiv durch ansonsten inerte synthetische Liganden aktiviert werden können1. Der chemogenetische Ansatz ist zu einem unverzichtbaren Werkzeug in den Neurowissenschaften geworden, da er es Forschern ermöglicht, die Konnektivität neuronaler Schaltkreise mit hoher Präzision zu untersuchen und unser Verständnis der zellulären Funktionen sowohl in vivo als auch in vitro durch die selektive Aktivierung oder Hemmung bestimmter Gehirnregionen oder Zelltypen zu verbessern 2,3.
Die stammzellbasierte Therapie stellt eine vielversprechende Strategie zur Behandlung neurodegenerativer Erkrankungen dar. Die Wirksamkeit von Transplantatzellen hängt von der richtigen Integration, dem Überleben und dem funktionellen Beitrag zum Wirtsgewebe ab. Unkontrollierte zelluläre Aktivität kann zu negativen Folgen führen, einschließlich Tumorgenese4; Dies erfordert eine präzise Kontrolle dieser Zellen nach der Transplantation. Die Nutzung der DREADD-Technologie in menschlichen reprogrammierten Stammzellen und abgeleiteten Neuronen bietet die Möglichkeit, die neuronale Aktivität durch die Verabreichung des Designermedikaments CNO 2,5 präzise zu steuern. Im Zusammenhang mit der Parkinson-Krankheit (PD), die durch den Verlust dopaminerger Neuronen gekennzeichnet ist, ist die Manipulation der Aktivität von aus Stammzellen stammenden dopaminergen Neuronen entscheidend für die Untersuchung ihrer synaptischen Inputs und Projektionsmuster in Nagetiermodellen 6,7,8,9,10. Die Integration von exzitatorischen hM3Dq- und inhibitorischen hM4Di-Rezeptoren in diese Modelle ermöglicht eine präzise Modulation der neuronalen Aktivität11,12.
Die Kombination von tierischen Verhaltensuntersuchungen und elektrophysiologischen Aufzeichnungen ermöglicht eine umfassende Bewertung der Auswirkungen der chemogenetischen Modulation auf transplantierte Zellen in vivo13. Verhaltensbewertungen, einschließlich der Apomorphin-induzierten Rotation, des Zylindertests und des Rotarod-Tests, bewerten die motorische Koordination und geben Einblicke in Veränderungen der motorischen Funktion, die mit experimentellen Modellen von PD14 verbunden sind. Elektrophysiologische Techniken, wie z. B. Patch-Clamp-Aufzeichnungen, ermöglichen die Echtzeitüberwachung synaptischer Reaktionen und Aktionspotentiale und bieten einen umfassenden Überblick darüber, wie sich transplantierte Zellen in bestehende neuronale Netzwerke integrieren15. Durch die Kombination von Verhaltensbewertungen mit elektrophysiologischen Bewertungen können wir untersuchen, wie sich die chemogenetische Modulation auf die Integration und Funktionalität dieser Zellen in den neuronalen Schaltkreisen des Wirts auswirkt16. Vorläufige Ergebnisse deuten darauf hin, dass die Verabreichung von CNOs die neuronale Aktivität in transplantierten Zellen effektiv moduliert, was zu verbesserten funktionellen Ergebnissen in Tiermodellen führt.
In diesem Protokoll wurden humane reprogrammierte Stammzellen so verändert, dass sie hM3Dq- oder hM4Di-Rezeptoren exprimieren, indem die CRISPR-Technologie (Clustered Regularly Interspaced Short Palindromic Repeats) verwendet wurde. Nach der Differenzierung modifizierter reprogrammierter Stammzellen in dopaminerge Vorläuferzellen des Mittelhirns wurden diese Zellen in Mausmodelle der Parkinson-Krankheit transplantiert, um ihre Integration und funktionelle Regulation innerhalb der neuronalen Schaltkreise des Wirts mithilfe von Verhaltensbewertungen und elektrophysiologischen Aufzeichnungen zu bewerten.
Protokoll
Alle Tierversuche wurden in Übereinstimmung mit den Richtlinien der Beijing Association for Laboratory Animal Science und der National Institutes of Health for the Care and Use of Laboratory Animals durchgeführt. Humane mononukleäre Zellen des peripheren Blutes (PBMCs) wurden von einem gesunden Spender mit schriftlicher Einverständniserklärung gewonnen, wie in einer früheren Studiebeschrieben 17.
1. Konstruktion von nicht-fusionsbasierten DREADD-Stammzelllinien
- Konstruieren Sie das DREADD-Donorplasmid.
HINWEIS: Die Aufschlussstellen sollten entsprechend der Vektorsequenz gestaltet werden. Die Fragmente sollten synthetisiert und mit 20-30 bp Überlappungen amplifiziert werden, die zu den Fragmentarmen passen, um die Assemblierungseffizienz zu verbessern.- Entwerfen und synthetisieren Sie das T2A-ZsGreen-Fragment (Fragment I).
- Amplifizieren Sie das hM3Dq/hM4Di-T2A-Fragment aus dem ursprünglichen Plasmid mittels PCR (Fragment II). Richten Sie die PCR-Reaktion (50 μl Gesamtvolumen) so ein, dass sie 1x PCR-Puffer, 200 μM jeder dNTP-Mischung, 0,5 μM Forward- und Reverse-Primer, 10-50 ng Plasmid-DNA-Template und 1,25 U High-Fidelity-DNA-Polymerase enthält. Führen Sie das Thermocycling unter folgenden Bedingungen durch: Erstdenaturierung bei 98 °C für 2,75 Minuten, um die Schablone vollständig zu denaturieren; 32 Amplifikationszyklen, bestehend aus 98 °C für 15 s (Denaturierung), 60 °C für 30 s (Primerglühen) und 72 °C für 60 s (Verlängerung); Endausdehnung bei 72 °C für 7 min.
- Verdauen Sie das Vektorplasmid mit dem entsprechenden Enzym, um den linearisierten Vektor zu erhalten. Reinigen Sie alle Fragmente durch Agarose-Gelelektrophorese und quantifizieren Sie sie mit einem Spektralphotometer.
HINWEIS: Hier wurde das ursprüngliche Plasmid mit MluI und SalI verdaut.
- Mischen Sie vorsichtig 50-100 ng des linearisierten Vektors, Fragment I und Fragment II in einem molaren Verhältnis von 1:3:3, zusammen mit 2x Cloning Mix. Die Mischung wird 1 h lang bei 55 °C inkubiert, um die homologe Rekombination durch Gibson-Assemblierung zu erleichtern.
- Fügen Sie die Guide-RNA, die auf die AAVS1-Stelle abzielt, in die BbsI-Stelle des pX458-Vektors ein.
HINWEIS: Führen Sie eine Sequenzverifizierung für alle konstruierten Plasmide mit Sanger-Sequenzierung durch. - Halten Sie die reprogrammierten Stammzellen in den Medien und streben Sie eine Zelldichte von 2-5 × 106 Zellen/ml an.
HINWEIS: Die hier verwendeten reprogrammierten Stammzellen wurden aus humanen PBMCs induziert, wie in einer früheren Studiebeschrieben 17. Die Überwachung der Zellgesundheit ist unerlässlich, da der Zellstatus die Effizienz der Elektroporation erheblich beeinflusst. Siehe Materialtabelle für einen Vergleich der Medien. - Mischen Sie 2-5 × 106 Zellen, 2 μg von jedem Donorplasmid (hM4Di-T2A-ZsGreen oder hM3Dq-T2A-ZsGreen) und 2 μg gRNA-Plasmid in 100 μl Elektroporationspuffer und überführen Sie es zur Elektroporation in eine Küvette.
HINWEIS: Stellen Sie sicher, dass die Mischung gut vermischt und frei von Blasen ist, bevor Sie mit der Elektroporation fortfahren. Optimieren Sie die Elektroporationsparameter basierend auf dem Zelltyp, um die Transfektionseffizienz zu maximieren, da verschiedene Geräte möglicherweise spezifische Anpassungen erfordern. - Stellen Sie den Nucleofector auf das B16-Programm ein und initiieren Sie die Elektroporation, um eine effiziente Transfektion der Ziel-iNSCs zu ermöglichen.
- 1 ml 50 μg/ml Poly-D-Lysin (PDL) in jede Vertiefung einer 6-Well-Platte geben und mindestens 2 Stunden bei Raumtemperatur inkubieren. Entfernen Sie sämtliches PDL aus den Vertiefungen und geben Sie 1,5 mL eines 5 μg/ml-Laminins in jede Vertiefung und inkubieren Sie bei 37 °C für 2-4 Stunden. Die beschichtete Platte sofort verwenden oder 1 Woche bei 2-8 °C lagern.
- Übertragen Sie die elektroporierten Zellen in Sechs-Well-Platten, die mit PDL und Laminin beschichtet wurden. Verteilen Sie ca. 500.000 Zellen pro Vertiefung in einem Volumen von 2 ml Kulturmedium pro Vertiefung.
- Verwenden Sie nach 72 Stunden Elektroporation ein Durchflusszytometer, um fluoreszierende positive Zellen zu sortieren.
- Resuspendieren Sie dissoziierte Zellen in DPBS und filtrieren Sie durch ein 70-μm-Zellsieb.
- Konfigurieren Sie den Sortierer mit FITC-Kanal für die ZsGreen-Erkennung. Nehmen Sie unbehandelte iNSCs als Negativkontrollen auf, um die Baseline-Fluoreszenz zu definieren.
- Kalibrieren Sie das Gerät mit 100-μm-Düsen bei einem Manteldruck von 20 psi. Implementieren Sie einen Vier-Wege-Sortiermodus für Reinheit mit Einzelzellbestätigung.
- Einzelne ZsGreen+ Zellen in 96-Well-Platten abfüllen, die mit PDL und Laminin vorbeschichtet sind, und 200 μl Kulturmedium pro Well hinzufügen, um das anfängliche Wachstum zu unterstützen. 7-14 Tage im Kulturmedium aufbewahren
HINWEIS: Überwachen Sie die Vertiefungen täglich auf Anzeichen von Wachstum und Fluoreszenz, die auf eine erfolgreiche Integration hinweisen, und markieren Sie die positive Vertiefung. - Isolieren Sie einzelne Klone, die Fluoreszenz aufweisen, mit einem inversen Fluoreszenzmikroskop, das mit einem GFP-Filterset ausgestattet ist. Bildschirmklone mit 10-facher Vergrößerung. Markieren Sie Vertiefungen, die monoklonale Kolonien enthalten, mit einem anhaltenden, einheitlichen ZsGreen-Signal. Erfassen Sie Hellfeld- und Fluoreszenzbilder, um Morphologie und Fluoreszenzintensität zu dokumentieren.
HINWEIS: Ersetzen Sie vor der Bildgebung frisches Medium, um die Hintergrundfluoreszenz zu reduzieren. Kolonien mit unregelmäßiger Morphologie oder Autofluoreszenz ausschließen. - Expandieren Sie die positiven Zellen auf beschichteten 48-Well-Platten. Dann in zwei Teile aufgeteilt, einen für die Kultur und den anderen für die Genotypisierung.
- Extrahieren Sie genomische DNA von Klonkandidaten. Bereiten Sie PCR-Reaktionen mit zwei Primerpaaren vor: ein Paar zur Bestätigung der Insertion des interessierenden Gens, das andere Primer zur Unterscheidung von homozygoten und heterozygoten Klonen. Führen Sie das Thermocycling unter folgenden Bedingungen durch: anfängliche Denaturierung bei 95 °C für 5 Minuten; 38 Zyklen von 95 °C für 30 s, 60 °C für 30 s und 72 °C für 45 s; Endausdehnung bei 72 °C für 7 min. Lösen Sie PCR-Produkte auf einem 2%igen Agarose-Gel auf.
HINWEIS: Die Grundierungen finden Sie in der Materialtabelle . Bestätigen Sie die Insertion des interessierenden Gens durch eine einzelne 650 bp-Bande (Primer 1). Heterozygote Klone werden durch das Vorhandensein einer einzelnen 630 bp-Bande und homozygote Klone durch das Fehlen dieser Bande identifiziert (Primer 2). - Expandieren Sie die positiven Zellen auf beschichteten Sechs-Well-Platten.
- Verdauen Sie die Zellen und waschen Sie sie in phosphatgepufferter Kochsalzlösung (PBS) durch Zentrifugation bei 250 × g für 3 Minuten. Frieren Sie die Zellen in Kryokonservierungsmedium mit 10 % DMSO bei einer Dichte von 2,5 × 106 Zellen pro Fläschchen ein.
2. Transplantierte aus DREADD-Stammzellen gewonnene Vorläuferzellen in Parkinson-Modellmäuse
- Verabreichen Sie immundefizienten Mäusen 30 Minuten vor der Operation eine intraperitoneale Injektion von Desipramin in einer Konzentration von 5 mg/ml (10 mg/kg). Betäuben Sie die Mäuse mit 2% Isofluran.
- Führen Sie einseitige stereotaktische Injektionen von 3 μl 6-Hydroxydopamin (6-OHDA, 5 μg/μl) durch. Richten Sie das rechte Corpus Striatum mit den folgenden Koordinaten aus: anteroposterior = 0,5 mm, lateral = 2,1 mm und vertikal = -3,2 mm.
HINWEIS: 6-OHDA sollte in Kochsalzlösung gelöst werden, die 0,2 % Ascorbinsäure enthält. Stellen Sie eine präzise Zielerfassung sicher, um die Variabilität des Schweregrads der Läsion zu minimieren.
ACHTUNG: 6-OHDA ist zytotoxisch und produziert reaktive Sauerstoffspezies, die zellulären Stress induzieren. Mit Vorsicht behandeln. - Verabreichen Sie eine intraperitoneale Injektion von Apomorphin (1 mg/kg, 0,5 mg/ml in Kochsalzlösung gelöst), um die Läsionen 4 Wochen nach der Operation zu validieren. Wählen Sie läsionierte Mäuse aus, die innerhalb eines Beobachtungszeitraums von 30 Minuten mehr als 100 kontralaterale Rotationen aufweisen, für nachfolgende Experimente.
HINWEIS: Apomorphin ist ein Dopaminrezeptoragonist, der überempfindliche postsynaptische Rezeptoren im denervierten Striatum direkt aktiviert und die bilaterale Aktivität des nigrostriatalen Schaltkreises aus dem Gleichgewicht bringt. Die Apomorphin-induzierte Rotation dient dazu, den Schweregrad einseitiger dopaminerger Läsionen durch 6-OHDA zu bewerten. - Keim- und Kulturzellen in beschichteten Sechs-Well-Platten (150.000 Zellen pro Well). Inkubieren Sie 10 Tage lang in Phase 1 Differenzierungsmedium und erneuern Sie das Medium alle 2 Tage. Wechseln Sie an Tag 11 zum Differenzierungsmedium der Phase 2 und setzen Sie die Inkubation bis zum 13. Tag fort, wobei Sie das Medium jeden Tag wechseln.
HINWEIS: Siehe Materialtabelle für einen Vergleich des Differenzierungsmediums. - Ernten Sie die Zellen an den Tagen 10 und 13 der Differenzierung und mischen Sie sie dann im Verhältnis 1:7. Suspendieren Sie die Mischung in einem Transplantationspuffer in einer Konzentration von 100.000 Zellen/μl.
HINWEIS: In der Materialtabelle finden Sie einen Vergleich des Transplantationspuffers. - Verabreichen Sie eine stereotaktische Injektion eines Gesamtvolumens von 4 μl der vorbereiteten Zellsuspension in eine Parkinson-Maus.
- Führen Sie den Zylindertest zu verschiedenen Zeitpunkten nach der Zelltransplantation durch. Stellen Sie einen Glasbecher (Durchmesser: 20 cm, Höhe: 30 cm) auf eine ebene, nicht reflektierende Fläche. Positionieren Sie eine Draufsichtkamera über dem Zylinder, um den vollen Durchmesser zu erfassen.
HINWEIS: Umschließen Sie den Zylinder mit schwarzen Vorhängen, um äußere visuelle Reize zu eliminieren. - Platzieren Sie jede Maus in der Mitte des Zylinders und starten Sie die gleichzeitige Videoaufzeichnung (3 Minuten/Sitzung). Bringen Sie die Maus nach der Sitzung wieder in ihren Ausgangskäfig zurück.
HINWEIS: Akklimatisieren Sie die Mäuse 30 Minuten lang unter Lichtbedingungen an den Testraum. Reinigen Sie den Zylinder zwischen den Versuchen mit 70 % Ethanol, um Geruchsreize zu beseitigen. - Beurteilen Sie die Bewegungsfähigkeit der oberen Gliedmaßen der Mäuse für 3 Minuten. Berechnen Sie die Anzahl der Wandkontakte, die von der beeinträchtigten Vordergliedmaße hergestellt wurden, im Verhältnis zur Gesamtzahl der Kontakte, die von beiden Vordergliedmaßen hergestellt wurden.
- Verabreichen Sie entweder Kochsalzlösung oder CNO in einer Dosierung von 1,2 mg/kg durch intraperitoneale Injektion. Durchführung des Zylindertests zur Beurteilung der Verhaltensmodulation im PD-Mausmodell nach CNO-Verabreichung
HINWEIS: Warten Sie eine Erholungsphase von ca. 40 Minuten, bevor Sie mit der Bewertung fortfahren.
3. In vivo elektrophysiologisches Profiling chemogenetisch modulierter Zellen
- Betäuben Sie Mäuse durch eine intraperitoneale Injektion von 250 mg/kg Pentobarbital. Entnehmen Sie das Gehirn und legen Sie es sofort in eiskalte künstliche Zerebrospinalflüssigkeit (s-ACSF) auf Saccharosebasis, um die zelluläre Integrität zu erhalten. Schneiden Sie das Gehirn mit einem Vibratom in 300-400 μm dicke Abschnitte.
HINWEIS: Arbeiten Sie schnell, um das Gehirn auf niedrigen Temperaturen zu halten und die Zellfunktion zu erhalten. Siehe Materialtabelle für den s-ACSF, der bei der Vorbereitung frischer Hirnschnitte verwendet wird. - Die Proben werden in eine mit ACSF gefüllte Kammer mit 95 % O2 und 5 % CO2 bei 34 °C gefüllt, wobei sichergestellt wird, dass diese Temperatur während des gesamten Experiments beibehalten wird.
HINWEIS: Siehe Materialtabelle für ACSF. - Laden Sie Glaspipetten mit vereister intrazellulärer Lösung mit einem Elektrodenwiderstand von 7 bis 10 MΩ.
HINWEIS: Siehe Materialtabelle für intrazelluläre Lösung. Glaspipetten werden aus Glaskapillaren mit Hilfe eines Mikropipettenabziehers18 hergestellt. Überprüfen Sie vor Beginn der Experimente die Glaspipetten auf Undichtigkeiten und stellen Sie sicher, dass die Spitzen scharf genug sind, um die Zellmembran zu durchdringen. Bereiten Sie Pipetten schnell vor, um Temperaturschwankungen in der intrazellulären Lösung zu minimieren. - Montieren Sie die Slices in einer untergetauchten Aufnahmekammer, die mit sauerstoffreichem ACSF bei 34 °C überflogen ist (2 ml/min). Positionieren Sie Pipetten mit motorisierten Mikromanipulatoren unter Infrarot-Differenzinterferenz-Kontrastmikroskopie. Stellen Sie eine Ganzzellkonfiguration her, indem Sie sanften Sog (Widerstand > 1 GΩ) auf gepatchte Neuronen in Transplantatregionen anwenden. Klemmen von Zellen bei -70 mV mit einem Patch-Clamp-Verstärker; Erfassung von Basis-sEPSCs für 8 min bei einer Abtastrate von 10 kHz (gefiltert bei 2 kHz) und kontinuierliche Überwachung des Zugriffswiderstands (Ziel: <20 MΩ; verwerfen, wenn >25 MΩ oder fluktuierend >10 %).
HINWEIS: Verwenden Sie beim Saugen angemessenen Druck, um eine unerwünschte Zellverschiebung oder einen Zellbruch zu vermeiden, und stellen Sie die Pipettenposition sorgfältig ein, um eine gute Abdichtung zu erzielen. Wenn die Qualität der Dichtung schlecht ist, entsorgen Sie die Pipette und versuchen Sie es erneut. - Geben Sie unmittelbar nach Abschluss der Basismessung 50 μM CNO in die Aufzeichnungskammer und setzen Sie die Datenaufzeichnung für weitere 16 Minuten fort, während Sie Änderungen der sEPSC-Frequenz oder -Amplitude beobachten.
- Führen Sie ein Auswaschverfahren durch, indem Sie die CNO-Lösung durch frisches ACSF ersetzen, um CNO effektiv aus der Aufnahmekammer zu entfernen. Setzen Sie die Aufzeichnung der Daten für den Rest des Experiments fort, um eine Erholung der synaptischen Aktivität zu bewerten.
Ergebnisse
Abbildung 1 zeigt die wichtigsten Schritte dieses methodischen Ansatzes für gentechnisch veränderte humane Stammzellen, die exzitatorische (hM3Dq) oder inhibitorische (hM4Di) DREADD-Rezeptoren exprimieren, um die funktionelle Integration und Modulation abgeleiteter dopaminerger Vorläufer in einem Mausmodell der Parkinson-Krankheit (PD) zu bewerten. Abbildung 2 zeigt die CRISPR/Cas9-vermittelte Gen-Knock-in-Strategie zur Einführung von Nicht-Fusionskonstrukten von hM4Di-T2A-ZsGreen und hM3Dq-T2A-ZsGreen in reprogrammierte Stammzellen und validiert durch Genotypisierung.
Abbildung 3 charakterisiert die funktionellen und synaptischen Effekte der chemogenetischen Modulation in transplantierten Zellen. Wir führten Verhaltensbewertungen und elektrophysiologische Aufzeichnungen durch, um die Auswirkungen der transplantierten DREADD-Zellen zu analysieren. Abbildung 3A zeigt die statistische Analyse der kontralateralen Bewegungsergebnisse der Vordergliedmaßen in einem Zylindertest über Kontroll-, hM4Di- und hM3Dq-Gruppen unter Kochsalzlösungs-, CNO- und postCNO-Auswaschungsbedingungen. Die Verabreichung von CNOs reduzierte die kontralaterale Beteiligung der Vordergliedmaßen bei hM4Di-transplantierten Tieren, verbesserte jedoch die motorische Leistung in der hM3Dq-Gruppe im Vergleich zu Kochsalzlösungskontrollen. Abbildung 3B-D zeigt die elektrophysiologische Analyse von spontanen exzitatorischen postsynaptischen Strömen (sEPSCs). Die hM4Dq-transplantierte Gruppe zeigte verlängerte Ereignisintervalle und reduzierte Spitzenamplituden, was auf synaptisches Silencing hinweist, während hM3Dq-transplantierte Zellen verkürzte Intervalle und erhöhte Amplituden aufwiesen, was mit einer verstärkten exzitatorischen Neurotransmission vereinbar ist. Abbildung 3E,F charakterisieren die transplantierten Zellen in vivo durch Immunfluoreszenz.

Abbildung 1: Chemogenetische Regulation in reprogrammierten Stammzell-abgeleiteten Vorläuferzellen zur Behandlung neurodegenerativer Erkrankungen im Mausmodell. Ein Flussdiagramm, das den methodischen Ansatz zur Etablierung eines Parkinson-Mausmodells und zur Bewertung von Verhaltensergebnissen über chemogenetische Regulation in reprogrammierten Stammzell-abgeleiteten Vorläuferzellen veranschaulicht. Schritt 1: Konstruktion von nicht-fusionellen DREADD-Stammzelllinien unter Verwendung der CRISPR-Technologie. Schritt 2: Etablierung des Parkinson-Mausmodells, gefolgt von der Transplantation von DREADD-Stammzell-Vorläuferzellen in das Gehirn. Schritt 3: Bewertung der verhaltensbezogenen und elektrophysiologischen Ergebnisse, um die erfolgreiche Integration und funktionelle Modulation der transplantierten Zellen zu überprüfen. Abkürzungen: CRISPR = clustered regularly interspaced short palindromic repeat; DREADD = Designer-Rezeptoren, die ausschließlich durch Designerdrogen aktiviert werden; PD = Parkinson-Krankheit; 6-OHDA = 6-Hydroxydopamin; CNO = Clozapin N-Oxid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: CRISPR/Cas9-vermitteltes Engineering von nicht-fusionellen DREADD-exprimierenden Stammzellen. (A) Flussdiagramm, das die CRISPR/Cas9-vermittelte Gen-Knock-in-Strategie für die Integration von nicht-fusionsbasierten hM4Di-T2A-ZsGreen- oder hM3Dq-T2A-ZsGreen-Konstrukten in reprogrammierte Stammzellen veranschaulicht. (B) Das Donorplasmid von nicht-fusionierendem hM4Di-T2A-ZsGreen oder hM3Dq-T2A-ZsGreen wurde durch eine mehrstufige Klonierungsstrategie modifiziert. Die Darstellung des Targeting-Allels veranschaulicht den Gen-Knock-in von nicht-fusionierendem hM4Di-T2A-ZsGreen oder hM3Dq-T2A-ZsGreen in reprogrammierten Stammzellen. Fluoreszenzbilder bestätigen, dass die gentechnisch veränderten Zellen eine homogene grüne Fluoreszenz aufweisen, was auf eine erfolgreiche Transgenexpression hinweist. Maßstabsbalken = 200 μm. (C) Die Ergebnisse der Genotypisierung bestätigen die erfolgreiche Integration der DREADD-Konstrukte in das Genom der Zielstammzelllinien. Diese Abbildung wurde von Wang et al.19 modifiziert. Abkürzungen: gRNA = Guide-RNA; FACS = fluoreszenzaktivierte Zellsortierung; DREADD = Designer-Rezeptoren, die ausschließlich durch Designerdrogen aktiviert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
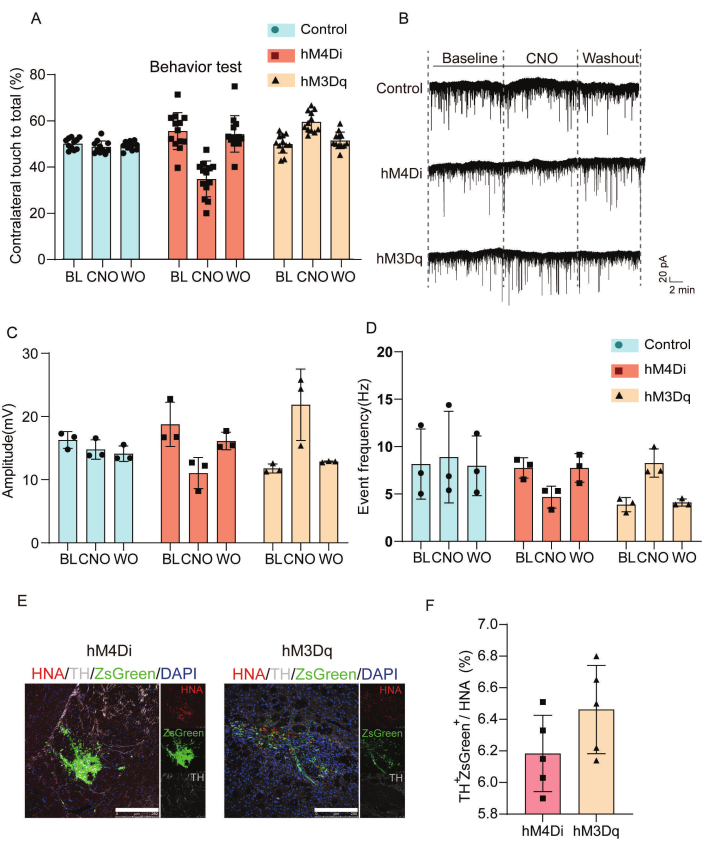
Abbildung 3: Bewertung der verhaltensbezogenen und elektrophysiologischen Ergebnisse zur Bestätigung der Integration und funktionellen Modulation von transplantierten Zellen. (A) Bewertung der Verhaltensmodulation im PD-Mausmodell nach CNO-Verabreichung; Detaillierte statistische Analyse zum Vergleich der Ergebnisse der kontralateralen Bewegung der Vordergliedmaßen im Zylindertest zwischen den Gruppen hM4Di, hM3Dq und Kontrollgruppen zu Studienbeginn und 8 Wochen nach der Transplantation, wobei die Auswertungen nach der Behandlung mit Kochsalzlösung und CNO sowie nach der Auswaschung nach CNO durchgeführt wurden. (B) Ganzzell-Patch-Clamp-Aufzeichnungen, die sEPSCs aus den Transplantaten von Kontroll-, hM4Di- oder hM3Dq-Gruppen zeigen, die zu Studienbeginn und während der CNO-Behandlung (50 μM) zusammen mit CNO-Auswaschphasen analysiert wurden. (C) Statistische Analyse der Peakamplitudenmessungen für die sEPSCs über Baseline, CNO und CNO-Washout. (D) Ereignishäufigkeitsstatistiken von sEPSCs, die aus den Transplantaten der Kontroll-, hM4Di- und hM3Dq-Gruppen aufgezeichnet wurden. (E)Charakterisierung von transplantierten Zellen in vivo durch Immunfluoreszenz. Maßstabsbalken = 250 μm. (F) Quantitative Analyse der Expression von Tyrosinhydroxylase und ZsGreen in hM4Di- und hM3Dq-Gruppen, relativ zur Expression des humanen nuklearen Antigens. Diese Abbildung wurde von Wang et al.19 modifiziert. Abkürzungen: BL = Baseline; CNO = Behandlung mit Clozapin N-oxid (CNO, 50 μM); WO = CNO auswaschen; PD = Parkinson-Krankheit; sEPSCs = spontane exzitatorische postsynaptische Ströme; TH = Tyrosinhydroxylase; HNA = humanes nukleäres Antigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Dieses Protokoll nutzte die CRISPR-Technologie, um menschliche reprogrammierte Stammzellen so zu entwickeln, dass sie exzitatorische hM3Dq- und inhibitorische hM4Di-Rezeptoren exprimieren. Die Modulation der neuronalen Aktivität durch CNO wurde durch Zelltransplantation in Mausmodelle der Parkinson-Krankheit untersucht, begleitet von Verhaltensbewertungen und elektrophysiologischen Aufzeichnungen.
Der erste entscheidende Schritt bei der Erzeugung stabil exprimierter, nicht-fusionsbedingter DREADD-Konstrukte in chemogenetisch reprogrammierten Stammzellen betrifft die T2A-ZsGreen bicistronische Kassette. Fusionsprotein-exprimierende Plasmide wie hM3Dq-mCherry und hM4Di-mCherry7 sind jedoch für diese induzierte neurale Stammzelllinie unwirksam. Um dies zu umgehen, haben wir ein Donorplasmid über Gibson-Assemblierung entwickelt, wie in den Protokollschritten 1.1-1.2 beschrieben.
Die Etablierung eines PD-Modells durch einseitige Verabreichung einer stereotaktischen Injektion von 6-OHDA in das Corpus Striatum immundefizienter Mäuse ist entscheidend für eine präzise in vivo Modulation. Während des gesamten Studienzeitraums, einschließlich Zelltransplantationen und medikamentöser Behandlungen, kann es bei einigen Tieren zu einer unerwarteten Sterblichkeit kommen. Daher sollte die anfängliche Stichprobengröße sorgfältig geplant werden, um eine ausreichende Anzahl für die Versuchsgruppen15,17 zu gewährleisten.
Mehrere Verhaltenstests in Bezug auf die motorische Funktion könnten verwendet werden, um die Verhaltensreaktionen der Tiere auf die DREADD-Modulation zu bewerten, einschließlich des Apomorphin-induzierten Rotationstests, des Zylindertests und des Rotarod-Tests. Hier haben wir den Zylindertest verwendet, der den Kontakt der Vordergliedmaßen mit den Wänden eines Zylinders20 quantitativ misst. Durch die Bewertung von Veränderungen in der Nutzung der oberen Extremitäten, die auf neuronale Schäden zurückzuführen sind, liefert der Zylindertest wertvolle Erkenntnisse darüber, wie CNO die motorische Funktion verbessern kann.
Die funktionelle Integration und synaptische Konnektivität von chemogenetisch umprogrammierten Spenderzellen innerhalb der neuronalen Schaltkreise des Wirts sind entscheidende Determinanten für die therapeutische Wirksamkeit bei neurodegenerativen Erkrankungen wie PD16. Ganzzell-Patch-Clamp-Aufnahmen, die nach der Transplantation an Hirnschnitten durchgeführt wurden, geben Aufschluss darüber, wie diese manipulierten Zellen mit nativen neuronalen Schaltkreisen interagieren. Mittelstachelige Neuronen (MSNs) im Striatum sind nachgeschaltete Ziele der dopaminergen Signalübertragung.
Mit Hilfe von Patch-Clamp-Aufzeichnungs-MSNs können wir genau beurteilen, wie sich DREADD-modifizierte Zellen in neuronale Schaltkreise integrieren und deren Funktion beeinflussen21. Die Zusammensetzung der künstlichen Zerebrospinalflüssigkeit (ACSF) für unterschiedliche Verfahrensphasen ist entscheidend für die Patch-Clamp. Während der Schnittvorbereitung wird dringend eine künstliche Zerebrospinalflüssigkeit (s-ACSF) auf Saccharosebasis empfohlen. Die hyperosmotischen Eigenschaften von Saccharose mildern die neuronale Schwellung während des Schnitts und halten gleichzeitig die für die zelluläre Lebensfähigkeit unerlässlichen Ionengradienten aufrecht. Die Elektrodenherstellung und die Qualitätskontrolle bestimmen direkt die Aufnahmestabilität. Borosilikatglaspipetten, die auf Spitzenwiderstände von 7-10 MΩ gezogen werden, stellen eine nicht verhandelbare Voraussetzung für nachhaltige sEPSC-Aufzeichnungen dar. Suboptimale Pipetten mit Widerständen unter 5 MΩ weisen einen beschleunigten Diffusionsaustausch zwischen Pipettenlösung und Zytoplasma auf, was zu einem fortschreitenden Stromabfall innerhalb von 3-5 Minuten führt.
Diese Studie zeigt jedoch, dass die chemogenetische Modulation des motorischen Verhaltens und der synaptischen Aktivität bei Parkinson-Mäusen durch manipuliertes iNSC-DAP, eine erhebliche prozedurale Attrition im Zusammenhang mit dual-stereotaktischen Interventionen und chronische CNO-Verabreichung die Notwendigkeit verfeinerter Xenotransplantationsparadigmen in immungeschwächten Modellen unterstreicht, insbesondere im Hinblick auf die chirurgische Standardisierung. Diese Strategie, die CRISPR/Cas9-vermitteltes DREADD-Rezeptor-Engineering mit der Abfrage auf Schaltkreisebene über Verhaltensassays (Zylindertest) und Ganzzell-Patch-Clamp-Elektrophysiologie kombiniert, liefert einen Neuronen-Subtyp-spezifischen Bauplan für die Neuromodulation und bietet ein vielseitiges Toolkit zur Weiterentwicklung präziser Zelltherapien für neurodegenerative Erkrankungen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde unterstützt von der Beijing Natural Science Foundation (7242068), der National Natural Science Foundation of China (82171250), dem Beijing Municipal Health Commission Fund (PXM2020_026283_000005) und dem Projekt für Technologieentwicklung der mit Peking verbundenen medizinischen Forschungsinstitute (11000023T000002036310).
Materialien
| Name | Company | Catalog Number | Comments |
| 2 x Rapid Taq Master Mix | Vazyme | P222-01 | used for genotyping analysis |
| 2×Seamless Cloning Mix | Biomed Gene Tech. | CL117-01 | used for plasmid construction |
| 6-OHDA | Sigma-Aldrich | H4381 | used for establishing PD mice model |
| AAVS1-Pur-CAG-EGFP | Addgene | 80945 | used as control |
| AAVS1-Pur-CAG-hM4Di-mCherry | Addgene | 80947 | original plasmid for construction of hM4Di-T2A-ZsGreen |
| AAVS1-Pur-CAG-hM3Dq-mCherry | Addgene | 80948 | original plasmid for construction of hM3Dq-T2A-ZsGreen |
| AAVS1-CAG-hM4Di-T2A-ZsGreen | N/A | N/A | Constructed donor plasmid based on #80947 |
| AAVS1-CAG-hM3Dq-T2A-ZsGreen | N/A | N/A | Constructed donor plasmid based on #80948 |
| Accutase | Invitrogen | A11105-01 | Used for digesting cells |
| AMAXA Nucleofector | Lonza | AAD-1001S | |
| Apomorphine | Sigma-Aldrich | A4393 | |
| Artificial cerebrospinal fluid (ACSF) | N/A | N/A | 125 mM NaCl, 2.5 mM KCl, 2 mM CaCl2, 1.25 mM NaH2PO4, 1 mM MgSO4, 25 mM glucose, and 26 mM NaHCO3 |
| Ascorbic acid | Sigma-Aldrich | 1043003 | |
| B-27 Supplement | Gibco | 17504044 | |
| BDNF | Peprotech | 450-02 | |
| cAMP | Sigma-Aldrich | D0627 | |
| CHIR99021 | Yeasen | 53003ES10 | |
| Clozapine-N-oxide | Enzo | BML-NS105 | |
| DAPT | Sigma-Aldrich | D5942 | |
| Desipramine | Sigma-Aldrich | D3900 | |
| D-glucose | Sigma-Aldrich | G5767 | |
| DMEM/F12 | Gibco | 11330-032 | |
| DMEM/F12 | Gibco | 11320-033 | |
| DMSO | Sigma-Aldrich | D2650 | |
| FGF8 | Peprotech | 100-25 | |
| GDNF | Peprotech | 450-10 | |
| GlutaMax | Gibco | 35050-061 | |
| Hank’s Balanced Salt Solution (HBSS) | Gibco | 14175095 | |
| Human leukemia inhibitory factor (hrLIF) | Millipore | LIF1010 | |
| Iced intracellular fluid | N/A | N/A | 130 mM K-gluconate, 16 mM KCl, 0.2 mM EGTA, 2 mM MgCl2, 10 mM HEPES, 4 mM Na2-ATP, 0.4 mM Na3-GTP, 0.3% of neurobiotin |
| KnockOut Serum Replacement | Gibco | 10828028 | |
| Laminin | Roche | 11243217001 | |
| Micropipette Puller | Sutter Instrument Company | P-1000 | |
| N-2 Supplement | Thermo Fisher | 17502048 | |
| Neurobasal-A Medium | Gibco | 10888-022 | |
| Non-essential amino acids (NEAA) | Gibco | 11140-050 | |
| PBS | Gibco | 10010023 | |
| pCLAMP 11 software suite | Molecular Devices | N/A | Patch-clamp electrophysiology data acquisition and analysis software |
| Phase 1 differentiation medium | N/A | N/A | 96% DMEM/F12 (Gibco, 11320-033), 1% B-27 Supplement, 1% N-2 Supplement, 1% NEAA, 1% GlutaMax, 1 µM SAG1, and 100 ng/mL FGF8 |
| Phase 2 differentiation medium | N/A | N/A | 96% DMEM/F12 (Gibco, 11320-033), 1% B-27 Supplement, 1% N-2 Supplement, 1% NEAA, and 1% GlutaMax, 10 ng/mL BDNF, 10 ng/mL GDNF, 1 ng/mL TGF-βIII, 10 µM DAPT, 0.2 mM ascorbic acid, and 0.5 mM cAMP. |
| Poly-D-lysine hydrobromide (PDL) | Sigma-Aldrich | P7886 | |
| Primers for genotyping | N/A | N/A | Insertion Foward: TCTTCACTCGCTGGGTTCCCTT; Insertion Reverse: CCTGTGGGAGGAAGAGAAGAGGT; Homozygosity Foward:CGTCTCCCTGGCTTTAGCCA; Homozygosity Reverse: GATCCTCTCTGGCTCCATCG |
| pX458 | Addgene | 152199 | |
| SAG1 | Enzo | ALX-270-426-M01 | |
| SB431542 | Yeasen | 53004ES50 | |
| sucrose-based artificial cerebrospinal fluid (s-ACSF) | 234 mM sucrose, 2.5 mM KCl, 26 mM NaHCO3, 1.25 mM NaH2PO4, 11 mM Dglucose, 0.5 mM CaCl2, and 10 mM MgSO4 | ||
| Stem cell culture media | N/A | N/A | 48% DMEM/F12 (Gibco, 11330-032) and 48% Neurobasal, with the addition of 1% B27, 1% N2, 1% NEAA, 1% GlutaMax, 10 ng/mL hrLIF, 2 µM SB431542, and 3 µM CHIR99021 |
| TGF-βIII | Peprotech | 100-36E | |
| Transplantation buffer | N/A | N/A | HBSS buffer with 5 g/L D-glucose, 100 ng/mL BDNF, 100 ng/mL GDNF, and 0.2 mM ascorbic Acid |
| Vibratome | Leica | VT1000 S | |
| Whole-cell patch-clamp | Molecular Devices | MultiClamp700B |
Referenzen
- Thiel, G. Designer receptors exclusively activated by designer drugs. Humana Press (2015).
- Roth, B. L. Dreadds for neuroscientists. Neuron. 89 (4), 683-694 (2016).
- Gomez, J. L. et al. Chemogenetics revealed: DREADD occupancy and activation via converted clozapine. Science. 357 (6350), 503-507 (2017).
- Chehelgerdi, M. et al. Exploring the promising potential of induced pluripotent stem cells in cancer research and therapy. Mol Cancer. 22 (1), 189 (2023).
- Song, J., Patel, R. V., Sharif, M., Ashokan, A., Michaelides, M. Chemogenetics as a neuromodulatory approach to treating neuropsychiatric diseases and disorders. Mol Ther. 30 (3), 990-1005 (2022).
- Parmar, M., Grealish, S., Henchcliffe, C. The future of stem cell therapies for Parkinson disease. Nat Rev Neurosci. 21 (2), 103-115 (2020).
- Dell'anno, M. T. et al. Remote control of induced dopaminergic neurons in Parkinsonian rats. J Clin Invest. 124 (7), 3215-3229 (2014).
- Chen, Y. et al. Chemical control of grafted human PSC-derived neurons in a mouse model of Parkinson's disease. Cell Stem Cell. 18 (6), 817-826 (2016).
- Bjorklund, A. Parmar, M. Dopamine cell therapy: From cell replacement to circuitry repair. J Parkinsons Dis. 11 (S2), S159-S165 (2021).
- Alcacer, C. et al. Chemogenetic stimulation of striatal projection neurons modulates responses to Parkinson's disease therapy. J Clin Invest. 127 (2), 720-734 (2017).
- Alexander, G. M. et al. Remote control of neuronal activity in transgenic mice expressing evolved G protein-coupled receptors. Neuron. 63 (1), 27-39 (2009).
- Stachniak, T. J., Ghosh, A., Sternson, S. M. Chemogenetic synaptic silencing of neural circuits localizes a hypothalamus midbrain pathway for feeding behavior. Neuron. 82 (4), 797-808 (2014).
- Kang, H. J., Minamimoto, T., Wess, J., Roth, B. L. Chemogenetics for cell-type-specific modulation of signalling and neuronal activity. Nat Rev Methods Primers. 3 (1), 93 (2023).
- Miyanishi, K. et al. Behavioral tests predicting striatal dopamine level in a rat hemi-Parkinson's disease model. Neurochem Int. 122, 38-46 (2019).
- Zheng, X. et al. Human IPSC-derived midbrain organoids functionally integrate into striatum circuits and restore motor function in a mouse model of Parkinson's disease. Theranostics. 13 (8), 2673-2692 (2023).
- Xiong, M. et al. Human stem cell-derived neurons repair circuits and restore neural function. Cell Stem Cell. 28 (1), 112-126.e116 (2021).
- Yuan, Y. et al. Dopaminergic precursors differentiated from human blood-derived induced neural stem cells improve symptoms of a mouse Parkinson's disease model. Theranostics. 8 (17), 4679 (2018).
- Zhu, M. et al. Preparation of acute spinal cord slices for whole-cell patch-clamp recording in substantia gelatinosa neurons. J Vis Exp. (143) (2019).
- Wang, X., Han, D., Zheng, T., Ma, J., Chen, Z. Modulation of human induced neural stem cell-derived dopaminergic neurons by DREADD reveals therapeutic effects on a mouse model of Parkinson's disease. Stem Cell Res Ther. 15 (1), 297 (2024).
- Iancu, R., Mohapel, P., Brundin, P., Paul, G. Behavioral characterization of a unilateral 6-OHDA-lesion model of Parkinson's disease in mice. Behav Brain Res. 162 (1), 1-10 (2005).
- Surmeier, D. J. et al. The role of dopamine in modulating the structure and function of striatal circuits. Prog Brain Res. 183, 148-167 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten