Method Article
Identifizierung von Knochenmark-Mikroumgebungspopulationen beim myelodysplastischen Syndrom und der akuten myeloischen Leukämie
In diesem Artikel
Zusammenfassung
Hier wird ein detailliertes Protokoll zur Isolierung und Charakterisierung von Knochenmark-Mikroumgebungspopulationen aus murinen Modellen myelodysplastischer Syndrome und akuter myeloischer Leukämie vorgestellt. Mit dieser Technik werden Veränderungen in der nicht-hämatopoetischen Knochenmarknische, einschließlich der endothelialen und mesenchymalen Stromazellen, mit Fortschreiten der Erkrankung identifiziert.
Zusammenfassung
Die Mikroumgebung des Knochenmarks besteht aus verschiedenen Zellpopulationen, wie mesenchymalen Stromazellen, Endothelzellen, Osteostammzellen und Fibroblasten, die hämatopoetische Stammzellen (HSCs) unterstützen. Neben der Unterstützung normaler HSCs spielt die Mikroumgebung des Knochenmarks auch eine Rolle bei der Entwicklung von hämatopoetischen Stammzellerkrankungen wie myelodysplastischen Syndromen (MDS) und akuter myeloischer Leukämie (AML). MDS-assoziierte Mutationen in HSCs führen zu einer Blockade der Differenzierung und zu einem fortschreitenden Knochenmarkversagen, insbesondere bei älteren Menschen. MDS kann oft zu einer therapieresistenten AML führen, einer Erkrankung, die durch eine schnelle Anhäufung unreifer myeloischer Blasten gekennzeichnet ist. Es ist bekannt, dass die Mikroumgebung des Knochenmarks bei Patienten mit diesen myeloischen Neoplasien verändert ist. In dieser Arbeit wird ein umfassendes Protokoll zur Isolierung und phänotypischen Charakterisierung von Mikroumgebungszellen des Knochenmarks aus murinen Modellen des myelodysplastischen Syndroms und der akuten myeloischen Leukämie beschrieben. Die Isolierung und Charakterisierung von Veränderungen in den Nischenpopulationen des Knochenmarks kann dazu beitragen, ihre Rolle bei der Initiierung und dem Fortschreiten der Krankheit zu bestimmen und kann zur Entwicklung neuartiger Therapeutika führen, die auf krebsfördernde Veränderungen in den Knochenmarkstromapopulationen abzielen.
Einleitung
Die Mikroumgebung des Knochenmarks besteht aus hämatopoetischen Zellen, nicht-hämatopoetischen Stromazellen und der extrazellulären Matrix 1,2. Diese Mikroumgebung kann die Selbsterneuerung hämatopoetischer Stammzellen fördern, die Differenzierung von Abstammungslinien regulieren und das Knochengewebe strukturell und mechanisch stützen 1,2,3,4,5. Die stromale Nische umfasst Osteolineage-Zellen, Fibroblasten, Nervenzellen und Endothelzellen6, während die hämatopoetische Nische aus den lymphatischen und myeloischen Populationen besteht 1,2,3. Neben der Unterstützung normaler HSCs kann die Mikroumgebung des Knochenmarks auch eine Rolle bei der Entwicklung von hämatopoetischen Stammzellerkrankungen wie MDS und AML spielen 7,8,9,10,11. Es wurde gezeigt, dass Mutationen in Osteolinienzellen die Entwicklung von MDS, AML und anderen myeloproliferativen Neoplasien fördern 10,12,13,14.
Myelodysplastische Syndrome sind eine Gruppe präleukämischer Erkrankungen, die durch Mutationen in hämatopoetischen Stammzellen entstehen. MDS ist häufig mit einer Blockade der HSZ-Differenzierung und der Produktion von dysplastischen Zellen verbunden, was häufig zu Knochenmarkversagen führen kann. MDS ist die am häufigsten diagnostizierte myeloische Neoplasie in den Vereinigten Staaten und mit einer 3-Jahres-Überlebensrate von 35 % bis 45 % verbunden15. MDS ist oft mit einem hohen Risiko für die Transformation in eine akute myeloische Leukämie verbunden. Dies kann eine tödliche Komplikation sein, da die MDS-abgeleitete AML gegen die meisten Therapien resistent ist und wahrscheinlich einen Rückfall erleidet. AML, die de novo aufgrund von Translokationen oder Mutationen im hämatopoetischen Stamm und in den Vorläuferzellen entsteht, ist häufig auch resistent gegen die Standard-Chemotherapie16,17. Da es sich bei MDS und AML in erster Linie um Erkrankungen älterer Menschen handelt, wobei die meisten im Alter von über 60 Jahren diagnostiziert werden, kommen die meisten Patienten für eine kurative Knochenmarktransplantation nicht in Frage. Es besteht daher ein erheblicher Bedarf, neue Regulatoren des Krankheitsverlaufs zu identifizieren. Da die Mikroumgebung des Knochenmarks bösartige Zellen unterstützen kann14, kann die Definition von Veränderungen in der Knochenmarknische mit dem Fortschreiten der Krankheit zur Identifizierung neuer Therapeutika führen, die darauf abzielen, den Umbau von Tumornischen zu hemmen. Es besteht daher ein erheblicher Bedarf, neue Regulatoren des Krankheitsverlaufs zu identifizieren. Zu diesem Zweck ist es von entscheidender Bedeutung, Veränderungen in den Stromazellpopulationen des Knochenmarks zu identifizieren und zu charakterisieren, die die malignen Zellen unterstützen können.
Es wurden mehrere Mausmodelle für AML und MDS entwickelt, die zur Untersuchung von Veränderungen in der Mikroumgebung des Knochenmarks während des Krankheitsbeginns und -fortschreitens verwendet werdenkönnen 6,1,19,20,21,22. Hier werden Protokolle zur Identifizierung von Veränderungen in den Knochenmark-Stromazellpopulationen unter Verwendung von murinen Modellen der retroviral induzierten AML 6,20 sowie des kommerziell erhältlichen Nup98-HoxD13 (NHD13)-Modells der Hochrisiko-MDS-zu-AML-Transformation19 beschrieben. Mäuse, denen de novo AML-Zellen transplantiert wurden, erliegen der Krankheit innerhalb von 20-30 Tagen6. Die NHD13-Mäuse entwickeln in der 15. bis 20. Woche Zytopenien und Knochenmarkdysplasie, die sich schließlich in AML umwandeln, und fast 75 % der Mäuse erliegen der Krankheit nach etwa 32 Wochen. Um die mikroumgebungsspezifischen Populationen des murinen Modells des Knochenmarks zu analysieren, werden Knochen entnommen, Knochenmark und Knochenspikulen mittels enzymatischer Verdauung verdaut und die Zellen dann durch magnetische Sortierung für CD45-/Ter119-nicht-hämatopoetische Populationen angereichert. Obwohl ähnliche Analysen bereits beschrieben wurden 11,13,22,23,24,25, konzentrieren sie sich oft entweder auf das Knochenmark oder den Knochen und beziehen Zellen aus beiden Quellen nicht in ihre Analysen ein. Die kombinierte Charakterisierung dieser Populationen in Verbindung mit Genexpressionsanalysen kann ein umfassendes Verständnis dafür liefern, wie die zelluläre hämatopoetische Mikroumgebung die Initiierung und das Fortschreiten der Krankheit unterstützt (Abbildung 1). Während sich das unten beschriebene Protokoll auf ein retroviral induziertes AML-Modell und ein genetisches MDS-Modell konzentriert, können diese Strategien leicht angepasst werden, um Veränderungen in der Knochenmarknische jedes Mausmodells von Interesse zu untersuchen.
Protokoll
Alle Tierversuche wurden in Übereinstimmung mit Protokollen durchgeführt, die vom University of Rochester University Committee on Animal Resources genehmigt wurden. Mäuse wurden in den Tierpflegeeinrichtungen der University of Rochester gezüchtet und gepflegt. Zur Modellierung des Hochrisiko-MDS wird das kommerziell erhältliche NHD13-Mausmodell19 verwendet. In diesem Modell werden Knochenmarkstromazellen in weiblichen NHD13-Mäusen im Alter von 8 Wochen vor dem Ausbruch der Krankheit analysiert. De novo AML wird wie zuvor beschrieben 6,11,20 erzeugt. Die Onkogene, die zur Induktion von AML verwendet werden, wie MLL-AF9 und NRas, sind mit GFP oder YFP markiert, was die Analyse der nicht-leukämischen GFP-Knochenmarkpopulationen mittels Durchflusszytometrie ermöglicht. Kurz gesagt, 10 Wochen alte weibliche C57BL/6J-Mäuse werden mit murinen GFP/YFP+ AML-Zellen transplantiert, und das Knochenmark wird 2 Wochen nach der Transplantation entnommen. Während in dieser Studie weibliche Mäuse zu Demonstrationszwecken verwendet werden, kann dieses Protokoll sowohl mit männlichen als auch mit weiblichen Mäusen durchgeführt werden. Sie kann auch entweder mit einem Oberschenkelknochen oder mit allen Röhrenknochen durchgeführt werden.
1. Entnahme des Knochenmarks
HINWEIS: Einzelheiten zum Präparationsprotokoll für Tiere finden Sie in Amend et al.26.
- Reinigen Sie den Mörser und den Stößel mit 70%igem Ethanol, spülen Sie sie mit gekühltem FACS-Puffer ab (Tabelle 1) und legen Sie sie zum Abkühlen auf Eis, bevor Sie mit der Ernte beginnen. Platzieren Sie außerdem den MACs-Puffer (Tabelle 1) auf der Bank, damit er Raumtemperatur erreichen kann.
- Euthanasieren Sie das Tier gemäß den institutionellen Richtlinien und Protokollen für die Tierpflege und -verwendung.
- Besprühen Sie die Maus auf dem Arbeitstisch gründlich mit 70%igem Ethanol, bis ihr Fell nass ist. Heben Sie mit einer Pinzette und einer gebogenen Schere die Haut am Bauch an und machen Sie zwei etwa 0,5 mm lange Schnitte auf beiden Seiten der Maus, seitlich vom Bauch. Als nächstes machen Sie einen 0,5 mm langen Schnitt distal vom Bauch. Ziehe nach unten, um die Haut und das Fell von den Beinen der Maus zu entfernen.
- Setzen Sie die Schere senkrecht zum Becken auf, drücken Sie sie nach unten, während Sie den Oberschenkelknochen mit einer Pinzette nach oben ziehen. Der Hüftkopf sollte sich vom Becken lösen. Femur und Tibia am patellofemoralen Gelenk trennen. Legen Sie die Knochen in FACS-Puffer in eine 6-Well-Platte auf Eis.
- Entfernen Sie Gewebe aus den Knochen mit Gewebe in Laborqualität und legen Sie die gereinigten Knochen in eine neue 6-Well-Platte mit frischem FACS-Puffer auf Eis.
- Geben Sie alle Knochen mit 2-5 ml FACS-Puffer in den Mörser, so dass alle Knochen in den Puffer eingetaucht sind (passen Sie das Volumen des Puffers an, je nachdem, wie viele Knochen Sie verarbeiten). Zerkleinern und zerkleinern Sie die Knochen mit dem Stößel in kreisenden Bewegungen, bis das Knochenmarkgewebe freigesetzt wird.
- Homogenisieren Sie das Knochenmark mit einer 3-ml-Spritze, indem Sie die Flüssigkeit aus dem Mörser herausziehen und herunterspülen.
- Ziehen Sie die Flüssigkeit mit einer 3-ml-Spritze aus dem Mörser und filtern Sie sie durch ein 70-μm-Zellsieb in ein 50-ml-Röhrchen auf Eis. Spülen Sie die Knochen-/Gewebestücke aus dem Filter mit FACS-Puffer zurück in den Mörtel und kehren Sie zu Schritt 1.7 zurück, um sie ein zweites Mal zu homogenisieren und abzuseihen. Dies stellt die Knochenmarkfraktion dar.
- Spülen Sie die verbleibenden Knochenstücke (Spikulen) mit FACS-Puffer zurück in den Mörtel und spülen Sie sie mit FACS-Puffer in ein 15-ml-Röhrchen, um eine maximale Zellausbeute zu gewährleisten. Dies ist die Knochenspikulenfraktion.
2. Verdauung des Knochenmarks
- Das Knochenmark wird bei 300 x g 5 min bei 4 °C zentrifugiert. Umfüllen und den Überstand verwerfen.
- Das Knochenmark wird in 2 ml der Knochenmarkverdauungsmischung (Tabelle 1) resuspendiert und in ein 15-ml-Röhrchen überführt. Bei 37 °C für 45 min auf einem Rotator inkubieren.
- Fügen Sie 10 ml FACS-Puffer hinzu, um die enzymatische Verdauung zu stoppen. Filtern Sie die Mischung durch ein 70-μm-Zellsieb in ein neues 50-ml-Röhrchen.
- Die Mischung wird bei 400 x g für 7 min bei 4 °C pelletiert.
- Resuspendieren Sie das Knochenmarkpellet in 1 ml Erythrozyten-Lysepuffer (siehe Materialtabelle). 4 Minuten auf Eis inkubieren.
- Fügen Sie 10 ml FACS-Puffer hinzu, um die Lyse zu stoppen. Filtern Sie die Mischung durch ein 70-μm-Zellsieb in ein neues 50-ml-Röhrchen.
- Die Mischung wird bei 300 x g für 5 min bei 4 °C pelletiert. Entfernen Sie den Überstand und resuspendieren Sie das Pellet in 100 μl FACS-Puffer.
3. Verdauung von Knochenspikulen
- Die Knochenspikulen aus Schritt 1.9 vortexen und absetzen lassen. Dekantieren Sie den Überstand und belassen Sie den Knochen an der Unterseite.
- Resuspendieren Sie die Knochenspikulen in 1 ml der Knochenspikulationsaufschlussmischung (Tabelle 1).
- Die Röhrchen für 60 min bei 37 °C auf einen Röhrchenrotator legen.
- Fügen Sie 10 ml FACS-Puffer hinzu, um die enzymatische Verdauung zu stoppen. Die Mischung wird durch ein 70-μm-Zellsieb in das 50-ml-Röhrchen mit Erythrozyten-lysiertem und verdautem Knochenmark filtriert.
4. Färbung
- Die Knochenspikulen und die Knochenmarkzellsuspension vorsichtig mischen.
- Verwenden Sie 10 μl der Zellsuspension, um die Anzahl der lebenden Zellen auf einem Hämozytometer unter Verwendung einer 0,4%igen Färbung auf Trypanblaubasis zu zählen, wie in den veröffentlichten Protokollen27 beschrieben. Sammeln Sie 50.000 Zellen für eine ungefärbte Kontrolle aus der Zellsuspension.
- Die restlichen Zellen werden bei 300 x g für 5 min bei 4 °C zentrifugiert. Entfernen Sie den Überstand und suspendieren Sie ihn in 100 μl FACS-Puffer.
HINWEIS: Antikörper können titriert werden, um die ideale Verdünnung zu bestimmen. Die Auswahl der Antikörper (Epitope und Fluorochrome) kann individuell angepasst werden. - Für die Färbung mit Antikörpern gegen magnetische Depletion fügen Sie FC Block (1 μl pro 25 x 106 Zellen), CD45-APC (10 μl pro 25 x 106 Zellen) und Ter119-APC (4 μl pro 25 x 106 Zellen) hinzu (siehe Materialtabelle).
- 20 Minuten auf Eis inkubieren. Mit FACS-Puffer waschen, 50.000 Zellen (~50 μl) für die APC-gefärbte Kontrolle entnehmen, 5 Minuten bei 4 °C bei 300 x g zentrifugieren und in 100 μl FACS-Puffer resuspendieren.
- Für die Färbung der Zellsuspension mit Mikrokügelchen für die magnetische Depletion fügen Sie mIgG (8 μl pro 25 x 106 Zellen) und Anti-APC-Mikrokügelchen (20 μl pro 25 x 106 Zellen) hinzu (siehe Materialtabelle).
- 20 Minuten auf Eis inkubieren. Mit 10 ml FACS-Puffer waschen, bei 300 x g 5 min bei 4 °C zentrifugieren.
5. Verarmung der Probe durch magnetische Sortierung
HINWEIS: Dieser Schritt wird mit einem handelsüblichen manuellen Magnetabscheider gemäß den Anweisungen des Herstellers durchgeführt. Dieser Schritt kann auch mit einem automatischen Separator durchgeführt werden (siehe Materialtabelle).
- Bereiten Sie die LD-Säule vor, indem Sie sie mit 2 ml MACs-Puffer waschen (Tabelle 1). Entsorgen Sie das Abwasser und wechseln Sie das Auffangröhrchen.
- Resuspendieren Sie bis zu 1 x 108 Zellen in MACs-Puffer und filtern Sie sie durch ein 5-ml-Reagenzglas mit einer 35-μm-Zellsiebkappe.
- Stellen Sie die LD-Säule auf den Magnetabscheiderständer. Positionieren Sie ein 5-ml-Reagenzglas unter der Säule, um das Eluat aufzufangen.
- Die Zellsuspension wird in die vorbereitete LD-Säule gegeben. Lassen Sie die negative Fraktion in das Sammelröhrchen durchfließen. Waschen Sie die Säule zweimal mit 1 ml MACs-Puffer und sammeln Sie das Eluat im selben Röhrchen. Dies ist der negative Bruch, der in Schritt 5.6 unten verwendet wird.
- Nehmen Sie die LD-Säule vom Magnetabscheiderständer und stellen Sie sie auf ein neues 5-ml-Reagenzglas. Verwenden Sie eine Pipette, um 3 ml Puffer in die Säule zu geben, um positiv markierte Zellen mit dem Säulenkolben auszuspülen.
- Die negativen und positiven Fraktionen werden bei 300 x g für 5 min bei 4 °C zentrifugiert. Resuspendieren Sie sie in 100 μl FACS-Puffer.
- Zählen Sie 10 μl der negativen und positiven Fraktionen mit 0,4 % Trypanblau. Die Volumina für die Osteo-Analyse/Endothel-Panel-Färbung unten basieren auf dieser Zellzahl.
- Verwenden Sie 50.000 lebende Zellen aus der positiven Fraktion für Durchflusszytometrie-Gating-Kontrollen.
6. Osteo-Analyse/Endothel-Panel-Färbung
HINWEIS: Die Kompensation sollte nach Standard-Durchflusszytometrieprotokollen durchgeführt werden, einschließlich aller geeigneten Färbe- und Gating-Kontrollen.
- Für die Färbung der CD45/Ter119-negativen Fraktion (1 μl jedes Antikörpers pro 1 x 106 Zellen) fügen Sie CD31-PE-Cy7, Sca-1-BV421, CD51-PE und CD140a-PE-Cy5 hinzu (siehe Materialtabelle).
- 20 Minuten auf Eis inkubieren. Mit 2 ml FACS-Puffer waschen, dann bei 300 x g 5 min bei 4 °C zentrifugieren.
- Fügen Sie 1 ml FACS-Puffer und eine 1:1000-Verdünnung PI für die Lebend-/Totfärbung hinzu und filtrieren Sie die Probe dann durch ein 5-ml-Reagenzglas mit einer 35-μm-Zellsiebkappe.
- Analysieren Sie die Zellen auf einem mehrfarbigen Durchflusszytometer.
Ergebnisse
Dieser Artikel beschreibt eine auf Durchflusszytometrie basierende Methode zur Analyse von Mikroumgebungspopulationen des Knochenmarks, wie z. B. der endothelialen und mesenchymalen Stromazellen, aus MDS- und Leukämie-Mausmodellen (Abbildung 1). Abbildung 2 zeigt die Gating-Strategie zur Detektion von Populationen von Interesse, beginnend mit der Selektion der Zellen (P1) in der verdauten und CD45/Ter119-abgereicherten Fraktion durch Vorwärts- und Seitenstreuprofil. Beispiele für das Gating von Zellen in einer Leukämieprobe sind in P1 (Abbildung 2A) dargestellt. Singuletts werden selektiert und Dubletten werden von dieser Analyse ausgeschlossen, P2 (Abbildung 2B). Abbildung 2C zeigt Gatter zur Selektion von Propidiumjod-negativen lebenden Zellen, P3. Um sich auf nicht-hämatopoetische Stromapopulationen zu konzentrieren, sollten Zellen, die CD45-/Ter119-, P4 (APC, Y-Achse) vs. SSC-A ausgewählt sind (Abbildung 2D). Diese anfängliche Gating-Strategie ist für alle zu analysierenden Proben üblich.
Das Leukämie-Mausmodell wird verwendet, um das Gating für Endothelzellen in den Abbildungen 2E-G zu veranschaulichen. Abbildung 2E zeigt CD31 positiv (Pe-Cy7, Y-Achse) vs. SSC-A-Zellen in einer Nicht-Krebs-Kontrollmaus sowie in einer leukämischen Maus, die durch P4 gesteuert wurde. Zellen, die positiv mit CD31 markiert sind, sind Endothelzellen, P5. In Abbildung 2F, die durch P5 gesteuert wird, werden arterioläre Endothelzellen als CD45-Ter119-CD31+Sca1+, P6 und sinusförmige Endothelzellen als CD45-Ter119-CD31+Sca1-, P7 (BV421, Y-Achse) vs. SSC-A.
Obwohl sich diese Analyse auf nicht-leukämische Mikroumgebungszellen des Knochenmarks konzentriert, ist sie auch hilfreich, um die Tumorlast zu bestimmen. Dies kann mit einem kleinen Bruchteil der unverdauten/nicht erschöpften Probe vor Beginn des Experiments erfolgen. In dieser experimentellen Kontrolle (gestrichelte Linien) Abbildung 2G repräsentiert P8 die Tumorlast in der Probe und P9 die nicht-krebsartigen Zellen.
Das MDS-Mausmodell wird verwendet, um die Analyse von mesenchymalen Stromazellen in Abbildung 2H-J zu veranschaulichen. Abbildung 2H zeigt CD31 negativ (Pe-Cy7, Y-Achse) vs. SSC-A-Zellen, angesteuert durch P4. Mesenchymale Stromazellpopulationen in einer Wildtyp-Kontrollmaus und einer MDS-Maus sind in Abbildung 2I dargestellt. Gesteuert durch P10 werden mesenchymale Stromazellen als CD45-Ter119-CD31-CD51+CD140a+, P113 (Pe, Y-Achse; Pe-Cy5, X-Achse). Die experimentelle Kontrolle (gestrichelte Linien), Abbildung 2J zeigt Beispiele für einfarbige Gating-Kontrollen, CD51-Einzelfärbung und CD140a-Einzelfärbung, die als Gate für die MSCs verwendet werden, die in den experimentellen Proben in Abbildung 2I gezeigt sind.
Diese Daten deuten darauf hin, dass sich arterioläre Endothelzellen in der AML-Mikroumgebung signifikant ausdehnen, mit einem gleichzeitigen Verlust in den sinusförmigen Endothelpopulationen (Abbildung 2F), was mit früheren Studien mit patienteneigenen Xenotransplantaten in immundefizienten Mäusen übereinstimmt28. Es ist wahrscheinlich, dass die geringe Ausdehnung der mesenchymalen Stromazellen, die bei den NHD13-Mäusen im Alter von 8 Wochen beobachtet wurde (Abbildung 2I), im Alter von 16-20 Wochen zunehmen kann, wenn diese Mäuse beginnen, Merkmale von MDS25 zu zeigen. Obwohl nur das Leukämiemodell zur Veranschaulichung der Endothelzellpopulation und das MDS-Mausmodell zur Veranschaulichung der mesenchymalen Stromazellpopulationen verwendet wird, können ähnliche Färbe- und Gating-Strategien verwendet werden, um die verschiedenen Mikroumgebungspopulationen in jedem dieser Modelle oder in jedem genetisch veränderten Mausmodell von Interesse zu analysieren.
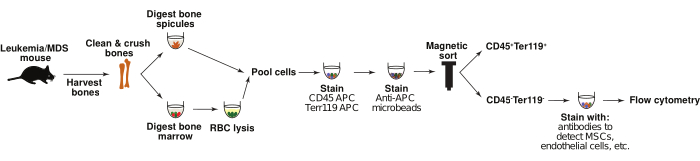
Abbildung 1: Isolierung von Stromazellen des Knochenmarks. Das Schema zeigt den Prozess der Isolierung von nicht-hämatopoetischen Knochenmarkstromazellen aus Kontroll- und leukämischen Mäusen. Kurz gesagt, Knochenspikule und Knochenmark werden getrennt voneinander verdaut und dann gepoolt. Die CD45-Ter119-Population wird durch magnetische Sortierung angereichert und mit Antikörper-Panels gegen Populationen von Interesse, wie mesenchymale Stromazellen und Endothelzellen, gefärbt. Anschließend werden die Zellen mittels Durchflusszytometrie analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
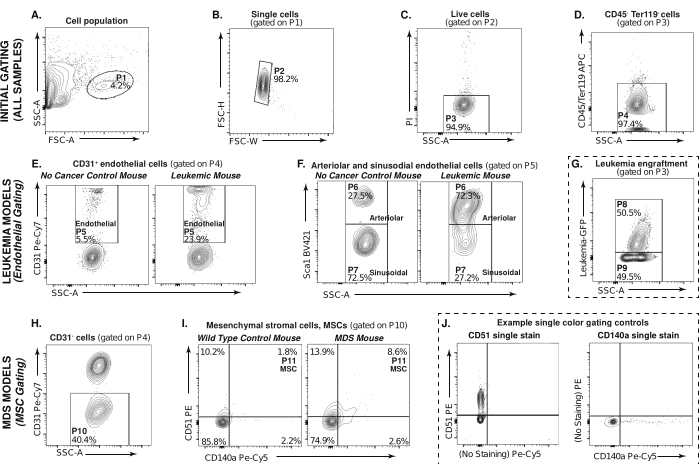
Abbildung 2: Gating-Strategie für nicht-hämatopoetische Knochenmark- und Stromazellen. (A-D) Durchflusszytometrie-Gating-Strategie zur Selektion von verdauten, CD45-/Ter119-, nicht-hämatopoetischen Populationen (P4) sowohl im Leukämie- als auch im MDS-Mausmodell. (E) Leukämie-Knochenmark-Endothelzellen (P5, CD31+) können unterteilt werden in (F) Sca1+ arteriolä (P6) oder Sca1- sinusoidale Endothelzellen (P7). (G) Leukämie-Transplantation in eine AML-Probe, wobei P8 für Transplantation und P9 für Nicht-Krebszellen steht. Gestrichelte Linien zeigen an, dass es sich bei diesem Diagramm um eine experimentelle Kontrolle handelt. (H) Gated durch P4, MDS-Knochenmark-CD31-Zellen, P10 (I) Mesenchymale Stromazellen (CD51+/CD140a+, P11). (J) Beispiel für einfarbige Gating-Kontrollen, CD51 (links) und CD140a (rechts), die zur Bestimmung der Gates für mesenchymale Stromazellen in (I) verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Lösung | Reagenz | Konzentration | Hinzuzufügender Betrag |
| FACS-Puffer | HBSS | 10-fach | 100 ml |
| EDTA | 0,5 Mio. | 4 ml | |
| Fötales Kälberserum | - | 50 ml | |
| Wasser | - | 848 ml | |
| Knochenmarkverdauungsmischung | HBSS | 1-fach | 2 ml |
| DNase 1 | 1 μg/ml in 1x DPBS | 20 μl | |
| Dispase II Pulver | - | 4 mg | |
| Kollagenase Typ IV | - | 2 mg | |
| Knochen-Spicule-Verdauungsmischung | DPBS | 1-fach | 320 ml |
| Kollagenase Typ 1 | - | 1 g | |
| Fötales Kälberserum | - | 80 ml | |
| MACs-Puffer | BSA | 66 g/mol | 5 g |
| DPBS | 10-fach | 100 ml | |
| EDTA | 0,5 Mio. | 4 ml | |
| Wasser | - | 896 L |
Tabelle 1: Zusammensetzung der in der vorliegenden Studie verwendeten Lösungen und Puffer.
Diskussion
Maus-Leukämiemodelle wurden in großem Umfang verwendet, um zellintrinsische und nischengesteuerte Signale zu identifizieren, die das Fortschreiten der aggressiven myeloischen Leukämie fördern 6,19,21. In dieser Arbeit wird ein umfassendes auf Durchflusszytometrie basierendes Protokoll zur Bestimmung der zellulären Zusammensetzung der Knochenmarkmikroumgebung in murinen Modellen von MDS und AML vorgestellt.
Vor der Erfassung durchflusszytometrischer Daten aus experimentellen Proben ist es wichtig, die Fluoreszenzüberlappung sorgfältig zu kompensieren. Es ist auch wichtig, alle geeigneten Färbe- und Anschnittkontrollen einzubeziehen. Diese Schritte ermöglichen es dem Versuchsleiter, zu bestätigen, dass die Färbung positiver oder negativer Antikörper eine genaue Expression von Zellmarkern von Interesse darstellt und kein Artefakt der Fluoreszenzspektralüberlappung oder Autofluoreszenz ist. Obwohl dieses Protokoll ausgewählte Zelloberflächenmarker beschreibt, können die Antikörper-Panels je nach experimentellem Bedarf erweitert werden. Zum Beispiel kann CD144 (Ve-Cadherin) in vivo vor der Entnahme der Mäuse verabreicht werden und als zusätzlicher spezifischer Marker für Endothelzellen dienen 5,29. Während die hier angegebenen Fluorophore und Antikörperklone verändert werden können, sollten Titrationen durchgeführt werden, um ihre ideale Verdünnung zu bestimmen. Um alle Zellpopulationen in der Knochenmarknische kategorisch zu definieren, kann die Einzelzell-RNA-Sequenzierung verwendet werden, um die Knochenmarknischenlandschaft während der MDS/AML-Initiierung und -Progression zu bestimmen 23,24,30.
Es ist wichtig, die Probe sorgfältig gemäß den in diesem Protokoll beschriebenen Schritten vorzubereiten. Bei der Homogenisierung der Knochen ist es entscheidend, dass Mörser und Stößel gekühlt sind und alle Schritte auf Eis durchgeführt werden, um den Zelltod zu verhindern und eine hohe Zellregeneration zu gewährleisten. Es ist sehr wichtig, vor dem Homogenisieren das gesamte Gewebe um die Knochen herum zu entfernen, um eine Kontamination anderer unerwünschter Zelltypen zu verhindern. Während der Erythrozytenlyse und der Verdauung ist es wichtig, die enzymatische Reaktion mit einer angemessenen Menge FACS-Puffer zu stoppen und gründlich in frischem Puffer zu resuspendieren, da die Zellen sonst weiter verdaut werden und schließlich absterben.
Die Trennung der Knochen- und Knochenmarkfraktionen ist unerlässlich, da die Knochenspikulen eine andere Mischung von Enzymen für den Verdauungspuffer benötigen und eine längere Zeit für die Verdauung benötigen. Bei der Knochenverdauung ist die Kollagenase Typ 1 nützlich für die Verdauung von Kollagenase-Fibrillen, die häufig in extrazellulärer Matrix und Kollagenfasern vorkommen31. Darüber hinaus bleiben einige Knochenmarkzellen, die sich in der Nähe des Endosteums befinden, nach der Homogenisierung am Knochen haften und werden erst durch enzymatische Verdauung freigesetzt. Bei der Verdauung von Knochenmark wird Kollagenase Typ IV verwendet, um die Basalmembran von Epithel- und Endothelzellen im Knochenmark zu verdauen32, während Dispase hauptsächlich Fibronektin31 spaltet. Das Knochenmark benötigt weniger Zeit für die Verdauung, da die Assoziationen zwischen Zellen und Matrix schwächer sind. Die Inkubation der Knochenmarkfraktion unter ähnlichen Bedingungen kann die interessierenden Populationen schädigen. Die Verwendung von zwei verschiedenen Aufschlusspuffern erhöht die Anzahl der zu erkennenden Stomapopulationen erheblich und bietet somit einen größeren Datensatz für die Analyse.
Die meisten verfügbaren Protokolle zur Analyse von Mikroumgebungen im Knochenmark von Mäusen verwenden eine Spritze, um das Knochenmark nur aus den Röhrenknochen zu spülen13,24. Die derzeitige Methode zum Zerkleinern von Knochen bietet die Möglichkeit, Daten von anderen Knochen wie dem Becken zu erfassen, die Probenvorbereitungszeit erheblich zu verkürzen und die Möglichkeit von Verletzungen durch scharfe Nadeln zu verringern. Angesichts der Tatsache, dass der Knochen aus reifen Osteoblasten und anderen Zellpopulationen besteht, die normale und bösartige hämatopoetische Zellen unterstützen können, ermöglicht diese Methode des Einschlusses von Zellen aus den Knochenspikulen eine genauere Darstellung der Mikroumgebung des Knochenmarks in der Probe. Während in einer früheren Studie Knochenmark und Knochenspikula getrennt verdaut wurden11, konnte keine Färbung von Osteolineage und Endothelzellen in derselben Probe nachgewiesen werden. In einer zweiten Studie wurden kommerziell erhältliche proprietäre Enzymmischungen zur Verdauung von Knochenmark und Knochenspikulenverwendet 23 und die Daten mit der deutlich teureren Einzelzell-RNA-Sequenzierungstechnologie analysiert. Diese Methode der Anreicherung für CD45-/Ter119-Zellen selektiert die interessierenden Zellen und reduziert die Zeit, die für die Datenerfassung auf dem Durchflusszytometer benötigt wird, erheblich. Im Vergleich zu anderen hochmodernen Methoden wie der Einzelzell-RNA-Sequenzierung ist diese auf Durchflusszytometrie basierende Methode zugänglicher, kostengünstiger und erfordert keine ausgefeilte Analyse durch ausgebildete Bioinformatiker 23,24,30.
Es ist wichtig zu beachten, dass dieses Protokoll verwendet werden kann, um die Mikroumgebung des Knochenmarks nicht nur von jedem der verfügbaren Mausmodelle für MDS und AML, sondern von jedem genetischen Mausmodell zu charakterisieren. Ähnliche Methoden können auch bei der Analyse der Veränderungen in der murinen Knochenmarknische von patientenabgeleiteten Xenograft-Modellen (PDX) wirksam sein. Diese Methoden können für Studien nützlich sein, die darauf abzielen, die Mechanismen zu bestimmen, durch die MDS/AML ihre Knochenmarknische beeinflussen. Angesichts der technischen Herausforderungen, die mit der Entnahme großer Mengen Knochenmark von menschlichen Patienten verbunden sind, sind diese Analysen von Mausmodellen ein effektives Werkzeug, um das Verständnis der malignen Knochenmarkmikroumgebung zu verbessern und ihre Rolle beim Fortschreiten der Krankheit zu definieren.
Offenlegungen
Es wurden keine Interessenkonflikte deklariert.
Danksagungen
Wir bedanken uns beim URMC Flow Cytometry Core. Diese Arbeit wurde durch den American Society of Hematology Scholar Award, den Preis der Leukemia Research Foundation und NIH-Stipendien R01DK133131 und R01CA266617 unterstützt, die an J.B.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 mL pipette Tips | Genesee Scientific | 24-165RL | |
| 1.7 mL Microcentrifuge Tubes | AVANT | L211511-CS | |
| 10 µL pipette Tips | Genesee Scientific | 24-140RL | |
| 10 mL Individually Wrapped Sterile Serological Pipettes | Globe scientific | 1760 | |
| 1000 mL Vacuum Filtration Flask | NEST | 344021 | |
| 15 mL Centrifuge Tube | VWR | 10026-076 | |
| 2 mL Aspirating Pipette | NEST | 325011 | |
| 200 µL pipette Tips | Genesee Scientific | 24-150-RL | |
| 25 mL Individually Wrapped Sterile Serological Pipettes | Globe scientific | 1780 | |
| 5 mL Individually Wrapped Sterile Serological Pipettes | Globe scientific | 1740 | |
| 5 mL Polystyrene Round-Bottom Tube 12 x 75 mm style | Falcon | 352054 | |
| 5 mL Polystyrene Round-Bottom Tube with Cell Strainer Cap 12 x 75 mm style | Falcon | 352235 | |
| 50 mL Centrifuge Tube | NEST | 602052 | |
| 6 Well, Flat Bottom with Low Evaporation Lid | Falcon | 353046 | |
| Absorbent Underpads with Waterproof Moisture Barrier | VWR | 56616-031 | |
| APC MicroBeads | Miltenyi | 130-090-855 | |
| autoMACS Pro Separator | Miltenyi Biotec GmBH | 4425745 | |
| BD Pharmingen Purified Rat Anti-Mouse CD16/CD32 (Mouse BD Fc Block) | BD Biosciences | 553141 | 0.5 mg/mL |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | 66.000 g/mol |
| Brilliant Violet 421 anti-mouse Ly-6A/E (Sca-1) Antibody (D7) | Invitrogen | 404-5981 | 0.2 mg/mL |
| C57BL/6J Mice | Jackson Laboratory | 664 | |
| Carbon Dioxide Gas Tank | Airgas | CD50 | |
| CD31 (PECAM-1) Monoclonal Antibody (390), PE-Cyanine7 | Invitrogen | 25-0311-82 | 0.2 mg/mL |
| CD45 Monoclonal Antibody (30-F11), APC | Invitrogen | 17-0451-82 | 0.2 mg/mL |
| Cell Strainer 70 µm Nylon | Falcon | 352350 | |
| Cole-Parmer Essentials Mortar and Pestle; Agate, 125 mL | Cole-Parmer | EW-63100-62 | |
| Collagenase from Clostridium histolyticum | Sigma-Aldrich | C5138-500MG | |
| Collagenase Type I | STEMCELL | 7415 | |
| Corning Mini Centrifuge | CORNING | 6770 | |
| Corning Stripettor Ultra Pipet Controller | Corning | 4099 | |
| Deoxyribonuclease I from bovine pancreas | Sigma-Aldrich | D4513 | |
| Dispase II, powder | Gibco | 117105041 | |
| DPBS 10x | gibco | 14200-075 | |
| eBioscience 1x RBC Lysis Buffer | Invitrogen | 00-4333-57 | |
| Ethanol absolute, KOPTEC, meets analytical specification of BP, Ph. Eur., USP (200 Proof) | VWR | 89125-174 | |
| Fine scissors - sharp | Fine Science Tools | 14061-10 | |
| Foundation B Fetal Bovine Serum | GeminiBio | 900-208 | |
| Gilson PIPETMAN L Pipette Starter Kits | FisherScientific | F167370G | |
| Graefe Forceps | Fine Science Tools | 11051-10 | |
| Hank's Balanced Salt Solution (HBSS) 10x | gibco | 14185-052 | |
| Hemocytometer | Fisher | 02-671-10 | |
| Incubator | BINDER | C150-UL | |
| Kimwipes | KIMTECH | K222101 | |
| LABGARD Class II, Type A2 Biological Safety Cabinet | Nuaire | NU-425-400 | |
| LD Columns | Miltenyi Biotec GmBH | 130-042-901 | |
| LSE Vortex Mixer | CORNING | 6775 | |
| LSRII/Fortessa/Symphony A1 | Becton, Dickinson and Company | 647800L6 | |
| MACS MULTI STAND | Miltenyi Biotec GmBH | 130-042-303 | |
| MACsmix Tube Rotator | Miltenyi Biotec GmBH | 130-090-753 | |
| mIgG | Millipore-Sigma | 18765-10mg | 2 mg/mL |
| Nup98-HoxD13 (NHD13) Mice | Jackson Laboratory | 010505 | |
| PE anti-mouse CD51 Antibody (RMV-7) | Biolegend | 104106 | 0.2 mg/mL |
| PE/Cyanine5 anti-mouse CD140a Antibody (RUO) | Biolegend | 135920 | 0.2 mg/mL |
| Penicillin-Streptomycin | Gibco | 15140122 | 10,000 U/mL |
| Plastipak 3 mL Syringe | Becton, Dickinson and Company | 309657 | |
| Propidium Iodide - 1.0 mg/mL Solution in Water | ThermoFisher Scientific | P3566 | |
| QuadroMACS Separator | Miltenyi Biotec GmBH | 130-090-976 | |
| Sorvall X Pro / ST Plus Series Centrifuge | Thermo Scientific | 75009521 | |
| TER-119 Monoclonal Antibody (TER-119), APC | Invitrogen | 17-5921-82 | 0.2 mg/mL |
| Trypan Blue Solution 0.4% | Gibco | 15-250-061 | |
| Ultrapure 0.5 M EDTA, pH 8.0 | Invitrogen | 15575-038 |
Referenzen
- Morrison, S. J., Scadden, D. T. The bone marrow niche for haematopoietic stem cells. Nature. 505 (7483), 327-334 (2014).
- Boulais, P. E., Frenette, P. S. Making sense of hematopoietic stem cell niches. Blood. 125 (17), 2621-2629 (2015).
- Pinho, S., Frenette, P. S. Haematopoietic stem cell activity and interactions with the niche. Nat Rev Mol Cell Biol. 20 (5), 303-320 (2019).
- Kfoury, Y., Scadden, D. T. Mesenchymal cell contributions to the stem cell niche. Cell Stem Cell. 16 (3), 239-253 (2015).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Bajaj, J., et al. Cd98-mediated adhesive signaling enables the establishment and propagation of acute myelogenous leukemia. Cancer Cell. 30 (5), 792-805 (2016).
- Konopleva, M. Y., Jordan, C. T. Leukemia stem cells and microenvironment: Biology and therapeutic targeting. J Clin Oncol. 29 (5), 591-599 (2011).
- Kim, Y. W., et al. Defective notch activation in microenvironment leads to myeloproliferative disease. Blood. 112 (12), 4628-4638 (2008).
- Walkley, C. R., et al. A microenvironment-induced myeloproliferative syndrome caused by retinoic acid receptor gamma deficiency. Cell. 129 (6), 1097-1110 (2007).
- Kode, A., et al. Leukaemogenesis induced by an activating β-catenin mutation in osteoblasts. Nature. 506 (7487), 240-244 (2014).
- Hanoun, M., et al. Acute myelogenous leukemia-induced sympathetic neuropathy promotes malignancy in an altered hematopoietic stem cell niche. Cell Stem Cell. 15 (3), 365-375 (2014).
- Raaijmakers, M. H., et al. Bone progenitor dysfunction induces myelodysplasia and secondary leukaemia. Nature. 464 (7290), 852-857 (2010).
- Frisch, B. J., et al. Functional inhibition of osteoblastic cells in an in vivo mouse model of myeloid leukemia. Blood. 119 (2), 540-550 (2012).
- Bajaj, J., Diaz, E., Reya, T. Stem cells in cancer initiation and progression. J Cell Biol. 219 (1), e201911053(2020).
- Sekeres, M. A., Taylor, J. Diagnosis and treatment of myelodysplastic syndromes: A review. Jama. 328 (9), 872-880 (2022).
- Zeisig, B. B., Kulasekararaj, A. G., Mufti, G. J., So, C. W. Snapshot: Acute myeloid leukemia. Cancer Cell. 22 (5), 698-698.e1 (2012).
- Krivtsov, A. V., Armstrong, S. A. Mll translocations, histone modifications and leukaemia stem-cell development. Nat Rev Cancer. 7 (11), 823-833 (2007).
- Yoshimi, A., et al. Coordinated alterations in rna splicing and epigenetic regulation drive leukaemogenesis. Nature. 574 (7777), 273-277 (2019).
- Lin, Y. W., Slape, C., Zhang, Z., Aplan, P. D. Nup98-hoxd13 transgenic mice develop a highly penetrant, severe myelodysplastic syndrome that progresses to acute leukemia. Blood. 106 (1), 287-295 (2005).
- Kwon, H. Y., et al. Tetraspanin 3 is required for the development and propagation of acute myelogenous leukemia. Cell Stem Cell. 17 (2), 152-164 (2015).
- Bajaj, J., et al. An in vivo genome-wide crispr screen identifies the rna-binding protein staufen2 as a key regulator of myeloid leukemia. Nat Cancer. 1 (4), 410-422 (2020).
- Krivtsov, A. V., et al. Transformation from committed progenitor to leukaemia stem cell initiated by mll-af9. Nature. 442 (7104), 818-822 (2006).
- Baryawno, N., et al. A cellular taxonomy of the bone marrow stroma in homeostasis and leukemia. Cell. 177 (7), 1915-1932.e16 (2019).
- Tikhonova, A. N., et al. The bone marrow microenvironment at single-cell resolution. Nature. 569 (7755), 222-228 (2019).
- Balderman, S. R., et al. Targeting of the bone marrow microenvironment improves outcome in a murine model of myelodysplastic syndrome. Blood. 127 (5), 616-625 (2016).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. JoVE. 110, e53936(2016).
- JoVE Science Education Database. Science Education Database. Basic Methods in Cellular and Molecular Biology. Using a Hemacytometer to Count Cells. , JoVE. (2023).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to acute myeloid leukemia progression and drug response. Blood. 128 (22), 2662(2016).
- Xu, C., et al. Stem cell factor is selectively secreted by arterial endothelial cells in bone marrow. Nat Commun. 9 (1), 2449(2018).
- Baccin, C., et al. Combined single-cell and spatial transcriptomics reveal the molecular, cellular and spatial bone marrow niche organization. Nat Cell Biol. 22 (1), 38-48 (2020).
- Ebrahimi Dastgurdi, M., Ejeian, F., Nematollahi, M., Motaghi, A., Nasr-Esfahani, M. H. Comparison of two digestion strategies on characteristics and differentiation potential of human dental pulp stem cells. Arch Oral Biol. 93, 74-79 (2018).
- Abreu-Velez, A. M., Howard, M. S. Collagen IV in normal skin and in pathological processes. N Am J Med Sci. 4 (1), 1-8 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten