Method Article
Etablierung von Organoiden aus dem menschlichen Zahn als mächtiges Werkzeug für die mechanistische Forschung und regenerative Therapie
In diesem Artikel
Zusammenfassung
Wir präsentieren ein Protokoll zur Entwicklung epithelialer Organoidkulturen ausgehend vom menschlichen Zahn. Die Organoide sind robust expandierbar und rekapitulieren die epithelialen Stammzellen des Zahnes, einschließlich ihrer Ameloblasten-Differenzierungskapazität. Das einzigartige Organoidmodell bietet ein vielversprechendes Werkzeug, um die Biologie der menschlichen Zahnmedizin (Stammzelle) mit Perspektiven für zahnregenerative Ansätze zu untersuchen.
Zusammenfassung
Zähne sind von entscheidender Bedeutung im Leben, nicht nur für den Kauen und die Sprache, sondern auch für das psychische Wohlbefinden. Das Wissen über die Entwicklung und Biologie des menschlichen Zahns ist knapp. Insbesondere ist nicht viel über die epithelialen Stammzellen des Zahnes und ihre Funktion bekannt. Es ist uns gelungen, ein neuartiges Organoidmodell zu entwickeln, das auf menschlichem Zahngewebe (d.h. Zahnfollikel, isoliert aus extrahierten Weisheitszähnen) basiert. Die Organoide sind robust und langfristig erweiterbar und rekapitulieren das vorgeschlagene epitheliale Stammzellkompartiment des menschlichen Zahnes sowohl in Bezug auf die Markerexpression als auch auf die funktionelle Aktivität. Insbesondere sind die Organoide in der Lage, einen Ameloblastendifferenzierungsprozess zu entfalten, wie er in vivo während der Amelogenese abläuft. Dieses einzigartige Organoidmodell wird ein leistungsfähiges Werkzeug zur Untersuchung nicht nur der Entwicklung des menschlichen Zahnes, sondern auch der Zahnpathologie bieten und den Weg für eine zahnregenerative Therapie ebnen. Der Ersatz verlorener Zähne durch einen biologischen Zahn, der auf diesem neuen Organoidmodell basiert, könnte eine attraktive Alternative zur derzeitigen Standardimplantation von synthetischen Materialien sein.
Einleitung
Zähne spielen eine wesentliche Rolle beim Kauen von Lebensmitteln, bei der Sprache und beim psychischen Wohlbefinden (Selbstbild). Der menschliche Zahn besteht aus hochmineralisiertem Gewebe unterschiedlicher Dichte und Härte1. Zahnschmelz, der Hauptbestandteil der Zahnkrone, ist das am höchsten mineralisierte Gewebe im menschlichen Körper. Während der Schmelzbildung (Amelogenese), wenn sich Zähne entwickeln, differenzieren sich zahnepitheliale Stammzellen (DESCs) in schmelzbildende Zellen (Ameloblasten). Einmal gebildet, wird der Zahnschmelz aufgrund des apoptotischen Verlustes der Ameloblasten zu Beginn des Zahnausbruchs selten repariert oder erneuert1. Die Wiederherstellung von beschädigtem Schmelzgewebe, wie es durch Trauma oder bakterielle Erkrankungen verursacht wird, wird derzeit mit synthetischen Materialien durchgeführt; Diese sind jedoch mit wichtigen Mängeln wie Mikroleckage, minderwertiger Osseointegration und Verankerung, begrenzter Lebensdauer und fehlender voll funktionsfähiger Reparaturkonfrontiert 2. Daher wäre eine robuste und zuverlässige Kultur menschlicher DESCs mit der Fähigkeit, Ameloblasten zu erzeugen und das Potenzial, mineralisiertes Gewebe zu produzieren, ein großer Schritt vorwärts im Bereich der dentalen Regenerative.
Das Wissen über den menschlichen DESC-Phänotyp und die biologische Funktion ist knapp 3,4,5. Interessanterweise wurde vorgeschlagen, dass DESCs menschlicher Zähne in den Epithelzellresten von Malassez (ERM) existieren, Zellclustern, die im Zahnfollikel (DF) vorhanden sind, der nicht durchgebrochene Zähne umgibt und im Parodontalband um die Wurzel vorhanden bleibt, sobald der Zahn ausbricht1. Es wurde festgestellt, dass ERM-Zellen, die mit Zahnpulpa kokultiviert werden, sich in ameloblastenähnliche Zellen differenzieren und schmelzähnliches Gewebeerzeugen 6. Tiefgreifende Studien über die spezifische Rolle von ERM-Zellen bei der (Re-) Erzeugung von Zahnschmelzzellen waren jedoch aufgrund des Mangels an zuverlässigen Studienmodellen begrenzt7. Aktuelle ERM-In-vitro-Kultursysteme werden durch eine begrenzte Lebensdauer und einen schnellen Verlust des Phänotyps unter den standardmäßig verwendeten 2D-Bedingungen 8,9,10,11,12 behindert. Daher ist ein handhabbares In-vitro-System zur originalgetreuen Erweiterung, Untersuchung und Differenzierung menschlicher DESCs dringend erforderlich.
In den letzten zehn Jahren wurde eine leistungsfähige Technik zur Züchtung epithelialer Stammzellen in vitro erfolgreich auf verschiedene Arten von (menschlichen) Epithelgeweben angewendet, um ihre Biologie sowie die Krankheit13,14,15,16 zu untersuchen. Diese Technologie ermöglicht es den epithelialen Stammzellen des Gewebes, sich selbst zu 3D-Zellkonstruktionen (d. H. Organoiden) zu entwickeln, wenn sie in ein extrazelluläres Matrix (ECM) -imitierendes Gerüst (typischerweise Matrigel) gesät und in einem definierten Medium kultiviert werden, das die Stammzell-Nischensignalisierung des Gewebes und / oder die Embryogenese repliziert. Typische Wachstumsfaktoren, die für die Organoidentwicklung benötigt werden, sind der epidermale Wachstumsfaktor (EGF) und die wingless-Typ MMTV Integration Site (WNT) Aktivatoren14,15,16. Die resultierenden Organoide zeichnen sich durch dauerhafte Genauigkeit bei der Nachahmung der ursprünglichen Epithelstammzellen des Gewebes sowie durch eine hohe Erweiterbarkeit unter Beibehaltung ihres Phänotyps und ihrer funktionellen Eigenschaften aus, wodurch die oft begrenzte primäre Verfügbarkeit von menschlichem Gewebe, wie sie in der Klinik erworben wurde, überwunden wird. Um Organoide zu etablieren, ist eine Isolierung der epithelialen Stammzellen aus dem heterogenen Gewebe (d.h. bestehend aus anderen Zelltypen wie mesenchymalen Zellen) vor der Kultivierung nicht erforderlich, da mesenchymale Zellen nicht an das ECM binden oder darin gedeihen, was schließlich zu rein epithelialen Organoiden führt 13,16,17,18,19 . Diese vielversprechende und vielseitige Technologie hat zur Entwicklung vielfältiger Organoidmodelle aus verschiedenen menschlichen Epithelgeweben geführt. Aus menschlichem Zahn gewonnene Organoide, die für die eingehende Untersuchung der Zahnentwicklung, -regeneration und -krankheit wertvoll sind, wurden jedoch noch nicht etabliert20,21. Kürzlich ist es uns gelungen, ein solches neues Organoidmodell zu entwickeln, das von DF-Gewebe aus dritten Molaren (Weisheitszähnen) ausgeht, die von jugendlichen Patienten19 extrahiert wurden.
Hier beschreiben wir das Protokoll zur Entwicklung epithelialer Organoidkulturen aus dem erwachsenen menschlichen Zahn (d.h. aus dem DF der dritten Molaren) (Abbildung 1A). Die resultierenden Organoide exprimieren ERM-assoziierte Stammheitsmarker und sind gleichzeitig langfristig erweiterbar. Interessanterweise ist im Gegensatz zu den meisten anderen Organoidmodellen der normalerweise benötigte EGF für eine robuste Organoidentwicklung und -entwicklung redundant. Interessanterweise zeigen die Stammheitsorganoide Ameloblastendifferenzierungseigenschaften, wodurch ERM/DESC-Merkmale und -Prozesse nachgeahmt werden, die in vivo auftreten. Das hier beschriebene neue und einzigartige Organoidmodell ermöglicht die Erforschung der DESC-Biologie, Plastizität und Differenzierungskapazität und öffnet die Tür für die ersten Schritte in Richtung zahnregenerativer Ansätze.
Protokoll
Alle hier beschriebenen Methoden wurden von der Ethikkommission Forschung UZ/KU Leuven (13/0104U) genehmigt. Extrahierte dritte Molaren (Weisheitszähne) wurden nach der informierten Zustimmung der Patienten erhalten.
1. Vorbereitungen
- Eine 48-Well-Kulturplatte für 15-20 h in einem 1,9% CO 2-Inkubator bei 37 °C vorwärmen.
- Verflüssigen Sie ein Matrigel aliquot (Wachstumsfaktor-reduziert; phenolrotfrei; weiter als Basalmembranmatrix bezeichnet; BMM) auf Eis (4 °C) für mindestens 2 h vor Schritt 2.1.
HINWEIS: Vermeiden Sie Frost-/Tauzyklen des BMM. Das BMM für 15-20 h auf Eis und aliquot bei 1 ml in Mikrozentrifugenröhrchen verflüssigen und bei -20 °C lagern. - Die Zentrifuge wird auf 4 °C abgekühlt.
- Bereiten Sie Medien vor und filtern Sie die Lösung durch einen 0,22-μm-Filter. Die folgenden Bände basieren auf der Sammlung der DFs aus allen vier dritten Molaren, die typischerweise gleichzeitig extrahiert werden.
- Bereiten Sie 20 ml DF-Entnahmemedium vor (Tabelle 1): mindestens essentielles Pigment Eagle (αMEM) mit 10% fetalem Rinderserum (FBS), 0,5% Amphotericin B und 1% Penicillin-Streptomycin. Übertragen Sie 4 ml DF-Sammelmedium auf ein 15-ml-Rohr und übertragen Sie die Röhrchen auf Eis.
HINWEIS: Pro Patient reicht ein 15-ml-Röhrchen für die Probenentnahme (d. h. für vier dritte Molaren) aus. - Machen Sie 20 ml Zahnorganoidmedium (weiter als TOM bezeichnet; Tabelle 2) unter Verwendung des serumfreien definierten Mediums (SFDM; Tabelle 3). TOM maximal 2 Wochen bei 4 °C lagern.
- Es werden 8 ml Dissoziationsmedium (Tabelle 4) hergestellt, d. h. phosphatgepufferte Kochsalzlösung (PBS), die Kollagenase VI (3 mg/ml) und Dispase II (4 mg/ml) enthält. Erwärmen Sie das Dissoziationsmedium vor Gebrauch mindestens 10 min in einem 37 °C warmen Wasserbad und geben Sie es in zwei 15 ml Röhrchen (4 ml pro Röhrchen) um, um vier DF-Gewebe zu dissoziieren.
- Bereiten Sie eine Waschplatte (12-Well) vor, die Folgendes enthält: drei Vertiefungen mit 70% EtOH, drei Vertiefungen mit PBS und drei Vertiefungen mit DF-Sammelmedium.
- Machen Sie 15 ml Medium A pro Probe (d. h. 5 ml pro zwei DFs pro Patienten; Tabelle 5).
HINWEIS: Die folgenden Schritte sollten unter sterilen Bedingungen durchgeführt werden.
- Bereiten Sie 20 ml DF-Entnahmemedium vor (Tabelle 1): mindestens essentielles Pigment Eagle (αMEM) mit 10% fetalem Rinderserum (FBS), 0,5% Amphotericin B und 1% Penicillin-Streptomycin. Übertragen Sie 4 ml DF-Sammelmedium auf ein 15-ml-Rohr und übertragen Sie die Röhrchen auf Eis.
2. Zahnfollikeldissoziation
- Sobald die dritten Molaren mit zugehörigen DFs im Sammelmedium (auf Eis) gesammelt sind, übertragen Sie den Inhalt der Röhrchen in eine Petrischale.
- Halten Sie den Zahn mit einer Pinzette fest und isolieren Sie den DF vorsichtig mit einer chirurgischen Klinge.
HINWEIS: Dieser Schritt erfordert Übung; Seien Sie vorsichtig mit der Klinge. - Um das restliche Blut von den DFs abzuwaschen, legen Sie das DF-Gewebe kurz in die erste Vertiefung von 70% EtOH (Waschplatte) für 20 s und dann für 20 s in die nächste EtOH-Vertiefung und weiter in die dritte EtOH-Vertiefung (maximal 1 min in 70% EtOH insgesamt; längere Inkubationszeit führt zu einer verringerten Zelllebensfähigkeit).
- Als nächstes fahren Sie mit dem Spülen in den drei PBS-Bohrlöchern fort (maximal 2 Minuten insgesamt).
- Spülen Sie die DFs in den drei verbleibenden DF-Auffangmittelvertiefungen (bis zu einem Maximum von insgesamt 20 min).
- Die gespülten DFs in eine neue Petrischale geben.
- Schneiden Sie ein kleines Stück eines der DFs (~5 mm2) zur Paraformaldehyd (PFA)-Fixierung (siehe Abschnitt 7) und lagern Sie es auf Eis (für maximal 6 h) in einem Mikrozentrifugenröhrchen mit 500 μL DF-Sammelmedium bis zur PFA-Fixierung.
- Zerkleinern Sie den Rest der DFs in kleinen Stücken (~ 1 mm2).
HINWEIS: Es wird empfohlen, die frisch isolierten DF-Gewebe sofort für die optimale Organoidbildung und Wachstumseffizienz zu verarbeiten, anstatt mit kryokonserviertem Gewebe zu beginnen, was zu einer geringeren Effizienz führt. Dennoch ist es möglich, primäres DF-Gewebe in diesem Schritt zu kryokonservieren (siehe Kryokonservierungsmedium und -protokoll in Abschnitt 5). - Die gehackten DFs in ein 15-ml-Röhrchen mit 4 ml vorgewärmtem Dissoziationsmedium geben und 2 h in einem 37 °C-Wasserbad inkubieren.
HINWEIS: Fügen Sie zwei DFs pro 4 ml vorgewärmtem Dissoziationsmedium (ein Röhrchen) hinzu, um eine optimale Dissoziation zu gewährleisten. - Alle 15 Minuten das DF-Dissoziationsmedium mit einer Pasteur-Glaspipette auf und ab vermischen, um den Gewebezerfall zu beschleunigen. Wenn DF-Stücke nicht mehr beobachtet werden (normalerweise nach 1 h), fahren Sie mit der DF-Dissoziation mit einer verengten feuerpolierten Pasteur-Pipette fort.
HINWEIS: Für eine optimale DF-Dissoziation wechseln Sie zu einer Pasteur-Pipette, die spätestens 1 h DF-Isolierung durch Feuerpolieren verengt wird.- In der Zwischenzeit werden 10 ml Medium A mit 50 μL DNase (0,2 mg/ml; Tabelle 5).
- Fügen Sie 5 ml Medium A (mit der DNase) zu jedem Röhrchen mit dissoziiertem DF hinzu und inkubieren Sie für 1 min bei Raumtemperatur (RT).
- Filtern Sie die Zellsuspension (die einzelne Zellen und kleine Zellklumpen enthält) durch ein 40-μm-Zellsieb, um die verbleibenden großen Fragmente und (den größten Teil) des faserigen Gewebes zu entfernen.
- Kombinieren Sie die verschiedenen Röhrchen mit dissoziiertem DF von einem Patienten in diesem Schritt.
- Spülen Sie den Filter mit 1 ml Medium A ab. Zentrifugieren Sie die gefilterte Zellsuspension bei 200 x g für 10 min bei 4 °C.
- Entfernen Sie den Überstand, suspendieren Sie das Pellet in 1 ml SFDM (Tabelle 3) und übertragen Sie die Zellsuspension in ein 1,5 ml Mikrozentrifugenröhrchen.
- Berechnen Sie die Zellkonzentration mit einem automatisierten Zellzähler. Vernachlässigen Sie die verbleibenden Zellklumpen.
- Zentrifuge bei 200 x g für 5 min bei 4 °C.
3. Etablierung der Zahnorganoidkultur (Abbildung 1A und Abbildung 2A)
- Berechnen Sie basierend auf der erhaltenen Zellzahl die Anzahl der Vertiefungen, die ausgesät werden können. Ein 20 μL Tröpfchen sollte 20.000 Zellen enthalten. Die endgültige Mischung besteht aus Zellsuspension und BMM im Verhältnis 30:70.
- Entfernen Sie die entsprechende Menge an Überstand und resuspendieren Sie in eiskaltem BMM, um ein Verhältnis von 70: 30 von BMM zu erhalten: Zellsuspension für die Beschichtung. Zum Beispiel, wenn 200.000 Zellen erhalten werden, platten 10 Tröpfchen von 20 μL. Entfernen Sie also den Überstand, bis 60 μL der Zellsuspension übrig sind, fügen Sie 140 μL eiskaltes BMM hinzu und suspendieren Sie es erneut.
- Suspendieren Sie langsam, um die Bildung von Luftblasen zu vermeiden. Sobald es in BMM resuspendiert ist, halten Sie das Mikrozentrifugenröhrchen auf Eis.
HINWEIS: Das Mikrozentrifugenröhrchen auf Eis zu halten, ist entscheidend, um eine BMM-Erstarrung zu vermeiden.
- Suspendieren Sie langsam, um die Bildung von Luftblasen zu vermeiden. Sobald es in BMM resuspendiert ist, halten Sie das Mikrozentrifugenröhrchen auf Eis.
- Pipettieren Sie die 20 μL BMM-Tröpfchen in der Mitte der Vertiefungen der vorgewärmten 48-Well-Kulturplatte.
- Drehen Sie die Platte auf den Kopf, legen Sie sie in einen 1,9% CO 2 -Inkubator (% CO2 nach SFDM-Puffer) und lassen Sie sie für mindestens 20 min bei 37 ° C erstarren.
- ROCK-Inhibitor (RI; 10 μM) und Amphotericin B (0,1%) in TOM geben und das Medium in einem 37 °C warmen Wasserbad vorwärmen.
HINWEIS: Amphotericin B ist lichtempfindlich. - Nehmen Sie die 48-Well-Platte aus dem Inkubator, stellen Sie sie aufrecht und fügen Sie 250 μL des vorbereiteten vorerwärmten Mediums zu jeder Vertiefung mit BMM-Tröpfchen / Zellen hinzu und bringen Sie die Platte in den 1,9% CO 2-Inkubator zurück.
- Um das Medium aufzufrischen (vorzugsweise alle 2-3 Tage), neigen Sie die 48-Well-Platte in einem Winkel von 45°, entfernen Sie vorsichtig das vorherige Medium, wobei Sie das BMM-Tröpfchen nicht berühren, und fügen Sie 250 μL neues vorgewärmtes TOM-Medium hinzu.
HINWEIS: Es wird empfohlen, mindestens dreimal pro Woche zu erfrischen, um eine Erschöpfung der wichtigsten Nährstoffe und Wachstumsfaktoren zu vermeiden. RI muss nur bei der ersten Aussaat und in den ersten Tagen jedes Durchgangs (siehe Abschnitt 4) zu TOM hinzugefügt werden, um den Zelltod durch Anoikis zu verhindern und das Organoidwachstum zu verstärken. Amphotericin B wird dem Medium nur während der Passage 0 (P0) zugesetzt, um eine mögliche Pilzkontamination zu blockieren.
4. Amplifikation und Durchgang der Zahnorganoidkultur (Abbildung 1B und Abbildung 2B)
- Passage der Organoide zwischen 10 und 14 Tagen Kultur.
HINWEIS: Vermeiden Sie es, über einen längeren Zeitraum zu kultivieren, da eine längere Kultur das Nachwachsen und die Durchlässigkeit von Organoiden einschränken kann. Nur bei der ersten Entwicklung (P0) können Organoide je nach Wachstumsrate bis zu 20 Tage kultiviert werden (bis maximal ±200 μm Durchmesser erreicht sind). - Entfernen Sie das Medium aus den Vertiefungen mit Organoiden, die durchgelassen werden müssen. Innerhalb einer Kulturbedingung können bis zu vier konfluente Bohrlöcher gebündelt werden (siehe Abbildung 2C).
- Um die Organoide zu sammeln, geben Sie 400 μL eiskaltes SFDM pro Vertiefung direkt auf das BMM-Tröpfchen und pipettieren Sie das Medium wiederholt auf und ab, bis sich das gesamte BMM-Tröpfchen gelöst hat.
- Falls Bohrlöcher gepoolt werden, übertragen Sie die 400 μL vom ersten Bohrloch zum nächsten (und so weiter), um die organoidhaltigen BMM-Tröpfchen aller Wells, die gepoolt werden müssen, zu entfernen.
- Die gelöste BMM-Organoid-Baugruppe wird in ein 1,5-ml-Mikrozentrifugenröhrchen überführt und Schritt 4.3 erneut wiederholt, bis alle Organoidstrukturen aus den Vertiefungen gesammelt sind.
- Zentrifuge bei 200 x g für 5 min bei 4 °C.
- Ein Aliquot TrypLE Express (ergänzt mit RI bei 5 μM) in einem 37 °C warmen Wasserbad vor. Pro Mikrozentrifugenröhrchen mit Organoiden wird ein Volumen von 400 μL TrypLE Express benötigt.
- Nach der Zentrifugation den Überstand entfernen und das Pellet in 400 μL TrypLE resuspendieren. Die Suspension in einem 37 °C warmen Wasserbad für 12 min ausbrüten.
- 400 μL eiskaltes SFDM hinzufügen, um das Enzym zu inaktivieren, und zentrifugieren Sie bei 200 x g für 5 min bei 4 °C. Entfernen Sie den Überstand.
- Beschichten Sie eine Spitze (zum Volumen siehe unten) mit eiskaltem SFDM vor und resuspendieren Sie das Organoidpellet.
- Um Organoidverlust zu vermeiden, verwenden Sie die gleiche (vorbeschichtete) Spitze so weit wie möglich, ohne die Sterilität zu gefährden.
HINWEIS: Das für die Resuspendierung des Pellets erforderliche SFDM-Volumen hängt von der Anzahl der erhaltenen Organoidstrukturen und der aktuellen Durchgangszahl ab. - Als Faustregel gilt, dass für Passagen P0 bis P2-3 (im Allgemeinen immer noch eine geringe Anzahl von Organoiden wachsend) ein Volumen von 200 μL SFDM verwenden. Aus Passagen verwenden P3-4 oder höher (im Allgemeinen eine größere Anzahl von Organoiden) ein Volumen von 700 μL SFDM.
ANMERKUNG: Beide Verfahren werden als "Low-Passage"- bzw. "High-Passage"-Methoden bezeichnet (siehe Schritte 4.10 und 4.11). Die korrekte Anwendung der verschiedenen Methoden ist entscheidend für eine effiziente Passage der Organoide. Bei der Methode der höheren Passage gehen Organoide leichter verloren und sollten nicht auf eine geringe Anzahl von Organoiden (d. H. Bei P0 bis P2-3) angewendet werden. Es wird jedoch die Methode der höheren Passage benötigt, um größere Mengen von Organoiden effizient zu dissoziieren.
- Um Organoidverlust zu vermeiden, verwenden Sie die gleiche (vorbeschichtete) Spitze so weit wie möglich, ohne die Sterilität zu gefährden.
- Low-Passage-Methode: Resuspendiert das Pellet in 200 μL eiskaltem SFDM. Drücken Sie die vollständig entleerte Pipettenspitze gegen den Boden des Mikrozentrifugenrohrs, um den Durchmesser zu reduzieren. Überprüfen Sie, ob der Durchmesser klein genug ist (langsamere Aspiration, der Fluss ist verzerrt, aber nicht blockiert). Pipette 5 Minuten lang auf und ab, um die Organoide mechanisch zu stören.
- Höhere Passage Methode: Resuspendiert das Pellet in 700 μL eiskaltem SFDM mit einer P1000-Spitze. Fügen Sie eine P200-Spitze (ohne Filter) auf diese P1000-Spitze und beschichten Sie sie mit eiskaltem SFDM vor. Verhindern Sie die Bildung von Luftblasen, indem Sie die Volumeneinstellung des Pipetts anpassen, um mindestens 90% des mittleren Volumens (mit Organoiden) anzustreben. Pipette 5 Minuten lang auf und ab, um die Organoide mechanisch zu stören.
- Prüfen Sie unter dem Lichtmikroskop (bei 4-facher Vergrößerung), ob hauptsächlich einzelne Zellen mit (nur) wenigen unverteilten Strukturen erhalten werden.
- Geben Sie 500 μL eiskaltes SFDM in das Mikrozentrifugenröhrchen und mischen Sie die dissoziierte Zellmischung vorsichtig mit dem frischen SFDM durch Pipettieren.
- Lassen Sie große undispergierte Strukturen für 10 Minuten sedimentieren, indem Sie das Mikrozentrifugenröhrchen vertikal auf Eis legen.
HINWEIS: Es ist notwendig, die unverteilten Strukturen zu entfernen, da sie die organoide Durchgangsfähigkeit negativ beeinflussen. Für die Organoid-Initiierung (P0) ist dieser Sedimentationsschritt nicht erforderlich. - Sammeln Sie den Überstand (~ 500-1.000 μL abhängig von der Passaging-Methode), der einzelne Zellen und kleine Zellcluster enthält; In ein neues Mikrozentrifugenröhrchen geben und bei 190 x g für 10 min bei 4 °C zentrifugieren.
- Berechnen Sie die Zellkonzentration mit einem automatisierten Zellzähler. Vernachlässigen Sie die verbleibenden Zellklumpen.
- Zählen Sie, wie viele Vertiefungen ausgesät werden können und berechnen Sie das geeignete Zellsuspensions/BMM-Verhältnis wie oben beschrieben (Abschnitt 3).
- Geben Sie 70% BMM in das Zellpellet und halten Sie das Mikrozentrifugenröhrchen auf Eis.
HINWEIS: Es ist wichtig, das Mikrozentrifugenröhrchen auf Eis zu halten, um eine BMM-Erstarrung zu vermeiden. - Fahren Sie mit Schritt 3.3 bis 3.7 fort und gehen Sie erneut zwischen Tag 10 und Tag 14 der Kultur über.
5. Kryokonservierung von Zahnorganoiden
- Sammeln und dissoziieren Sie die Organoide, wie oben erwähnt, für die Übertragung (Schritt 4). Zellsuspension bei 200 x g für 5 min bei 4 °C zentrifugieren.
- Entsorgen Sie den Überstand und suspendieren Sie das Pellet in 1 ml Kryokonservierungsmedium, das SFDM (70%), FBS (20%) und DMSO (10%) enthält.
- Übertrage die Suspension in ein Kryovie und lege sie auf Eis. Legen Sie die Kryozellen in eine Kryobox und geben Sie sie auf -80 ° C (für mindestens 4 h).
- Innerhalb von 1 Monat die gefrorenen Proben zur Langzeitlagerung (>12 Monate) in flüssigen Stickstoff überführen.
6. Auftauen von kryokonservierten Zahnorganoiden
- Bevor Sie mit dem Auftauen beginnen, legen Sie ein 15-ml-Röhrchen pro Kryo auf Eis, das 10 ml SFDM mit 20% FBS enthält.
- Entfernen Sie das Kryo aus dem flüssigen Stickstoff und legen Sie es auf Eis.
HINWEIS: Führen Sie die folgenden Schritte so schnell wie möglich durch und vermeiden Sie eine zu lange Auftauzeit (>5 min) sowie zu lange Intervalle zwischen den Schritten (>5 min), da eine solche Verlängerung das Überleben der Zellen verringert. - Legen Sie die Kryofrüchte in ein warmes Wasserbad (37 °C), bis sie aufgetaut sind (~1-2 min).
- Sofort den Inhalt des Kryovials in das eiskalte 15-ml-Röhrchen und die Zentrifuge bei 200 x g für 5 min bei 4 °C überführen.
- Entfernen Sie 9 ml des Überstands und zentrifugieren Sie die restlichen 1 ml bei 200 x g für 5 min bei 4 °C. Entfernen Sie den Überstand und waschen Sie das Pellet mit 1 ml eiskaltem SFDM.
- Übertragen Sie die Zellsuspension auf ein 1,5-ml-Mikrozentrifugenröhrchen und zählen Sie die Zellen mit einem automatisierten Zellzähler.
- Zählen Sie, wie viele Vertiefungen ausgesät werden können und berechnen Sie das geeignete Zellsuspensions/BMM-Verhältnis wie oben beschrieben (Abschnitt 3).
- Zentrifuge bei 200 x g für 5 min bei 4 °C. Schwebe das Pellet im Verhältnis 70:30 von BMM:TOM und halte es auf Eis.
- Fahren Sie mit Schritt 3.3 bis 3.7 fort und gehen Sie zwischen Tag 10 und Tag 14 der Kultur über.
7. Fixierung und Paraffin-Einbettung von Zahnorganoiden
HINWEIS: Dieses Verfahren (einschließlich der Abschnitte 8 und 9) kann auch auf das primäre DF-Gewebe angewendet werden.
- Fixierung von Zahnorganoiden in PFA
- Entfernen Sie das Medium aus jedem Well, wie in Schritt 4.2.
- Sammeln Sie die Organoide, indem Sie den BMM-Tröpfchen entfernen (Schritt 4.3). Überführen Sie die BMM-Organoid-Mischung in ein 1,5 ml Mikrozentrifugenröhrchen.
- Zentrifuge bei 200 x g für 5 min bei 4 °C.
- Entfernen Sie den Überstand und fügen Sie 500 μL 4% PFA hinzu und inkubieren Sie für mindestens 30 min (maximal 1 h) bei RT mit sanftem Mischen auf einem Orbitalschüttler.
ACHTUNG: Verwenden Sie die chemische Haube, wenn Sie mit PFA arbeiten. - Zentrifuge bei 200 x g für 5 min bei 4 °C. Entfernen Sie den Überstand und spülen Sie das Organoid-Pellet mit PBS für 10 min bei RT mit leichtem Schütteln.
- Das Organoid-Pellet (Schritt 7.1.5) wird zweimal zusätzlich gewaschen. Drehen Sie die fixierten Organoide bei 200 x g für 5 min bei 4 °C nach unten.
- Resuspendiert das Pellet in 500 μL von 70% EtOH (in deionisiertem Wasser). Lagern Sie die Organoide bis zu 1 Monat in 70% EtOH bei 4 °C.
- Agarose- und Paraffin-Einbettung von Zahnorganoiden
HINWEIS: Um Organoide effizient in Paraffin einzubetten, ist ein zusätzlicher Schritt der Einbettung in Agarose erforderlich.- Die PFA-fixierten Organoide zentrifugieren Sie in 70% EtOH bei 200 x g für 5 min bei 4 °C. Entfernen Sie den Überstand.
- Bereiten Sie eine 2% ige Agaroselösung in 30 ml PBS in einer Glasflasche vor. Erwärmen Sie das Agarose-PBS-Gemisch in einer Mikrowelle, bis eine gelartige Struktur beobachtet wird (ca. 2,5 min bei 600 W).
- Fügen Sie parallel 30 ml PBS zu einer anderen Glasflasche hinzu und wärmen Sie sich in der Mikrowelle auf (ca. 2,5 min bei 600 W). Lassen Sie die Agaroselösung 1 min abkühlen.
- Schneiden Sie das Ende einer P200-Spitze ab, um präzise mit der Agaroselösung arbeiten zu können und Luftblasen zu vermeiden.
- Wärmen Sie die P200-Spitze im warmen PBS vor, indem Sie einige Male auf und ab pipettieren.
- 150 μL vorgewärmte Agaroselösung in das Organoidpellet geben und das Organoid-Agarose-Gemisch vorsichtig (mit minimaler Reuspension) pipettieren, während Luftblasen vermieden werden.
HINWEIS: Die minimale Resuspension ermöglicht eine bessere Lokalisierung der Organoide im Agarosegel bei Mikrotomschnitten, da die Organoide dann näher beieinander liegen. - Übertragen Sie das Organoid-Agarose-Gemisch sofort auf die gleiche Mikrozentrifugen-Röhrchenkappe, legen Sie das Röhrchen horizontal und lassen Sie die Agarose erstarren (~ 20 min bei RT). Beschriften Sie in der Zwischenzeit die Kassetten.
- Nach dem Erstarren das Agarosegel mit einer Pinzette von der Mikrozentrifugenkappe entfernen und auf eine beschriftete Kassette geben.
- Die agarosehaltige Kassette in ein Becherglas mit 50% EtOH in entionisiertem Wasser geben. Decken Sie das Becherglas mit Parafilm ab, um eine Verdunstung des EtOH zu vermeiden.
HINWEIS: Das Volumen des Becherglases hängt von der Anzahl der Kassetten ab. - Verarbeiten Sie die Proben in einem Tissue-Prozessor über Nacht.
HINWEIS: Da sich Paraffin bei RT verfestigt, müssen die folgenden Schritte schnell durchgeführt werden. - Heizblock im Einbettarbeitsplatz für 15 min vorwärmen.
- Entfernen Sie das organoidhaltige Agarosegel, das in der Kassette eingeschlossen ist, und legen Sie es in den vorgewärmten Heizblock. Entfernen Sie die Kappe von der Kassette und legen Sie sie zur späteren Verwendung beiseite.
- Füllen Sie den restlichen Heizblock mit Paraffin.
HINWEIS: Überprüfen Sie mit einer Pinzette, ob sich das organoidhaltige Agarosegel noch am Boden des Heizblocks befindet. Aufgrund seines geringen Gewichts kann es anfangen zu schweben, was zu einem Probenverlust führt. - Legen Sie die Kassettenkappe auf den Heizblock. Lassen Sie es 30 min auf einer kalten Platte erstarren. Entfernen Sie den Heizblock und lagern Sie die Paraffinblöcke bei 4 °C.
8. Mikrotomschnitt und Färbung von Zahnorganoiden (Abbildung 2B und Abbildung 3A-C)
- Mikrotomschnitt von organoidhaltigen Paraffinblöcken
- Schneiden Sie 5 μm-Abschnitte der Zahnorganoide in die Paraffinblöcke mit einem Mikrotom.
- Legen Sie die Paraffinscheiben auf einen Objektträger aus Glas. Legen Sie den Objektträger aus Glas auf eine warme Platte bei 37 °C und bedecken Sie ihn mit einer Pasteur-Pipette mit deionisiertem Wasser.
- Lassen Sie das Mikroskopglas über Nacht trocknen.
- Färbeung von Zahnorganoidschnitten
- Entparaffinisieren Sie die Organoidabschnitte (auf dem Mikroskopglasobjektträger) im Ofen für 1 h bei 58 °C.
- Rehydrieren Sie die Organoidabschnitte (auf dem Objektträger) in abnehmender EtOH-Serie innerhalb der chemischen Haube in der folgenden Reihenfolge:
Xylol 2x für je 3 min
100% EtOH 2x für je 3 min
95% EtOH 2x für je 3 min
90% EtOH 2x für je 3 min
70% EtOH 3x für je 3 min
ACHTUNG: Verwenden Sie die chemische Haube, wenn Sie mit Xylol arbeiten. - Spülen Sie den Mikroskop-Glasobjektträger in Leitungswasser für 5 min bei RT. Dann spülen Sie es in PBS für 5 Minuten bei RT.
- Führen Sie eine Antigenentnahme durch, indem Sie die Organoidabschnitte (auf dem Objektträger) in einen vorerwärmten Citratpuffer (10 mM Zitronensäure in entionisiertem Wasser mit pH 6; in einem Kunststoffbehälter; vorgewärmt für 10 min in einem 95 °C Wasserbad) für 30 min in einem 95 °C Wasserbad.
- Lassen Sie den Objektträger des Mikroskopglases bei RT 20 min abkühlen. Spülen Sie den Objektträger in PBS für 5 min bei RT und dann in PBS mit 0,1% Triton-X (PBT) für 5 min bei RT.
- Verwenden Sie einen Markierungsstift, um eine hydrophobe Barriere an den Rändern jedes Objektträgers zu erzeugen.
- Block für mindestens 1 h bei RT in Blockpuffer (mit 1,5 mg/ml Glycin, 2 mg/ml Albumin-Rinderserum (BSA) gelöst in PBT) plus 10% Eselserum.
HINWEIS: Typischerweise werden 300 μL Sperrpuffer plus 10% Eselserum pro Mikroskopglasobjektträger benötigt. - Legen Sie den Objektträger aus Glas in eine feuchte Kammer. Verwenden Sie eine luftdichte Box, in die alle Dias passen, und befeuchten Sie ein paar Papiertaschentücher mit Wasser, um sie am Boden der Box zu platzieren. Dadurch wird verhindert, dass die Objektträger bei den nachfolgenden Färbeschritten austrocknen.
- Entfernen Sie den Sperrpuffer, fügen Sie den primären Antikörper (Tabelle 6), der im Blockpuffer hergestellt wurde, plus 1% Eselserum hinzu und inkubieren Sie den mit Antikörperlösung bedeckten Objektträger über Nacht in der feuchten Kammer bei 4 °C. Wiederholen Sie bei Bedarf den Markierungsstiftrand.
- Waschen Sie den Objektträger dreimal in PBT bei RT für 10 Minuten mit sanftem Mischen auf einem Orbitalschüttler (75-150 U / min).
- Fügen Sie den sekundären Antikörper (Tabelle 6) hinzu, der in Blockpuffer plus 1% Eselserum hergestellt wurde, und inkubieren Sie für 1 h in der feuchten Kammer bei RT.
- Entfernen Sie den Sperrpuffer und waschen Sie den Objektträger dreimal in PBT bei RT für 10 Minuten mit sanftem Mischen auf einem Orbitalschüttler.
- Fügen Sie Antifade-Montagemedium mit DAPI (1 bis 3 Tröpfchen) auf einen Glasdeckel und montieren Sie dieses auf den Organoidabschnitten (auf dem Objektträger aus Glas). Fahren Sie mit der Bildgebung fort; Lagern Sie Dias für maximal 1 Woche bei 4 °C.
9. RNA-Extraktion und RT-qPCR von Zahnorganoiden (Abbildung 2B und Abbildung 3D)
- RNA-Extraktion von Zahnorganoiden
- Entfernen Sie das Medium aus jeder Vertiefung (Schritt 4.2).
- Sammeln Sie die Organoide, indem Sie das BMM-Tröpfchen entfernen (Schritt 4.3), übertragen Sie das BMM-Organoid-Gemisch in ein 1,5-ml-Mikrozentrifugenröhrchen und zentrifugieren Sie bei 200 x g für 5 min bei 4 °C.
- Entfernen Sie den Überstand, resuspendieren Sie kräftig in 350 μL von 1% β-Mercaptoethanol, gelöst in Lysepuffer (Tabelle der Materialien), und legen Sie es auf Eis.
ACHTUNG: Führen Sie alle Schritte mit β-Mercaptoethanol in einer chemischen Haube durch.
HINWEIS: Proben können bei diesem Schritt bis zu 1 Monat bei -80 °C gelagert werden. Tauen Sie die Proben auf Eis auf, bevor Sie mit Schritt 9.1.4 fortfahren. - Wirbeln Sie die Proben solange, bis keine organoiden Strukturen mehr beobachtet werden.
- Fahren Sie mit der RNA-Extraktion mit dem RNA-Extraktionskit (Table of Materials) gemäß den Anweisungen des Herstellers fort.
- RT-qPCR von Zahnorganoiden (Tabelle 7)
- Reverse-Transkribieren (RT) der RNA19 mit dem Reverse-Transkriptions-Kit (Table of Materials) gemäß den Anweisungen des Herstellers.
- Analysieren Sie die resultierenden cDNA-Proben mit SYBR Green-basierter quantitativer PCR (qPCR)19 unter Verwendung eines Real-Time PCR-Systems (Table of Materials).
Ergebnisse
Entwicklung von Zahnorganoiden
Wir bieten ein detailliertes Protokoll zur Etablierung von Organoidkulturen aus menschlichem DF-Gewebe, das nach der Weisheitszahnextraktion gewonnen wurde (Abbildung 1A). Isolierte DF ist enzymatisch und mechanisch dissoziiert. Die erhaltenen Zellen werden innerhalb von BMM in Medien kultiviert, die empirisch für eine optimale Organoidentwicklung und -wachstum (Zahnorganoidmedium; TOM)19.
Die Organoide entwickeln sich typischerweise innerhalb von 2 Wochen nach der DF-Zellaussaat (P0; Abbildung 2A). Die Organoide sind langzeitexpandierbar (bisher bis zu 11 Passagen) (Abbildung 2B, dargestellt auf P4). Die Aussaat von etwa 20.000 Zellen pro BMM-Tröpfchen (sowohl an P0 als auch an weiteren Passagen) ergibt eine optimale Dichte an Organoiden (Abbildung 2C), während die Aussaat höherer Zellzahlen zu suboptimalem Organoidwachstum führt (d. h. kleinere Organoide mit zu hoher Dichte), da nicht genügend Platz zum Wachsen vorhanden ist (Abbildung 2D). Die schließlich optimierten Kulturbedingungen ermöglichen die Entwicklung von Organoiden aus DF-Proben mit einem Wirkungsgradvon 100% 19.
Charakterisierung und Validierung von Zahnorganoiden
Die entwickelten Organoide zeigen ein dichtes Aussehen und enthalten Zellen mit einem hohen nukleozytoplasmatischen Verhältnis, wie es in ähnlicher Weise in ERM-Zellen7 beobachtet wurde (Abbildung 3A). Darüber hinaus und in weiterer Analogie exprimieren die Organoide den ERM-Marker Cytokeratin 14 (CK14)22 und bestätigen damit ihren epithelialen Ursprung (Abbildung 3B) sowie andere vorgeschlagene ERM-Marker (wie P63, CD44 und ITGα612,22,23 (Abbildung 3B). Darüber hinaus exprimieren Organoide SOX2, einen bekannten DESC-Marker bei Mäusen, der auch im Epithel der sich entwickelnden menschlichen Zähne vorhanden ist (Abbildung 3B)1. Interessanterweise wird Amelogenin (AMELX), der Hauptbestandteil der Schmelzmatrix, der auch in den Organoiden exprimiert wird, auch im ERM24 nachgewiesen (Abbildung 3C). Die Expression von noch anderen ERM/Stammheitsmarkern wird in unserer aktuellen Studie19 beschrieben und kann verwendet werden, um die erhaltenen Organoide weiter zu zertifizieren. Darüber hinaus behalten die Organoide ihren ERM/Stammheitsphänotyp während des Passagings, unter anderem durch stabile Expression von ERM/Stammzellmarkern (Abbildung 3D). Schließlich zeigen die von Zähnen abgeleiteten Organoide eine Differenzierungskapazität zu Ameloblasten(-ähnlichen) Zellen, die auch zur Validierung der erhaltenen Organoidkulturen angewendet werden können, wobei die Expression von reifen Ameloblastenmarkern wie Odontogen-Ameloplast-assoziiertem Protein (ODAM) und Amelotin (AMTN) nach dem Transfer auf das Differenzierungsmedium gezeigt wird (siehe19).

Abbildung 1: Schematischer Arbeitsablauf der Entwicklung, Charakterisierung und Anwendung von Zahnorganoiden. (A) Zahnorganoidentwicklung aus Zahnfollikelgewebe (DF), das aus nicht ausgebrochenen dritten Molaren isoliert wurde. (B) Amplifikation, Charakterisierung und Anwendungspotenzial von Zahnorganoiden. d, Tag; P, Passage. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
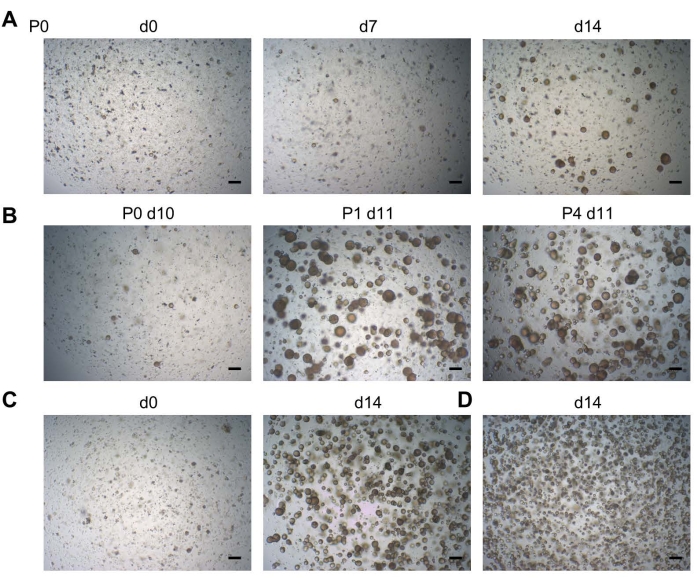
Abbildung 2: Entwicklung der Zahnorganoide . (A) Organoidentwicklung aus DF-Gewebe. Repräsentative Hellfeldbilder, die an verschiedenen Tagen (d) nach der Aussaat (P0; P, Passage; 2,5-fach). (B) Hellfeldbilder, die eine robuste Durchlässigkeit einer Zahnorganoidlinie zeigen (2,5x). (C) Hellfeldbilder, die eine Zahnorganoidlinie unmittelbar beim Passieren zeigen (d0; links; 2,5x), die mit einer Dichte von 20.000 Zellen pro Bohrung ausgesät ist, und die resultierende konfluierende Organoidkultur, die bereit ist, durchgelassen zu werden (d14; rechts; 2,5x). (D) Hellfeldbilder, die eine Zahnorganoidlinie zeigen, die mit einer Dichte von >20.000 Zellen gesät wurde, was zu kleineren Organoiden mit zu hoher Dichte bei d14 (2,5x) führte. Maßstabsstäbe: 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Charakterisierung des Zahnorganoids. (A) Hellfeldbilder von DF-abgeleiteten Organoidkulturen bei verschiedenen Vergrößerungen, die dichte Strukturen zeigen, die sich nach 14 Tagen in TOM entwickelt haben (P4; 5-20x)). Hämatoxylin- und Eosinfärbung eines Organoids (P1, Tag 11). Box ist vergrößert. Pfeile zeigen Zellen mit einem hohen nukleozytoplasmatischen Verhältnis an. (B) Immunfluoreszenzfärbung für Epithel-/ERM-/Stammheitsmarker in TOM-gezüchteten Organoiden (20x). (C) Immunfluoreszenzfärbung für Amelogenin (AMELX) in TOM-gezüchtetem Organoid (20x). DAPI (blau) wurde verwendet, um Kerne zu kennzeichnen. (D) Genexpression (relativ zu GAPDH) von ERM/Stammheitsmarkern in P1 und P5 TOM-gezüchteten Organoiden am Tag 14 der Kultur (Mittelwert ± SEM; n = 3 biologische Replikate). Maßstabsstäbe: 50 μm, sofern nicht anders angegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Entnahmemedium für Zahnfollikel (DF) | |
| Name | Konzentration |
| Minimaler essentieller mittlerer Adler (αMEM) | |
| Fetales Rinderserum (FBS) | 10% |
| Amphotericin B | 0.5% |
| Penicillin-Streptomycin (Pen/Streptokokken) | 1% |
Tabelle 1: Entnahmemedium für Zahnfollikel (DF) Die Tabelle listet die Bestandteile des DF-Sammlungsmediums auf.
| Zahnorganoid-Medium (TOM) | |
| Name | Konzentration |
| Serumfreies definiertes Medium (SFDM) | Tabelle 3 zur Zusammensetzung siehe Tabelle 3 |
| A83-01 | 0,5 μM |
| B27 (ohne Vitamin A) | 2% |
| Cholera-Toxin | 100 ng/ml |
| FGF2 (= Basis-FGF) | 20 ng/ml |
| FGF8 | 200 ng/ml |
| FGF10 | 100 ng/ml |
| L-Glutamin | 2 mM |
| IGF-1 | 100 ng/ml |
| Nr. N2 | 1% |
| N-Acetyl L-Cystein | 1,25 mM |
| Nicotinamid | 10 mM |
| Birne | 100 ng/ml |
| RSPO1 | 200 ng/ml |
| SB202190 (p38i) | 10 μM |
| Pst | 100 ng/ml |
| WNT3a | 200 ng/ml |
Tabelle 2: Zahnorganoid-Medium (TOM). Die Tabelle listet die Bestandteile und ihre jeweiligen Konzentrationen auf, die zur Herstellung des Zahnorganoidmediums erforderlich sind.
| Serumfreies definiertes Medium (SFDM) (pH 7,3) | |
| Name | Konzentration |
| Steril H2O | |
| DMEM 1:1 F12 ohne Fe | 16,8 g/l |
| Transferrin | 5 mg/l |
| Insulin aus der Bauchspeicheldrüse des Rinds | 5 mg/l |
| Penicillin G Natriumsalz | 35 mg/l |
| Streptomycinsulfatsalz | 50 mg/l |
| Ethanol absolut, ≥99,8% (EtOH) | 600 μL/L |
| Katalase aus Rinderleber | 50 μL/L |
| Natriumhydrogencarbonat (NaHCO3) | 1 g/l |
| Albumin Rinder (Zellkulturqualität) | 5 g/l |
Tabelle 3: Serumfreies definiertes Medium (SFDM) (pH 7,3). Die Tabelle listet die Zusammensetzung des serumfrei definierten Mediums auf.
| Dissoziationsmedium | |
| Name | Konzentration |
| Phosphatgepufferte Kochsalzlösung (PBS) | |
| Kollagenase IV | 3 mg/ml |
| Dispase II | 4 mg/ml |
Tabelle 4: Dissoziationsmedium Liste der Bestandteile und ihrer erforderlichen Konzentrationen zur Herstellung des Dissoziationsmediums.
| Medium A (pH 7,3) | |
| Name | Konzentration |
| Steril H2O | |
| DMEM-Pulver mit hohem Glukosegehalt | 13,38 g/l |
| HEPES | 5,958 g/l |
| Natriumpyruvat (C3H3NaO3) | 110 mg / l |
| Penicillin G Natriumsalz | 35 mg/l |
| Streptomycinsulfatsalz | 50 mg/l |
| Natriumchlorid (NaCl) | 0,5 g/l |
| Natriumhydrogencarbonat (NaHCO3) | 1 g/l |
| Albumin Rinder (Zellkulturqualität) | 3 g/l |
| Dnase* | 0,2 mg/ml |
| *Hinzufügen, wenn erwähnt |
Tabelle 5: Medium A (pH 7,3). Die Tabelle listet die Konzentration der Bestandteile auf, die zur Herstellung von Medium A verwendet werden.
| Primäre Antikörper | ||
| Name | Gastgeber | Konzentration |
| AMELX | Maus | 1:100 |
| CD44 | Maus | 1:200 |
| CK14 | Maus | 1:200 |
| ITGA6 | Kaninchen | 1:200 |
| S. 63 | Kaninchen | 1:1000 |
| SOX2 | Kaninchen | 1:2000 |
| Sekundarische Antikörper | ||
| Name | Gastgeber | Konzentration |
| Maus IgG (Alexa 555) | Esel | 1:1000 |
| Kaninchen IgG (Alexa 488) | Esel | 1:1000 |
Tabelle 6: Liste der Antikörper und ihrer Verdünnungen. Die Tabelle listet die Antikörper und ihre jeweiligen Verdünnungen auf, die in dieser Studie verwendet wurden.
| Zündkapseln | ||
| Gen | Vorwärts-Primer | Reverse Primer |
| GAPDH | GGTATCGTGGAAGGACTCATGAC | ATGCCAGTGAGCTTCCCGTTCTCAG |
| S. 63 | CAACGCAGTAGACACCATTTCC | CCCAAAACCTTCTCGCTTGTT |
| ITGA6 | GGCGGTGTTATGTCCTGAGTC | AATCGCCCATCACAAAAGCTC |
| SOX2 | GCTGGGACATGTGAAGTCTG | GKKCTGTGGTTACCTCTCCCT |
| PITX2 | CAGCGGACTCACTTTACCAG | ATTCTTGAACCAAACCCGGAC |
Tabelle 7: Liste der Primer Die Tabelle listet die in dieser Studie verwendeten Primer von GAPDH, P63, ITGA6, SOX2 und PITX2 auf.
Diskussion
Dieses Protokoll beschreibt die effiziente und reproduzierbare Erzeugung von Organoiden ausgehend vom menschlichen Zahn. Unseres Wissens ist dies die erste Methodik zur Etablierung von (epithelialen) Organoiden des aktuellen Konzepts, ausgehend von menschlichem Zahngewebe. Die Organoide sind langfristig expandierbar und zeigen einen Phänotyp der Zahnepithelstammigkeit, der DESCs dupliziert, die zuvor im ERM-Kompartiment des DF7 berichtet wurden. Darüber hinaus replizieren die Organoide funktionelle DESC/ERM-Eigenschaften, einschließlich der Entfaltung eines Ameloblast-Differenzierungsprozesses 7,25,26. Die Ergebnisse sind robust, da vergleichbare Ergebnisse mit unabhängigen Organoidlinien für Patientengefunden wurden 19.
Bei der Ausführung dieses Zahnorganoidprotokolls müssen mehrere kritische Punkte berücksichtigt werden. Erstens ist die Zugabe von Rho-assoziierter Kinase (ROCK) -Inhibitor Y-27632 bei der ersten Aussaat und unmittelbar nach jedem Durchgang unerlässlich, um zu verhindern, dass die einzelnen Zellen einer Anoikis27 unterzogen werden. Darüber hinaus wird Amphotericin B in allen Medienerfrischungen während P0 benötigt, um (orales) Pilzwachstum zu vermeiden. Zweitens wird empfohlen, die frisch isolierten DF-Gewebe sofort für die optimale Organoidbildung und Wachstumseffizienz zu verarbeiten, anstatt mit kryokonserviertem Gewebe zu beginnen, was zu einer geringeren Effizienz führt. Drittens, wenn Sie eine kryokonservierte Organoidlinie für die Kultivierung auftauen, führen Sie Schritte so schnell wie möglich durch und vermeiden Sie eine zu lange Auftauzeit sowie zu lange Intervalle zwischen den Schritten, da die Zeitverlängerung das Zellüberleben verringert. Viertens sollte beachtet werden, dass die Anzahl der Organoide bei der frühen Passage (P0-P3) begrenzt bleiben kann, auch weil nur eine begrenzte Anzahl von ERM (Stamm-) Zellen in den spezifischen isolierten DF-Gewebeproben vorhanden sein kann. Daher sollten die Organoidkulturen bei der frühen Passage mit Sorgfalt und Rücksicht behandelt werden. Daher wird empfohlen, (i) eine schnelle Expansion der Organoidkultur zu vermeiden (d.h. erst bei 1:3 oder mehr ab P3-4 und vor 1:0,5 oder 1:1 mit der Spaltung zu beginnen); ii) die geeignete Spaltmethode (niedriger Durchgang - höhere Passage) wie beschrieben anwenden. In diesem Zusammenhang wird empfohlen, nicht ausgebrochene Weisheitszähne junger jugendlicher Patienten (15 bis 19 Jahre alt) zu sammeln, da die Anzahl der ERM-Zellen mit der Zahnentwicklung und dem Altervon 28 Jahren abnimmt. Fünftens führen verbleibende unverteilte Hartgewebefragmente aus dem DF-Gewebe in der Zellsuspension (auch nach der Filterung) dazu, dass der BMM-Abfall weniger stabil ist und sich während der Kultur eher löst. Ein höherer BMM-Anteil (z. B. 80%) wird empfohlen, wenn mehrere unverteilte Hartgewebefragmente in der dissoziierten DF-Zellsuspension beobachtet werden. Sechstens wird dringend empfohlen, die Organoide zwischen Tag 10 und Tag 14 der Kultur zu passieren, da eine längere Kultur die Erweiterbarkeit der Organoide aufgrund der weniger optimalen Dissoziation negativ beeinflusst. Wenn Organoide aus dem einen oder anderen Grund länger als 14 Tage kultiviert werden, könnten die TryplE Express-Menge und die Inkubationszeit für die Organoiddissoziation für eine effiziente Dissoziation verlängert werden, obwohl 15 Minuten enzymatische Exposition nicht überschritten werden sollten. Im gleichen Zusammenhang muss das Kulturmedium alle 2-3 Tage aufgefrischt werden, um eine Erschöpfung der Nährstoffe und des Wachstumsfaktors zu verhindern. Für den Fall, dass sich Organoide nicht richtig ausdehnen, sollte man sich unabhängig von den oben genannten kritischen Punkten darauf konzentrieren, alle Werkzeuge (BMM, eiskaltes SFDM für die Vorbeschichtungsspitze, Mikrozentrifugenröhrchen), die während des Passierens auf Eis verwendet werden, zu halten. Darüber hinaus ist es entscheidend, die verschiedenen Passaging-Methoden (Low-Passage- und High-Passage-Methode) für eine effiziente Passage der Organoide korrekt anzuwenden.
Zuvor berichteten andere Gruppen über ein In-vitro-Wachstum von primärem humanem DESC/ERM-Gewebe 8,9,10,11,12,21. Kulturen waren jedoch hauptsächlich 2D (Monolayer) und nicht 3D, wie dieses Organoidmodell, und zeigten außerdem nur kurzfristiges Wachstum und Phänotypretention. Alternativ wurden oft (spontan) immortalisierte Zellen verwendet, die jedoch nicht physiologisch sind und nur eine begrenzte Ähnlichkeit mit dem Gewebe oder den Ursprungszellen aufweisen. Darüber hinaus wurden diese Zelllinien aus embryonalem Gewebe und/oder von Tieren gewonnen. Des Weiteren wird die Ameloblastendifferenzierung entweder nicht oder nur eingeschränkt dokumentiert. Somit bietet das hier vorgestellte Organoidmodell mehrere Vorteile: (i) getreue Rekapitulation des Ursprungsgewebes/der Ursprungszellen, (ii) langfristig erweiterbar, (iii) in 3D kultiviert, die die In-vivo-Konfiguration, (iv) des menschlichen Ursprungs und des postnatalen Alters besser repräsentieren und (v) in der Lage ist, sich in reife Zahnzellen (Ameloblastenzelltyp) zu differenzieren (siehe19).
Auf diese Weise haben wir ein wertvolles Forschungsinstrument generiert, über das noch nie berichtet wurde, das mehrere interessante Anwendungen enthält (Abbildung 1B). Die Organoide können angewendet werden, um die menschliche DESC/ERM-Stammheit und -Plastizität zu untersuchen. Es bietet die Möglichkeit, durch Immunfluoreszenz-, Genexpressions- und (Einzelzell-)transkriptomische Analysen weitere Einblicke in die Biologie der immer noch rätselhaften ERM-Zellpopulation zu gewinnen. Darüber hinaus eignen sich Organoide besonders für die Modellierung menschlicher Krankheiten, um pathogenetische Mechanismen zu entschlüsseln, (neue) therapeutische Ziele zu identifizieren und Wirkstoffforschungs- und Screening-Tools zu generieren29. Genauer gesagt kann dieses Modell auf odontogene Zysten angewendet werden (für die kein zuverlässiges Forschungsmodell verfügbar ist), die mit organischen Organoiden aus gesundem Zahn verglichen werden können. Darüber hinaus kann dieser Zahnorganoid-Ansatz genutzt werden, um Zahnerkrankungen zu modellieren und zu untersuchen, die von den Auswirkungen von Bakterien bis hin zu genetischen Mutationen im Zusammenhang mit Zahnanomalien (wie Mutationen in P63, die mit modernsten Gen-Editing-Methoden wie CRISPR-Cas eingeführt werden könnten)30 reichen, was schließlich zu potenziellen und neuartigen therapeutischen Zielen und Behandlungen führt. Andere Anwendungen des Zahnorganoidprotokolls können Biobanking (derzeit bereits für Zahnpulpa verfügbar, wie die Future Health Biobank)31 sein, um Organoidlinien von vielfältigen Personen und Krankheiten zu sammeln (z. B. für Grundlagen- und translationale Forschung wie Arzneimittelscreening). Darüber hinaus wurden kürzlich mehrere Berichte über zusammengesetzte Organoidmodelle veröffentlicht, die nicht nur epitheliale, sondern auch andere Zelltypen des Ursprungsgewebes enthalten32,33. Da die Zahnzusammensetzung ziemlich komplex ist und mesenchymale, immune und Endothelzellen beherbergt, ist die Anwendung dieses epithelialen Organoidmodells in Kombination mit diesen Zelltypen, um ihr in vivo-Gegenstück detaillierter darzustellen, eine ansprechende Perspektive. Dieses System ermöglicht es auch, die Amelogenese im menschlichen Zahn zu erforschen, die derzeit nur wenig verstanden wird, aber sicherlich auf epithelial-mesenchymale Wechselwirkungen angewiesen ist. Es wird erwartet, dass die Entschlüsselung der Ameloblastenentwicklung einen wichtigen Sprung nach vorne in der zahnmedizinischen wissenschaftlichen und klinischen Welt darstellt, da die Produktion von Zahnschmelz, dem Hauptbestandteil unserer Zähne, ein sehr verfolgtes Ziel bei der Reparatur von Zahngewebe ist. Darüber hinaus kann die in dieser Studie beschriebene Organoidmodellierung den Beginn der Bildung von mineralisiertem Gewebe in vitro bedeuten und den Weg für die Entwicklung eines biotechnologisch hergestellten Zahnes (oder zumindest von Teilen) für die Ersatztherapie ebnen.
Eine der Einschränkungen des Organoidmodells besteht darin, dass es ausschließlich die epitheliale Komponente des Gewebes darstellt. Wie oben ausführlich beschrieben, könnte dieser Mangel jedoch durch die Zugabe anderer Zell-/Gewebetypen, wie z.B. des dentalen Mesenchyms19, behoben werden. Ein weiterer Aspekt, der als Einschränkung erkannt werden kann, ist die Herkunft des hier verwendeten BMM (Matrigel). Dieses BMM wird von einem Sarkom (Engelbrecht-Holm-Schwarm) einer Maus abgeleitet und muss daher ersetzt werden, bevor der Organoidansatz in die Klinik übersetzt wird. In jüngster Zeit wurden mehrere Anstrengungen unternommen, um Matrigel durch synthetische Hydrogele34,35 zu ersetzen. Es ist jedoch mehr Forschung erforderlich, um Organoide in solchen nicht-natürlichen Gelen erfolgreich zu züchten. Obwohl die Organoid-Technologie einen interessanten Ansatz für die zukünftige dentale regenerative Therapie bietet - zum Beispiel die Entwicklung eines biotechnologisch hergestellten Zahnes -, sollten ethische Fragen hinsichtlich der Privatsphäre von Zellspendern sowie der Kommerzialisierung von menschlichen Organoiden und daraus gewonnenen Geweben aufgeworfen werden. Bisher wurden keine Schlussfolgerungen hinsichtlich der Vermarktung von Organoiden für regenerative Zweckegezogen 36. Zahnpulpa-Biobanken sind auf dem Vormarsch31, ebenso wie Organoid-Biobanken aus mehreren, hauptsächlich krebsartigen Geweben, für Drogenscreening-Zwecke. Angesichts der Tatsache, dass Organoide nicht als Zellen, Gameten, Gewebe oder Organe kategorisiert werden können (die alle gesetzlich geregelt sind), besteht die dringende Notwendigkeit, ihren rechtlichen Status für ihre Verwendung in klinischen, wissenschaftlichen oder kommerziellen Umgebungen darzustellen. Obwohl die Organoide gezeigt haben, dass sie mineralisiertes Gewebe ablagern, wenn sie subkutan in vivo19 transplantiert werden, sind weitere Studien erforderlich, um ihr Potenzial zur Ablagerung von Zahnschmelz ähnlich dem eines natürlichen menschlichen Zahnes zu analysieren.
Insgesamt stellt das neu entwickelte Organoidmodell ein vielversprechendes, wertvolles Werkzeug zur Untersuchung der Biologie des menschlichen Zahnes (Stammzell) und der Amelogenese dar, die beide derzeit nur wenig erforscht sind, mit Zukunftsperspektiven in Richtung Zahnkrankheitsmodellierung und regenerative Therapien.
Offenlegungen
Der korrespondierende Autor stellt sicher, dass alle Autoren alle Interessenkonflikte offengelegt haben.
Danksagungen
Wir danken allen Mitarbeiterinnen und Mitarbeitern der Mund-, Kiefer- und Gesichtschirurgie (MKA) der UZ Leuven sowie den Patienten für ihre unschätzbare Hilfe beim Sammeln frisch extrahierter dritter Molaren. Wir danken auch Dr. Reinhilde Jacobs und Dr. Elisabeth Tijskens für ihre Hilfe bei der Probensammlung. Diese Arbeit wurde durch Zuschüsse der KU Leuven (BOF) und der FWO-Flanders (G061819N) unterstützt. L.H. ist ein FWO Ph.D. Fellow (1S84718N).
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL Microcentrifuge tube | Eppendorf | 30120.086 | |
| 15 mL Centrifuge tube | Corning | 430052 | |
| 2-Mercaptoethanol | Sigma-Aldrich | M-6250 | |
| 48-well flat bottom plates | Corning | 3548 | |
| 50 mL Centrifuge tube | Corning | 430290 | |
| A83-01 | Sigma-Aldrich | SML0788 | |
| Agarose | Lonza | 50004 | |
| Albumin Bovine (cell culture grade) | Serva | 47330.03 | |
| AMELX antibody | Santa Cruz | sc-365284 | |
| Amphotericin B | Gibco | 15200018 | |
| B27 (without vitamin A) | Gibco | 12587-010 | |
| Cassette | VWR | 7202191 | |
| Catalase from bovine liver | Sigma-Aldrich | C100 | |
| CD44 antibody | Abcam | ab34485 | |
| Cell strainer, 40 µm | Falcon | 352340 | |
| Cholera Toxin | Sigma-Aldrich | C8052 | |
| Citric acid | Sigma-Aldrich | C0759 | |
| CK14 antibody | Thermo Fisher Scientific | MA5-11599 | |
| Collagenase IV | Gibco | 17104-019 | |
| Cover glass | VWR | 6310146 | |
| Cryobox | Thermo Scientific | 5100-0001 | |
| Cryovial | Thermo Fisher Scientific | 375353 | |
| Dimethylsulfoxide (DMSO) | Sigma-Aldrich | D2650 | |
| Dispase II | Sigma-Aldrich | D4693 | |
| DMEM 1:1 F12 without Fe | Invitrogen | 074-90715A | |
| DMEM powder high glucose | Gibco | 52100039 | |
| Dnase | Sigma-Aldrich | D5025-15KU | |
| Donkey serum | Sigma-Aldrich | D9663 - 10ML | |
| Embedding workstation, 220 to 240 Vac | Thermo Fisher Scientific | 12587976 | |
| Ethanol absolute, ≥99.8% (EtOH) | Fisher Chemical | E/0650DF/15 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F7524 | |
| FGF10 | Peprotech | 100-26 | |
| FGF2 (= basic FGF) | R&D Systems | 234-FSE-025 | |
| FGF8 | Peprotech | AF-100-25 | |
| GenElute Mammaliam Total RNA Miniprep Kit | Sigma-Aldrich | RTN350-1KT | Includes 1% β-mercaptoethanol dissolved in lysis buffer |
| Glass Pasteur pipette | Niko Mechanisms | 170-40050 | |
| Glycine | VWR | 101194M | |
| HEPES | Sigma-Aldrich | H4034 | |
| IGF-1 | PeproTech | 100-11 | |
| InSolution Y-27632 (ROCK inhibitor, RI) | Sigma-Aldrich | 688001 | |
| Insulin from bovine pancreas | Sigma-Aldrich | I6634 | |
| ITGA6 antibody | Sigma-Aldrich | HPA012696 | |
| L-Glutamine | Gibco | 25030024 | |
| Matrigel (growth factor-reduced; phenol red-free) | Corning | 15505739 | |
| Microscope slide | Thermo Fisher Scientific | J1800AMNZ | |
| Millex-GV Syringe Filter Unit, 0.22 μm | Millipore | SLGV033R | |
| Minimum essential medium eagle (αMEM) | Sigma-Aldrich | M4526 | |
| mouse IgG (Alexa 555) secondary antibody | Thermo Fisher Scientific | A-31570 | |
| N2 | Gibco | 17502-048 | |
| N-acetyl L-cysteine | Sigma-Aldrich | A7250 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Noggin | PeproTech | 120-10C | |
| P63 antibody | Abcam | ab124762 | |
| Pap Pen | Sigma-Aldrich | Z377821-1EA | Marking pen |
| Paraformaldehyde (PFA), 16% | Merck | 8.18715 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penicillin-streptomycin (Pen/Strep) | Gibco | 15140-122 | |
| Petri dish | Corning | 353002 | |
| Phosphate buffered saline (PBS) | Gibco | 10010-015 | |
| Pipette (P20, P200, P1000) | Eppendorf or others | 2231300006 | |
| Plastic transfer pipette (3.5 mL) | Sarstedt | 86.1171.001 | |
| Rabbit IgG (Alexa 488) secondary antibody | Thermo Fisher Scientific | A21206 | |
| RSPO1 | PeproTech | 120-38 | |
| SB202190 (p38i) | Biotechne (Tocris) | 1264 | |
| Scalpel (surgical blade) | Swann-Morton | 207 | |
| SHH | R&D Systems | 464-SH-200 | |
| Silicone molds (Heating block) | VWR | 720-1918 | |
| Sodium Chloride (NaCl) | BDH | 102415K | |
| Sodium Hydrogen Carbonate (NaHCO3) | Merck | 106329 | |
| Sodium-pyruvate (C3H3NaO3) | Sigma-Aldrich | P-5280 | |
| SOX2 antibody | Abcam | ab92494 | |
| StepOnePlus | Thermo Fisher Scientific | Real-Time PCR System | |
| Stericup-GP, 0.22 µm | Millipore | SCGPU02RE | |
| Steriflip-GP Sterile Centrifuge Tube Top Filter Unit, 0.22 μm | Millipore | SCGP00525 | |
| Sterile 1000 μL pipette tips with filter | Greiner | 740288 | |
| Sterile 20 μL pipette tips with filter | Greiner | 774288 | |
| Sterile 200 μL pipette tips with and without filter | Greiner | 739288 | |
| Sterile H2O | Fresenius | B230531 | |
| Streptomycin sulfate salt | Sigma-Aldrich | S6501 | |
| Superscript III first-strand synthesis supermix | Invitrogen | 11752-050 | Reverse transcription kit |
| Tissue processor | Thermo Scientific | 12505356 | |
| Transferrin | Serva | 36760.01 | |
| Triton X-100 | Sigma | T8787-50ML | |
| TrypLE express | Gibco | 12605-010 | |
| Vectashield mounting medium+DAPI | Labconsult NV | H-1200 | Antifade mounting medium with DAPI |
| WNT3a | Biotechne (Tocris) | 5036-WN-500 | |
| Xylenes, 99%, for biochemistry and histology | VWR | 2,89,75,325 |
Referenzen
- Yu, T., Klein, O. D. Molecular and cellular mechanisms of tooth development, homeostasis and repair. Development (Cambridge). 147 (2), (2020).
- Arrow, P. Dental enamel defects, caries experience and oral health-related quality of life: a cohort study. Australian Dental Journal. 62 (2), 165-172 (2017).
- Mitsiadis, T. A., Orsini, G., Jimenez-Rojo, L. Dental Stem Cells for Tooth Regeneration. Dental Stem Cells: Regenerative Potential. Stem Cell Biology and Regenerative Potential. Stem Cell Biology and Regenerative Medicine. Zavan, B., Bressan, E. , Humana Press. Cham. (2016).
- Mitsiadis, T. A., Orsini, G. Editorial: a new era in dentistry: stem cell-based approaches for tooth and periodontal tissue regeneration. Frontiers in Physiology. 7, 357(2016).
- Miran, S., Mitsiadis, T. A., Pagella, P. Innovative dental stem cell-based research approaches: the future of dentistry. Stem Cells International. 2016, 7231038(2016).
- Shinmura, Y., Tsuchiya, S., Hata, K. I., Honda, M. J. Quiescent epithelial cell rests of malassez can differentiate into ameloblast-like cells. Journal of Cellular Physiology. 217 (3), 728-738 (2008).
- Davis, E. M. A review of the epithelial cell rests of Malassez on the bicentennial of their description. Journal of Veterinary Dentistry. 35 (4), 290-298 (2018).
- Athanassiou-Papaefthymiou, M., Papagerakis, P., Papagerakis, S. Isolation and characterization of human adult epithelial stem cells from the periodontal ligament. Journal of Dental Research. 94 (11), 1591-1600 (2015).
- Kim, G. -H., et al. Differentiation and establishment of dental epithelial-like stem cells derived from human ESCs and iPSCs. International Journal of Molecular Sciences. 21 (12), 1-16 (2020).
- Nam, H., et al. Establishment of Hertwig's epithelial root sheath/ epithelial rests of malassez cell line from human periodontium. Molecules and Cells. 37 (7), 562-567 (2014).
- Nam, H., et al. Expression profile of the stem cell markers in human hertwig's epithelial root sheath/Epithelial rests of Malassez cells. Molecules and Cells. 31 (4), 355-360 (2011).
- Tsunematsu, T., et al. Human odontogenic epithelial cells derived from epithelial rests of Malassez possess stem cell properties. Laboratory Investigation; A Journal of Technical Methods and Pathology. 96 (10), 1063-1075 (2016).
- Artegiani, B., Clevers, H. Use and application of 3D-organoid technology. Human Molecular Genetics. 27 (2), 99-107 (2018).
- Boretto, M., et al. Patient-derived organoids from endometrial disease capture clinical heterogeneity and are amenable to drug screening. Nature Cell Biology. 21 (8), 1041-1051 (2019).
- Cox, B., et al. Organoids from pituitary as a novel research model toward pituitary stem cell exploration. Journal of Endocrinology. 240 (2), 287-308 (2019).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Boretto, M., et al. Development of organoids from mouse and human endometrium showing endometrial epithelium physiology and long-term expandability. Development (Cambridge). 144 (10), 1775-1786 (2017).
- Schutgens, F., Clevers, H. Human organoids: tools for understanding biology and treating diseases). Annual Review of Pathology. 15, 211-234 (2020).
- Hemeryck, L., et al. Organoids from human tooth showing epithelial stemness phenotype and differentiation potential. Cellular and Molecular Life Sciences. 79 (3), 153(2022).
- Gao, X., Wu, Y., Liao, L., Tian, W. Oral organoids: progress and challenges. Journal of Dental Research. 100 (5), 454-463 (2021).
- Binder, M., et al. Novel strategies for expansion of tooth epithelial stem cells and ameloblast generation. Scientific Reports. 10 (1), 4963(2020).
- Xiong, J., Mrozik, K., Gronthos, S., Bartold, P. M. Epithelial cell rests of malassez contain unique stem cell populations capable of undergoing epithelial-mesenchymal transition. Stem Cells and Development. 21 (11), 2012-2025 (2012).
- Luan, X., Ito, Y., Diekwisch, T. G. H. Evolution and development of Hertwig's epithelial root sheath. Developmental Dynamics. 235 (5), 1167-1180 (2006).
- Fukumoto, S., et al. New insights into the functions of enamel matrices in calcified tissues. Japanese Dental Science Review. 50 (2), 47-54 (2014).
- Consolaro, A., Consolaro, M. F. M. O. ERM functions, EGF and orthodontic movement or Why doesn't orthodontic movement cause alveolodental ankylosis. Dental Press Journal of Orthodontics. 15 (2), 24-32 (2010).
- Guajardo, G., et al. Immunohistochemical localization of epidermal growth factor in cat paradental tissues during tooth movement. American Journal of Orthodontics and Dentofacial Orthopedics. 118 (2), 210-219 (2000).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Gonçalves, J., Sasso-Cerri, E., Cerri, P. Cell death and quantitative reduction of rests of Malassez according to age. Journal of Periodontal Research. 43 (4), 478-481 (2008).
- Kim, J., Koo, B. -K., Knoblich, J. A. Human organoids: Model systems for human biology and medicine. Nature Reviews. Molecular Cell Biology. 21 (10), 571-584 (2020).
- Razmi, M. T., Narang, T., Handa, S. ADULT (acro-dermato-ungual-lacrimal-tooth) syndrome: a case report from India. Indian Dermatology Online Journal. 9 (3), 194(2018).
- Future Health Biobank. , Available from: https://futurehealthbiobank.com/ch-en/ (2022).
- Schreurs, R. R. C. E., Baumdick, M. E., Drewniak, A., Bunders, M. J. In vitro co-culture of human intestinal organoids and lamina propria-derived CD4+ T cells. STAR Protocols. 2 (2), 100519(2021).
- Fiorini, E., Veghini, L., Corbo, V. Modeling cell communication in cancer with organoids: Making the complex simple. Frontiers in Cell and Developmental Biology. 8, 166(2020).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
- Zhang, Y., et al. Polyisocyanide hydrogels as a tunable platform for mammary gland organoid formation. Advanced Science. 7 (18), 2001797(2020).
- Mollaki, V. Ethical challenges in organoid use. BioTech. 10 (3), 12(2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten