Method Article
Messung der Eigenschaften des periodischen Membranskeletts des Axon-Anfangssegments mittels 3D-strukturierter Beleuchtungsmikroskopie (3D-SIM)
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt eine Methode zur Visualisierung und Messung von Aktinringen und anderen Komponenten des periodischen Membranskeletts des Axon-Anfangssegments unter Verwendung von kultivierten Ratten-Hippocampus-Neuronen und 3D-strukturierter Beleuchtungsmikroskopie (3D-SIM).
Zusammenfassung
Das Axon-Anfangssegment (AIS) ist der Ort, an dem Aktionspotentiale ausgehen und bildet einen Transportfilter und eine Diffusionsbarriere, die zur Aufrechterhaltung der neuronalen Polarität beitragen, indem sie somato-dendritische Fracht sortieren. Ein periodisches Membranskelett (MPS), das aus periodischen Aktinringen besteht, bietet ein Gerüst zur Verankerung verschiedener AIS-Proteine, einschließlich Strukturproteinen und verschiedener Ionenkanäle. Obwohl neuere proteomische Ansätze eine beträchtliche Anzahl neuartiger AIS-Komponenten identifiziert haben, fehlen Details über die Struktur des MPS und die Rollen seiner einzelnen Komponenten. Der Abstand zwischen einzelnen Aktinringen im MPS (~ 190 nm) erfordert den Einsatz von hochauflösenden Mikroskopietechniken, um die strukturellen Details des MPS aufzulösen. Dieses Protokoll beschreibt eine Methode zur Verwendung kultivierter Hippocampus-Neuronen von Ratten, um die genaue Lokalisation eines AIS-Proteins im MPS relativ zu submembranösen Aktinringen mittels 3D-strukturierter Beleuchtungsmikroskopie (3D-SIM) zu untersuchen. Darüber hinaus wird ein analytischer Ansatz beschrieben, um die Periodizität einzelner Komponenten und ihre Position relativ zu Aktinringen quantitativ zu beurteilen.
Einleitung
Das Axon-Anfangssegment (AIS) ist eine kurze, einzigartig spezialisierte Region des proximalen Axons von Wirbeltierneuronen1. Das AIS umfasst einen Transportfilter und eine Diffusionsbarriere, die für die Aufrechterhaltung der neuronalen Polarität durch Sortierung somato-dendritischer Fracht2,3,4,5,6,7 unerlässlich sind. Darüber hinaus ermöglicht die einzigartige Struktur des AIS, Cluster von spannungsgesteuerten Ionenkanälen unterzubringen, die seine Funktion als Ort der Initiierung von Aktionspotentialen erleichtern8. Den einzigartigen Funktionen des AIS liegt ein hochstabiler Strukturkomplex zugrunde. Die Forschung in den letzten zehn Jahren hat das Vorhandensein eines periodischen Membranskeletts (MPS) gezeigt, das Aktinringe enthält, die durch Spektrin verbunden sind und ein Gerüst für die Verankerung verschiedener AIS-Proteine bieten9,10.
Der Abstand zwischen den Aktinringen im MPS (~190 nm)9,10 liegt unter der Auflösungsgrenze der herkömmlichen Lichtmikroskopie. Frühe Versuche, die Elektronenmikroskopie zur Visualisierung des MPS zu verwenden, waren nicht erfolgreich, da die harten Vorbereitungsverfahren die Struktur des MPS nicht erhalten konnten. Daher haben sich hochauflösende Mikroskopietechniken als von unschätzbarem Wert erwiesen, um einige der strukturellen Details des MPS11 zu erläutern. Das Verständnis des AIS-Strukturkomplexes, der Identität und Funktionen seiner Komponenten und seiner räumlich-zeitlichen Regulation ist jedoch noch unvollständig. Jüngsten proteomischen Studien gelang es, eine beträchtliche Liste von Proteinen zu erstellen, die in der Nähe von Strukturkomponenten des AIS lokalisiert sind12,13. Dennoch fehlen Details zu ihrer Funktion und ihrem genauen Platz im AIS-Komplex. Daher dienen hochauflösende Mikroskopietechniken als wesentliches Werkzeug, um die genauen Positionen dieser Proteine relativ zu anderen MPS-Komponenten zu untersuchen und ihre Funktionen zu untersuchen. Mehrere Lichtmikroskopietechniken können Auflösungen erreichen, die über der Beugungsgrenze des Lichts liegen, einige sind sogar in der Lage, einzelne Moleküle zu lokalisieren. Viele dieser Techniken erfordern jedoch in der Regel spezielle Fluorophore oder Bildgebungspuffer, und die Bildaufnahme ist oft zeitintensiv14.
Die strukturierte 3D-Beleuchtungsmikroskopie (3D-SIM) erfordert aufgrund ihrer Benutzerfreundlichkeit und einfachen Anforderungen an die Probenvorbereitung keine speziellen Reagenzien für die Bildgebung oder Probenvorbereitung, funktioniert gut mit einer Vielzahl von Fluorophoren und Proben, kann problemlos in mehreren Farben implementiert werden und ist in der Lage, Live-Cell-Imaging zu erstellen15. Während die bestmögliche Auflösung der SIM-Karte (~ 120 nm) im Vergleich zu vielen anderen Super-Resolution-Techniken niedrig ist, ist sie für viele Anwendungen ausreichend (z. B. zum Auflösen der Komponenten des MPS in Neuronen). Daher ist es wichtig, die Anforderung für bestimmte Anwendungen zu berücksichtigen, um festzustellen, ob SIM eine geeignete Wahl ist oder ob eine noch höhere Auflösung erforderlich ist. Hier wird ein Protokoll zur Verwendung von kultivierten Hippocampus-Neuronen und 3D-strukturierter Beleuchtungsmikroskopie (3D-SIM) beschrieben, um die Position und Organisation von mutmaßlichen AIS-Proteinen relativ zu Aktinringen im MPS zu untersuchen, wie es in Abouelezz et al.16 implementiert ist.
Protokoll
Primäre Hippocampus-Neuronen, die in diesen Experimenten verwendet wurden16, wurden ab dem Embryonaltag 17 Wistar-Rattenembryonen beiderlei Geschlechts gemäß den ethischen Richtlinien der Universität Helsinki und dem finnischen Recht geerntet.
1. Probenvorbereitung
- Lassen Sie auf High-Fidelity-Glasdeckgläsern die Hippocampus-Neuronen der Ratte 14 Tage lang (14 Tage in vitro) unter spärlichen Kulturbedingungen (~ 5.000-10.000 Zellen / cm2) wachsen.
HINWEIS: Die Verwendung von spärlichen Kulturen reduziert die Wahrscheinlichkeit von überlappenden Neuriten, was zur Quantifizierung des MPS beiträgt. - Fixieren Sie Neuronen mit 4% Paraformaldehyd für 12 Minuten bei Raumtemperatur. Waschen Sie das Deckglas einmal in 0,2% BSA in PBS (BSA-PBS), dann für 10 min in einer 1% igen Lösung von Triton-X in PBS bei Raumtemperatur inkubieren. Einmal in BSA-PBS waschen.
- Verdünnte Anti-Ankyrin-G-Antikörper (1:200) in BSA-PBS. Antikörperlösung in das Deckglas geben und über Nacht bei 4 °C inkubieren. Fügen Sie optional Hühner-Anti-MAP2-Antikörper (siehe Materialtabelle) zur Antikörperlösung hinzu, um die somato-dendritische Domäne abzugrenzen.
- Waschen Sie die Zellen einmal in BSA-PBS, gefolgt von 0,1% Triton-X in PBS, und waschen Sie dann eine letzte Wäsche in BSA-PBS.
- Verdünnte fluorophormarkierte Anti-Maus-Sekundärantikörper (1:200) (siehe Materialtabelle) in BSA-PBS, in das Deckglas geben und bei Raumtemperatur 1 h inkubieren. Waschen Sie die Zellen einmal in BSA-PBS, einmal in 0,1% Triton-X in PBS, dann einmal in PBS.
- Bereiten Sie eine 1 μM-Lösung aus markiertem Phalloidin in PBS vor (siehe Materialtabelle) und fügen Sie sie den Zellen hinzu. Inkubiere für 2 h bei Raumtemperatur. Waschen Sie die Zellen einmal in PBS, einmal in 0,1% Triton-X in PBS, dann einmal in PBS.
HINWEIS: AlexaFluor488-markiertes Phalloidin wurde hier verwendet, aber auch andere Tags funktionieren. - Um das Deckglas auf einem Glasobjektträger zu montieren, tragen Sie einen Tropfen Montagemedien auf einen Glasträger auf, tauchen Sie den Deckglas in deionisiertes Wasser und tupfen Sie ihn mit einem weichen Papiertuch ab (um überschüssiges Wasser zu entfernen).
- Legen Sie das Deckglas auf den Glasschieber. Bei Raumtemperatur für 24 h inkubieren.
HINWEIS: Es wurden keine nachteiligen Auswirkungen auf die Probe unter Verwendung von fest aushärtenden Montagemedien (Brechungsindex 1,47 nach Aushärtung) beobachtet.
- Legen Sie das Deckglas auf den Glasschieber. Bei Raumtemperatur für 24 h inkubieren.
2. Bildgebung
- Wenn möglich, konsultieren Sie vor der Bildgebung das Personal der Mikroskopieanlage. Verwenden Sie einen Tauchölrechner (siehe Materialtabelle), um das für die Probe geeignete Tauchöl auszuwählen.
- Sobald die Proben fertig sind, stellen Sie sicher, dass die Deckgläser sauber und frei von Rückständen oder überschüssigen Montagemedien sind. Verwenden Sie bei Bedarf eine in Wasser oder Ethanol getauchte Baumwollspitze zur Reinigung. Legen Sie die Probe in ein 3D-SIM-fähiges Mikroskop (siehe Materialtabelle) und suchen Sie eine Zelle zum Abbilden.
- Passen Sie die Leistung der relevanten Laserlinien und die Belichtungszeiten an, um das Signal-Rausch-Verhältnis zu maximieren, ohne die Probe signifikant zu bleichen.
HINWEIS: Ein gutes Signal-Rausch-Verhältnis zeigt ein klares, gitterartiges Erscheinungsbild, das sich auf die Probe konzentriert und zu genauen hochauflösenden Rekonstruktionen führt. 488 nm und 640 nm Laserlinien wurden verwendet, um F-Aktin bzw. Ankyrin G zu visualisieren. - Legen Sie die oberen und unteren Grenzen der Probe in der Z-Dimension fest und fahren Sie fort, einen Stapel zu erfassen.
HINWEIS: Hier wurde eine Z-Schritt-Größe von 125 nm verwendet. - Stellen Sie für eine erfolgreiche SIM-Rekonstruktion sicher, dass die Mikroskopie-Software (siehe Materialtabelle) über eine optische Übertragungsdatei (OTF) verfügt, die für die verwendeten Farbstoffe geeignet ist.
HINWEIS: Diese werden in der Regel von spezialisiertem Personal erstellt und gewartet. In Ermangelung eines funktionsfähigen OTF stehen auch Open-Source-Tools zur Verfügung, um Rekonstruktionen auf der Grundlage von Schätzungen durchzuführen17. Bei dieser Arbeit wurden aktuelle OTF-Dateien verwendet, die von spezialisiertem Personal kontrolliert wurden. - Führen Sie den Rekonstruktionsalgorithmus auf dem Stapel aus, um eine hochaufgelöste Rekonstruktion zu erhalten. Überprüfen Sie die Rekonstruktionen auf bekannte Artefakte und passen Sie gegebenenfalls die Parameter an, um sie zu korrigieren.
HINWEIS: Die Standardalgorithmusparameter liefern in der Regel genaue Ergebnisse. Viele dieser Artefakte beinhalten einige sich wiederholende Muster: gestreifte Linien zusammen mit mehreren Richtungen auf einer Z-Ebene, "Ghosting " (wiederholte Merkmale, die in verschiedenen Z-Ebenen auftreten) oder ein sechseckiges "Wabenmuster", das in einigen Bereichen auftaucht. Diese Artefakte können oft mit einer besseren Probenvorbereitung, einem besseren Signal-Rausch-Verhältnis, der Anpassung der Parameter des Rekonstruktionsalgorithmus oder der Sicherstellung, dass der Brechungsindex des gewählten Tauchöls für das Montagemedium und die Probe geeignet ist, behoben werden. Eine ausführlichere Erläuterung von SIM-Artefakten und ein frei verfügbares Tool zur Überprüfung auf das Vorhandensein von Artefakten finden Sie unter Ball et al. 18 - Vergleichen Sie die SIM-Rekonstruktion mit einem Weitfeldbild, um Verbesserungen in der Auflösung zu beobachten.
- Verwenden Sie beim Ausführen einer mehrfarbigen SIM-Karte den Ausrichtungsalgorithmus, um die verschiedenen Kanäle korrekt auszurichten, sobald eine zufriedenstellende Rekonstruktion fertig ist.
HINWEIS: Der Ausrichtungsalgorithmus verwendet eine Ausrichtungsreferenzdatei, die mit einer Mikrokugelperlenvorbereitung gemäß den Anweisungen des Mikroskopherstellers generiert wurde.
3. Bildanalyse
- Aktinringe im MPS haben ein unverwechselbares periodisches Aussehen. Erstellen Sie mit einer Bildanalysesoftware (siehe Tabelle der Materialien) ein Projektionsbild mit maximaler Intensität unter Verwendung aller Fokusebenen, in denen Aktinringe sichtbar sind.
- Zeichnen Sie auf dem Projektionsbild mit maximaler Intensität eine senkrechte Linie über sichtbare benachbarte Ringe und zeichnen Sie die Fluoreszenzintensität zusammen mit der Funktion "Plot Profile" der Software auf.
- Um die mittlere Zwischenspitzenentfernung zu berechnen, notieren Sie sich die lokalen Maxima im Linienprofil und messen Sie den Abstand zwischen einzelnen benachbarten fluoreszierenden Intensitätsspitzen.
HINWEIS: Dies kann leicht berechnet werden, zum Beispiel mit der Funktion 'findpeaks' in MATLAB oder der (Open-Source) GNU Octave Plattform. - Um die Kolokalisation verschiedener Proteine mit Aktinringen im MPS zu bewerten, führen Sie ein Kolokalisierungsanalyseverfahren19,20,21 auf SIM-Rekonstruktionen von Aktin und dem Kandidatenprotein durch. Ziehen Sie manuell eine Auswahl, um das AIS als Region von Interesse für das EzColocalization-Plugin in der Softwareplattform zu definieren19, und führen Sie die Analyse aus, um den Pearson-Korrelationskoeffizienten (PCC) der Kolokalisierung zu berechnen19,20,21. Ein PCC-Wert nahe 1 weist auf eine starke Kolokalisation hin.
Hinweis: Dieses Protokoll ist in Abbildung 1 zusammengefasst.
Ergebnisse
Unter Verwendung von kultivierten Ratten-Hippocampus-Neuronen und 3D-SIM wird ein Protokoll beschrieben, um Aktinringe und andere Komponenten des MPS im AIS zu visualisieren und zu messen. Rekonstruktionen von Bildstapeln zeigten eine deutliche Periodizität von Aktinringen (Abbildung 2A). In unseren Händen betrug die mittlere Zwischenspitzendistanz der Aktinringe im MPS, visualisiert mit Alexa 488-markiertem Phalloidin, 190,36 ± 1,7 nm (Mittelwert ± SEM). Dies steht im Einklang mit dem zuvor berichteten durchschnittlichen Abstand von ~ 190 nm zwischen den Aktinringen im MPS. In ähnlicher Weise wurde ein Anti-Ankyrin-G-Antikörper verwendet, um Ankyrin G sichtbar zu machen (Abbildung 2B). Die Kolokalisation von Ankyrin G und F-Aktin wurde im AIS unter Verwendung eines Kolokalisierungsanalyseverfahrens getestet, um den PCC der Kolokalisierung zu berechnen19,20,21. Die PCC der Kolokalisation von Ankyrin G und F-Aktinfluoreszenz betrug 0,36 ± 0,03 (Mittelwert ± SEM, Abbildung 2B). Da Ankyrin G und Aktinringe βIV-Spektrin an verschiedene Domänen binden, zeigen sie keine signifikante Kolokalisation. Diese Daten wurden von Abouelezz et al.16 übernommen.

Abbildung 1: Schematische Darstellung des Protokolls für die Probenvorbereitung für die 3D-SIM-Bildgebung. Hippocampus-Neuronen werden aus Rattenembryonen geerntet, dissoziiert und 14 Tage lang auf Glasdeckgläsern in Kultur wachsen gelassen. Die Zellen werden dann mit markiertem Phalloidin und geeigneten Antikörpern fixiert und gefärbt und dann auf Glasobjektträgern für die 3D-SIM-Bildgebung montiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
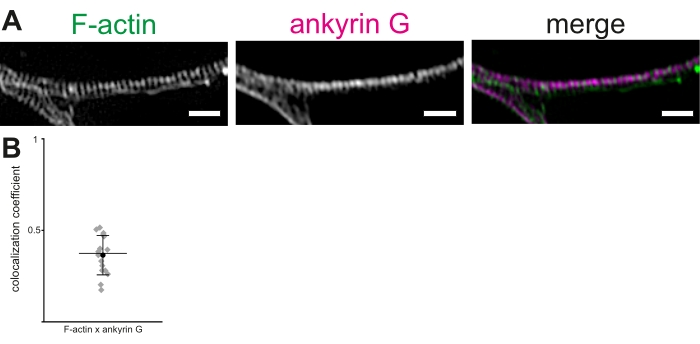
Abbildung 2: 3D-SIM-Rekonstruktion des periodischen Membranskeletts (MPS) im Axon-Anfangssegment (AIS). (A) F-Aktin (grün) und Ankyrin G (Magenta) zeigen eine regelmäßige Verteilung im AIS, visualisiert durch 3D-SIM. Skalenbalken = 1 μm. (B) Pearson's coefficients of correlation (PCC) of colocalization of ankyrin G with F-actin in the MPS in the AIS. Der mittlere PCC von Ankyrin G betrug 0,36 (schwarzer Kreis). Graue Diamanten stellen einzelne Zellen dar (n = 16), die mittlere Linie den Median, Fehlerbalken stellen das 25. und 75. Perzentil dar. Die Daten stammen von Abouelezz et al.16Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Das hier beschriebene Protokoll bietet eine Methode zur Visualisierung und Messung von MPS-Proteinen mit der Super-Resolution-Technik. Da Aktinringe und andere MPS-Komponenten eine Periodizität von ~190 nm9,10 aufweisen, können herkömmliche beugungsbegrenzte Bildgebungsansätze die Details des MPS nicht offenbaren. Mehrere Mikroskopie-Setups können beugungsbegrenzte Strukturen in Superauflösung auflösen, und SIM stellt eine robuste und unkomplizierte Option dar. Wichtig ist, dass SIM mit den am weitesten verbreiteten Fluorophoren kompatibel ist und eine erhebliche Flexibilität bietet. Darüber hinaus ist SIM wirksam bei der Visualisierung potenziell dunkler Strukturen im MPS, wie Aktinringe in mit Latrunculin behandelten Neuronen22, sowie bei lebenden Neuronen15.
Ein wesentlicher Aspekt für den Erfolg dieses Protokolls ist die Wahrung der Integrität des MPS in erster Linie. Die kritischsten Schritte zu einer erfolgreichen SIM-Bildgebung treten während der Probenvorbereitung auf. Zum Beispiel liefern moderate Fixierung (4% PFA für 12 min bei Raumtemperatur) und starke Permeabilisierung (1% Triton-X für 10 min) die besten Ergebnisse. Es ist wichtig zu bedenken, dass harte Behandlungen, die für bestimmte Präparate erforderlich sein können, sich negativ auf die strukturelle Integrität des MPS auswirken können. Daher ist es vielleicht am besten, eine Modifikation solcher Behandlungen in Betracht zu ziehen, um die Erhaltung des MPS zu maximieren.
Der andere entscheidende Faktor, der bei der Vorbereitung von Proben für die Bildgebung zu berücksichtigen ist, ist die Maximierung der Markierungsdichte. Im Idealfall würde jedes Molekül markiert und detektiert. Zum Beispiel wird der in diesem Experiment verwendete Anti-Ankyrin-G-Antikörper häufig verwendet, um das AIS zu kennzeichnen. Es bietet eine hervorragende Leistung in der konventionellen Fluoreszenzmikroskopie, selbst wenn es bei einer Verdünnung von 1:1000 verwendet und bei Raumtemperatur nur 1 h inkubiert wird. Um jedoch eine gute Markierungsdichte für die hochauflösende Mikroskopie zu erhalten, ist es sehr effektiv, diese 5-fache Konzentration (1:200) zu verwenden und den Antikörper über Nacht bei 4 °C zu inkubieren. Während die spezifischen Anforderungen für jeden Antikörper oder jede Markierungstechnik variieren und experimentell bestimmt werden müssen, ist es vielleicht hilfreich, dies im Hinterkopf zu behalten.
Darüber hinaus ist es wichtig zu beachten, dass sich das Erreichen eines hohen Signal-Rausch-Verhältnisses gut für genaue und erfolgreiche SIM-Rekonstruktionen eignet. Eine gute Faustregel ist, dass das Rastermuster in den einzelnen Bildern sichtbar sein sollte, sobald die SIM-Modalität aktiviert ist. Dies ist jedoch nicht immer möglich.
Schließlich ist es wichtig zu beachten, dass SIM zu den schwächsten Superauflösungstechniken bei der Auflösung von Leistung gehört14. Während es also im Allgemeinen ausreicht, um Periodizität und Gesamtorganisation von MPS-Proteinen aufzudecken, ist es deutlich weniger in der Lage, Details über ihre Interaktionen zu liefern als die stochastische optische Rekonstruktionsmikroskopie (STORM)10. Darüber hinaus beschränkt sich der hier beschriebene Nutzen der Technik auf die Untersuchung von Proteinen, für die ein spezifischer, leistungsfähiger Antikörper verfügbar ist. Dies kann jedoch teilweise durch exogene Expression markierter Proteine umgangen werden15.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Dr. Pirta Hotulainen wird für ihre kritischen Kommentare gewürdigt, die für die Vorbereitung dieses Manuskripts von unschätzbarem Wert sind. Dr. Rimante Minkeviciene ist für ihre Hilfe bei der Vorbereitung der neuronalen Kulturen bekannt, die für die ursprünglichen Experimente verwendet wurden. Die gesamte Bildgebung wurde in der Biomedicum Imaging Unit durchgeführt. Diese Arbeit wurde von der Akademie von Finnland (D.M., SA 266351) und dem Doktorandenprogramm Brain & Mind (A.A.) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 3524 | |
| 4% Paraformaldehyde | |||
| Alexa-488 Phalloidin | ThermoFisher | A12379 | |
| Alexa-647 anti-mouse | ThermoFisher | A31571 | |
| Anti-Ankyrin G antibody | UC Davis/NIH NeuroMab Facility, Clone 106/36 | 75-146 | |
| Anti-MAP2 antibody | Merck Millipore | AB5543 | |
| B-27 | Invitrogen | 17504044 | |
| Bovine Serum Albumin (BSA) | BioWest | P6154 | |
| Deltavision OMX SR | GE Healthcare Life Sciences | N/A | |
| Fiji software package | ImageJ | ||
| GNU Octave | GNU | ||
| High performance coverslips | Marienfeld | 117530 | |
| Immersion Oil Calculator | Cytiva Life Sciences | https://tinyurl.com/ImmersionOilCalculator | |
| L-Glutamine | VWR | ICNA1680149 | |
| MATLAB R2020a | Mathworks | ||
| Neurobasal media | Invitrogen | 21103049 | |
| OMX SR | Delta Vision OMX | ||
| Primocin | InvivoGen | ant-pm-1 | |
| ProLong Gold mounting media | Invitrogen | P10144 | |
| softWoRx Deconvolution | Cytiva Life Sciences | ||
| Superfrost Slides | Epredia | ISO 8037/1 | |
| TetraSpeck microspheres 0.1 µm | ThermoFisher | T7279 | |
| Triton-X | Sigma | X100 |
Referenzen
- Leterrier, C. The Axon initial segment, 50 years later: A nexus for neuronal organization and function. Current Topics in Membranes. 77, 185-233 (2016).
- Leterrier, C., Dargent, B. No pasaran! Role of the axon initial segment in the regulation of protein transport and the maintenance of axonal identity. Seminars in Cell & Developmental Biology. 27, 44-51 (2014).
- Brachet, A., et al. Ankyrin G restricts ion channel diffusion at the axonal initial segment before the establishment of the diffusion barrier. Journal of Cell Biology. 191 (2), 383-395 (2010).
- Nakada, C., et al. Accumulation of anchored proteins forms membrane diffusion barriers during neuronal polarization. Nature Cell Biology. 5 (7), 626-632 (2003).
- Song, A. H., et al. A selective filter for cytoplasmic transport at the axon initial segment. Cell. 136 (6), 1148-1160 (2009).
- Sun, X., et al. Selective filtering defect at the axon initial segment in Alzheimer's disease mouse models. Proceedings of the National Academy of Sciences of the United States of America. 111 (39), 14271-14276 (2014).
- Winckler, B., Forscher, P., Mellman, I. A diffusion barrier maintains distribution of membrane proteins in polarized neurons. Nature. 397 (6721), 698-701 (1999).
- Kole, M. H., et al. Action potential generation requires a high sodium channel density in the axon initial segment. Nature Neuroscience. 11 (2), 178-186 (2008).
- Xu, K., Zhong, G., Zhuang, X. Actin, spectrin, and associated proteins form a periodic cytoskeletal structure in axons. Science. 339 (6118), 452-456 (2013).
- Leterrier, C., et al. Nanoscale architecture of the axon initial segment reveals an organized and robust scaffold. Cell Reports. 13 (12), 2781-2793 (2015).
- Leterrier, C. The axon initial segment: An updated viewpoint. The Journal of Neuroscience. 38 (9), 2135-2145 (2018).
- Hamdan, H., et al. Mapping axon initial segment structure and function by multiplexed proximity biotinylation. Nature Communications. 11 (1), 100 (2020).
- Zhou, R., et al. Proteomic and functional analyses of the periodic membrane skeleton in neurons. bioRxiv. , (2020).
- Valli, J., et al. Seeing beyond the limit: A guide to choosing the right super-resolution microscopy technique. Journal of Biological Chemistry. 297 (1), 100791 (2021).
- Wang, T., et al. Radial contractility of actomyosin rings facilitates axonal trafficking and structural stability. Journal of Cell Biology. 219 (5), 201902001 (2020).
- Abouelezz, A., Stefen, H., Segerstråle, M., Micinski, D., Minkeviciene, R., Lahti, L., Hardeman, E., Gunning, P., Hoogenraad, C., Taira, T., Fath, T., Hotulainen, P. Tropomyosin Tpm3.1 is required to maintain the structure and function of the axon initial segment. iScience. 23 (5), 101053 (2020).
- Muller, M., Monkemoller, V., Hennig, S., Hubner, W., Huser, T. Open-source image reconstruction of super-resolution structured illumination microscopy data in ImageJ. Nature Communications. 7, 10980 (2016).
- Ball, G., et al. SIMcheck: a toolbox for successful super-resolution structured illumination microscopy. Scientific Reports. 5, 15915 (2015).
- Stauffer, W., Sheng, H., Lim, H. N. EzColocalization: An ImageJ plugin for visualizing and measuring colocalization in cells and organisms. Scientific Reports. 8 (1), 15764 (2018).
- Dunn, K. W., Kamocka, M. M., McDonald, J. H. A practical guide to evaluating colocalization in biological microscopy. American Journal of Physiology-Cell Physiology. 300 (4), 723-742 (2011).
- Lahti, L. Data analysis supplement to the research article Abouelezz, Stefen, Segerstråle, Micinski, Minkeviciene, Lahti, Hardeman, Gunning, Hoogenraad, Taira, Fath, & Hotulainen. Tropomyosin Tpm3.1 Is Required to Maintain the Structure and Function of the Axon Initial Segment. , (2021).
- Abouelezz, A., Micinski, D., Lipponen, A., Hotulainen, P. Sub-membranous actin rings in the axon initial segment are resistant to the action of latrunculin. Journal of Biological Chemistry. 400 (9), 1141-1146 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten