Method Article
Überwachung der eIF4F-Montage durch Messung der eIF4E-eIF4G-Interaktion in lebenden Zellen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Messung der eIF4E-eIF4G-Interaktion in lebenden Zellen vor, das es dem Anwender ermöglichen würde, die medikamentöse Störung der komplexen EIF4F-Dynamik in Screening-Formaten zu bewerten.
Zusammenfassung
Die Bildung des eIF4F-Komplexes hat sich als der wichtigste nachgeschaltete Knoten für die Konvergenz von Signalsignalen erwiesen, die häufig beim Menschen onkoogene Aktivierung erfahren. eIF4F ist ein Cap-Bindungskomplex, der an der mRNA-ribossomeRekrutierungsphase der TranslationInitiation beteiligt ist. In vielen zellulären und präklinischen Krebsarten führt die Deregulierung von eIF4F zu einer verstärkten Übersetzung spezifischer mRNA-Teilmengen, die an der Proliferation und dem Überleben von Krebs beteiligt sind. eIF4F ist ein heterotrimerkomplex, der aus der Kappenbindungs-Untereinheit eIF4E, dem Helicase eIF4A und der Gerüstuntereinheit eIF4G gebaut wurde. Entscheidend für die Montage aktiver eIF4F-Komplexe ist die Protein-Protein-Wechselwirkung zwischen eIF4E- und eIF4G-Proteinen. In diesem Artikel beschreiben wir ein Protokoll zur Messung der eIF4F-Assembly, das den Status der eIF4E-eIF4G-Interaktion in lebenden Zellen überwacht. Der eIF4e:4G zellbasierte Protein-Protein-Interaktionstest ermöglicht auch eine genaue und zuverlässige Bewertung medikamentös induzierter Veränderungen in der komplexen Integrität von eIF4F. Wir stellen uns vor, dass diese Methode zur Überprüfung der Aktivität kommerziell erhältlicher Verbindungen oder zum weiteren Screening neuartiger Verbindungen oder Modalitäten angewendet werden kann, die die Bildung des eIF4F-Komplexes effizient stören.
Einleitung
Die Kontrolle der Genexpression spielt eine zentrale Rolle bei der korrekten Ausführung zellulärer Programme wie Wachstumsproliferation und Differenzierung. Ein regulatorischer Kontrollmechanismus kann entweder auf der Ebene der Gentranskription oder auf der Ebene der mRNA-Translation ausgeübt werden. In den letzten zehn Jahren ist es immer offensichtlicher geworden, dass die translationale Kontrolle durch Modulation des Initiationsprozesses und nicht durch die späteren Schritte der Dehnung und Beendigung die Synthese bestimmter Teilmengen von Proteinen, die eine breite Palette biologischer Funktionen spielen, fein regulieren kann.
Erhöhte Übersetzung von mRNAs, die am Überleben beteiligt sind, antiautophagische und anti-apoptotische Reaktionen wurden in mehrere Krebsarten verwickelt und wurden auch ursächlich mit der abnormen Aktivierung oder dem Ausdruck von Translationsinitiationsfaktoren in Verbindung gebracht1.
Der eIF4F-Komplex ist ein Master-Regulator der Übersetzungsinitiierung. Durch die Bindung der Cap-Struktur am 5' Ende von mRNAs treibt eIF4F die anfängliche mRNA-ribossome Rekrutierung voran und erhöht damit die mRNA-Translationseffizienz von schwach übersetzten eukaryotischen mRNAs2. EIF4F vermittelte Übersetzung von krebsbedingten mRNAs wurde für viele Krebsmodelle berichtet, die eine abnorme Aktivierung von RAS/MAPK- oder AKT/TOR-Signalwegen beherbergen, was darauf hindeutet, dass Krebszellen eIF4F hochregulieren, um ihre eigene proneoplastische Aktivität zu steigern. Die Störung dieser Einspeiseschleife durch Hemmung der eIF4F-Komplexbildung ist damit eine sehr vielversprechende therapeutische Strategie3,4.
Der eIF4F-Komplex besteht aus (i) eIF4E, der kappenbindenden Untereinheit von eIF4F, die mit der Cap-Struktur der 5' UTR von mRNA interagiert, (ii) eIF4A, dem RNA-Helicase und (iii) eIF4G, dem Gerüstprotein, das sowohl mit eIF4A als auch mit eIF4E interagiert und schließlich die 40S-Riboso-Subunit5rekrutiert. EIF4G-Assoziation mit eIF4E ist der preisbegrenzende Schritt für die Montage funktioneller eIF4F-Komplexe und wird durch die eIF4E-bindenden Proteine (4EBPs, Mitglieder 1, 2 und 3)negativ reguliert. Durch den Wettbewerb mit eIF4G-Bindung an eIF4E über eine Schnittstelle, die aus kanonischen und nicht-kanonischen eIF4E-Bindungssequenzen7,8,9 (Region aa 604-646 auf Human eIF4E) besteht, reduziert 4EBP den Pool von eIF4E, der aktiv an der Übersetzung und Der Verhinderung von eIF4F-Komplexbildung beteiligt ist. Das Zusammenspiel dieser Protein-Protein-Wechselwirkungen wird hauptsächlich durch das Säugetierziel der rapamycin (mTOR)-vermittelten Phosphorylierung von 4EBP reguliert. Bei mitogenen Reizen phosphorisiert mTOR die Mitglieder der 4E-BP-Proteinfamilie direkt, verringert ihre Verbindung mit eIF4E und fördert damit die EIF4E-eIF4G-Interaktion und die Bildung funktioneller eIF4F-Komplexe10.
Trotz der großen Anstrengungen bei der Entwicklung von Verbindungen, die auf die komplexe Integrität von eIF4F abzielen, hat das Fehlen von Assays zur Messung der direkten Störung der eIF4E-eIF4G-Interaktion in lebenden Zellen die Suche nach zellulären aktiven Schlagverbindungen eingeschränkt. Wir haben einen Luziferase-Assay auf Basis eines Coelenterazin-Analogas (z. B. Nanoluc-basierter Komplementierungs-Assay) angewendet, um den Status der eIF4F-Integrität durch die eIF4E-eIF4G-Interaktion in Echtzeit zu überwachen. Das Luziferase-Komplementierungsproteinsystem besteht aus einem 18 kDa-Proteinfragment (SubA) und 11 Aminosäure-Peptidfragment (SubB), das für minimale Selbstassoziation und Stabilität optimiert ist11. Einmal als Fusionsprodukt mit dem menschlichen EIF4E und der eIF4E-Interaktionsdomäne aus dem menschlichen eiF4G1 (aa 604-646) ausgedrückt, werden die beiden interagierenden Proteine das SubA- und SubB-Fragment in die Nähe voneinander bringen und die Bildung der aktiven Luziferase induzieren, die in Gegenwart eines zelldurchlässigen Substrats schließlich ein helles Leuchtsignal erzeugen wird (Abbildung 1). Wir haben an anderer Stelle über den Bau und die Validierung des EIF4E:eIF4G604-646 Ergänzungssystems16berichtet.
Hier beschreiben wir, wie das komplementäre System eIF4E:eIF4G604-646 (auf Anfrage erhältlich) angewendet werden kann, um 4EBP1-vermittelte eIF4E-eIF4G-Störungen in lebenden Zellen genau zu messen. Darüber hinaus zeigen wir seinen Nutzen, indem wir die Wirkung mehrerer mTOR-Inhibitoren messen, die sich derzeit in klinischen Studien als potenzielle Krebstherapeutika befinden12. Da Off-Target-Effekte oft die medikamentöse Aktivität verschleiern, beschreiben wir auch, wie die Vielseitigkeit der EIF4E:eIF4G604-646 Systemmessung mit orthogonalen Messungen der zellulären Lebensfähigkeit erweitert werden kann, um diese zu berücksichtigen.
Protokoll
HEK293 Zelllinie wurde für das Protokoll verwendet und wurde in Dulbecco modifizierte Adler Medium ergänzt mit 10% Fetal Bovine Serum, 2 mM L-Glutamin, und 100 U/mL Penicillin/Streptomycin kultiviert. Die Zellen wurden bei 37 °C mit 5%CO2 in einer befeuchteten Umgebung kultiviert.
1. Quantitative Bewertung der komplexen Störung des eIF4F über eIF4E:eIF4G604-646 Complementation Assay
- Zellkultur und transiente Transfektion von eIF4E:eIF4G604-646 Komplementierungstest
- Verwenden Sie frisch aufgetaute Zellen mit weniger als 20 Passagen für alle Experimente. Bestimmen Sie am ersten Tag die Zellennummer mithilfe eines Standardzellenzählers, und zählen Sie die gesamten lebensfähigen Zellen mit der Trypan Blue Exclusion Methode13.
- Samen 6-Well-Platten mit 0,9 - 1,2 x 106 HEK 293 Zellen pro Brunnen in 2 ml Standardwachstumsmedium.
HINWEIS: Um die beste Transfektionseffizienz innerhalb der oben angegebenen Zelllinien zu erreichen, stellen Sie sicher, dass die vergoldeten Zellen am Tag nach der Aussaat 70-90% konfluent sind. - Am Morgen des 2. Tages werden Zellen mit SubA-eIF4E und eIF4G604-646-SubB-Plasmid mit einem lipidbasierten Transfektionsreagenz (siehe Materialtabelle) wie unten beschrieben, mittransfekt.
- Verdünnen Sie 9 l Liposomenlösung in einem Rohr, das 125 l reduziertes Serummedium ohne Phenolrot enthält (siehe Materialtabelle)für jede Transfektion und inkubieren Bei Raumtemperatur für 5 min.
- Bereiten Sie den Master-Mix der DNA vor, indem Sie für jedes Transfektionsrohr 3 g jedes Plasmids in 125 l reduziertem Serummedium verdünnen.
- Fügen Sie dem DNA-Master-Mix-Rohr 12 l Enhancer-Reagenzien hinzu, mischen Sie sie gut und fügen Sie sofort die DNA hinzu: Enhancer Master-Mix zu jeder Tube verdünnter Liposomen im Verhältnis 1:1. 15 min bei Raumtemperatur inkubieren.
- Fügen Sie den DNA-Lipid-Komplex zu jedem Brunnen hinzu und inkubieren Sie die Zellen in einem 37 °C-Inkubator mit 5%CO2 für 24 h.
- Am Morgen des 3. Tages, spülen Sie jeden Brunnen mit 1 ml PBS.
- Entfernen Sie die PBS und inkubieren Zellen in jedem Brunnen mit 0,3 ml Trypsin für 5 min bei 37 °C.
- Neutralisieren Sie Trypsin, indem Sie 2 ml des reduzierten Serummediums ohne Phenolrot, das 0% FCS in jedem Brunnen enthält, hinzufügen und transfizierte Zellen in ein 15 ml-Rohr übertragen.
HINWEIS: Phenolrot kann die Luziferase-Aktivität stören; Daher müssen die Zellen von diesem Schritt an in Medium ohne Phenolrot behandelt werden. - Zellen für 5 min bei 290 x g abdrehen und das Medium ansaugen. Setzen Sie die Zellen in 2 ml des reduzierten Serummediums ohne Phenolrot mit 0% FCS aus. Zählen Sie wie in Schritt 1.1 beschrieben.
- Samen transfizierte HEK 293 Zellen in 96 gut undurchsichtigen Platten mit einer Dichte von 30.000 Zellen pro Bohrkörper in 90 l Medium ohne Phenolrot, das 0% FCS enthält. Um 3 technische Replikationen innerhalb des gleichen Experiments für 3 verschiedene Verbindungen zu erhalten, säen 60 Brunnen der Platten mit Zellen, die die Brunnen an den Rändern ausschließen.
- Unmittelbar nach dem Aussaat der transfizierten Zellen 10 L mit 10 % DMSO-Verbindungslösung hinzufügen (siehe Schritt 1.2).
- Compound-Vorbereitung
- Bereiten Sie 1 mM Compound-Aktienlösungen vor, indem Sie die Zinsverbindung in 100% DMSO (v/v) auflösen. Um 3 Replikationen für jede zusammengesetzte Titration zu erhalten, verwenden Sie 8 l der 1 mM Compound-Stammlösung.
HINWEIS: Das Volumen von 1 mM Stammmasse verwendet wird, ist mehr als das, was es benötigt wird. Dies geschieht, um Pipettierfehler zu berücksichtigen, falls vorhanden. - Führen Sie eine 2-fache serielle Verdünnung der Stammverbindungslösung mit 4 l des 1 mM-Bestands in 4 l von 100 % DMSO für jeden Titrationspunkt durch.
HINWEIS: Für eine vollständige zusammengesetzte Titration führen Sie 9 serielle Verdünnungen aus dem Ausgangsbestand in 100% DMSO durch. Wenn mehrere Verbindungen getestet werden müssen, verwenden Sie eine 96-Well-Platte und eine Mehrkanalpipette, um diesen Schritt und nachfolgende Verdünnungen zu erleichtern. - Fügen Sie für jeden Punkt der 2-fachen seriellen Verdünnung 36 l Sterilwasser der HPLC-Klasse hinzu, um eine 40-L-L-10-fache Arbeitslösung in 10 % DMSO (v/v) zu erstellen (d. h. 100 bis 0,39 M 10 % DMSO-Stammserie, die zu einer von 10 bis 0,039 M,in 1 % DMSO führt, wenn zellenzugesetzt wird). Wie für die Behandlungskontrolle, bereiten Sie auch eine 10% DMSO nur Lagerlösung in HPLC-Grade sterilen Wasser.
- Fügen Sie den Zellen in der 96 gut opaken Platte 10 l 10 l 10 l 10 l 10-L-Arbeitslösungen hinzu, um die beabsichtigte Endkonzentration mit einer Rest-DMSO-Konzentration von 1% (v/v) bei einem Gesamtvolumen von 100 l zu erzielen und 3 h bei 37 °C mit 5% v/v atmosphärischem CO2zu brüten.
- Bereiten Sie 1 mM Compound-Aktienlösungen vor, indem Sie die Zinsverbindung in 100% DMSO (v/v) auflösen. Um 3 Replikationen für jede zusammengesetzte Titration zu erhalten, verwenden Sie 8 l der 1 mM Compound-Stammlösung.
- Luziferase-Komplementierung und Lebensfähigkeitstest
- Nach 3 h Arzneimittel herstellung das Luziferase-Substratreagenz durch Kombination von 1 Volumen Substrat mit 19 Volumen des Verdünnungsreagenzes (siehe Herstelleranleitung).
HINWEIS: Der Luziferase-Test wird mit einem handelsüblichen Kit durchgeführt (siehe Tabelle der Materialien). - Verwenden Sie eine Mehrkanalpipette, um sofort 25 L Substratreagenz hinzuzufügen.
- Schütteln Sie die Platte bei 350 Umdrehungen von Euro/ Mittel 50 min auf einem Orbital-Shaker bei Raumtemperatur.
- Bewerten Sie die Lumineszenz mit einem Plattenleser. Stellen Sie dazu den Spiegelleser auf Lumineszenz und den Emissionsfilter auf 455. Verwenden Sie eine Messhöhe von 6,5 mm mit einer Messzeit von 1 s.
- Um die Zelllebensfähigkeit zu beschwichtigen, fügen Sie 33 L des Lebensfähigkeits-Assay-Reagenz hinzu und messen Sie dann die Lumineszenz nach 15 min bei Raumtemperatur mit dem Plattenleser erneut.
- Bewerten Sie die Lumineszenz mit dem Plattenleser, indem Sie den Spiegelleser auf Lumineszenz und den Emissionsfilter auf 600 nm einstellen. Verwenden Sie eine Messhöhe von 6,5 mm mit einer Messzeit von 1 s.
HINWEIS: Die Lebensfähigkeit wird durch Messung des intrazellulären ATP-Spiegels bei der Zelllyse gemäß den Anweisungen des Herstellers beurteilt. Multiplexing ist in diesem Fall möglich, da der Lebensfähigkeitstest eine andere Luziferase mit einer anderen Emissionswellenlänge verwendet. - Verwenden Sie die Daten, um die IC50-Werte jeder Verbindung durch Kurve zu bestimmen, die die Daten an eine 4-Parameter-Anpassungskurvengleichung anpasst:
Y = ((A-D)/(1+((x/C)))) + D
Die numerischen Werte D und A müssen im Kurvenanpassungsprozess eingeschränkt werden:
D = Leuchtwert auf der Y-Achse für minimale Kurvenasymptote oder minimale theoretische Reaktionsstufe, die vom eIF4E:4G-Komplementierungstest erwartet wird.
A = Lumineszenzwert auf der Y-Achse für maximale Kurvenasymptote oder maximale theoretische Antwortvariablen, die vom eIF4E:4G-Komplementierungstest erwartet werden.
ANMERKUNG: Der Wert für die minimale Reaktion wird aus der 1% DMSO (v/v) Kontrollbehandlung abgeleitet, und der Wert für die maximale Reaktion wird von der maximalen Reaktion eines mTOR-Inhibitors mit hoher Affinität abgeleitet, der ohne Auswirkungen auf die Zelllebensfähigkeit plateaued hat.
- Nach 3 h Arzneimittel herstellung das Luziferase-Substratreagenz durch Kombination von 1 Volumen Substrat mit 19 Volumen des Verdünnungsreagenzes (siehe Herstelleranleitung).
2. Korrelierende eIF4E:eIF4G604-646 Assay-Hemmung mit eIF4F-komplexer Störung in Zellen
- Um zu bestätigen, dass das vom Test gemessene Signal der physikalischen Störung der eIF4E-eIF4G-Wechselwirkung durch die Verbindung, Samenzellen, wie in Schritt 1.1 beschrieben, entspricht.
- Ersetzen Sie am Tag danach das Medium durch 1 ml reduziertes Serummedium, das kein Phenolrot enthält, und brüten sie 4 h durch die Verbindung von Interesse. Stellen Sie eine Rest-DMSO-Konzentration von 1% (v/v) bei einem Gesamtvolumen von 1 ml sicher.
ANMERKUNG: Um eine Korrelation mit dem Ergebnis zu ermöglichen, verwenden Sie zusammengesetzte Konzentrationen am Anfang, Mittelpunkt und Endpunkt der gemessenen Komplementierungs-Assay-Titrationskurve. - Lyse-Zellen und durchgeführt m7GTP ziehen nach unten, um eIF4F und eIF4E:4EBP1 Komplexe zu isolieren. Detaillierte Verfahren zur Durchführung des m7GTP Pull-Down-Experiments finden Sie an anderer Stelle14.
- Detektieren Sie m7GTP-gebundene eIF4G-, eIF4E- und 4EBP1-Proteinspiegel durch Western-Blot-Analyse und korrelieren Sie dann mit eIF4F-komplexer Störung mit Komplement-Assay-Signalhemmung.
Ergebnisse
Um die Empfindlichkeit des Komplementierungssystems eIF4E:eIF4G604-646 zu validieren, wurde die 4EBP1-vermittelte Hemmung der eIF4F-Komplexbaugruppe mit mTOR-Inhibitoren bewertet. Durch hemmende mTORC1-Kinase-abhängige Phosphorylierung der 4EBP-Proteinfamilie verbessert die mTOR-Hemmung die 4EBP1-Assoziation mit eIF4E und damit eIF4F-Demontage15. Es wurden zwei Klassen mechanistisch unterschiedlicher MTOR-Kinasen getestet: Rapalogs (z.B. Rapamycin), die allosterische Inhibitoren von mTORC1 sind, aber keine mTORC2- und ATP-Wettbewerbshemmer (z. B. PP242), die sowohl mTORC1 als auch mTORC2 kinase-katalytische Aktivität gezielt hemmen.
HEK293-Zellen wurden mit dem komplementationssystem eIF4E:eIF4G604-646, wie in Schritt 1 beschrieben, transfiziert. Nach 24 h Transfektion wurden die Zellen wieder gesät und mit den mTOR-Inhibitoren PP242 und Rapamycin (wie in Schritt 1.2 beschrieben) behandelt. Vier Stunden nach der Behandlung wurde die Lumineszenz, wie zuvor beschrieben, bewertet, gefolgt von der Zelllebensfähigkeit. Wie in Abbildung 2dargestellt, erzeugt PP242 eine dosisabhängige Hemmung des Signals mit einem berechneten IC50 von 0,72 ± 0,04 m, während ein IC50 von 6,88 ± 0,88 m für Ramamycin abgeleitet wird ( Abbildung2A). Die Platten wurden dann für den Zelllebensfähigkeitstest multiplexiert (Abbildung 2D). Diese Analyse zeigt, dass weder PP242 noch Rapamycin eine signifikante Abnahme der Zelllebensfähigkeit bewirken, was beweist, dass die Abnahme der Lumineszenz im Komplementierungssystem eIF4E:eIF4G604-646 nicht auf den unspezifischen Zelltod, sondern auf eine Störung der EIF4E:4G-Wechselwirkung zurückzuführen ist.
Ein m7GTP-Pulldown-Experiment, das durch Inkubation untransfizierter Zellen mit zusammengesetzten Konzentrationen durchgeführt wurde, die den Anfangs-, Mittel- und Endpunkten der gemessenen Titrationskurve in Abbildung 2A entsprechen, zeigt, dass 4EBP1-vermittelte Störungen der endogenen eIF4E-eIF4G-Wechselwirkung mit dem gemessenen EIF4E:eIF4G604-646 Assaysignal korrelieren (Abbildung 2B, 2C). In Übereinstimmung mit diesen Ergebnissen zeigt sich PP242 als ein stärkerer Inhibitor der gesamten 4EBP1-Phosphorylierung als Rapamycin unter den experimentellen Bedingungen, die in HEK 293-Zellen getestet wurden (Abbildung 3A), während beide Inhibitoren einen Einfluss auf die mTOR-Signalisierung zeigten, wobei Ramamycin gegen mTORC1-Substrate und PP242, die sowohl auf mTORC1 als auch für mTORC2 abzielten, aktiver war (Abbildung 3B).
Zusammengenommen zeigten diese Ergebnisse, dass PP242 die komplexe Bildung von eIF4F effektiver stört als Rapamycin in HEK293-Zellen und zeigen ferner, dass das EIF4E:eIF4G604-646-System die komplexe EIF4F-Komplexzusammensetzung in lebenden Zellen genau messen kann.
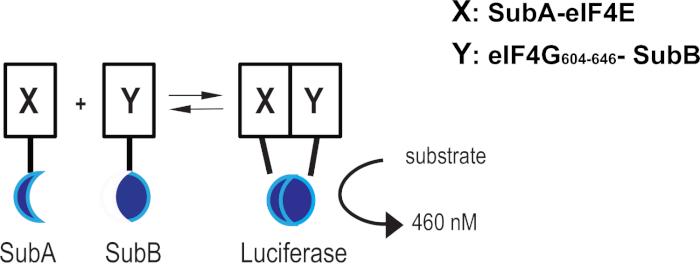
Abbildung 1: eIF4E:eIF4G604-646 Komplementierungssystem. Schematische Darstellung zeigt, wie die Wechselwirkung von Protein X (eIF4E) und Protein Y (eIF4G604-646) subA- und SubB-Fusionen in die Nähe zueinander kommen und die aktive Luziferase rekonstituieren kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: 4EBP1-vermittelte Störung des eIF4F-Komplexes. (A) PP242 und Rapamycin eIF4E:eIF4G604-646 Assay Titration modellierung der Wechselwirkung zwischen eIF4E und eIF4G in transfizierten HEK 293 Zellen. (B,C) Western Blot-Analyse, die den endogene Gehalt an eIF4E, eIF4G und 4EBP1 in HEK 293-Extrakten und in m7GTP zeigt, ziehen nach inkubierender Zellen mit unterschiedlicher Konzentration von PP242 bzw. Rapamycin nach unten. (D) Behandelte Zellen in A, bei denen Multiplexe für Zelllebensfähigkeit und Lumineszenz bewertet wurden. Alle Werte stehen für mittelwert ± SD (n=3). Diese Zahl wurde von16geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
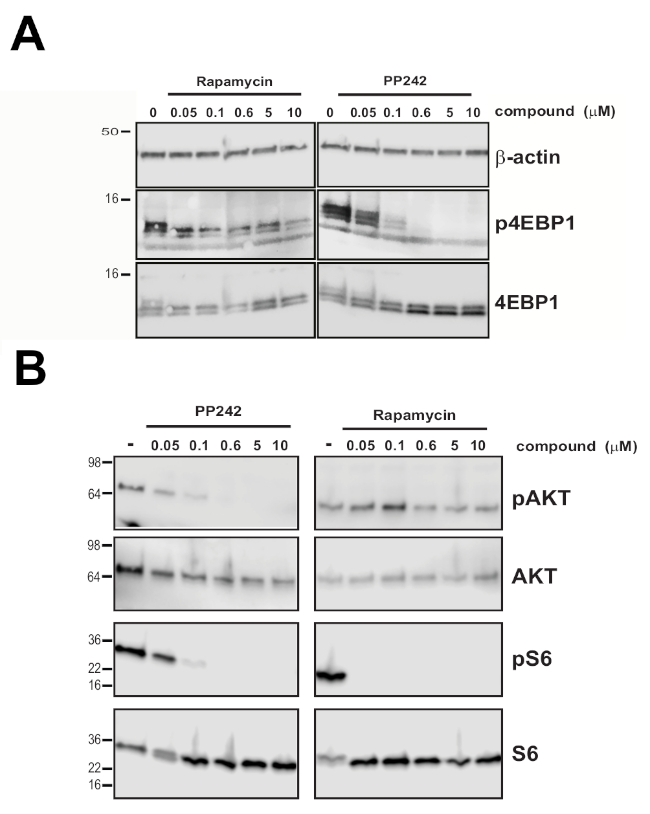
Abbildung 3: Differentialwirkung der mTOR-Hemmung auf 4EBP1-Phoshorylierung. (A) Western Blot Analyse des Phosphorylierungsstatus von 4EBP1 in nicht transfizierten HEK293-Zellen, die mit einer angegebenen Konzentration von PP242 und Rapamycin behandelt wurden. (B) Western Blot Analyse des AKT- und S6-Phosphorylierungsstatus in nicht transfizierten HEK293-Zellen, die entweder mit den dualen MTORC1/2-Aktiv-Standortinhibitoren PP242 oder dem allosterischen Inhibitor von mTORC1 Rapamycin behandelt wurden. Beta-Actin wurde für die Ladekontrolle sowie insgesamt 4EBP1, AKT und S6 visualisiert. Diese Zahl wurde von16geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die in diesem Artikel beschriebene Methode nutzt einen luziferase-basierten Komplementierungstest zur quantitativen Überwachung der komplexen EIF4F-Montage durch direkte Messung der eIF4G-eIF4E-Interaktion in lebenden Zellen. Wir haben Einzelheiten für die Verwendung des eIF4E-eIF4G-Komplementierungssystems zur Verfügung gestellt und auch gezeigt, dass das System bei der Messung der medikamentös induzierten 4EBP1-vermittelten Dissoziation der eIF4E-eIF4G-Interaktion16extrem genau ist. Um den Durchsatz dieses Assays zu erleichtern, wurde das in diesem Artikel beschriebene Versuchsaufbau für eine 96-Well-Mikroplattenformat-Nutzung entwickelt.
Für optimale Ergebnisse sollten bei der Durchführung des Assays zwei kritische Schritte berücksichtigt werden. Erstens sollte die Transfektionseffizienz zwischen den Experimenten ähnlich bleiben. Dies kann durch die Verwendung von Zellen mit geringer Durchgangszahl und durch rigoroses Zählen von Zellen am Tag der Aussaat sichergestellt werden. Die Zellkonfluenkantinz sollte auch vor der DNA-Transfektion beurteilt werden, da es nicht empfohlen wird, Zellen mit lipidbasiertem Transfektionsreagenz zu transfekieren, wenn die Zellkonfluenz weniger als 70-90% beträgt. Zweitens ist es wichtig, den Komplementierungstest mit einem Zelllebensfähigkeitstest zu multiplexen. Einige Verbindungen können das Luziferase-Signal in erster Linie durch die Verringerung der Anzahl der lebensfähigen Zellen durch schädliche Effekte beeinflussen. Daher ist es wichtig, die Lebensfähigkeit der Zelle unmittelbar nach dem EIF4E:eIF4G604-646 Komplementierungstest zu messen, um außerhalb des Ziels und unspezifische Auswirkungen anzugehen.
Protein-Protein-Schnittstellen, wie die zwischen eIF4E und eIF4G, die frei von hydrophoben Spalten sind und relativ groß und planar sind, werden im Allgemeinen von herkömmlichen kleinen Molekültherapeutika (<500 MW)17als "nicht medikamentös" angesehen. So wächst das Interesse an der Entwicklung neuartiger Modalitäten, die effizient mit diesen Oberflächen (z. B. makrozyklische und peptidomimetische Verbindungen) interagieren. Viele dieser neuartigen Modalitäten sind jedoch nicht von Natur aus in der Lage, die Zellmembran zu überqueren und ihr Ziel zu erreichen. Um diese Probleme zu umgehen, forschen viele Forschungsgruppen an neuen Strategien zur chemischen Optimierung und Zellabgabe. Wir stellen uns vor, dass der eIF4E:eIF4G604-646 Live-Zell-PPI-Assay und andere ähnliche PPI-abgeleitete Assays eine zentrale Rolle bei der Förderung dieser Strategien und deren Validierung spielen werden.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch das Kernbudget des p53lab (BMSI, A*STAR) und des JCO VIP Grants (A*STAR) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| Cell culture microplate 96 well, F-Bottom | greiner bio-one | 655083 | |
| Cell titer Glo 2.0 | PROMEGA | G9241 | |
| Envision Multilabel Reader | PerkinElmer | not applcable | |
| Finnpipette F2 Multichannel Pipettes 12-channels 30-300 ml | Thermo Fisher Scientific | 4662070 | |
| Finnpipette F2 Multichannel Pipettes 12-channels 5-50 ml | Thermo Fisher Scientific | 4662050 | |
| FUGENE6 | PROMEGA | E2692 | |
| Lipofectamine 3000 | Thermo Fisher Scientific | L3000015 | |
| NanoBiT PPI Starter Systems | PROMEGA | N2014 | |
| Optimem I Reduced Serum Mediun, no phenol red | Thermo Fisher Scientific | 11058021 | |
| Orbital shaker | Eppendorf | not appicable | |
| γ-Aminophenyl-m7GTP (C10-spacer)-Agarose | Jena Bioscience | AC-155S |
Referenzen
- Silvera, D., Formenti, S. C., Schneider, R. J. Translational control in cancer. Nature Reviews Cancer. 10 (4), 254-266 (2010).
- Gebauer, F., Hentze, M. W. Molecular Mechanism of translational control. Nature Reviews Molecular Cell Biology. 10, 827-835 (2004).
- Bhat, M., et al. Targeting the translation machinery in cancer. Nature Reviews Drug Discovery. 14 (4), 261-278 (2015).
- Pelletier, J., Graff, J., Ruggero, D., Sonenberg, N. Targeting the eIF4F translation initiation complex: a critical nexus for cancer development. Cancer Research. 75 (2), 250-263 (2015).
- Montanaro, L., Pandolfi, P. P. Initiation of mRNA translation in oncogenesis: the role of eIF4E. Cell Cycle. 11, 1387-1389 (2004).
- Merrick, W. C. eIF4E: A retrospective. Journal of Biological Chemistry. 290 (40), 24091-24099 (2015).
- Marcotrigiano, J., Gingras, A. C., Sonenberg, N., Burley, S. K. Cap-dependent translation initiation in eukaryotes is regulated by a molecular mimic of eIF4G. Molecular Cell. 3 (6), 707-716 (1999).
- Umenaga, Y., Paku, K. S., In, Y., Ishida, T., Tomoo, K. Identification and function of the second eIF4E-binding region in N-terminal domain of eIF4G: comparison with eIF4E-binding protein. Biochemical and Biophysical Research Communication. 414 (3), 462-467 (2011).
- Grüner, S., et al. The Structures of eIF4E-eIF4G Complexes Reveal an Extended Interface to Regulate Translation Initiation. Molecular Cell. 64 (3), 467-479 (2016).
- Gingras, A. C., et al. Hierarchical phosphorylation of the translation inhibitor 4E-BP1. Genes and Development. 15 (21), 2852-2864 (2001).
- Dixon, A. S., et al. NanoLuc Complementation Reporter Optimized for Accurate Measurement of Protein Interactions in Cells. ACS Chemical Biology. 11 (2), 400-408 (2016).
- Sun, S. Y. mTOR kinase inhibitors as potential cancer therapeutic drugs. Cancer Letters. 340 (1), 1-8 (2013).
- Strober, W. Trypan blue exclusion test of cell viability. Current Protocols in Immunology. , Appendix 3: Appendix 3B (2001).
- Sekiyama, N., et al. Molecular mechanism of the dual activity of 4EGI-1: Dissociating eIF4G from eIF4E but stabilizing the binding of unphosphorylated 4E-BP1. Proceedings of the National Academy of Science U. S. A. 112 (30), e4036-e4045 (2015).
- Muller, D., et al. 4E-BP restrains eIF4E phosphorylation. Translation. 1 (2), e25819(2013).
- Frosi, Y., Usher, R., Lian, D. T. G., Lane, D. P., Brown, C. J. Monitoring flux in signalling pathways through measurements of 4EBP1-mediated eIF4F complex assembly. BMC Biology. (1), 40(2019).
- Ran, X., Gestwicki, J. E. Inhibitors of protein-protein interactions (PPIs): An analysis of scaffold choices and buried surface area. Current Opinion in Chemical Biology. 44, 75-86 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten