Method Article
胰腺导管细胞系中 pH 值改变的癌基因表达分析
摘要
本研究描述了一种通过胰腺导管细胞系的 RNA-seq 分析来探索 pH 值改变对癌基因表达影响的方案。
摘要
癌症相关死亡的第四大原因胰腺导管腺癌 (PDAC) 的五年生存率为 12%。这种疾病预后不良,其特征是刚性基质微环境,这代表了其治疗的切实挑战。慢性胰腺炎患者患 PDAC 的风险要高 10 倍;在这些患者中,由于碳酸氢盐功能不足和炎症环境,导管 pH 值从 pH 8.0 降至 pH 6.0。我们的目标是了解在慢性胰腺炎中观察到的酸性环境对胰腺导管细胞系中癌基因表达的影响。
因此,将 80% 汇合的人胰腺导管上皮细胞在 pH 6.0 至 pH 7.2 下孵育 6 h。处理来自细胞的总 RNA 以富集样品中的总 mRNA。通过生物重复的下一代测序 (NGS) 评估基因表达。借助在线工具进行 RNA-seq 分析,鉴定出差异表达基因 (FCs < ± 2.0);在 pH 6.0 、 6.5 和 7.0 时,分别有 90 、 148 和 109 个上调基因和 20 、 14 和 23 个下调基因。4 个癌基因在 pH 6.0 时上调,7 个在 pH 6.5 时上调,7 个在 pH 7.0 时上调。在 pH 6.0、pH 6.5 和 pH 7.0 时上调的常见基因是淋巴细胞特异性蛋白酪氨酸激酶 (LCK) [pH 6.0,FC:2.93;pH 6.5,FC:2.93;pH 7.0,FC:3.32]、FGR 原癌基因、Src 家族酪氨酸激酶 (FGR) [pH 6.0,FC:4.17;pH 6.5,FC:5.25;pH 7.0,FC:5.09] 和具有 SH3 结构域的 ArfGAP, 锚蛋白重复序列和 PH 结构域 3 (ASAP3) [pH 6.0,FC:2.37;pH 6.5,FC:3.84;pH 7.0,FC:2.51]。酸性环境触发原癌基因的激活,这可能触发慢性胰腺炎的肿瘤起始。
引言
胰腺导管腺癌 (PDAC) 是原发癌之一,就相关死亡人数而言,在癌症中排名第四1。PDAC 患者的死亡率明显较高,根据最近的研究,五年生存率仅为 12%。患有慢性胰腺炎(一种导致胰腺炎症的疾病2)的患者更有可能(大约 15-16 倍)发展为 PDAC3。有多种类型的 CP,病因不同,例如酒精相关、遗传性和特发性 CP4。众所周知,CP 期间存在的条件会导致钙化结石的形成、癌细胞的发展,甚至糖尿病。遗传性慢性胰腺炎患者的 PDAC 患病率很高5。
CP 患者胰腺内的一些生理状况可能有利于其他疾病的开始;这些因素包括炎症环境、活性消化酶和胰液的酸性 pH值 6。在炎症领域正在进行广泛的研究,因为炎症会给器官的正常功能造成混乱 7,8,9。然而,pH 调节的不平衡是另一种可能引发细胞不受控制生长的现象10。健康人的胰液是碱性的,有助于中和胃产生的酸性食糜。相比之下,由于碳酸氢盐分泌不足,CP 患者的胰液保持酸性。低水平的碳酸氢盐不能完全中和果汁,这会导致导管内出现微酸性环境11.
早期研究表明,癌细胞适应酸性环境并在其中茁壮成长12。在这种情况下,慢性胰腺炎过程中存在的条件表明这些细胞的增殖和转移具有有利的环境13。因此,我们的研究旨在评估人胰腺导管细胞的形态变化,并评估微酸性条件下的癌基因表达。
研究方案
1. 细胞系的复苏
- 获取人正常胰腺导管细胞系 (HPDEC/H6C7) 并在新鲜培养基中恢复细胞。
- 在水浴中于 37 °C 解冻细胞。
- 新鲜制备完全培养基并使用注射器过滤器过滤。
注:完全培养基含有补充有 10% 胎牛血清 (FBS) 的 Dulbecco 改良 Eagle 培养基 (DMEM)。 - 将 6 mL 完全培养基加入新鲜的 15 mL 锥形管中,加入解冻的细胞悬液,并充分混合。
- 将悬浮液以 1,800 × g 离心 6 分钟。弃去上清液,向沉淀中加入 1 mL 培养基,轻轻混匀。
注:培养基含有 DMEM,含 10% 胎牛血清 (FBS) 和 1% 笔/链球菌抗生素。 - 将细胞逐滴加入含有 4 mL 培养基的 T-25 培养瓶中,并在 37 °C、5% CO2 下孵育。
2. 改变培养基的 pH 值
- 制备 100 mL pH 值为 6.0、6.5 和 7.0 的 0.5 M 磷酸钠缓冲储备液,如 表 1 所示。
- 用 pH 计检查 pH 值,并使用 1 N HCl/1 M NaOH 进行调节。
- 使用 0.45 μm 注射器过滤器在层流罩中对缓冲液进行过滤灭菌。
注意:缓冲液可以储存在 4 °C 直至进一步使用。 - 向 80 mL 培养基中加入 20 mL 无菌 0.5 M 磷酸钠缓冲液,制备 pH 值为 6.0、6.5 和 7.0 的培养基。
- 将 pH 值改变的培养基加入 85-90% 汇合的人胰腺导管细胞中,并在明场显微镜下以 1 小时的间隔检查形态变化 6 小时。
3. RNA 分离
注:从细胞中分离 RNA 必须使用无菌手套,并且必须使用 100% 乙醇对表面进行去污。
- 取出培养基并用磷酸盐缓冲盐水 (PBS) 冲洗细胞。
- 将板放在冰上,向孔中加入 PBS,然后用细胞刮刀刮擦细胞。
注意:添加的 PBS 量应足以覆盖细胞以进行刮擦。无需添加大量 PBS。 - 将悬浮液收集在新鲜管中,并以 8,000 × 离心 10 分钟。丢弃上清液。
- 向沉淀中加入含有 β-巯基乙醇的裂解缓冲液,并在冰上上上下移液 5-10 分钟。
- 向裂解物中加入等体积的 70% 乙醇并充分混合。不要涡旋。
- 将溶液加入硅胶膜柱中,以 >8,000 × 离心 30 秒,然后弃去流出液。
- 向样品中加入洗涤缓冲液,并以 >8,000 × 离心 30 秒。将空管以 >8,000 ×离心 1 分钟。
- 向色谱柱中加入 30-50 μL 不含核酸酶的水,并在室温下放置 5 分钟。将色谱柱以 >8,000 × 离心 5 分钟。
注:RNA 样品可以储存在 -20 °C 直至进一步使用。
4. RNA-seq/转录组
- RNA 完整性评估
- 取 1 μL (25-500 ng) 并与 5 μL 染料缓冲液混合,检查生物分析仪中 RNA 的完整性。
- 将溶液以 500 × g 涡旋 1 分钟,然后短暂旋转。
- 在 72 °C 下对每个样品运行一步聚合酶链反应 (PCR) 3 分钟,然后取管并在冰上孵育 2 分钟,然后用生物分析仪分析。
- 去除 rRNA 的总 RNA
- 向新的 PCR 管中加入 5 μg 总 RNA、50 μL 杂交缓冲液和 1-3 μL 探针混合物。
- 执行以下步骤:第一步,在 70 °C 下变性 10 分钟;第二步,在 37 °C 下杂交 20 分钟;并在 37 °C 下孵育直至使用。
- 在新鲜试管中,加入 500 μL 磁珠和样品;将混合物放在磁力架上 1 分钟,直到溶液变得透明。
- 小心丢弃上清液。取出试管,加入 500 μL 无核酸酶的水洗涤珠子,然后将混合物放回磁力架上 1 分钟。溶液澄清后去除上清液。
- 重复该过程 5-7 次。加入 200 μL 杂交缓冲液并适当混合。将试管在 37 °C 下保持 5 分钟。
- 向上述混合物中加入 100 μL RNA 探针,轻轻混合并在 37 °C 下孵育混合物 15 分钟。
- 短暂旋转管子,然后将其放在磁力架上 1 分钟。
- 将含有 rRNA 耗尽的 RNA 的透明上清液收集在新管中。
- RNA 片段化
- 在新的 PCR 管中,加入 8-10 μL 去除 rRNA 的总 RNA (500 ng/g)、1 μL 10x 缓冲液和 1 μL RNase III。
- 通过敲击和短暂旋转来混合溶液。
- 将试管置于热循环仪中,在 37 °C 下放置 10 分钟。
- 在新鲜试管中,加入 5 μL 核酸结合微珠,然后加入 90 μL 结合溶液浓缩液。
- 向上述混合物中加入 30 μL RNA 片段和 150 μL 100% 乙醇。通过移液 10 次混合样品,并在室温下孵育 5 分钟。
- 将试管放在磁力架上,直到溶液变得透明。取出并丢弃上清液。
- 当试管放在磁力架上时,用 150 μL 乙醇清洗珠子;让混合物静置 30 秒,然后去除上清液。
- 将试管放在磁力架上 1-2 分钟,让剩余的乙醇蒸发。
- 向珠子中加入 12 μL 预热的无核酸酶水,并通过移液 10 次混合样品。
- 在室温下孵育样品 1 分钟。将试管放在磁力架上,澄清后收集上清液。
- 文库制备和 cDNA 制备
- 向 PCR 管中加入 3 μL 片段化的 RNA 样品,然后加入 5 μL 杂交混合物。通过移液几次混合样品,然后短暂旋转样品。
- 将试管在热循环仪中于 65 °C 下放置 10 分钟,然后在 30 °C 下放置 5 分钟。
- 向上述溶液中加入 10 μL 连接缓冲液和 2 μL 连接酶混合物。将反应物在热循环仪中于 30 °C 孵育 1 小时。
- 将逆转录酶预混液加入连接混合物中,并在 70 °C 下孵育 10 分钟,然后在冰上孵育。
- cDNA 扩增
- 将 6 μL cDNA (500 ng) 添加到新的 PCR 管中,然后从含有 46 μL 高保真聚合酶和 1 μL 3' 条形码引物的试管中加入 46 μL 条形码 PCR 混合物。
- 将PCR条件设置如下:第一阶段,在94°C下保持2分钟;第二阶段,在 94 °C 下循环 30 秒,在 50 °C 下循环 30 秒;第三阶段,在 94 °C 下循环 30 s,在 62 °C 下循环 30 s,在 68 °C 下循环 30 s。将反应物在 68 °C 下保持 5 分钟。
- 重复步骤 4.3.5 至 4.3.10 纯化扩增的 cDNA,并用上一步获得的 PCR 产物替换片段化的 RNA 样品。
- 在测序仪 (NGS) 上分析纯化的 RNA 样品,以生成 FASTq 文件形式的数据。
5. 生物信息学
- 通过复制粘贴链接打开在线可用的 Galaxy 工具:https://usegalaxy.org 进入浏览器的搜索选项卡。
- 首次访问此站点时,通过单击 登录 或右上角的 注册 选项来注册个人资料。
- 创建账号后,填写公网名称/用户名和密码登录账号。
- 通过单击左侧面板上的 上传 选项,上传 RNA-seq 分析后获得的 FASTq 文件。
- 从左侧面板的 tools 部分中选择 cutadapt 工具。此工具将修剪低质量序列。
注意:此文件现在可用于映射。 - 单击左侧面板上的 工具 按钮,然后查找 RNA 星形工具。填写详细信息如下:使用 cutadapt 的输出文件,选择 参考基因组 作为 人类参考基因组 (hg38),并将 带注释的连接周围的基因组序列长度 更改为 36。选择输出作为 基因计数 ,然后单击右上角的 运行工具 选项。
注意:此工具将生成一个 BAM 文件。 - 从工具选项中选择 featurecounts 工具,并上传 RNA 星形分析后获得的 BAM 文件。将 minimum mapping quality per read 选项设置为 10,然后单击右上角的 run tool 选项。
- 从右侧面板打开 featurecount 输出文件,并将数据复制到电子表格中。通过将测试中获得的读取计数数除以使用命令 =log (fold change, 2) 在控件中获得的读取计数数来计算相对于对照的倍数变化。
注意:<-2.0 的倍数变化被认为表示下调,而 2.0 >倍数变化被认为是上调。
结果
形态变化
人胰腺导管细胞在酸性培养基中孵育改变了细胞的形态。在酸性条件下,汇合和紧密堆积的细胞模式导致细胞分散,并且细胞死亡随着时间的推移而变得突出。与正常含培养基的细胞相比,导管细胞缩小且体积减小。孵育 6 小时后观察到死亡率发生剧烈变化。24 小时后,细胞数量减少,细胞似乎处于应激状态(图 1、 图 2 和 图 3)。因此,在进一步分析之前,在酸性培养基中将孵育时间延长至 6 h。
RNA-seq 分析
转录组的原始文件可在 Mendeley Data (https://data.mendeley.com/datasets/kd994sftcs/1) 上获得,DOI:10.17632/kd994sftcs.1。NGS 数据显示,在 pH 6.0 时共有 1,389 个基因,在 pH 6.5 时共有 1,346 个基因,在 pH 7.0 时共有 1,427 个基因(补充表 S1)。当考虑失调基因的倍数变化时,在 pH 6.0 、 6.5 和 7.0 时,上调基因的数量分别为 90 、 148 和 109 个。此外,20 、 14 和 23 个基因分别在 pH 6.0 、 6.5 和 7.0 下调 (图 4)。在不同 pH 值水平下上调的癌基因和肿瘤抑制基因如 表 2 和 表 3 所示。在所有三个 pH 水平上调的三个癌基因(图 5)是淋巴细胞特异性蛋白酪氨酸激酶 (LCK)、FGR 原癌基因、Src 家族酪氨酸激酶 (FGR) 和具有 SH3 结构域、锚蛋白重复序列和 PH 结构域 3 的 ArfGAP (ASAP3)。已知 LCK 参与肿瘤细胞迁移的调节,并参与癌症干细胞的自我更新14。Src 家族激酶的激活与癌症的发生和进展有关15。发现 FGR 过表达与肿瘤快速进展有关,导致白血病16 、淋巴瘤17 和多形性胶质母细胞瘤18 等癌症患者的生存率低。ASAP3 被证明在肝细胞癌的细胞增殖中发挥作用19 以及细胞迁移和侵袭20。
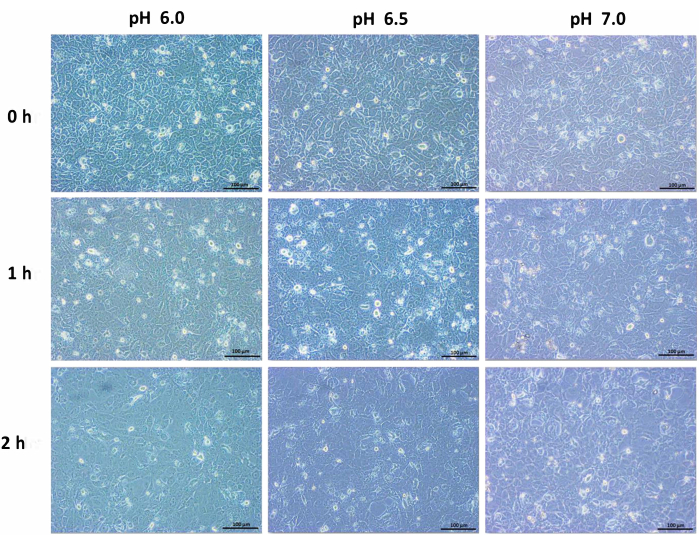
图 1:在 pH 6.0、pH 6.5 和 pH 7.0 下孵育并在改变的培养基中孵育 0 小时、1 小时和 2 小时的人胰腺导管细胞系的 10 倍放大图像。 比例尺 = 100 μm。 请点击此处查看此图的较大版本。
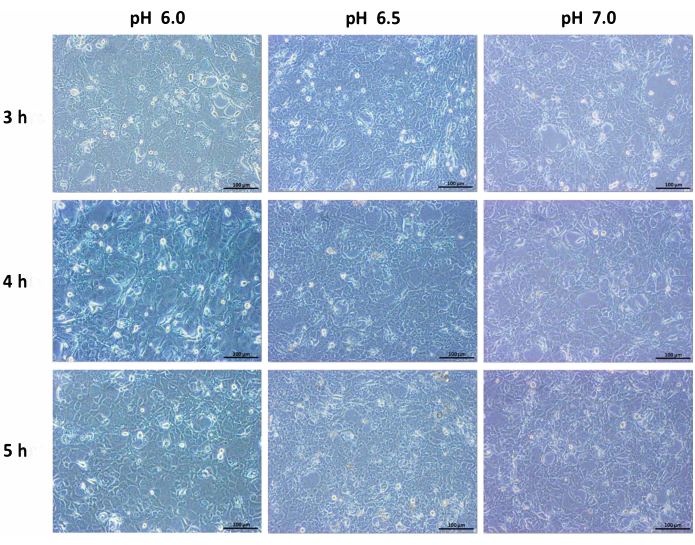
图 2:在 pH 6.0、pH 6.5 和 pH 7.0 下孵育并在改变的培养基中孵育 3 小时、4 小时和 5 小时的人胰腺导管细胞系的 10 倍放大图像。 比例尺 = 100 μm。 请点击此处查看此图的较大版本。

图 3:在 pH 6.0、pH 6.5 和 pH 7.0 下孵育并在改变的培养基中孵育 6 小时和 22 小时的人胰腺导管细胞系的 10 倍放大图像。 比例尺 = 100 μm。 请点击此处查看此图的较大版本。

图 4:pH 6.0、pH 6.5 和 pH 7.0 下失调基因总数的条形图,描绘了在相应 pH 值下上调和下调的基因总数。 请单击此处查看此图的较大版本。
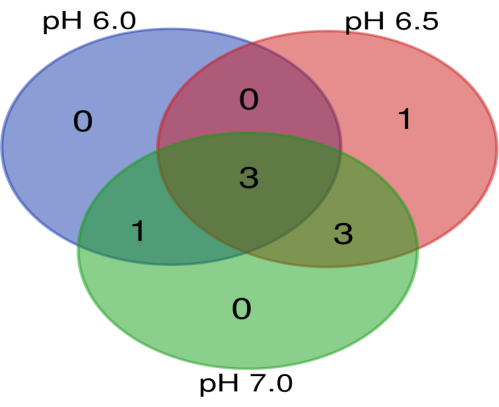
图 5:代表在 pH 6.0、6.5 和 7.0 下测定的常见癌基因的维恩图。 发现在所有 pH 值下通常上调的癌基因是 LCK 、 FGR 和 ASAP3。 请单击此处查看此图的较大版本。
| Sr. No. | 最终 pH 值 | 化学的 | 数量 |
| 1 | pH 值 6.0 | Na2HPO4 Diabasic | 1.835 克 |
| NaH2PO4 单元 | 5.955 克 | ||
| 2 | pH 值 6.5 | Na2HPO4 Diabasic | 4.789 克 |
| NaH2PO4 单元 | 4.434 克 | ||
| 3 | pH 值 7.0 | Na2HPO4 Diabasic | 7.743 克 |
| NaH2PO4 单元 | 2.913 克 |
表 1:磷酸钠缓冲液储备液组成。
| 癌基因 | |||
| 基因 | pH 值 6.0 FC | pH 值 6.5 FC | pH 值 7.0 FC |
| LCK 公司 | 2.93 | 2.93 | 3.32 |
| TNFRSF14 | 3.91 | 1 | 2.58 |
| FGR | 4.17 | 5.25 | 5.09 |
| 尽快 3 | 2.37 | 3.84 | 2.51 |
| MYCL | 1 | 2.94 | 2.69 |
| TAL1 | 1 | 3.05 | 2.36 |
| VTCN1 型 | 1 | 2.25 | 1 |
| EPHA10 | 1 | 2.58 | 2.09 |
表 2:在不同 pH 值下上调的癌基因列表和基因的倍数变化。
| 肿瘤抑制基因 | |||
| 基因 | pH 值 6.0 FC | pH 值 6.5 FC | pH 值 7.0 FC |
| PTCH2 | 5.52 | 6.73 | 6.41 |
| DIRAS3 | 1 | 2.22 | 1 |
| VTCN1 型 | 1 | 2.25 | 1 |
表 3:在不同 pH 值下上调的肿瘤抑制基因列表和基因的倍数变化。
补充表 S1:pH 6 与 7.2、pH 6.5 与 7.2 以及 pH 7 与 pH 7.2 时上调和下调的基因。请点击此处下载此文件。
讨论
开发该方法是为了确定酸性条件对生物重复中非癌性人胰腺导管上皮细胞的影响。培养基 pH 值的变化导致细胞处于应激状态,并观察到形态的变化。将细胞在 pH 6.0、6.5 或 7.0 下培养 24 小时,并每小时监测一次,以优化孵育期以进行进一步实验。我们观察到孵育 4 小时后形态发生变化,在酸性 pH 下孵育 6 小时后细胞死亡率增加。因此,在进行基因表达分析之前,我们将细胞孵育 6 小时。
具有 log2 倍变化的差异表达基因 (DEG) 证明了酸性 pH 值对导管细胞基因表达的影响。癌基因 LCK 、 FGR 和 ASAP3 的表达增加表明酸性 pH 值对导管上皮细胞基因表达的影响。在酸性 pH 条件下癌基因和肿瘤抑制基因表达失调表明 pH 值可能在炎症条件下的肿瘤发生中发挥作用 7,8。
该方法的关键步骤是导管细胞在酸性 pH 下的孵育时间,因为与癌细胞相比,非癌细胞容易受到培养条件变化的影响,这成为进行长孵育期实验的限制因素12。这项研究的一个局限性是我们使用了人胰腺导管细胞系。尽管存在这一限制,我们还是评估了酸性 pH 条件下基因表达的改变,以模拟慢性胰腺炎。
治疗癌症的主要缺点是在癌症过程中形成的微环境和基质。抗癌药物具有杀死癌细胞的能力,但不能侵入微环境,导致药物疗效低下。任何癌症的微环境因素之一是癌症代谢和进展过程中诱导的酸性条件20。可以通过这种方法评估细胞行为变化和对特定类型癌症的药物有效性。可以通过下一代测序评估基因表达,并确定潜在的药物靶标。
披露声明
作者没有需要声明的利益冲突。
致谢
Renuka Goudshelwar 感谢 DBT 为她提供奖学金。Renuka Goudshelwar 感谢奥斯马尼亚大学(海得拉巴)生物化学系的帮助,以及 V. V. Ravikanth 博士在分析 NGS 数据时提供的指导。M. Sasikala 博士感谢印度政府卫生部 ICMR 提供的财政援助(拨款号 94/2020/5582/Proteomics-Adhoc/BMS)。
材料
| Name | Company | Catalog Number | Comments |
| 15 mL graduated centrifuge tubes (Falcon) | Tarsons | 500031 | used for sample preparation |
| 50 mL graduated centrifuge tubes (Falcon) | Tarsons | 500041 | used for sample preparation |
| Agilent 2100 Bioanalyzer instrument | Agilent | Agilent G2938A | Instrment for RNA quality assessment |
| Antibiotic Antimycotic Solution 100x liquid | Himedia | A002-100 mL | Used to prevent contamination |
| Cell Scraper with rotatable blade | Himedia | TCP223 | Scraping and collecting the cells |
| CO2 incubator | New Brunswick | Galaxy 170 S | Used for incubating the culture |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose | Himedia | AL007S-500 mL | Medium used for culturing the cells |
| Dulbecco's Phosphate Buffered saline 1x | Himedia | TL1006-500mL | cell washing |
| Galaxy | (https://usegalaxy.org) | Online tool for processing NSG Data | |
| HI FBS (origin: Australia) | Gibco | 10100-147 | Used for the growth of cells |
| Human Pancreatic Ductal Epithelial Cell Line (HPDEC/ H6C7) | Addex Bio | T0018001 | Pancreatic ductal cell line |
| Ion Total RNA-Seq Kit v2 | Thermo fisher scientific | 4475936 | RNA sample preparation kit |
| Laminar Air Flow | |||
| Na2HPO4 Diabasic | Sigma Aldrich | S3264-250G | Sodium phosphate buffer preparation |
| NaH2PO4 Monobasic | Sigma Aldrich | S3139-250G | Sodium phosphate buffer preparation |
| NovaSeq 6000 | Illumina | 3376672 | Sequencer |
| Nunclon Delta Surface (6-well plate) | Thermo Scientific | 140675 | Culturing the cells |
| Refrigerated benchtop Centrifuge | Thermo Scientific | Sorvall ST 8R | Used for centrifugation |
| RiboMinus Eukaryote System v2 | Thermo fisher scientific | A15026 | rRNA depletion kit |
| Rneasy Mini kit | Qiagen | 74104 | Kit for isolating Total RNA from cells |
| Thermal cycler | Eppendorf | E950040025 | PCR reaction |
| Water Bath | Equitron | #8406M | Thawing of sample |
参考文献
- Franck, C., et al. Advanced pancreatic ductal adenocarcinoma: Moving forward. Cancers (Basel). 12 (7), 1955 (2020).
- Kong, X., Sun, T., Kong, F., Du, Y., Li, Z. Chronic pancreatitis and pancreatic cancer. Gastrointest Tumors. 1 (3), 123-134 (2014).
- Jura, N., Archer, H., Bar-Sagi, D. Chronic pancreatitis, pancreatic adenocarcinoma and the black box in-between. Cell Res. 15 (1), 72-77 (2005).
- Kloppel, G. Chronic pancreatitis, pseudotumors and other tumor-like lesions. Mod Pathol. 20 (Suppl 1), S113-S131 (2007).
- Schneider, A., Whitcomb, D. C. Hereditary pancreatitis: a model for inflammatory diseases of the pancreas. Best Pract Res Clin Gastroenterol. 16 (3), 347-363 (2002).
- Boedtkjer, E., Pedersen, S. F. The acidic tumor microenvironment as a driver of cancer. Annu Rev Physiol. 82, 103-126 (2020).
- Coussens, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Balkwill, F., Mantovani, A. Inflammation and cancer: back to Virchow. Lancet. 357 (9255), 539-345 (2001).
- Clevers, H. At the crossroads of inflammation and cancer. Cell. 118 (6), 671-674 (2004).
- Kato, , et al. Acidic extracellular microenvironment and cancer. Cancer Cell Int. 13 (1), 89 (2013).
- Hegyi, P., Maléth, J., Venglovecz, V., Rakonczay, Z. Pancreatic ductal bicarbonate secretion: challenge of the acinar acid load. Front Physiol. 2, 36 (2011).
- Ibrahim-Hashim, A., Estrella, V. Acidosis and cancer: from mechanism to neutralization. Cancer Metastasis Rev. 38 (1-2), 149-155 (2019).
- Vakkila, J., Lotze, M. T. Inflammation and necrosis promote tumour growth. Nat Rev Immunol. 4 (8), 641-648 (2004).
- Zepecki, J. P., et al. Regulation of human glioma cell migration, tumor growth, and stemness gene expression using a Lck targeted inhibitor. Oncogene. 38 (10), 1734-1750 (2019).
- Kim, R. K., et al. Role of lymphocyte-specific protein tyrosine kinase (LCK) in the expansion of glioma-initiating cells by fractionated radiation. Biochem Biophys Res Commun. 402 (4), 631-636 (2010).
- Hu, Y., et al. Requirement of Src kinases Lyn, Hck and Fgr for BCR-ABL1-induced B-lymphoblastic leukemia but not chronic myeloid leukemia. Nat Genet. 36 (5), 453-461 (2004).
- Sharp, N. A., Luscombe, M. J., Clemens, M. J. Regulation of c-fgr proto-oncogene expression in Burkitt's lymphoma cells: effect of interferon treatment and relationship to EBV status and c-myc mRNA levels. Oncogene. 4 (8), 1043-1046 (1989).
- Hui, A. B., Lo, K. W., Yin, X. L., Poon, W. S., Ng, H. K. Detection of multiple gene amplifications in glioblastoma multiforme using array-based comparative genomic hybridization. Lab Invest. 81 (5), 717-723 (2001).
- Fang, Z. Y., et al. Proteomic identification and functional characterization of a novel ARF6 GTPase-activating protein, ACAP4. Mol Cell Proteomics. 5 (8), 1437-1449 (2006).
- Okabe, H., et al. Isolation of development and differentiation enhancing factor-like 1 (DDEFL1) as a drug target for hepatocellular carcinomas. Int J Oncol. 24 (1), 43-48 (2004).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。