Method Article
来自单个分子的时间分辨荧光各向异性表征生物分子的局部灵活性
摘要
在这里,我们提出了在共聚焦显微镜模式下使用单分子水平的时间分辨荧光各向异性研究生物分子的局部灵活性和动力学的方案。
摘要
我们描述了一种使用共聚焦显微镜在单分子水平进行时间分辨荧光各向异性的方案,以研究 FoxP1 转录因子的脱氧核糖核酸 (DNA) 结合叉头 (FKH) 结构域的局部灵活性和动力学。FoxP1 通过三维结构域交换 (3D-DS) 机制进行二聚化,形成有或没有 DNA 的无序中间体。由于 3D-DS 涉及一个固有的无序区域,因此了解其行为对于阐明 FoxP1 的结构和功能特性至关重要。使用单半胱氨酸标记的 FoxP1,我们进行了单分子荧光各向异性 (smFA) 实验,应用动态各向异性光子分布分析 (daPDA) 和时间分辨各向异性突发方差分析 (traBVA) 方法来探测局部灵活性和动力学。该协议为 smFA 测量提供了详细的分步指南,强调时间分辨分析、方差和概率分布技术,以捕获不同时间尺度的结构动力学。这种方法使我们能够将动力学和异质性与 FoxP1 二聚化和 DNA 结合联系起来,突出了表征该转录因子的复杂作用机制。
引言
生物分子的功能活性取决于其分子柔韧性和结构动力学 1,2,3。自然地,生物分子会经历持续的热波动,从快速运动到影响其功能的长期构象变化(图 1)4。在生物分子中,局部骨架运动有助于更大规模的整体运动,包括酶的铰链弯曲和运动蛋白的显着构象变化。核磁共振 (NMR)5、X 射线晶体学6 和低温电子显微镜 (cryo-EM)7 等结构测定方法揭示了各种生物分子中的多种构象。然而,将局部波动与生物分子的大构象动力学及其在功能中的作用联系起来大多尚未探索。关联动力学和结构可能具有挑战性,尤其是对于固有无序蛋白 (IDP) 8,9,10。与结构化蛋白不同,IDP 不保持稳定的三级结构。相反,它们会经历具有相似自由能水平的广泛构象变化,从而能够进行广泛的生物活动11,12。
已经采用了几种实验方法,通过探测蛋白质的分子灵活性来研究蛋白质的构象动力学 1,13,14,15,16。其中,NMR 因其能够在各种时间尺度(从几十皮秒到几小时)提供原子级分辨率而脱颖而出12。然而,由于高自由度和大尺寸蛋白质,确定大分子柔韧性仍然具有挑战性;因此,NMR 通常仅限于研究约 100 kDa 的生物分子17。
鉴于 IDP 等高动态蛋白的结构复杂性,已经开发了额外的方法学进步来探索局部和远程构象空间以了解其功能11。单分子多参数荧光光谱 (smMFS)18,19,20,21,22 提供了有关生物分子的广泛信息,提供了有关其功能、构象动力学、结合状态和化学计量学的重要见解。然而,解释从生物分子获得的大量结构数据具有挑战性,分子动力学、荧光团行为和分子的复杂行为等因素会使数据分析进一步复杂化 23,24,25,26,27,28。
我们采用单分子荧光各向异性 (smFA) 作为评估沿生物分子骨架的局部和全局动力学的稳健方法(图 1A)。荧光各向异性首先由 Perrin29 描述,并由 Weber30,31 作为生物分析工具32 引入,后来随着时间分辨荧光技术的出现和检测器灵敏度的提高,它被适用于单分子研究 33,34,35,36,37.smFA 跨越了广泛的时间尺度(从皮秒到几小时),并补充了从单分子 Förster 共振能量转移 (smFRET) 实验中获得的数据38。
smFA 可以以各种格式可视化,以提取有关生物分子动力学的关键信息(图 1B)。时间分辨荧光各向异性衰减是一维直方图,可捕获皮秒到纳秒时间尺度上的动态39,40。二维单分子直方图将荧光寿命与单个分子的各向异性相关联,可以揭示各向异性状态的异质性,并在共聚焦实验 (~ms) 的观察时间内提供对潜在动力学的直观见解41,42。为了研究亚毫秒动力学,可以使用动态各向异性光子分布分析 (daPDA),而时间分辨各向异性突发方差分析 (traBVA) 提供了一种可靠的方法来确认毫秒43 附近的特定动力学(图 1B)。
这些方法补充了更传统的工具,例如偏振分辨荧光相关光谱 (pFCS),它具有更宽的光谱 44,45,46,47。总体而言,只要考虑适当的校准,smFA 的多种数据分析工具有助于识别局部和全局构象变化。
在这里,我们应用 smFA 来研究人 FoxP1 转录因子 48,49,50,51 的 DNA 结合。由于其多肽链的内在无序性质,这种蛋白质采用结构域交换二聚体,其受到显着影响,具体取决于蛋白质的四级状态和 DNA 的存在。我们生成了不同的单半胱氨酸突变体以用 BODIPY-FL 标记,进行了 smFA 实验,并使用了 daPDA 和 trBVAa。这种方法使我们能够将动力学和异质性与 FoxP1 二聚化和 DNA 结合联系起来,突出了表征该转录因子的复杂作用机制。
研究方案
注:选择合适的荧光基团对于 smFA 实验至关重要。根据可用的反应性基团,可以通过用荧光标记物修饰蛋白质中的氨基酸或核酸中的核苷酸碱基,在位点特异性位置标记生物分子。在有机染料52 中,Alexa Fluor、Cy、BODIPY 和 Janelia Farms 家族是 smFA 最受欢迎的选择,这要归功于它们的长荧光寿命、光稳定性和高量子产率。BODIPY-FL 因其延长的荧光寿命、卓越的量子产率和短的连接接头而受到青睐。此外,替代荧光团通常用于药物筛选,其中首选散装技术53。嵌合荧光蛋白也可用于活细胞各向异性实验和成像,尽管存在较低动态范围的限制。
1. 缓冲液制备
注意:进行实验室实验时,请戴上手套、护目镜和实验室外套。
- 标准缓冲液(20 mM 4-(2-羟乙基)-1-哌嗪乙磺酸 [HEPES],pH 7.8,150 mM NaCl):将 2.38 g HEPES、4.38 g NaCl 溶于 400 mL 超纯水中,调节 pH 至 7.8,使最终体积达到 500 mL。
- 裂解缓冲液(20 mM HEPES,pH 7.8、150 mM NaCl、0.1 mM 苯甲基磺酰氟 [PMSF]、10 μg/mL DNAse):将 2.38 g HEPES、4.38 g NaCl 和 8.71 mg PMSF 和终浓度为 10 μg/mL DNAse 溶于 400 mL 超纯水中,调节 pH 至 7.8,使最终体积为 500 mL。
- 平衡缓冲液(50 mM 磷酸盐缓冲盐水 [PBS]、150 mM NaCl 和 10 mM 咪唑;pH 7.4):将 5.05 g Na2HPO4、0.85 g NaH2PO4、4.38 g NaCl 和 0.34 g 咪唑溶于 400 mL 超纯水中,调节 pH 至 7.4,使最终体积为 500 mL。
- 洗涤缓冲液(50 mM PBS、150 mM NaCl 和 30 mM 咪唑;pH 7.4):将 5.05 g Na2HPO4、0.85 g NaH2PO4、4.38 g NaCl 和 1.02 g 咪唑溶于 400 mL 超纯水中,调节 pH 至 7.4,使最终体积为 500 mL。
- 洗脱缓冲液(50 mM PBS、150 mM NaCl 和 250 mM 咪唑;pH 7.4):将 5.05 g Na2HPO4、0.85 g NaH2PO4、4.38 g NaCl 和 8.5 g 咪唑溶于 400 mL 超纯水中,调节 pH 至 7.4,使最终体积为 500 mL。
- PBS 缓冲液(50 mM 磷酸钠缓冲盐水,150 mM NaCl,pH 7.4)。将 5.05 g Na2HPO4、0.85 g NaH2PO4 和 4.38 g NaCl 溶于 400 mL 超纯水中,将 pH 调节至 7.4,使最终体积达到 500 mL。
- 通过使用 0.22 μm 孔径过滤器过滤溶液来消毒所有缓冲液,并将缓冲液储存在室温 (RT) 下。
- 使用木炭过滤器过滤标准缓冲液以进行单分子采集。
2. 荧光探针
- BODIPY-FL:通过添加最终体积为 1.29 mL 的新鲜开封的二甲基亚砜 (DMSO) 来溶解 5 mg 含有 BODIPY-FL 的小瓶,以获得 10 mM BODIPY-FL 的最终浓度。
- 罗丹明 110:通过添加最终体积的 1 mL 新鲜打开的 DMSO 来溶解 3.67 mg 罗丹明 110,得到最终浓度为 10 mM 罗丹明 110。
注:当暴露在光线下时,需要避免使用荧光探针。始终使用感光管(琥珀色)并用铝箔包裹。如果 DMSO 已经打开,则必须将其保存在干燥器中以延长其保质期。 - 准备制备的荧光探针的小体积等分试样,并将其储存在 -20 °C 直至进一步使用。
注意:避免冻融循环以提高其标记效率。
3. 校准测量
- 在具有 4 个检测通道和 2 个脉冲激光器(蓝色 485 nm 和红色 640 nm)的自制偏振分辨装置上进行多参数荧光检测 (MFD) 实验54。但是,对于各向异性测量,请使用蓝色激光和两个检测器通道。
- 打开检测器通道(平行和垂直)和蓝色激光功率。
- 确保激光功率设置为 60 μW 并打开时间相关单光子计数 (TCSPC) 软件36 控制面板。
- 将 1 μL 的 100 nM 罗丹明 110 和 49 μL 蒸馏水混合,使最终浓度为 2 nM 的罗丹明 110。将 50 μL 的 2 nM 罗丹明 110 添加到盖玻片的中心以进行校准测量。
- 接下来,在显微镜物镜顶部添加一滴浸液(油或水,但要注意共享相同的折射率),以提高显微镜的分辨率。
- 将盖玻片放在物镜顶部,确保水滴位于显微镜物镜的中心。
- 将图像平面调整为位于溶液内部和玻璃表面上方。
- 调整旋钮以找到第二个明亮的焦点并转到一个半点。将激光聚焦在玻璃和液体的界面上。
- 通过调整针孔位置 (70 μm) 来最大化检测到的光子数量,同时监测光子计数率。
- 在时间标记的时间分辨(TTTR 或 T3)模式面板中打开软件,然后单击 Start 开始按钮。记录计数率 120 秒,并使用采集软件保存 Rhodamine 110.ptu 文件格式。考虑到浓度 (2 nM),这个采集时间应该足够了。
- 对于背景测量,在盖玻片中心加入 50 μL 蒸馏水,然后重复步骤 3.5-3.10。但是,记录光子计数率 300 秒并保存 water.ptu 文件。
- 对于其他背景测量,在盖玻片中心添加 50 μL 标准缓冲液,然后重复步骤 3.5-3.10。记录 300 秒的光子计数率并保存 标准 buffer.ptu 文件。然后,使用突发积分荧光寿命 (BIFL) 分析软件分析数据。
4. 校准和数据分析
- 打开 BIFL 软件,然后单击自动窗口中 的确认设置 。接下来,单击 Get parameters from file(从文件中获取参数),然后单击 OK(确定)。
- 对于要分析的测量,请单击 Data path array 并选择它。
- 接下来,加载 Green scatter 等数组的水测量 water.ptu 文件。同样,为 Green background (绿色背景) 选择标准 buffer.ptu 文件。对于 Green thick(绿色浓厚),选择 Rhodamine 110.ptu。
- 在单个分子选择参数下,单击 Next(下一步),然后单击 Adjust(调整)以显示一个新的弹出窗口。单击阈值以更改光子间到达时间,并为平均光子间到达时间 (dt) 选择单分子事件时间。接下来,单击 min. # 选择每个单分子事件的最小光子数,然后单击 Return 关闭弹出窗口。然后,单击 OK。
- 接下来,单击 Color fit parameters(颜色拟合参数)以调整绿色的初始荧光寿命,绿色颜色是使用荧光衰减参数生成的。通过修改 From 和 To 值来调整 Prompt 和 Delay 值。然后单击 Return 关闭弹出窗口。然后,单击 OK。
- 单击 Save 以处理 ASCII 文件并将其保存在选定的文件夹中。然后,使用 ChiSurf22、光子分布分析55,56 或突发方差分析57,58 处理时间分辨荧光各向异性衰减数据。
注意:有关如何使用 ChiSurf、PDA 和 BVA 的示例数据和分步说明分别在 github.com/Fluorescence-Tools/chisurf、github.com/Fluorescence-Tools/tttrlib、www.mpc.hhu.de/en/software/mfd-fcs-and-mfis 和 github.com/SMB-Lab/feda_tools 上提供。实验数据可在 Zenodo (10.5281/zenodo.13371503) 获得。
5. FoxP1 蛋白制备
- 重组 FoxP1 细菌过表达
- 在快速变化定点聚合酶链反应 (PCR) 诱变后,转化到 大肠 杆菌 C41 细菌细胞中。
- 准备 LB 培养基并高压灭菌以对其进行消毒。
- 通过加入 5 mL 含有 5 μL (100 μg/mL) 氨苄青霉素的 LB 培养基中,预接种单个大 肠杆菌 C41 菌落。在 37 °C 下在培养箱振荡器上孵育过夜。
- 第二天,通过将过夜预接种物以 1:500 的比例加入 500 mL 含有预添加抗生素的 LB 培养基中,接种大规模细菌培养物。
- 通过测量培养物在 600 nm 处的吸光度来监测培养物的生长。
- 当光密度达到~0.5-0.7之间时,通过加入终浓度为0.5mM异丙基-β-d-1-硫代吡喃半乳糖苷(IPTG)来诱导蛋白质表达,并将培养物在15°C下在培养箱振荡器上保持过夜。
- 在达到 1.4-1.6 范围内的光密度 (600 nm) 后,通过在 4 °C 下以 3000 g 离心 20 分钟来收获细菌细胞。 弃去上清液,将沉淀储存在 -20 °C 直至使用。
- 重组 FoxP1 纯化
- 使用任何裂解方法(如超声处理、液体匀浆、法式压榨等)添加裂解缓冲液裂解大肠 杆菌 C41 细胞。
- 将裂解物在 4 °C 下以 14000 g 离心 10 分钟。
- 对于 His6 标签蛋白,洗涤 Ni2+-NTA 亲和柱,并使用快速蛋白质液相色谱 (FPLC) 与 NiSO4 平衡。
- 将 His6 标记的 FoxP1 蛋白加载到平衡的 Ni2+-NTA 亲和柱中。
- 使用洗脱缓冲液的线性梯度从 Ni2+-NTA 柱中洗脱 FoxP1 蛋白。
- 蛋白质洗脱后,进行透析以进行缓冲液置换。将蛋白质加入不含咪唑的平衡缓冲液中,并使用磁力搅拌器在 4 °C 下透析过夜。
注:如果蛋白质使用其他方法纯化,请相应地进行缓冲液更换。 - 要去除 His6 标签,将烟草蚀刻病毒 (TEV) 蛋白酶(TEV:FoxP1 的比例为 1:100)加入透析过夜。
注:His6 标签消化也可以通过添加柱上 TEV 蛋白酶消化来进行。此步骤将提高目标蛋白质的产量。 - 第二天,重复 5.2.3-5.2.5 中的步骤。但是,这一次,在洗涤缓冲液而不是洗脱缓冲液中洗脱蛋白质。
- 通过添加 PBS 缓冲液将蛋白质浓缩至适当的体积,并考虑蛋白质的消光系数,使用 280 nm 处的吸光度定量蛋白质。
- 重组 FoxP1 标记
- 将 50-100 μM FoxP1 蛋白与 10 倍摩尔过量的二硫苏糖醇 (DTT) 或三(2-羧乙基)膦 (TCEP) 在 RT 下孵育 30 分钟,最终体积为 500 μL。
- 使用 PD 10 脱盐柱进行缓冲液置换。使用色谱柱适配器将 PD10 色谱柱放入 50 mL 离心管中。
- 通过添加 5 mL PBS 缓冲液平衡 PD10 色谱柱,并以 1000 × g 离心 2 分钟。重复此步骤 3 次。
- 将 PD10 色谱柱保存在新的 50 mL 试管中,并向色谱柱中加入 2 mL PBS 缓冲液。然后,将步骤 1 中的 500 μL FoxP1 蛋白添加到色谱柱中。通过离心 1000 × g 2 分钟洗脱蛋白质并收集蛋白质。
- 通过将蛋白质加入超离心过滤器 (10 kDa MWCO) 中浓缩蛋白质,以 7500 × g 离心 10 分钟,然后收集洗脱液。
- 测量蛋白质浓度,在蛋白质中加入相当于 30% 半胱氨酸浓度的 BODIPY,并将其在 4 °C 下放在旋转器上 2 小时。
- 如步骤 5.3.2-5.3.5 中所述进行缓冲液交换并浓缩蛋白质。
- 使用分光光度计或比色测定法(如 Bradford 或 Lowry 方法)检查蛋白质浓度,并通过 500 nm 处的吸光度检查染料浓度。
- 使用以下公式测量标记程度:
(A染料/ε染料)/(蛋白质分子量/每毫升蛋白质毫克数)= 染料摩尔数/蛋白质摩尔数
其中 A染料 和 ε染料 是染料在最大吸光度波长处的吸光度值和染料的摩尔消光系数,分别为59。在 280 nm 处测量时,蛋白质的实际吸光度必须通过以下公式校正:
A蛋白 = A280-(A染料·CF280)
其中 CF280 是指定染料的校正因子,考虑到其在 280 nm 处的内禀吸光度59。
注:虽然我们专注于半胱氨酸修饰蛋白的表达和纯化,但必须仔细考虑将半胱氨酸残基战略性地引入蛋白质中。
6. 显微镜样品室制备
- 将 495 μL 超纯水和 5 μL(0.01% v/v 终浓度)的 Tween-20(非离子表面活性剂)添加到腔室载玻片的孔中并混合。在 RT 下孵育 30 分钟以上。
- 用超纯水轻轻清洗腔室两次并擦干。
注意:腔室已准备好用于实验。
7. 单分子荧光各向异性实验
- 首先,校准 smFA 仪器。通过测量拉曼散射光或通过测量具有短寿命和适当发射光谱的染料(例如,赤藓红或孔雀石绿)的荧光,另外被饱和碘化钾 KI 溶液淬灭,确定仪器响应函数。
- 为了校正背景荧光,请对缓冲溶液进行测量。通过从快速旋转的染料中获取数据来校准平行和垂直检测器的相对灵敏度(G 因子)。在这里,使用 2 nM 罗丹明 110 校准检测器60。
- 对于单体 FoxP1 的 smFA 实验,将 100 pM BODIPY 标记的 FoxP1 和 100 nM 未标记的 FoxP1 添加到 500 μL 标准缓冲液(20 mM HEPES,20 mM NaCl,pH 7.8)中。
- 对于使用 DNA 和单体 FoxP1 的滴定测定,将 400 nM DNA 添加到 100 pM BODIPY 标记/100 nM 未标记的蛋白质混合物中,并在室温下孵育 10 分钟。
- 对于二聚体 FoxP1 上的 smFA 实验,在标准缓冲液中将 500 nM 未标记的 FoxP1 添加到 100 pM BODIPY 标记的 FoxP1 中。接下来,在 37 °C 下孵育 30 分钟。
- 对于使用 DNA 和二聚体 FoxP1 的滴定测定,将 2000 nM DNA 添加到 100 pM BODIPY 标记/500 nM 未标记的蛋白质混合物中,并在 RT 下孵育 10 分钟。
- 开始测量并在 BIFL 软件中检查 30 秒记录中的突发量是否在 60-90 之间。如果样品浓度更高,请稀释样品,直到达到该值。此步骤可确保单分子条件。
- 在不存在或存在 DNA 的情况下,开始对单体 FoxP1 或二聚体 FoxP1 进行 smFA 每次测量至少 4 小时。
- 分析第 4 节中讨论的 smFA 测量值。
结果
荧光各向异性是由荧光团吸收和发射偶极矩的相对方向引起的。当荧光团暴露于偏振光时,具有与入射光的电场矢量对齐的吸收过渡矩的荧光团被优先激发(光选择)。因此,激发态种群变得部分取向,其中很大一部分激发分子的跃迁矩与偏振激发光的电场矢量对齐61。荧光团由于它们的布朗运动而旋转。因此,发射跃迁矩也会旋转,从而导致对荧光各向异性的时间依赖性。这种效应可用于测量荧光分子的旋转运动、检测结合事件、表征荧光团的环境以及捕获分子动力学。
单分子实验是确定样品异质性的独特优势。利用单分子灵敏度和荧光各向异性,为多参数荧光光谱增加了另一个维度。在典型的单分子共聚焦显微镜(图 2)20,21 中,当使用脉冲激光器时,可以通过基于强度或时间分辨来确定荧光各向异性。
为了考虑共聚焦显微镜62中高数值孔径物镜的去偏振效应,时间分辨各向异性35,63的正确形式由下式给出
 (1)
(1)
其中 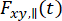 和
和 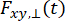 是波长 x 激发后第 y 个检测通道中的时间分辨荧光强度,对于平行和垂直偏振 L1 和 L2,并且是由于这些测量中使用的高数值孔径 (NA) 物镜,是描述平行和垂直信号之间混合的因子35,62,64.染料的平行
是波长 x 激发后第 y 个检测通道中的时间分辨荧光强度,对于平行和垂直偏振 L1 和 L2,并且是由于这些测量中使用的高数值孔径 (NA) 物镜,是描述平行和垂直信号之间混合的因子35,62,64.染料的平行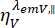 检测通道 和垂直检测通道
检测通道 和垂直检测通道  的检测效率差异用检测效率
的检测效率差异用检测效率 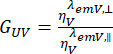 的比率 进行校正。GUV 也称为 G 因子。
的比率 进行校正。GUV 也称为 G 因子。
时间分辨荧光各向异性可以使用多指数衰减进行建模,以解释荧光团附着在更大的生物分子上,如
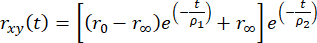 , (2)
, (2)
其中 r0 是荧光团依赖性的基本各向异性(通常为 r0 = 0.38), r∞ 是残余各向异性,ρ1 和 ρ2 分别是快速(荧光团的局部运动)和慢速(大分子的全局运动)旋转相关时间。
在单分子各向异性测量中(图 2),使用突发积分荧光寿命 (BIFL) 分析记录光子到达时间以识别单个发射器33,35。使用运行平均值对光子间到达时间 (Δt) 进行平滑处理,然后绘制以帮助可视化。这些时间的直方图拟合了半高斯,以确定来自背景的光子的平均值和标准差。任意阈值(设置为标准偏差的倍数)用于过滤掉单个事件,同时识别每个爆发中的第一个和最后一个光子。然后对每个爆发中的光子进行积分以进行进一步分析,其中包括使用方程 1 和 2 或通过最大似然估计器35 计算时间分辨和基于强度的稳态荧光各向异性。由于单分子事件中的光子数量有限,最大似然估计器只考虑单个指数分量,不再进一步讨论。
在单分子事件的二维直方图中,平均荧光寿命 (τ) 和各向异性 (rxy) 可以通过 Perrin 方程29,61 相关联,以获得 (ρ) 作为平均旋转时间。
 (3)
(3)
通过“子集成”(se) 分析可以更确定地获得特定的 ρ 值,其中不同爆发的光子被积分到组合的时间分辨荧光各向异性衰减中,该衰减可以通过优化方程 2 的参数来分析实验衰减 (seTRFA)。时间分辨各向异性可以解决在 ns 时间范围内发生的荧光发射内与生物分子的旋转运动(局部和全局)相关的异质性和动力学。
为了检测单分子事件(亚毫秒级)内的动力学,我们引入了时间分辨各向异性突发方差分析 (traBVA)57。在 traBVA 中,对于包含 Mi 连续光子分段的光子暴,暴发的超额各向异性方差 (s2) 为
 . (4)
. (4)
对于单个各向异性状态,方差 σ2 仅由散粒噪声65 (sn: √N,其中 N 是光子数)产生
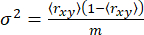 (5)
(5)
其中 m 是突发中的光子数。因此,为了确定各向异性中的其他方差,我们可以将由于构象异质性引起的超额各向异性方差 (S2) 定义为方程 4 和 5 之间的差异。
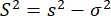 (6)
(6)
为了捕捉在单个分子的观察中发生的动力学并考虑方差近似,可以使用动态各向异性光子分布分析 (daPDA)55,56。在 daPDA 中,荧光强度通过遵循表示为二项式分布的条件概率 ()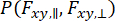 进行建模。
进行建模。
 (7)
(7)
一起,以及遵循 Poisson 分布的背景计数率的估计值
 (8)
(8)
其中  是每个设置时间窗口的平均背景光子数。平行和垂直背景计数
是每个设置时间窗口的平均背景光子数。平行和垂直背景计数  和
和  ,可以使用缓冲样品作为参考进行测量。实验确定的荧光各向异性通过最小化品质因数 χ2 来优化,每个偏振通道的荧光强度分布可以包括动力学变化。
,可以使用缓冲样品作为参考进行测量。实验确定的荧光各向异性通过最小化品质因数 χ2 来优化,每个偏振通道的荧光强度分布可以包括动力学变化。
提供的分析例程和数据表示提供了一种解释所收集数据的全面方法。尽管该协议主要侧重于共聚焦测量,其仅限于捕获从纳秒到毫秒的各向异性变化,但可以采用全内反射显微镜来监测较长时间尺度上的荧光各向异性,从而实现时间序列分析66。对于单分子共聚焦测量,我们重点介绍了多维直方图的使用,这些直方图为观察到的集合创建了独特的指纹。从选定的群体重建的时间分辨荧光衰减可以在纳秒尺度上跟踪荧光各向异性的演变(图 3)。光子分布分析 55,56 和突发方差分析 (BVA)57,58 还可以捕获时间分辨衰减和多维直方图之间的中间时间尺度的动态。虽然该协议不包括偏振荧光相关光谱 (FCS) 的使用,无论是否具有脉冲激发67,68,它可以桥接纳秒到毫秒的时间尺度,但相同的数据可用于计算 FCS69,尽管这超出了所提出的协议的范围。如果进行此类实验,建议更长的样品测量时间。
这种方法已应用于人类 FoxP 蛋白等复杂系统,为了解其作用机制所涉及的运动提供了有价值的见解。FoxP 蛋白是参与大脑和肺发育等多个生理方面的转录因子;重要的是,不同的突变已被公认为会损害这些蛋白的功能70,71。使用 FoxP1 的 DNA 结合结构域作为模型,我们生成了不同的单半胱氨酸突变体,以引入 BODIPY-FL 染料作为运动的跟踪器(图 4A)。事实上,我们评估了二聚化和 DNA 结合作为该蛋白质主要结构调节因子的影响。使用 smFA 方法,我们生成了 2D-smFA 图,并在单体和二聚体条件下为每个突变体制作了 traBVA 和 daPDA。我们展示了一个研究的单个突变体的实例(图 4)。在确定高和低旋转相关时间方面,所有突变体的各向异性行为都是相似的,因此,推测的、无序的和折叠的集合。尽管如此,就每个集合的分数和动力学而言,它在所有突变体中也高度异质,证明了受二聚化和 DNA 结合影响的不同顺序到无序过渡变化,并显示了沿链的结构动力学的高分辨率描述(图 5)。
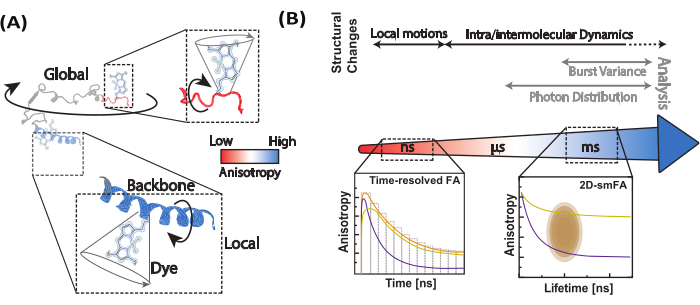
图 1:生物分子的动态范围和荧光各向异性方法。 (A) 沿目标生物分子骨架拴系到不同位置的小荧光团的各向异性为局部结构动力学提供信息。(B) 通过荧光强度衰减(时间分辨荧光各向异性,FA)和共聚焦单分子显微镜数据的单分子直方图探测的时间尺度。 请单击此处查看此图的较大版本。

图 2:单分子荧光各向异性数据配准和处理。 (A) 使用配备单个线性偏振激发激光器(在本例中为蓝色)的共聚焦单分子显微镜分析自由扩散的分子。在光束偏振器将信号分成两个偏振(平行于  激发源和垂直
激发源和垂直  于激发源)后,两个检测器检测到荧光发射(在本例中为绿色)。(B) 每个检测到的光子由三个参数表征:微观时间、宏观时间和通道类型。数据以时间标记时间解析 (TTTR) 格式72.(C) 选择并处理单个分子的爆发以提取荧光参数,包括每个观察到的分子的荧光各向异性。(D) 数据以多种方式表示,包括荧光各向异性与荧光寿命和时间分辨各向异性衰减的二维图。这些表示允许对荧光寿命、旋转相关时间和系统异质性进行视觉和定量测定。 请单击此处查看此图的较大版本。
于激发源)后,两个检测器检测到荧光发射(在本例中为绿色)。(B) 每个检测到的光子由三个参数表征:微观时间、宏观时间和通道类型。数据以时间标记时间解析 (TTTR) 格式72.(C) 选择并处理单个分子的爆发以提取荧光参数,包括每个观察到的分子的荧光各向异性。(D) 数据以多种方式表示,包括荧光各向异性与荧光寿命和时间分辨各向异性衰减的二维图。这些表示允许对荧光寿命、旋转相关时间和系统异质性进行视觉和定量测定。 请单击此处查看此图的较大版本。
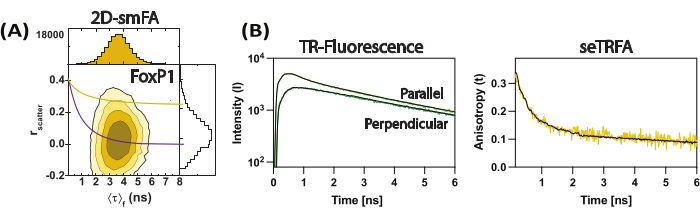
图 3:FoxP1 结构域交换二聚体的代表性数据。 (A) 荧光各向异性 (r散射) 与每个分子的平均荧光寿命的相关性(以等值线图表示)。两个旋转分量的单个 Perrin 方程的叠加,作为分子的集合平均值的代表,分别考虑 ρ1 和 ρ2 为 0.2 ns 和 8.5 ns。(B) 子集成时间分辨荧光衰减用于计算样品的时间分辨荧光各向异性。使用公式 2 拟合可解析荧光各向异性的局部和全局分量。 请单击此处查看此图的较大版本。

图 4:使用单分子荧光各向异性 (smFA) 监测的亚毫秒级 FoxP1 动力学。 (A) 单体 FoxP1 结构的卡通表示。(B) 二维直方图说明了动态异质性,揭示了通过时间分辨荧光各向异性识别的两个不同的旋转相关时间。时间分辨各向异性突发方差分析 (traBVA) 揭示了一小部分具有超额方差(方程 6)的事件,这些事件表现出较大的各向异性。使用光子分布分析 (PDA) 的定量动态各向异性分析可进一步提取此过程的汇率。 请单击此处查看此图的较大版本。

图 5:筛选 FoxP1 在二聚化过程中的局部和全局运动。 (A) 卡通表示将单体 FoxP1 与其二聚体形式进行了比较。(B) 显示了在单体和二聚体条件下每个位置的平均超额方差,较大的超额方差表示各向异性的变化越显著。(C) 使用光子分布分析 (PDA) 的动态各向异性分析有助于确定 DNA 不存在(绿色)和存在(黄色)的群体分数(深色时高各向异性,浅色时低各向异性)。在这种方法中,估计了局部和全局行为之间转换的速率(未显示),揭示了 FoxP1 经历了部分展开。 请单击此处查看此图的较大版本。
讨论
对于单分子荧光各向异性实验,仔细考虑所选荧光团的光物理特性至关重要。这些特性包括发射波长(必须与检测系统一致)和激发波长(应与可用的脉冲激光器兼容)。为了优化动态范围,荧光基团应具有相对于分子旋转扩散时间的较长荧光寿命。这对于跟踪旋转动力学和荧光团偶极子相对于目标生物分子的键合/方向至关重要。此外,亮度、光稳定性和量子产率对于产生具有稳定信噪比的强信号至关重要。由于这些原因,BODIPY-FL 在几项研究中被选为荧光团 39,40,42。
筛选生物分子的骨架动力学通常需要蛋白质标记,通常通过位点特异性标记来实现。这通常是通过引入残留物进行靶向化学修饰来实现的。最常见的方法是在感兴趣的位置引入半胱氨酸,它们的巯基侧链可以用马来酰亚胺或碘乙酰胺等试剂选择性修饰。较少见的是,卤化苄和溴甲基酮用于形成硫醚键。也可以靶向其他氨基酸侧链,但它们在蛋白质中的丰度不太常用。然而,也可以使用其他方法,如非天然氨基酸73。正确选择标记位点对于最大限度地减少对所研究生物分子的干扰至关重要,并且必须采取适当的控制措施。例如,如果标记分子用于结合测定,则互补的无标记方法应验证荧光基团不会影响结合亲和力。
在确定合适的样品并实施最佳标记策略后,下一步是确保共聚焦显微镜正确对准和校准,以进行单分子实验。该协议描述了如何确定进一步分析所需的因子。校准仪器后,下一步是测量样品并处理数据,以从检测到的光子中提取尽可能多的信息。 如图 2 所示,关键参数(如微时间、宏观时间和通道类型)可用于使用典型的 TCSPC 电子设备进行进一步分析和可视化。
单分子荧光光谱的最新进展可广泛用于研究来自生物分子异质系综的结构信息。然而,相对较少的研究利用荧光各向异性提供的见解,需要一个完整的蛋白质模型来推导出生物分子的结构动力学。因此,揭示几种转录因子的结构域间和蛋白质-蛋白质相互作用的动力学具有挑战性。
总之,单分子荧光各向异性实验提供了有关生物分子骨架局部和全局运动的补充信息,这对于理解其功能至关重要。
披露声明
所有作者都声明,他们对本文的内容没有竞争性的经济利益。
致谢
这项工作得到了 FONDECYT 赠款 11200729 和 FONDEQUIP EQM200202 对 E.M.、NIH R15CA280699 R01GM151334 和 NSF CAREER MCB 1749778 对 HS 的奖励的支持。NK 感谢克莱姆森大学博士后奖学金计划的支持。
材料
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
参考文献
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。