Method Article
Anisotropie de fluorescence résolue dans le temps à partir de molécules uniques pour caractériser la flexibilité locale dans les biomolécules
Dans cet article
Résumé
Ici, nous présentons le protocole pour étudier la flexibilité locale et la dynamique des biomolécules en utilisant l’anisotropie de fluorescence résolue en temps au niveau de la molécule unique en mode microscopie confocale.
Résumé
Nous décrivons un protocole pour effectuer une anisotropie de fluorescence résolue en temps au niveau de la molécule unique à l’aide de la microscopie confocale afin d’étudier la flexibilité locale et la dynamique du domaine de la tête de fourche de liaison à l’acide désoxyribonucléique (ADN) du facteur de transcription FoxP1. FoxP1 se dimérise par un mécanisme d’échange de domaine tridimensionnel (3D-DS), formant un intermédiaire désordonné avec ou sans ADN. Étant donné que la 3D-SD implique une région intrinsèquement désordonnée, la compréhension de son comportement est cruciale pour élucider les propriétés structurelles et fonctionnelles de FoxP1. À l’aide d’un FoxP1 marqué à la cystéine unique, nous avons mené des expériences d’anisotropie de fluorescence à molécule unique (smFA), en appliquant des approches d’analyse dynamique de distribution de photons d’anisotropie (daPDA) et d’analyse de variance en rafale d’anisotropie résolue en temps (traBVA) pour sonder la flexibilité et la dynamique locales. Ce protocole fournit un guide détaillé, étape par étape, pour les mesures d’AFSm, en mettant l’accent sur les analyses résolues en temps, la variance et les techniques de distribution de probabilité pour capturer la dynamique structurelle à différentes échelles de temps. Cette approche nous a permis de relier la dynamique et l’hétérogénéité à la dimérisation de FoxP1 et à la liaison à l’ADN, mettant en évidence le mécanisme d’action complexe qui caractérise ce facteur de transcription.
Introduction
L’activité fonctionnelle des biomolécules dépend de leur flexibilité moléculaire et de leur dynamique structurale 1,2,3. Naturellement, les biomolécules subissent des fluctuations thermiques constantes, allant de mouvements rapides à des changements conformationnels à long terme influençant leur fonction (Figure 1)4. Dans les biomolécules, les mouvements locaux du squelette contribuent à des mouvements globaux à plus grande échelle, y compris la flexion des charnières dans les enzymes et des changements conformationnels significatifs dans les protéines motrices. Les méthodes de détermination de structure telles que la résonance magnétique nucléaire (RMN)5, la cristallographie aux rayons X6 et la microscopie électronique cryogénique (cryo-EM)7 ont révélé de multiples conformations dans diverses biomolécules. Néanmoins, le lien entre les fluctuations locales et la grande dynamique conformationnelle des biomolécules et leur rôle dans la fonction sont pour la plupart inexplorés. Il peut être difficile de relier la dynamique et la structure, en particulier pour les protéines intrinsèquement désordonnées (IDP)8,9,10. Contrairement aux protéines structurées, les IDP ne maintiennent pas une structure tertiaire stable. Au lieu de cela, ils subissent d’importants changements de conformation avec des niveaux d’énergie libre similaires, permettant un large éventail d’activités biologiques11,12.
Plusieurs approches expérimentales ont été employées pour étudier la dynamique conformationnelle des protéines en sondant leur flexibilité moléculaire 1,13,14,15,16. Parmi celles-ci, la RMN se distingue par sa capacité à fournir une résolution de niveau atomique sur différentes échelles de temps, de quelques dizaines de picosecondes à plusieurs heures12. Cependant, la détermination de la flexibilité macromoléculaire reste difficile en raison des degrés de liberté élevés et pour les protéines de grande taille ; ainsi, la RMN se limite souvent à l’étude de biomolécules d’environ 100 kDa17.
Compte tenu de la complexité structurelle des protéines hautement dynamiques comme les IDP, des avancées méthodologiques supplémentaires ont été développées pour explorer l’espace conformationnel local et à longue portée afin de comprendre leur fonction11. La spectroscopie de fluorescence multiparamètre à molécule unique (smMFS)18,19,20,21,22 offre des informations détaillées sur les biomolécules, fournissant des informations cruciales sur leur fonction, la dynamique conformationnelle, les états de liaison et la stœchiométrie. Cependant, l’interprétation de la grande quantité de données structurelles obtenues à partir de biomolécules est un défi, et des facteurs tels que la dynamique moléculaire, le comportement des fluorophores et le comportement complexe des molécules peuvent compliquer davantage l’analyse des données 23,24,25,26,27,28.
Nous utilisons l’anisotropie de fluorescence à molécule unique (smFA) comme méthode robuste pour évaluer la dynamique locale et globale le long de l’épine dorsale des biomolécules (Figure 1A). L’anisotropie de fluorescence, décrite pour la première fois par Perrin29 et introduite par Weber30,31 en tant qu’outil bioanalytique32, a ensuite été adaptée pour des études de molécules uniques avec l’avènement des techniques de fluorescence résolues en temps et l’augmentation de la sensibilité des détecteurs 33,34,35,36,37. smFA couvre une large gamme d’échelles de temps - de la picoseconde à plusieurs heures - et complète les données obtenues à partir d’expériences smFRET (Förster Resonance Energy Transfer) à molécule unique38.
L’AGm peut être visualisé dans divers formats pour extraire des informations critiques sur la dynamique biomoléculaire (Figure 1B). Les décroissances de l’anisotropie de fluorescence résolues dans le temps sont des histogrammes unidimensionnels qui capturent la dynamique sur l’échelle de temps de la picoseconde à la nanoseconde39,40. Les histogrammes bidimensionnels à molécule unique, qui corrèlent la durée de vie de la fluorescence avec l’anisotropie pour des molécules individuelles, peuvent révéler l’hétérogénéité de l’état d’anisotropie et fournir des informations visuelles sur la dynamique potentielle dans le temps d’observation dans les expériences confocales (~ms)41,42. Pour étudier la dynamique inférieure à la milliseconde, l’analyse dynamique de la distribution des photons de l’anisotropie (daPDA) peut être utilisée, tandis que l’analyse de variance de l’anisotropie résolue en temps (traBVA) offre une méthode robuste pour confirmer une dynamique spécifique autour de quelques millisecondes43 (Figure 1B).
Ces méthodes complètent des outils plus traditionnels, tels que la spectroscopie de corrélation de fluorescence résolue par polarisation (pFCS), qui a un spectre plus large 44,45,46,47. Dans l’ensemble, plusieurs outils d’analyse de données pour les AGm facilitent l’identification des changements conformationnels locaux et globaux, à condition qu’un étalonnage approprié soit pris en compte.
Ici, nous appliquons la smFA pour étudier la liaison à l’ADN du facteur de transcription humainFoxP1 48,49,50,51. Cette protéine adopte des dimères échangés en raison de la nature intrinsèquement désordonnée de sa chaîne polypeptidique, qui est notablement affectée en fonction de l’état quaternaire de la protéine et de la présence d’ADN. Nous avons généré différents mutants mono-cystéine à marquer avec BODIPY-FL, effectué des expériences smFA et utilisé daPDA et trBVAa. Cette approche nous a permis de relier la dynamique et l’hétérogénéité à la dimérisation de FoxP1 et à la liaison à l’ADN, mettant en évidence le mécanisme d’action complexe qui caractérise ce facteur de transcription.
Protocole
REMARQUE : Le choix du fluorophore approprié est essentiel pour les expériences d’AFm. Les biomolécules peuvent être marquées à des positions spécifiques au site, soit en modifiant les acides aminés dans les protéines, soit les bases nucléotidiques dans les acides nucléiques avec des marqueurs fluorescents, en fonction des groupes réactifs disponibles. Parmi les colorants organiques52, les familles Alexa Fluor, Cy, BODIPY et Janelia Farms sont les choix les plus populaires pour les smFA, grâce à leur longue durée de vie en fluorescence, leur photostabilité et leurs rendements quantiques élevés. BODIPY-FL est souvent privilégié pour sa durée de vie prolongée en fluorescence, son rendement quantique supérieur et son linker de connexion court. De plus, les fluorophores alternatifs sont couramment utilisés dans le criblage de médicaments où les techniques en vrac sont préférées53. Les protéines fluorescentes chimériques peuvent également être utilisées pour des expériences d’anisotropie et d’imagerie sur des cellules vivantes, bien qu’il y ait une limitation d’une plage dynamique plus faible.
1. Préparation du tampon
REMARQUE : Portez des gants, des lunettes de protection et une blouse de laboratoire lorsque vous effectuez des expériences de laboratoire.
- Tampon étalon (20 mM d’acide 4-(2-hydroxyéthyl)-1-pipérazineéthananesulfonique [HEPES], pH 7,8, 150 mM de NaCl) : Dissoudre 2,38 g de HEPES, 4,38 g de NaCl dans 400 mL d’eau ultra-pure, ajuster le pH à 7,8 et porter le volume final à 500 mL.
- Tampon de lyse (20 mM HEPES, pH 7,8, 150 mM NaCl, 0,1 mM de fluorure de phénylméthylsulfonyle [PMSF], 10 μg/mL d’ADNASE) : Dissoudre 2,38 g de HEPES, 4,38 g de NaCl et 8,71 mg de PMSF et une concentration finale de 10 μg/mL d’ADNASE dans 400 mL d’eau ultrapure, ajuster le pH à 7,8 et porter le volume final à 500 mL.
- Tampon d’équilibrage (50 mM de solution saline tamponnée au phosphate [PBS], 150 mM de NaCl et 10 mM d’imidazole ; pH 7,4) : Dissoudre 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl et 0,34 g d’imidazole dans 400 mL d’eau ultra-pure, ajuster le pH à 7,4 et porter le volume final à 500 mL.
- Tampon de lavage (50 mM de PBS, 150 mM de NaCl et 30 mM d’imidazole ; pH 7,4) : Dissoudre 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl et 1,02 g d’imidazole dans 400 mL d’eau ultra-pure, ajuster le pH à 7,4 et porter le volume final à 500 mL.
- Tampon d’élution (50 mM de PBS, 150 mM de NaCl et 250 mM d’imidazole ; pH de 7,4) : Dissoudre 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl et 8,5 g d’imidazole dans 400 ml d’eau ultra-pure, ajuster le pH à 7,4 et porter le volume final à 500 ml.
- Tampon PBS (50 mM de solution saline tamponnée au phosphate de sodium, 150 mM de NaCl, pH 7,4). Dissoudre 5,05 g de Na2HPO4, 0,85 g de NaH2PO4 et 4,38 g de NaCl dans 400 ml d’eau ultra-pure, ajuster le pH à 7,4 et porter le volume final à 500 ml.
- Stérilisez tous les tampons en filtrant la solution à l’aide de filtres à pores de 0,22 μm et conservez les tampons à température ambiante (RT).
- Filtrez le tampon standard à l’aide d’un filtre à charbon pour l’acquisition d’une seule molécule.
2. Sondes fluorescentes
- BODIPY-FL : Dissoudre un flacon de 5 mg contenant BODIPY-FL en ajoutant un volume final de 1,29 mL de diméthylsulfoxyde (DMSO) fraîchement ouvert pour obtenir la concentration finale de 10 mM DE BODIPY-FL.
- Rhodamine 110 : Dissoudre 3,67 mg de Rhodamine 110 en ajoutant un volume final de 1 mL de DMSO fraîchement ouvert pour obtenir la concentration finale de 10 mM de Rhodamine 110.
REMARQUE : Les sondes fluorescentes doivent être évitées lorsqu’elles sont exposées à la lumière. Utilisez toujours un tube sensible à la lumière (de couleur ambrée) et enveloppez-le de papier d’aluminium. Si le DMSO est déjà ouvert, il doit être conservé dans le dessiccateur pour augmenter sa durée de conservation. - Préparez des aliquotes de petit volume de la sonde fluorescente préparée et conservez-la à -20 °C jusqu’à ce qu’elle soit utilisée ultérieurement.
REMARQUE : Évitez les cycles de gel-dégel pour améliorer l’efficacité de son étiquetage.
3. Mesures d’étalonnage
- Réalisez des expériences de détection de fluorescence multiparamètre (MFD) sur une configuration de polarisation maison résolue avec 4 canaux de détection et 2 lasers pulsés (bleu 485 nm et rouge 640 nm)54. Cependant, pour les mesures d’anisotropie, utilisez le laser bleu et deux canaux de détection.
- Allumez les canaux du détecteur (parallèle et perpendiculaire) et l’alimentation du laser bleu.
- Assurez-vous que la puissance du laser est réglée sur 60 μW et ouvrez le panneau de commande du logiciel TCSPC (Time-Correlated Single Photon Counting Counting (TCSPC)36 .
- Mélangez 1 μL de 100 nM de Rhodamine 110 et 49 μL d’eau distillée pour obtenir une concentration finale de 2 nM de Rhodamine 110. Ajoutez 50 μL de rhodamine 110 2 nM au centre du verre de protection pour les mesures d’étalonnage.
- Ensuite, ajoutez une goutte de liquide d’immersion (soit de l’huile, soit de l’eau, mais en prenant soin de partager le même indice de réfraction) sur la lentille de l’objectif du microscope pour augmenter le pouvoir de résolution du microscope.
- Placez le verre de protection sur la lentille de l’objectif et assurez-vous que la gouttelette d’eau est au centre de l’objectif du microscope.
- Ajustez le plan de l’image pour qu’il soit à l’intérieur de la solution et au-dessus de la surface du verre.
- Ajustez le bouton pour trouver le deuxième point focal lumineux et tournez-le sur un point et demi. Focalisez le laser à l’interface du verre et du liquide.
- Maximisez le nombre de photons détectés en ajustant la position du sténopé (70 μm) tout en surveillant le taux de comptage des photons.
- Ouvrez le logiciel dans le panneau du mode TTTR ou T3 et cliquez sur le bouton Démarrer . Enregistrez le taux de comptage pendant 120 s et sauvegardez le format de fichier Rhodamine 110.ptu à l’aide du logiciel d’acquisition. Ce temps d’acquisition devrait être suffisant compte tenu de la concentration (2 nM).
- Pour les mesures de fond, ajoutez 50 μL d’eau distillée au centre du verre de couverture et répétez les étapes 3.5-3.10. Cependant, enregistrez le taux de comptage des photons pendant 300 s et enregistrez le fichier water.ptu .
- Pour d’autres mesures de fond, ajoutez 50 μL de tampon standard au centre de la vitre de protection et répétez les étapes 3.5 à 3.10. Enregistrez le taux de comptage des photons pendant 300 s et enregistrez le fichier buffer.ptu standard . Ensuite, analysez les données à l’aide d’un logiciel d’analyse de la durée de vie de la fluorescence d’intégration en rafale (BIFL).
4. Calibrage et analyse des données
- Ouvrez le logiciel BIFL et cliquez sur confirmer la configuration dans la fenêtre automatique. Ensuite, cliquez sur obtenir les paramètres du fichier, puis sur OK.
- Pour la mesure à analyser, cliquez sur le tableau de chemins d’accès aux données et sélectionnez-le .
- Ensuite, chargez le fichier water.ptu de mesure de l’eau pour les tableaux tels que Green scatter. De même, sélectionnez le fichier buffer.ptu standard pour Fond vert. Pour Green thick, sélectionnez Rhodamine 110.ptu.
- Sous les paramètres de sélection d’une molécule unique, cliquez sur Suivant, puis sur Ajuster pour afficher une nouvelle fenêtre contextuelle. Cliquez sur le seuil pour modifier l’heure d’arrivée des interphotons et sélectionnez l’heure de l’événement de la molécule unique pour le temps d’arrivée moyen des interphotons (dt). Ensuite, cliquez sur min. # pour sélectionner le nombre minimum de photons par événement moléculaire, puis fermez la fenêtre contextuelle en cliquant sur Retour. Cliquez ensuite sur OK.
- Ensuite, cliquez sur Paramètres d’ajustement des couleurs pour ajuster la durée de vie initiale de la fluorescence du vert, couleurs générées avec les paramètres de décroissance de la fluorescence. Ajustez les valeurs Invite et Délai en modifiant les valeurs De et À. Fermez ensuite la fenêtre contextuelle en cliquant sur Retour. Cliquez ensuite sur OK.
- Cliquez sur Enregistrer pour traiter les fichiers ASCII et les enregistrer dans un dossier sélectionné. Ensuite, traitez les données pour la décroissance de l’anisotropie de fluorescence résolue en temps à l’aide de ChiSurf22, de l’analyse de distribution de photons 55,56 ou de l’analyse de variance en rafale57,58.
REMARQUE : Des données exemplaires et des descriptions étape par étape sur l’utilisation de ChiSurf, PDA et BVA sont disponibles sur github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis et github.com/SMB-Lab/feda_tools, respectivement. Les données expérimentales sont disponibles sur Zenodo (10.5281/zenodo.13371503).
5. Préparation de protéines FoxP1
- Surexpression bactérienne recombinante de FoxP1
- Effectuez la transformation en cellules bactériennes E. coli C41 après mutagenèse par réaction en chaîne par polymérase (PCR) à changement rapide.
- Préparez le milieu LB et l’autoclave pour le stériliser.
- Préinoculer une seule colonie d’E. coli C41 en l’ajoutant dans 5 mL de milieu LB contenant 5 μL (100 μg/mL) d’ampicilline. Laisser incuber à 37 °C pendant la nuit sur un agitateur d’incubateur.
- Le lendemain, inoculer une culture bactérienne à grande échelle en ajoutant du pré-inoculum pendant la nuit dans 500 ml de milieu LB avec un antibiotique pré-ajouté dans un rapport de 1:500.
- Surveiller la croissance de la culture en mesurant l’absorbance de la culture à 600 nm.
- Lorsque la densité optique atteint entre ~0,5 et 0,7, induire l’expression de la protéine en ajoutant une concentration finale de 0,5 mM d’isopropyl-β-d-1-thiogalactopyranoside (IPTG) et maintenir la culture à 15 °C sur un agitateur d’incubateur pendant la nuit.
- Après avoir atteint une densité optique (600 nm) dans la gamme 1,4-1,6, récolter les cellules bactériennes par centrifugation à 3000 g pendant 20 min à 4 °C. Jetez le surnageant et conservez la pastille à -20 °C jusqu’à utilisation.
- Purification recombinante FoxP1
- Lyse les cellules E. coli C41 en ajoutant un tampon de lyse à l’aide de toutes les méthodes de lyse telles que la sonication, l’homogénéisation liquide, la presse française, etc.
- Centrifuger le lysat à 14000 g pendant 10 min à 4 °C.
- Pour les protéines marquées à6 sons, laver la colonne d’affinité Ni2+-NTA et équilibrer avec NiSO4 à l’aide de la chromatographie liquide protéique rapide (FPLC).
- Chargez la protéine FoxP1 marquée à6 His dans la colonne d’affinité Ni2+-NTA équilibrée.
- Éluer la protéine FoxP1 de la colonne de Ni2+-NTA en utilisant un gradient linéaire du tampon d’élution.
- Une fois la protéine éluée, effectuez une dialyse pour l’échange de tampon. Ajouter les protéines dans un tampon d’équilibrage sans imidazole et dialyser pendant la nuit à l’aide d’un agitateur magnétique à 4 °C.
REMARQUE : Si la protéine est purifiée à l’aide d’une méthode différente, effectuez l’échange de tampon en conséquence. - Pour retirer la baliseHis 6, ajoutez la protéase du virus de la gravure du tabac (TEV) (rapport 1:100 TEV :FoxP1) à la dialyse pendant la nuit.
REMARQUE : Sa digestion à6 étiquettes peut également être effectuée en ajoutant une digestion par protéase TEV sur colonne. Cette étape permettrait d’améliorer le rendement de la protéine d’intérêt. - Le lendemain, répétez les étapes 5.2.3-5.2.5. Cependant, cette fois, éluez la protéine dans le tampon de lavage plutôt que dans le tampon d’élution.
- Concentrez la protéine à un volume approprié en ajoutant un tampon PBS et quantifiez la protéine en utilisant l’absorbance à 280 nm, en tenant compte du coefficient d’extinction de la protéine.
- Marquage recombinant FoxP1
- Incuber 50 à 100 μM de protéine FoxP1 avec un excès molaire de 10 fois plus de dithiothréitol (DTT) ou de tris (2-carboxyéthyl)phosphine (TCEP) avec un volume final de 500 μL à RT pendant 30 min.
- Effectuez l’échange de tampon à l’aide des colonnes de dessalement 10. Placez la colonne PD10 dans un tube à centrifuger de 50 ml à l’aide d’un adaptateur de colonne.
- Équilibrez la colonne PD10 en ajoutant 5 mL de tampon PBS et centrifugez à 1000 × g pendant 2 min. Répétez cette étape 3 fois.
- Conservez la colonne PD10 dans un tube frais de 50 ml et ajoutez 2 ml de tampon PBS dans la colonne. Ensuite, ajoutez 500 μL de protéine FoxP1 de l’étape 1 dans la colonne. Éluer la protéine en centrifugeant 1000 × g pendant 2 min et recueillir la protéine.
- Concentrez la protéine en l’ajoutant dans des filtres ultra-centrifuges (10 kDa MWCO), centrifugez à 7500 × g pendant 10 min et récupérez l’élu.
- Mesurez la concentration en protéines, ajoutez BODIPY avec 30 % de l’équivalent de la concentration en cystéine dans la protéine, et placez-le sur un rotateur pendant 2 h à 4 °C.
- Effectuez l’échange de tampon et concentrez la protéine comme indiqué aux étapes 5.3.2-5.3.5.
- Vérifiez la concentration en protéines à l’aide d’un spectrophotomètre ou d’un test colorimétrique, tel que la méthode de Bradford ou de Lowry, et la concentration du colorant par absorbance à 500 nm.
- Mesurez le degré d’étiquetage à l’aide de la formule suivante :
(colorant A/colorant ε)/(poids moléculaire de la protéine/milligramme de protéine par millilitre) = moles de colorant/moles de protéine
oùColorant A et εcolorant sont la valeur d’absorbance du colorant et le coefficient d’extinction molaire du colorant à la longueur d’onde d’absorbance maximale, respectivement59. Lorsqu’elle est mesurée à 280 nm, l’absorbance réelle de la protéine doit être corrigée par la formule suivante :
Uneprotéine = A280-(Uncolorant· CF280)
où CF280 est le facteur de correction pour le colorant spécifié, compte tenu de son absorbance intrinsèque à 280 nm59.
REMARQUE : Bien que nous nous concentrions sur l’expression et la purification d’une protéine modifiée à la cystéine, une attention particulière doit être accordée à l’introduction stratégique de résidus de cystéine dans la protéine.
6. Préparation de la chambre d’échantillonnage du microscope
- Ajouter 495 μL d’eau ultra-pure et 5 μL (concentration finale de 0,01 % v/v) de Tween-20 (tensioactif non ionique) dans le puits d’une lame de chambre et mélanger. Incuber sur 30 min à RT.
- Lavez doucement la chambre avec de l’eau ultra-pure deux fois et séchez-la.
REMARQUE : La chambre est prête à être utilisée pour l’expérience.
7. Expérience d’anisotropie de fluorescence à molécule unique
- Tout d’abord, calibrez l’instrument smFA. Déterminer la fonction de réponse de l’instrument, soit en mesurant la lumière diffusée Raman, soit en mesurant la fluorescence d’un colorant ayant une courte durée de vie et un spectre d’émission approprié (par exemple, l’érythrosine ou le vert de malachite) éteint en outre par une solution saturée d’iodure de potassium, KI.
- Pour corriger la fluorescence de fond, effectuez des mesures d’une solution tampon. Calibrez la sensibilité relative des détecteurs parallèles et perpendiculaires (facteur G) en acquérant des données à partir d’un colorant à rotation rapide. Ici, 2 nM de Rhodamine 110 ont été utilisés pour calibrer les détecteurs60.
- Pour les expériences d’AFMm sur FoxP1 monomère, ajoutez 100 pM de FoxP1 marqué BODIPY et 100 nM de FoxP1 non marqué à 500 μL de tampon étalon (20 mM HEPES, 20 mM NaCl, pH 7,8) dans une lame de chambre.
- Pour un dosage par titrage avec de l’ADN et du FoxP1 monomère, ajoutez 400 nM d’ADN à 100 pM de mélange de protéines BODIPY / 100 nM non marquées et incubez à RT pendant 10 min.
- Pour les expériences smFA sur FoxP1 dimérique, ajoutez 500 nM non marqués FoxP1 à 100 pM BODIPY marqués dans le tampon standard. Ensuite, incubez à 37 °C pendant 30 min.
- Pour un dosage avec de l’ADN et du FoxP1 dimérique, ajoutez 2000 nM d’ADN à 100 pM de mélange de protéines BODIPY / 500 nM non marquées et incubez à RT pendant 10 min.
- Lancez les mesures et vérifiez dans le logiciel BIFL que le nombre de rafales dans un enregistrement de 30 s est compris entre 60 et 90. Si l’échantillon est plus concentré, diluez-le jusqu’à ce qu’il atteigne cette valeur. Cette étape garantit des conditions monomoléculaires.
- Commencer l’analyse par smFA à chaque mesure pour le FoxP1 monomère ou le FoxP1 dimérique en l’absence ou en présence d’ADN pendant au moins 4 h.
- Analysez les mesures d’AGSM comme indiqué à la section 4.
Résultats
L’anisotropie de fluorescence résulte de l’orientation relative de l’absorption du fluorophore et des moments dipolaires d’émission. Lorsque les fluorophores sont exposés à la lumière polarisée, les fluorophores dont les moments de transition d’absorption sont alignés sur le vecteur de champ électrique de la lumière incidente sont préférentiellement excités (photosélection). Par conséquent, la population à l’état excité devient partiellement orientée, avec une fraction significative des molécules excitées dont les moments de transition sont alignés avec le vecteur de champ électrique de la lumière excitante polarisée61. Les fluorophores tournent en raison de leur mouvement brownien. Ainsi, le moment de transition d’émission tourne également, ce qui entraîne une dépendance temporelle à l’anisotropie de fluorescence. Cet effet peut être utilisé pour mesurer les mouvements de rotation des molécules fluorescentes, détecter les événements de liaison, caractériser l’environnement du fluorophore et capturer la dynamique moléculaire.
Les expériences sur une seule molécule sont particulièrement bien placées pour déterminer l’hétérogénéité de l’échantillon. Tirer parti de la sensibilité d’une molécule unique et de l’anisotropie de fluorescence ajoute une autre dimensionnalité à la spectroscopie de fluorescence multiparamètre. Dans un microscope confocal à molécule unique typique (Figure 2)20,21, l’anisotropie de fluorescence peut être déterminée en fonction de l’intensité ou résolue dans le temps lorsque des lasers pulsés sont utilisés.
Pour considérer les effets dépolarisants de l’objectif à haute ouverture numérique dans un microscope confocal62, la forme correcte de l’anisotropie résolue en temps35,63 est donnée par
 (1)
(1)
où 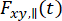 et sont
et sont 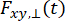 l’intensité de fluorescence résolue en temps dans le y-ième canal de détection après excitation à la longueur d’onde x, pour la polarisation parallèle et perpendiculaire L1 et L2 et sont des facteurs qui décrivent le mélange entre les signaux parallèles et perpendiculaires en raison de l’objectif à grande ouverture numérique (AN) utilisé dans ces mesures35,62, N° 64. Les différences d’efficacité de détection du canal de détection parallèle,
l’intensité de fluorescence résolue en temps dans le y-ième canal de détection après excitation à la longueur d’onde x, pour la polarisation parallèle et perpendiculaire L1 et L2 et sont des facteurs qui décrivent le mélange entre les signaux parallèles et perpendiculaires en raison de l’objectif à grande ouverture numérique (AN) utilisé dans ces mesures35,62, N° 64. Les différences d’efficacité de détection du canal de détection parallèle, 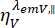 , et perpendiculaire,
, et perpendiculaire,  , pour le colorant sont corrigées par le rapport des efficacités de détection,
, pour le colorant sont corrigées par le rapport des efficacités de détection, 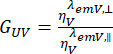 . Le GUV est également appelé facteur G.
. Le GUV est également appelé facteur G.
L’anisotropie de fluorescence résolue en temps peut être modélisée à l’aide d’une décroissance multiexponentielle pour tenir compte du fait que le fluorophore est attaché à une biomolécule plus grande comme
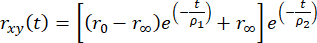 , (2)
, (2)
où r0 est l’anisotropie fondamentale dépendante des fluorophores (typiquement r0 = 0,38), r∞ est l’anisotropie résiduelle, et ρ1 et ρ2 sont respectivement des temps de corrélation de rotation rapides (mouvements locaux du fluorophore) et lents (mouvement global de la macromolécule).
Dans les mesures d’anisotropie d’une seule molécule (Figure 2), les temps d’arrivée des photons sont enregistrés pour identifier les émetteurs individuels à l’aide de l’analyse de la durée de vie de fluorescence intégrée en rafale (BIFL)33,35. Les temps d’arrivée interphotoniques (Δt) sont lissés à l’aide d’une moyenne mobile, puis tracés pour faciliter la visualisation. L’histogramme de ces temps est équipé d’une demi-gaussienne pour déterminer la moyenne et l’écart-type des photons provenant de l’arrière-plan. Un seuil arbitraire, fixé à des multiples de l’écart-type, est utilisé pour filtrer les événements individuels tout en identifiant le premier et le dernier photon de chaque sursaut. Les photons de chaque sursaut sont ensuite intégrés pour une analyse plus approfondie, qui comprend le calcul de l’anisotropie de fluorescence à l’état stationnaire résolue en temps et basée sur l’intensité à l’aide des équations 1 et 2 ou via un estimateur de vraisemblance maximale35. En raison du nombre limité de photons dans les événements à molécule unique, l’estimateur du maximum de vraisemblance ne prend en compte qu’une seule composante exponentielle et ne sera pas discuté plus en détail.
Dans un histogramme bidimensionnel d’événements de molécules uniques, la durée de vie moyenne de fluorescence (τ) et l’anisotropie (rxy) peuvent être liées par l’équation de Perrin29,61 pour obtenir (ρ) comme temps de rotation moyen.
 (3)
(3)
Des valeurs spécifiques de ρ peuvent être obtenues avec une certitude plus élevée par l’analyse des « sous-ensembles » (se) où les photons de différents sursauts sont intégrés dans une décroissance combinée de l’anisotropie de fluorescence résolue en temps qui peut être analysée en optimisant les paramètres de l’équation 2 à la décroissance expérimentale (seTRFA). L’anisotropie résolue dans le temps peut résoudre l’hétérogénéité et la dynamique associées aux mouvements de rotation (locaux et globaux) des biomolécules dans l’émission de fluorescence qui se produit dans le ns laps de temps.
Pour détecter la dynamique au sein d’événements à molécule unique (à l’échelle de la milliseconde), nous avons introduit l’analyse de variance en rafale d’anisotropie résolue en temps (traBVA)57. Dans traBVA, pour un sursaut de photons contenant Mi segments de photons consécutifs, l’excès de variance d’anisotropie (s2) pour les sursauts est de
 . (4)
. (4)
Pour un seul état anisotrope, la variance σ2 provient uniquement du bruit de tir65 (sn : √N, où N est le nombre de photons)
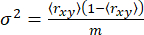 (5)
(5)
où m est le nombre de photons dans une rafale. Par conséquent, pour identifier une variance supplémentaire dans l’anisotropie, nous pouvons définir la variance d’anisotropie excédentaire (S2) due à l’hétérogénéité conformationnelle comme la différence entre les équations 4 et 5.
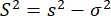 (6)
(6)
Pour capturer la dynamique qui se produit lors de l’observation de molécules individuelles et considérer l’approximation de la variance, l’analyse dynamique de la distribution des photons par anisotropie (daPDA)55,56 peut être utilisée. Dans la daPDA, l’intensité de fluorescence est modélisée en suivant une probabilité conditionnelle (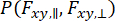 ) exprimée sous forme de distribution binomiale.
) exprimée sous forme de distribution binomiale.
 (7)
(7)
Ensemble, avec une estimation du taux de dénombrement de fond qui suit une distribution de Poisson
 (8)
(8)
où 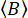 est le nombre moyen de photons d’arrière-plan par fenêtre de temps définie. Les comptages d’arrière-plan parallèles et perpendiculaires,
est le nombre moyen de photons d’arrière-plan par fenêtre de temps définie. Les comptages d’arrière-plan parallèles et perpendiculaires,  et
et 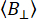 , peuvent être mesurés à l’aide d’échantillons tampons comme référence. L’anisotropie de fluorescence déterminée expérimentalement est optimisée en minimisant un facteur de mérite χ2 avec une distribution de l’intensité de fluorescence par canal de polarisation qui peut inclure des changements cinétiques.
, peuvent être mesurés à l’aide d’échantillons tampons comme référence. L’anisotropie de fluorescence déterminée expérimentalement est optimisée en minimisant un facteur de mérite χ2 avec une distribution de l’intensité de fluorescence par canal de polarisation qui peut inclure des changements cinétiques.
Les routines d’analyse et les représentations de données fournies offrent une approche globale de l’interprétation des données collectées. Bien que ce protocole se concentre principalement sur les mesures confocales, qui sont limitées dans la capture des changements d’anisotropie de la nanoseconde à la milliseconde, il est possible d’adopter un microscope à réflexion interne totale pour surveiller l’anisotropie de fluorescence sur des échelles de temps plus longues, permettant ainsi l’analyse de séries temporelles66. Pour les mesures confocales d’une seule molécule, nous mettons en évidence l’utilisation d’histogrammes multidimensionnels qui créent une empreinte unique de l’ensemble observé. Les désintégrations de fluorescence résolues en temps, reconstruites à partir de populations sélectionnées, peuvent suivre l’évolution de l’anisotropie de fluorescence à l’échelle de la nanoseconde (Figure 3). L’analyse de la distribution des photons55,56 et l’analyse de variance en rafale (BVA)57,58 peuvent également capturer la dynamique à des échelles de temps intermédiaires entre les décroissances résolues en temps et les histogrammes multidimensionnels. Bien que ce protocole ne couvre pas l’utilisation de la spectroscopie de corrélation de fluorescence de polarisation (FCS), avec ou sans excitation pulsée67,68, qui peut faire le pont entre des échelles de temps de la nanoseconde à la milliseconde, les mêmes données peuvent être utilisées pour calculer la FCS69, bien que cela ne relève pas du champ d’application du protocole présenté. Si de telles expériences sont entreprises, il est recommandé d’allonger le temps de mesure de l’échantillon.
Cette approche a été appliquée à un système complexe comme les protéines FoxP humaines, fournissant des informations précieuses sur les mouvements impliqués dans leur mécanisme d’action. Les protéines FoxP sont des facteurs de transcription impliqués dans plusieurs aspects physiologiques tels que le développement du cerveau et des poumons ; Il est important de noter que différentes mutations ont été reconnues comme altérant la fonction de ces protéines70,71. En utilisant le domaine de liaison à l’ADN de FoxP1 comme modèle, nous avons généré différents mutants monocystéine pour introduire un colorant BODIPY-FL comme traqueur des mouvements (Figure 4A). En fait, nous avons évalué l’effet de la dimérisation et de la liaison à l’ADN en tant que régulateurs structurels majeurs de cette protéine. En utilisant l’approche smFA, nous avons généré des graphiques 2D-smFA et réalisé des traBVA et daPDA pour chaque mutant dans des conditions monomères et dimériques. Nous montrons l’exemple d’un des mutants uniques étudiés (Figure 4). Le comportement de l’anisotropie est similaire chez tous les mutants en termes de détermination des temps de corrélation de rotation élevés et faibles et, par conséquent, des ensembles présumés, désordonnés et pliés. Pourtant, il est également très hétérogène chez tous les mutants en termes de fraction et de cinétique de chaque ensemble, mettant en évidence différents changements de transition de l’ordre au désordre influencés par la dimérisation et la liaison à l’ADN, et montre la description à haute résolution de la dynamique structurelle le long de la chaîne (Figure 5).
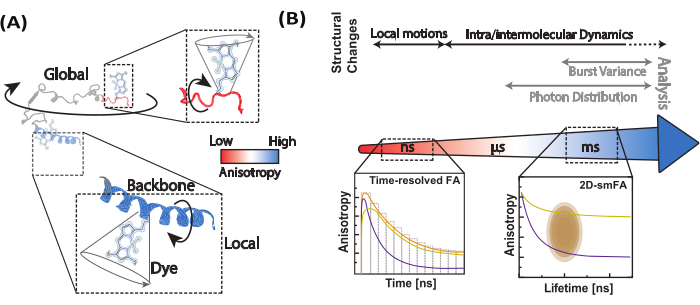
Figure 1 : Gamme dynamique des biomolécules et méthodes d’anisotropie de fluorescence. (A) L’anisotropie de petits fluorophores attachés à diverses positions le long de l’épine dorsale de la biomolécule d’intérêt informe la dynamique structurale locale. (B) Échelles de temps sondées par les décroissances d’intensité de fluorescence (anisotropie de fluorescence résolue en temps, FA) et histogrammes monomoléculaires de données de microscope confocal monomoléculaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Enregistrement et traitement des données d’anisotropie de fluorescence d’une molécule unique. (A) Les molécules à diffusion libre sont analysées à l’aide d’un microscope confocal à molécule unique équipé d’un seul laser d’excitation à polarisation linéaire (bleu dans notre cas). L’émission de fluorescence (verte dans notre cas) est détectée par deux détecteurs après qu’un polariseur de faisceau ait divisé le signal en deux polarisations (parallèle, 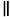 , et perpendiculaire,
, et perpendiculaire,  à la source d’excitation). (B) Chaque photon détecté est caractérisé par trois paramètres : le micro-temps, le macro-temps et le type de canal. Les données sont stockées dans un format TTTR (Time-Tagged Time-Solved)72. (C) Des rafales de molécules individuelles sont sélectionnées et traitées pour extraire les paramètres de fluorescence, y compris l’anisotropie de fluorescence pour chaque molécule observée. (D) Les données sont représentées de plusieurs façons, y compris des graphiques bidimensionnels de l’anisotropie de fluorescence en fonction de la durée de vie de la fluorescence et des décroissances de l’anisotropie résolues dans le temps. Ces représentations permettent de déterminer à la fois visuellement et quantitativement la durée de vie de la fluorescence, les temps de corrélation de rotation et l’hétérogénéité du système. Veuillez cliquer ici pour voir une version agrandie de cette figure.
à la source d’excitation). (B) Chaque photon détecté est caractérisé par trois paramètres : le micro-temps, le macro-temps et le type de canal. Les données sont stockées dans un format TTTR (Time-Tagged Time-Solved)72. (C) Des rafales de molécules individuelles sont sélectionnées et traitées pour extraire les paramètres de fluorescence, y compris l’anisotropie de fluorescence pour chaque molécule observée. (D) Les données sont représentées de plusieurs façons, y compris des graphiques bidimensionnels de l’anisotropie de fluorescence en fonction de la durée de vie de la fluorescence et des décroissances de l’anisotropie résolues dans le temps. Ces représentations permettent de déterminer à la fois visuellement et quantitativement la durée de vie de la fluorescence, les temps de corrélation de rotation et l’hétérogénéité du système. Veuillez cliquer ici pour voir une version agrandie de cette figure.
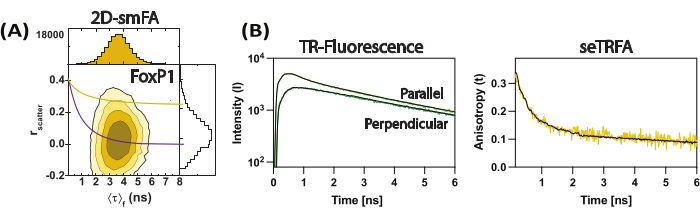
Figure 3 : Données représentatives pour le dimère à échange de domaine FoxP1. (A) Corrélation de l’anisotropie de fluorescence (diffusion r) avec la durée de vie moyenne de fluorescence par molécule sous forme de graphique de contour. Superposition d’une seule équation de Perrin pour deux composantes de rotation comme représentative de la moyenne d’ensemble de la molécule, en considérant ρ1 et ρ2 de 0,2 ns et 8,5 ns, respectivement. (B) Les décroissances de fluorescence résolues en temps de sous-ensembles sont utilisées pour calculer l’anisotropie de fluorescence résolue en temps de l’échantillon. L’ajustement avec l’équation 2 a résolu les composantes locales et globales de l’anisotropie de fluorescence. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Dynamique sub-milliseconde de FoxP1 surveillée à l’aide de l’anisotropie de fluorescence à molécule unique (smFA). (A) Une représentation graphique de la structure monomère de FoxP1. (B) Un histogramme bidimensionnel illustre l’hétérogénéité dynamique, révélant deux temps de corrélation de rotation distincts identifiés par anisotropie de fluorescence résolue en temps. L’analyse de variance en rafale d’anisotropie résolue dans le temps (traBVA) révèle un petit sous-ensemble d’événements avec une variance excessive (équation 6) qui présentent une grande anisotropie. L’analyse quantitative dynamique de l’anisotropie à l’aide de l’analyse de la distribution des photons (PDA) permet d’extraire les taux de change de ce processus. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Projection des mouvements locaux et globaux de FoxP1 pendant la dimérisation. (A) Une représentation de dessin animé compare le monomère FoxP1 à sa forme dimérique. (B) L’excès de variance moyen par emplacement dans des conditions monomères et dimériques est indiqué, avec une variance excédentaire plus importante indiquant des changements plus significatifs de l’anisotropie. (C) L’analyse dynamique de l’anisotropie à l’aide de l’analyse de la distribution des photons (PDA) permet de déterminer les fractions de population (anisotropie élevée dans les couleurs sombres et anisotropie faible dans les couleurs claires) en l’absence (vert) et la présence (jaune) d’ADN. Dans cette approche, des taux (non présentés) ont été estimés pour les transitions entre les comportements locaux et globaux, révélant que FoxP1 subit un déploiement partiel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Pour les expériences d’anisotropie de fluorescence à molécule unique, il est crucial d’examiner attentivement les propriétés photophysiques du fluorophore choisi. Ces propriétés comprennent la longueur d’onde d’émission, qui doit s’aligner sur le système de détection, et la longueur d’onde d’excitation, qui doit être compatible avec les lasers pulsés disponibles. Pour optimiser la plage dynamique, le fluorophore doit avoir une longue durée de vie de fluorescence par rapport au temps de diffusion en rotation de la molécule. Ceci est essentiel pour suivre la dynamique de rotation et la liaison/orientation du dipôle du fluorophore par rapport à la biomolécule d’intérêt. De plus, la luminosité, la photostabilité et le rendement quantique sont essentiels pour produire des signaux forts avec un rapport signal/bruit stable. Pour ces raisons, BODIPY-FL a été choisi comme fluorophore dans plusieurs études 39,40,42.
Le criblage de la dynamique de base des biomolécules nécessite souvent un marquage des protéines, généralement réalisé par un marquage spécifique au site. Cela se fait généralement en introduisant un résidu pour une modification chimique ciblée. L’approche la plus courante consiste à introduire des cystéines à des positions d’intérêt, où leurs chaînes latérales thiols peuvent être modifiées sélectivement avec des réactifs tels que les maléimides ou les iodoacétamides. Moins fréquemment, les halogénures benzyliques et les bromométhylcétones sont utilisés pour former des liaisons thioéther. D’autres chaînes latérales d’acides aminés peuvent également être ciblées, mais leur abondance en protéines est moins couramment utilisée. Cependant, des approches alternatives, comme les acides aminés non naturels, peuvent également être utilisées73. Le choix approprié du site pour l’étiquetage est crucial pour minimiser l’interférence avec la biomolécule à l’étude, et des contrôles appropriés doivent être mis en place. Par exemple, si la molécule marquée est utilisée dans des tests de liaison, les méthodes complémentaires sans marquage doivent vérifier que les fluorophores n’ont pas d’impact sur l’affinité de liaison.
Après avoir identifié l’échantillon approprié et mis en œuvre la stratégie de marquage optimale, l’étape suivante consiste à s’assurer que le microscope confocal est correctement aligné et calibré pour les expériences sur une seule molécule. Le protocole décrit comment déterminer le facteur requis pour une analyse plus approfondie. Une fois l’instrument étalonné, l’étape suivante consiste à mesurer l’échantillon et à traiter les données pour extraire autant d’informations que possible des photons détectés. Les paramètres clés, tels que le micro-temps, le macro-temps et le type de canal, comme le montre la figure 2, peuvent être utilisés pour une analyse et une visualisation plus poussées à l’aide de l’électronique TCSPC typique.
Les progrès récents de la spectroscopie de fluorescence à molécule unique peuvent être largement utilisés pour étudier les informations structurelles provenant d’ensembles hétérogènes de biomolécules. Cependant, relativement peu d’études exploitent les informations fournies par l’anisotropie de fluorescence, et un modèle protéique complet est nécessaire pour dériver la dynamique structurelle des biomolécules. Par conséquent, il est difficile de démêler la dynamique des interactions interdomaines et protéine-protéine de plusieurs facteurs de transcription.
En conclusion, les expériences d’anisotropie de fluorescence à molécule unique offrent des informations complémentaires sur les mouvements locaux et globaux du squelette biomoléculaire, qui sont essentielles pour comprendre sa fonction.
Déclarations de divulgation
Tous les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrent avec le contenu de cet article.
Remerciements
Ce travail a été soutenu par les subventions FONDECYT 11200729 et FONDEQUIP EQM200202 à E.M., NIH R15CA280699 R01GM151334 et NSF CAREER MCB 1749778 bourses à HS. NK a reconnu le soutien du programme de bourses postdoctorales de l’Université Clemson.
matériels
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Références
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.