Method Article
Анизотропия флуоресценции с временным разрешением из одиночных молекул для характеристики локальной гибкости биомолекул
В этой статье
Резюме
В данной работе представлен протокол исследования локальной гибкости и динамики биомолекул с использованием анизотропии флуоресценции с временным разрешением на уровне одной молекулы в режиме конфокальной микроскопии.
Аннотация
Мы описываем протокол проведения анизотропии флуоресценции с временным разрешением на уровне одной молекулы с использованием конфокальной микроскопии для исследования локальной гибкости и динамики домена вилочной головки (FKH), связывающей дезоксирибонуклеиновую кислоту (ДНК) транскрипционного фактора FoxP1. FoxP1 димеризуется с помощью трехмерного механизма переключения доменов (3D-DS), образуя неупорядоченный промежуточный продукт с ДНК или без нее. Поскольку 3D-DS включает в себя внутренне неупорядоченную область, понимание ее поведения имеет решающее значение для выяснения структурных и функциональных свойств FoxP1. Используя меченный одним цистеином FoxP1, мы провели эксперименты с одномолекулярной флуоресцентной анизотропией (smFA), применяя методы динамического анализа распределения фотонов (daPDA) и дисперсионного анализа вспышек анизотропии с временным разрешением (traBVA) для зондирования локальной гибкости и динамики. Этот протокол предоставляет подробное пошаговое руководство по измерениям smFA, уделяя особое внимание анализу с временным разрешением, дисперсии и методам распределения вероятностей для захвата структурной динамики в различных временных масштабах. Этот подход позволил нам связать динамику и гетерогенность с димеризацией FoxP1 и связыванием ДНК, подчеркнув сложный механизм действия, характеризующий этот транскрипционный фактор.
Введение
Функциональная активность биомолекул зависит от их молекулярной гибкости и структурной динамики 1,2,3. Естественно, биомолекулы испытывают постоянные тепловые колебания, начиная от быстрых движений и заканчивая долгосрочными конформационными изменениями, влияющими на их функцию (рис. 1). В биомолекулах локальные движения позвоночника способствуют более масштабным глобальным движениям, включая изгиб шарниров в ферментах и значительные конформационные изменения в моторных белках. Методы определения структуры, такие как ядерный магнитный резонанс (ЯМР)5, рентгеновская кристаллография6 и криогенная электронная микроскопия (крио-ЭМ)7, выявили множественные конформации в различных биомолекулах. Тем не менее, связь локальных флуктуаций с большой конформационной динамикой биомолекул и их роль в функционировании в основном не изучены. Соотнесение динамики и структуры может быть сложной задачей, особенно для внутренне неупорядоченных белков (IDP)8,9,10. В отличие от структурированных белков, IDP не поддерживают стабильную третичную структуру. Вместо этого они претерпевают обширные конформационные изменения с одинаковыми уровнями свободной энергии, что делает возможным широкий спектр биологическойактивности.
Для исследования конформационной динамики белков было использовано несколько экспериментальных подходов путем изучения их молекулярной гибкости 1,13,14,15,16. Среди них ЯМР выделяется своей способностью обеспечивать разрешение на атомном уровне в различных временных масштабах, от десятков пикосекунд до нескольких часов12. Тем не менее, определение гибкости макромолекул остается сложной задачей из-за высоких степеней свободы и для белков большого размера; таким образом, ЯМР часто ограничивается изучением биомолекул с массой около 100 кДа17.
Учитывая структурную сложность высокодинамичных белков, таких как IDP, были разработаны дополнительные методологические достижения для исследования локального и дальнего конформационного пространства для пониманияих функции. Одномолекулярная многопараметрическая флуоресцентная спектроскопия (smMFS)18,19,20,21,22 предоставляет обширную информацию о биомолекулах, обеспечивая важнейшее понимание их функции, конформационной динамики, состояний связывания и стехиометрии. Тем не менее, интерпретация огромного количества структурных данных, полученных от биомолекул, является сложной задачей, а такие факторы, как молекулярная динамика, поведение флуорофоров и сложное поведение молекул, могут еще больше усложнить анализ данных 23,24,25,26,27,28.
Мы используем одномолекулярную флуоресцентную анизотропию (smFA) в качестве надежного метода оценки локальной и глобальной динамики вдоль основы биомолекул (рис. 1A). Флуоресцентная анизотропия, впервые описанная Перрином29 и введенная Вебером 30,31 в качестве биоаналитического инструмента32, была позже адаптирована для исследований на одной молекуле с появлением методов флуоресценции с временным разрешением и увеличением чувствительности детекторов 33,34,35,36,37 . smFA охватывает широкий диапазон временных масштабов — от пикосекунд до нескольких часов — и дополняет данные, полученные в экспериментах по резонансному переносу энергии Фёрстера (smFRET)на основе одной молекулы.
smFA может быть визуализирована в различных форматах для извлечения важной информации о биомолекулярной динамике (рис. 1B). Распады анизотропии флуоресценции с временным разрешением представляют собой одномерные гистограммы, которые фиксируют динамику в масштабах от пикосекунды до наносекунды39,40. Двумерные одномолекулярные гистограммы, которые коррелируют время жизни флуоресценции с анизотропией для отдельных молекул, могут выявить неоднородность состояния анизотропии и дать визуальное представление о динамике потенциала за время наблюдения в конфокальных экспериментах (~мс)41,42. Для изучения динамики за доли миллисекунды можно использовать анализ динамического анизотропного распределения фотонов (daPDA), в то время как анализ дисперсии всплесков с временным разрешением (traBVA) предлагает надежный метод подтверждения конкретной динамики в течение43 миллисекунд (рис. 1B).
Эти методы дополняют более традиционные инструменты, такие как флуоресцентная корреляционная спектроскопия (pFCS) с поляризационным разрешением, которая имеет более широкий спектр 44,45,46,47. В целом, несколько инструментов анализа данных для smFA облегчают выявление локальных и глобальных конформационных изменений при условии правильной калибровки.
Здесь мы применяем smFA для изучения связывания ДНК транскрипционного фактора FoxP1 человека 48,49,50,51. Этот белок использует димер со сменой доменов из-за внутренне неупорядоченной природы его полипептидной цепи, которая заметно подвержена влиянию в зависимости от четвертичного состояния белка и наличия ДНК. Мы сгенерировали различные одиночные цистеиновые мутанты для мечения с помощью BODIPY-FL, провели эксперименты с smFA и использовали daPDA и trBVAa. Такой подход позволил нам связать динамику и гетерогенность с димеризацией FoxP1 и связыванием ДНК, подчеркнув сложный механизм действия, характеризующий этот фактор транскрипции.
протокол
Примечание: Выбор правильного флуорофора имеет важное значение для экспериментов с smFA. Биомолекулы могут быть помечены в сайт-специфичных позициях либо путем модификации аминокислот в белках, либо нуклеотидных оснований в нуклеиновых кислотах флуоресцентными маркерами, в зависимости от доступных реакционноспособных групп. Среди органических красителейсемейства 52 семейства Alexa Fluor, Cy, BODIPY и Janelia Farms являются наиболее популярными вариантами для smFA благодаря их длительному сроку службы флуоресценции, фотостабильности и высокому квантовому выходу. BODIPY-FL часто предпочитают за увеличенный срок службы флуоресценции, превосходный квантовый выход и короткий соединительный линкер. Кроме того, альтернативные флуорофоры обычно используются при скрининге лекарственных средств, где предпочтение отдается объемным методам53. Химерные флуоресцентные белки также могут быть использованы для экспериментов по анизотропии живых клеток и визуализации, хотя существует ограничение в виде более низкого динамического диапазона.
1. Подготовка буфера
ПРИМЕЧАНИЕ: Во время проведения лабораторных экспериментов надевайте перчатки, защитные очки для глаз и лабораторный халат.
- Стандартный буфер (20 мМ 4- (2-гидроксиэтил)-1-пиперазинетансульфоновая кислота [HEPES], pH 7,8, 150 mM NaCl): растворить 2,38 г HEPES, 4,38 г NaCl в 400 мл ультрачистой воды, отрегулировать pH до 7,8 и сделать конечный объем 500 мл.
- Буфер для лизиса (20 мМ HEPES, pH 7,8, 150 мМ NaCl, 0,1 мМ фенилметилсульфонилфторид [PMSF], 10 мкг/мл ДНК): растворите 2,38 г HEPES, 4,38 г NaCl и 8,71 мг PMSF и конечную концентрацию 10 мкг/мл ДНКазы в 400 мл сверхчистой воды, отрегулируйте pH до 7,8 и доведите конечный объем до 500 мл.
- Уравновешивающий буфер (50 мМ фосфатно-солевого буфера [PBS], 150 мМ NaCl и 10 мМ имидазола; pH 7,4): растворить 5,05 г Na2HPO4, 0,85 г2PO4, 4,38 г NaCl и 0,34 г имидазола в 400 мл ультрачистой воды, отрегулировать pH до 7,4 и довести конечный объем до 500 мл.
- Промывочный буфер (50 мМ PBS, 150 мМ NaCl и 30 мМ имидазола; pH 7,4): 5,05 г Na2HPO4, 0,85 г2PO4, 4,38 г NaCl и 1,02 г имидазола в 400 мл сверхчистой воды, отрегулировать pH до 7,4 и сделать конечный объем 500 мл.
- Элюирующий буфер (50 мМ PBS, 150 мМ NaCl и 250 мМ имидазол; pH 7,4): 5,05 г Na2HPO4, 0,85 г2PO4, 4,38 г NaCl и 8,5 г имидазола в 400 мл сверхчистой воды, отрегулируйте pH до 7,4 и доведите конечный объем до 500 мл.
- Буфер PBS (50 мМ фосфатно-натрий-фосфатного буферного раствора, 150 мМ NaCl, pH 7,4). Растворите 5,05 г Na2HPO4, 0,85 г2PO4 и 4,38 г NaCl в 400 мл ультрачистой воды, отрегулируйте pH до 7,4 и доведите окончательный объем до 500 мл.
- Стерилизуйте все буферы, отфильтровав раствор с помощью фильтров с размером пор 0,22 мкм, и храните буферы при комнатной температуре (RT).
- Отфильтруйте стандартный буфер с помощью угольного фильтра для получения отдельных молекул.
2. Флуоресцентные щупы
- BODIPY-FL: Растворите 5 мг флакона, содержащего BODIPY-FL, добавив 1,29 мл свежеоткрытого диметилсульфоксида (ДМСО), чтобы получить конечную концентрацию 10 мМ BODIPY-FL.
- Родамин 110: Растворите 3,67 мг Родамина 110, добавив окончательный объем 1 мл только что открытого ДМСО, чтобы получить концентрацию 10 мМ Родамина 110.
ПРИМЕЧАНИЕ: Следует избегать использования флуоресцентных зондов при воздействии света. Всегда используйте светочувствительную трубку (янтарного цвета) и оберните ее алюминиевой фольгой. Если ДМСО уже открыт, его необходимо хранить в эксикаторе для увеличения срока годности. - Приготовьте небольшие по объему аликвоты подготовленного флуоресцентного зонда и храните его при температуре -20 °С до дальнейшего использования.
ПРИМЕЧАНИЕ: Избегайте циклов замораживания-размораживания, чтобы повысить эффективность маркировки.
3. Калибровочные измерения
- Проводите эксперименты по многопараметрическому детектированию флуоресценции (МФУ) на собственной установке с поляризационным разрешением с 4 каналами детектирования и 2 импульсными лазерами (синим 485 нм и красным 640 нм)54. Однако для измерения анизотропии используйте синий лазер и два канала детектора.
- Включите каналы детектора (параллельные и перпендикулярные) и синюю мощность лазера.
- Убедитесь, что мощность лазера установлена на 60 μВт, и откройте панель управления программного обеспечения36 программного обеспечения для подсчета одиночных фотонов с корреляцией по времени (TCSPC).
- Смешайте 1 мкл 100 нМ Родамина 110 и 49 мкЛ дистиллированной воды до получения конечной концентрации 2 нМ Родамина 110. Добавьте 50 мкл 2 нМ Rhodamine 110 в центр покровного стекла для калибровочных измерений.
- Затем добавьте каплю иммерсионной жидкости (масляной или водяной, но с учетом одинакового показателя преломления) на верхнюю часть линзы объектива микроскопа, чтобы увеличить разрешающую способность микроскопа.
- Поместите покровное стекло на верхнюю часть линзы объектива и убедитесь, что капля воды находится в центре объектива микроскопа.
- Отрегулируйте плоскость изображения так, чтобы она находилась внутри раствора и над поверхностью стекла.
- Отрегулируйте ручку, чтобы найти вторую яркую точку фокусировки и поверните на полторы точки. Сфокусируйте лазер на границе раздела стекла и жидкости.
- Увеличьте количество обнаруженных фотонов, регулируя положение точечного отверстия (70 мкм) и контролируя скорость подсчета фотонов.
- Откройте программу в режиме TTTR или T3 с временными метками и нажмите кнопку «Пуск ». Запишите скорость подсчета в течение 120 с и сохраните файл в формате Rhodamine 110.ptu с помощью программного обеспечения для сбора данных. Этого времени сбора должно быть достаточно, учитывая концентрацию (2 нМ).
- Для фоновых измерений добавьте 50 μL дистиллированной воды в центр покровного стекла и повторите шаги 3,5-3,10. Тем не менее, запишите скорость подсчета фотонов в течение 300 с и сохраните файл water.ptu .
- Для других фоновых измерений добавьте 50 мкл стандартного буфера в центр покровного стекла и повторите шаги 3.5-3.10. Запишите скорость подсчета фотонов за 300 с и сохраните стандартный файл buffer.ptu . Затем проанализируйте данные с помощью программного обеспечения для анализа времени жизни флуоресценции с пакетной интеграцией (BIFL).
4. Калибровка и анализ данных
- Откройте программное обеспечение BIFL и нажмите на кнопку «Подтвердить настройку» в автоматическом окне. Далее нажимаем на получение параметров из файла, затем ОК.
- Чтобы проанализировать измерение, нажмите на массив путей к данным и выберите его.
- Затем загрузите файл water.ptu для измерения воды для массивов, таких как Green scatter. Аналогично выберите стандартный файл buffer.ptu для зеленого фона. В поле Зеленый густой выберите Rhodamine 110.ptu.
- В параметрах выбора одной молекулы нажмите кнопку Далее, а затем кнопку Настроить, чтобы отобразить новое всплывающее окно. Нажмите Threshold, чтобы изменить время прихода межфотонов, и выберите время события одной молекулы для среднего времени прихода межфотонов (dt). Затем нажмите мин. #, чтобы выбрать минимальное количество фотонов на событие одной молекулы, затем закройте всплывающее окно, нажав Return. Затем нажмите OK.
- Затем щелкните Параметры подгонки цвета, чтобы настроить начальное время жизни флуоресценции для зеленого цвета, цвета, которые генерируются с параметрами затухания флуоресценции. Настройте значения Prompt и Delay, изменив значения From и To. Затем закройте всплывающее окно, нажав Return. Затем нажмите OK.
- Нажмите кнопку Сохранить, чтобы обработать файлы ASCII и сохранить их в выбранной папке. Затем обработайте данные для затухания анизотропии флуоресценции с временным разрешением с помощью ChiSurf22, анализа распределения фотонов55,56 или дисперсионного анализавсплесков 57,58.
ПРИМЕЧАНИЕ: Примерные данные и пошаговые описания использования ChiSurf, PDA и BVA доступны на github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis и github.com/SMB-Lab/feda_tools соответственно. Экспериментальные данные доступны на сайте Zenodo (10.5281/zenodo.13371503).
5. Белковый препарат FoxP1
- Гиперэкспрессия рекомбинантных бактерий FoxP1
- Выполните трансформацию в бактериальные клетки E. coli C41 после быстрого мутагенеза полимеразной цепной реакции (ПЦР) с быстрым изменением сайта.
- Подготовьте среду LB и автоклав для ее стерилизации.
- Предварительно инокулируйте одну колонию E. coli C41 путем добавления в 5 мл LB среды, содержащей 5 мкл (100 мкг/мл) ампициллина. Дайте инкубировать при температуре 37 °C в течение ночи на шейкере инкубатора.
- На следующий день сделайте посев бактерий в больших количествах, добавив в течение ночи предварительный посев в 500 мл среды LB с предварительно добавленным антибиотиком в соотношении 1:500.
- Следите за ростом культуры, измеряя поглощение культуры при длине волны 600 нм.
- Когда оптическая плотность достигнет ~0,5-0,7, индуцируйте экспрессию белка, добавив конечную концентрацию 0,5 мМ изопропил-β-d-1-тиогалактопиранозида (IPTG) и держите культуру при 15 °C в шейкере инкубатора в течение ночи.
- После достижения оптической плотности (600 нм) в диапазоне 1,4-1,6 бактериальные клетки собирают центрифугированием при 3000 g в течение 20 мин при 4 °C. Выбросьте надосадочную жидкость и храните гранулу при температуре -20 °C до использования.
- Рекомбинантная очистка FoxP1
- Лизировать клетки E. coli C41 путем добавления лизисного буфера с использованием любых методов лизиса, таких как ультразвуковая обработка, гомогенизация жидкости, френч-пресс и т.д.
- Центрифугируйте лизат при 14000 г в течение 10 мин при 4 °C.
- Для белков, помеченныхHis 6, промойте колонку сродства Ni2+-NTA и уравновесьте NiSO4 с помощью быстрой жидкостной хроматографии белка (FPLC).
- Загрузите белок FoxP1, помеченныйHis 6, в уравновешенную колонку сродства Ni2+-NTA.
- Элюирование белка FoxP1 из колонки Ni2+-NTA с использованием линейного градиента элюирующего буфера.
- После того, как белок будет элюирован, проведите диализ для буферного обмена. Добавьте белок в уравновешивающий буфер без имидазола и диализируйте в течение ночи с помощью магнитной мешалки при 4 °C.
ПРИМЕЧАНИЕ: Если белок очищается с использованием другого метода, проведите замену буфера соответствующим образом. - Чтобы удалить 6-метку His, добавьте протеазу вируса травления табака (TEV) (соотношение TEV:FoxP1 1:100) в диализ на ночь.
ПРИМЕЧАНИЕ: Егорасщепление с 6 тегами также может быть выполнено путем добавления расщепления протеазы на колонке TEV. Этот шаг улучшит выход интересующего белка. - На следующий день повторите шаги из пунктов 5.2.3-5.2.5. Однако на этот раз элюируйте белок в буфере для промывки, а не в буфере для элюирования.
- Сконцентрируйте белок до соответствующего объема, добавив буфер PBS, и количественно оцените белок, используя абсорбцию при длине волны 280 нм, учитывая коэффициент экстинкции белка.
- Рекомбинантная маркировка FoxP1
- Инкубируйте 50-100 мкМ белка FoxP1 с 10-кратным молярным избытком либо дитиотреитола (DTT), либо триса (2-карбоксиэтил)фосфина (TCEP) с конечным объемом 500 мкл при RT в течение 30 мин.
- Замену буфера выполняют с помощью обессоливающих колонн PD 10. Поместите колонку PD10 в центрифужную пробирку объемом 50 мл с помощью адаптера для колонки.
- Уравновесьте колонку PD10, добавив 5 мл буфера PBS, и центрифугируйте при 1000 × г в течение 2 минут. Повторите этот шаг 3 раза.
- Храните колонку PD10 в свежей пробирке объемом 50 мл и добавьте в колонку 2 мл буфера PBS. Затем добавьте в колонку 500 мкл белка FoxP1 из шага 1. Разбавьте белок центрифугированием 1000 × г в течение 2 мин и соберите белок.
- Сконцентрируйте белок, добавив его в ультрацентробежные фильтры (10 кДа MWCO), центрифугируйте при 7500 × г в течение 10 мин и соберите элюй.
- Измерьте концентрацию белка, добавьте BODIPY с 30% эквивалентом концентрации цистеина в белке и поместите его на ротатор на 2 ч при температуре 4 °C.
- Проведите буферный обмен и сконцентрируйте белок, как описано в шагах 5.3.2-5.3.5.
- Проверьте концентрацию белка с помощью спектрофотометра или колориметрического анализа, такого как метод Брэдфорда или Лоури, а также концентрацию красителя по абсорбции на длине волны 500 нм.
- Измерьте степень маркировки по следующей формуле:
(Краситель/εкраситель)/(молекулярная масса белка/миллиграмм белка на миллилитр) = моли красителя/моли белка
Где ADye и εDye — значение поглощения красителя и коэффициент молярного затухания красителя на максимальной длине волны поглощения, соответственно59. При измерении при длине волны 280 нм реальную абсорбцию белка необходимо корректировать по следующей формуле:
Белок = A 280-(Aкраситель· CF280)
Где CF280 — поправочный коэффициент для указанного красителя, учитывая его собственную абсорбцию при 280 нм59.
Примечание: В то время как мы сосредоточены на экспрессии и очистке белка, модифицированного цистеином, необходимо тщательно рассмотреть вопрос о стратегическом введении остатков цистеина в белок.
6. Подготовка камеры для образцов под микроскопом
- Добавьте 495 мкл сверхчистой воды и 5 мкл (0,01% v/v конечной концентрации) Tween-20 (неионогенное поверхностно-активное вещество) в лунку камерного стекла и перемешайте. Инкубировать в течение 30 минут при RT.
- Дважды аккуратно промойте камеру ультрачистой водой и высушите ее.
ПРИМЕЧАНИЕ: Камера готова к использованию для эксперимента.
7. Эксперимент по анизотропии флуоресценции одной молекулы
- Во-первых, откалибруйте прибор smFA. Определите функцию отклика прибора либо путем измерения комбинационного рассеянного света, либо путем измерения флуоресценции красителя с коротким временем жизни и соответствующим спектром излучения (например, эритрозина или малахитового зеленого), дополнительно гасимого насыщенным раствором йодида калия, KI.
- Чтобы сделать поправку на фоновую флуоресценцию, выполните измерения буферного раствора. Откалибруйте относительную чувствительность параллельных и перпендикулярных детекторов (G-фактор) путем получения данных от быстро вращающегося красителя. Здесь для калибровки детекторов 110 использовался 2 нМ Rhodamine60.
- Для экспериментов с smFA на мономерном FoxP1 добавьте 100 пМ меченого BODIPY FoxP1 и 100 нМ немеченного FoxP1 в 500 мкл стандартного буфера (20 мМ HEPES, 20 мМ NaCl, pH 7,8) в предметное стекло.
- Для титрования с ДНК и мономером FoxP1 добавьте 400 нМ ДНК в 100 пМ смесь немеченых белков BODIPY/100 нМ и инкубируйте при РТ в течение 10 минут.
- Для экспериментов с smFA на димерном FoxP1 добавьте 500 нМ немеченого FoxP1 к 100 пМ меченого BODIPY FoxP1 в стандартном буфере. Затем инкубируйте при температуре 37 °C в течение 30 минут.
- Для титрования с ДНК и димерным FoxP1 добавьте 2000 нМ ДНК в смесь 100 пМ BODIPY-меченых/500 нМ немеченых белков и инкубируйте при ЛТ в течение 10 мин.
- Запустите измерения и проверьте в программном обеспечении BIFL, что количество пакетов в записи за 30 секунд составляет от 60 до 90. Если образец более концентрированный, разбавьте его до тех пор, пока он не достигнет этого значения. Этот шаг обеспечивает одномолекулярные условия.
- Начинайте каждое измерение smFA для мономерного FoxP1 или димерного FoxP1 при отсутствии или присутствии ДНК в течение не менее 4 ч.
- Проанализируйте измерения smFA, как описано в разделе 4.
Результаты
Анизотропия флуоресценции возникает из-за относительной ориентации поглощения флуорофора и дипольных моментов излучения. При воздействии на флуорофоры поляризованного света преимущественно возбуждаются флуорофоры с переходными моментами поглощения, выровненными по вектору электрического поля падающего света (фотоселекция). Следовательно, популяция возбужденного состояния становится частично ориентированной, при этом у значительной части возбужденных молекул переходные моменты выровнены по вектору электрического поля поляризованного возбуждающего света61. Флуорофоры вращаются за счет своего броуновского движения. Таким образом, переходный момент излучения также вращается, что приводит к зависимости от анизотропии флуоресценции во времени. Этот эффект может быть использован для измерения вращательных движений флуоресцентных молекул, обнаружения событий связывания, характеристики среды флуорофора и захвата молекулярной динамики.
Эксперименты с одной молекулой обладают уникальными возможностями для определения гетерогенности образца. Использование преимуществ чувствительности к одной молекуле и анизотропии флуоресценции придает многопараметрической флуоресцентной спектроскопии еще одну размерность. В типичном одномолекулярном конфокальном микроскопе (рис. 2)20,21 анизотропия флуоресценции может быть определена с помощью интенсивности или с временным разрешением при использовании импульсных лазеров.
Для рассмотрения деполяризующих эффектов объектива с высокой числовой апертурой в конфокальном микроскопе62 правильная форма анизотропии35,63 с временным разрешением определяется как
 (1)
(1)
где 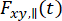 и
и 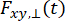 – интенсивность флуоресценции с временным разрешением в y-м канале детектирования после возбуждения на длине волны x, для параллельной и перпендикулярной поляризации l1 и l2 и – коэффициенты, описывающие смешивание между параллельными и перпендикулярными сигналами из-за объектива с высокой числовой апертурой (NA), используемого в этих измерениях35,62, 64. Различия в эффективности детектирования параллельного,
– интенсивность флуоресценции с временным разрешением в y-м канале детектирования после возбуждения на длине волны x, для параллельной и перпендикулярной поляризации l1 и l2 и – коэффициенты, описывающие смешивание между параллельными и перпендикулярными сигналами из-за объектива с высокой числовой апертурой (NA), используемого в этих измерениях35,62, 64. Различия в эффективности детектирования параллельного, 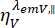 , и перпендикулярного канала детектирования,
, и перпендикулярного канала детектирования,  , для красителя корректируются с помощью отношения эффективности детектирования,
, для красителя корректируются с помощью отношения эффективности детектирования, 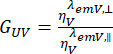 . GUV также называют G-фактором.
. GUV также называют G-фактором.
Анизотропия флуоресценции с временным разрешением может быть смоделирована с использованием мультиэкспоненциального распада, чтобы объяснить присоединение флуорофора к более крупной биомолекуле как
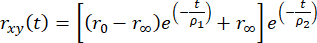 , (2)
, (2)
где r0 — флуорофорно-зависимая фундаментальная анизотропия (обычно r0 = 0,38), R∞ — остаточная анизотропия, а ρ1 и ρ2 — быстрые (локальные движения флуорофора) и медленные (глобальное движение макромолекулы) времени корреляции вращения соответственно.
При измерениях анизотропии одиночных молекул (рис. 2) регистрируется время прихода фотонов для идентификации отдельных излучателей с помощью анализа времени жизни флуоресценции с интегрированной в всплеск (BIFL)33,35. Время прихода межфотонов (Δt) сглаживается с помощью скользящего среднего, а затем строится на графике для облегчения визуализации. Гистограмма этих времен снабжена половиной Гаусса для определения среднего и стандартного отклонения фотонов, происходящих от фона. Произвольный порог, установленный на уровне, кратном стандартному отклонению, используется для фильтрации отдельных событий при идентификации первого и последнего фотонов в каждом всплеске. Фотоны в каждом всплеске затем интегрируются для дальнейшего анализа, который включает в себя расчет стационарной анизотропии флуоресценции с временным разрешением и интенсивностью с использованием уравнений 1 и 2 или с помощью оценки максимального правдоподобия35. Из-за ограниченного числа фотонов в событиях с одной молекулой оценка максимального правдоподобия учитывает только одну экспоненциальную компоненту и не будет обсуждаться далее.
В двумерной гистограмме событий с одной молекулой среднее время жизни флуоресценции (τ) и анизотропия (rxy) могут быть связаны уравнениемПеррена 29,61 для получения (ρ) в качестве среднего времени вращения.
 (3)
(3)
Конкретные значения ρ могут быть получены с большей достоверностью с помощью анализа «субансамбля» (se), в котором фотоны различных всплесков интегрированы в комбинированный анизотропный распад флуоресценции с временным разрешением, который может быть проанализирован путем оптимизации параметров уравнения 2 до экспериментального распада (seTRFA). Анизотропия с временным разрешением может разрешать гетерогенность и динамику, связанные с вращательными движениями (локальными и глобальными) биомолекул в пределах излучения флуоресценции, которое происходит в течение ns-временного интервала.
Для обнаружения динамики в событиях одной молекулы (в субмиллисекундном масштабе) мы ввели анизотропию с временным разрешением Burst Variance Analysis (traBVA)57. В traBVA для фотонного всплеска, содержащего Mi последовательных сегментов фотонов, дисперсия избыточной анизотропии (s2) для пакетов равна
 . (4)
. (4)
Для одного анизотропного состояния дисперсия σ2 возникает исключительно из-за шума дроби65 (sn: √N, где N — количество фотонов)
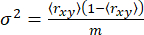 (5)
(5)
где m — количество фотонов в пакете. Следовательно, чтобы выявить дополнительную дисперсию в анизотропии, мы можем определить избыточную дисперсию анизотропии (S2) из-за конформационной неоднородности как разность между уравнениями 4 и 5.
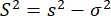 (6)
(6)
Чтобы зафиксировать динамику, происходящую при наблюдении за отдельными молекулами, и рассмотреть приближение дисперсии, можно использовать анализ распределения фотонов с динамической анизотропией (daPDA)55,56. В daPDA интенсивность флуоресценции моделируется с помощью условной вероятности (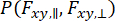 ), выраженной в виде биномиального распределения.
), выраженной в виде биномиального распределения.
 (7)
(7)
Вместе с оценкой скорости фонового подсчета, которая следует за распределением Пуассона
 (8)
(8)
где 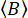 — среднее количество фоновых фотонов за заданное временное окно. Параллельный и перпендикулярный фоновые значения
— среднее количество фоновых фотонов за заданное временное окно. Параллельный и перпендикулярный фоновые значения 
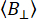 и могут быть измерены с использованием буферных выборок в качестве эталона. Экспериментально определенная анизотропия флуоресценции оптимизируется путем минимизации добротности χ2 с распределением интенсивности флуоресценции на поляризационный канал, которое может включать кинетические изменения.
и могут быть измерены с использованием буферных выборок в качестве эталона. Экспериментально определенная анизотропия флуоресценции оптимизируется путем минимизации добротности χ2 с распределением интенсивности флуоресценции на поляризационный канал, которое может включать кинетические изменения.
Предоставленные процедуры анализа и представления данных предлагают комплексный подход к интерпретации собранных данных. Хотя этот протокол в первую очередь ориентирован на конфокальные измерения, которые ограничены в регистрации изменений анизотропии от наносекунд до миллисекунд, можно использовать микроскоп с полным внутренним отражением для мониторинга анизотропии флуоресценции в более длительных временных масштабах, что позволяет проводить анализ временных рядов66. Для одномолекулярных конфокальных измерений мы выделяем использование многомерных гистограмм, которые создают уникальный отпечаток наблюдаемого ансамбля. Затухания флуоресценции с временным разрешением, реконструированные из выбранных популяций, могут отслеживать эволюцию анизотропии флуоресценции в наносекундном масштабе (рис. 3). Анализ распределения фотонов55,56 и дисперсионный анализ всплесков (BVA)57,58 также могут фиксировать динамику на промежуточных временных масштабах между распадами с временным разрешением и многомерными гистограммами. Хотя этот протокол не охватывает использование поляризационной флуоресцентной корреляционной спектроскопии (FCS) с импульсным возбуждением67,68 или без него, которая может соединять наносекундный и миллисекундный временные масштабы, те же данные могут быть использованы для вычисления FCS69, хотя это выходит за рамки представленного протокола. Если такие эксперименты проводятся, рекомендуется увеличить время измерения образца.
Этот подход был применен к такой сложной системе, как человеческие белки FoxP, что дает ценную информацию о движениях, участвующих в механизме их действия. Белки FoxP являются транскрипционными факторами, участвующими в нескольких физиологических аспектах, таких как развитие мозга и легких; Важно отметить, что различные мутации были признаны нарушающими функцию этих белков70,71. Используя ДНК-связывающий домен FoxP1 в качестве модели, мы сгенерировали различные мутанты с одним цистеином для введения красителя BODIPY-FL в качестве трекера движений (рис. 4A). Фактически, мы оценили эффект димеризации и связывания ДНК как основных структурных регуляторов этого белка. Используя подход smFA, мы сгенерировали 2D-smFA графики и изготовили traBVA и daPDA для каждого мутанта в мономерных и димерных условиях. Мы показываем пример одного из изученных одиночных мутантов (рис. 4). Поведение анизотропии сходно у всех мутантов с точки зрения определения высокого и низкого времени корреляции вращения и, следовательно, предполагаемых, неупорядоченных и свернутых ансамблей. Тем не менее, он также очень гетероген у всех мутантов с точки зрения фракции и кинетики каждого ансамбля, свидетельствуя о различных изменениях перехода от порядка к беспорядку под влиянием димеризации и связывания ДНК, и показывает описание структурной динамики вдоль цепи с высоким разрешением (рис. 5).
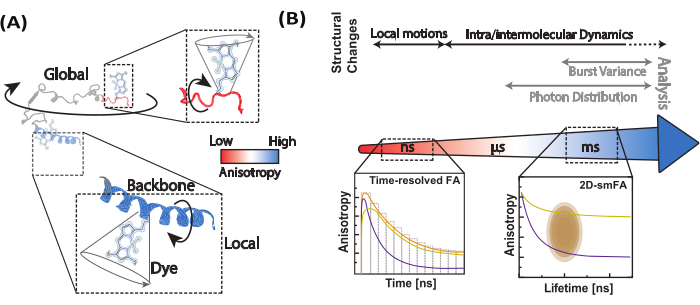
Рисунок 1: Динамический диапазон биомолекул и методы флуоресцентной анизотропии. (А) Анизотропия малых флуорофоров, привязанных к различным положениям вдоль хребта исследуемой биомолекулы, определяет локальную структурную динамику. (B) Временные шкалы, исследуемые по затуханиям интенсивности флуоресценции (анизотропия флуоресценции с временным разрешением, FA) и одномолекулярным гистограммам данных конфокального одномолекулярного микроскопа. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 2: Регистрация и обработка данных об анизотропии флуоресценции одной молекулы. (А) Свободно диффундирующие молекулы анализируются с помощью конфокального одномолекулярного микроскопа, оснащенного одним лазером линейно поляризованного возбуждения (в нашем случае синим). Флуоресцентное излучение (в нашем случае зеленое) регистрируется двумя детекторами после того, как поляризатор луча расщепляет сигнал на две поляризации (параллельную, 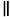 , и перпендикулярную,
, и перпендикулярную,  , к источнику возбуждения). (B) Каждый обнаруженный фотон характеризуется тремя параметрами: микровременем, макровременем и типом канала. Данные хранятся в формате TTTR (Time-Tagged Time-Resolved (TTTR)72. (C) Всплески отдельных молекул отбираются и обрабатываются для извлечения параметров флуоресценции, включая анизотропию флуоресценции для каждой наблюдаемой молекулы. (D) Данные представлены различными способами, включая двумерные графики зависимости анизотропии флуоресценции от времени жизни флуоресценции и распада анизотропии с временным разрешением. Эти представления позволяют как визуально, так и количественно определять время жизни флуоресценции, время корреляции вращения и неоднородность системы. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
, к источнику возбуждения). (B) Каждый обнаруженный фотон характеризуется тремя параметрами: микровременем, макровременем и типом канала. Данные хранятся в формате TTTR (Time-Tagged Time-Resolved (TTTR)72. (C) Всплески отдельных молекул отбираются и обрабатываются для извлечения параметров флуоресценции, включая анизотропию флуоресценции для каждой наблюдаемой молекулы. (D) Данные представлены различными способами, включая двумерные графики зависимости анизотропии флуоресценции от времени жизни флуоресценции и распада анизотропии с временным разрешением. Эти представления позволяют как визуально, так и количественно определять время жизни флуоресценции, время корреляции вращения и неоднородность системы. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
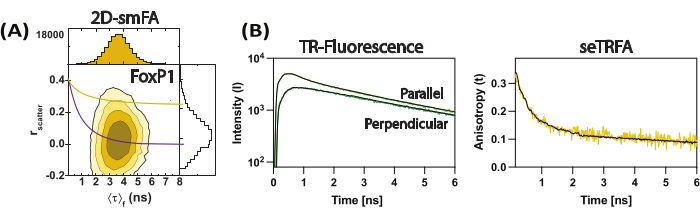
Рисунок 3: Репрезентативные данные для димера FoxP1 с заменой доменов. (A) Корреляция анизотропии флуоресценции (rрассеяния) со средним временем жизни флуоресценции на молекулу в виде контурного графика. Наложение одного уравнения Перрена для двух вращательных компонент в качестве репрезентативного ансамблевого среднего молекулы с учетом ρ1 и ρ2 0,2 нс и 8,5 нс соответственно. (B) Субансамблевые затухания флуоресценции с временным разрешением используются для вычисления анизотропии флуоресценции образца с временным разрешением. Соответствие уравнению 2 позволило разрешить локальную и глобальную составляющие анизотропии флуоресценции. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 4: Динамика FoxP1 за доли миллисекунды отслеживается с помощью одномолекулярной флуоресцентной анизотропии (smFA). (A) Мультяшное представление мономерной структуры FoxP1. (B) Двумерная гистограмма иллюстрирует динамическую неоднородность, выявляя два различных времени корреляции вращения, идентифицированных с помощью анизотропии флуоресценции с временным разрешением. Анализ дисперсии всплесков с временным разрешением (traBVA) выявляет небольшое подмножество событий с избыточной дисперсией (уравнение 6), которые демонстрируют большую анизотропию. Количественный анализ динамической анизотропии с использованием анализа распределения фотонов (PDA) позволяет дополнительно извлечь обменные курсы для этого процесса. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 5: Экранирование локальных и глобальных движений FoxP1 во время димеризации. (A) В мультфильме сравнивается мономерный FoxP1 с его димерной формой. (B) Показана средняя избыточная дисперсия для каждого местоположения в мономерных и димерных условиях, при этом большая избыточная дисперсия указывает на более значительные изменения в анизотропии. (C) Анализ динамической анизотропии с использованием анализа распределения фотонов (PDA) помогает определить популяционные доли (высокая анизотропия в темных цветах и низкая анизотропия в светлом цвете) при отсутствии (зеленый) и присутствии (желтый) ДНК. В этом подходе были оценены скорости (не показаны) для переходов между локальным и глобальным поведением, что показывает, что FoxP1 претерпевает частичное развертывание. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обсуждение
Для экспериментов по анизотропии флуоресценции одной молекулы крайне важно тщательно рассмотреть фотофизические свойства выбранного флуорофора. Эти свойства включают в себя длину волны излучения, которая должна совпадать с системой детектирования, и длину волны возбуждения, которая должна быть совместима с имеющимися импульсными лазерами. Для оптимизации динамического диапазона флуорофор должен иметь длительное время жизни флуоресценции относительно времени вращательной диффузии молекулы. Это имеет решающее значение для отслеживания вращательной динамики и связи/ориентации диполя флуорофора относительно интересующей биомолекулы. Кроме того, яркость, фотостабильность и квантовый выход имеют важное значение для получения сильных сигналов со стабильным соотношением сигнал/шум. По этим причинам BODIPY-FL был выбран в качестве флуорофора в нескольких исследованиях 39,40,42.
Скрининг динамики основной массы биомолекул часто требует мечения белков, что обычно достигается с помощью сайт-специфичного мечения. Обычно это делается путем введения остатка для целенаправленной химической модификации. Наиболее распространенным подходом является введение цистеинов в интересующие позиции, где их тиоловые боковые цепи могут быть селективно модифицированы такими реагентами, как малеимиды или йодоацетамиды. Реже бензиловые галогениды и бромметилкетоны используются для образования тиоэфирных связей. Другие боковые цепи аминокислот также могут быть мишенью, но их обилие в белках используется реже. Тем не менее, альтернативные подходы, такие как неестественные аминокислоты, также могут быть использованы73. Правильный выбор места для мечения имеет решающее значение для минимизации вмешательства в исследуемую биомолекулу, и должны быть приняты соответствующие меры контроля. Например, если меченая молекула используется в анализах связывания, комплементарные безметочные методы должны убедиться, что флуорофоры не влияют на аффинность связывания.
После определения подходящего образца и реализации оптимальной стратегии мечения следующим шагом является обеспечение правильной юстировки и калибровки конфокального микроскопа для экспериментов с одиночными молекулами. В протоколе описано, как определить необходимый фактор для дальнейшего анализа. После калибровки прибора следующим шагом является измерение образца и обработка данных, чтобы извлечь как можно больше информации из обнаруженных фотонов. Ключевые параметры, такие как микровремя, макровремя и тип канала, как показано на рисунке 2, могут быть использованы для дальнейшего анализа и визуализации с использованием типичной электроники TCSPC.
Последние достижения в области флуоресцентной спектроскопии одиночных молекул могут быть широко использованы для изучения структурной информации из гетерогенных ансамблей биомолекул. Тем не менее, относительно небольшое количество исследований использует идеи, полученные в результате флуоресцентной анизотропии, и для получения структурной динамики биомолекул требуется полная белковая модель. Таким образом, разгадка динамики междоменных и белок-белковых взаимодействий нескольких транскрипционных факторов является сложной задачей.
В заключение следует отметить, что эксперименты с флуоресцентной анизотропией одной молекулы дают дополнительную информацию о локальных и глобальных движениях биомолекулярного каркаса, которые имеют решающее значение для понимания его функции.
Раскрытие информации
Все авторы заявляют, что у них нет конкурирующих финансовых интересов с содержанием данной статьи.
Благодарности
Эта работа была поддержана грантами FONDECYT 11200729 и FONDEQUIP EQM200202 E.M., NIH R15CA280699 R01GM151334 и NSF CAREER MCB 1749778 наградами HS. NK выразил признательность за поддержку со стороны программы стипендий для постдокторантов Университета Клемсона.
Материалы
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Ссылки
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. . Principles of Protein X-Ray Crystallography. , (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701 (2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. a. M. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. a. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676 (2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854 (2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724 (2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. . Fluorescence Correlation Spectroscopy: Theory and Applications. , (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375 (2020).
- Takahashi, H., Takahashi, K., Liu, F. -. C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302 (2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623 (2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116 (2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1 (2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077 (2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004 (2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -. J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -. O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. . Time tagged time-resolved fluorescence data collection in life sciences. , (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
ISSN 2578-2037
Авторские права © 2025 MyJoVE Corporation. Все права защищены
Мы используем файлы cookie для улучшения качества работы на нашем веб-сайте.
Продолжая пользоваться нашим веб-сайтом или нажимая кнопку «Продолжить», вы соглашаетесь принять наши файлы cookie.