Method Article
אניזוטרופיה פלואורסצנטית שנפתרה בזמן ממולקולות בודדות לאפיון גמישות מקומית בביו-מולקולות
In This Article
Summary
כאן, אנו מציגים את הפרוטוקול לחקר הגמישות והדינמיקה המקומית של ביומולקולות באמצעות אניזוטרופיה פלואורסצנטית שנפתרה בזמן ברמת מולקולה בודדת במצב מיקרוסקופיה קונפוקלית.
Abstract
אנו מתארים פרוטוקול לביצוע אניזוטרופיה פלואורסצנטית שנפתרה בזמן ברמת המולקולה הבודדת באמצעות מיקרוסקופיה קונפוקלית כדי לחקור את הגמישות והדינמיקה המקומית של תחום ראש המזלג המחייב חומצה דאוקסיריבונוקלאית (DNA) (FKH) של גורם השעתוק FoxP1. FoxP1 מתעמעם באמצעות מנגנון החלפת תחום תלת מימדי (3D-DS), ויוצר ביניים לא מסודר עם או בלי DNA. מכיוון ש-3D-DS מערב אזור לא מסודר באופן מהותי, הבנת התנהגותו חיונית להבהרת התכונות המבניות והתפקודיות של FoxP1. באמצעות FoxP1 עם תווית ציסטאין יחידה, ערכנו ניסויים באניזוטרופיה פלואורסצנטית של מולקולה אחת (smFA), תוך יישום גישות ניתוח התפלגות פוטון של אניזוטרופיה דינמית (daPDA) וניתוח שונות פרץ של אניזוטרופיה (traBVA) שנפתרו בזמן כדי לחקור גמישות ודינמיקה מקומיות. פרוטוקול זה מספק מדריך מפורט שלב אחר שלב למדידות smFA, תוך שימת דגש על ניתוחים שנפתרו בזמן, שונות וטכניקות חלוקת הסתברות כדי ללכוד דינמיקה מבנית על פני טווחי זמן שונים. גישה זו אפשרה לנו לקשר דינמיקה והטרוגניות לדימריזציה של FoxP1 וקשירת DNA, תוך הדגשת מנגנון הפעולה המורכב המאפיין גורם שעתוק זה.
Introduction
הפעילות התפקודית של ביומולקולות תלויה בגמישות המולקולרית שלהן ובדינמיקה המבנית שלהן 1,2,3. באופן טבעי, ביומולקולות חוות תנודות תרמיות קבועות, החל מתנועות מהירות ועד לשינויים קונפורמטיביים ארוכי טווח שמשפיעים על תפקודן (איור 1)4. בביומולקולות, תנועות עמוד השדרה המקומיות תורמות לתנועות גלובליות בקנה מידה גדול יותר, כולל כיפוף צירים באנזימים ושינויים קונפורמטיביים משמעותיים בחלבונים מוטוריים. שיטות לקביעת מבנה כגון תהודה מגנטית גרעינית (NMR)5, קריסטלוגרפיה של קרני רנטגן6 ומיקרוסקופ אלקטרונים קריוגני (cryo-EM)7 חשפו קונפורמציות מרובות בביומולקולות שונות. עם זאת, חיבור התנודות המקומיות לדינמיקה קונפורמציונית גדולה של ביומולקולות ותפקידן בתפקוד לא נחקר ברובו. קשר בין דינמיקה ומבנה יכול להיות מאתגר, במיוחד עבור חלבונים עם הפרעות מהותיות (IDPs)8,9,10. בניגוד לחלבונים מובנים, IDPs אינם שומרים על מבנה שלישוני יציב. במקום זאת, הם עוברים שינויים קונפורמטיביים נרחבים עם רמות אנרגיה חופשית דומות, המאפשרים מגוון רחב של פעילויות ביולוגיות11,12.
מספר גישות ניסיוניות שימשו כדי לחקור את הדינמיקה הקונפורמטיבית של חלבונים על ידי בחינת הגמישות המולקולרית שלהם 1,13,14,15,16. בין אלה, NMR בולט ביכולתו לספק רזולוציה ברמה האטומית על פני טווחי זמן שונים, מעשרות פיקו-שניות ועד מספר שעות12. עם זאת, קביעת הגמישות המקרומולקולרית נותרה מאתגרת בשל דרגות החופש הגבוהות ועבור חלבונים בגודל גדול; לפיכך, NMR מוגבל לעתים קרובות לחקר ביומולקולות של כ-100 kDa17.
בהתחשב במורכבות המבנית של חלבונים דינמיים מאוד כמו IDPs, פותחו התקדמות מתודולוגית נוספת כדי לחקור מרחב קונפורמציה מקומי וארוך טווח כדי להבין את תפקודם11. ספקטרוסקופיה פלואורסצנטית רב-פרמטרית של מולקולה אחת (smMFS)18,19,20,21,22 מציעה מידע נרחב על ביומולקולות, ומספקת תובנות מכריעות לגבי תפקודן, דינמיקה קונפורמטיבית, מצבי קשירה וסטוכיומטריה. עם זאת, פירוש הכמות העצומה של נתונים מבניים המתקבלים מביומולקולות הוא מאתגר, וגורמים כמו דינמיקה מולקולרית, התנהגות פלואורופור והתנהגות מורכבת של מולקולות יכולים לסבך עוד יותר את ניתוח הנתונים 23,24,25,26,27,28.
אנו משתמשים באניזוטרופיה פלואורסצנטית של מולקולה בודדת (smFA) כשיטה חזקה להערכת דינמיקה מקומית וגלובלית לאורך עמוד השדרה של ביומולקולות (איור 1A). אניזוטרופיה פלואורסצנטית, שתוארה לראשונה על ידי פרין29 והוצגה על ידי וובר30,31 ככלי ביואנליטי32, הותאמה מאוחר יותר למחקרים של מולקולה בודדת עם הופעתן של טכניקות פלואורסצנטיות שנפתרו בזמן ועלייה ברגישות הגלאים 33,34,35,36,37. smFA משתרע על פני טווח רחב של טווחי זמן - מפיקו-שניות ועד מספר שעות - ומשלים את הנתונים שהתקבלו מניסויי העברת אנרגיה תהודה של Förster (smFRET) של מולקולה בודדת38.
ניתן לדמיין smFA בפורמטים שונים כדי לחלץ מידע קריטי על דינמיקה ביו-מולקולרית (איור 1B). דעיכות אניזוטרופיות פלואורסצנטיות שנפתרו בזמן הן היסטוגרמות חד-ממדיות הלוכדות דינמיקה בקנה מידה של פיקו-שנייה עד ננו-שנייה39,40. היסטוגרמות דו-ממדיות של מולקולה אחת, המתאמות את אורך החיים הקרינה עם אניזוטרופיה עבור מולקולות בודדות, יכולות לחשוף הטרוגניות של מצב אניזוטרופיה ולספק תובנות חזותיות לגבי דינמיקה פוטנציאלית בתוך זמן התצפית בניסויים קונפוקליים (~ms)41,42. עבור חקר דינמיקה תת-אלפית השנייה, ניתן להשתמש בניתוח התפלגות פוטונים אניזוטרופיים דינמיים (daPDA), בעוד שניתוח שונות פרץ אניזוטרופיה (traBVA) מציע שיטה חזקה לאישור דינמיקה ספציפית סביב אלפיות שנייה43 (איור 1B).
שיטות אלו משלימות כלים מסורתיים יותר, כגון ספקטרוסקופיה של מתאם פלואורסצנטי (pFCS), בעלת ספקטרום רחב יותר 44,45,46,47. בסך הכל, כלי ניתוח נתונים מרובים עבור smFA מקלים על זיהוי שינויים קונפורמטיביים מקומיים וגלובליים, בתנאי שנלקח בחשבון כיול מתאים.
כאן, אנו מיישמים smFA כדי לחקור את קשירת ה-DNA של גורם השעתוק האנושי FoxP1 48,49,50,51. חלבון זה מאמץ דימר מוחלף תחום בשל האופי הלא מסודר מהותי של שרשרת הפוליפפטיד שלו, המושפע במיוחד בהתאם למצב הרבעוני של החלבון ונוכחות ה-DNA. יצרנו מוטציות שונות של ציסטאין בודד לתיוג עם BODIPY-FL, ביצענו ניסויי smFA והשתמשנו ב-daPDA ו-trBVAa. גישה זו אפשרה לנו לקשר דינמיקה והטרוגניות לדימריזציה של FoxP1 ולקשירת ה-DNA, תוך הדגשת מנגנון הפעולה המורכב המאפיין גורם שעתוק זה.
Protocol
הערה: בחירת הפלואורופור המתאים חיונית לניסויי smFA. ניתן לתייג ביומולקולות במיקומים ספציפיים לאתר על ידי שינוי חומצות אמינו בחלבונים או בסיסי נוקלאוטידים בחומצות גרעין עם סמנים פלואורסצנטיים, בהתאם לקבוצות התגובתיות הזמינות. מבין הצבעים האורגניים52, משפחות Alexa Fluor, Cy, BODYPY ו-Janelia Farms הן הבחירות הפופולריות ביותר עבור smFA, הודות לחיי הקרינה הארוכים שלהם, יציבות האור והתשואות הקוונטיות הגבוהות שלהם. BODIPY-FL מועדף לעתים קרובות בשל אורך החיים הקרינה הממושך שלו, התפוקה הקוונטית המעולה והמקשר הקצר. בנוסף, פלואורופורים אלטרנטיביים משמשים בדרך כלל בבדיקת תרופות בהן עדיפות טכניקות בתפזורת53. חלבונים פלואורסצנטיים כימריים יכולים לשמש גם לניסויים באניזוטרופיה של תאים חיים והדמיה, אם כי קיימת מגבלה של טווח דינמי נמוך יותר.
1. הכנת חיץ
הערה: ללבוש כפפות, משקפי מגן עיניים ומעיל מעבדה בזמן ביצוע ניסויי מעבדה.
- מאגר סטנדרטי (20 מ"מ 4- (2-הידרוקסיאתיל)-1-פיפרזינאתיאנסולפונית חומצה [HEPES], pH 7.8, 150 מ"מ NaCl): ממיסים 2.38 גרם HEPES, 4.38 גרם NaCl ב-400 מ"ל מים טהורים במיוחד, מכוונים את ה-pH ל-7.8 והופכים את הנפח הסופי ל-500 מ"ל.
- מאגר ליזה (20 מ"מ HEPES, pH 7.8, 150 מ"מ NaCl, 0.1 מ"מ פניל-מתיל-סולפוניל פלואוריד [PMSF], 10 מיקרוגרם/מ"ל DNAse): ממיסים 2.38 גרם HEPES, 4.38 גרם NaCl ו-8.71 מ"ג PMSF וריכוז סופי של 10 מיקרוגרם/מ"ל DNAse ב-400 מ"ל מים טהורים במיוחד, התאימו את ה-pH ל-7.8 והגיעו לנפח הסופי ל-500 מ"ל.
- מאגר שיווי משקל (50 מ"מ תמיסת מלח חוצצת פוספט [PBS], 150 מ"מ NaCl ו-10 מ"מ אימידזול; pH 7.4): ממיסים 5.05 גרם של Na2HPO4, 0.85 גרם NaH2PO4, 4.38 גרם NaCl ו-0.34 גרם אימידזול ב-400 מ"ל מים טהורים במיוחד, התאימו את ה-pH ל-7.4 והפכו את הנפח הסופי ל-500 מ"ל.
- מאגר כביסה (50 מ"מ PBS, 150 מ"מ NaCl ו-30 מ"מ אימידזול; pH 7.4): ממיסים 5.05 גרם של Na2HPO4, 0.85 גרם של NaH2PO4, 4.38 גרם של NaCl ו-1.02 גרם של אימידזול ב-400 מ"ל של מים טהורים במיוחד, התאימו את ה-pH ל-7.4 והפכו את הנפח הסופי ל-500 מ"ל.
- מאגר אלוטיון (50 מ"מ PBS, 150 מ"מ NaCl ו-250 מ"מ אימידזול; pH 7.4): ממיסים 5.05 גרם של Na2HPO4, 0.85 גרם של NaH2PO4, 4.38 גרם NaCl ו-8.5 גרם אימידזול ב-400 מ"ל מים טהורים במיוחד, התאימו את ה-pH ל-7.4 והפכו את הנפח הסופי ל-500 מ"ל.
- מאגר PBS (50 מ"מ נתרן פוספט מלוח, 150 מ"מ NaCl, pH 7.4). ממיסים 5.05 גרם של Na2HPO4, 0.85 גרם של NaH2PO4 ו-4.38 גרם NaCl ב-400 מ"ל מים טהורים במיוחד, התאימו את ה-pH ל-7.4 והפכו את הנפח הסופי ל-500 מ"ל.
- עקר את כל המאגרים על ידי סינון התמיסה באמצעות מסננים בגודל נקבוביות של 0.22 מיקרומטר ואחסן את המאגרים בטמפרטורת החדר (RT).
- סנן את המאגר הסטנדרטי באמצעות מסנן פחם לרכישת מולקולה בודדת.
2. בדיקות פלואורסצנטיות
- BODIPY-FL: יש להמיס בקבוקון של 5 מ"ג המכיל BODIPY-FL על ידי הוספת נפח סופי של 1.29 מ"ל של דימתיל סולפוקסיד (DMSO) שנפתח לאחרונה כדי לקבל את הריכוז הסופי של 10 מ"מ BODIPY-FL.
- רודמין 110: ממיסים 3.67 מ"ג של רודמין 110 על ידי הוספת נפח סופי של 1 מ"ל של DMSO שנפתח לאחרונה כדי לקבל את הריכוז הסופי של 10 מ"מ רודמין 110.
הערה: יש להימנע מבדיקות פלורסנט בעת חשיפה לאור. השתמש תמיד בצינור רגיש לאור (צבע ענבר) ועטוף אותו בנייר אלומיניום. אם DMSO כבר נפתח, יש לשמור אותו במייבש כדי להגדיל את חיי המדף שלו. - הכן כמויות בנפח קטן של הגשושית הפלואורסצנטית המוכנה ואחסן אותה בטמפרטורה של -20 מעלות צלזיוס עד לשימוש נוסף.
הערה: הימנע ממחזורי הקפאה-הפשרה כדי לשפר את יעילות התיוג שלו.
3. מדידות כיול
- בצע ניסויים בזיהוי פלואורסצנטי רב-פרמטרי (MFD) על מערך פתרון קיטוב ביתי עם 4 ערוצי זיהוי ו-2 לייזרים פועמים (כחול 485 ננומטר ואדום 640 ננומטר)54. עם זאת, למדידות אניזוטרופיה, השתמש בלייזר הכחול ובשני ערוצי גלאים.
- הפעל את ערוצי הגלאי (מקבילים וניצבים) ואת כוח הלייזר הכחול.
- ודא שעוצמת הלייזר מוגדרת ל-60 μW ופתח את לוח הבקרה של תוכנת ספירת פוטונים בודדים (TCSPC)36 .
- מערבבים 1 מיקרוליטר של 100 ננומטר רודמין 110 ו -49 מיקרוליטר מים מזוקקים ליצירת ריכוז סופי של 2 ננומטר רודמין 110. הוסף 50 מיקרוליטר של 2 ננומטר רודמין 110 למרכז זכוכית הכיסוי למדידות כיול.
- לאחר מכן, הוסף טיפה של נוזל טבילה (שמן או מים, אך הקפידו לחלוק את אותו מקדם שבירה) על גבי עדשת האובייקט של המיקרוסקופ כדי להגביר את כוח הרזולוציה של המיקרוסקופ.
- הנח את זכוכית הכיסוי על גבי עדשת המטרה וודא שטיפות המים נמצאות במרכז מטרת המיקרוסקופ.
- כוונן את מישור התמונה כך שיהיה בתוך התמיסה ומעל משטח הזכוכית.
- כוונן את הכפתור כדי למצוא את נקודת המוקד הבהירה השנייה ופנה לנקודה וחצי. מקד את הלייזר בממשק הזכוכית והנוזל.
- מקסם את מספר הפוטונים שזוהו על ידי התאמת מיקום חור הסיכה (70 מיקרומטר) תוך ניטור קצב ספירת הפוטונים.
- פתח את התוכנה בלוח המצב שנפתר בזמן (TTTR, או T3) ולחץ על כפתור התחל . רשום את קצב הספירה עבור 120 שניות ושמור את פורמט הקובץ Rhodamine 110.ptu באמצעות תוכנת הרכישה. זמן רכישה זה אמור להספיק בהתחשב בריכוז (2 ננומטר).
- למדידות רקע, הוסף 50 מיקרוליטר מים מזוקקים למרכז זכוכית הכיסוי וחזור על השלבים 3.5-3.10. עם זאת, רשום את קצב ספירת הפוטונים למשך 300 שניות ושמור את הקובץ water.ptu .
- למדידות רקע אחרות, הוסף 50 מיקרוליטר של מאגר סטנדרטי למרכז זכוכית הכיסוי וחזור על שלבים 3.5-3.10. רשום את קצב ספירת הפוטונים למשך 300 שניות ושמור קובץ buffer.ptu סטנדרטי . לאחר מכן, נתח את הנתונים באמצעות תוכנת ניתוח אורך חיים פלואורסצנטי של שילוב פרץ (BIFL).
4. כיול וניתוח נתונים
- פתח את תוכנת BIFL ולחץ על אשר את ההגדרה מהחלון האוטומטי. לאחר מכן, לחץ על קבל פרמטרים מהקובץ ולאחר מכן על אישור.
- כדי לנתח את המדידה, לחץ על מערך נתיבי הנתונים ובחר בו.
- לאחר מכן, טען את מדידת המים water.ptu file עבור מערכים כגון פיזור ירוק. באופן דומה, בחר את הקובץ הסטנדרטי buffer.ptu עבור רקע ירוק. עבור ירוק עבה, בחר רודמין 110.ptu.
- תחת הפרמטרים לבחירת מולקולה בודדת, לחץ על הבא ולאחר מכן על התאם כדי להציג חלון מוקפץ חדש. לחץ על סף כדי לשנות את זמן ההגעה הבין-פוטוני ולבחור זמן אירוע של מולקולה בודדת עבור זמן ההגעה הממוצע בין פוטונים (dt). לאחר מכן, לחץ על min. # כדי לבחור את המספר המינימלי של פוטונים לכל אירוע של מולקולה בודדת, ולאחר מכן סגור את החלון המוקפץ על ידי לחיצה על Return. לאחר מכן, לחץ על אישור.
- לאחר מכן, לחץ על פרמטרים של התאמת צבע כדי להתאים את אורך החיים הקרינה הראשוני עבור ירוק, צבעים הנוצרים עם פרמטרים של דעיכת הקרינה. התאם את ערכי הבקשה והעיכוב על-ידי שינוי הערכים מ ועד. לאחר מכן סגור את החלון המוקפץ על-ידי לחיצה על Return. לאחר מכן, לחץ על אישור.
- לחץ על שמור כדי לעבד את קבצי ASCII ולשמור אותם בתיקייה שנבחרה. לאחר מכן, עבד את הנתונים עבור דעיכת אניזוטרופיה פלואורסצנטית שנפתרה בזמן באמצעות ChiSurf22, ניתוח התפלגות פוטונים 55,56 או ניתוח שונות פרץ57,58.
הערה: נתונים לדוגמה ותיאורים שלב אחר שלב כיצד להשתמש ב-ChiSurf, PDA ו-BVA זמינים ב-github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis ו-github.com/SMB-Lab/feda_tools, בהתאמה. נתוני הניסוי זמינים ב-Zenodo (10.5281/zenodo.13371503).
5. הכנת חלבון FoxP1
- ביטוי יתר חיידקי רקומביננטי FoxP1
- לבצע טרנספורמציה לתאי חיידק E. coli C41 לאחר מוטגנזה של תגובת שרשרת פולימראז (PCR) המכוונת לשינוי מהיר.
- הכן מדיית LB וחיטוי כדי לעקר אותה.
- יש לחסן מראש מושבה אחת של E. coli C41 על ידי הוספה ל-5 מ"ל של מדיום LB המכיל 5 מיקרוליטר (100 מיקרוגרם/מ"ל) אמפיצילין. יש לדגור בטמפרטורה של 37 מעלות צלזיוס למשך הלילה על שייקר אינקובטור.
- למחרת, חסנו תרבית חיידקים בקנה מידה גדול על ידי הוספת חיסון מקדים למשך הלילה ל-500 מ"ל של חומר LB עם אנטיביוטיקה שנוספה מראש ביחס של 1:500.
- עקוב אחר צמיחת התרבות על ידי מדידת ספיגת התרבות ב-600 ננומטר.
- כאשר הצפיפות האופטית מגיעה בין ~0.5-0.7, השרה את ביטוי החלבון על ידי הוספת ריכוז סופי של 0.5 מ"מ איזופרופיל-β-d-1-thiogalactopyranoside (IPTG) ושמור את התרבית על 15 מעלות צלזיוס על שייקר אינקובטור למשך הלילה.
- לאחר השגת צפיפות אופטית (600 ננומטר) בטווח של 1.4-1.6, קצרו את תאי החיידקים על ידי צנטריפוגה ב-3000 גרם למשך 20 דקות ב-4 מעלות צלזיוס. השליכו את הסופרנטנט ואחסנו את הגלולה בטמפרטורה של -20 מעלות צלזיוס עד לשימוש.
- טיהור רקומביננטי FoxP1
- תאי Lyse E. coli C41 על ידי הוספת מאגר ליזה בכל שיטות ליזה כגון סוניקציה, הומוגניזציה נוזלית, עיתונות צרפתית וכו'.
- צנטריפוגה את הליזאט ב-14000 גרם למשך 10 דקות ב-4 מעלות צלזיוס.
- עבור6 החלבונים המתויגים שלו, שטפו את עמודת הזיקה Ni2+-NTA ושיווי משקל עם NiSO4 באמצעות כרומטוגרפיה נוזלית מהירה של חלבון (FPLC).
- טען את חלבון ה-FoxP1 בעל6 התיוגים שלו לתוך עמודת הזיקה המאוזנת Ni2+-NTA.
- מסלק את חלבון FoxP1 מהעמודה Ni2+-NTA באמצעות שיפוע ליניארי של מאגר הפליטה.
- לאחר הסרת החלבון, בצע דיאליזה להחלפת חיץ. מוסיפים חלבון למאגר שיווי משקל ללא אימידזול ומבצעים דיאליזה למשך הלילה באמצעות מערבל מגנטי בטמפרטורה של 4 מעלות צלזיוס.
הערה: אם החלבון מטוהר בשיטה אחרת, בצע את החלפת החיץ בהתאם. - כדי להסיר אתתג 6 שלו, הוסף פרוטאז של וירוס תחריט טבק (TEV) (יחס של 1:100 של TEV:FoxP1) לדיאליזה למשך הלילה.
הערה: ניתן לבצע את עיכול6 התגים שלו גם על ידי הוספת עיכול פרוטאז TEV בעמודה. צעד זה ישפר את תפוקת החלבון המבוקש. - למחרת, חזור על השלבים בין 5.2.3-5.2.5. עם זאת, הפעם, הסירו את החלבון במאגר הכביסה ולא את מאגר הפליטה.
- רכז את החלבון לנפח מתאים על ידי הוספת מאגר PBS וכימת את החלבון באמצעות ספיגה ב-280 ננומטר, בהתחשב במקדם ההכחדה של החלבון.
- תיוג רקומביננטי FoxP1
- דגירה של 50-100 מיקרומטר חלבון FoxP1 עם עודף מולרי פי 10 של דיתיותרייטול (DTT) או טריס (2-קרבוקסיאתיל)פוספין (TCEP) עם נפח סופי של 500 מיקרוליטר ב-RT למשך 30 דקות.
- בצע החלפת מאגר באמצעות עמודות התפלה PD 10. הנח את עמוד PD10 לתוך צינור צנטריפוגה של 50 מ"ל באמצעות מתאם עמודה.
- אזנו את עמודת PD10 על ידי הוספת 5 מ"ל של מאגר PBS וצנטריפוגה ב-1000 × גרם למשך 2 דקות. חזור על שלב זה 3 פעמים.
- שמור את עמודת ה-PD10 בצינור טרי של 50 מ"ל והוסף 2 מ"ל של מאגר PBS לעמודה. לאחר מכן, הוסף 500 מיקרוליטר של חלבון FoxP1 משלב 1 לעמודה. יש לסלק את החלבון על ידי צנטריפוגה של 1000 × גרם למשך 2 דקות ולאסוף את החלבון.
- רכזו את החלבון על ידי הוספתו למסננים אולטרה צנטריפוגליים (10 kDa MWCO), צנטריפוגה ב-7500 × גרם למשך 10 דקות, ואספו את האlute.
- מדוד את ריכוז החלבון, הוסף BODIPY עם 30% שווה ערך לריכוז הציסטאין בחלבון, והנח אותו על מסובב למשך שעתיים ב-4 מעלות צלזיוס.
- בצע חילופי חיץ ורכז את החלבון כפי שנדון בשלבים 5.3.2-5.3.5.
- בדוק את ריכוז החלבון באמצעות ספקטרופוטומטר או בדיקה קולורימטרית, כגון שיטת ברדפורד או לאורי, ואת ריכוז הצבע בספיגה ב-500 ננומטר.
- מדוד את מידת התיוג באמצעות הנוסחה הבאה:
(צבע/ε צבע)/(משקל מולקולרי של חלבון/מיליגרם חלבון למיליליטר) = שומות של צבע/שומות של חלבון
כאשרA Dye ו-ε Dye הם ערך הספיגה של הצבע ומקדם ההכחדה המולרי של הצבע באורך גל הספיגה המרבי, בהתאמה59. כאשר נמדד ב-280 ננומטר, יש לתקן את הספיגה האמיתית של החלבון על ידי הנוסחה הבאה:
חלבון = A 280-(A Dye· CF280)
כאשר CF280 הוא גורם התיקון עבור הצבע שצוין, בהתחשב בספיגה הפנימית שלו ב-280 ננומטר59.
הערה: בעוד שאנו מתמקדים בביטוי וטיהור של חלבון שעבר שינוי בציסטאין, יש לשקול היטב את ההחדרה האסטרטגית של שאריות ציסטאין לחלבון.
6. הכנת תא דגימה במיקרוסקופ
- הוסף 495 מיקרוליטר של מים טהורים במיוחד ו-5 מיקרוליטר (ריכוז סופי של 0.01% v/v) של Tween-20 (חומר פעילי שטח לא יוני) לבאר של מגלשת תא וערבב. דגירה מעל 30 דקות ב-RT.
- שטפו את החדר בעדינות במים טהורים במיוחד פעמיים וייבשו אותו.
הערה: החדר מוכן לשימוש לניסוי.
7. ניסוי אניזוטרופיה פלואורסצנטית של מולקולה אחת
- ראשית, כייל את מכשיר ה-smFA. קבע את פונקציית תגובת המכשיר, על ידי מדידת אור מפוזר של ראמאן או על ידי מדידת הקרינה של צבע בעל אורך חיים קצר וספקטרום פליטה מתאים (למשל, אריתרוזין או ירוק מלכיט) המרווה בנוסף על ידי תמיסת אשלגן יודיד רווי, KI.
- כדי לתקן פלואורסצנטיות ברקע, בצע מדידות של תמיסת חיץ. כייל את הרגישות היחסית של הגלאים המקבילים והניצבים (G-factor) על ידי רכישת נתונים מצבע המסתובב במהירות. כאן, 2 ננומטר רודמין 110 שימש לכיול הגלאים60.
- עבור ניסויי smFA על FoxP1 מונומרי, הוסף 100 pM BODIPY עם תווית FoxP1 ו-100 ננומטר ללא תווית FoxP1 ל-500 מיקרוליטר של מאגר סטנדרטי (20 מ"מ HEPES, 20 מ"מ NaCl, pH 7.8) בשקופית תא.
- לבדיקת טיטרציה עם DNA ו-FoxP1 מונומרי, הוסף 400 ננומטר DNA לתערובת חלבונים 100 pM עם תווית BODIPY/100 ננומטר ללא תווית ודגירה ב-RT למשך 10 דקות.
- עבור ניסויי smFA על FoxP1 דימרי, הוסף FoxP1 ללא תווית של 500 ננומטר ל-FoxP1 עם תווית BODIPY של 100 pM במאגר הסטנדרטי. לאחר מכן, דגרו בטמפרטורה של 37 מעלות צלזיוס למשך 30 דקות.
- לבדיקת טיטרציה עם DNA ודימרי FoxP1, הוסף 2000 ננומטר DNA לתערובת חלבונים 100 pM BODIPY / 500 ננומטר ללא תווית ודגירה ב-RT למשך 10 דקות.
- התחל את המדידות ובדוק בתוכנת BIFL שכמות ההתפרצויות ברשומה של 30 שניות היא בין 60-90. אם הדגימה מרוכזת יותר, יש לדלל אותה עד שתגיע לערך זה. שלב זה מבטיח תנאים של מולקולה אחת.
- התחל smFA בכל מדידה עבור FoxP1 מונומרי או FoxP1 דימרי בהיעדר או נוכחות של DNA למשך 4 שעות לפחות.
- נתח את מדידות ה-smFA כפי שנדון בסעיף 4.
תוצאות
אניזוטרופיה פלואורסצנטית נובעת מהכיוון היחסי של בליעת הפלואורופור ומומנטי דיפול הפליטה. כאשר פלואורופורים נחשפים לאור מקוטב, פלואורופורים עם מומנטי מעבר בליעה מיושרים עם וקטור השדה החשמלי של האור הפוגע נרגשים באופן מועדף (בחירת תמונות). כתוצאה מכך, אוכלוסיית המצב הנרגש הופכת למכוונת חלקית, כאשר לחלק ניכר מהמולקולות הנרגשות יש את רגעי המעבר שלהן מיושרים עם וקטור השדה החשמלי של האור המרגש המקוטב61. פלואורופורים מסתובבים עקב התנועה הבראונית שלהם. לפיכך, גם רגע מעבר הפליטה מסתובב, וכתוצאה מכך תלות בזמן באניזוטרופיה פלואורסצנטית. ניתן להשתמש באפקט זה כדי למדוד את תנועות הסיבוב של מולקולות פלואורסצנטיות, לזהות אירועי קשירה, לאפיין את סביבת הפלואורופור וללכוד דינמיקה מולקולרית.
ניסויים של מולקולה בודדת ערוכים באופן ייחודי לקבוע את ההטרוגניות של הדגימה. ניצול הרגישות של מולקולה בודדת ואניזוטרופיה פלואורסצנטית מוסיף מימד נוסף לספקטרוסקופיה פלואורסצנטית רב-פרמטרית. במיקרוסקופ קונפוקלי טיפוסי של מולקולה אחת (איור 2)20,21, ניתן לקבוע אניזוטרופיה פלואורסצנטית באמצעות מבוססת עוצמה או פתרון בזמן כאשר משתמשים בלייזרים פועמים.
כדי לשקול את ההשפעות הדפולריזציה של מטרת הצמצם המספרי הגבוה במיקרוסקופ קונפוקלי62, הצורה הנכונה של האניזוטרופיה שנפתרה בזמן35,63 ניתנת על ידי
 (1)
(1)
כאשר 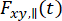 והם
והם 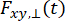 עוצמת הקרינה שנפתרה בזמן בערוץ הזיהוי y-th לאחר עירור באורך הגל x, עבור הקיטוב המקביל והניצב l1 ו-l 2 והם גורמים המתארים את הערבוב בין האותות המקבילים והניצבים עקב מטרת הצמצם המספרי הגבוה (NA) המשמשת במדידות אלה35,62, 64. הבדלים ביעילות הגילוי של תעלת הגילוי המקבילה,
עוצמת הקרינה שנפתרה בזמן בערוץ הזיהוי y-th לאחר עירור באורך הגל x, עבור הקיטוב המקביל והניצב l1 ו-l 2 והם גורמים המתארים את הערבוב בין האותות המקבילים והניצבים עקב מטרת הצמצם המספרי הגבוה (NA) המשמשת במדידות אלה35,62, 64. הבדלים ביעילות הגילוי של תעלת הגילוי המקבילה, 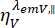 , והמאונכת,
, והמאונכת,  עבור הצבע מתוקנים עם היחס בין יעילות הגילוי,
עבור הצבע מתוקנים עם היחס בין יעילות הגילוי, 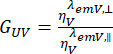 . ה-GUV מכונה גם גורם ה-G.
. ה-GUV מכונה גם גורם ה-G.
ניתן לעצב את האניזוטרופיה הקרינה שנפתרה בזמן באמצעות דעיכה רב-מעריכית כדי להסביר את הצמדת הפלואורופור לביו-מולקולה גדולה יותר כ
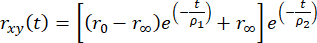 , (2)
, (2)
כאשר R0 היא אניזוטרופיה בסיסית תלויה בפלואורופור (בדרך כלל R0 = 0.38), R∞ היא האניזוטרופיה השיורית, ו-ρ1 ו-ρ2 הם זמני מתאם סיבוביים מהירים (תנועות מקומיות של הפלואורופור) ואיטיים (תנועה גלובלית של המקרומולקולה), בהתאמה.
במדידות אניזוטרופיה של מולקולה בודדת (איור 2), זמני הגעת הפוטונים נרשמים כדי לזהות פולטים בודדים באמצעות ניתוח אורך חיים פלואורסצנטי משולב פרץ (BIFL)33,35. זמני ההגעה בין הפוטונים (Δt) מוחלקים באמצעות ממוצע רץ ולאחר מכן משורטטים כדי לסייע בהדמיה. ההיסטוגרמה של זמנים אלה מצוידת בחצי גאוס כדי לקבוע את הממוצע וסטיית התקן של פוטונים שמקורם ברקע. סף שרירותי, המוגדר בכפולות של סטיית התקן, משמש לסינון אירועים בודדים תוך זיהוי הפוטונים הראשונים והאחרונים בכל פרץ. פוטונים בתוך כל פרץ משולבים לאחר מכן לניתוח נוסף, הכולל חישוב אניזוטרופיה פלואורסצנטית במצב יציב שנפתרה בזמן ומבוססת עוצמה באמצעות משוואות 1 ו-2 או באמצעות אומדן סבירות מקסימלית35. בשל המספר המוגבל של פוטונים באירועים של מולקולה בודדת, אומדן הסבירות המקסימלית מתייחס רק לרכיב מעריכי יחיד ולא יידון בהמשך.
בהיסטוגרמה דו-ממדית של אירועי מולקולה בודדת, ניתן לקשר את אורך החיים הקרינה הממוצע (τ) ואת האניזוטרופיה (rxy) על ידי משוואת פרין29,61 לקבלת (ρ) כזמן סיבוב ממוצע.
 (3)
(3)
ניתן להשיג ערכי ρ ספציפיים בוודאות גבוהה יותר על ידי ניתוח "תת-אנסמבל" (se) שבו הפוטונים של התפרצויות שונות משולבים בדעיכה אנזוטרופית פלואורסצנטית משולבת שנפתרה בזמן שניתן לנתח על ידי אופטימיזציה של פרמטרים של משוואה 2 לדעיכה הניסיונית (seTRFA). אניזוטרופיה שנפתרה בזמן יכולה לפתור הטרוגניות ודינמיקה הקשורות לתנועות סיבוביות (מקומיות וגלובליות) של הביו-מולקולות בתוך פליטת הקרינה המתרחשת במסגרת הזמן ns.
כדי לזהות דינמיקה בתוך אירועי מולקולה בודדת (בסולם תת-אלפית השנייה), הצגנו ניתוח שונות פרץ אנזוטרופיה (traBVA)57. ב-traBVA, עבור פרץ פוטון המכיל מקטעי פוטון עוקבים M, שונות האניזוטרופיה העודפת (s2) עבור התפרצויות היא
 . (4)
. (4)
עבור מצב אנזוטרופי יחיד, השונות σ2 נובעת אך ורק מרעש זריקה65 (sn: √N, כאשר N הוא מספר הפוטונים)
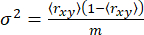 (5)
(5)
כאשר m הוא מספר הפוטונים בפרץ. לפיכך, כדי לזהות שונות נוספת באניזוטרופיה, אנו יכולים להגדיר את שונות האניזוטרופיה העודפת (S2) עקב הטרוגניות קונפורמציה כהבדל בין משוואות 4 ו-5.
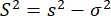 (6)
(6)
כדי ללכוד דינמיקה המתרחשת בתוך התבוננות במולקולות בודדות ולשקול את קירוב השונות, ניתן להשתמש בניתוח התפלגות פוטון אניזוטרופיה דינמית (daPDA)55,56. ב-daPDA, עוצמת הקרינה מעוצבת על ידי מעקב אחר הסתברות מותנית (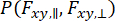 ) המבוטאת כהתפלגות בינומיאלית.
) המבוטאת כהתפלגות בינומיאלית.
 (7)
(7)
יחד, עם אומדן של קצב ספירת הרקע העוקב אחר התפלגות פואסון
 (8)
(8)
כאשר  הוא המספר הממוצע של פוטוני רקע לחלון זמן מוגדר. ניתן למדוד את ספירת הרקע המקבילה והניצבת,
הוא המספר הממוצע של פוטוני רקע לחלון זמן מוגדר. ניתן למדוד את ספירת הרקע המקבילה והניצבת,  ו
ו  - , באמצעות דגימות חיץ כהפניה. האניזוטרופיה הקרינה שנקבעה בניסוי מותאמת על ידי מזעור נתון הכשרון χ2 עם התפלגות עוצמת הקרינה לכל ערוץ קיטוב שיכול לכלול שינויים קינטיים.
- , באמצעות דגימות חיץ כהפניה. האניזוטרופיה הקרינה שנקבעה בניסוי מותאמת על ידי מזעור נתון הכשרון χ2 עם התפלגות עוצמת הקרינה לכל ערוץ קיטוב שיכול לכלול שינויים קינטיים.
שגרות הניתוח וייצוגי הנתונים המסופקים מציעים גישה מקיפה לפירוש הנתונים שנאספו. למרות שפרוטוקול זה מתמקד בעיקר במדידות קונפוקליות, המוגבלות בלכידת שינויים באניזוטרופיה מננו-שניות לאלפיות השנייה, ניתן לאמץ מיקרוסקופ השתקפות פנימית כוללת כדי לנטר אניזוטרופיה פלואורסצנטית על פני טווחי זמן ארוכים יותר, מה שמאפשר ניתוח סדרות זמן66. עבור מדידות קונפוקליות של מולקולה אחת, אנו מדגישים את השימוש בהיסטוגרמות רב-ממדיות היוצרות טביעת אצבע ייחודית של ההרכב הנצפה. דעיכות פלואורסצנטיות שנפתרו בזמן, משוחזרות מאוכלוסיות נבחרות, יכולות לעקוב אחר האבולוציה של אניזוטרופיה פלואורסצנטית בקנה מידה ננו-שניות (איור 3). ניתוח התפלגות פוטונים55,56 וניתוח שונות פרץ (BVA)57,58 יכולים גם ללכוד דינמיקה בטווחי זמן ביניים בין דעיכות שנפתרו בזמן והיסטוגרמות רב-ממדיות. בעוד שפרוטוקול זה אינו מכסה את השימוש בספקטרוסקופיה של מתאם פלואורסצנטי קיטוב (FCS), עם או בלי עירור פועם67,68, שיכול לגשר על טווחי הזמן של ננו-שנייה לאלפית השנייה, ניתן להשתמש באותם נתונים כדי לחשב FCS69, אם כי זה נופל מחוץ לתחום הפרוטוקול המוצג. אם מתבצעים ניסויים כאלה, מומלץ זמן מדידת דגימה ארוך יותר.
גישה זו יושמה על מערכת מורכבת כמו חלבוני FoxP אנושיים, ומספקת תובנות חשובות לגבי התנועות המעורבות במנגנון הפעולה שלהם. חלבוני FoxP הם גורמי שעתוק המעורבים במספר היבטים פיזיולוגיים כגון התפתחות המוח והריאות; חשוב לציין, מוטציות שונות הוכרו כפוגעות בתפקוד החלבונים הללו70,71. באמצעות תחום קשירת ה-DNA של FoxP1 כמודל, יצרנו מוטציות שונות של ציסטאין בודד כדי להציג צבע BODIPY-FL כגשש לתנועות (איור 4A). למעשה, הערכנו את השפעת הדימריזציה וקשירת ה-DNA כמווסתים מבניים עיקריים של חלבון זה. באמצעות גישת smFA, יצרנו עלילות 2D-smFA ויצרנו traBVA ו-daPDA עבור כל מוטציה בתנאים מונומריים ודימריים. אנו מראים דוגמה לאחד המוטאנטים הבודדים שנחקרו (איור 4). התנהגות האניזוטרופיה דומה בכל המוטאנטים מבחינת קביעת זמני מתאם סיבוביים גבוהים ונמוכים, ולכן הרכבים משוערים, לא מסודרים ומקופלים. ובכל זאת, הוא גם הטרוגני מאוד בכל המוטאנטים מבחינת השבר והקינטיקה של כל הרכב, מה שמעיד על שינויים שונים במעבר מסדר להפרעה המושפעים מהדימריזציה וקשירת ה-DNA, ומציג את התיאור ברזולוציה גבוהה של הדינמיקה המבנית לאורך השרשרת (איור 5).
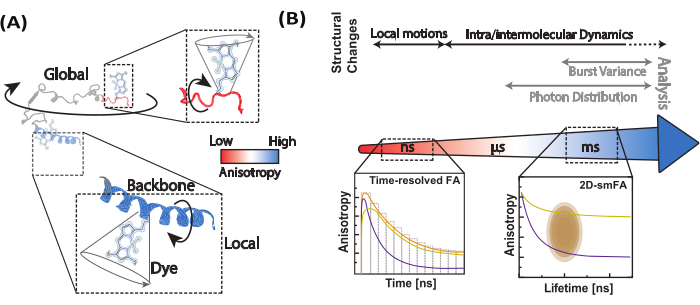
איור 1: טווח דינמי של ביומולקולות ושיטות אניזוטרופיה פלואורסצנטיות. (A) האניזוטרופיה של פלואורופורים קטנים הקשורים למיקומים שונים לאורך עמוד השדרה של הביו-מולקולה המעניינת מודיעה על דינמיקה מבנית מקומית. (B) טווחי זמן שנבדקו על ידי דעיכות בעוצמת הקרינה (אנאיזוטרופיה פלואורסצנטית שנפתרה בזמן, FA) והיסטוגרמות של מולקולה בודדת של נתוני מיקרוסקופ קונפוקלי של מולקולה אחת. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2: רישום ועיבוד נתוני אניזוטרופיה פלואורסצנטית של מולקולה אחת. (A) מולקולות מפוזרות באופן חופשי מנותחות באמצעות מיקרוסקופ קונפוקלי חד-מולקולי שמצויד בלייזר עירור מקוטב ליניארי יחיד (כחול במקרה שלנו). פליטת פלואורסצנטיות (ירוק במקרה שלנו) מזוהה על ידי שני גלאים לאחר שמקטב קרן מפצל את האות לשני קיטובים (מקביל,  , וניצב,
, וניצב,  למקור העירור). (B) כל פוטון שזוהה מאופיין בשלושה פרמטרים: זמן מיקרו, זמן מאקרו וסוג ערוץ. הנתונים מאוחסנים בתבנית Time-Tagged Time-Resolved (TTTR)72. (C) התפרצויות של מולקולות בודדות נבחרות ומעובדות כדי לחלץ פרמטרים פלואורסצנטיים, כולל אניזוטרופיה פלואורסצנטית עבור כל מולקולה שנצפתה. (D) הנתונים מיוצגים במספר דרכים, כולל תרשימים דו-ממדיים של אניזוטרופיה פלואורסצנטית לעומת אורך חיים פלואורסצנטי ודעיכת אניזוטרופיה שנפתרה בזמן. ייצוגים אלה מאפשרים קביעה חזותית וכמותית של אורך החיים הקרינה, זמני מתאם סיבובי והטרוגניות של המערכת. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
למקור העירור). (B) כל פוטון שזוהה מאופיין בשלושה פרמטרים: זמן מיקרו, זמן מאקרו וסוג ערוץ. הנתונים מאוחסנים בתבנית Time-Tagged Time-Resolved (TTTR)72. (C) התפרצויות של מולקולות בודדות נבחרות ומעובדות כדי לחלץ פרמטרים פלואורסצנטיים, כולל אניזוטרופיה פלואורסצנטית עבור כל מולקולה שנצפתה. (D) הנתונים מיוצגים במספר דרכים, כולל תרשימים דו-ממדיים של אניזוטרופיה פלואורסצנטית לעומת אורך חיים פלואורסצנטי ודעיכת אניזוטרופיה שנפתרה בזמן. ייצוגים אלה מאפשרים קביעה חזותית וכמותית של אורך החיים הקרינה, זמני מתאם סיבובי והטרוגניות של המערכת. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
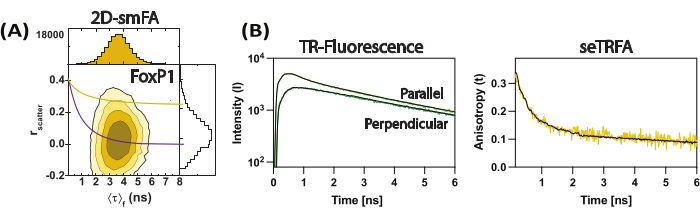
איור 3: נתונים מייצגים עבור דימר החלפת דומיין FoxP1. (A) מתאם של האניזוטרופיה הקרינה (פיזור r) כנגד אורך החיים הקרינה הממוצע לכל מולקולה כעלילת קווי מתאר. שכבת-על של משוואת פרין בודדת עבור שני רכיבי סיבוב כמייצגת את ממוצע האנסמבל של המולקולה, בהתחשב ב-ρ1 ו-ρ 2 של 0.2 ns ו-8.5 ns, בהתאמה. (B) דעיכות פלואורסצנטיות שנפתרו בזמן של תת-אנסמבל משמשות לחישוב האניזוטרופיה הקרינה שנפתרה בזמן של הדגימה. התאמה למשוואה 2 פתרה את המרכיבים המקומיים והגלובליים של אניזוטרופיה פלואורסצנטית. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 4: דינמיקת FoxP1 תת-אלפית שנייה מנוטרת באמצעות אניזוטרופיה פלואורסצנטית של מולקולה אחת (smFA). (A) ייצוג מצויר של מבנה FoxP1 המונומרי. (B) היסטוגרמה דו-ממדית מדגימה הטרוגניות דינמית, וחושפת שני זמני מתאם סיבוביים נפרדים שזוהו באמצעות אניזוטרופיה פלואורסצנטית שנפתרה בזמן. אנזוטרופיה שנפתרה בזמן ניתוח שונות פרץ (traBVA) חושף תת-קבוצה קטנה של אירועים עם שונות עודפת (Eq. 6) המציגים אניזוטרופיה גדולה. ניתוח אנזוטרופיה דינמית כמותית באמצעות ניתוח התפלגות פוטונים (PDA) מחלץ עוד יותר את שערי החליפין לתהליך זה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 5: סינון התנועות המקומיות והגלובליות של FoxP1 במהלך דימריזציה. (A) ייצוג מצויר משווה את ה-FoxP1 המונומרי לצורתו הדימרית. (B) מוצגת שונות עודפת ממוצעת למיקום בתנאים מונומריים ודימריים, כאשר שונות עודפת גדולה יותר מצביעה על שינויים משמעותיים יותר באניזוטרופיה. (C) ניתוח אניזוטרופיה דינמית באמצעות ניתוח התפלגות פוטונים (PDA) מסייע לקבוע שברי אוכלוסייה (אניזוטרופיה גבוהה בצבעים כהים ואניזוטרופיה נמוכה בצבע בהיר) בהיעדר (ירוק) ונוכחות (צהוב) של DNA. בגישה זו, שיעורים (לא מוצגים) הוערכו עבור מעברים בין התנהגויות מקומיות וגלובליות, וחשפו כי FoxP1 עובר התפתחות חלקית. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
עבור ניסויי אניזוטרופיה פלואורסצנטיים של מולקולה אחת, חיוני לשקול היטב את התכונות הפוטופיזיקליות של הפלואורופור הנבחר. מאפיינים אלה כוללים את אורך גל הפליטה, שחייב להתיישר עם מערכת הגילוי, ואורך גל העירור, שאמור להיות תואם ללייזרים הפועמים הזמינים. כדי לייעל את הטווח הדינמי, הפלואורופור צריך להיות בעל אורך חיים פלואורסצנטי ארוך ביחס לזמן הדיפוזיה הסיבובית של המולקולה. זה קריטי למעקב אחר דינמיקת סיבוב וקישור/כיוון של הדיפול של הפלואורופור ביחס לביו-מולקולה המעניינת. בנוסף, בהירות, יציבות פוטו-יציבות ותפוקה קוונטית חיוניים להפקת אותות חזקים עם יחס אות לרעש יציב. מסיבות אלה, BODIPY-FL נבחר כפלואורופור במספר מחקרים 39,40,42.
סינון הדינמיקה של עמוד השדרה של ביומולקולות דורש לעתים קרובות תיוג חלבון, המושג בדרך כלל באמצעות תיוג ספציפי לאתר. זה נעשה בדרך כלל על ידי הכנסת שאריות לשינוי כימי ממוקד. הגישה הנפוצה ביותר היא הכנסת ציסטאין למיקומים מעניינים, שם ניתן לשנות את שרשראות הצד של התיול שלהם באופן סלקטיבי עם ריאגנטים כגון מלימידים או יודואצטמידים. פחות נפוץ, הלידים בנזיליים וקטונים ברומו-מתיל משמשים ליצירת קשרי תיואתר. ניתן גם למקד שרשראות צד אחרות של חומצות אמינו, אך השפע שלהן בחלבונים פחות נפוץ. עם זאת,ניתן להשתמש גם בגישות אלטרנטיביות, כמו חומצות אמינו לא טבעיות. בחירת אתר נכונה לתיוג היא חיונית כדי למזער את ההפרעה לביו-מולקולה הנחקרת, ויש לקיים בקרות מתאימות. לדוגמה, אם המולקולה המסומנת משמשת במבחני קשירה, שיטות משלימות ללא תווית צריכות לוודא שהפלואורופורים אינם משפיעים על זיקת הקשירה.
לאחר זיהוי הדגימה המתאימה ויישום אסטרטגיית התיוג האופטימלית, השלב הבא הוא לוודא שהמיקרוסקופ הקונפוקלי מיושר ומכויל כראוי לניסויים של מולקולה בודדת. הפרוטוקול מתאר כיצד לקבוע את הגורם הנדרש להמשך ניתוח. לאחר כיול המכשיר, השלב הבא הוא למדוד את הדגימה ולעבד את הנתונים כדי לחלץ כמה שיותר מידע מהפוטונים שזוהו. הפרמטרים המרכזיים, כגון מיקרו-זמן, זמן מאקרו וסוג ערוץ, כפי שמוצג באיור 2, יכולים לשמש להמשך ניתוח והדמיה באמצעות אלקטרוניקה TCSPC טיפוסית.
ניתן להשתמש בהתקדמות האחרונה בספקטרוסקופיה פלואורסצנטית של מולקולה בודדת כדי לחקור מידע מבני מהרכבים הטרוגניים של ביומולקולות. עם זאת, מעט יחסית מחקרים ממנפים את התובנות שמספקת אניזוטרופיה פלואורסצנטית, ונדרש מודל חלבון שלם כדי לגזור את הדינמיקה המבנית של ביומולקולות. לכן, חשיפת הדינמיקה של אינטראקציות בין-דומיינים וחלבון-חלבון של מספר גורמי שעתוק היא מאתגרת.
לסיכום, ניסויי אניזוטרופיה פלואורסצנטיים של מולקולה אחת מציעים מידע משלים על התנועות המקומיות והגלובליות של עמוד השדרה הביו-מולקולרי, שהם קריטיים להבנת תפקודו.
Disclosures
כל המחברים מצהירים כי אין להם אינטרסים כלכליים מתחרים עם תוכן מאמר זה.
Acknowledgements
עבודה זו נתמכה על ידי מענקי FONDECYT 11200729 ו-FONDEQUIP EQM200202 ל-E.M., NIH R15CA280699 R01GM151334 ו-NSF CAREER MCB 1749778 פרסים ל-HS. NK הכירה בתמיכה מתוכנית המלגות לפוסט-דוקטורט של אוניברסיטת קלמסון.
Materials
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
References
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. . Principles of Protein X-Ray Crystallography. , (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701 (2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. a. M. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. a. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676 (2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854 (2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724 (2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. . Fluorescence Correlation Spectroscopy: Theory and Applications. , (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375 (2020).
- Takahashi, H., Takahashi, K., Liu, F. -. C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302 (2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623 (2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116 (2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1 (2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077 (2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004 (2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -. J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -. O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. . Time tagged time-resolved fluorescence data collection in life sciences. , (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ISSN 2578-2037
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.
