Method Article
Rekombinaz polimeraz amplifikasyonu/Cas12a tabanlı bir sistem kullanılarak Acinetobacter baumannii enfeksiyonlarının hızlı ve spesifik tespiti
Bu Makalede
Özet
Acinetobacter baumannii'nin hızlı ve hassas tespitini kolaylaştırmak için, A. baumannii enfeksiyonlarını tanımlamak için LbaCas12a endonükleaz ile birlikte Rekombinaz Polimeraz Amplifikasyonu (RPA) kullanan bir protokol sunuyoruz.
Özet
Gram negatif bir bakteri olan Acinetobacter baumannii, yüksek ölüm oranlarına sahip ciddi enfeksiyonlara neden olmasıyla ünlüdür. A. baumannii'nin hızlı ve doğru tespiti, hızlı tedavi, etkili enfeksiyon kontrolü ve antibiyotik direncinin azaltılması için çok önemlidir. Bununla birlikte, A. baumannii'nin yerinde hızlı ve kolay tespiti için uygun bir yöntem yoktur. DNA Endonükleaz Hedefli CRISPR Trans Raportör (DETECTR) sistemi, Cas12a'nın hedefe özgü tanıma yeteneklerini Rekombinaz Polimeraz Amplifikasyonunun (RPA) izotermal amplifikasyon verimliliği ile entegre ederek A. baumannii tespitine hızlı, kesin ve hassas bir yaklaşım sunar. Bu protokol, LbaCas12a endonükleaz ile birlikte RPA kullanılarak A. baumannii'nin tespitini detaylandırır. Bu makalede aşağıdaki adımlar açıklanmaktadır: DNA'nın ekstraksiyonu, belirli bir DNA dizisinin seçimi, primer ve CRISPR RNA (crRNA) tasarımı, pozitif rekombinant plazmidin yapımı, Cas12a-RPA testinin kurulumu, RPA amplifikasyon sisteminin optimizasyonu, gerçek zamanlı PCR cihazı gibi bir floresan tespit aracı kullanılarak RPA-CRISPR/Cas12a testinin görselleştirilmesi, ve duyarlılık ve özgüllük değerlendirmesinin değerlendirilmesi.
Giriş
Klinik mikrobiyolojide, Acinetobacter baumannii enfeksiyonlarının saptanması önemli bir zorluk teşkil etmektedir. Bu gram negatif bakteri, özellikle bağışıklığı baskılanmış hastalarda yüksek ölüm oranlarında bile ciddi klinik semptomlarla enfeksiyonlara neden olabilir1. Patojen enfeksiyonları için geleneksel kültür tabanlı tespit yöntemleri zaman alıcıdır ve duyarlılıktan yoksun olabilir, bu da antibiyotik tedavisini geciktirebilir ve hasta sonuçlarını tehlikeye atabilir. A. baumannii'nin hızlı ve doğru bir şekilde tanımlanması, etkili tedavi ve salgın kontrolü için çok önemlidir. Bu ihtiyacı karşılamak için nükleik asit amplifikasyonunu kullanan moleküler teknikler araştırılmıştır. Bununla birlikte, bu yaklaşımlar genellikle gelişmiş termal döngü ekipmanına ihtiyaç duyar ve iyi eğitimli teknisyenler ve iyi kurulmuş laboratuvarlar tarafından sınırlandırılabilir. Bu zorlukların üstesinden gelmek için, araştırmalar giderek daha fazla izotermal amplifikasyon yöntemlerigeliştirmeye odaklanmaktadır 2,3,4.
Rekombinaz Polimeraz Amplifikasyonu (RPA), Piepenburg ve ark. 5 tarafından oluşturulan ve polimeraz zincir reaksiyonuna (PCR) benzer şekilde ancak sıcaklık döngüsüne ihtiyaç duymadan DNA'yı amplifiye etmek için kullanılan bir yöntemdir. Bu yöntem 50 mM Tris (pH 7.5), 100 mM potasyum asetat, 14 mM magnezyum asetat, 2 mM DTT, %5 PEG20000 (yüksek moleküler ağırlıklı polietilen glikol), 200 μM dNTP, 3 mM ATP, 50 mM fosfokreatin, 100 μg/mL kreatin kinaz, 120 μg/mL UvsX, 30 μg/mL UvsY, 900 μg/mL Gp32, 30 μg/mL Bsu LF, 450 nM primerler ve DNA şablonları. Amplifikasyon işlemi, rekombinaz proteini UvsX'in 3 mM ATP varlığında primerlere bağlanması ve %5 PEG20000 bir rekombinaz-primer kompleksi oluşturmasıyla başlar. Bu kompleks daha sonra primerlerin çift sarmallı DNA üzerindeki homolog dizilerle kombinasyonunu kolaylaştırır.
UvsY'nin yardımıyla, rekombinaz UvsX, astar ve şablon ipliği arasındaki alışverişi kolaylaştırır ve hedef DNA'nın bir zincirinin yer değiştirmesine neden olur. Gp32, tek sarmallı DNA yapısının korunmasına yardımcı olur. Son olarak, rekombinaz ayrışır ve DNA ipliklerinin yerini alabilen bir DNA polimeraz (Bsu LF), primerin 3' ucuna bağlanır ve onu deoksiribonükleosit trifosfatların (dNTP'ler) varlığında uzatır. Bu işlem döngüsel olarak tekrarlanır ve üstel amplifikasyon sağlanır. Tüm amplifikasyon işlemleri 20-40 dakika içinde ve 37 °C ile 42 °C arasındaki nispeten sabit sıcaklıklarda tamamlanabilir. Bu sıcaklık aralığı, RPA'nın minimalist bir ortamda yürütülmesine izin veren fizyolojik sıcaklıklarla yaklaşık olarak aynıdır ve RPA'yı DNA tespiti ve analizi için çok yönlü ve verimli bir araç haline getirir.
Kümelenmiş düzenli aralıklı kısa palindromik tekrarlar (CRISPR) ve CRISPR ile ilişkili protein (CRISPR-Cas) sistemi, bakteri ve arkelerde Sınıf I ve Sınıf II sistemleri kapsayan adaptif bir bağışıklık mekanizması olarak işlev görür. Sınıf II, CRISPR RNA (crRNA) tarafından yönlendirilen hedef DNA veya RNA'yı tanımlayan ve parçalayan Cas12 (a, b, f), Cas13 (a, b) ve Cas14 gibi proteinleri içerir6,7,8 . LbaCas12a (veya Cpf1), crRNA güdümlü bir DNA endonükleazdır. crRNA, Cas proteinleri ile kompleks oluşturduğu CRISPR-Cas sistemi içinde yol gösterici bir RNA görevi görür. Hedef DNA ile eşleşmek için aralayıcı bölgesini kullanır ve protein kompleksini hedef dizilere etkili bir şekilde yönlendirir. 5'-UAAUUUCUACUAAGUGUAGAU-3' dizisi, tüm LbaCas12a crRNA'larında sabit bir element olan korunmuş crRNA tekrarı olarak hizmet eder. Bu tekrarı takiben, amaçlanan DNA hedefine göre farklılık gösteren hedefe özgü bir segment gelir. Bu hedefleme dizisinin uzunluğu 18 ila 24 nükleotid arasında değişir.
PAM (Protospacer-Bitişik Motif) dizisi (V, A, C veya G olabilen TTTV), DNA hedefinin tamamlayıcı olmayan zincirinin 5' ucunda bulunur. Cas12a, PAM dizisinde hedef çift sarmallı DNA'yı keser. Daha sonra, aktive edilmiş Cas proteinleri, tek sarmallı DNA'nın (ssDNA) spesifik olmayan trans-bölünmesini gerçekleştirir9,10,11,12. Bu nedenle, bir florofor ve söndürücü ile etiketlenmiş ssDNA, bölünmeye uğrar ve bu da teminat bölünmesini takiben bir floresan sinyalinin yayılmasına neden olur 8,13. Floresan yoğunluğu, hassas nükleik asit tespiti için bir floresan okuyucu kullanılarak ölçülebilir14. Özellikle, Cas12a, Protospacer-Bitişik Motifin (PAM) tanınmasına bağlı olarak hedef dizilerin bağlanması ve bölünmesi ile DNA analizi için yaygın olarak kullanılırken, Cas13a böyle bir gereklilikten bağımsız olarak çalışır.
CRISPR-Cas sistemleri 15,16,17, tek bir reaksiyonda bölünmeyi kolaylaştırır 18,19,20,21. Yukarıdaki ikisini birleştirmek, nükleik asit tespiti için A. baumannii-DETECTR (LbaCas12a-Hedeflenen A. baumannii'nin Etkin Tespiti) olarak bilinen sağlam bir araç oluşturabilir 22,23,24. Bu protokol, A. baumannii'nin 16s rDNA geni içindeki bir crRNA kılavuzlu diziyi spesifik olarak hedeflemek ve parçalamak için Cas12a'nın DNaz aktivitesi ile birlikte RPA'nın kullanımını gösterir, böylece patojenin hassas ve hassas bir şekilde tespit edilmesini sağlar.
A. baumannii-DETECTR yöntemi, geleneksel tespit yöntemlerine göre çeşitli avantajlar sunar25,26. İlk olarak, RPA'nın izotermal özelliği, termal döngüleyiciden bağımsız amplifikasyon mekanizması5 aracılığıyla operasyonel prosedürleri kolaylaştırır ve maliyetleri düşürür. İkinci olarak, Cas12a endonükleaz, crRNA aracılı hedefleme ve Watson-Crick baz eşleşmesi7 yoluyla testin özgüllüğünü arttırır. Son olarak, A. baumannii-DETECTR'in tek tüp yönteminin kullanılması sadece zaman kazandırmakla kalmaz, aynı zamanda amplikon kontaminasyonu riskini de önemli ölçüde azaltır19.
Protokol, DNA ekstraksiyonunun ekstraksiyonu, hedef DNA dizilerinin seçimi, primerlerin ve crRNA'ların tasarımı, pozitif rekombinant plazmitlerin inşası, Cas12a-RPA testinin kurulması, RPA amplifikasyonunun optimize edilmesinin optimizasyonu ve Gerçek zamanlı PCR makinesi gibi floresan algılama cihazları araçları kullanılarak sonuçların görselleştirilmesi, duyarlılık ve özgüllük değerlendirmeleri ile tamamlayın.
A. baumannii-DETECTR yöntemi, zamanında ve etkili tedaviyi, sağlam enfeksiyon kontrolünü ve antibiyotik direncinin yayılmasını kontrol altına alarak hasta sonuçlarını iyileştirmede umut vaat etmektedir. Bu protokol, Cas12a-RPA teknolojisinin uygulanması için kapsamlı bir kılavuz görevi görebilir ve çeşitli sağlık hizmeti ortamlarında faydasını artırabilir. Bununla birlikte, tutarlı ve güvenilir sonuçlar elde etmek için her adımın belirli laboratuvar koşullarına ve numune çeşitliliğine göre optimize edilmesi gerektiğine dikkat etmek çok önemlidir.
Protokol
1. A. baumannii -DETECTR'ın inşası
NOT: A. baumannii-DETECTR'in yapımı, primer ve crRNA tasarımını, pozitif rekombinant plazmit yapısının inşasını, reaksiyon çözeltilerinin hazırlanmasını, RPA ile izotermal DNA amplifikasyonunu ve RPA-CRISPR/Cas12a testinin görselleştirilmesini içeren dört aşamalı bir süreçtir. DETECTR testinin şeması Şekil 1'de gösterilmiştir.
- Primer ve crRNA tasarımı (Ek Dosya 1'e bakınız)
- Astar tasarım ilkeleri temelinde tasarım araçlarını kullanarak 12 çift astar tasarlayın. On iki çift primer (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R3, F2R1, F2R2, F2R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3) oluşturmak için dört ileri astar (F0-F3) ve üç ters astar (R1-R3) elde edin.
- Primerin 5' ucunun, sürekli bir G gerilmesi yerine ilk 3-5 nükleotid içinde C veya T bazları içerdiğinden emin olun. Ek olarak, amplifikasyonu artırmak için 3' ucundaki son üç nükleotidin ideal olarak G ve C bazları olduğundan emin olun.
- Saç tokası oluşumunu önlemek için kendi kendini tamamlayan diziler olmayan ve aralarında dörtten fazla tamamlayıcı veya homolog baz olmayan astarlar tasarlayın. Astar dimerizasyonunu önlemek için 3' uç tamamlayıcılığından uzak durun.
- Primerlerin etkinliğini ve özgüllüğünü değerlendirmek için Ulusal Biyoteknoloji Bilgi Merkezi'nin (NCBI) Primer-BLAST aracını kullanarak her bir primer çiftini özgüllük açısından değerlendirin.
NOT: Primer-BLAST, tasarlanan primerlerle eşleşen dizileri bulmak için NCBI veritabanını kullanabilir ve kullanıcıların primerlerin etkinliğini ve özgüllüğünü değerlendirmesine yardımcı olabilir. - Astar taramasının ardından, sabit dizi olarak LbaCas12a crRNA iskele dizisini (5'-UAAUUUCUACUAAGUGUAGAU-3') kullanarak CRISPR RNA'yı (crRNA) veya kılavuz RNA'yı (gRNA) tasarlayın. Hedefe özel bir segment (5'-GUUAAUACCUAGAGAUAGUG-3') dahil edin, PAM motifinin tamamlayıcı olmayan telin 5' ucunda yer aldığından emin olun.
NOT: Tüm oligonükleotidlerin ve crRNA'nın dizileri Tablo 1'de sunulmuştur.
- Astar tasarım ilkeleri temelinde tasarım araçlarını kullanarak 12 çift astar tasarlayın. On iki çift primer (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R3, F2R1, F2R2, F2R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3) oluşturmak için dört ileri astar (F0-F3) ve üç ters astar (R1-R3) elde edin.
- Pozitif rekombinant plazmit yapısı
- A . baumannii 16s rDNA-F (ileri primer) ve A. baumannii 16s rDNA-R (ters primer) primer setini kullanarak 16s rDNA segmentini amplifiye edin.
- 1 μL 10 μM F ve 10 μM R, 10 μL 5x HızlıpFu Tamponu, 1 μL HızlıpFu Polimeraz, 4 μL 2.5 mM dNTP, 1 μL 10 ng/μL A. baumannii genomik DNA ve 32 μL Nükleaz İçermeyen Su 50 μL PCR sistemine ekleyin.
- PCR döngü koşullarını aşağıdaki gibi ayarlayın: 5 dakika boyunca 95 ° C'de ilk denatürasyon, ardından 30 saniye boyunca 95 ° C'de 33 döngü denatürasyon, 55 ° C'de 30 saniye tavlama ve 72 ° C'de 1 dakika ve 20 saniye uzatma. Son olarak, son uzatma için 72°C'de 10 dakika gerçekleştirin.
- Pozitif PCR ürününü saflaştırmak için üreticinin talimatlarına göre bir DNA Saflaştırma Kiti kullanın. Daha sonra, saflaştırılmış DNA'yı BamHI ve SalI enzimleri ile sindirilmiş olan pUC57 vektörüne klonlayın.
NOT: Bu işlem, korunmuş bir bölge içeren pozitif bir rekombinant plazmitin oluşturulmasıyla sonuçlanacaktır. - Pozitif rekombinant plazmiti, bir kopya / μL ila 107 kopya / μL'lik bir konsantrasyon elde etmek için sterilize edilmiş ddH2O ile on kat artışlarla seri olarak seyreltin. Sonraki deneyler için seyreltilmiş plazmidi -20 °C'de saklayın.
- A . baumannii 16s rDNA-F (ileri primer) ve A. baumannii 16s rDNA-R (ters primer) primer setini kullanarak 16s rDNA segmentini amplifiye edin.
- Çözelti hazırlama
- Çözelti A'yı aşağıdaki gibi hazırlayın: her reaksiyon için 29.5 μL Primer Serbest Rehidrasyon tamponu, 2.4 μL 10 μM A. baumannii-F ve 10 μM A. baumannii-R ve 12.2μL ddH2O ekleyerek 46.5 μL'lik bir nihai hacim elde edin. Ardından, reaksiyon karışımını RPA Kitinden enzim reaksiyon tüplerine ekleyin. Karıştırmak için pipetleyin.
NOT: A. baumannii-F ve R'nin primerleri, A. baumannii genini hedef alan testte kullanılır. Sıra için lütfen Tablo 1'e bakın. - Çözelti B'yi aşağıdaki gibi hazırlayın: her reaksiyon için 0.24 μL 10 μM ssDNA raporlayıcı, 2 μL 10x Cas12a Reaksiyon Tamponu, 1 μL 1 μM crRNA, 1 μL 1 μM Cas12a ve 10.76μL ddH2Oekleyin 15 μL'lik bir nihai hacim elde etmek için.
NOT: Sondalar (ssDNA muhabiri) satın alındı ve 5' ucunda bir 5-Karboksifloresein (FAM) florofor ve 3' ucunda bir Kara Delik Söndürücü 1 (BHQ1) söndürücü ile etiketlenmiş tek sarmallı bir DNA raportöründen oluşuyordu. Tespit edilmesi gereken çok sayıda numune olması durumunda, önemli miktarda çözelti B hazırlayın ve ardından numuneleri daha sonra alikotlara bölün.
- Çözelti A'yı aşağıdaki gibi hazırlayın: her reaksiyon için 29.5 μL Primer Serbest Rehidrasyon tamponu, 2.4 μL 10 μM A. baumannii-F ve 10 μM A. baumannii-R ve 12.2μL ddH2O ekleyerek 46.5 μL'lik bir nihai hacim elde edin. Ardından, reaksiyon karışımını RPA Kitinden enzim reaksiyon tüplerine ekleyin. Karıştırmak için pipetleyin.
- İzotermal DNA amplifikasyonu (RPA)
- Farklı primer çiftleri ile çözelti A'ya 107 kopya / μL'lik 1 μL pozitif rekombinant plazmit ekleyin; İyice karıştırın. Daha sonra çözeltiye 2,5 μL 280 mM Magnezyum Asetat (MgOAc) ekleyin ve iyice karıştırın.
NOT: MgOAc eklenir eklenmez RPA reaksiyonları başlar. - 39 °C'de 20 dakika inkübe edin. 20 dakika sonra, bir saflaştırma kiti ile RPA kullanarak farklı primerlerle amplifiye edilmiş ürünleri saflaştırın ve ardından en iyi primer çifti olarak en parlak ve en geniş bandı seçmek için temiz amplikonları 120 V'ta %1'lik bir agaroz jel üzerinde 10 dakika boyunca çalıştırın.
NOT: Düşük şablon kopya sayısı için, 4 dakika sonra EP tüpünü, vorteksini çıkarın ve kısa bir süre döndürün; Ardından 16 dakika daha ısıtma cihazında değiştirin. Amplifikasyondan sonra, çalışma yüzeylerinin amplikonlarla kirlenmesini önlemek için tüpler çok dikkatli bir şekilde açılmalıdır. Bu önlem, sonraki deneysel prosedürlerde yanlış pozitif sonuçlara neden olma riskini azaltacaktır.
- Farklı primer çiftleri ile çözelti A'ya 107 kopya / μL'lik 1 μL pozitif rekombinant plazmit ekleyin; İyice karıştırın. Daha sonra çözeltiye 2,5 μL 280 mM Magnezyum Asetat (MgOAc) ekleyin ve iyice karıştırın.
- RPA amplifikasyon sisteminin optimizasyonu
NOT: Önceki adımda seçilen optimal primer çifti F3R3, optimal primer konsantrasyonunu ve süresini taramak için RPA amplifikasyon reaksiyonu için farklı nihai konsantrasyonlara ayarlanmıştır. NC, karşılık gelen primer konsantrasyonu için boş bir kontroldür.- Çözelti A'daki diğer bileşenlerin konsantrasyonlarını sabit ve toplam hacmi 46.5 μL'de koruyun. 0.40 μM (2.0 μL), 0.44 μM (2.2 μL), 0.48 μM (2.4 μL) ve 0.52 μM (2.6 μL) 'de 10 μM F3 ve 10 μM R3 nihai primer konsantrasyonlarını elde etmek için RNaz içermeyen ddH2Ohacmini buna göre ayarlayın, sırasıyla.
- 39 °C'de farklı sürelerde inkübe edin: 10 dk, 20 dk, 30 dk.
- RPA ürünlerini bir DNA Saflaştırma Kiti kullanarak temizleyin ve bantların özelliklerini analiz ederek optimum primer konsantrasyonunu ve inkübasyon sürelerini belirlemek için 120 V'ta %1'lik bir agaroz jel üzerinde 10 dakika boyunca elektroforez gerçekleştirin.
- RPA-CRISPR/Cas12a testinin görselleştirilmesi
- 5 μL RPA ürününü B çözeltisine ekleyin ve homojen olana kadar iyice karıştırın.
- Numuneleri floresan kantitatif PCR cihazına yerleştirin, FAM kanalını ayarlayın ve değerleri 37 °C'de 60 dakika, 60x boyunca toplam 60 dakika boyunca her dakika okuyun.
NOT: Yeşil floresan sinyali birkaç gün sürebilir. Sistemin etkili bir şekilde çalıştığından emin olmak için, DNA ve RNaz içermeyen bir test yapmak daha iyidir.
2. RPA-CRISPR/Cas12a tabanlı tespit platformunun özgüllük değerlendirmesi
NOT: A. baumannii-DETECTR'in özgüllüğünü test etmek için A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Cockerella burnetii, Serratia'nın nükleik asitleri ve insan örnekleri DETECTR testine tabi tutulmuştur.
- Referans verilen DNA Kitini kullanarak veya bakterileri suda kaynatarak izole ederek çeşitli türlerden elde edilen DNA şablonlarını kullanın. Tüm şablonların eşit konsantrasyonlara sahip olduğundan emin olun. Negatif kontrol olarak su kullanın.
- F3R3 primerleri içeren Çözelti A'ya 10 ng veya 1 μL DNA şablonu ekleyin. 2,5 μL MgOAc ekleyin, iyice karıştırın ve karışımı 39 °C'de 20 dakika inkübe edin.
- 20 dakika sonra, önceki adımdaki üründen 5 μL ürünü çözelti B'ye ekleyin ve karıştırın.
- Floresan kantitatif PCR cihazına yerleştirin, kanalı FAM olarak ayarlayın ve değeri 37 °C'de dakikada bir kez, 60 dakika boyunca toplam 60 x boyunca okuyun.
3. RPA-CRISPR/Cas12a tabanlı algılama platformunun duyarlılık değerlendirmesi
- Reaksiyon için şablon olarak 1-107 kopya / μL'lik bir konsantrasyonda hazırlanmış pozitif bir rekombinant plazmit kullanın. Negatif kontrol için su kullanın.
- 2.2-2.4 adımlarını izleyerek yöntemi gerçekleştirin.
NOT: RPA-F3 ve RPA-R3 primer seti, pozitif rekombinant plazmitten 174 baz çiftinden oluşan daha kısa bir parçayı amplifiye etmek için tasarlanmıştır.
4. Numune UV ışığı ve LFS tespiti altındayken yapılan hazırlıklar
- Gerekli sayıda test şeridini çıkarın ve uygun şekilde etiketleyin.
- Amplifiye edilmiş ürünlerin 10 μL'sini sırasıyla 2. ve 3. adımlardan ayrı PCR tüplerine aktarın. Ardından, her tüpe 40 μL ddH2O ekleyin.
NOT: Kendi kendine hazırlanmış bir reaksiyon sistemi kullanılıyorsa, optimum seyreltme oranının araştırılması önerilir. - Testi gerçekleştirin.
- Floresan varlığını değerlendirmek için güçlendirilmiş ürünleri içeren PCR tüpünü bir transilluminatöre tabi tutun.
- Test şeritlerini, numune pedi ucu aşağı bakacak şekilde PCR tüplerine yerleştirin.
- Sonucu okumak için oda sıcaklığında 5-10 dakika bekletin. Bu süreden sonra, devam etmeden önce sıvı seviyesinin maksimum çizgiyi aşmadığından emin olun.
- T çizgisi renk göstermiyorsa sonuçları negatif olarak yorumlayın. T çizgisi çıplak gözle görülebiliyorsa, nükleik asit probunun Cas enzimi tarafından parçalandığını ve Cas enziminin aktive edildiğini gösteren sonuçları pozitif olarak yorumlayın. Ne C çizgisi ne de T çizgisi renk göstermiyorsa sonucun geçersiz olduğunu düşünün.
Sonuçlar
Bu çalışmada, A. baumannii'nin hızlı ve güvenilir saha tanımlaması için RPA ve CRISPR-Cas12a sisteminin izotermal amplifikasyon verimliliğini entegre eden A. baumannii-DETECTR adlı yeni, taşınabilir bir tanı platformu sunuyoruz. DETECTR testinin şeması Şekil 1'de gösterilmiştir.
On iki çift astar, tasarım araçları kullanılarak tasarlanmıştır.astar tasarım ilkeleri temelinde. Dört ileri primer (F0-F3) ve üç ters primer (R1-R3) elde edildi, böylece 12 çift primer (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3). 20 dakika boyunca 39 ° C'de RPA amplifikasyonunun ardından, RPA PCR ürünleri, optimal primer kombinasyonunu belirlemek için 10 dakika boyunca 120 V'ta %1'lik bir agaroz jel üzerinde analiz edildi. Primer çifti F3R3, en parlak ve en yoğun bantları sergileyerek en etkili olarak ortaya çıktı (Şekil 2).
Optimum primer çifti F3R3'ü belirledikten sonra, diğer tüm bileşenleri sabit tutarak RPA amplifikasyonu için nihai primer konsantrasyonlarını ve reaksiyon sürelerini optimize ettik. Bulgularımız, amplifikasyon verimliliğinin 0.44 μM'lik bir primer konsantrasyonunda ve 20 dakikalık bir reaksiyon süresinde zirveye ulaştığını ortaya koydu (Şekil 3). Şablon olarak 107 kopya/μL konsantrasyonda pozitif bir rekombinant plazmit kullanarak, crRNA-1 için floresan yoğunluğunda zamana bağlı bir artış gözlemlerken, crRNA-2 zaman içinde böyle bir artış göstermedi. Sonuç olarak, A. baumannii'nin tespiti için crRNA olarak crRNA-1 seçildi (Şekil 4).
A. baumannii-DETECTR'nin özgüllüğü, çeşitli bakteri suşlarından izole edilen nükleik asitler kullanılarak değerlendirildi. PCR tahlilleri sırasında, yalnızca A. baumannii genomu hedeflendiğinde önemli bir floresan sinyali gözlenirken, diğer bakteri kaynaklarından genomik DNA kullanıldığında floresan tespit edilmedi (Şekil 5). Bu sonuçlar, A. baumannii-F3 ve R3 primer çiftinin, DETECTR testinde A. baumannii için özgüllük sergilediğini doğrulamaktadır, bu da A. baumannii-DETECTR'nin bu patojeni güvenilir bir şekilde tespit edebileceğini göstermektedir.
A. baumannii-DETECTR testinin duyarlılığını değerlendirmek için, A. baumannii DNA'sı ile bir dizi seyreltme gerçekleştirdik. Kopya sayısı aşağıdaki formül kullanılarak hesaplandı: kopya sayısı (kopya/μL) = 6.02 × 1023 x P (ng/μL) x 10-9 / (plazmit baz sayısı x 660) (burada P, plazmit konsantrasyonunu temsil eder). Tahlil, 1 kopya / μL'ye kadar algılama kabiliyetini gösterdi ve bu da yerinde uygulamalar için çok uygun, son derece hassas bir teşhis aracı olarak potansiyelini gösterdi (Şekil 6). Bu nedenle, A. baumannii-DETECTR dikkate değer bir hassasiyet sergiler.
Bu protokolde, hedef DNA dizilerinin tanımlanmasını kolaylaştırmak için CRISPR/Cas12a aracılı DETECTR algılama platformunu yanal akış şeridi (LFS) teknolojisi ile de entegre ettik. LFS bazlı testin duyarlılığını ve özgüllüğünü değerlendirdik. LFS kullanılarak yapılan bir özgüllük testi, diğer bakteri türleriyle çapraz reaktivite belirtisi göstermedi ve bu da yöntemin yüksek özgüllüğünü gösterdi (Şekil 7A). Ayrıca, 107 kopya / μL'den tek bir kopya / μL'ye kadar bir dizi seyreltmede A. baumannii'yi tespit etmek için LFS'yi kullandık. Özellikle, test edilen tüm numuneler, UV ışığı altındaki sonuçlarla uyumlu olan pozitif sonuçlar vermiştir (Şekil 7B) (Şekil 7C, D). Toplu olarak, bu sonuçlar, platformumuzun saha ortamlarında A. baumannii için hızlı, sağlam ve hassas bir teşhis cihazı olarak hizmet ettiğinin altını çizmektedir.

Şekil 1: A. baumannii-DETECTR iş akışı. Kısaltmalar: RPA =R ekombinaz Polimeraz Amplifikasyonu; DETECTR = DNA endonükleaz hedefli CRISPR trans muhabiri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Astarların tasarımı ve seçimi. RPA reaksiyonu için primerlerin optimal kombinasyonu, agaroz jel elektroforezinden elde edilen sonuçlara dayanarak tanımlandı. Primer çifti F3R3, parlak ve yoğun bantlarla en iyi performansı gösterdi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: RPA için primer konsantrasyonu ve amplifikasyon süresinin optimizasyonu. 1-4: Primer konsantrasyonları 0.40 μM, 0.44 μM, 0.48 μM, 0.52 μM. Amplifikasyon süreleri sırasıyla 10 dakika, 20 dakika ve 30 dakikadır. Amplifikasyon etkisi, primer konsantrasyonu 0.44 μM ve reaksiyon süresi 20 dakika olduğunda en yüksek seviyeye ulaşır. Kısaltmalar: M = 8000 DNA Marker; RPA = Rekombinaz Polimeraz Amplifikasyonu; NC = boş kontrol. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
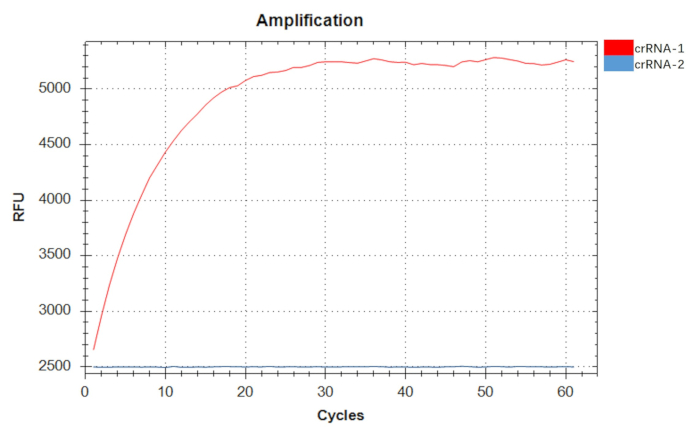
Şekil 4: İki crRNA'nın (crRNA-1 ve crRNA-2) taranması. crRNA-1 tespitinin floresan yoğunluğu zamanla artarken, crRNA-2 tespitinin floresan yoğunluğu zamanla artmadı ve önemli bir tespit aktivitesi göstermedi. crRNA-1, A. baumannii'nin tespiti için crRNA olarak kullanıldı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
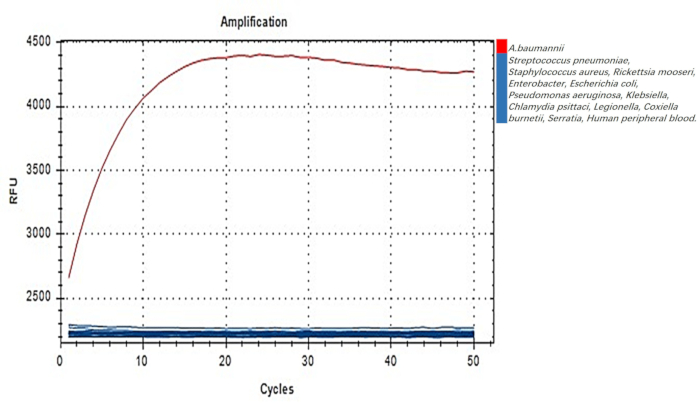
Şekil 5: A. baumannii-DETECTR'ın özgüllüğünün test edilmesi. A. baumannii-DETECTR'nin amplifikasyonu, A. baumannii-F3 ve R3 primer çifti kullanılarak, A. baumannii'den ve diğer bakteri türlerinden ekstrakte edilen genomik DNA kullanılarak elde edildi. Kısaltma: DETECTR = DNA endonükleaz hedefli CRISPR trans muhabiri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
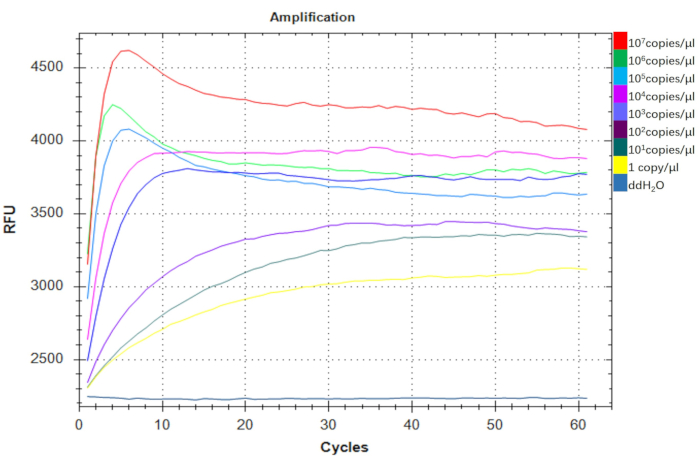
Şekil 6: A. baumannii-DETECTR'ın hassasiyetinin test edilmesi. pUC57-16s rDNA pozitif rekombinant plazmitleri, görsel floresan bazlı RPA-CRISPR / Cas12a testi kullanılarak A. baumannii için tespit sınırını değerlendirmek için hedefler olarak görev yaptı. Tahlil, tutarlı primerler kullanarak bir kopya ila 107 kopya/μL aralığını başarıyla tespit etti. Tek kopya grubundaki floresan yoğunluğu, ddH2O kontrol grubuna kıyasla önemli ölçüde yüksekti. Kısaltmalar: DETECTR = DNA endonükleaz hedefli CRISPR trans muhabir; RPA = Rekombinaz Polimeraz Amplifikasyonu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
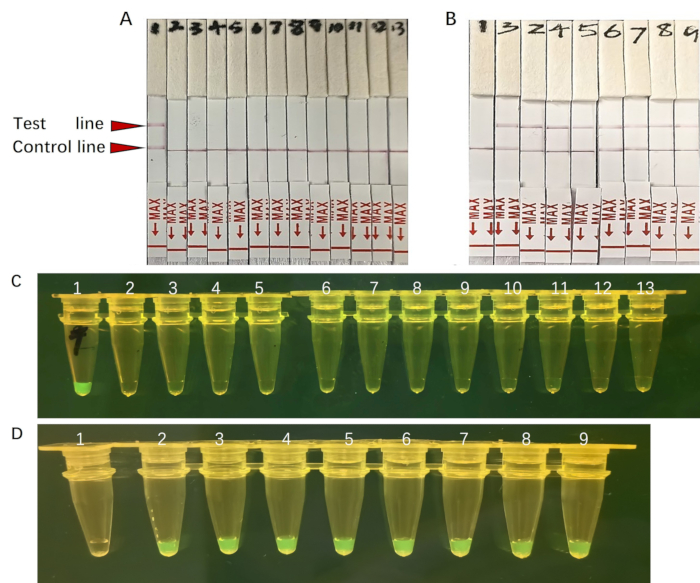
Şekil 7: UV ışığı ve LFS tespiti altında numunenin floresan sinyali.Plazmid standartları ile A. baumannii-DETECTR reaksiyonu. (A-C) A. baumannii-DETECTR ile farklı patojenler tespit edildi. Sadece A. baumannii başarıyla tespit edildi. 1-13, A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Coxiella burnetii, Serratia, İnsan çevre birimini temsil eder. (B-D) A. baumannii-DETECTR tarafından tespit edilen on kat artışlarla seri olarak seyreltilmiş pozitif rekombinant plazmit. Negatif kontrol dışında hepsi tespit edildi. 1-9 negatif kontrolü, 1 kopya/μL, 101 kopya/μL, 102 kopya/μL, 103 kopya/μL, 104 kopya/μL, 105 kopya/μL, 106 kopya/μL, 107 kopya/μL. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Hayır | Ad | Sıra (5'-3') | |||
| 1 | crRNA-1 | uaauuucuacuaaguguagauGUUAAUACCUAGAGAUAGUG | |||
| 2 | crRNA-2 | uaauuucuacuaaguguagauGUUGGUAUUCCGGAAGAAGU | |||
| 3 | crRNA-1-DNA | GTTAATACCTAGAGATAGTG | |||
| 4 | crRNA-2-DNA | GTTGGTATTCCGGAAGAAGT | |||
| 5 | Katranlı dizi | TAACTGAAGAGTTTGATCATGGCTCAGATTGAACGCTGG CGGCAGGCTTAACACATGCAAGTCGAGCGGGGGAAGGT AGCTTGCTACTGGACCTAGCGGCGGACGGGTGAGTAATG CTTAGTAGGAATCTGCCTATTAGTGGGGGACAACATCGAAA GGGATGCTAATACCGCATACGTCCTACGGGAGAAAGCAG GGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCG GATTAGCTAGTTGGTGGGGTAAAGGCCTACCAAGGCGAC GATCTGTAGCGGGTCTGAGAGGATGATCCGCCACACTGG GACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGT GGGGAATATTGGACAATGGGGGGAACCCTGATCCAGCCAT GCCGCGTGTGTGTGAAGAAGGCCTTATGGTTGTAAAGCACTT TAAGCGAGGAGGAGGCTACTTTAGTTAATACCTAGAGATAG TGGACGTTACTCGCAGAATAAGCACCGGCTAACTCTGTGC CAGCAGCCGCGGTAATACAGAGGGTGCGAGCGTTAATCGG ATTTACTGGGCGTAAAGCGTGCGTAGGCGGCTTATTAAGTC GGATGTGAAATCCCCGAGCTTAACTTGGGAATTGCATTCGAT ACTGGTGAGCTAGAGTATGGGAGAGGATGGTAGAATTCCAG GTGTAGCGGTGAAATGCGTAGAGATCTGGAGGAATACCGAT GGCGAAGGCAGCCATCTGGCCTAATACTGACGCTGAGGTAC GAAAGCATGGGGAGCAAACAGGATTAGATACCCTGGTAGTC CATGCCGTAAACGATGTCTACTAGCCGTTGGGGCCTTTGAGG CTTTAGTGGCGCAGCTAACGCGATAAGTAGACCGCCTGGGGA GTACGGTCGCAAGACTAAAACTCAAATGAATTGACGGGGGCC CGCACAAGCGGTGGAGCATGTGGTTTAATTCGATGCAACGC GAAGAACCTTACCTGGCCTTGACATACTAGAAACTTTCCAGA GATGGATTGGTGCCTTCGGGAATCTAGATACAGGTGCTGCAT GGCTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCC CGCAACGAGCGCAACCCTTTTCCTTTGCCAGCATTTCG GATGGGAACTTTAAGGATACTGCCAGTGACAAACTGGAGGAA GGCGGGGACGACGTCAAGTCATCATGGCCCTTACGGCCAGG GCTACACACGTGCTACAATGGTCGGTACAAAGGGTTGCTACA CAGCGATGTGATGCTAATCTCAAAAAGCCGATCGTAGTCCGG ATTGGAGTCTGCAACTCGACTCCATGAAGTCGGAATCGCTAG TAATCGCGGATCAGAATGCCGCGGTGAATACGTTCCCGGGCC TTGTACACACCGCCCGTCACACCATGGGAGTTTGTTGCACCA GAAGTAGCTAGCCTAACTGCAAAGAGGGCGGTTACCACGGTG TGGCCGATGACTGGGGTGAAGTCGTAACAAGGTAGCCGTAGG GGAACCTGCGGCTGGATCACCTCCTTA | |||
| 6 | A.baumannii-F0 | CGGGAGGCAGCAGTGGGGGAATATTGGACAAT | |||
| 7 | A.baumannii-F1 | AAGAAGGCCTTATGGTTGTAAAGCACTTTAA | |||
| 8 | A.baumannii -F2 | GGTTGTAAAGCACTTTAAGCGAGGAGGAGGCTA | |||
| 9 | A.baumannii -F3 | AAGGCCTTATGGTTGTAAAGCACTTTAAGCGA | |||
| 10 | A.baumannii-R1 | ACAGAGTTAGCCGGTGCTTATTCTGCGAGTAA | |||
| 11 | A.baumannii-R2 | AATCCGATTAACGCTCGCACCCTCTGTATTA | |||
| 12 | A.baumannii-R3 | AGTTAGCCGGTGCTTATTCTGCGAGTAACGTC | |||
| 13 | ssDNA-FQ | ssDNA-FQ FAM-TTTATTT-BHQ1 | |||
| 14 | A.baumannii-16s rDNA-F | 5′-CGCGGATCCTAACTGAAGAGTTTGATCATGG-3" | |||
| 15 | A.baumannii-16s rDNA-R | 5′-ACGCGTCGACTAAGGAGGTGATCCAGC-3". | |||
Tablo 1: Bu çalışmada kullanılan nükleik asitler. Burada kullanılan nükleik asit, 5' ucunda 5-Karboksifloresein ile konjuge edilmiş tek sarmallı DNA'dan ve 3' ucunda floresan söndürücü Kara Delik Söndürücü 1'den oluşan ssDNA-FQ'dur. Kısaltmalar: F = florofor; Q = söndürücü.
Ek Dosya 1: Primer ve crRNA'nın tasarımı.Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
A. baumannii enfeksiyonları için geleneksel tanı yöntemleri, onları daha az erişilebilir ve bakım noktası testleri için uygun kılan çeşitli kısıtlamalara sahiptir27. Örneğin, PCR iyi bir duyarlılığa ve özgüllüğe sahiptir, ancak profesyoneller tarafından çalıştırılması gereken özel termal döngü ekipmanı ve karmaşık iş akışları gerektirir. Burada sunulan, RPA ve CRISPR-LbaCas12a genom düzenlemesini A. baumannii-DETECTR testi olarak bilinen yeniden birleştirme ile birleştiren yeni yöntem, verimli genetik manipülasyona izin verir. Acinetobacter baumannii enfeksiyonlarının yanı sıra diğer bakteriyel patojenlerin teşhisi için hızlı, hassas ve spesifik bir tespit yöntemi sağlar. İş akışı Şekil 1'de gösterilmiştir.
Yöntemimiz, mevcut tanı tekniklerinin eksikliklerinin üstesinden gelerek hastanın iyileşmesini kolaylaştırması ve A. baumannii'nin yayılmasını azaltması gereken enfeksiyonların teşhisinde ve yönetiminde özellikle avantajlıdır. A. baumannii-DETECTR sisteminin A. baumannii'yi yüksek özgüllük ve duyarlılıkla tanımladığı bulunmuştur (Şekil 5 ve Şekil 6). Bulgularımız, gerçek zamanlı değerlendirme sırasında yalnızca A. baumannii genomunun pozitif bir floresan kıvrım değişikliği sergilediğini, diğer tüm türlerin genomlarının ise negatif sonuçlar gösterdiğini ortaya koydu (Şekil 5).
RPA-CRISPR/Cas12a tespit platformunun hassasiyetini değerlendirmek için, A. baumannii için pozitif olan rekombinant plazmitler pUC57-16s rDNA, sistemin tespit limitini (LOD) belirlemek için hedef olarak kullanıldı. Floresan bazlı RPA-CRISPR / Cas12a testinde, tespit mikrolitre başına bir kopya ile mikrolitre başına 107 kopya arasında değişmiştir ve negatif kontrol grubuna kıyasla 1 kopya / μL grubunda floresan yoğunluğunda kayda değer bir artış gözlenmiştir (Şekil 6). Daha da önemlisi, gerçek zamanlı tanı için, algılamanın sonuçları, gerçek zamanlı tanı için gerekli olan laboratuvar tabanlı ekipmandan bağımsız olarak, elde taşınan taşınabilir bir cihaz aracılığıyla doğrudan görselleştirilebilir.
Protokol26 ile ilgili olarak birkaç kritik faktör göz önünde bulundurulmalıdır. İlk olarak, A. baumannii-DETECTR testinin yüksek hassasiyeti göz önüne alındığında, numune toplama işlemi sırasında çapraz kontaminasyonu önlemek önemlidir. Ek olarak, reaksiyon reaktifleri doğrudan güneş ışığına maruz kalmaktan korunmalıdır. Tüm prosedür adımlarına bağlı kalmak, mümkün olan en iyi sonuçları elde etmek için çok önemlidir. Pratik uygulamalarda A. baumannii-DETECTR sisteminin uygun işlevselliğini doğrulamak için A. baumannii'ye özgü gen fragmanlarını barındıran bir plazmitten oluşan pozitif bir kontrol dahil edilmelidir.
Protokol, parametrelerin belirli laboratuvar koşulları ve primer konsantrasyonu, reaksiyon süresi ve diğer değişkenler gibi numune türleri için optimize edilebileceğini göstermektedir. Bu deneyde, en yüksek amplifikasyon verimliliğini ve özgüllüğünü üretmek için RPA amplifikasyonu için optimum primer kombinasyonu, primer konsantrasyonları ve reaksiyon süreleri seçilmiştir. Bu, agaroz jel elektroforezi ile belirlendi (Şekil 2 ve Şekil 3). Ek olarak, protokolde doğruluğu sağlamak için kontrol olarak A. baumannii genomunun korunmuş bir bölgesini içeren pozitif bir rekombinant plazmit kullanıldı. Ticari bakteriyel genomik DNA ekstraksiyon kitlerinin, bu protokolde DNA ekstraksiyonu için standartlaştırılmış ve güvenilir bir yöntem sağladığını belirtmekte fayda var. Bununla birlikte, gelecekteki çalışmalarda, klinik veya çevresel numuneler gibi farklı numune türleri, A. baumannii DNA'sını etkili bir şekilde izole etmek için özel ekstraksiyon yöntemleri28 gerektirebilir.
Pozitif kontrolden tespit edilebilir floresan sinyallerinin yokluğunda, reaksiyon karışımında reaktiflerin değiştirilmesini gerektiren inhibitörlerin bulunması muhtemeldir. Tersine, floresan yoğunluğu net farklılaşma için yetersizse, inkübasyon süresinin uzatılması faydalı olabilir. Bununla birlikte, uzun süreli inkübasyon da yanlış pozitiflere neden olabilir26.
A. baumannii için mevcut tespit metodolojileri, pahalı PCR ekipmanının, sabit operasyonel konumların ve uzman personelin kullanılmasını gerektirmektedir. Buna karşılık, burada önerilen yöntem, A. baumannii'nin saha ortamlarında doğru ve hassas teşhisini kolaylaştırarak daha geniş bir demografi için erişilebilir olmasını sağlar.
İzotermal amplifikasyonun özellikleri ve floresan yoluyla toplanan veriler, A. baumannii-DETCTER iş akışını basit ve düşük ekipman gereksinimleri haline getirmenin yanı sıra uzak bölgelerde veya salgınlar sırasında hızlı patojen tespiti gerçekleştirmeyi mümkün kılar. Yeni bir izotermal nükleik asit amplifikasyon tekniği olan RPA-LFS 29,30,31,32, operasyonel basitliği ve azaltılmış zamansal talepleri nedeniyle viral, bakteriyel, fungal ve parazitik patojenlerin tespitinde önemli bir ilgi görmüştür. Protokol, ultraviyole (UV) ışık veya Transilluminatör ve yanal akış şeritleri (LFS) algılama altında numunenin floresan sinyalini detaylandırır. Görsel inceleme kısa sürede sonuçlandığı için yüksek hassasiyetli cihazlara ve uzman teknisyenlere bağımlılığı önleyebilir (Şekil 7), böylece pahalı ve karmaşık laboratuvar ekipmanlarına ve operasyonel nüfusa olan ihtiyacı ortadan kaldırır. Ayrıca, tek tüp yöntemi sadece zaman kazandırmakla kalmaz, aynı zamanda amplikon kontaminasyonu riskini de azaltır33.
Gelecekteki çalışmalar, daha verimli ve daha düşük maliyetli DNA ekstraksiyon tekniklerinin araştırılması gibi, çeşitli numune türleri ve farklı çevresel koşullar için uygulanabilir hale getirmek için yöntemi geliştirmeyi amaçlamalıdır. A. baumannii-DETECTR'in kullanımı sadece klinik tanı için değil, aynı zamanda çevresel değerlendirme, salgınların izlenmesi ve yönetimi için de kullanılabilir, bu da toplulukların ve hastanelerin A. baumannii'yi hızlı bir şekilde tanımlamasını sağlar. Ayrıca, crRNA ve primerleri modifiye ederek, bu moleküler tanı yaklaşımı, Streptococcus pneumoniae, Staphylococcus aureus ve Enterobacter gibi çeşitli solunum yolu bakteriyel enfeksiyonlarının tespiti için uyarlanabilir.
Özetle, A. baumannii-DETECTR, yüksek hassasiyeti ve özgüllüğü, kullanıcı dostu tasarımı ve taşınabilirliği sayesinde A. baumannii'yi mümkün olduğunca hızlı ve doğru bir şekilde tanımlayabilir. Bu yöntem, zamanında ve etkili tedaviyi, enfeksiyon kontrolünü kolaylaştırarak ve antibiyotik direncinin çoğalmasını azaltarak hasta sağlığı sonuçlarını iyileştirebilir ve aynı zamanda A. baumannii enfeksiyonlarının artan tehdidiyle mücadele edebilir.
Açıklamalar
Yazarlar rekabet eden çıkarları olmadığını beyan ederler.
Teşekkürler
Bu çalışma, Çin'in Jilin Eyaleti Bilim ve Teknoloji Geliştirme Planı Projesi (20240305027YY) ve Jilin İl Maliye Bakanlığı (JLSWSRCZX2023-55, JLSWSRCZX2021-041) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| -20 °C Freezer | Haier | HYCD-290 | China |
| Agarose Basic | BioFroxx | 1110GR100 | China |
| All oligonucleotides and crRNA were synthesized by company | www.comatebio.com | ||
| Cas12a cutting substrate - ssDNA - fluorescent type | EZassay Biotech. Co. Ltd. | DNA-FAM-BHQ | China |
| Cas12a cutting substrate - ssDNA - test paper type | EZassay Biotech. Co. Ltd. | DNA-FAM-BIO | China |
| Electrophoresis apparatus | BIO-RAD | POWER PAC1000 | USA |
| Fluorescence quantitative PCR instrument | BIO-RAD | CFX Connect | USA |
| Gel Imaging System | BIO-RAD | Gel Doc 2000 | USA |
| https://ezassay.com/rna | |||
| https://www.ezassay.com/primer | |||
| https://www.ncbi.nlm.nih.gov/tools/primer-blast | |||
| Lateral flow paper strip (Biotin/FAM) | EZassay Biotech. Co. Ltd. | HD-FMBO | China |
| LbaCas12a (Cpf1) enhanced protein | EZassay Biotech. Co. Ltd. | CAS-12E-001 | China |
| LED Transilluminator | LABGIC | BL-20 | China |
| Magnesium acetate, MgOAc | TwistDx | TABAS03KIT | UK |
| Microcentrifuge | allsheng | Mini-6k | China |
| PCR strip tubes | PCR strip tubes | PST-0208-FT-C | China |
| TGrade Dry Bath Incubator | Tiangen biochemical technology | OSE-DB-01 | China |
| Tianamp Bacteria DNA Kit | Tiangen biochemical technology | DP302-02 | China |
| TIANamp Bacteria DNA Kit | TIANGEN BIOTECH (BEIJING) CO.; LTD. | DP302 | China |
| TransStart FastPfu DNA Polymerase | TransGen Biotech. Co. Ltd. | AP221 | China |
| TwistAmp Basic Kit | TwistDX | TABAS03KIT | UK |
| Universal DNA Purification Kit | Tiangen biochemical technology | DP214-03 | China |
Referanslar
- Lenie, D., Alexandr, N., Harald, S. An increasing threat in hospitals: Multidrug-resistant Acinetobacter baumannii. Nat Rev Microbiol. 5 (12), 939-951 (2007).
- Li, P., et al. Rapid detection of Acinetobacter baumannii and molecular epidemiology of carbapenem-resistant A. baumannii in two comprehensive hospitals of Beijing, China. Front Microbiol. 6, 997 (2015).
- Wu, X., et al. A diagnostic test that uses isothermal amplification and lateral flow detection sdaA can detect tuberculosis in 60 min. J Appl Microbiol. 130 (6), 2102-2110 (2020).
- Huang, B., et al. A cas12a-based fluorescent microfluidic system for rapid on-site human papillomavirus diagnostics. Appl Microbiol Biotechnol. 107 (20), 6287-6297 (2023).
- Piepenburg, O., Williams, C. H., Stemple, D. L., Armes, N. A. DNA detection using recombination proteins. Plos Biology. 4 (7), 1115-1121 (2006).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), aaf5573 (2016).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), eaar6245 (2018).
- Xiong, D., et al. Rapid detection of SARS-CoV-2 with CRISPR-Cas12a. PLoS Biol. 18 (12), e3000978 (2020).
- Gootenberg, J. S., et al. Nucleic acid detection with crispr-Cas13a/C2C2. Science. 356 (6336), 438-442 (2017).
- Harrington, L. B., et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science. 362 (6416), 839-842 (2018).
- Li, S. Y., et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA. Cell Res. 28 (4), 491-493 (2018).
- Wang, B., et al. Cas12aVDet: A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem. 91 (19), 12156-12161 (2019).
- Huang, Z., et al. Ultra-sensitive and high-throughput crispr-p owered COVID-19 diagnosis. Biosens Bioelectron. 164, 112316 (2020).
- Zhang, W. S., et al. Reverse transcription recombinase polymerase amplification coupled with CRISPR-Cas12a for facile and highly sensitive colorimetric SARS-CoV-2 detection. Anal Chem. 93 (8), 4126-4133 (2021).
- Marraffini Luciano, A. CRISPR-Cas immunity in prokaryotes. Nature. 526 (7571), 55-61 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), aad5147 (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 13 (11), 722-736 (2015).
- Li, S. Y., et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discovery. 4 (1), 20 (2018).
- Li, L., Li, S., Wu, N., Wu, J., Wang, J. Holmesv2: A CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol. 8 (10), 2228-2237 (2019).
- Liang, M., Li, Z., Wang, W., Liu, J., Zhang, L. X. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules. Nat Communications. 10 (1), 3672 (2019).
- Aquino-Jarquin, G. CRISPR-Cas14 is now part of the artillery for gene editing and molecular diagnostic. Nanomedicine. 18, 428-431 (2019).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Swarts, D. C., John, V. D. O., Jinek, M. Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a. Mol Cell. 66 (2), 221-233 (2017).
- Jiang, Y., et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun. 8, 15179 (2017).
- Yan, M. Y., et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol. 83 (17), e00947-e01017 (2017).
- Ashraf, A., et al. A novel multiplex pcr assay for simultaneous detection of nine clinically significant bacterial pathogens associated with bovine mastitis. Mol Cell Probes. 33, 57-64 (2017).
- Zhu, L., et al. A rapid on-site visualization platform based on RPA coupled with CRISPR-Cas12a for the detection of genetically modified papaya 'huanong no.1'. Talanta. 277, 126437 (2024).
- Zheng, C., et al. Rapid developments in lateral flow immunoassay for nucleic acid detection. Analyst. 146 (5), 1514-1528 (2021).
- Wang, Y., et al. Establishment and clinical application of a RPA-LFS assay for detection of capsulated and non-capsulated Haemophilus influenzae. Front Cell Infect Microbiol. 12, 878813 (2022).
- Wang, F., et al. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strips for detecting Candida albicans. Anal Biochem. 633, 114428 (2021).
- Ma, B., et al. A simple and efficient method for potential point-of-care diagnosis of human papillomavirus genotypes: Combination of isothermal recombinase polymerase amplification with lateral flow dipstick and reverse dot blot. Anal Bioanal Chem. 411 (28), 7451-7460 (2019).
- Sun, Y., Yu, L., Liu, C., Ye, S., Huang, W. One-tube SARS-CoV-2 detection platform based on RT-RPA and CRISPR/Cas12a. J Transl Med. 19 (1), 74 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır