Method Article
Schneller und spezifischer Nachweis von Acinetobacter baumannii-Infektionen mit einem Rekombinase-Polymerase-Amplifikations-/Cas12a-basierten System
In diesem Artikel
Zusammenfassung
Um den schnellen und präzisen Nachweis von Acinetobacter baumannii zu ermöglichen, stellen wir ein Protokoll vor, das die Rekombinase-Polymerase-Amplifikation (RPA) in Verbindung mit der LbaCas12a-Endonuklease zur Identifizierung von A. baumannii-Infektionen verwendet.
Zusammenfassung
Acinetobacter baumannii, ein gramnegatives Bakterium, ist berüchtigt dafür, schwere Infektionen mit hohen Sterblichkeitsraten zu verursachen. Ein schneller und genauer Nachweis von A. baumannii ist entscheidend für eine schnelle Behandlung, eine wirksame Infektionskontrolle und die Eindämmung von Antibiotikaresistenzen. Es gibt jedoch keine geeignete Methode, um A . baumannii schnell und einfach vor Ort nachweisen zu können. Das DNA Endonuclease Targeted CRISPR Trans Reporter (DETECTR) System bietet einen schnellen, präzisen und sensitiven Ansatz für den Nachweis von A. baumannii , indem es die zielspezifischen Erkennungsfähigkeiten von Cas12a mit der isothermen Amplifikationseffizienz der Rekombinase-Polymerase-Amplifikation (RPA) integriert. Dieses Protokoll beschreibt den Nachweis von A. baumannii mittels RPA in Kombination mit LbaCas12a-Endonuklease. Folgende Schritte werden in diesem Artikel beschrieben: Extraktion der DNA, Auswahl einer spezifischen DNA-Sequenz, Design des Primers und der CRISPR-RNA (crRNA), Konstruktion eines positiven rekombinanten Plasmids, Aufbau des Cas12a-RPA-Assays, Optimierung des RPA-Amplifikationssystems, Visualisierung des RPA-CRISPR/Cas12a-Assays mit einem Fluoreszenzdetektionswerkzeug wie z.B. einem Real-Time-PCR-Instrument, und Bewertung der Sensitivität und Spezifitätsbewertung.
Einleitung
In der klinischen Mikrobiologie stellt der Nachweis von Acinetobacter baumannii-Infektionen eine große Herausforderung dar. Dieses gramnegative Bakterium kann Infektionen mit schweren klinischen Symptomen verursachen, auch bei hohen Mortalitätsraten, insbesondere bei immungeschwächten Patienten1. Herkömmliche kulturbasierte Nachweismethoden für Erregerinfektionen sind zeitaufwändig und möglicherweise nicht empfindlich, was die Antibiotikabehandlung verzögern und die Patientenergebnisse beeinträchtigen kann. Die schnelle und genaue Identifizierung von A. baumannii ist entscheidend für eine wirksame Behandlung und Ausbruchskontrolle. Um diesen Bedarf zu decken, wurden molekulare Techniken mit Nukleinsäureamplifikation erforscht. Diese Ansätze erfordern jedoch oft hochentwickelte Temperaturwechselgeräte und können durch gut ausgebildete Techniker und gut etablierte Labore eingeschränkt werden. Um diese Herausforderungen zu bewältigen, konzentriert sich die Forschung zunehmend auf die Entwicklung isothermer Amplifikationsmethoden 2,3,4.
Die Rekombinase-Polymerase-Amplifikation (RPA) ist eine Methode, die von Piepenburg et al . 5 etabliert wurde und zur Amplifikation von DNA verwendet wird, ähnlich der Polymerase-Kettenreaktion (PCR), jedoch ohne dass Temperaturzyklen erforderlich sind. Diese Methode umfasst 50 mM Tris (pH 7,5), 100 mM Kaliumacetat, 14 mM Magnesiumacetat, 2 mM DTT, 5% PEG20000 (ein Polyethylenglykol mit hohem Molekulargewicht), 200 μM dNTPs, 3 mM ATP, 50 mM Phosphokreatin, 100 μg/mL Kreatinkinase, 120 μg/mL UvsX, 30 μg/mL UvsY, 900 μg/mL Gp32, 30 μg/mL Bsu LF, 450 nM Primer und DNA-Vorlagen. Der Amplifikationsprozess beginnt mit der Bindung des Rekombinase-Proteins UvsX an Primer in Gegenwart von 3 mM ATP und 5% PEG20000 Bildung eines Rekombinase-Primer-Komplexes. Dieser Komplex ermöglicht dann die Kombination von Primern mit den homologen Sequenzen auf der doppelsträngigen DNA.
Mit Hilfe von UvsY erleichtert die Rekombinase UvsX den Austausch zwischen dem Primer und dem Template-Strang, was zur Verdrängung eines Strangs der Ziel-DNA führt. Gp32 trägt zur Aufrechterhaltung der einzelsträngigen DNA-Struktur bei. Schließlich dissoziiert die Rekombinase, und eine DNA-Polymerase (Bsu LF), die in der Lage ist, DNA-Stränge zu verdrängen, bindet an das 3'-Ende des Primers und verlängert ihn in Gegenwart von Desoxyribonukleosidtriphosphaten (dNTPs). Dieser Prozess wird zyklisch wiederholt, wodurch eine exponentielle Verstärkung erreicht wird. Alle Amplifikationsprozesse können innerhalb von 20-40 min und bei relativ konstanten Temperaturen zwischen 37 °C und 42 °C abgeschlossen werden. Dieser Temperaturbereich ist in etwa identisch mit physiologischen Temperaturen, was die Durchführung von RPA in einer minimalistischen Umgebung ermöglicht, was RPA zu einem vielseitigen und effizienten Werkzeug für den DNA-Nachweis und die DNA-Analyse macht.
Das System der Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) und CRISPR-assoziierten Proteine (CRISPR-Cas) fungiert als adaptiver Immunmechanismus in Bakterien und Archaeen und umfasst Systeme der Klassen I und II. Klasse II umfasst Proteine wie Cas12 (a, b, f), Cas13 (a, b) und Cas14, die Ziel-DNA oder -RNA identifizieren und spalten, die von CRISPR-RNA (crRNA) gesteuert wird6,7,8 . LbaCas12a (oder Cpf1) ist eine crRNA-gesteuerte DNA-Endonuklease. Die crRNA dient als Leit-RNA innerhalb des CRISPR-Cas-Systems, wo sie mit Cas-Proteinen komplexiert. Es nutzt seine Spacerregion, um sich mit der Ziel-DNA zu paaren und den Proteinkomplex effektiv zu den Zielsequenzen zu lenken. Die Sequenz 5'-UAAUUUCUACUAAGUGUAGAU-3' dient als konservierte crRNA-Wiederholung, ein konstantes Element in allen LbaCas12a-crRNAs. Auf diese Wiederholung folgt ein zielspezifisches Segment, das sich je nach beabsichtigtem DNA-Ziel unterscheidet. Diese Zielsequenz ist zwischen 18 und 24 Nukleotide lang.
Die PAM-Sequenz (Protospacer-Adjacent Motif) (TTTV, wobei V A, C oder G sein kann) befindet sich am 5'-Ende des nicht-komplementären Strangs des DNA-Ziels. Der Cas12a-Schnitt zielt auf doppelsträngige DNA an der PAM-Sequenz ab. Anschließend führen die aktivierten Cas-Proteine eine unspezifische trans-Spaltung von einzelsträngiger DNA (ssDNA) durch9,10,11,12. Somit erfährt die mit einem Fluorophor und Quencher markierte ssDNA eine Spaltung, was zur Emission eines Fluoreszenzsignals nach der Kollateralspaltung führt 8,13. Die Fluoreszenzintensität kann mit einem Fluoreszenz-Reader für eine präzise Nukleinsäuredetektion quantifiziert werden14. Insbesondere wird Cas12a in großem Umfang für die DNA-Analyse verwendet, wobei seine Bindung und Spaltung von Zielsequenzen von der Erkennung des Protospacer-Nachbarmotivs (PAM) abhängt, während Cas13a unabhängig von einer solchen Anforderung arbeitet.
CRISPR-Cas-Systeme 15,16,17 erleichtern die Spaltung innerhalb einer einzigen Reaktion 18,19,20,21. Durch die Kombination der beiden oben genannten kann ein robustes Werkzeug geschaffen werden, das als A. baumannii-DETECTR (LbaCas12a-Enabled Detection of Targeted A. baumannii) für den Nukleinsäurenachweisbekannt ist 22,23,24. Dieses Protokoll demonstriert die Verwendung von RPA in Verbindung mit der DNase-Aktivität von Cas12a, um eine crRNA-gesteuerte Sequenz innerhalb des 16s-rDNA-Gens von A. baumannii spezifisch zu adressieren und zu spalten, wodurch ein sensitiver und präziser Nachweis des Erregers ermöglicht wird.
Die A. baumannii-DETECTR-Methode bietet gegenüber herkömmlichen Nachweismethoden mehrere Vorteile25,26. Erstens rationalisiert die isotherme Eigenschaft von RPA die Betriebsabläufe und senkt die Kosten durch seinen Thermocycler-unabhängigen Verstärkungsmechanismus5. Zweitens erhöht die Cas12a-Endonuklease die Spezifität des Assays durch crRNA-vermitteltes Targeting und Watson-Crick-Basenpaarung7. Schließlich spart die Ein-Röhrchen-Methode von A. baumannii-DETECTR nicht nur Zeit, sondern reduziert auch das Risiko einer Amplikonkontamination erheblich19.
Das Protokoll beschreibt die Schritte für die Extraktion der DNA-Extraktion, die Auswahl von Ziel-DNA-Sequenzen, das Design von Primern und crRNAs, die Konstruktion positiver rekombinanter Plasmide, die Einrichtung des Cas12a-RPA-Assays, die Optimierung der Optimierung der RPA-Amplifikation und die Visualisierung der Visualisierung der Ergebnisse unter Verwendung von Fluoreszenzdetektionsinstrumenten, wie z. B. einer Real-Time-PCR-Maschine. Komplett mit Sensitivitäts- und Spezifitätsbewertungen.
Die A. baumannii-DETECTR-Methode ist vielversprechend für die Verbesserung der Patientenergebnisse, indem sie eine rechtzeitige und effektive Behandlung, eine robuste Infektionskontrolle und die Eindämmung der Ausbreitung von Antibiotikaresistenzen ermöglicht. Dieses Protokoll kann als umfassender Leitfaden für die Implementierung der Cas12a-RPA-Technologie dienen und ihren Nutzen in verschiedenen Bereichen des Gesundheitswesens verbessern. Es ist jedoch wichtig zu beachten, dass jeder Schritt auf der Grundlage der spezifischen Laborbedingungen und der Vielfalt der Proben optimiert werden sollte, um konsistente und zuverlässige Ergebnisse zu gewährleisten.
Protokoll
1. Aufbau des A. baumannii -DETECTR
HINWEIS: Die Konstruktion von A. baumannii-DETECTR ist ein vierstufiger Prozess, der das Design von Primer und crRNA, die Konstruktion der Konstruktion des positiven rekombinanten Plasmids, die Herstellung von Reaktionslösungen, die isotherme DNA-Amplifikation durch RPA und die Visualisierung des RPA-CRISPR/Cas12a-Assays umfasst. Das Schema des DECERT-Assays ist in Abbildung 1 dargestellt.
- Design von Primer und crRNA (siehe Ergänzende Datei 1)
- Entwerfen Sie 12 Paar Grundierungen mit Designwerkzeugen auf der Grundlage von Primer-Designprinzipien. Besorgen Sie sich vier Vorwärts-Primer (F0-F3) und drei Rückwärts-Primer (R1-R3), um zwölf Primerpaare zusammenzustellen (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- Stellen Sie sicher, dass das 5'-Ende des Primers C- oder T-Basen innerhalb der ersten 3-5 Nukleotide enthält, anstatt einer kontinuierlichen G-Dehnung. Stellen Sie außerdem sicher, dass die letzten drei Nukleotide am 3'-Ende idealerweise G- und C-Basen sind, um die Amplifikation zu verbessern.
- Entwerfen Sie Primer ohne selbstkomplementäre Sequenzen, um Haarnadelbildung zu vermeiden, und mit nicht mehr als vier komplementären oder homologen Basen dazwischen. Halten Sie sich von der Komplementarität des 3'-Endes fern, um eine Primer-Dimerisierung zu vermeiden.
- Bewerten Sie jedes Primerpaar auf Spezifität mit dem Primer-BLAST-Tool des National Center for Biotechnology Information (NCBI), um die Wirksamkeit und Spezifität der Primer zu bewerten.
HINWEIS: Primer-BLAST kann die NCBI-Datenbank nutzen, um Sequenzen zu finden, die mit den entworfenen Primern übereinstimmen, und hilft dem Benutzer, die Wirksamkeit und Spezifität der Primer zu beurteilen. - Nach dem Primer-Screening entwerfen Sie die CRISPR-RNA (crRNA) oder Guide-RNA (gRNA) unter Verwendung der LbaCas12a crRNA-Gerüstsequenz (5'-UAAUUUCUACUAAGUGUAGAU-3') als fixierte Sequenz. Integrieren Sie ein zielspezifisches Segment (5'-GUUAAUACCUAGAGAUAGUG-3'), um sicherzustellen, dass sich das PAM-Motiv am 5'-Ende des nicht-komplementären Strangs befindet.
HINWEIS: Die Sequenzen aller Oligonukleotide und crRNA sind in Tabelle 1 aufgeführt.
- Entwerfen Sie 12 Paar Grundierungen mit Designwerkzeugen auf der Grundlage von Primer-Designprinzipien. Besorgen Sie sich vier Vorwärts-Primer (F0-F3) und drei Rückwärts-Primer (R1-R3), um zwölf Primerpaare zusammenzustellen (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- Positiver rekombinanter Plasmidaufbau
- Amplifizieren Sie das 16s rDNA-Segment mit dem Primer-Set A. baumannii 16s rDNA-F (Forward-Primer) und A. baumannii 16s rDNA-R (Reverse-Primer).
- Geben Sie 1 μl 10 μM F und 10 μM R, 10 μl 5x FastpFu Puffer, 1 μl FastpFu Polymerase, 4 μl 2,5 mM dNTPs, 1 μl 10 ng/μl genomische DNA von A. baumannii und 32 μl nukleasefreies Wasser in das 50 μl PCR-System.
- Die Bedingungen für den PCR-Zyklus sind wie folgt einzustellen: anfängliche Denaturierung bei 95 °C für 5 min, gefolgt von 33 Denaturierungszyklen bei 95 °C für 30 s, Glühen bei 55 °C für 30 s und Verlängerung bei 72 °C für 1 min und 20 s. Zum Schluss 10 min bei 72°C für die abschließende Verlängerung durchführen.
- Um das positive PCR-Produkt aufzureinigen, verwenden Sie ein DNA-Aufreinigungskit gemäß den Anweisungen des Herstellers. Klonieren Sie danach die gereinigte DNA in den pUC57-Vektor, der mit BamHI- und SalI-Enzymen verdaut wurde.
HINWEIS: Dieser Prozess führt zur Erzeugung eines positiven rekombinanten Plasmids, das eine konservierte Region enthält. - Das positive rekombinante Plasmid wird seriell in zehnfachen Schritten mit sterilisiertem ddH2O verdünnt, um eine Konzentration von einer Kopie/μl auf 107 Kopien/μl zu erreichen. Lagern Sie das verdünnte Plasmid bei -20 °C für nachfolgende Experimente.
- Amplifizieren Sie das 16s rDNA-Segment mit dem Primer-Set A. baumannii 16s rDNA-F (Forward-Primer) und A. baumannii 16s rDNA-R (Reverse-Primer).
- Vorbereitung der Lösung
- Lösung A wird wie folgt vorbereitet: Für jede Reaktion werden 29,5 μl primerfreier Rehydrationspuffer, 2,4 μl 10 μM A. baumannii-F und 10 μM A. baumannii-R sowie 12,2μl ddH2O hinzugefügt, um ein Endvolumen von 46,5 μl zu erreichen. Geben Sie als nächstes das Reaktionsgemisch in die Enzymreaktionsröhrchen aus dem RPA-Kit. Pipette zum Mischen.
HINWEIS: Die Primer von A. baumannii-F und R werden in dem Assay verwendet, der auf das A. baumannii-Gen abzielt. Die Reihenfolge entnehmen Sie bitte Tabelle 1 . - Bereiten Sie Lösung B wie folgt vor: Fügen Sie für jede Reaktion 0,24 μl 10 μM ssDNA-Reporter, 2 μl 10x Cas12a-Reaktionspuffer, 1 μl 1 μM crRNA, 1 μl 1 μM Cas12a und 10,76μl ddH2O hinzu, um ein Endvolumen von 15 μl zu erreichen.
HINWEIS: Die Sonden (ssDNA-Reporter) wurden gekauft und bestanden aus einem einzelsträngigen DNA-Reporter, der am 5'-Ende mit einem 5-Carboxyfluorescein (FAM)-Fluorophor markiert war, und einem Black Hole Quencher 1 (BHQ1)-Quencher am 3'-Ende. Bei zahlreichen Proben, die detektiert werden müssen, ist eine beträchtliche Menge Lösung B vorzubereiten und die Proben später in Aliquote aufzuteilen.
- Lösung A wird wie folgt vorbereitet: Für jede Reaktion werden 29,5 μl primerfreier Rehydrationspuffer, 2,4 μl 10 μM A. baumannii-F und 10 μM A. baumannii-R sowie 12,2μl ddH2O hinzugefügt, um ein Endvolumen von 46,5 μl zu erreichen. Geben Sie als nächstes das Reaktionsgemisch in die Enzymreaktionsröhrchen aus dem RPA-Kit. Pipette zum Mischen.
- Isotherme DNA-Amplifikation (RPA)
- 1 μl des positiven rekombinanten Plasmids von 107 Kopien/μl wird zu Lösung A mit unterschiedlichen Primerpaaren gegeben; Gründlich mischen. Geben Sie dann 2,5 μl 280 mM Magnesiumacetat (MgOAc) in die Lösung und mischen Sie gut.
HINWEIS: RPA-Reaktionen starten, sobald MgOAc hinzugefügt wird. - Bei 39 °C 20 min inkubieren. Nach 20 Minuten reinigen Sie die mit verschiedenen Primern amplifizierten Produkte unter Verwendung von RPA mit einem Aufreinigungskit und lassen Sie dann die sauberen Amplikons 10 Minuten lang mit einem 1%igen Agarosegel bei 120 V laufen, um die hellste und breiteste Bande als bestes Primerpaar auszuwählen.
HINWEIS: Bei einer niedrigen Vorlagenkopienzahl nach 4 Minuten das EP-Rohr entfernen, den Vortex verwenden und kurz drehen; Setzen Sie es dann für weitere 16 Minuten in das Heizgerät ein. Nach der Amplifikation sollten die Röhrchen mit äußerster Vorsicht geöffnet werden, um eine Kontamination der Arbeitsflächen mit Amplikons zu vermeiden. Diese Vorsichtsmaßnahme verringert das Risiko, in nachfolgenden experimentellen Verfahren falsch positive Ergebnisse zu induzieren.
- 1 μl des positiven rekombinanten Plasmids von 107 Kopien/μl wird zu Lösung A mit unterschiedlichen Primerpaaren gegeben; Gründlich mischen. Geben Sie dann 2,5 μl 280 mM Magnesiumacetat (MgOAc) in die Lösung und mischen Sie gut.
- Optimierung des RPA-Amplifikationssystems
HINWEIS: Das im vorherigen Schritt ausgewählte optimale Primerpaar F3R3 wurde für die RPA-Amplifikationsreaktion auf unterschiedliche Endkonzentrationen eingestellt, um die optimale Primerkonzentration und -zeit herauszufiltern. NC ist eine Blindkontrolle für die entsprechende Primerkonzentration.- Halten Sie die Konzentrationen der anderen Komponenten konstant und das Gesamtvolumen von 46,5 μl in Lösung A. Passen Sie das Volumen von RNase-freiem ddH2O entsprechend an, um die endgültigen Primerkonzentrationen von 10 μM F3 und 10 μM R3 bei 0,40 μM (2,0 μL), 0,44 μM (2,2 μL), 0,48 μM (2,4 μL) und 0,52 μM (2,6 μL) zu erreichen. beziehungsweise.
- Inkubieren Sie bei 39 °C für verschiedene Zeiten: 10 min, 20 min, 30 min.
- Reinigen Sie die RPA-Produkte mit einem DNA-Aufreinigungskit und führen Sie eine Elektrophorese an einem 1%igen Agarosegel bei 120 V für 10 Minuten durch, um die optimale Primerkonzentration und Inkubationszeiten durch Analyse der Eigenschaften der Banden zu ermitteln.
- Visualisierung des RPA-CRISPR/Cas12a-Assays
- 5 μl des RPA-Produkts in Lösung B geben und gründlich mischen, bis eine homogene Masse entsteht.
- Legen Sie die Proben in das quantitative Fluoreszenz-PCR-Instrument, stellen Sie den FAM-Kanal ein und lesen Sie die Werte jede Minute für 60 Minuten bei 37 °C, 60x für insgesamt 60 Minuten.
HINWEIS: Das grüne Fluoreszenzsignal kann mehrere Tage dauern. Um sicherzustellen, dass das System effektiv funktioniert, ist es besser, einen DNA- und RNase-freien Test durchzuführen.
2. Spezifitätsbewertung der RPA-CRISPR/Cas12a-basierten Nachweisplattform
HINWEIS: Um die Spezifität von A. baumannii-DETECTR zu testen, wurden die Nukleinsäuren von A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionellen, Cockerella burnetii, Serratia und Proben beim Menschen dem DETECTR-Test unterzogen.
- Verwenden Sie DNA-Vorlagen, die von verschiedenen Spezies gewonnen wurden, indem Sie das referenzierte DNA-Kit verwenden oder sie isolieren, indem Sie die Bakterien im Wasser kochen. Stellen Sie sicher, dass alle Vorlagen die gleichen Konzentrationen aufweisen. Verwenden Sie Wasser als Negativkontrolle.
- Geben Sie 10 ng oder 1 μl DNA-Templates zu Lösung A, die F3R3-Primer enthält. 2,5 μl MgOAc zugeben, gründlich mischen und die Mischung 20 Minuten lang bei 39 °C inkubieren.
- Nach 20 Minuten 5 μl des Produkts aus dem vorherigen Schritt zu Lösung B geben und mischen.
- Legen Sie es in das quantitative Fluoreszenz-PCR-Instrument, stellen Sie den Kanal auf FAM und lesen Sie den Wert einmal pro Minute bei 37 °C ab, insgesamt 60 x für 60 Minuten.
3. Bewertung der Sensitivität der RPA-CRISPR/Cas12a-basierten Detektionsplattform
- Als Vorlage für die Reaktion wird ein positives rekombinantes Plasmid verwendet, das auf eine Konzentration von 1-107 Kopien/μl hergestellt wurde. Verwenden Sie Wasser für die Negativkontrolle.
- Führen Sie die Methode aus, indem Sie die Schritte 2.2-2.4 ausführen.
HINWEIS: Das Primer-Set RPA-F3 und RPA-R3 wurde entwickelt, um ein kürzeres Fragment von 174 Basenpaaren aus dem positiven rekombinanten Plasmid zu amplifizieren.
4. Vorbereitungen, während die Probe unter UV-Licht und LFS-Detektion steht
- Nehmen Sie die benötigte Anzahl an Teststreifen heraus und beschriften Sie diese entsprechend.
- Übertragen Sie 10 μl der amplifizierten Produkte aus Schritt 2 und 3 in separate PCR-Röhrchen. Geben Sie dann 40 μl ddH2O in jedes Röhrchen.
HINWEIS: Bei Verwendung eines selbst vorbereiteten Reaktionssystems wird empfohlen, das optimale Verdünnungsverhältnis zu untersuchen. - Führen Sie den Test durch.
- Das PCR-Röhrchen mit den amplifizierten Produkten wird einem Transilluminator unterzogen, um das Vorhandensein von Fluoreszenz zu beurteilen.
- Führen Sie die Teststreifen mit dem Ende des Probenpads nach unten in die PCR-Röhrchen ein.
- Lassen Sie es 5-10 min bei Raumtemperatur stehen, um das Ergebnis abzulesen. Stellen Sie nach dieser Zeit sicher, dass der Flüssigkeitsstand die maximale Linie nicht überschreitet, bevor Sie fortfahren.
- Interpretieren Sie die Ergebnisse als negativ, wenn die T-Linie keine Farbe anzeigt. Interpretieren Sie die Ergebnisse als positiv, wenn die T-Linie mit bloßem Auge sichtbar ist, was darauf hinweist, dass die Nukleinsäuresonde durch das Cas-Enzym gespalten und das Cas-Enzym aktiviert wurde. Betrachten Sie das Ergebnis als ungültig, wenn weder die C-Linie noch die T-Linie Farbe aufweist.
Ergebnisse
In dieser Studie stellen wir eine neuartige, tragbare Diagnoseplattform namens A. baumannii-DETECTR vor, die die isotherme Verstärkungseffizienz von RPA- und CRISPR-Cas12a-Systemen zur schnellen und zuverlässigen Feldidentifizierung von A. baumannii integriert. Das Schema des DECERT-Assays ist in Abbildung 1 dargestellt.
Zwölf Paar Primer wurden mit Hilfe von Designwerkzeugen auf der Grundlage von Primer-Designprinzipien entworfen. Es wurden vier Vorwärts-Primer (F0-F3) und drei Reverse-Primer (R1-R3) erhalten, was zu 12 Primerpaaren führte (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3). Nach der RPA-Amplifikation bei 39 °C für 20 min wurden die RPA-PCR-Produkte 10 Minuten lang auf einem 1%igen Agarosegel bei 120 V analysiert, um die optimale Primerkombination zu bestimmen. Das Primerpaar F3R3 erwies sich als das effektivste und wies die hellsten und dichtesten Bänder auf (Abbildung 2).
Nachdem wir das optimale Primerpaar F3R3 identifiziert hatten, optimierten wir als Nächstes die endgültigen Primerkonzentrationen und Reaktionszeiten für die RPA-Amplifikation, wobei alle anderen Komponenten konstant blieben. Unsere Ergebnisse zeigten, dass die Amplifikationseffizienz bei einer Primerkonzentration von 0,44 μM und einer Reaktionszeit von 20 min ihren Höhepunkt erreichte (Abbildung 3). Unter Verwendung eines positiven rekombinanten Plasmids in einer Konzentration von 107 Kopien/μl als Template beobachteten wir einen zeitabhängigen Anstieg der Fluoreszenzintensität für crRNA-1, während crRNA-2 im Laufe der Zeit keinen solchen Anstieg zeigte. Daher wurde crRNA-1 als crRNA für den Nachweis von A. baumannii ausgewählt (Abbildung 4).
Die Spezifität von A. baumannii-DETECTR wurde anhand von Nukleinsäuren untersucht, die aus einer Vielzahl von Bakterienstämmen isoliert wurden. Während der PCR-Assays wurde ein signifikantes Fluoreszenzsignal nur beobachtet, wenn das Genom von A. baumannii angegriffen wurde, während bei der Verwendung genomischer DNA aus anderen bakteriellen Quellen keine Fluoreszenz nachgewiesen wurde (Abbildung 5). Diese Ergebnisse bestätigen, dass das Primerpaar A. baumannii-F3 und R3 im DETECTR-Assay Spezifität für A. baumannii aufweist, was darauf hindeutet, dass A. baumannii-DETECTR diesen Erreger zuverlässig nachweisen kann.
Um die Sensitivität des A. baumannii-DETECTR-Assays zu beurteilen, führten wir eine Reihe von Verdünnungen mit A. baumannii-DNA durch. Die Kopienzahl wurde mit der folgenden Formel berechnet: Kopienzahl (Kopien/μL) = 6,02 × 1023 x P (ng/μL) x 10-9 / (Anzahl der Plasmidbasen x 660) (wobei P die Plasmidkonzentration darstellt). Der Assay demonstrierte die Fähigkeit, bis zu 1 Kopie/μl zu detektieren, was auf sein Potenzial als äußerst empfindliches Diagnosewerkzeug hinweist, das sich gut für Vor-Ort-Anwendungen eignet (Abbildung 6). Damit weist der A. baumannii-DETECTR eine bemerkenswerte Sensitivität auf.
In diesem Protokoll haben wir auch die CRISPR/Cas12a-vermittelte DETECTR-Detektionsplattform mit der Lateral-Flow-Strip-Technologie (LFS) integriert, um die Identifizierung von Ziel-DNA-Sequenzen zu erleichtern. Wir untersuchten die Sensitivität und Spezifität des LFS-basierten Assays. Ein Spezifitätsassay, der mit LFS durchgeführt wurde, zeigte keine Anzeichen einer Kreuzreaktivität mit anderen Bakterienarten, was die hohe Spezifität der Methode belegt (Abbildung 7A). Darüber hinaus verwendeten wir LFS, um A. baumannii über einen Verdünnungsbereich von 10bis 7 Kopien/μl bis hinunter zu einer einzigen Kopie/μl nachzuweisen. Bemerkenswert ist, dass alle getesteten Proben positive Ergebnisse lieferten (Abbildung 7B), die mit den Ergebnissen unter UV-Licht übereinstimmten (Abbildung 7C, D). Zusammengenommen unterstreichen diese Ergebnisse, dass unsere Plattform als schnelles, robustes und sensitives Diagnoseinstrument für A. baumannii im Feld dient.

Abbildung 1: Arbeitsablauf von A. baumannii-DETECTR. Abkürzungen: RPA =R Ecombinase Polymerase Amplifikation; DETECTR = DNA-Endonuklease zielgerichteter CRISPR-Trans-Reporter. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Design und Auswahl der Grundierungen. Die optimale Kombination von Primern für die RPA-Reaktion wurde auf der Grundlage der Ergebnisse der Agarose-Gelelektrophorese identifiziert. Das Primerpaar F3R3 schnitt am besten mit hellen und dichten Bändern ab. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Die Optimierung der Primerkonzentration und der Amplifikationszeit für RPA. 1-4: Die Primerkonzentrationen 0,40 μM, 0,44 μM, 0,48 μM, 0,52 μM. Die Verstärkungszeiten betragen jeweils 10 min, 20 min und 30 min. Der Amplifikationseffekt erreicht seinen höchsten, wenn die Konzentration des Primers 0,44 μM und die Reaktionszeit 20 min beträgt. Abkürzungen: M = 8000 DNA Marker; RPA = Rekombinase-Polymerase-Amplifikation; NC = leere Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
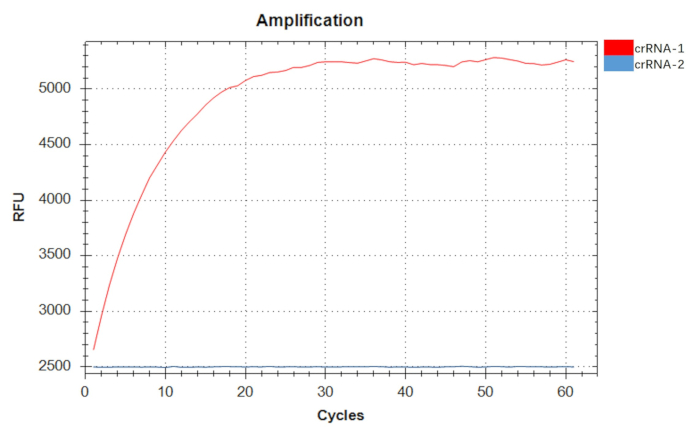
Abbildung 4: Screening von zwei crRNAs (crRNA-1 und crRNA-2). Die Fluoreszenzintensität des crRNA-1-Nachweises nahm mit der Zeit zu, während die des crRNA-2-Nachweises mit der Zeit nicht zunahm und keine signifikante Nachweisaktivität zeigte. crRNA-1 wurde als crRNA für den Nachweis von A. baumannii verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
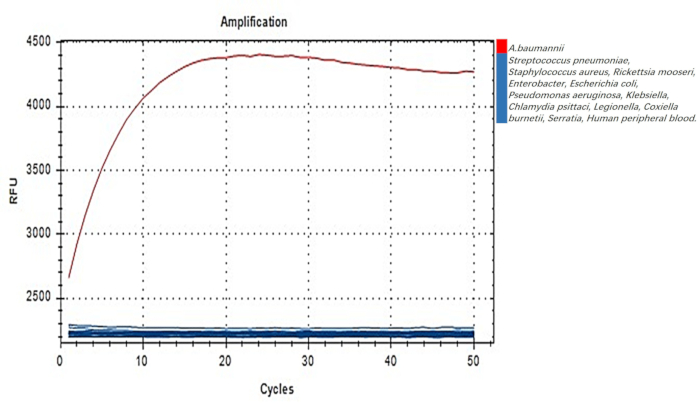
Abbildung 5: Test der Spezifität des A. baumannii-DETECTR. Die Amplifikation von A. baumannii-DETECTR wurde unter Verwendung des Primerpaares A. baumannii-F3 und R3 unter Verwendung genomischer DNA erreicht, die sowohl von A. baumannii als auch von anderen Bakterienarten extrahiert wurde. Abkürzung: DETECTR = DNA Endonuclease Targeted CRISPR Trans Reporter. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
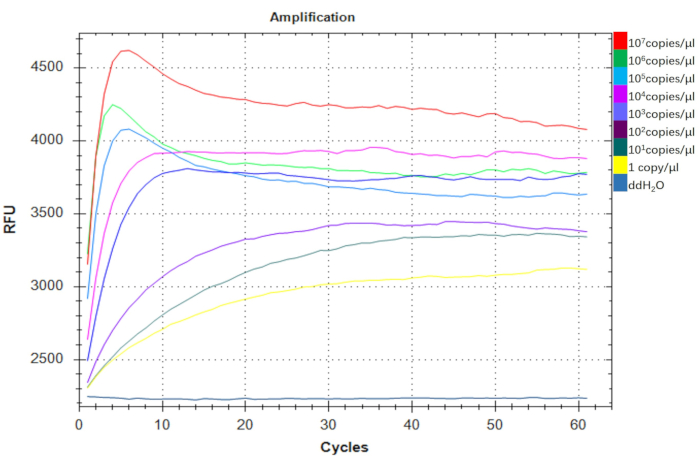
Abbildung 6: Testen der Sensitivität des A. baumannii-DETECTR. Die pUC57-16s rDNA-positiven rekombinanten Plasmide dienten als Targets, um die Nachweisgrenze für A. baumannii mit dem visuellen Fluoreszenz-basierten RPA-CRISPR/Cas12a-Assay zu bewerten. Der Assay konnte einen Bereich von einer Kopie bis 107 Kopien/μl unter Verwendung konsistenter Primer erfolgreich nachweisen. Die Fluoreszenzintensität in der Ein-Kopien-Gruppe war im Vergleich zur ddH2O-Kontrollgruppe signifikant erhöht. Abkürzungen: DETECTR = DNA Endonuclease Targeted CRISPR Trans Reporter; RPA = Rekombinase-Polymerase-Amplifikation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
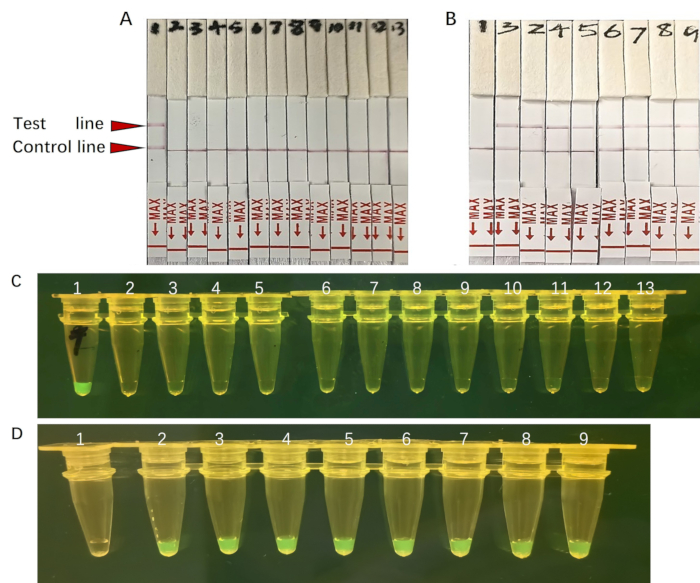
Abbildung 7: Das Fluoreszenzsignal der Probe unter UV-Licht und LFS-Detektion. A. baumannii-DETECTR-Reaktion mit Plasmidstandards. (A-C) Verschiedene Erreger wurden mit A. baumannii-DETECTR nachgewiesen. Nur A. baumannii konnte erfolgreich nachgewiesen werden. 1-13 repräsentieren A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Coxiella burnetii, Serratia, Human peripheral. (B-D) Das positive rekombinante Plasmid mit seriell verdünntem in zehnfachen Schritten, nachgewiesen mit dem A. baumannii-DETECTR. Alle wurden mit Ausnahme der Negativkontrolle nachgewiesen. 1-9 stehen für Negativkontrolle, 1 Kopie/μL, 101 Kopien/μL, 102 Kopien/μL, 103 Kopien/μL, 104 Kopien/μL, 105 Kopien/μL, 106 Kopien/μL, 107 Kopien/μL. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Nein | Name | Sequenz (5'-3') | |||
| 1 | crRNA-1 | uaauuucuacuaaguguagauGUUAAUACCUAGAGAUAGUG | |||
| 2 | crRNA-2 | uaauuucuacuaaguguagauGUUGGUAUUCCGGAAGAAGU | |||
| 3 | crRNA-1-DNA | GTTAATACCTAGAGATAGTG | |||
| 4 | crRNA-2-DNA | GTTGGTATTCCGGAAGAAGT | |||
| 5 | Geplante Sequenz | TAACTGAAGAGTTTGATCATGGCTCAGATTGAACGCTGG CGGCAGGCTTAACACATGCAAGTCGAGCGGGGGAAGGT AGCTTGCTACTGGACCTAGCGGCGGACGGGTGAGTAATG CTTAGGAATCTGCCTATTAGTGGGGGACAACATCTCGAAA GGGATGCTAATACCGCATACGTCCTACGGGAGAAAGCAG GGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCG GATTAGCTAGTTGGTGGGGTAAAGGCCTACCAAGGCGAC GATCTGTAGCGGGTCTGAGAGGATGATCCGCCACACTGG GACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGT GGGGAATATTGGACAATGGGGGGAACCCTGATCCAGCCAT GCCGCGTGTGTGAAGAAGGCCTTATGGTTGTAAAGCACTT TAAGCGAGGAGGAGGCTACTTTAGTTAATACCTAGAGATAG TGGACGTTACTCGCAGAATAAGCACCGGCTAACTCTGTGC CAGCAGCCGCGGTAATACAGAGGGTGCGAGCGTTAATCGG ATTTACTGGGCGTAAAGCGTGCGTAGGCGGCTTATTAAGTC GGATGTGAAATCCCCGAGCTTAACTTGGGAATTGCATTCGAT ACTGGTGAGCTAGAGTATGGGAGAGGATGGTAGAATTCCAG GTGTAGCGGTGAAATGCGTAGAGATCTGGAGGAATACCGAT GGCGAAGGCAGCCATCTGGCCTAATACTGACGCTGAGGTAC GAAAGCATGGGGAGCAAACAGGATTAGATACCCTGGTAGTC CATGCCGTAAACGATGTCTACTAGCCGTTGGGGCCTTTGAGG CTTTAGTGGCGCAGCTAACGCGATAAGTAGACCGCCTGGGGA GTACGGTCGCAAGACTAAAACTCAAATGAATTGACGGGGGCC CGCACAAGCGGTGGAGCATGTGGTTTAATTCGATGCAACGC GAAGAACCTTACCTGGCCTTGACATACTAGAAACTTTCCAGA GATGGATTGGTGCCTTCGGGAATCTAGATACAGGTGCTGCAT GGCTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCC CGCAACGAGCGCAACCCTTTTCCTTACTTGCCAGCATTTCG GATGGGAACTTTAAGGATACTGCCAGTGACAAACTGGAGGAA GGCGGGGACGACGTCAAGTCATGGCCCTTACGGCCAGG GCTACACACGTGCTACAATGGTCGGTACAAAGGGTTGCTACA CAGCGATGTGATGCTAATCTCAAAAAGCCGATCGTAGTCCGG ATTGGAGTCTGCAACTCGACTCCATGAAGTCGGAATCGCTAG TAATCGCGGATCAGAATGCCGCGGTGAATACGTTCCCGGGCC TTGTACACACCGCCCGTCACACCATGGGAGTTTGTTGCACCA GAAGTAGCTAGCCTAACTGCAAAGAGGGCGGTTACCACGGTG TGGCCGATGACTGGGGTGAAGTCGTAACAAGGTAGCCGTAGG GGAACCTGCGGCTGGATCACCTCCTTA | |||
| 6 | A.baumannii-F0 | CGGGAGGCAGCAGTGGGGAATATTGGACAAT | |||
| 7 | A.baumannii-F1 | AAGAAGGCCTTATGGTTGTAAAGCACTTTAA | |||
| 8 | A.baumannii -F2 | GGTTGTAAAGCACTTTAAGCGAGGAGGAGGCTA | |||
| 9 | A.baumannii -F3 | AAGGCCTTATGGTTGTAAAGCACTTTAAGCGA | |||
| 10 | A.baumannii-R1 | ACAGAGTTAGCCGGTGCTTATTCTGCGAGTAA | |||
| 11 | A.baumannii-R2 | AATCCGATTAACGCTCGCACCCTGTATTA | |||
| 12 | A.baumannii-R3 | AGTTAGCCGGTGCTTATTCTGCGAGTAACGTC | |||
| 13 | ssDNA-FQ | ssDNA-FQ FAM-TTTATTT-BHQ1 | |||
| 14 | A.baumannii-16s rDNA-F | 5′-CGCGGATCCTAACTGAAGAGTTTGATCATGG-3′ | |||
| 15 | A.baumannii-16s rDNA-R | 5′-ACGCGTCGACTAAGGAGGTGATCCAGC-3′. | |||
Tabelle 1: In dieser Studie verwendete Nukleinsäuren. Bei der hier verwendeten Nukleinsäure handelt es sich um ssDNA-FQ, die aus einzelsträngiger DNA besteht, die am 5'-Ende mit einem 5-Carboxyfluorescein und am 3'-Ende mit dem Fluoreszenzquencher Black Hole Quencher 1 konjugiert ist. Abkürzungen: F = Fluorophor; Q = Quencher.
Ergänzende Datei 1: Design von Primern und crRNA.Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Traditionelle Diagnosemethoden für A . baumannii-Infektionen unterliegen verschiedenen Einschränkungen, die sie für Point-of-Care-Tests weniger zugänglich und praktikabel machen27. Zum Beispiel hat die PCR eine gute Sensitivität und Spezifität, erfordert aber spezielle Geräte für den thermischen Kreislauf und komplexe Arbeitsabläufe, die von Fachleuten bedient werden müssen. Die hier vorgestellte neuartige Methode, die RPA- und CRISPR-LbaCas12a-Genom-Editing mit Rekombinierung koppelt, der sogenannte A. baumannii-DETECTR-Assay, ermöglicht eine effiziente genetische Manipulation. Es bietet eine schnelle, empfindliche und spezifische Nachweismethode für Acinetobacter baumannii-Infektionen sowie für die Diagnose anderer bakterieller Krankheitserreger. Der Workflow ist in Abbildung 1 dargestellt.
Unsere Methode ist besonders vorteilhaft bei der Diagnose und Behandlung von Infektionen, was die Genesung der Patienten erleichtern und die Ausbreitung von A. baumannii eindämmen soll, indem die Unzulänglichkeiten bestehender Diagnosetechniken überwunden werden. Es wurde festgestellt, dass das A. baumannii-DETECTR-System A. baumannii mit hoher Spezifität und Sensitivität identifizieren konnte (Abbildung 5 und Abbildung 6). Unsere Ergebnisse zeigten, dass nur das Genom von A. baumannii während der Echtzeitbewertung eine positive Änderung der Fluoreszenzfaltung aufwies, während die Genome aller anderen Spezies negative Ergebnisse zeigten (Abbildung 5).
Um die Sensitivität der RPA-CRISPR/Cas12a-Detektionsplattform zu bewerten, wurden rekombinante Plasmide pUC57-16s rDNA, die positiv für A. baumannii sind, als Targets zur Bestimmung der Nachweisgrenze (LOD) des Systems verwendet. Im fluoreszenzbasierten RPA-CRISPR/Cas12a-Assay reichte die Detektion von einer Kopie pro Mikroliter bis zu 107 Kopien pro Mikroliter, wobei in der 1-Kopien/μl-Gruppe im Vergleich zur negativen Kontrollgruppe eine bemerkenswerte Verbesserung der Fluoreszenzintensität beobachtet wurde (Abbildung 6). Wichtig für die Echtzeitdiagnose ist, dass die Ergebnisse der Erkennung direkt über ein tragbares Handgerät visualisiert werden können, unabhängig von Laborgeräten, was für die Echtzeitdiagnose unerlässlich ist.
In Bezug auf das Protokoll müssen mehrere kritische Faktoren berücksichtigt werden26. Erstens ist es wichtig, Kreuzkontaminationen während des Probenentnahmeprozesses zu verhindern, da der A. baumannii-DETECTR-Assay sehr empfindlich ist. Zusätzlich sollten die Reaktionsreagenzien vor direkter Sonneneinstrahlung geschützt werden. Die Einhaltung aller Verfahrensschritte ist entscheidend, um das bestmögliche Ergebnis zu erzielen. Eine Positivkontrolle, bestehend aus einem Plasmid, das A . baumannii-spezifische Genfragmente enthält, sollte eingebaut werden, um die ordnungsgemäße Funktionalität des A. baumannii-DETECTR-Systems in praktischen Anwendungen zu überprüfen.
Das Protokoll zeigt, dass Parameter für bestimmte Laborbedingungen und Probentypen optimiert werden können, wie z. B. Primerkonzentration, Reaktionszeit und andere Variablen. In diesem Experiment wurden die optimale Primerkombination, die Primerkonzentrationen und die Reaktionszeiten für die RPA-Amplifikation ausgewählt, um die höchste Amplifikationseffizienz und Spezifität zu erzielen. Dies wurde mittels Agarose-Gelelektrophorese bestimmt (Abbildung 2 und Abbildung 3). Zusätzlich wurde ein positives rekombinantes Plasmid, das eine konservierte Region des A . baumannii-Genoms enthält, als Kontrolle verwendet, um die Genauigkeit des Protokolls zu gewährleisten. Es ist erwähnenswert, dass kommerzielle bakterielle genomische DNA-Extraktionskits eine standardisierte und zuverlässige Methode für die DNA-Extraktion in diesem Protokoll bieten. In zukünftigen Studien könnten jedoch verschiedene Probentypen, wie z. B. klinische oder Umweltproben, spezifische Extraktionsmethoden28 erfordern, um A . baumannii-DNA wirksam zu isolieren.
In Ermangelung nachweisbarer Fluoreszenzsignale von der Positivkontrolle ist es wahrscheinlich, dass Inhibitoren in dem Reaktionsgemisch vorhanden sind, was einen Wechsel der Reagenzien erforderlich macht. Ist die Fluoreszenzintensität für eine klare Differenzierung nicht ausreichend, kann eine Verlängerung der Inkubationsdauer von Vorteil sein. Eine längere Inkubation kann jedoch auch zu falsch positiven Ergebnissen führen26.
Die derzeitigen Nachweismethoden für A. baumannii erfordern den Einsatz teurer PCR-Geräte, fester Einsatzorte und spezialisiertes Personal. Im Gegensatz dazu ermöglicht die hier vorgeschlagene Methode eine genaue und empfindliche Diagnose von A. baumannii im Freiland und macht sie für eine breitere Bevölkerungsgruppe zugänglich.
Die Eigenschaften der isothermen Amplifikation und die durch Fluoreszenz gewonnenen Daten machen den Arbeitsablauf von A. baumannii-DETCTER einfach und stellen geringe Anforderungen an die Ausrüstung und ermöglichen einen schnellen Erregernachweis in abgelegenen Gebieten oder bei Ausbrüchen. RPA-LFS 29,30,31,32, eine neuartige isotherme Nukleinsäure-Amplifikationstechnik, hat aufgrund ihrer einfachen Bedienung und des geringeren Zeitaufwands beim Nachweis von viralen, bakteriellen, pilzlichen und parasitären Krankheitserregern erheblich an Bedeutung gewonnen. Das Protokoll beschreibt das Fluoreszenzsignal der Probe unter ultraviolettem (UV) Licht oder Transilluminator- und Lateral-Flow-Strips-Detektion (LFS). Es kann die Abhängigkeit von hochpräzisen Instrumenten und spezialisierten Technikern vermeiden, da die visuelle Inspektion in kurzer Zeit erfolgt (Abbildung 7), wodurch der Bedarf an teuren und komplexen Laborgeräten und der Betriebsbevölkerung entfällt. Darüber hinaus ist die Ein-Röhrchen-Methode nicht nur zeitsparend, sondern verringert auch das Risiko einer Amplikonkontamination33.
Zukünftige Studien sollten darauf abzielen, die Methode zu verbessern, um sie für verschiedene Arten von Proben und unterschiedliche Umweltbedingungen nutzbar zu machen, z. B. durch die Erforschung effizienterer und kostengünstigerer DNA-Extraktionstechniken. Die Verwendung von A. baumannii-DETECTR dient nicht nur der klinischen Diagnose, sondern kann auch zur Umweltbewertung, Überwachung und zum Management von Ausbrüchen verwendet werden, so dass Gemeinden und Krankenhäuser A . baumannii schnell identifizieren können. Darüber hinaus kann dieser molekulardiagnostische Ansatz durch Modifikation der crRNA und der Primer für den Nachweis verschiedener bakterieller Atemwegsinfektionen wie Streptococcus pneumoniae, Staphylococcus aureus und Enterobacter angepasst werden.
Zusammenfassend lässt sich sagen, dass A. baumannii-DETECTR A . baumannii aufgrund seiner hohen Sensitivität und Spezifität, seines benutzerfreundlichen Designs und seiner Portabilität so schnell und genau wie möglich identifizieren kann. Diese Methode kann die Gesundheitsergebnisse der Patienten verbessern, indem sie eine rechtzeitige und wirksame Behandlung, Infektionskontrolle und die Eindämmung der Ausbreitung von Antibiotikaresistenzen erleichtert und gleichzeitig die zunehmende Bedrohung durch A . baumannii-Infektionen bekämpft.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden Interessen haben.
Danksagungen
Diese Arbeit wurde unterstützt durch das Projekt für den Wissenschafts- und Technologieentwicklungsplan der Provinz Jilin, China (20240305027YY) und das Finanzministerium der Provinz Jilin (JLSWSRCZX2023-55, JLSWSRCZX2021-041).
Materialien
| Name | Company | Catalog Number | Comments |
| -20 °C Freezer | Haier | HYCD-290 | China |
| Agarose Basic | BioFroxx | 1110GR100 | China |
| All oligonucleotides and crRNA were synthesized by company | www.comatebio.com | ||
| Cas12a cutting substrate - ssDNA - fluorescent type | EZassay Biotech. Co. Ltd. | DNA-FAM-BHQ | China |
| Cas12a cutting substrate - ssDNA - test paper type | EZassay Biotech. Co. Ltd. | DNA-FAM-BIO | China |
| Electrophoresis apparatus | BIO-RAD | POWER PAC1000 | USA |
| Fluorescence quantitative PCR instrument | BIO-RAD | CFX Connect | USA |
| Gel Imaging System | BIO-RAD | Gel Doc 2000 | USA |
| https://ezassay.com/rna | |||
| https://www.ezassay.com/primer | |||
| https://www.ncbi.nlm.nih.gov/tools/primer-blast | |||
| Lateral flow paper strip (Biotin/FAM) | EZassay Biotech. Co. Ltd. | HD-FMBO | China |
| LbaCas12a (Cpf1) enhanced protein | EZassay Biotech. Co. Ltd. | CAS-12E-001 | China |
| LED Transilluminator | LABGIC | BL-20 | China |
| Magnesium acetate, MgOAc | TwistDx | TABAS03KIT | UK |
| Microcentrifuge | allsheng | Mini-6k | China |
| PCR strip tubes | PCR strip tubes | PST-0208-FT-C | China |
| TGrade Dry Bath Incubator | Tiangen biochemical technology | OSE-DB-01 | China |
| Tianamp Bacteria DNA Kit | Tiangen biochemical technology | DP302-02 | China |
| TIANamp Bacteria DNA Kit | TIANGEN BIOTECH (BEIJING) CO.; LTD. | DP302 | China |
| TransStart FastPfu DNA Polymerase | TransGen Biotech. Co. Ltd. | AP221 | China |
| TwistAmp Basic Kit | TwistDX | TABAS03KIT | UK |
| Universal DNA Purification Kit | Tiangen biochemical technology | DP214-03 | China |
Referenzen
- Lenie, D., Alexandr, N., Harald, S. An increasing threat in hospitals: Multidrug-resistant Acinetobacter baumannii. Nat Rev Microbiol. 5 (12), 939-951 (2007).
- Li, P., et al. Rapid detection of Acinetobacter baumannii and molecular epidemiology of carbapenem-resistant A. baumannii in two comprehensive hospitals of Beijing, China. Front Microbiol. 6, 997(2015).
- Wu, X., et al. A diagnostic test that uses isothermal amplification and lateral flow detection sdaA can detect tuberculosis in 60 min. J Appl Microbiol. 130 (6), 2102-2110 (2020).
- Huang, B., et al. A cas12a-based fluorescent microfluidic system for rapid on-site human papillomavirus diagnostics. Appl Microbiol Biotechnol. 107 (20), 6287-6297 (2023).
- Piepenburg, O., Williams, C. H., Stemple, D. L., Armes, N. A. DNA detection using recombination proteins. Plos Biology. 4 (7), 1115-1121 (2006).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), aaf5573(2016).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), eaar6245(2018).
- Xiong, D., et al. Rapid detection of SARS-CoV-2 with CRISPR-Cas12a. PLoS Biol. 18 (12), e3000978(2020).
- Gootenberg, J. S., et al. Nucleic acid detection with crispr-Cas13a/C2C2. Science. 356 (6336), 438-442 (2017).
- Harrington, L. B., et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science. 362 (6416), 839-842 (2018).
- Li, S. Y., et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA. Cell Res. 28 (4), 491-493 (2018).
- Wang, B., et al. Cas12aVDet: A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem. 91 (19), 12156-12161 (2019).
- Huang, Z., et al. Ultra-sensitive and high-throughput crispr-p owered COVID-19 diagnosis. Biosens Bioelectron. 164, 112316(2020).
- Zhang, W. S., et al. Reverse transcription recombinase polymerase amplification coupled with CRISPR-Cas12a for facile and highly sensitive colorimetric SARS-CoV-2 detection. Anal Chem. 93 (8), 4126-4133 (2021).
- Marraffini Luciano, A. CRISPR-Cas immunity in prokaryotes. Nature. 526 (7571), 55-61 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), aad5147(2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 13 (11), 722-736 (2015).
- Li, S. Y., et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discovery. 4 (1), 20(2018).
- Li, L., Li, S., Wu, N., Wu, J., Wang, J. Holmesv2: A CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol. 8 (10), 2228-2237 (2019).
- Liang, M., Li, Z., Wang, W., Liu, J., Zhang, L. X. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules. Nat Communications. 10 (1), 3672(2019).
- Aquino-Jarquin, G. CRISPR-Cas14 is now part of the artillery for gene editing and molecular diagnostic. Nanomedicine. 18, 428-431 (2019).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Swarts, D. C., John, V. D. O., Jinek, M. Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a. Mol Cell. 66 (2), 221-233 (2017).
- Jiang, Y., et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun. 8, 15179(2017).
- Yan, M. Y., et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol. 83 (17), e00947-e01017 (2017).
- Ashraf, A., et al. A novel multiplex pcr assay for simultaneous detection of nine clinically significant bacterial pathogens associated with bovine mastitis. Mol Cell Probes. 33, 57-64 (2017).
- Zhu, L., et al. A rapid on-site visualization platform based on RPA coupled with CRISPR-Cas12a for the detection of genetically modified papaya 'huanong no.1'. Talanta. 277, 126437(2024).
- Zheng, C., et al. Rapid developments in lateral flow immunoassay for nucleic acid detection. Analyst. 146 (5), 1514-1528 (2021).
- Wang, Y., et al. Establishment and clinical application of a RPA-LFS assay for detection of capsulated and non-capsulated Haemophilus influenzae. Front Cell Infect Microbiol. 12, 878813(2022).
- Wang, F., et al. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strips for detecting Candida albicans. Anal Biochem. 633, 114428(2021).
- Ma, B., et al. A simple and efficient method for potential point-of-care diagnosis of human papillomavirus genotypes: Combination of isothermal recombinase polymerase amplification with lateral flow dipstick and reverse dot blot. Anal Bioanal Chem. 411 (28), 7451-7460 (2019).
- Sun, Y., Yu, L., Liu, C., Ye, S., Huang, W. One-tube SARS-CoV-2 detection platform based on RT-RPA and CRISPR/Cas12a. J Transl Med. 19 (1), 74(2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten