Method Article
Detecção rápida e específica de infecções por Acinetobacter baumannii usando um sistema baseado em amplificação da polimerase recombinase/Cas12a
Neste Artigo
Resumo
Para facilitar a detecção rápida e precisa de Acinetobacter baumannii, apresentamos um protocolo que emprega a amplificação da polimerase recombinase (RPA) em conjunto com a endonuclease LbaCas12a para identificar infecções por A. baumannii .
Resumo
Acinetobacter baumannii, uma bactéria gram-negativa, é notória por causar infecções graves com altas taxas de mortalidade. A detecção rápida e precisa de A. baumannii é crucial para o tratamento imediato, controle eficaz de infecções e redução da resistência a antibióticos. No entanto, não existe um método adequado para detecção rápida e fácil no local de A. baumannii. O sistema DNA Endonuclease Targeted CRISPR Trans Reporter (DETECTR) oferece uma abordagem rápida, precisa e sensível para a detecção de A. baumannii , integrando os recursos de reconhecimento específicos do alvo de Cas12a com a eficiência de amplificação isotérmica da amplificação da polimerase recombinase (RPA). Este protocolo detalha a detecção de A. baumannii usando RPA combinada com endonuclease LbaCas12a. As seguintes etapas são descritas neste artigo: extração de DNA, seleção de uma sequência específica de DNA, projeto de primer e RNA CRISPR (crRNA), construção de plasmídeo recombinante positivo, configuração do ensaio Cas12a-RPA, otimização do sistema de amplificação RPA, visualização do ensaio RPA-CRISPR/Cas12a usando uma ferramenta de detecção de fluorescência, como um instrumento de PCR em tempo real, e avaliação da sensibilidade e especificidade.
Introdução
Em microbiologia clínica, a detecção de infecções por Acinetobacter baumannii apresenta um desafio significativo. Essa bactéria gram-negativa pode causar infecções com sintomas clínicos graves, mesmo com altas taxas de mortalidade, principalmente entre pacientes imunocomprometidos1. Os métodos tradicionais de detecção baseados em cultura para infecções por patógenos são demorados e podem não ter sensibilidade, o que pode atrasar o tratamento com antibióticos e comprometer os resultados dos pacientes. A identificação rápida e precisa de A. baumannii é crucial para o tratamento eficaz e o controle de surtos. Técnicas moleculares que empregam amplificação de ácidos nucleicos têm sido exploradas para atender a essa necessidade. No entanto, essas abordagens geralmente precisam de equipamentos sofisticados de ciclagem térmica e podem ser limitadas por técnicos bem treinados e laboratórios bem estabelecidos. Para superar esses desafios, a pesquisa está cada vez mais focada no desenvolvimento de métodos de amplificação isotérmica 2,3,4.
A amplificação da polimerase recombinase (RPA) é um método estabelecido por Piepenburg et al 5 e usado para amplificar o DNA, semelhante à reação em cadeia da polimerase (PCR), mas sem a necessidade de ciclos de temperatura. Este método inclui 50 mM de Tris (pH 7,5), 100 mM de acetato de potássio, 14 mM de acetato de magnésio, 2 mM de DTT, 5% de PEG20000 (um polietilenoglicol de alto peso molecular), 200 μM dNTPs, 3 mM de ATP, 50 mM de fosfocreatina, 100 μg/mL de creatina quinase, 120 μg/mL de UvsX, 30 μg/mL de UvsY, 900 μg/mL de Gp32, 30 μg/mL de Bsu LF, Primers de 450 nM e modelos de DNA. O processo de amplificação começa com a proteína recombinase UvsX ligando-se aos primers na presença de 3 mM de ATP e 5% PEG20000 formando um complexo recombinase-primer. Este complexo facilita então a combinação de primers com as sequências homólogas no DNA de fita dupla.
Com a ajuda do UvsY, a recombinase UvsX facilita a troca entre o primer e a fita molde, resultando no deslocamento de uma fita do DNA alvo. A gp32 ajuda a manter a estrutura do DNA de fita simples. Finalmente, a recombinase se dissocia e uma DNA polimerase (Bsu LF) capaz de deslocar as fitas de DNA se liga à extremidade 3 'do primer, alongando-a na presença de trifosfatos desoxirribonucleosídeos (dNTPs). Este processo é repetido ciclicamente, alcançando amplificação exponencial. Todos os processos de amplificação podem ser concluídos em 20-40 min e em temperaturas relativamente constantes entre 37 °C e 42 °C. Essa faixa de temperatura é aproximadamente idêntica às temperaturas fisiológicas, o que permite que a RPA seja conduzida em um ambiente minimalista, tornando a RPA uma ferramenta versátil e eficiente para detecção e análise de DNA.
O sistema de repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) e proteínas associadas a CRISPR (CRISPR-Cas) funciona como um mecanismo imunológico adaptativo em bactérias e archaea, abrangendo sistemas de Classe I e Classe II. A classe II inclui proteínas como Cas12 (a, b, f), Cas13 (a, b) e Cas14, que identificam e clivam o DNA ou RNA alvo guiado pelo RNA CRISPR (crRNA)6,7,8. LbaCas12a (ou Cpf1) é uma endonuclease de DNA guiada por crRNA. O crRNA serve como um RNA guia dentro do sistema CRISPR-Cas, onde se complexa com as proteínas Cas. Ele aproveita sua região espaçadora para emparelhar com o DNA alvo, direcionando efetivamente o complexo de proteínas para as sequências alvo. A sequência 5'-UAAUUUCUACUAAGUGUAGAU-3' serve como a repetição conservada de crRNA, um elemento constante em todos os crRNAs LbaCas12a. Após essa repetição, há um segmento específico do alvo que difere com base no alvo de DNA pretendido. Essa sequência de direcionamento varia de 18 a 24 nucleotídeos de comprimento.
A sequência PAM (Protospacer-Adjacent Motif) (TTTV, onde V pode ser A, C ou G) está localizada na extremidade 5' da fita não complementar do alvo de DNA. O Cas12a corta o DNA de fita dupla alvo na sequência PAM. Posteriormente, as proteínas Cas ativadas executam transclivagem não específica de DNA de fita simples (ssDNA) 9 , 10 , 11 , 12 . Assim, o ssDNA marcado com um fluoróforo e um supressor sofre clivagem, resultando na emissão de um sinal fluorescente após a clivagem colateral 8,13. A intensidade da fluorescência pode ser quantificada usando um leitor de fluorescência para detecção precisa de ácidos nucleicos14. Notavelmente, Cas12a é amplamente utilizado para análise de DNA, com sua ligação e clivagem de sequências-alvo dependentes do reconhecimento do Motivo Adjacente ao Protoespaçador (PAM), enquanto Cas13a opera independentemente de tal requisito.
Os sistemas CRISPR-Cas 15,16,17 facilitam a clivagem em uma única reação 18,19,20,21. A combinação dos dois acima pode criar uma ferramenta robusta conhecida como A. baumannii-DETECTR (LbaCas12a-Enabled Detection of Targeted A. baumannii) para detecção de ácido nucleico 22,23,24. Este protocolo demonstra o uso de RPA em conjunto com a atividade DNase de Cas12a para direcionar e clivar especificamente uma sequência guiada por crRNA dentro do gene 16s rDNA de A. baumannii, permitindo assim a detecção sensível e precisa do patógeno.
O método A. baumannii-DETECTR apresenta várias vantagens em relação aos métodos convencionaisde detecção 25,26. Em primeiro lugar, a característica isotérmica do RPA agiliza os procedimentos operacionais e reduz os custos por meio de seu mecanismo de amplificação independente do termociclador5. Em segundo lugar, a endonuclease Cas12a aumenta a especificidade do ensaio por meio de direcionamento mediado por crRNA e emparelhamento de bases Watson-Crick7. Por fim, o uso do método de um tubo de A. baumannii-DETECTR não apenas economiza tempo, mas também reduz significativamente o risco de contaminação do amplicon19.
O protocolo descreve as etapas para a extração de DNA, a seleção de sequências de DNA alvo, o design de primers e crRNAs, a construção de plasmídeos recombinantes positivos, o estabelecimento da configuração do ensaio Cas12a-RPA, a otimização da otimização da amplificação de RPA e a visualização dos resultados com o uso de ferramentas de instrumentos de detecção de fluorescência, como uma máquina de PCR em tempo real, completo com avaliações de sensibilidade e especificidade.
O método A. baumannii-DETECTR é promissor para melhorar os resultados dos pacientes, facilitando o tratamento oportuno e eficaz, o controle robusto da infecção e a contenção da disseminação da resistência aos antibióticos. Este protocolo pode servir como uma diretriz abrangente para a implementação da tecnologia Cas12a-RPA, aumentando sua utilidade em vários ambientes de saúde. No entanto, é crucial observar que cada etapa deve ser otimizada com base nas condições laboratoriais específicas e na variedade de amostras para garantir resultados consistentes e confiáveis.
Protocolo
1. Construção do A. baumannii -DETECTR
NOTA: A construção de A. baumannii-DETECTR é um processo de quatro etapas envolvendo o projeto de primer e crRNA, a construção de plasmídeo recombinante positivo, a preparação de soluções de reação, amplificação isotérmica de DNA por RPA e a visualização do ensaio RPA-CRISPR/Cas12a. O esquema do ensaio DETECTR é ilustrado na Figura 1.
- Desenho do primer e do RNAcr (ver Arquivo Suplementar 1)
- Projete 12 pares de primers usando ferramentas de design com base nos princípios de design do Primer. Obtenha quatro primers diretos (F0-F3) e três primers reversos (R1-R3) para compor doze pares de primers (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- Certifique-se de que a extremidade 5' do primer contenha bases C ou T nos primeiros 3-5 nucleotídeos em vez de um trecho G contínuo. Além disso, certifique-se de que os últimos três nucleotídeos na extremidade 3 'sejam idealmente bases G e C para aumentar a amplificação.
- Projete primers sem sequências auto-complementares para evitar a formação de grampos de cabelo e com não mais do que quatro bases complementares ou homólogas entre eles. Evite a complementaridade da extremidade 3' para evitar a dimerização do primer.
- Avalie cada par de primers quanto à especificidade usando a ferramenta Primer-BLAST do National Center for Biotechnology Information (NCBI) para avaliar a eficácia e especificidade dos primers.
NOTA: O Primer-BLAST pode utilizar o banco de dados do NCBI para encontrar sequências que correspondam aos primers projetados, ajudando os usuários a avaliar a eficácia e a especificidade dos primers. - Após a triagem do primer, projete o RNA CRISPR (crRNA) ou RNA guia (gRNA) usando a sequência de andaime de crRNA LbaCas12a (5'-UAAUUUCUACUAAGUGUAGAU-3') como a sequência fixa. Incorpore um segmento específico do alvo (5'-GUUAAUACCUAGAGAUAGUG-3'), garantindo que o motivo PAM esteja localizado na extremidade 5' da fita não complementar.
NOTA: As sequências de todos os oligonucleotídeos e crRNA são apresentadas na Tabela 1.
- Projete 12 pares de primers usando ferramentas de design com base nos princípios de design do Primer. Obtenha quatro primers diretos (F0-F3) e três primers reversos (R1-R3) para compor doze pares de primers (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- Construção de plasmídeo recombinante positivo
- Amplifique o segmento de rDNA 16s usando o conjunto de primers A. baumannii 16s rDNA-F (primer direto) e A. baumannii 16s rDNA-R (primer reverso).
- Adicione 1 μL de 10 μM F e 10 μM R, 10 μL de tampão 5x FastpFu, 1 μL de FastpFu Polimerase, 4 μL de 2,5 mM dNTPs, 1 μL de 10 ng/μL de DNA genômico de A. baumannii e 32 μL de água livre de nuclease ao sistema de PCR de 50 μL.
- Defina as condições de ciclo de PCR da seguinte forma: desnaturação inicial a 95 °C por 5 min, seguida por 33 ciclos de desnaturação a 95 °C por 30 s, recozimento a 55 °C por 30 s e extensão a 72 °C por 1 min e 20 s. Por fim, execute 10 min a 72°C para a extensão final.
- Para purificar o produto de PCR positivo, use um Kit de Purificação de DNA de acordo com as instruções do fabricante. Em seguida, clone o DNA purificado no vetor pUC57, que foi digerido com as enzimas BamHI e SalI.
NOTA: Este processo resultará na criação de um plasmídeo recombinante positivo contendo uma região conservada. - Diluir o plasmídeo recombinante positivo em série em incrementos de dez vezes com ddH2O esterilizado para atingir uma concentração de uma cópia/μL a 107 cópias/μL. Armazenar o plasmídeo diluído a -20 °C para experiências subsequentes.
- Amplifique o segmento de rDNA 16s usando o conjunto de primers A. baumannii 16s rDNA-F (primer direto) e A. baumannii 16s rDNA-R (primer reverso).
- Preparação da solução
- Prepare a Solução A da seguinte forma: para cada reação, adicione 29,5 μL de tampão de reidratação sem primer, 2,4 μL de 10 μM de A. baumannii-F e 10 μM de A. baumannii-R e 12,2μL de ddH2O para atingir um volume final de 46,5 μL. Vórtice e gire brevemente. Em seguida, adicione a mistura de reação aos tubos de reação enzimática do Kit RPA. Pipeta para misturar.
NOTA: Os primers de A. baumannii-F e R são usados no ensaio que tem como alvo o gene A. baumannii . Consulte a Tabela 1 para obter a sequência. - Prepare a Solução B da seguinte forma: para cada reação, adicione 0,24 μL de repórter de ssDNA 10 μM, 2 μL de tampão de reação Cas12a 10x, 1 μL de crRNA 1 μM, 1 μL de Cas12a 1 μM e 10,76μL de ddH2O para atingir um volume final de 15 μL.
NOTA: As sondas (repórter ssDNA) foram compradas e consistiam em um repórter de DNA de fita simples que foi marcado com um fluoróforo de 5-carboxifluoresceína (FAM) na extremidade 5 'e um quencher Black Hole Quencher 1 (BHQ1) na extremidade 3 '. No caso de numerosas amostras que necessitem de detecção, preparar uma quantidade substancial da solução B e, em seguida, dividir as amostras em alíquotas posteriormente.
- Prepare a Solução A da seguinte forma: para cada reação, adicione 29,5 μL de tampão de reidratação sem primer, 2,4 μL de 10 μM de A. baumannii-F e 10 μM de A. baumannii-R e 12,2μL de ddH2O para atingir um volume final de 46,5 μL. Vórtice e gire brevemente. Em seguida, adicione a mistura de reação aos tubos de reação enzimática do Kit RPA. Pipeta para misturar.
- Amplificação isotérmica de DNA (RPA)
- Adicionar 1 μL do plasmídeo recombinante positivo de 107 cópias/μL à solução A com diferentes pares de iniciadores; Homogeneizar. Em seguida, adicione 2,5 μL de acetato de magnésio (MgOAc) 280 mM à solução e misture bem.
NOTA: As reações de RPA começam assim que o MgOAc é adicionado. - Incubar a 39 °C durante 20 min. Após 20 min, purifique os produtos amplificados com diferentes primers usando RPA com um kit de purificação e, em seguida, execute os amplicons limpos em um gel de agarose a 1% a 120 V por 10 min para selecionar a banda mais brilhante e larga como o melhor par de primers.
NOTA: Para um número baixo de cópias do modelo, após 4 min, remova o tubo EP, o vórtice e gire brevemente; em seguida, substitua-o no dispositivo de aquecimento por mais 16 min. Após a amplificação, os tubos devem ser abertos com extremo cuidado para evitar a contaminação das superfícies de trabalho com amplicons. Essa precaução reduzirá o risco de induzir resultados falsos positivos em procedimentos experimentais subsequentes.
- Adicionar 1 μL do plasmídeo recombinante positivo de 107 cópias/μL à solução A com diferentes pares de iniciadores; Homogeneizar. Em seguida, adicione 2,5 μL de acetato de magnésio (MgOAc) 280 mM à solução e misture bem.
- Otimização do sistema de amplificação RPA
NOTA: O par de primers ideal F3R3 selecionado na etapa anterior foi definido para diferentes concentrações finais para a reação de amplificação de RPA para filtrar a concentração e o tempo ideais do primer. NC é um controle em branco para a concentração de primer correspondente.- Manter constantes as concentrações dos outros componentes e o volume total de 46,5 μL na solução A. Ajustar o volume de ddH2O livre de RNase em conformidade para atingir as concentrações finais de 10 μM F3 e 10 μM R3 a 0,40 μM (2,0 μL), 0,44 μM (2,2 μL), 0,48 μM (2,4 μL) e 0,52 μM (2,6 μL), respectivamente.
- Incubar a 39 °C por diferentes momentos: 10 min, 20 min, 30 min.
- Limpe os produtos RPA usando um kit de purificação de DNA e realize eletroforese em um gel de agarose a 1% a 120 V por 10 min para identificar a concentração ideal do primer e os tempos de incubação analisando as características das bandas.
- Visualização do ensaio RPA-CRISPR/Cas12a
- Adicione 5 μL do produto RPA na solução B e misture bem até ficar homogêneo.
- Coloque as amostras no instrumento de PCR quantitativo de fluorescência, defina o canal FAM e leia os valores a cada minuto por 60 min a 37 °C, 60x para um total de 60 min.
NOTA: O sinal de fluorescência verde pode durar vários dias. Para garantir que o sistema funcione de forma eficaz, é melhor realizar um teste livre de DNA e RNase.
2. Avaliação da especificidade da plataforma de deteção baseada em RPA-CRISPR/Cas12a
NOTA: Para testar a especificidade de A. baumannii-DETECTR, os ácidos nucléicos de A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Cockerella burnetii, Serratia e amostras humanas foram submetidos ao teste DETECTR.
- Use modelos de DNA obtidos de várias espécies usando o Kit de DNA referenciado ou isolando-os fervendo as bactérias na água. Certifique-se de que todos os modelos tenham concentrações iguais. Use a água como controle negativo.
- Adicione 10 ng ou 1 μL de moldes de DNA à Solução A contendo primers F3R3. Adicione 2,5 μL de MgOAc, misture bem e incube a mistura a 39 °C por 20 min.
- Após 20 min, adicione 5 μL do produto da etapa anterior à solução B e misture.
- Coloque no instrumento de PCR quantitativo de fluorescência, defina o canal para FAM e leia o valor uma vez a cada minuto a 37 ° C, para um total de 60 x por 60 min.
3. Avaliação de sensibilidade da plataforma de detecção baseada em RPA-CRISPR/Cas12a
- Utilizar um plasmídeo recombinante positivo, que tenha sido preparado para uma concentração de 1-107 cópias/μl, como modelo para a reacção. Use água para o controle negativo.
- Execute o método seguindo as etapas 2.2-2.4.
NOTA: O conjunto de primers RPA-F3 e RPA-R3 foi projetado para amplificar um fragmento mais curto de 174 pares de bases do plasmídeo recombinante positivo.
4. Preparações enquanto a amostra está sob luz UV e detecção LFS
- Retire o número necessário de tiras de teste e rotule-as adequadamente.
- Transfira 10 μL dos produtos amplificados das etapas 2 e 3 para tubos de PCR separados, respectivamente. Em seguida, adicione 40 μL de ddH2O a cada tubo.
NOTA: Se estiver usando um sistema de reação auto-preparado, recomenda-se explorar a taxa de diluição ideal. - Execute o teste.
- Submeter o tubo de PCR que contém os produtos amplificados a um transiluminador para avaliar a presença de fluorescência.
- Insira as tiras de teste nos tubos de PCR com a extremidade da almofada de amostra voltada para baixo.
- Deixe repousar em temperatura ambiente por 5 a 10 minutos para ler o resultado. Após este tempo, certifique-se de que o nível do líquido não exceda a linha máxima antes de prosseguir.
- Interprete os resultados como negativos se a linha T não mostrar cor. Interprete os resultados como positivos se a linha T for visível a olho nu, indicando que a sonda de ácido nucleico foi clivada pela enzima Cas e a enzima Cas foi ativada. Considere o resultado inválido se nem a linha C nem a linha T mostrarem cor.
Resultados
Neste estudo, apresentamos uma nova plataforma de diagnóstico portátil chamada A. baumannii-DETECTR, que integra a eficiência de amplificação isotérmica do sistema RPA e CRISPR-Cas12a para identificação de campo rápida e confiável de A. baumannii. O esquema do ensaio DETECTR é ilustrado na Figura 1.
Doze pares de primers foram projetados usando ferramentas de design com base nos princípios de design de primers. Foram obtidos quatro primers diretos (F0-F3) e três iniciadores reversos (R1-R3), resultando em 12 pares de primers (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3). Após a amplificação de RPA a 39 °C por 20 min, os produtos de PCR de RPA foram analisados em um gel de agarose a 1% a 120 V por 10 min para determinar a combinação ideal de primers. O par de primers F3R3 emergiu como o mais eficaz, exibindo as bandas mais brilhantes e densas (Figura 2).
Tendo identificado o par de primers ideal F3R3, otimizamos as concentrações finais de primers e os tempos de reação para a amplificação de RPA, mantendo todos os outros componentes constantes. Nossos achados revelaram que a eficiência de amplificação atingiu o pico em uma concentração de primer de 0,44 μM e um tempo de reação de 20 min (Figura 3). Usando um plasmídeo recombinante positivo a uma concentração de 107 cópias/μL como molde, observamos um aumento dependente do tempo na intensidade de fluorescência para crRNA-1, enquanto crRNA-2 não mostrou tal aumento ao longo do tempo. Consequentemente, o crRNA-1 foi selecionado como o crRNA para a detecção de A. baumannii (Figura 4).
A especificidade de A. baumannii-DETECTR foi avaliada usando ácidos nucléicos isolados de uma gama diversificada de cepas bacterianas. Durante os ensaios de PCR, um sinal de fluorescência significativo só foi observado quando o genoma de A. baumannii foi direcionado, enquanto nenhuma fluorescência foi detectada ao usar DNA genômico de outras fontes bacterianas (Figura 5). Esses resultados confirmam que o par de primers A. baumannii-F3 e R3 exibe especificidade para A. baumannii no ensaio DETECTR, indicando que A. baumannii-DETECTR pode detectar esse patógeno de forma confiável.
Para avaliar a sensibilidade do ensaio A . baumannii-DETECTR, realizamos uma série de diluições com DNA de A. baumannii . O número de cópias foi calculado usando a seguinte fórmula: número de cópias (cópias/μL) = 6,02 × 1023 x P (ng/μL) x 10-9 / (número de bases plasmídicas x 660) (onde P representa a concentração de plasmídeo). O ensaio demonstrou a capacidade de detectar até 1 cópia/μL, indicando seu potencial como uma ferramenta de diagnóstico extremamente sensível, adequada para aplicações no local (Figura 6). Assim, o A. baumannii-DETECTR exibe uma sensibilidade notável.
Neste protocolo, também integramos a plataforma de detecção DETECTR mediada por CRISPR/Cas12a com a tecnologia de faixa de fluxo lateral (LFS) para facilitar a identificação de sequências de DNA alvo. Avaliamos a sensibilidade e especificidade do ensaio baseado em LFS. Um ensaio de especificidade realizado usando LFS não revelou sinais de reatividade cruzada com outras espécies bacterianas, demonstrando a alta especificidade do método (Figura 7A). Além disso, utilizamos LFS para detectar A. baumannii em uma faixa de diluições de 10a 7 cópias/μL até uma única cópia/μL. Notavelmente, todas as amostras testadas produziram resultados positivos (Figura 7B), que estavam alinhados com os resultados sob luz ultravioleta (Figura 7C, D). Coletivamente, esses resultados ressaltam que nossa plataforma serve como um instrumento de diagnóstico rápido, robusto e sensível para A. baumannii em ambientes de campo.

Figura 1: Fluxo de trabalho de A. baumannii-DETECTR. Abreviaturas: RPA =R ecombinase Amplificação da Polimerase; DETECTR = Repórter CRISPR Trans direcionado à endonuclease de DNA. Clique aqui para ver uma versão maior desta figura.

Figura 2: Design e seleção de primers. A combinação ideal de primers para a reação RPA foi identificada com base nos resultados obtidos da eletroforese em gel de agarose. O par de primers F3R3 teve melhor desempenho com bandas brilhantes e densas. Clique aqui para ver uma versão maior desta figura.

Figura 3: A otimização da concentração do primer e do tempo de amplificação para RPA. 1-4: As concentrações do primer 0,40 μM, 0,44 μM, 0,48 μM, 0,52 μM. Os tempos de amplificação são respectivamente 10 min, 20 min e 30 min. O efeito de amplificação atinge seu ponto mais alto quando a concentração de primer é de 0,44 μM e o tempo de reação é de 20 min. Abreviaturas: M = 8000 Marcador de DNA; RPA = amplificação da polimerase recombinase; NC = controle em branco. Clique aqui para ver uma versão maior desta figura.
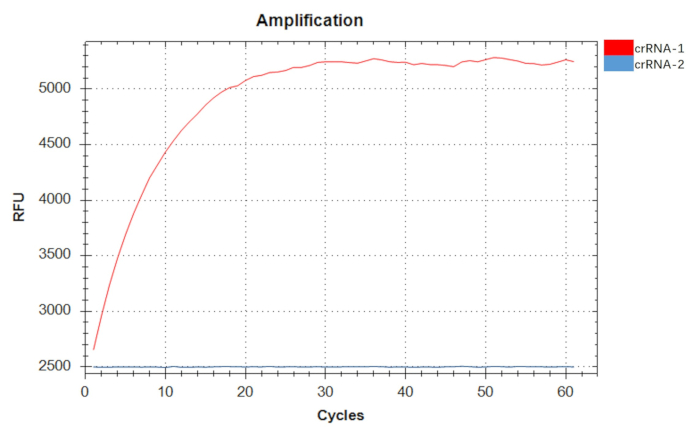
Figura 4: Triagem de dois crRNAs (crRNA-1 e crRNA-2). A intensidade de fluorescência da detecção de crRNA-1 aumentou com o tempo, enquanto a de detecção de crRNA-2 não aumentou com o tempo, não mostrando atividade de detecção significativa. O crRNA-1 foi usado como crRNA para a detecção de A. baumannii. Clique aqui para ver uma versão maior desta figura.
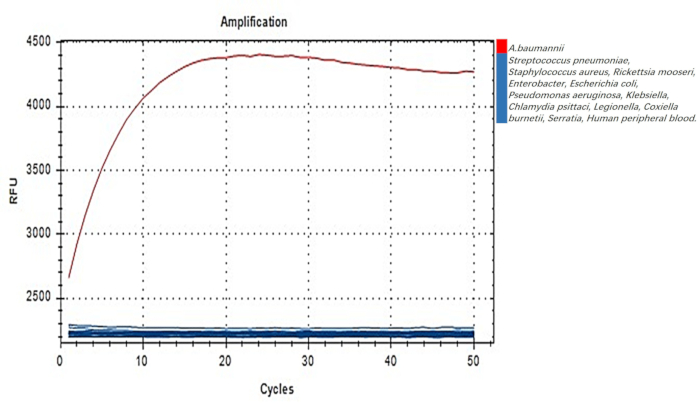
Figura 5: Testando a especificidade do A. baumannii-DETECTR. A amplificação de A. baumannii-DETECTR foi obtida usando o par de primers A. baumannii-F3 e R3, utilizando DNA genômico extraído de A. baumannii, bem como de outras espécies bacterianas. Abreviatura: DETECTR = DNA Endonuclease Direcionado CRISPR Trans Reporter. Clique aqui para ver uma versão maior desta figura.
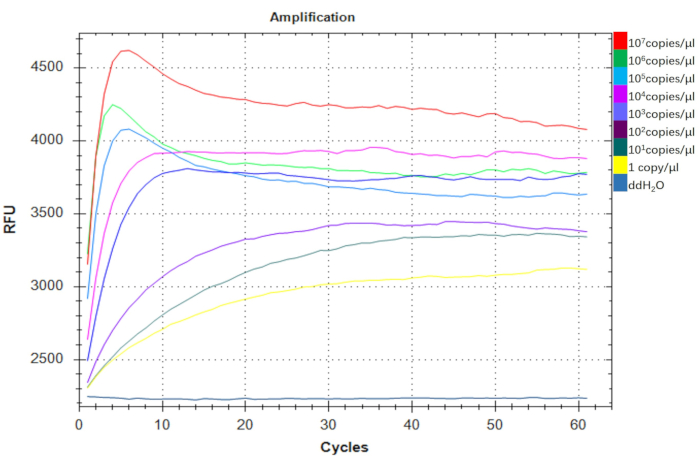
Figura 6: Testando a sensibilidade do A. baumannii-DETECTR. Os plasmídeos recombinantes positivos para rDNA pUC57-16s serviram como alvos para avaliar o limite de detecção de A. baumannii usando o ensaio RPA-CRISPR/Cas12a baseado em fluorescência visual. O ensaio detectou com sucesso uma faixa de uma cópia a 107 cópias/μL usando primers consistentes. A intensidade de fluorescência no grupo de uma cópia foi significativamente elevada em comparação com o grupo controle ddH2O. Abreviaturas: DETECTR = Repórter Trans CRISPR direcionado à endonuclease de DNA; RPA = amplificação da polimerase recombinase. Clique aqui para ver uma versão maior desta figura.
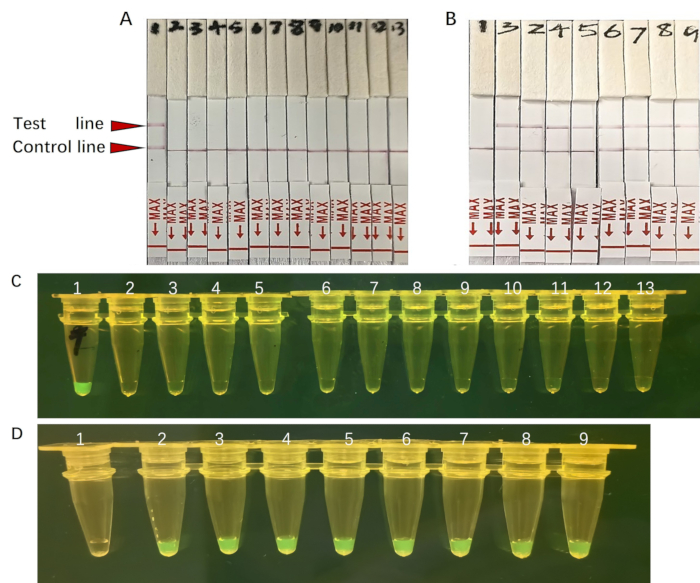
Figura 7: O sinal de fluorescência da amostra sob luz UV e detecção LFS. Reação de A. baumannii-DETECTR com padrões de plasmídeo. (AC) Diferentes patógenos foram detectados por A. baumannii-DETECTR. Apenas A. baumannii foi detectado com sucesso. 1-13 representam A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Coxiella burnetii, Serratia, Periférico humano. (B-D) O plasmídeo recombinante positivo com diluição em série em incrementos de dez vezes detectado pelo A. baumannii-DETECTR. Todos foram detectados, exceto o controle negativo. 1-9 representam controle negativo, 1 cópia/μL, 101 cópias/μL, 102 cópias/μL, 103 cópias/μL, 104 cópias/μL, 105 cópias/μL, 106 cópias/μL, 107 cópias/μL. Clique aqui para ver uma versão maior desta figura.
| Não | Nome | Sequência (5'-3') | |||
| 1 | crRNA-1 | uaauucuacuaaguguagauGUUAAUACCUAGAGAUAGUG | |||
| 2 | crRNA-2 | uauucuacuaaguguagauGUUGGUAUUCCGGAAGAAGU | |||
| 3 | crRNA-1-DNA | GTTAATACCTAGAGATAGTG | |||
| 4 | crRNA-2-DNA | GTTGGTATTCCGGAAGAAGT | |||
| 5 | Sequência | TAACTGAAGAGTTTGATCATGGCTCAGATTGAACGCTGG CGGCAGGCTTAACACATGCAAGTCGAGCGGGGGAAGGT AGCTTGCTACTGGACCTAGCGGCGGACGGGTGAGTAATG CTTAGGAATCTGCCTATTAGTGGGGGACAACATCTCGAAA GGGATGCTAATACCGCATACGTCCTACGGGAGAAAGCAG GGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCG GATTAGCTAGTTGGTGGGGTAAAGGCCTACCAAGGCGAC GATCTGTAGCGGGTCTGAGAGGATGATCCGCCACACTGG GACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGT GGGGAATATTGGACAATGGGGGGAACCCTGATCCAGCCAT GCCGCGTGTGTGTGAAGAAGGCCTTATGGTTGTAAAGCACTT TAAGCGAGGAGGAGGCTACTTTAGTTAATACCTAGAGATAG TGGACGTTACTCGCAGAATAAGCACCGGCTAACTCTGTGC CAGCAGCCGCGGTAATACAGAGGGTGCGAGCGTTAATCGG ATTTACTGGGCGTAAAGCGTGCGTAGGCGGCTTATTAAGTC GGATGTGAAATCCCCGAGCTTAACTTGGGAATTGCATTCGAT ACTGGTGAGCTAGAGTATGGGAGAGGATGGTAGAATTCCAG GTGTAGCGGTGAAATGCGTAGAGATCTGGAGGAATACCGAT GGCGAAGGCAGCCATCTGGCCTAATACTGACGCTGAGGTAC GAAAGCATGGGGAGCAAACAGGATTAGATACCCTGGTAGTC CATGCCGTAAACGATGTCTACTAGCCGTTGGGGCCTTTGAGG CTTTAGTGGCGCAGCTAACGCGATAAGTAGACCGCCTGGGGA GTACGGTCGCAAGACTAAAACTCAAATGAATTGACGGGGGCC CGCACAAGCGGTGGAGCATGTGGTTTAATTCGATGCAACGC GAAGAACCTTACCTGGCCTTGACATACTAGAAACTTTCCAGA GATGGATTGGTGCCTTCGGGAATCTAGATACAGGTGCTGCAT GGCTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCC CGCAACGAGCGCAACCCTTTTCCTTACTTGCCAGCATTTCG GATGGGAACTTTAAGGATACTGCCAGTGACAAACTGGAGGAA GGCGGGGACGACGTCAAGTCATCATGGCCCTTACGGCCAGG GCTACACACGTGCTACAATGGTCGGTACAAAGGGTTGCTACA CAGCGATGTGATGCTAATCTCAAAAAGCCGATCGTAGTCCGG ATTGGAGTCTGCAACTCGACTCCATGAAGTCGGAATCGCTAG TAATCGCGGATCAGAATGCCGCGGTGAATACGTTCCCGGGCC TTGTACACACCGCCCGTCACACCATGGGAGTTTGTTGCACCA GAAGTAGCTAGCCTAACTGCAAAGAGGGCGGTTACCACGGTG TGGCCGATGACTGGGGTGAAGTCGTAACAAGGCCGTAGG GGAACCTGCGGCTGGATCACCTCCTTA | |||
| 6 | A.baumannii-F0 | CGGGAGGCAGCAGTGGAATATTGGACAAT | |||
| 7 | A.baumannii-F1 | AAGAAGGCCTTATGGTTGTAAAGCACTTTAA | |||
| 8 | A.baumannii -F2 | GGTTGTAAAGCACTTTAAGCGAGGAGGAGGCTA | |||
| 9 | A.baumannii -F3 | AAGGCCTTATGGTTGTAAAGCACTTTAAGCGA | |||
| 10 | A.baumannii-R1 | ACAGAGTTAGCCGGTGCTTATTCTGCGAGTAA | |||
| 11 | A.baumannii-R2 | AATCCGATTAACGCTCGCACCCTCTGTATTA | |||
| 12 | A.baumannii-R3 | AGTTAGCCGGTGCTTATTCTGCGAGTAACGTC | |||
| 13 | ssDNA-FQ | ssDNA-FQ FAM-TTTATTT-BHQ1 | |||
| 14 | A.baumannii-16s rDNA-F | 5 ′ - CGCGGATCCTAACTGAAGAGTTTGATCATGG-3 ′ | |||
| 15 | A.baumannii-16s rDNA-R | 5′-ACGCGTCGACTAAGGAGGTGATCCAGC-3′. | |||
Tabela 1: Ácidos nucléicos utilizados neste estudo. O ácido nucléico usado aqui é o ssDNA-FQ, que é composto de DNA de fita simples conjugado com uma 5-carboxifluoresceína na extremidade 5' e o quencher de fluorescência Black Hole Quencher 1 na extremidade 3'. Abreviaturas: F = fluoróforo; Q = quencher.
Arquivo Suplementar 1: Projeto de primers e crRNA.Clique aqui para ver uma versão maior desta figura.
Discussão
Os métodos tradicionais de diagnóstico para infecções por A. baumannii têm várias restrições que os tornam menos acessíveis e viáveis para testes no local de atendimento27. Por exemplo, a PCR tem boa sensibilidade e especificidade, mas requer equipamentos especializados de ciclagem térmica e fluxos de trabalho complexos que devem ser operados por profissionais. O novo método apresentado aqui, que combina RPA e edição do genoma CRISPR-LbaCas12a com recombinação, conhecido como ensaio A. baumannii-DETECTR, permite manipulação genética eficiente. Ele fornece um método de detecção rápido, sensível e específico para infecções por Acinetobacter baumannii , bem como para o diagnóstico de outros patógenos bacterianos. O fluxo de trabalho é ilustrado na Figura 1.
Nosso método é particularmente vantajoso no diagnóstico e gerenciamento de infecções, o que deve facilitar a recuperação do paciente e mitigar a disseminação de A. baumannii , superando as deficiências das técnicas de diagnóstico existentes. O sistema A. baumannii-DETECTR identificou A. baumannii com alta especificidade e sensibilidade (Figura 5 e Figura 6). Nossos achados revelaram que apenas o genoma de A. baumannii exibiu uma mudança positiva na dobra de fluorescência durante a avaliação em tempo real, enquanto os genomas de todas as outras espécies apresentaram resultados negativos (Figura 5).
Para avaliar a sensibilidade da plataforma de detecção RPA-CRISPR/Cas12a, plasmídeos recombinantes pUC57-16s rDNA, que são positivos para A. baumannii, foram empregados como alvos para determinar o limite de detecção (LOD) do sistema. No ensaio RPA-CRISPR/Cas12a baseado em fluorescência, a detecção variou de uma cópia por microlitro a 107 cópias por microlitro, com um aumento notável na intensidade de fluorescência observado no grupo de 1 cópia/μL em comparação com o grupo de controle negativo (Figura 6). É importante ressaltar que, para o diagnóstico em tempo real, os resultados da detecção podem ser visualizados diretamente por meio de um dispositivo portátil portátil, independente do equipamento laboratorial, o que é essencial para o diagnóstico em tempo real.
Vários fatores críticos devem ser considerados em relação ao protocolo26. Em primeiro lugar, é essencial evitar a contaminação cruzada durante o processo de coleta da amostra, dada a alta sensibilidade do ensaio A. baumannii-DETECTR. Além disso, os reagentes de reação devem ser protegidos da exposição direta à luz solar. A adesão a todas as etapas processuais é crucial para alcançar os melhores resultados possíveis. Um controle positivo, consistindo em um plasmídeo que abriga fragmentos de genes específicos de A. baumannii, deve ser incorporado para verificar a funcionalidade adequada do sistema A. baumannii-DETECTR em aplicações práticas.
O protocolo mostra que os parâmetros podem ser otimizados para condições laboratoriais específicas e tipos de amostras, como concentração de primer, tempo de reação e outras variáveis. Neste experimento, a combinação ideal de primer, concentrações de primer e tempos de reação para amplificação de RPA foram selecionados para produzir a maior eficiência e especificidade de amplificação. Isso foi determinado por eletroforese em gel de agarose (Figura 2 e Figura 3). Além disso, um plasmídeo recombinante positivo, contendo uma região conservada do genoma de A. baumannii , foi usado como controle para garantir a precisão do protocolo. Vale a pena notar que os kits comerciais de extração de DNA genômico bacteriano fornecem um método padronizado e confiável para extração de DNA neste protocolo. No entanto, em estudos futuros, diferentes tipos de amostras, como amostras clínicas ou ambientais, podem exigir métodos de extração específicos28 para isolar efetivamente o DNA de A. baumannii .
Na ausência de sinais de fluorescência detectáveis do controlo positivo, é provável que estejam presentes inibidores na mistura de reacção, necessitando de uma mudança de reagentes. Por outro lado, se a intensidade da fluorescência for insuficiente para uma diferenciação clara, estender a duração da incubação pode ser benéfico. No entanto, a incubação prolongada também pode resultar em falsos positivos26.
As metodologias atuais de detecção de A. baumannii exigem o uso de equipamentos de PCR caros, locais operacionais fixos e pessoal especializado. Em contraste, o método aqui proposto facilita o diagnóstico preciso e sensível de A. baumannii em ambientes de campo, tornando-o acessível a um grupo demográfico mais amplo.
As características da amplificação isotérmica e os dados coletados por fluorescência tornam o fluxo de trabalho de A. baumannii-DETCTER simples e com baixa necessidade de equipamento, além de possibilitar a detecção rápida de patógenos em áreas remotas ou durante surtos. RPA-LFS 29,30,31,32, uma nova técnica isotérmica de amplificação de ácidos nucleicos, ganhou força significativa na detecção de patógenos virais, bacterianos, fúngicos e parasitários devido à sua simplicidade operacional e demandas temporais reduzidas. O protocolo detalha o sinal de fluorescência da amostra sob luz ultravioleta (UV) ou detecção de Transiluminador e tiras de fluxo lateral (LFS). Pode evitar a dependência de instrumentos de alta precisão e técnicos especializados, pois a inspeção visual resulta em um curto espaço de tempo (Figura 7), eliminando assim a necessidade de equipamentos laboratoriais caros e complexos e a população operacional. Além disso, o método de um tubo não apenas economiza tempo, mas também diminui o risco de contaminação do amplicon33.
Estudos futuros devem ter como objetivo melhorar o método para torná-lo viável para vários tipos de amostras e diferentes condições ambientais, como explorar técnicas de extração de DNA mais eficientes e de menor custo. O uso do A. baumannii-DETECTR não é apenas para diagnóstico clínico, mas também pode ser usado para avaliação ambiental, monitoramento e gerenciamento de surtos, permitindo que comunidades e hospitais identifiquem rapidamente A. baumannii. Além disso, ao modificar o crRNA e os primers, essa abordagem diagnóstica molecular pode ser adaptada para a detecção de várias infecções bacterianas respiratórias, como Streptococcus pneumoniae, Staphylococcus aureus e Enterobacter.
Em resumo, A. baumannii-DETECTR pode identificar A. baumannii da forma mais rápida e precisa possível devido à sua alta sensibilidade e especificidade, design amigável e portabilidade. Esse método pode melhorar os resultados de saúde do paciente, facilitando o tratamento oportuno e eficaz, o controle de infecções e mitigando a proliferação da resistência a antibióticos, ao mesmo tempo em que enfrenta a crescente ameaça de infecções por A. baumannii .
Divulgações
Os autores declaram que não têm interesses conflitantes.
Agradecimentos
Este trabalho foi apoiado pelo Projeto do Plano de Desenvolvimento de Ciência e Tecnologia da Província de Jilin, China (20240305027YY) e pelo Departamento de Finanças da Província de Jilin (JLSWSRCZX2023-55, JLSWSRCZX2021-041).
Materiais
| Name | Company | Catalog Number | Comments |
| -20 °C Freezer | Haier | HYCD-290 | China |
| Agarose Basic | BioFroxx | 1110GR100 | China |
| All oligonucleotides and crRNA were synthesized by company | www.comatebio.com | ||
| Cas12a cutting substrate - ssDNA - fluorescent type | EZassay Biotech. Co. Ltd. | DNA-FAM-BHQ | China |
| Cas12a cutting substrate - ssDNA - test paper type | EZassay Biotech. Co. Ltd. | DNA-FAM-BIO | China |
| Electrophoresis apparatus | BIO-RAD | POWER PAC1000 | USA |
| Fluorescence quantitative PCR instrument | BIO-RAD | CFX Connect | USA |
| Gel Imaging System | BIO-RAD | Gel Doc 2000 | USA |
| https://ezassay.com/rna | |||
| https://www.ezassay.com/primer | |||
| https://www.ncbi.nlm.nih.gov/tools/primer-blast | |||
| Lateral flow paper strip (Biotin/FAM) | EZassay Biotech. Co. Ltd. | HD-FMBO | China |
| LbaCas12a (Cpf1) enhanced protein | EZassay Biotech. Co. Ltd. | CAS-12E-001 | China |
| LED Transilluminator | LABGIC | BL-20 | China |
| Magnesium acetate, MgOAc | TwistDx | TABAS03KIT | UK |
| Microcentrifuge | allsheng | Mini-6k | China |
| PCR strip tubes | PCR strip tubes | PST-0208-FT-C | China |
| TGrade Dry Bath Incubator | Tiangen biochemical technology | OSE-DB-01 | China |
| Tianamp Bacteria DNA Kit | Tiangen biochemical technology | DP302-02 | China |
| TIANamp Bacteria DNA Kit | TIANGEN BIOTECH (BEIJING) CO.; LTD. | DP302 | China |
| TransStart FastPfu DNA Polymerase | TransGen Biotech. Co. Ltd. | AP221 | China |
| TwistAmp Basic Kit | TwistDX | TABAS03KIT | UK |
| Universal DNA Purification Kit | Tiangen biochemical technology | DP214-03 | China |
Referências
- Lenie, D., Alexandr, N., Harald, S. An increasing threat in hospitals: Multidrug-resistant Acinetobacter baumannii. Nat Rev Microbiol. 5 (12), 939-951 (2007).
- Li, P., et al. Rapid detection of Acinetobacter baumannii and molecular epidemiology of carbapenem-resistant A. baumannii in two comprehensive hospitals of Beijing, China. Front Microbiol. 6, 997 (2015).
- Wu, X., et al. A diagnostic test that uses isothermal amplification and lateral flow detection sdaA can detect tuberculosis in 60 min. J Appl Microbiol. 130 (6), 2102-2110 (2020).
- Huang, B., et al. A cas12a-based fluorescent microfluidic system for rapid on-site human papillomavirus diagnostics. Appl Microbiol Biotechnol. 107 (20), 6287-6297 (2023).
- Piepenburg, O., Williams, C. H., Stemple, D. L., Armes, N. A. DNA detection using recombination proteins. Plos Biology. 4 (7), 1115-1121 (2006).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), aaf5573 (2016).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), eaar6245 (2018).
- Xiong, D., et al. Rapid detection of SARS-CoV-2 with CRISPR-Cas12a. PLoS Biol. 18 (12), e3000978 (2020).
- Gootenberg, J. S., et al. Nucleic acid detection with crispr-Cas13a/C2C2. Science. 356 (6336), 438-442 (2017).
- Harrington, L. B., et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science. 362 (6416), 839-842 (2018).
- Li, S. Y., et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA. Cell Res. 28 (4), 491-493 (2018).
- Wang, B., et al. Cas12aVDet: A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem. 91 (19), 12156-12161 (2019).
- Huang, Z., et al. Ultra-sensitive and high-throughput crispr-p owered COVID-19 diagnosis. Biosens Bioelectron. 164, 112316 (2020).
- Zhang, W. S., et al. Reverse transcription recombinase polymerase amplification coupled with CRISPR-Cas12a for facile and highly sensitive colorimetric SARS-CoV-2 detection. Anal Chem. 93 (8), 4126-4133 (2021).
- Marraffini Luciano, A. CRISPR-Cas immunity in prokaryotes. Nature. 526 (7571), 55-61 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), aad5147 (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 13 (11), 722-736 (2015).
- Li, S. Y., et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discovery. 4 (1), 20 (2018).
- Li, L., Li, S., Wu, N., Wu, J., Wang, J. Holmesv2: A CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol. 8 (10), 2228-2237 (2019).
- Liang, M., Li, Z., Wang, W., Liu, J., Zhang, L. X. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules. Nat Communications. 10 (1), 3672 (2019).
- Aquino-Jarquin, G. CRISPR-Cas14 is now part of the artillery for gene editing and molecular diagnostic. Nanomedicine. 18, 428-431 (2019).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Swarts, D. C., John, V. D. O., Jinek, M. Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a. Mol Cell. 66 (2), 221-233 (2017).
- Jiang, Y., et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun. 8, 15179 (2017).
- Yan, M. Y., et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol. 83 (17), e00947-e01017 (2017).
- Ashraf, A., et al. A novel multiplex pcr assay for simultaneous detection of nine clinically significant bacterial pathogens associated with bovine mastitis. Mol Cell Probes. 33, 57-64 (2017).
- Zhu, L., et al. A rapid on-site visualization platform based on RPA coupled with CRISPR-Cas12a for the detection of genetically modified papaya 'huanong no.1'. Talanta. 277, 126437 (2024).
- Zheng, C., et al. Rapid developments in lateral flow immunoassay for nucleic acid detection. Analyst. 146 (5), 1514-1528 (2021).
- Wang, Y., et al. Establishment and clinical application of a RPA-LFS assay for detection of capsulated and non-capsulated Haemophilus influenzae. Front Cell Infect Microbiol. 12, 878813 (2022).
- Wang, F., et al. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strips for detecting Candida albicans. Anal Biochem. 633, 114428 (2021).
- Ma, B., et al. A simple and efficient method for potential point-of-care diagnosis of human papillomavirus genotypes: Combination of isothermal recombinase polymerase amplification with lateral flow dipstick and reverse dot blot. Anal Bioanal Chem. 411 (28), 7451-7460 (2019).
- Sun, Y., Yu, L., Liu, C., Ye, S., Huang, W. One-tube SARS-CoV-2 detection platform based on RT-RPA and CRISPR/Cas12a. J Transl Med. 19 (1), 74 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados