Method Article
זיהוי מהיר וספציפי של זיהומי אצינטובקטר באומני באמצעות הגברת פולימראז רקומבינאז/מערכת מבוססת Cas12a
In This Article
Summary
כדי להקל על הזיהוי המהיר והמדויק של Acinetobacter baumannii, אנו מציגים פרוטוקול המשתמש בהגברה רקומבינאז פולימראז (RPA) בשילוב עם אנדונוקלאז LbaCas12a לזיהוי זיהומי A. baumannii .
Abstract
Acinetobacter baumannii, חיידק גרם שלילי, ידוע לשמצה בגרימת זיהומים חמורים עם שיעורי תמותה גבוהים. זיהוי מהיר ומדויק של A. baumannii הוא חיוני לטיפול מהיר, בקרת זיהום יעילה וריסון עמידות לאנטיביוטיקה. עם זאת, אין שיטה מתאימה לזיהוי מהיר וקל באתר של A. baumannii. מערכת ה-DNA Endonuclease Targeted CRISPR Trans Reporter (DETECTR) מציעה גישה מהירה, מדויקת ורגישה לזיהוי A. baumannii על ידי שילוב יכולות הזיהוי הספציפיות למטרה של Cas12a עם יעילות ההגברה האיזותרמית של הגברת פולימראז רקומבינאז (RPA). פרוטוקול זה מפרט את הזיהוי של A. baumannii באמצעות RPA בשילוב עם אנדונוקלאז LbaCas12a. השלבים הבאים מתוארים במאמר זה: מיצוי DNA, בחירת רצף DNA ספציפי, תכנון פריימר ו-CRISPR RNA (crRNA), בניית פלסמיד רקומביננטי חיובי, הגדרת בדיקת Cas12a-RPA, אופטימיזציה של מערכת ההגברה RPA, הדמיה של בדיקת RPA-CRISPR/Cas12a באמצעות כלי זיהוי פלואורסצנטי כגון מכשיר PCR בזמן אמת, והערכת רגישות והערכת ספציפיות.
Introduction
במיקרוביולוגיה קלינית, איתור זיהומי Acinetobacter baumannii מהווה אתגר משמעותי. חיידק גרם שלילי זה יכול לגרום לזיהומים עם תסמינים קליניים חמורים, אפילו עם שיעורי תמותה גבוהים, במיוחד בקרב חולים מדוכאי חיסון1. שיטות זיהוי מסורתיות מבוססות תרבית לזיהומים פתוגנים גוזלות זמן ועשויות להיות חסרות רגישות, מה שעלול לעכב את הטיפול האנטיביוטי ולפגוע בתוצאות המטופלים. זיהוי מהיר ומדויק של A. baumannii הוא חיוני לטיפול יעיל ולבקרת התפרצויות. טכניקות מולקולריות המשתמשות בהגברה של חומצות גרעין נחקרו כדי לענות על צורך זה. עם זאת, גישות אלו זקוקות לרוב לציוד רכיבה תרמי מתוחכם ועשויות להיות מוגבלות על ידי טכנאים מאומנים היטב ומעבדות מבוססות היטב. כדי להתגבר על אתגרים אלה, המחקר מתמקד יותר ויותר בפיתוח שיטות הגברה איזותרמיות 2,3,4.
הגברת פולימראז רקומבינאז (RPA) היא שיטה שהוקמה על ידי Piepenburg et al 5 ומשמשת להגברת DNA, בדומה לתגובת שרשרת פולימראז (PCR) אך ללא צורך במחזור טמפרטורה. שיטה זו כוללת 50 מ"מ טריס (pH 7.5), 100 מ"מ אשלגן אצטט, 14 מ"מ מגנזיום אצטט, 2 מ"מ DTT, 5% PEG20000 (פוליאתילן גליקול במשקל מולקולרי גבוה), 200 מיקרומטר dNTPs, 3 מ"מ ATP, 50 מ"מ פוספוקריאטין, 100 מיקרוגרם/מ"ל קריאטין קינאז, 120 מיקרוגרם/מ"ל UvsX, 30 מיקרוגרם/מ"ל UvsY, 900 מיקרוגרם/מ"ל Gp32, 30 מיקרוגרם/מ"ל Bsu LF, 450 ננומטר פריימרים ותבניות DNA. תהליך ההגברה מתחיל עם החלבון הרקומבינאז UvsX שנקשר לפריימרים בנוכחות 3 מ"מ ATP ו-5% PEG20000 יצירת קומפלקס רקומבינאז-פריימר. קומפלקס זה מקל על השילוב של פריימרים עם הרצפים ההומולוגיים על ה-DNA הדו-גדילי.
בעזרת UvsY, ה-UvsX הרקומבינאז מקל על ההחלפה בין הפריימר לגדיל התבנית, וכתוצאה מכך תזוזה של גדיל אחד של ה-DNA המטרה. Gp32 מסייע בשמירה על מבנה ה-DNA החד-גדילי. לבסוף, הרקומבינאז מתנתק, ו-DNA פולימראז (Bsu LF) המסוגל לעקור גדילי DNA נקשר לקצה ה-3' של הפריימר, ומאריך אותו בנוכחות דאוקסיריבונוקלאוזידים טריפוספטים (dNTPs). תהליך זה חוזר על עצמו באופן מחזורי, ומשיג הגברה אקספוננציאלית. ניתן להשלים את כל תהליכי ההגברה תוך 20-40 דקות ובטמפרטורות קבועות יחסית בין 37 מעלות צלזיוס ל-42 מעלות צלזיוס. טווח טמפרטורות זה זהה בערך לטמפרטורות פיזיולוגיות, מה שמאפשר לבצע RPA בסביבה מינימליסטית, מה שהופך את RPA לכלי רב-תכליתי ויעיל לזיהוי וניתוח DNA.
מערכת החזרות הפלינדרומיות הקצרות (CRISPR) ומערכת החלבון הקשור ל-CRISPR (CRISPR-Cas) מתפקדת כמנגנון חיסוני אדפטיבי בחיידקים וארכאונים, הכוללים מערכות Class I ו-Class II. Class II כולל חלבונים כגון Cas12 (a, b, f), Cas13 (a, b) ו-Cas14, המזהים ומבקעים DNA או RNA מטרה מונחה על ידי CRISPR RNA (crRNA)6,7,8. LbaCas12a (או Cpf1) הוא אנדונוקלאז DNA מונחה crRNA. ה-crRNA משמש כ-RNA מנחה בתוך מערכת CRISPR-Cas, שם הוא מתחבר עם חלבוני Cas. הוא ממנף את אזור המרווח שלו כדי לזווג עם ה-DNA של המטרה, ולמעשה מנווט את קומפלקס החלבון לרצפי המטרה. הרצף 5'-UAAUUUCUACUAAGUGAUGAU-3' משמש כחזרת crRNA משומרת, אלמנט קבוע בכל ה-crRNAs של LbaCas12a. לאחר חזרה זו מופיע קטע ספציפי למטרה השונה בהתאם ליעד ה-DNA המיועד. רצף מטרה זה נע בין 18 ל-24 נוקלאוטידים.
רצף ה-PAM (Protospacer-Adjacent Motif) (TTTV, כאשר V יכול להיות A, C או G) ממוקם בקצה 5' של הגדיל הלא משלים של יעד ה-DNA. ה-Cas12a חותך את ה-DNA הדו-גדילי ברצף ה-PAM. לאחר מכן, חלבוני ה-Cas המופעלים מבצעים טרנס-פיצול לא ספציפי של DNA חד-גדילי (ssDNA)9,10,11,12. לפיכך, ssDNA המסומן עם פלואורופור ומרווה עובר מחשוף, וכתוצאה מכך פליטה של אות פלואורסצנטי לאחר מחשוף צדדי 8,13. ניתן לכמת את עוצמת הקרינה באמצעות קורא פלואורסצנטי לזיהוי מדויק של חומצות גרעין14. יש לציין כי Cas12a נמצא בשימוש נרחב לניתוח DNA, כאשר הקישור והפיצול של רצפי המטרה שלו מותנים בזיהוי המוטיב הסמוך לפרוטו-ספייסר (PAM), בעוד ש-Cas13a פועל ללא תלות בדרישה כזו.
מערכות CRISPR-Cas 15,16,17 מקלות על מחשוף בתוך תגובה אחת 18,19,20,21. שילוב של שני הנ"ל יכול ליצור כלי חזק המכונה A. baumannii-DETECTR (LbaCas12a-Enabled Detection of Targeted A. baumannii) לזיהוי חומצות גרעין 22,23,24. פרוטוקול זה מדגים את השימוש ב-RPA בשילוב עם פעילות ה-DNase של Cas12a כדי למקד ולבקע באופן ספציפי רצף מונחה crRNA בתוך גן ה-rDNA 16s של A. baumannii, ובכך לאפשר זיהוי רגיש ומדויק של הפתוגן.
שיטת A. baumannii-DETECTR מציגה מספר יתרונות על פני שיטות זיהוי קונבנציונליות25,26. ראשית, המאפיין האיזותרמי של RPA מייעל את ההליכים התפעוליים ומפחית עלויות באמצעות מנגנון ההגברה הבלתי תלוי במחזור התרמי שלו5. שנית, האנדונוקלאז Cas12a מגדיל את הספציפיות של הבדיקה באמצעות מיקוד בתיווך crRNA וזיווג בסיס ווטסון-קריק7. לבסוף, שימוש בשיטת הצינור האחד של A. baumannii-DETECTR לא רק חוסך זמן אלא גם מפחית משמעותית את הסיכון לזיהום אמפליקון19.
הפרוטוקול מתאר שלבים למיצוי מיצוי DNA, בחירת רצפי DNA מטרה, תכנון פריימרים ו-crRNAs, בניית פלסמידים רקומביננטיים חיוביים, הקמת מבחן Cas12a-RPA, אופטימיזציה של אופטימיזציה של הגברת RPA והדמיה של תוצאות באמצעות כלים לזיהוי פלואורסצנטי כגון מכונת PCR בזמן אמת, עם הערכות רגישות וספציפיות.
שיטת A. baumannii-DETECTR מבטיחה בשיפור תוצאות המטופלים על ידי הקלה על טיפול בזמן ויעיל, בקרת זיהום חזקה ובלימת התפשטות עמידות לאנטיביוטיקה. פרוטוקול זה יכול לשמש כהנחיה מקיפה ליישום טכנולוגיית Cas12a-RPA, ולשפר את התועלת שלה במסגרות בריאות שונות. עם זאת, חשוב לציין שכל שלב צריך להיות אופטימלי על סמך תנאי מעבדה ספציפיים ומגוון הדגימות כדי להבטיח תוצאות עקביות ואמינות.
Protocol
1. בניית ה-A. baumannii -DETECTR
הערה: הבנייה של A. baumannii-DETECTR היא תהליך בן ארבעה שלבים הכולל תכנון של פריימר ו-crRNA, בניית בניית פלסמיד רקומביננטי חיובי, הכנת פתרונות תגובה, הגברת DNA איזותרמית על ידי RPA והדמיה של בדיקת RPA-CRISPR/Cas12a. הסכימה של מבחן ה-DETECTR מודגמת באיור 1.
- תכנון פריימר ו-crRNA (ראה קובץ משלים 1)
- תכנון 12 זוגות פריימרים באמצעות כלי עיצוב על בסיס עקרונות עיצוב פריימר. השג ארבעה פריימרים קדימה (F0-F3) ושלושה פריימרים הפוכים (R1-R3) כדי להרכיב שנים עשר זוגות פריימרים (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- ודא שקצה ה-5' של הפריימר מכיל בסיסי C או T בתוך 3-5 הנוקלאוטידים הראשונים במקום מתיחת G רציפה. בנוסף, ודא ששלושת הנוקלאוטידים האחרונים בקצה 3' הם באופן אידיאלי בסיסי G ו-C כדי לשפר את ההגברה.
- תכנן פריימרים ללא רצפים משלימים את עצמם כדי למנוע היווצרות סיכות ראש ועם לא יותר מארבעה בסיסים משלימים או הומולוגיים ביניהם. התרחק מהשלמת קצה 3' כדי למנוע דימריזציה של פריימר.
- העריכו את הספציפיות של כל זוג פריימרים באמצעות כלי Primer-BLAST של המרכז הלאומי למידע ביוטכנולוגי (NCBI) כדי להעריך את היעילות והספציפיות של הפריימרים.
הערה: Primer-BLAST יכול להשתמש במסד הנתונים של NCBI כדי למצוא רצפים התואמים את הפריימרים המתוכננים, ולעזור למשתמשים להעריך את היעילות והספציפיות של הפריימרים. - לאחר הקרנת הפריימר, תכנן את ה-CRISPR RNA (crRNA) או ה-RNA המנחה (gRNA) באמצעות רצף פיגומי LbaCas12a crRNA (5'-UAAUUUCUAAGUGUAGAU-3') כרצף הקבוע. שלב קטע ספציפי למטרה (5'-GUUAAUACCUAGAGAUAGUG-3'), וודא שמוטיב ה-PAM ממוקם בקצה 5' של הגדיל הלא משלים.
הערה: הרצפים של כל האוליגונוקלאוטידים וה-crRNA מוצגים בטבלה 1.
- תכנון 12 זוגות פריימרים באמצעות כלי עיצוב על בסיס עקרונות עיצוב פריימר. השג ארבעה פריימרים קדימה (F0-F3) ושלושה פריימרים הפוכים (R1-R3) כדי להרכיב שנים עשר זוגות פריימרים (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3).
- בניית פלסמיד רקומביננטית חיובית
- הגבירו את מקטע ה-rDNA של 16 שניות באמצעות ערכת הפריימר A. baumannii 16s rDNA-F (פריימר קדימה) ו-A. baumannii 16s rDNA-R (פריימר הפוך).
- הוסף 1 μL של 10 μM F ו-10 μM R, 10 μL של מאגר pFu מהירפי 5, 1 μL של pFu פולימראז מהיר, 4 μL של 2.5 mM dNTPs, 1 μL של 10 ng/μL A. baumannii DNA גנומי ו-32 μL של מים נטולי נוקלאז למערכת 50 μL PCR.
- הגדר את תנאי הרכיבה של PCR כדלקמן: דנטורציה ראשונית ב-95 מעלות צלזיוס למשך 5 דקות, ואחריה 33 מחזורי דנטורציה ב-95 מעלות צלזיוס למשך 30 שניות, חישול ב-55 מעלות צלזיוס למשך 30 שניות, והארכה ב-72 מעלות צלזיוס למשך דקה ו-20 שניות. לבסוף, בצע 10 דקות ב-72 מעלות צלזיוס להארכה הסופית.
- כדי לטהר את מוצר ה-PCR החיובי, השתמש בערכת טיהור DNA בהתאם להוראות היצרן. לאחר מכן, שכפל את ה-DNA המטוהר לווקטור pUC57, שעוכל עם אנזימי BamHI ו-SalI.
הערה: תהליך זה יביא ליצירת פלסמיד רקומביננטי חיובי המכיל אזור שמור. - לדלל את הפלסמיד הרקומביננטי החיובי באופן סדרתי במרווחים של פי עשרה עם ddH2O מעוקר כדי להשיג ריכוז של עותק אחד/מיקרוליטר ל-107 עותקים/מיקרוליטר. אחסן את הפלסמיד המדולל בטמפרטורה של -20 מעלות צלזיוס לניסויים הבאים.
- הגבירו את מקטע ה-rDNA של 16 שניות באמצעות ערכת הפריימר A. baumannii 16s rDNA-F (פריימר קדימה) ו-A. baumannii 16s rDNA-R (פריימר הפוך).
- הכנת פתרון
- הכן את פתרון A באופן הבא: עבור כל תגובה, הוסף 29.5 מיקרוליטר של מאגר התייבשות ללא פריימר, 2.4 מיקרוליטר של 10 מיקרומטר A. baumannii-F ו-10 מיקרומטר A. baumannii-R, ו-12.2מיקרוליטר של ddH2O כדי להשיג נפח סופי של 46.5 מיקרוליטר מערבולת וסובב לזמן קצר. לאחר מכן, הוסף את תערובת התגובה לצינורות תגובת האנזים מערכת RPA. פיפטה לערבב.
הערה: הפריימרים של A. baumannii-F ו-R משמשים בבדיקה המכוונת לגן A. baumannii . אנא עיין בטבלה 1 לרצף. - הכן את פתרון B באופן הבא: עבור כל תגובה, הוסף 0.24 μL של 10 μM ssDNA reporter, 2 μL של 10x Cas12a Reaction Buffer, 1 μL של 1 μM crRNA, 1 μL של 1 μM Cas12a ו-10.76μL של ddH2O כדי להשיג נפח סופי של 15 μL.
הערה: הבדיקות (מדווח ssDNA) נרכשו והורכבו ממדווח DNA חד-גדילי שתויג עם פלואורופור 5-Carboxyfluorescein (FAM) בקצה 5' ומרווה חור שחור 1 (BHQ1) בקצה 3'. במקרה של דגימות רבות הזקוקות לזיהוי, הכינו כמות משמעותית של תמיסה B, ולאחר מכן חלקו את הדגימות למנות מאוחר יותר.
- הכן את פתרון A באופן הבא: עבור כל תגובה, הוסף 29.5 מיקרוליטר של מאגר התייבשות ללא פריימר, 2.4 מיקרוליטר של 10 מיקרומטר A. baumannii-F ו-10 מיקרומטר A. baumannii-R, ו-12.2מיקרוליטר של ddH2O כדי להשיג נפח סופי של 46.5 מיקרוליטר מערבולת וסובב לזמן קצר. לאחר מכן, הוסף את תערובת התגובה לצינורות תגובת האנזים מערכת RPA. פיפטה לערבב.
- הגברת DNA איזותרמית (RPA)
- הוסף 1 מיקרוליטר של הפלסמיד הרקומביננטי החיובי של 107 עותקים/מיקרוליטר לתמיסה A עם זוגות פריימר שונים; מערבבים היטב. לאחר מכן, הוסיפו לתמיסה 2.5 מיקרוליטר של 280 מ"מ מגנזיום אצטט (MgOAc) וערבבו היטב.
הערה: תגובות RPA מתחילות ברגע שמוסיפים MgOAc. - יש לדגור בטמפרטורה של 39 מעלות צלזיוס למשך 20 דקות. לאחר 20 דקות, טהר את המוצרים המוגברים עם פריימרים שונים באמצעות RPA עם ערכת טיהור, ולאחר מכן הפעל את המגפליקונים הנקיים על ג'ל אגרוז 1% ב-120 וולט למשך 10 דקות כדי לבחור את הרצועה הבהירה והרחבה ביותר כזוג הפריימר הטוב ביותר.
הערה: למספר עותק תבנית נמוך, לאחר 4 דקות, הסר את צינור ה-EP, המערבולת וסובב לזמן קצר; לאחר מכן החלף אותו במכשיר החימום למשך 16 דקות נוספות. לאחר ההגברה, יש לפתוח את הצינורות בזהירות רבה כדי למנוע זיהום משטחי עבודה במגברים. אמצעי זהירות זה יפחית את הסיכון לגרימת תוצאות חיוביות כוזבות בהליכים ניסיוניים הבאים.
- הוסף 1 מיקרוליטר של הפלסמיד הרקומביננטי החיובי של 107 עותקים/מיקרוליטר לתמיסה A עם זוגות פריימר שונים; מערבבים היטב. לאחר מכן, הוסיפו לתמיסה 2.5 מיקרוליטר של 280 מ"מ מגנזיום אצטט (MgOAc) וערבבו היטב.
- אופטימיזציה של מערכת הגברה RPA
הערה: זוג הפריימר האופטימלי F3R3 שנבחר בשלב הקודם הוגדר לריכוזים סופיים שונים עבור תגובת הגברה של RPA כדי לסנן את ריכוז הפריימר והזמן האופטימליים. NC הוא בקרה ריקה עבור ריכוז הפריימר המתאים.- שמור על ריכוזי הרכיבים האחרים קבועים ועל הנפח הכולל של 46.5 מיקרוליטר בתמיסה A. התאם את נפח ה-ddH2O נטול RNase בהתאם כדי להשיג את ריכוזי הפריימר הסופיים של 10 μM F3 ו-10 μM R3 ב-0.40 μM (2.0 μL), 0.44 μM (2.2 μL), 0.48 μM (2.4 μL) ו-0.52 μM (2.6 μL), בהתאמה.
- דגירה בטמפרטורה של 39 מעלות צלזיוס לזמנים שונים: 10 דקות, 20 דקות, 30 דקות.
- נקו את מוצרי ה-RPA באמצעות ערכת טיהור DNA ובצעו אלקטרופורזה על ג'ל אגרוז 1% ב-120 וולט למשך 10 דקות כדי לזהות את ריכוז הפריימר וזמני הדגירה האופטימליים על ידי ניתוח מאפייני הרצועות.
- הדמיה של בדיקת RPA-CRISPR/Cas12a
- מוסיפים 5 מיקרוליטר ממוצר ה- RPA לתמיסה B ומערבבים היטב עד לקבלת תערובת אחידה.
- הנח את הדגימות במכשיר ה-PCR הכמותי הקרינה, הגדר את ערוץ ה-FAM וקרא את הערכים כל דקה במשך 60 דקות ב-37 מעלות צלזיוס, פי 60 בסך הכל 60 דקות.
הערה: אות פלואורסצנטי ירוק יכול להימשך מספר ימים. כדי לוודא שהמערכת פועלת ביעילות, עדיף לבצע בדיקת DNA ו-RNase ללא DNASE.
2. הערכת ספציפיות של פלטפורמת הזיהוי מבוססת RPA-CRISPR/Cas12a
הערה: כדי לבדוק את הספציפיות של A. baumannii-DETECTR, חומצות הגרעין של A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Cockerella burnetii, Serratia ודגימות אנושיות הועברו לבדיקת DETECTR.
- השתמשו בתבניות דנ"א שהתקבלו ממינים שונים על ידי שימוש בערכת הדנ"א המוזכרת או בידודן על ידי הרתחת החיידקים במים. ודא שלכל התבניות יש ריכוזים שווים. השתמש במים כבקרה שלילית.
- הוסף 10 ננוגרם או 1 מיקרוליטר של תבניות DNA לפתרון A המכיל פריימרים F3R3. מוסיפים 2.5 מיקרוליטר MgOAc, מערבבים היטב ודוגרים את התערובת בחום של 39 מעלות צלזיוס למשך 20 דקות.
- לאחר 20 דקות מוסיפים 5 מיקרוליטר מהמוצר מהשלב הקודם לתמיסה B ומערבבים.
- הנח במכשיר ה-PCR הכמותי הקרינה, הגדר את הערוץ ל-FAM וקרא את הערך אחת לדקה ב-37 מעלות צלזיוס, בסך הכל 60 x למשך 60 דקות.
3. הערכת רגישות של פלטפורמת הזיהוי מבוססת RPA-CRISPR/Cas12a
- השתמש בפלסמיד רקומביננטי חיובי, שהוכן לריכוז של 1-107 עותקים/מיקרוליטר, כתבנית לתגובה. השתמש במים לשליטה שלילית.
- בצע את השיטה על ידי ביצוע שלבים 2.2-2.4.
הערה: ערכת הפריימר RPA-F3 ו-RPA-R3 נועדה להגביר שבר קצר יותר של 174 זוגות בסיסים מהפלסמיד הרקומביננטי החיובי.
4. הכנות בזמן שהדגימה נמצאת תחת אור UV וזיהוי LFS
- הוצא את המספר הנדרש של רצועות בדיקה ותייג אותן כראוי.
- העבירו 10 מיקרוליטר מהמוצרים המוגברים משלבים 2 ו-3 לצינורות PCR נפרדים, בהתאמה. לאחר מכן, הוסף 40 מיקרוליטר של ddH2O לכל צינור.
הערה: אם משתמשים במערכת תגובה בהכנה עצמית, מומלץ לבחון את יחס הדילול האופטימלי. - בצע את הבדיקה.
- הכניסו את צינור ה-PCR המכיל את המוצרים המוגברים לטרנס-מאיר כדי להעריך את נוכחות הקרינה.
- הכנס את רצועות הבדיקה לתוך צינורות ה-PCR כשקצה כרית הדגימה פונה כלפי מטה.
- תן לו לעמוד בטמפרטורת החדר למשך 5-10 דקות כדי לקרוא את התוצאה. לאחר זמן זה, ודא שמפלס הנוזל אינו חורג מהקו המקסימלי לפני שתמשיך הלאה.
- פרש את התוצאות כשליליות אם קו T אינו מציג צבע. פרש את התוצאות כחיוביות אם קו ה-T נראה לעין בלתי, מה שמצביע על כך שבדיקת חומצת הגרעין נבקעה על ידי האנזים Cas, והאנזים Cas הופעל. קחו בחשבון את התוצאה כלא חוקית אם לא בקו C ולא בקו T מופיע צבע.
תוצאות
במחקר זה, אנו מציגים פלטפורמת אבחון חדשה וניידת בשם A. baumannii-DETECTR, המשלבת את יעילות ההגברה האיזותרמית של מערכת RPA ו-CRISPR-Cas12a לזיהוי שדה מהיר ואמין של A. baumannii. הסכימה של מבחן ה-DETECTR מודגמת באיור 1.
שנים עשר זוגות פריימרים תוכננו באמצעות כלי עיצוב על בסיס עקרונות עיצוב פריימר. התקבלו ארבעה פריימרים קדמיים (F0-F3) ושלושה פריימרים הפוכים (R1-R3), וכתוצאה מכך נוצרו 12 זוגות פריימרים (F0R1, F0R2, F0R3, F1R1, F1R2, F1R3, F2R1, F2R2, F2R3, F3R1, F3R2, F3R3). לאחר הגברת RPA ב-39 מעלות צלזיוס למשך 20 דקות, מוצרי RPA PCR נותחו על ג'ל אגרוז 1% ב-120 וולט למשך 10 דקות כדי לקבוע את שילוב הפריימר האופטימלי. זוג הפריימר F3R3 התגלה כיעיל ביותר, ומציג את הפסים הבהירים והצפופים ביותר (איור 2).
לאחר שזיהינו את זוג הפריימר האופטימלי F3R3, לאחר מכן ביצענו אופטימיזציה של ריכוזי הפריימר הסופיים וזמני התגובה להגברת ה-RPA, תוך שמירה על כל שאר הרכיבים קבועים. הממצאים שלנו גילו שיעילות ההגברה הגיעה לשיא בריכוז פריימר של 0.44 מיקרומטר וזמן תגובה של 20 דקות (איור 3). באמצעות פלסמיד רקומביננטי חיובי בריכוז של 107 עותקים/מיקרוליטר כתבנית, ראינו עלייה תלוית זמן בעוצמת הקרינה עבור crRNA-1, בעוד ש-crRNA-2 לא הראה עלייה כזו לאורך זמן. כתוצאה מכך, crRNA-1 נבחר כ-crRNA לגילוי A. baumannii (איור 4).
הספציפיות של A. baumannii-DETECTR הוערכה באמצעות חומצות גרעין שבודדו ממערך מגוון של זני חיידקים. במהלך מבחני ה-PCR, אות פלואורסצנטי משמעותי נצפה רק כאשר הגנום של A. baumannii היה ממוקד, בעוד שלא זוהתה פלואורסצנטיות בעת שימוש בדנ"א גנומי ממקורות חיידקים אחרים (איור 5). תוצאות אלו מאשרות כי זוג הפריימר A. baumannii-F3 ו-R3 מציג ספציפיות עבור A. baumannii בבדיקת DETECTR, מה שמצביע על כך ש-A. baumannii-DETECTR יכול לזהות פתוגן זה באופן מהימן.
כדי להעריך את הרגישות של בדיקת A. baumannii-DETECTR, ביצענו סדרה של דילולים עם ה-DNA של A. baumannii . מספר העותק חושב באמצעות הנוסחה הבאה: מספר עותק (עותקים/מיקרוליטר) = 6.02 × 1023 x P (ng/μL) x 10-9 / (מספר בסיסי הפלסמיד x 660) (כאשר P מייצג את ריכוז הפלסמיד). הבדיקה הדגימה את היכולת לזהות עד עותק 1/μL, מה שמצביע על הפוטנציאל שלו ככלי דיאגנוסטיקה רגיש להפליא, המתאים היטב עבור יישומים באתר (איור 6). לפיכך, ה-A. baumannii-DETECTR מפגין רגישות יוצאת דופן.
בפרוטוקול זה, שילבנו גם את פלטפורמת הזיהוי DETECTR בתיווך CRISPR/Cas12a עם טכנולוגיית רצועת זרימה רוחבית (LFS) כדי להקל על זיהוי רצפי DNA מטרה. הערכנו את הרגישות והספציפיות של הבדיקה המבוססת על LFS. בדיקת ספציפיות שנערכה באמצעות LFS לא הראתה סימנים של תגובתיות צולבת עם מיני חיידקים אחרים, מה שמדגים את הספציפיות הגבוהה של השיטה (איור 7A). יתר על כן, השתמשנו ב-LFS כדי לזהות A. baumannii בטווח של דילולים מ-107 עותקים/מיקרוליטר ועד עותק בודד/מיקרוליטר בודד. יש לציין שכל הדגימות שנבדקו הניבו תוצאות חיוביות (איור 7B), שהיו תואמות את התוצאות תחת אור UV (איור 7C, D). באופן קולקטיבי, תוצאות אלה מדגישות כי הפלטפורמה שלנו משמשת כמכשיר אבחון מהיר, חזק ורגיש עבור A. baumannii בסביבות שדה.

איור 1: זרימת עבודה של A. baumannii-DETECTR. קיצורים: RPA = הגברת פולימראז אקומבינאז R; DETECTR = DNA אנדונוקלאז ממוקד CRISPR Trans Reporter. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2: תכנון ובחירה של פריימרים. השילוב האופטימלי של פריימרים לתגובת RPA זוהה על סמך התוצאות שהתקבלו מאלקטרופורזה של ג'ל אגרוז. זוג הפריימר F3R3 ביצע את הביצועים הטובים ביותר עם רצועות בהירות וצפופות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 3: אופטימיזציה של ריכוז פריימר וזמן הגברה עבור RPA. 1-4: ריכוזי הפריימר 0.40 מיקרומטר, 0.44 מיקרומטר, 0.48 מיקרומטר, 0.52 מיקרומטר. זמני ההגברה הם 10 דקות, 20 דקות ו-30 דקות בהתאמה. אפקט ההגברה מגיע לשיאו כאשר ריכוז הפריימר הוא 0.44 מיקרומטר וזמן התגובה הוא 20 דקות. קיצורים: M = 8000 סמן DNA; RPA = הגברת פולימראז רקומבינאז; NC = שליטה ריקה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
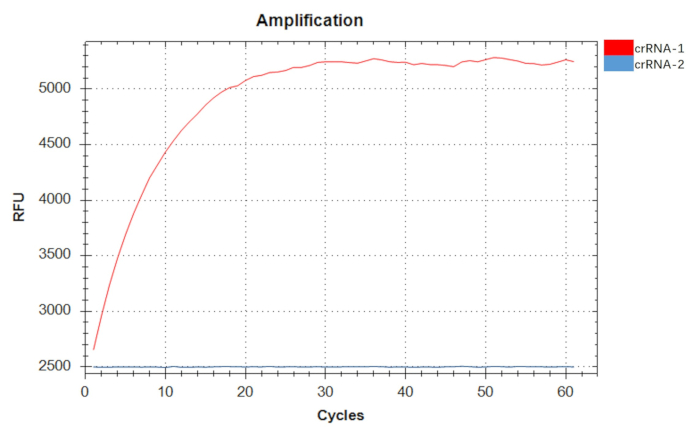
איור 4: הקרנה של שני crRNAs (crRNA-1 ו-crRNA-2). עוצמת הקרינה של זיהוי crRNA-1 עלתה עם הזמן, בעוד זו של זיהוי crRNA-2 לא עלתה עם הזמן, ולא הראתה פעילות זיהוי משמעותית. crRNA-1 שימש כ-crRNA לגילוי A. baumannii. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
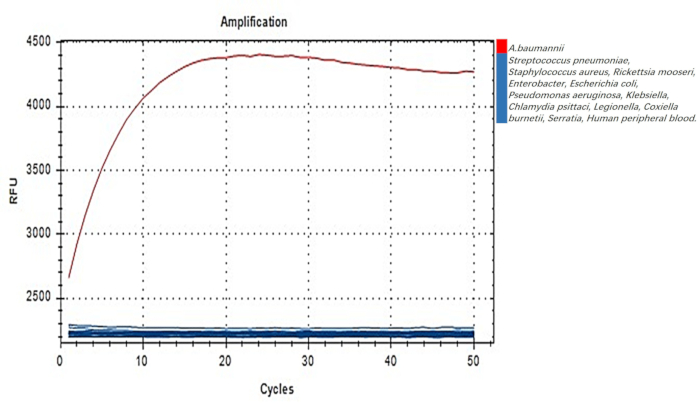
איור 5: בדיקת הספציפיות של ה-A. baumannii-DETECTR. ההגברה של A. baumannii-DETECTR הושגה באמצעות צמד הפריימרים A. baumannii-F3 ו-R3, תוך שימוש ב-DNA גנומי שהופק מ-A. baumannii כמו גם ממיני חיידקים אחרים. קיצור: DETECTR = DNA אנדונוקלאז ממוקד CRISPR Trans Reporter. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
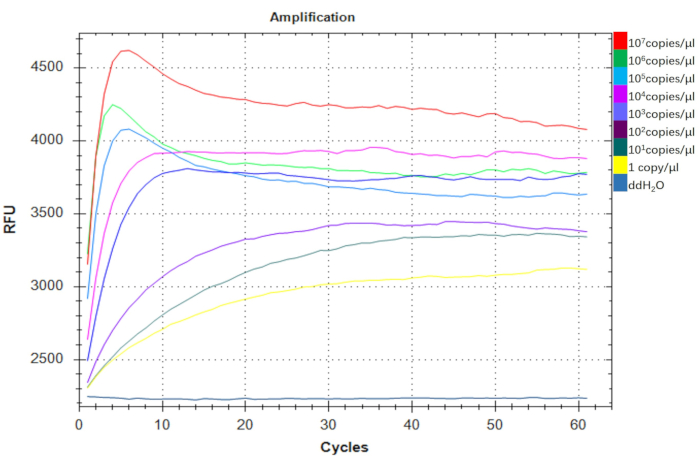
איור 6: בדיקת הרגישות של A. baumannii-DETECTR. הפלסמידים הרקומביננטיים החיוביים pUC57-16s rDNA שימשו כמטרות להערכת גבול הזיהוי של A. baumannii באמצעות בדיקת RPA-CRISPR/Cas12a מבוססת פלואורסצנטיות חזותית. הבדיקה זיהתה בהצלחה טווח של עותק אחד עד 107 עותקים/מיקרוליטר באמצעות פריימרים עקביים. עוצמת הקרינה בקבוצת העותק היחיד הייתה גבוהה משמעותית בהשוואה לקבוצת הביקורת ddH2O. קיצורים: DETECTR = DNA Endonuclease Targeted CRISPR Trans Reporter; RPA = הגברת פולימראז רקומבינאז. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
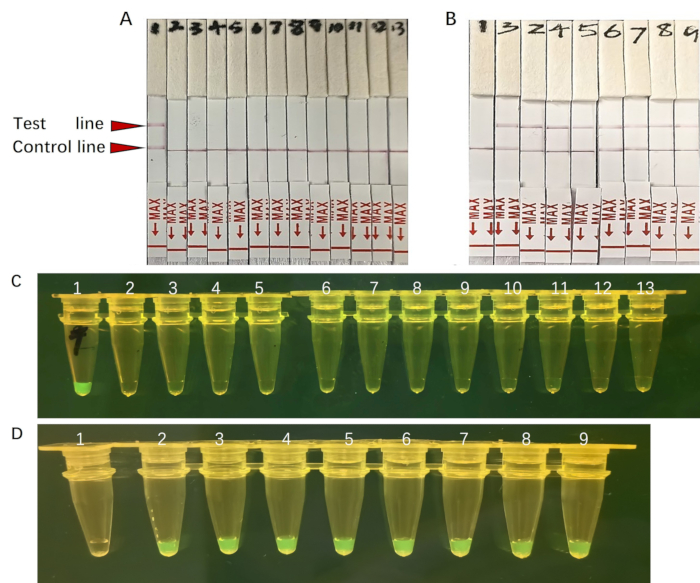
איור 7: אות הקרינה של הדגימה תחת אור UV וגילוי LFS.A. תגובת baumannii-DETECTR עם תקני פלסמיד. (א-ג) פתוגנים שונים זוהו על ידי A. baumannii-DETECTR. רק A. baumannii זוהה בהצלחה. 1-13 מייצגים את A. baumannii, Streptococcus pneumoniae, Staphylococcus aureus, Rickettsia mooseri, Enterobacter, Escherichia coli, Pseudomonas aeruginosa, Klebsiella, Chlamydia psittaci, Legionella, Coxiella burnetii, Serratia, היקפי אנושי. (B-D) הפלסמיד הרקומביננטי החיובי עם דילול סדרתי במרווחים של פי עשרה שזוהה על ידי A. baumannii-DETECTR. כולם זוהו למעט הביקורת השלילית. 1-9 מייצגים בקרה שלילית, 1 עותק/מיקרוליטר, 101 עותקים/מיקרוליטר, 102 עותקים/מיקרוליטר, 103 עותקים/מיקרוליטר, 104 עותקים/מיקרוליטר, 105 עותקים/מיקרוליטר, 106 עותקים/מיקרוליטר, 107 עותקים/מיקרוליטר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| לא | שם | רצף (5'-3') | |||
| 1 | crRNA-1 | uaauuucuacuaaguguagauGUUAAUACCUAGAGAUAGUG | |||
| 2 | crRNA-2 | uaauuucuacuaaguguagauGUUGGUAUUCCGGAAGAAGU | |||
| 3 | crRNA-1-DNA | GTTAATACCTAGAGATAGTG | |||
| 4 | crRNA-2-DNA | GTTGGTATTCCGGAAGAAGT | |||
| 5 | רצף מסומן | TAACTGAAGAGTTTGATCATGGCTCAGATTGAACGCTGG CGGCAGGCTTAACACATGCAAGTCGAGCGGGGGAAGGT AGCTTGCTACTGGACCTAGCGGCGGACGGGTGAGTAATG CTTAGGAATCTGCCTATTAGTGGGGGACAACATCTCGAAA GGGATGCTAATACCGCATACGTCCTACGGGAGAAAGCAG GGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCG GATTAGCTAGTTGGTGGTTAAAGGCCTACCAAGGCGAC GATCTGTAGCGGGTCTGAGAGGATCCGCCACACTGG GACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGT GGGATATTGGACAATGGGGGACCCTGATCCAGCCAT GCCGCGTGTGTGAAGAAGGCCTTATGGTTGTAAAGCACTT TAAGCGAGGAGGAGGCTACTTTAGTTAATACCTAGAGATAG TGGACGTTACTCCAGAATAAGCACCGGCTAACTCTGTGC CAGCAGCCGCGGTAATACAGAGGGTGCGAGCGTTAATCGG ATTTACTGGGCGTAAAGCGTGCGGTAGGCGGCTTATTAAGTC GGATGTGAAATCCCCGAGCTTAACTTGGGAATTGCATTCGAT ACTGGTGAGCTAGAGTATGGGAGAGGATGGTAGAATTCCAG GTGTAGCGGTGAAATGCGTAGAGATCTGGAGGAATACCGAT GGCGAAGGCAGCCATCTGGCCTAATACTGACGCTGAGGTAC GAAAGCATGGGGAGCAAACAGGATTAGATACCCTGGTAGTC CATGCCGTAAACGATGTCTACTAGCCGTTGGGGCCTTGAGG CTTTAGTGGCGCAGCTAACGCGATAAGTAGACCGCCTGGGGA GTACGGTCGCAAGACTAAAACTCAAATGAATTGACGGGGGCC CGCACAAGCGGTGGAGCATGTGTTTTAATTCGATGCAACGC GAAGAACCTTACCTGGCCTTGACATACTAGAAACTTTCCAGA GATGGATTGGTGCCTTCGGGAATCTAGATACAGGTGCTGCAT GGCTCGTCAGCTCGTCGTCGTGAGATGTTGGTTTAAGTCC CGCAACGAGCGCAACCCTTTTTCTTACTTGCCAGCATTTCG GATGGGAACTTTAAGGATACTGCCAGTGACAAACTGGAGGAA GGCGGACGACGTCAAGTCATCATGGCCCTTACGGCCAGG GCTACACACGTGCTACAATGGTCGGTACAAAGGGTTGCTACA CAGCGATGATGCTAATCTCAAAAAGCCGATCGTAGTCCGG ATTGGAGTCTGCAACTCGACTCCATGAAGTCGGAATCGCTAG TAATCGCGGATCAGAATGCCGCGGTGAATACGTTCCCGGGCC TTGTACACACCGCCCGTCACACACCATGGGAGTTTGTTGCACCA GAAGTAGCTAGCCTAACTGCAAAGAGGGCGGTTACCACGGTG TGGCCGATGACTGGGGTGAAGTCGTAACAAGGTAGCCGTAGG GGAACCTGCGGCTGGATCACCTCCTTA | |||
| 6 | א.באומני-ו0 | CGGGAGGCAGCAGTGGGGAATATTGGACAAT | |||
| 7 | A.baumannii-F1 | AAGAAGGCCTTATGGTTGTAAAGCACTTTAA | |||
| 8 | A.baumannii -F2 | GGTTGTAAAGCACTTTAAGCGAGGAGGGCTA | |||
| 9 | A.baumannii -F3 | AAGGCCTTATGGTTGTAAAGCACTTTAAGCGA | |||
| 10 | A.baumannii-R1 | ACAGAGTTAGCCGGTGCTTATTCTGCGAGTAA | |||
| 11 | A.baumannii-R2 | AATCCGATTAACGCTCCCACCCTCTGTATTA | |||
| 12 | A.baumannii-R3 | AGTTAGCCGGTGCTTATTCTGCGAGTAACGTC | |||
| 13 | ssDNA-FQ | ssDNA-FQ FAM-TTTATTT-BHQ1 | |||
| 14 | A.baumannii-16s rDNA-F | 5′-CGCGGATCCTAACTGAAGAGTTTGATCATGG-3′ | |||
| 15 | A.baumannii-16s rDNA-R | 5′-ACGCGTCGACTAAGGAGGTGATCCAGC-3′. | |||
טבלה 1: חומצות גרעין ששימשו במחקר זה. חומצת הגרעין המשמשת כאן היא ssDNA-FQ, המורכבת מ-DNA חד-גדילי מצומד עם 5-Carboxyfluorescein בקצה 5' ומרווה החור השחור הקרינה 1 בקצה ה-3'. קיצורים: F = פלואורופור; Q = מרווה.
קובץ משלים 1: תכנון פריימרים ו-crRNA.אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
לשיטות אבחון מסורתיות לזיהומי A. baumannii יש מגבלות שונות שהופכות אותן לפחות נגישות ואפשריות לבדיקות נקודתיות27. לדוגמה, ל-PCR יש רגישות וספציפיות טובות אך מחייב ציוד מיוחד לרכיבה תרמית ותהליכי עבודה מורכבים שחייבים להיות מופעלים על ידי אנשי מקצוע. השיטה החדשנית המוצגת כאן, המשלבת עריכת גנום RPA ו-CRISPR-LbaCas12a עם רקומבינריה, הידועה בשם מבחן A. baumannii-DETECTR, מאפשרת מניפולציה גנטית יעילה. הוא מספק שיטת זיהוי מהירה, רגישה וספציפית לזיהומי Acinetobacter baumannii , כמו גם לאבחון פתוגנים חיידקיים אחרים. זרימת העבודה מוצגת באיור 1.
השיטה שלנו מועילה במיוחד באבחון וניהול זיהומים, מה שאמור להקל על החלמת המטופלים ולהפחית את התפשטות A. baumannii על ידי התגברות על החסרונות של טכניקות האבחון הקיימות. מערכת A. baumannii-DETECTR נמצאה כמזהה A . baumannii עם סגוליות ורגישות גבוהות (איור 5 ואיור 6). הממצאים שלנו חשפו שרק הגנום של A. baumannii הראה שינוי חיובי בקפל הקרינה במהלך ההערכה בזמן אמת, בעוד שהגנומים של כל המינים האחרים הראו תוצאות שליליות (איור 5).
כדי להעריך את הרגישות של פלטפורמת הזיהוי RPA-CRISPR/Cas12a, פלסמידים רקומביננטיים pUC57-16s rDNA, שהם חיוביים ל-A. baumannii, שימשו כמטרות לקביעת גבול הזיהוי (LOD) של המערכת. בבדיקת RPA-CRISPR/Cas12a מבוססת פלואורסצנטיות, הגילוי נע בין עותק אחד למיקרוליטר ל-10,7 עותקים למיקרוליטר, עם שיפור ניכר בעוצמת הקרינה שנצפה בקבוצת העתק/מיקרוליטר 1 בהשוואה לקבוצת הביקורת השלילית (איור 6). חשוב לציין, לאבחון בזמן אמת, ניתן להמחיש את תוצאות הזיהוי ישירות באמצעות מכשיר נייד כף יד, ללא תלות בציוד מבוסס מעבדה, החיוני לאבחון בזמן אמת.
יש לקחת בחשבון מספר גורמים קריטיים בנוגע לפרוטוקול26. ראשית, חיוני למנוע זיהום צולב במהלך תהליך איסוף הדגימות, בהתחשב ברגישות הגבוהה של בדיקת A. baumannii-DETECTR. בנוסף, יש להגן על ריאגנטים מפני חשיפה ישירה לאור השמש. הקפדה על כל השלבים הפרוצדורליים היא חיונית להשגת התוצאות הטובות ביותר האפשריות. יש לשלב בקרה חיובית, המורכבת מפלסמיד המכיל שברי גנים ספציפיים ל-A. baumannii, כדי לאמת את הפונקציונליות הנכונה של מערכת A. baumannii-DETECTR ביישומים מעשיים.
הפרוטוקול מראה שניתן לבצע אופטימיזציה של פרמטרים עבור תנאי מעבדה ספציפיים וסוגי דגימות, כגון ריכוז פריימר, זמן תגובה ומשתנים אחרים. בניסוי זה, נבחרו שילוב הפריימר האופטימלי, ריכוזי הפריימר וזמני התגובה להגברת RPA כדי לייצר את יעילות ההגברה והספציפיות הגבוהות ביותר. זה נקבע על ידי אלקטרופורזה של ג'ל אגרוז (איור 2 ואיור 3). בנוסף, פלסמיד רקומביננטי חיובי, המכיל אזור שמור בגנום A. baumannii שימש כבקרה להבטחת דיוק בפרוטוקול. ראוי לציין כי ערכות מיצוי DNA גנומיות חיידקיות מסחריות מספקות שיטה סטנדרטית ואמינה למיצוי DNA בפרוטוקול זה. עם זאת, במחקרים עתידיים, סוגי דגימות שונים, כגון דגימות קליניות או סביבתיות, עשויים לדרוש שיטות מיצוי ספציפיות28 כדי לבודד ביעילות את ה-DNA של A. baumannii.
בהיעדר אותות פלואורסצנטיים הניתנים לזיהוי מהבקרה החיובית, סביר להניח כי מעכבים קיימים בתערובת התגובה, מה שמחייב החלפת ריאגנטים. לעומת זאת, אם עוצמת הקרינה אינה מספיקה להתמיינות ברורה, הארכת משך הדגירה עשויה להועיל. עם זאת, דגירה ממושכת עלולה לגרום גם לתוצאות חיוביות כוזבות26.
מתודולוגיות זיהוי נוכחיות עבור A. baumannii מחייבות שימוש בציוד PCR יקר, מיקומים תפעוליים קבועים וכוח אדם מיוחד. לעומת זאת, השיטה המוצעת כאן מאפשרת אבחון מדויק ורגיש של A. baumannii במסגרות שדה, מה שהופך אותו לנגיש לדמוגרפיה רחבה יותר.
המאפיינים של הגברה איזותרמית ונתונים שנאספו באמצעות פלואורסצנטיות הופכים את זרימת העבודה של A. baumannii-DETCTER לדרישות ציוד פשוטות ונמוכות, כמו גם מאפשרים לבצע זיהוי מהיר של פתוגנים באזורים מרוחקים או במהלך התפרצויות. RPA-LFS 29,30,31,32, טכניקה חדשה להגברת חומצות גרעין איזותרמיות, צברה אחיזה משמעותית באיתור פתוגנים ויראליים, חיידקיים, פטרייתיים וטפילים בשל פשטותה התפעולית והפחתת הדרישות הזמניות. הפרוטוקול מפרט את אות הקרינה של הדגימה תחת אור אולטרה סגול (UV) או Transilluminator וזיהוי רצועות זרימה רוחביות (LFS). זה יכול למנוע תלות במכשירים בעלי דיוק גבוה וטכנאים מומחים, שכן בדיקה ויזואלית מביאה לתוצאה תוך זמן קצר (איור 7), ובכך לבטל את הצורך בציוד מעבדה יקר ומורכב ואת האוכלוסייה המבצעית. יתר על כן, שיטת הצינור האחד לא רק חוסכת זמן אלא גם מפחיתה את הסיכון לזיהום אמפליקון33.
מחקרים עתידיים צריכים לשאוף לשפר את השיטה כדי להפוך אותה למעשית עבור סוגים שונים של דגימות ותנאים סביבתיים שונים, כגון חקר טכניקות מיצוי DNA יעילות יותר ובעלות נמוכה יותר. השימוש ב-A. baumannii-DETECTR אינו מיועד רק לאבחון קליני אלא יכול לשמש גם להערכה סביבתית, ניטור וניהול התפרצויות, מה שמאפשר לקהילות ולבתי חולים לזהות במהירות את A. baumannii. יתר על כן, על ידי שינוי ה-crRNA והפריימרים, ניתן להתאים גישה אבחנתית מולקולרית זו לאיתור זיהומים חיידקיים שונים בדרכי הנשימה, כגון Streptococcus pneumoniae, Staphylococcus aureus ו-Enterobacter.
לסיכום, A. baumannii-DETECTR יכול לזהות את A. baumannii במהירות ובדייקנות האפשרית בשל הרגישות והספציפיות הגבוהות שלו, העיצוב הידידותי למשתמש והניידות שלו. שיטה זו יכולה לשפר את תוצאות בריאות המטופל על ידי הקלה על טיפול יעיל ומהיר, בקרת זיהומים והפחתת התפשטות העמידות לאנטיביוטיקה, תוך התמודדות עם האיום הגובר של זיהומי A. baumannii .
Disclosures
המחברים מצהירים שאין להם אינטרסים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי פרויקט תוכנית פיתוח המדע והטכנולוגיה של מחוז ג'ילין, סין (20240305027YY) ומחלקת הכספים של מחוז ג'ילין (JLSWSRCZX2023-55, JLSWSRCZX2021-041).
Materials
| Name | Company | Catalog Number | Comments |
| -20 °C Freezer | Haier | HYCD-290 | China |
| Agarose Basic | BioFroxx | 1110GR100 | China |
| All oligonucleotides and crRNA were synthesized by company | www.comatebio.com | ||
| Cas12a cutting substrate - ssDNA - fluorescent type | EZassay Biotech. Co. Ltd. | DNA-FAM-BHQ | China |
| Cas12a cutting substrate - ssDNA - test paper type | EZassay Biotech. Co. Ltd. | DNA-FAM-BIO | China |
| Electrophoresis apparatus | BIO-RAD | POWER PAC1000 | USA |
| Fluorescence quantitative PCR instrument | BIO-RAD | CFX Connect | USA |
| Gel Imaging System | BIO-RAD | Gel Doc 2000 | USA |
| https://ezassay.com/rna | |||
| https://www.ezassay.com/primer | |||
| https://www.ncbi.nlm.nih.gov/tools/primer-blast | |||
| Lateral flow paper strip (Biotin/FAM) | EZassay Biotech. Co. Ltd. | HD-FMBO | China |
| LbaCas12a (Cpf1) enhanced protein | EZassay Biotech. Co. Ltd. | CAS-12E-001 | China |
| LED Transilluminator | LABGIC | BL-20 | China |
| Magnesium acetate, MgOAc | TwistDx | TABAS03KIT | UK |
| Microcentrifuge | allsheng | Mini-6k | China |
| PCR strip tubes | PCR strip tubes | PST-0208-FT-C | China |
| TGrade Dry Bath Incubator | Tiangen biochemical technology | OSE-DB-01 | China |
| Tianamp Bacteria DNA Kit | Tiangen biochemical technology | DP302-02 | China |
| TIANamp Bacteria DNA Kit | TIANGEN BIOTECH (BEIJING) CO.; LTD. | DP302 | China |
| TransStart FastPfu DNA Polymerase | TransGen Biotech. Co. Ltd. | AP221 | China |
| TwistAmp Basic Kit | TwistDX | TABAS03KIT | UK |
| Universal DNA Purification Kit | Tiangen biochemical technology | DP214-03 | China |
References
- Lenie, D., Alexandr, N., Harald, S. An increasing threat in hospitals: Multidrug-resistant Acinetobacter baumannii. Nat Rev Microbiol. 5 (12), 939-951 (2007).
- Li, P., et al. Rapid detection of Acinetobacter baumannii and molecular epidemiology of carbapenem-resistant A. baumannii in two comprehensive hospitals of Beijing, China. Front Microbiol. 6, 997 (2015).
- Wu, X., et al. A diagnostic test that uses isothermal amplification and lateral flow detection sdaA can detect tuberculosis in 60 min. J Appl Microbiol. 130 (6), 2102-2110 (2020).
- Huang, B., et al. A cas12a-based fluorescent microfluidic system for rapid on-site human papillomavirus diagnostics. Appl Microbiol Biotechnol. 107 (20), 6287-6297 (2023).
- Piepenburg, O., Williams, C. H., Stemple, D. L., Armes, N. A. DNA detection using recombination proteins. Plos Biology. 4 (7), 1115-1121 (2006).
- Abudayyeh, O. O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), aaf5573 (2016).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), eaar6245 (2018).
- Xiong, D., et al. Rapid detection of SARS-CoV-2 with CRISPR-Cas12a. PLoS Biol. 18 (12), e3000978 (2020).
- Gootenberg, J. S., et al. Nucleic acid detection with crispr-Cas13a/C2C2. Science. 356 (6336), 438-442 (2017).
- Harrington, L. B., et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science. 362 (6416), 839-842 (2018).
- Li, S. Y., et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA. Cell Res. 28 (4), 491-493 (2018).
- Wang, B., et al. Cas12aVDet: A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem. 91 (19), 12156-12161 (2019).
- Huang, Z., et al. Ultra-sensitive and high-throughput crispr-p owered COVID-19 diagnosis. Biosens Bioelectron. 164, 112316 (2020).
- Zhang, W. S., et al. Reverse transcription recombinase polymerase amplification coupled with CRISPR-Cas12a for facile and highly sensitive colorimetric SARS-CoV-2 detection. Anal Chem. 93 (8), 4126-4133 (2021).
- Marraffini Luciano, A. CRISPR-Cas immunity in prokaryotes. Nature. 526 (7571), 55-61 (2015).
- Mohanraju, P., et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353 (6299), aad5147 (2016).
- Makarova, K. S., et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 13 (11), 722-736 (2015).
- Li, S. Y., et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discovery. 4 (1), 20 (2018).
- Li, L., Li, S., Wu, N., Wu, J., Wang, J. Holmesv2: A CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol. 8 (10), 2228-2237 (2019).
- Liang, M., Li, Z., Wang, W., Liu, J., Zhang, L. X. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules. Nat Communications. 10 (1), 3672 (2019).
- Aquino-Jarquin, G. CRISPR-Cas14 is now part of the artillery for gene editing and molecular diagnostic. Nanomedicine. 18, 428-431 (2019).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Swarts, D. C., John, V. D. O., Jinek, M. Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a. Mol Cell. 66 (2), 221-233 (2017).
- Jiang, Y., et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun. 8, 15179 (2017).
- Yan, M. Y., et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol. 83 (17), e00947-e01017 (2017).
- Ashraf, A., et al. A novel multiplex pcr assay for simultaneous detection of nine clinically significant bacterial pathogens associated with bovine mastitis. Mol Cell Probes. 33, 57-64 (2017).
- Zhu, L., et al. A rapid on-site visualization platform based on RPA coupled with CRISPR-Cas12a for the detection of genetically modified papaya 'huanong no.1'. Talanta. 277, 126437 (2024).
- Zheng, C., et al. Rapid developments in lateral flow immunoassay for nucleic acid detection. Analyst. 146 (5), 1514-1528 (2021).
- Wang, Y., et al. Establishment and clinical application of a RPA-LFS assay for detection of capsulated and non-capsulated Haemophilus influenzae. Front Cell Infect Microbiol. 12, 878813 (2022).
- Wang, F., et al. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strips for detecting Candida albicans. Anal Biochem. 633, 114428 (2021).
- Ma, B., et al. A simple and efficient method for potential point-of-care diagnosis of human papillomavirus genotypes: Combination of isothermal recombinase polymerase amplification with lateral flow dipstick and reverse dot blot. Anal Bioanal Chem. 411 (28), 7451-7460 (2019).
- Sun, Y., Yu, L., Liu, C., Ye, S., Huang, W. One-tube SARS-CoV-2 detection platform based on RT-RPA and CRISPR/Cas12a. J Transl Med. 19 (1), 74 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved