Method Article
Çeşitli hücre tiplerinde nükleer cisimlerdeki dinamik değişiklikleri analiz etmek için çok yönlü bir boru hattı
Bu Makalede
Özet
Bu yöntem, insan T lenfositlerinde çeşitli nükleer organizasyon paternleri ile protein dağılımını değerlendirmek için bir immünofloresan protokolünü ve miktar tayini boru hattını tanımlar. Bu protokol, numune hazırlamadan başlayarak Fiji'de yarı otomatik analizin yürütülmesine kadar devam eden ve bir Google Colab not defteri tarafından veri işleme ile sonuçlanan adım adım rehberlik sağlar.
Özet
Transkripsiyonel kontrol gibi çeşitli nükleer süreçler, immünofloresan tekniği ile ayırt edilebilen odaklar olarak bilinen ayrı yapılar içinde meydana gelir. Bu odakların dinamiklerini çeşitli hücresel koşullar altında mikroskopi yoluyla araştırmak, hücresel kimliği ve işlevleri yöneten moleküler mekanizmalar hakkında değerli bilgiler verir. Bununla birlikte, farklı hücre tiplerinde immünofloresan testleri yapmak ve bu odakların montajı, difüzyonu ve dağılımındaki değişiklikleri değerlendirmek çok sayıda zorluk ortaya çıkarmaktadır. Bu zorluklar, numune hazırlama, görüntüleme verilerini analiz etmek için parametrelerin belirlenmesi ve önemli veri hacimlerinin yönetimindeki karmaşıklıkları kapsar. Ayrıca, mevcut görüntüleme iş akışları genellikle yetkin kullanıcılar için uyarlanır ve bu nedenle erişilebilirliği daha geniş bir kitleye sınırlar.
Bu çalışmada, ilgilenilen herhangi bir protein ve hücre tipi için özelleştirilebilen, farklı insan birincil T hücre tiplerinde nükleer proteinleri araştırmak için uyarlanmış optimize edilmiş bir immünofloresan protokolü sunuyoruz. Ayrıca, ister farklı odaklar oluştursunlar, ister dağınık bir nükleer dağılım sergilesinler, protein boyamayı tarafsız bir şekilde ölçmek için bir yöntem sunuyoruz.
Önerilen yöntemimiz, Jython'da geliştirilen ve Fiji'de yürütülebilen yarı otomatik bir boru hattından yararlanarak, hücresel boyamadan analize kadar kapsamlı bir kılavuz sunmaktadır. Ayrıca, veri yönetimini kolaylaştırmak için bir Google Colab not defterinde herkesin erişebileceği kullanıcı dostu bir Python komut dosyası sağlıyoruz. Yaklaşımımız, farklı bağlamlarda çeşitli nükleer organizasyon modellerine sahip proteinler için son derece bilgilendirici immünofloresan analizleri sağlamada etkinlik göstermiştir.
Giriş
Ökaryotik genomun organizasyonu, nükleer cisimler veya yoğunlaşmalar2 adı verilen özel bölmelerde meydana gelebilecek çeşitli nükleer işlevleri koordine eden çok sayıda epigenetik modifikasyon 1 katmanı tarafındanyönetilir. Bu yapılar içinde transkripsiyon başlatma 3, RNA işleme 4,5,6, DNA onarımı 7,8, ribozom biyogenezi 9,10,11 ve heterokromatin regülasyonu12,13 gibi süreçler gerçekleşir. Nükleer cisimlerin düzenlenmesi, faz ayırmailkelerinin rehberliğinde hücresel gereksinimleri karşılamak için hem uzamsal hem de zamansal boyutlarda ayarlama yapar 14,15. Sonuç olarak, bu cisimler, işlevsel bileşenlerin bir araya getirildiği ve söküldüğü, boyut ve mekansal dağılımda değişikliklere uğradığı geçici fabrikalar olarak işlev görür. Bu nedenle, nükleer proteinlerin özelliklerini, cisim oluşturma eğilimleri ve farklı hücresel koşullardaki uzamsal düzenlemeleri de dahil olmak üzere mikroskopi ile anlamak, işlevsel rolleri hakkında değerli bilgiler sunar. Floresan mikroskobu, nükleer proteinleri incelemek için yaygın olarak kullanılan bir yöntemdir, floresan antikorlar aracılığıyla tespit edilmelerine veya bir floresan protein raportörü16,17 ile hedefleri doğrudan ifade etmelerine izin verir.
Bu bağlamda, nükleer cisimler, kayda değer bir küresellik derecesine sahip parlak odaklar veya punkta olarak görünür ve bu da onları çevredeki ortamdan kolayca ayırt edilebilir hale getirir16,18. STORM ve PALM gibi süper çözünürlük teknikleri, gelişmiş çözünürlük (10 nm'ye kadar)19 sağlayarak, belirli kondensatların yapısının ve bileşiminin daha hassas bir şekilde karakterize edilmesini sağlar20. Bununla birlikte, erişilebilirlikleri, ekipman masrafları ve veri analizi için gereken özel becerilerle sınırlıdır. Bu nedenle, konfokal mikroskopi, çözünürlük ve daha geniş kullanım arasındaki elverişli dengesi nedeniyle popülerliğini korumaktadır. Bu popülerlik, doğru segmentasyon için kapsamlı işlem sonrası prosedürlere olan gereksinimi, araştırma enstitülerinde yaygın olarak kullanılabilirliğini, etkili edinme süresini ve tipik olarak verimli olan numune hazırlamayı azaltan odak dışı ışığın doğal olarak kaldırılmasıyla kolaylaştırılır. Bununla birlikte, çeşitli hücresel koşullar boyunca immünofloresan tahlilleri kullanılarak protein dağılımının, montajının veya difüzyonunun doğru bir şekilde ölçülmesi, mevcut birçok yöntemin değişen dağılım modellerine sahip proteinler için uygun parametrelerin seçilmesi konusunda rehberlik gerektirmemesi nedeniyle zorluklar doğurmaktadır21. Ayrıca, ortaya çıkan büyük veri hacminin ele alınması, veri analizi konusunda sınırlı deneyime sahip kullanıcılar için göz korkutucu olabilir ve sonuçların biyolojik önemini potansiyel olarak tehlikeye atabilir.
Bu zorlukların üstesinden gelmek için, çeşitli organizasyon modelleriyle protein boyamayı ölçmek için tarafsız bir yöntem sağlamayı amaçlayan, immünofloresan hazırlama ve veri analizi için ayrıntılı bir adım adım protokol sunuyoruz (Şekil 1). Bu yarı otomatik işlem hattı, hesaplama ve görüntüleme analizinde sınırlı uzmanlığa sahip kullanıcılar için tasarlanmıştır. İki yerleşik Fiji eklentisinin işlevlerini birleştirir: FindFoci22 ve 3D suite23. FindFoci'nin kesin odak tanımlama yeteneğini, 3B paket tarafından sunulan 3B alandaki nesne tanımlama ve segmentasyon özellikleriyle entegre ederek, yaklaşımımız her bir edinme alanı için kanal başına iki CSV dosyası oluşturur. Bu dosyalar, hücre başına odak sayısı, odakların nükleer merkeze olan uzaklığı ve dağınık protein boyama için tanıttığımız homojenlik katsayısı (IC) gibi çeşitli sinyal dağılımı türlerine uygun metriklerin hesaplanmasını kolaylaştıran tamamlayıcı bilgiler içerir. Ayrıca, veri işleme becerilerine sahip kullanıcılar için veri ekstrapolasyonunun zaman alıcı olabileceğini kabul ediyoruz. Bu süreci kolaylaştırmak için, toplanan tüm ölçümleri her deneme için otomatik olarak tek bir dosyada derleyen bir Python betiği sağlıyoruz. Kullanıcılar, herhangi bir programlama dili yazılımı yüklemeye gerek kalmadan bu komut dosyasını çalıştırabilir. Python komut dosyalarının doğrudan tarayıcıda yazılmasına izin veren bulut tabanlı bir platform olan Google Colab'da yürütülebilir bir kod sağlıyoruz. Bu, yöntemimizin sezgisel olmasını ve anında kullanım için kolayca erişilebilir olmasını sağlar.
Protokolümüzün iki nükleer proteinin sinyal dağılımındaki değişiklikleri analiz etmede ve ölçmedeki etkinliğini gösteriyoruz: Bromodomain içeren protein 4 (BRD4) ve zeste-12'nin baskılayıcısı (SUZ12). BRD4, polimeraz II'ye bağlı transkripsiyonel başlatma24,25 ile ilişkili kondensatlar oluşturduğu bilinen Mediator kompleksi içinde iyi belgelenmiş bir koaktivatör proteindir. SUZ12, H3K27me3 histon modifikasyonu 26,27'nin birikimini düzenlemekten sorumlu Polycomb Baskılayıcı Kompleks2'nin (PRC2) bir protein bileşenidir. Bu proteinler, iki farklı hücre tipi içinde farklı desenler sergiler: hareketsiz ve yavaş transkripsiyonel aktivite oranları sergileyen taze izole edilmiş insan CD4+ naif T hücreleri ve in vitro farklılaşmış TH1 CD4 + hücreleri, artan transkripsiyon gösteren özelleşmiş, çoğalan efektör hücreler28.
Protokol
İnsan örneklerinin araştırma amaçlı kullanımı, Fondazione Istituto di Ricovero e Cura a Carattere Scientifico (IRCCS) Cà Granda Ospedale Maggiore Policlinico (Milano) Etik Kurulları tarafından onaylandı ve tüm deneklerden bilgilendirilmiş onam alındı (yetki numaraları: 708_2020). Protokol üç ana bölüme ayrılmıştır: immünofloresan yürütme, görüntü elde etme ve görüntü analizi. Ortalama olarak, tamamlanması 4 iş günü gerektirir (Şekil 1).
1. İmmünofloresan hazırlama
NOT: Bu immünofloresan protokolü, fiksasyon ve geçirgenlik koşullarını ayarlayarak çeşitli hücre tipleri ve protein hedefleri için kolayca özelleştirilebilir. İmmünofloresan preparatının tamamlanması tipik olarak 3 günden az sürer ve primer antikor inkübasyon süresi antikor kalitesine ve hedef proteine bağlı olarak değişir (Şekil 1).
- Numune hazırlama
- İnsan periferik kan mononükleer hücrelerini (PBMC'ler), üreticinin talimatlarına göre (Malzeme Tablosu) yaklaşık 1.077 g / mL yoğunluklu bir ortam aracılığıyla yoğunluk gradyanlı bir santrifüjleme yoluyla izole edin.
- CD4+ T hücrelerini, üreticinin talimatlarını izleyerek manyetik boncuklarla PBMC'lerden izole edin (Malzeme Tablosu).
- Hücreleri CD4, CD45RA ve CD45RO (Malzeme Tablosu) için antikorlarla boyayın ve başka bir yerde açıklandığı gibi saf CD4+ T hücrelerinin CD4 + / CD45RA + / CD45RO- hücreleri olarak FACS sıralaması ile devam edin29,30.
NOT: Bu protokolde kullanılan FACS sıralayıcının özellikleri için Malzeme Tablosuna bakın. - Naif CD4 + T hücrelerinin, 29'da tarif edildiği gibi T yardımcı 1'e (TH1 CD4 + hücreleri) farklılaşmasını indükleyin. Kısaca, 1.5 x 106 hücre/mL FACS ile sıralanmış naif CD4+ T hücrelerinin TH1 besiyerinde kültürlenmesi ve 1:1 oranında anti-CD3/anti-CD28 manyetik boncuklarla uyarılması. Hücreleri sayın ve her 2-3 günde bir 1.5 × 106 hücre / mL'ye ulaştıklarında bölün (T, H,1 orta bileşimi için Tablo 1'e bakınız).
- 29'da tarif edildiği gibi 7 günlük farklılaşmadan sonra efektör fonksiyon sitokinlerinin salgılanmasını değerlendirin.
- Hücre fiksasyonu ve geçirgenlik
NOT: Tüm bu işlemler küçük uyarlamalarla 30,31'de tarif edilmiştir.- Optimum hücre yapışmasını sağlamak için, cam lamellere (10 mm, kalınlık 1,5 H) kaplama solüsyonu (Tablo 1) ile aşağıdaki gibi işlem yapın:
- Cam lamelleri önce damıtılmış su (ddH2O) ile yıkayarak, ardından %70 etanol (EtOH) ile durulayarak ve kurumaya bırakarak temizleyin.
- Yıkanmış lamelleri 1.2.1.3-1.3 adımları için 24 oyuklu bir plakaya yerleştirin.
- Cam lamel üzerine 200 μL'lik bir damla kaplama solüsyonu uygulayın. 5 dakika sonra damlayı çıkarın ve kurumaya bırakın.
- Lamelleri 200 μL ddH2O damla uygulayarak yıkayın. 5 dakika sonra damlayı çıkarın ve havayla kurutun.
- 3 x adım 1.2.1.3-1.2.1.4'ü tekrarlayın.
- Naif CD4 + T hücrelerini ve TH1 CD4 + hücrelerini 1 × 106 hücre / mL konsantrasyonda 1x fosfat tamponlu salin (PBS) içinde yeniden askıya alın.
NOT: Belirtilen konsantrasyon, insan primer T lenfositleri gibi küçük hücreler için özel olarak tavsiye edilir. Yapışık hücreler için cam kaplama işlemi gereksizdir. Bunun yerine, uygun hücre kültürü ortamını kullanarak hücreleri doğrudan cam yüzey üzerinde büyütmeye devam edin. - Cam kapak kızağı üzerine 200 μL'lik bir damla hücre süspansiyonu uygulayın. Hücrelerin 30 dakika oda sıcaklığında (RT) tohumlanmasına izin verin; Ardından, damlayı çıkarın.
- Hücreleri yeni filtrelenmiş %3 paraformaldehit (PFA çözeltisi, Tablo 1) ile RT'de 10 dakika sabitleyin.
- Cam lamelini TPBS (Tablo 1) ile RT'de 3 x 5 dakika yıkayın.
- TPBS'yi çıkarın ve RT'de 10 dakika boyunca geçirgenleştirme çözeltisi (Tablo 1) ekleyin.
- Geçirgenleştirme solüsyonunu atın ve numuneyi 1 ° C'de 1 saatten gece boyunca (AÇIK) 4 ° C'de saklama solüsyonunda (Tablo 4) inkübe edin.
NOT: Bu aşamada, protokol güvenli bir şekilde durdurulabilir ve cam kapak kaymaları, 3-4 hafta boyunca 24 çok kuyulu bir plakada saklama solüsyonunda saklanabilir.
- Optimum hücre yapışmasını sağlamak için, cam lamellere (10 mm, kalınlık 1,5 H) kaplama solüsyonu (Tablo 1) ile aşağıdaki gibi işlem yapın:
- İmmünofloresan
- (İsteğe bağlı) Lameli 24 oyuklu plakadan çıkarın ve kuru buz üzerinde 30 saniye boyunca hızla dondurun, RT'de çözün ve ardından cam lameli önceden doldurulmuş bir kuyuda saklama solüsyonu ile yıkayın.
- (İsteğe bağlı) 3 x adım 1.3.1'i tekrarlayın.
- RT'de 5 dakika geçirgenlik solüsyonunda yıkayın. Ardından, RT'de TPBS ile 2 x 5 dakika yıkayın.
- RT'de 12 dakika boyunca 0.1 N HCl'de inkübe edin.
- 1x PBS'de iki hızlı yıkama gerçekleştirin.
NOT: Donma ve çözülme adımları ve HCl muamelesi dahil olmak üzere belirtilen hücre geçirgenliği koşulları, yoğun şekilde paketlenmiş kromatin ile karakterize edilen hücrelerde nükleer bileşenlerin boyanması için idealdir. Bununla birlikte, daha az sıkıştırılmış kromatin ile karakterize edilen hücrelerle uğraşırken, bu adımların azaltılması veya kaçınılması tavsiye edilir. Ek olarak, sitoplazmik bileşenleri hedeflemek için HCl tedavisini azaltmayı veya ortadan kaldırmayı düşünün. - Hücreleri, antikor seyreltme tamponunda seyreltilmiş birincil antikor (BRD4, 1:500 veya SUZ12, 1:100) ile inkübe edin (Tablo 1) (her cam lamel için 200 μL) 4 ° C'de AÇIK.
NOT: İmmünofloresan, deneysel ihtiyaçlara, ikincil antikorlara ve tespit sistemlerine bağlı olarak birden fazla birincil antikorun çoğullanmasıyla gerçekleştirilebilir. - RT'de hafif çalkalayarak PBS-T (Tablo 3) ile 5 x 1 dakika yıkayın.
- Hücreleri, RT'de 1 saat boyunca antikor seyreltme tamponunda (her cam lamel için 200 μL) seyreltilmiş ikincil antikor ile inkübe edin.
NOT: Deneysel ihtiyaçlara ve mevcut tespit sistemlerine en uygun ikincil antikoru seçin. Her ikincil antikorun, birincil antikorun türetildiği türü hedeflediğinden emin olun. Ek olarak, mikroskobun filtreleri ve ışık kaynağı ile uyumlu floroforları seçin. Seçilen florofor ile nükleer lekenin emisyon spektrumu arasındaki spektral örtüşmeyi önlemek çok önemlidir. - RT'de PBS-T ile 3 x 5 dakika hafif çalkalayarak yıkayın.
- RT'de 5 dakika boyunca 1x PBS'de seyreltilmiş 1 ng / mL 4 ', 6-diamidino-2-fenilindol (DAPI) ile boyayın.
NOT: Alternatif boyalar, ikincil antikorların emisyon spektrumu ile örtüşmedikleri sürece nükleer boyama için kullanılabilir. - 1x PBS'de birkaç hızlı yıkama gerçekleştirin.
- Cam lamelli, antifade montaj ortamı ile bir mikroskopi slaytına monte edin.
2. Görüntü edinme
NOT: Görüntü alma süresi, cihaza ve seçilen ayarlara bağlıdır.
- Konfokal mikroskop edinimi
- Konfokal mikroskop kullanarak, z'de 0,25 μm'lik bir adım boyutu ve 0,1-0,2 μm'lik piksel boyutu ayarlayarak 3D görüntüler yakalayın.
NOT: 63x 1,4 NA Yağ objektifimizle optimum ışık kırınımı sınırlı çözünürlük elde etmek için iğne deliği boyutunu 0,8 AU, 2x satır ortalaması ve çerçeve boyutunu 1024 × 1024 piksel olarak ayarladık. Uyarma lazeri ve kanal edinme sekansı, kullanılan floroforlar arasındaki paraziti veya karışmayı önlemek için kasıtlı olarak seçildi. Bununla birlikte, parametrelerin ayarlanması, spesifik mikroskop ve numune özelliklerine göre uyarlanmalıdır. Bu protokolde kullanılan konfokal mikroskobun teknik özellikleri için Malzeme Tablosuna bakın. - Biyolojik replikat başına yaklaşık 50 hücreyi kapsayacak şekilde tutarlı sayıda rastgele alan elde edin.
- Konfokal mikroskop kullanarak, z'de 0,25 μm'lik bir adım boyutu ve 0,1-0,2 μm'lik piksel boyutu ayarlayarak 3D görüntüler yakalayın.
3. Görüntü analizi
- Yazılım kurulumu
- Fiji'nin mevcut son sürümünü resmi Fiji İndirme sayfasından (https://imagej.net/software/fiji/downloads) indirin ve yükleyin.
- 3D Suite'i Fiji güncelleme sitesi aracılığıyla veya 3D Suite web sitesindeki (https://mcib3d.frama.io/3d-suite-imagej/#download) talimatları izleyerek manuel olarak yükleyin.
- GDSC'yi (FindFoci) Fiji güncelleme sitesi aracılığıyla veya GitHub deposundaki (https://github.com/aherbert/gdsc) yönergeleri izleyerek manuel olarak yükleyin.
- TIFF dönüştürme
- "convert_to_TIFF.py" komut dosyasını indirin (Ek Dosya 1).
- Komut dosyasını Fiji'ye sürükleyip bırakın ve kodu çalıştırın.
- Görüntülenen panelde, denemenin depolandığı yola göz atın. Dönüştürülen TIFF dosyalarını içeren alt klasör aynı deneme klasöründe oluşturulur.
- 3D Manager'da ayarları başlatın.
- Eklentiler | 3DSuite | 3D Yönetici Seçenekleri'ne tıklayarak 3D Yöneticisi seçenek panelini açın.
- 3D Yöneticisi seçenek penceresinde, aşağıdaki ölçümlere karşılık gelen onay kutularını seçin: Hacim (birim), Ortalama Gri Değer, Sınırlayıcı kutu (pix), Std Dev Gri Değeri, Centroid (pix) ve Centroid (birim).
- Aşağıdaki seçenekleri işaretleyin: XY kenarlarındaki nesneleri hariç tut ve Z kenarlarındaki nesneleri hariç tut ve Tamam'ı tıklatın.
NOT: Başlangıçta uzamsal ve nicel bilgi dosyalarında depolanacak metrikleri yapılandırmak için bu adımın yalnızca bir kez yapılması gerekir. Seçilen metrikler, boru hattının düzgün çalışmasını sağlamak için gereklidir. Tartışma bölümünde daha ayrıntılı olarak açıklandığı gibi isteğe bağlı ek ölçümler bu adıma dahil edilebilir.
- FindFoci GUI'de parametreleri ayarlayın.
NOT: Yarı otomatik işlem hattını en uygun parametrelerle yapılandırmak için bu adımın yalnızca bir kez yapılması gerekir ve bu parametreler daha sonra komut dosyalarına dahil edilecektir.- Test görüntüsünü açın. Yığın da dahil olmak üzere protein kanalını çoğaltın (Ctrl + Shift + D), hiper yığın onay kutusunu işaretleyin ve Kanallar (c) kutusunda uygun kanal numarasını belirtin ve uygun şekilde yeniden adlandırın.
- Eklentiler | Makrolar | Kayıt.
- FindFoci eklentisini açın (Eklentiler | Genel Müdürlük Mali Müşavirlik | Odak Bul | FindFoci GUI) ve "Görüntü" açılır menüsünden analiz edilecek görüntüyü seçin.
- Parametreleri aşağıdaki gibi ayarlayın (Şekil 2A): Gauss bulanıklığı = 1.5; arka plan yöntemi = SD yukarıdaki ortalama; arka plan parametresi = 9; arama yöntemi = zirvenin kesri - arka plan; arama parametresi = 0.7; tepe yöntemi = arka planın üzerindeki göreli; tepe parametresi = 0.2; minimum boyut = 5; Maksimum tepe = 1.000.000.
NOT: Belirtilen parametreler vaka çalışmalarımıza göre seçilmiştir ve diğer boyama prosedürleri için uygun olmayabilir. - Odak tanımlamasını geliştirmek için aşağıdaki parametreleri ayarlayın:
- Gauss bulanıklığı , odakları daha iyi segmentlere ayırmak için yumuşatmanın kapsamını tanımlar. Odak çapına (piksel) yakın tutun.
- Arka plan parametresi, arka planı odak sinyalinden ayırt etmek için bir eşik ayarlar. Daha katı eşikler uygulamak için değerleri artırın.
- Arama parametresi , sinyal tanımaya dahil edilen tepe noktasından gelen floresan yüzdesini tanımlar. Değerleri, floresan tepe noktasından daha uzak alanları içerecek şekilde azaltın.
- Tepe parametresi, iki sinyal tepe noktasının sürekli veya ayrılmış olarak kabul edilme derecesini belirler. Değerin düşürülmesi, tepe noktalarının ayrılmasına neden olur.
- Maksimum tepe noktaları, tanımlanabilir maksimum odak sayısını belirtir. Görüntüdeki tüm odakları dahil etmek için yüksek sayılar ayarlayın.
- FindFoci'yi çalıştırın ve tırnak işaretleri hariç olmak üzere seçilen parametreleri içeren kaydedici penceresinde (Adım 3.4.2, Şekil 2B) görünen dizeyi kopyalayın. Ayarlarla ilgili daha fazla bilgi için eklenti kılavuzu talimatına 22 bakın.
- Nükleer protein miktar tayin boru hattı
- "nuclear_prot_q.py" betiğini indirin (Ek Dosya 2).
- Komut dosyasını Fiji'ye sürükleyip bırakın ve kodu yürütmek için çalıştır'a tıklayın.
- Görüntüleri işlemek için görüntülenen iletişim kutusundaki talimatları izleyin.
- Çekirdek kanalı: DAPI kanalına (veya herhangi bir nükleer boyamaya) karşılık gelen numarayı girin.
- Çekirdek Gauss Bulanıklığı: segmentasyon için görüntüyü bulanıklaştırmak için gereken sigma değerini girin.
NOT: Bu parametreyi çekirdek çapına (yani 5-6 μm) daha yakın tutun. Homojen olmayan boyama için daha yüksek sigma değerleri endikedir. - Protein kanalı: ilgilenilen boyama kanalına karşılık gelen sayıyı girin.
- FindFoci Parametresi: makro kayıt adımından elde edilen dizeyi 3.4.6 pasajına yapıştırın (Şekil 2B).
- (İsteğe bağlı) Kalite kontrol: segmentli çekirdeklerin kalitesini kontrol edin. Bu, komut dosyasını duraklatacak ve oluşturulan her nükleer ilgi alanının (ROI) manuel olarak kontrol edilmesine izin verecektir.
- Bir görüntü dizini seçin: Analiz edilecek TIFF dosyalarını içeren klasöre gitmek için Gözat düğmesini tıklayın.
- Tüm kutular derlendikten sonra, yürütmeye devam etmek için Tamam'a tıklayın.
- Bir çekirdek doğru şekilde bölümlere ayrılmamışsa ve kalite gereksinimlerini karşılayamıyorsa, Şekil 2C'de gösterildiği gibi silin veya değiştirin.
- ROIManager3D penceresindeki ROI listesini kontrol edin; liste boşsa, birleştirme penceresini seçin ve yöneticiyi yenilemek için 3D'yi Ölç'ü tıklatın. Ardından, Quantify 3D sonuç tablosunu kapatın. Çekirdek kanalını seçin ve Canlı-ROI'yi AÇIK konuma tıklayın.
- Aynı çekirdeğe ait ROI'yi seçin ve Birleştir'e basın veya istenmeyen çekirdekler olması durumunda Sil'e basın.
- Tümünü seç'e tıklayın ve Maske onay penceresinde Tamam'a tıklayarak analize devam edin.
- Sonuçlar için, 3.5.3.6 adımında belirtilen dosya yolu içinde, analiz için kullanılan parametrenin kayıtlarını içeren bir txt dosyası içeren Niceleme klasörüne bakın.
- Google Colab'da İşlem Hattı
- "final_nuclear_protein_metrics.ipynb" not defterini indirin (Ek Dosya 3).
- Not defterini Google Colab'da (https://colab.research.google.com/) açın.
- Her bir resim alanının .csv dosyalarını içeren tüm klasörleri Google Drive'da tercih ettiğiniz bir klasöre yükleyin.
- Not defterinde, sonuç alt klasörlerinin depolandığı klasörün yolunu belirtin ve tüm hücreleri çalıştırın. Kodun derlenmesi tamamlandığında, derlenen tüm verileri içeren son elektronik tablo dosyası, .csv dosyalarının yüklendiği klasörde oluşturulur.
Sonuçlar
Bu yöntemde özetlenen protokol, insan birincil T hücrelerindeki nükleer protein boyamasındaki değişikliklerin görselleştirilmesini ve niceliklerinin belirlenmesini kolaylaştırır ve çeşitli hücre tipleri ve protein hedefleri için özelleştirilebilir. Vaka çalışmaları olarak, naif ve TH1 CD4 + hücrelerinde BRD4 ve SUZ12'nin boyanmasını gerçekleştirdik ve analiz ettik.
BRD4, hem hareketsiz naif hem de farklılaşmış TH1 CD4 + hücrelerinde iyi noktalı bir boyama modeli gösterir ve her iki hücre tipi için odak tanımlaması için tasarlanmış parametrelerin kullanılmasını sağlar (Şekil 3A). Boru hattımız, çekirdek içindeki BRD4 odaklarının sayısı, hacmi, floresan sinyali (ortalama floresan yoğunluğu (MFI) olarak ifade edilir), kapsamı ve uzamsal düzenlemesi dahil olmak üzere çeşitli parametrelerin nicelleştirilmesine izin verir (Şekil 3B-F). Ek olarak, odakların nükleer hacim yüzdesini belirlemek için otomatik bir hesaplama başlattık ve farklı nükleer boyutlara sahip hücreler arasında karşılaştırmayı kolaylaştırdık (Şekil 3E).
Son olarak, odakların nükleer merkeze olan mesafelerini haritalama seçeneği sunarak nükleer konumlarının anlaşılmasına yardımcı olduk (Şekil 3F). Kantifikasyondan elde edilen sonuçlar, TH1 ve hareketsiz CD4 + T hücreleri arasındaki BRD4 odaklarında, odak sayısı, boyutu, parlaklığı ve hacmindeki bir artışın yanı sıra dağılım ve lokalizasyonlarındaki farklılıkları kapsayan dikkate değer değişiklikleri vurgulamaktadır. Bu bulgular, aktive edilmiş / çoğalan T hücrelerinin hareketsiz muadilleri28 ile karşılaştırıldığında artan transkripsiyonel aktivitesi ile tutarlıdır.
Buna karşılık, SUZ12 yoğuşma oluşumuna katılmaz. Araştırmamızda, iki hücresel durum arasında SUZ12'nin boyama modelinde önemli bir eşitsizlik gözlemledik: naif T hücrelerinde noktasal bir model, TH1 CD4 + hücrelerinde yaygın bir modele dönüşür (Şekil 4A). Protein davranışındaki bu önemli değişim, T, H,CD4 + hücrelerinde nokta tanımlaması büyük ölçüde başarısız olduğundan, aynı parametreleri kullanan odak özelliklerinin karşılaştırılabilirliğini engeller (Şekil 4A). Farklı hücresel koşullar arasında immünofloresan boyamayı karşılaştırırken genellikle hem edinim hem de analiz parametrelerinde tutarlılığın korunması tavsiye edilirken, ilgilenilen hedefte önemli değişiklikler olması durumunda ayarlamalar gerekli hale gelir.
Bu özel senaryoda, Şekil 4B'de gösterildiği gibi, arka plan parametrelerine ayarlama, Gauss bulanıklığı ve arama parametreleri (protokol adımı 3.4.5'te önerildiği gibi) dahil olmak üzere çeşitli parametre kombinasyonları ile deneyler yapılmasına rağmen, FindFoci, proteinin doğal dağılım doğası nedeniyle gerçek sinyal zirvelerini önemsiz olanlardan ayırt etmede zorluklarla karşılaşır. Ayrıca, nükleer MFI gibi geleneksel ölçümlerin yanıltıcı olabileceğini vurguluyoruz (Şekil 4C), sinyal dağılımındaki değişiklikleri dikkate almadan.
Bu nedenle, bu tür değişiklikleri ölçmek için, homojenlik katsayısı (IC) olarak adlandırılan protein difüzyonunu karakterize etmek için bu protokolde tanıttığımız bir metrik olan varyasyon katsayısının kullanılmasını öneriyoruz. IC, SUZ12 odaklarının TH1 CD4 + hücrelerindeki difüzyonunu, yanlış segmentasyon yaklaşımlarıyla tanımlamalarını zorlamadan doğru bir şekilde yakalar (bkz. Şekil 4D). Son olarak, DAPI'yi her iki hücre koşulunda eşit olarak dağılmış halde kalan bir kontrol olarak kullanarak, bu parametrenin etkinliğini daha da doğrularız (Şekil 4E).
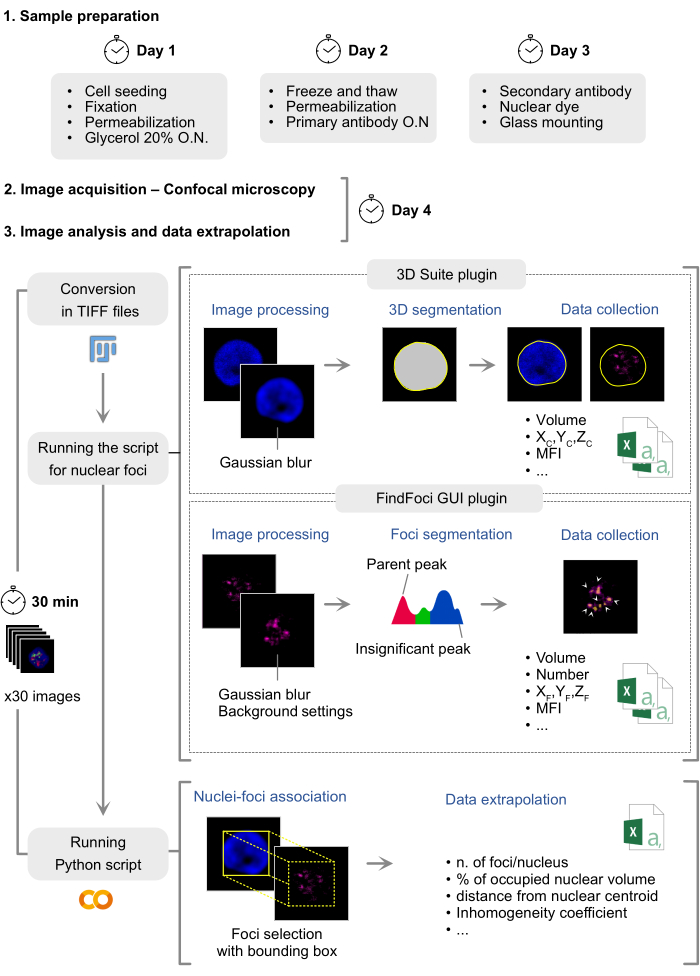
Şekil 1: İmmünofloresan hazırlama ve veri analizinin şematik gösterimi. Nükleer protein immünofloresansının boyanması ve analiz edilmesi için protokol üç ana aşamadan oluşur: numune hazırlama, görüntü elde etme ve analiz, yaklaşık 4 iş gününü kapsar. Komut dosyası, çekirdek segmentasyonunu gerçekleştirmek için 3D Suite ve protein kanalını işlemek için FindFoci olmak üzere iki eklenti kullanır. Odaklar ve çekirdek ile ilgili mekansal ve nicel bilgiler ayrı csv dosyalarında toplanır. Google Colab'da yürütülebilir bir komut dosyası, çekirdek sınırlama kutusunu kullanarak odak ölçümlerini karşılık gelen çekirdeklerle bağlar ve toplanan verilere dayalı olarak türetilmiş ölçümleri hesaplar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
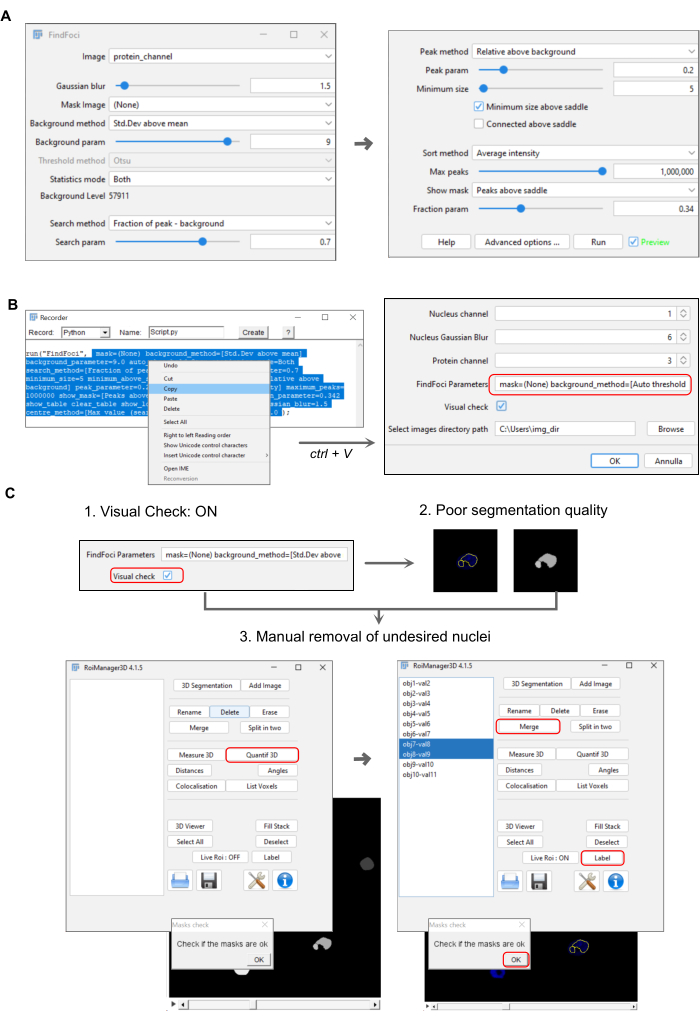
Şekil 2: Nükleer protein miktar tayini boru hattı ayarlarının gösterimi. (A) Odakların tanımlanması için parametreleri gösteren FindFoci açılır penceresi (protokol bölümü 3.4.3). (B) FindFoci'nin seçilen parametrelerini kaydeden makroyu temsil eden açılır pencere. Kopyalanan dize başlangıç penceresine yapıştırılır (protokol adımları 3.4.6-3.5.3.4). (C) 3D yöneticiyi kullanarak ilgilenilen nükleer bölgelerin manuel olarak çıkarılması veya değiştirilmesi için çekirdek segmentasyonu kalite kontrol adımını gösteren panel (protokol adımları 3.5.5.1 -3.5.5.3). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
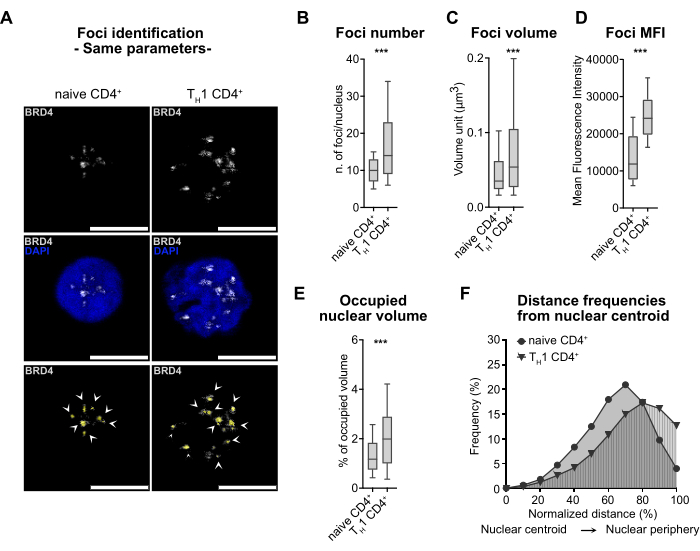
Şekil 3: BRD4 odak analizlerine dayalı farklı hücresel koşullar arasındaki karşılaştırmalar. (A) İnsan primer naif CD4 + T hücrelerinde ve TH1 CD4 + hücrelerinde BRD4'ün (gri) immünofloresan boyanmasının temsili konfokal floresan mikroskobu görüntüleri. Çekirdekler DAPI (mavi) ile karşı boyanır. Orijinal büyütme 63x; ölçek çubuğu = 5 μm. Boru hattı tarafından tanımlanan alt, BRD4 odakları beyaz ok uçları ile işaretlenir ve sarı (alt) ile karşılanır. (B) Naif ve TH1 CD4+ hücrelerinde (n = 2 kişi) BRD4 odak sayısını/çekirdeğini temsil eden kutu grafiği. (C) Saf ve TH1CD4+ hücrelerinde (n = 2 birey) BRD4 odaklarının hacminin (μm 3) kutu grafiği temsili. (D) BRD4 odaklarının MFI odaklarının saf ve TH1 CD4+ hücrelerinde (n = 2 kişi) kutu grafiği temsili. (E) Toplam çekirdek hacmine (n = 2 kişi) göre BRD4 odakları tarafından işgal edilen nükleer hacmin yüzdesini temsil eden kutu grafiği. (F) BRD4 odaklarının nükleer merkezden nükleer çevreye olan mesafe frekanslarının temsili (n = 2 kişi). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
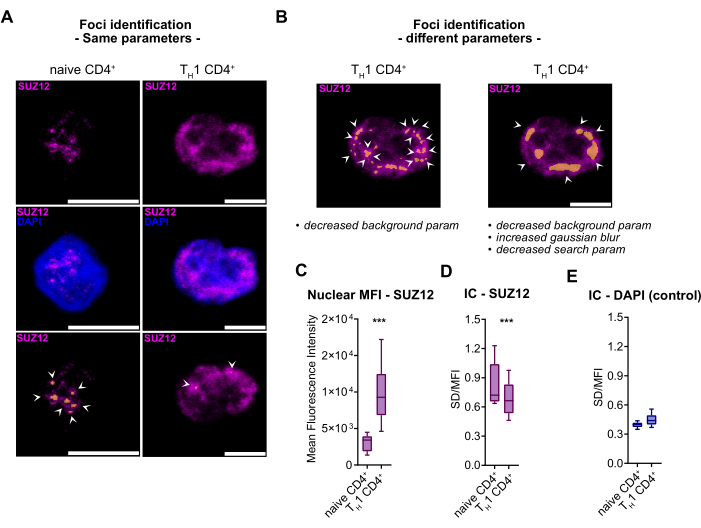
Şekil 4: Homojenlik katsayısı, SUZ12 immünofloresan sinyallerinde bir noktadan dağınık bir modele geçişi ölçmek için bir metrik . (A) İnsan primer naif CD4 + T hücrelerinde ve TH1 CD4 + 'da SUZ12 (macenta) için immünofloresan boyamanın temsili konfokal floresan mikroskobu görüntüleri Hücre. Çekirdekler DAPI (mavi) ile karşı boyanır. Orijinal büyütme 63x; ölçek çubuğu = 5 μm. Altta, boru hattı tarafından tanımlanan SUZ12 odakları beyaz bir ok ucu ile işaretlenmiş ve turuncu (B) ile karşılanmıştır. İki farklı parametre ayarı ile beyaz oklar ve turuncu maske ile gösterilen TH1 CD4 + hücrelerinde yanlış odak tanımlama örnekleri. (C) Naif ve TH1 CD4+ hücrelerinde (n = 2 birey) SUZ12'nin nükleer MFI'sinin kutu grafiği temsili. (D) SUZ12 IC'nin naif ve TH1 CD4+ hücrelerinde (n = 2 kişi) kutu grafiği gösterimi. (E) DAPI IC'nin saf ve TH1 CD4 + hücrelerinde (n = 2 kişi) kutu grafiği gösterimi. Kısaltmalar: DAPI = 4',6-diamidino-2-fenilindol; MFI = ortalama floresan yoğunluğu; IC = homojenlik katsayısı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Çözüm | Kompozisyon | Yorumlar/Açıklama | |
| TH1 orta | GlutaMAX-I ile RPMI, %10 (h/h) Fetal Sığır Serumu (FBS), %1 (h/h) esansiyel olmayan amino asitler, 1 mM sodyum piruvat, 50 IU/mL penisilin, 50 μg/mL streptomisin, 20 IU/mL rekombinant IL-2, 10 ng/mL rekombinant IL-12, 2 mg/mL nötralize edici anti-IL-4. | Adım 1.1.4. | |
| Kaplama çözeltisi | ddH2O içinde% 0.1 poli-L-lizin | Adım 1.2.1. | |
| PBS-T Serisi | 1x PBS/%0.1 TWEEN 20 pH 7.0 | Adım 1.3. | |
| Fiksasyon için PFA çözümü | % 0.1 PBS-TWEEN içinde seyreltilmiş% 3 paraformaldehit (PFA) | Adım 1.2.4. | |
| TPBS (TPBS) | % 0.05 Triton X-100, 1x PBS'de seyreltilmiş | Adım 1.2., 1.3. | |
| Geçirgenlik çözümü | 1x PBS'de seyreltilmiş% 0.5 TPBS | Adım 1.2., 1.3. | |
| Depolama çözümü | % 20 gliserol/1x PBS | Adım 1.2.7., 1.3.1. | |
| Antikor seyreltme tamponu | % 0.1 PBS-TWEEN /% 2 Keçi Serumu /% 1 BSA | Adım 1.3.6., 1.3.8. | |
Tablo 1: Bu protokolde kullanılan ortam ve tamponların bileşimi.
Ek Dosya 1: "convert_to_TIFF.py". Bu dosya, herhangi bir görüntü dosyasını (örn. ND2, LIF, vb.) doğru niceleme işlemleri için gerekli olan TIFF dosyalarına dönüştürmek için komut dosyasını içerir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 2: "nuclear_prot_q.py". Bu dosya, en az iki kanal (çekirdek boyama, nükleer protein boyama) içeren görüntüleri ölçmeye ve nicelleştirmeye izin veren komut dosyasını içerir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 3: "final_nuclear_protein metrics.ipynb". Bu dosya, giriş olarak "nuclear_prot_q.py" çıktısını kullanarak tüm ilgili parametrelerin bir özetini ayıklayan ve tek bir Excel dosyasında derleyen bir Jupyter not defteri içerir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Bu çalışmada, insan T lenfositlerindeki nükleer proteinler üzerinde immünofloresan deneyleri yapmak için bir yöntem sunuyoruz. Bu yöntem, daha önce tarif edildiği gibi, fiksasyon ve geçirgenlik aşamalarında küçük değişiklikler yoluyla çeşitli hücre tipleriyle kullanım için esneklik sunar30,31.
Görüntüleme iş akışımız, özellikle FindFoci ve 3D Suite22,23 olmak üzere literatürde özetlenen yerleşik teknikler üzerine kuruludur. Önceki yayınlardan farklı olarak, yarı otomatik bir boru hattı için Fiji'ye entegre edilmiş, titiz parametre takibi ve görsel kalite kontrollerine olanak tanıyan yürütülebilir bir Jython kodu sağlıyoruz. Özellikle, protokol bölümü 3.5.3.5'te çekirdek segmentasyonunun kalitesini düzenlemek için isteğe bağlı yeteneği tanıttık ve protokol bölümü 3.5.5'te düzeltici bir adım uyguladık. Google Colab'da yürütülebilir komut dosyası ile kullanıcılar, kapsamlı ölçümler alabilir ve kendi özel biyolojik senaryoları için en uygun olanları seçebilir.
Sağlanan analiz hattı Jython'da (Java + Python) yazılmıştır ve yürütme için Fiji'nin yüklenmesini ve iki farklı eklentinin kurulmasını gerektirir: 3D Suite ve FindFoci (Şekil 1). Boru hattı, görüntünün TIFF formatına dönüştürülmesi ve çekirdek segmentasyonunu geliştirmek için çekirdekleri içeren kanalın bir Gauss bulanıklığı filtresi ile ön işlenmesiyle başlar. 3D Suite'i hem i) nükleer hacim, merkez ve sınırlayıcı kutu koordinatları (nesneyi çevreleyen ve odakları sağ çekirdeğe ilişkilendirmek için kullanılan en küçük kutuyu temsil eder) gibi uzamsal bilgileri hem de ii) MFI ve sinyalin standart sapması dahil olmak üzere nicel bilgileri yakalamaya yönlendiriyoruz. Daha sonra, boru hattı, odak sinyalindeki yerel maksimumları tanımlamak için FindFoci'yi kullanır, küresel eşikleme yöntemlerine (genellikle kullanıcının takdirine dayanan ve kesinlikten yoksun olabilen) bağımlılığı ortadan kaldırır ve odaklara göre hem uzamsal hem de nicel bilgileri kaydeder. Bu nedenle çıkış klasörü, uzamsal ("M_.csv") ve nicel ("Q_.csv") ölçümleri kapsayan kanal başına iki CSV dosyası içerecektir.
Toplanan ve türetilen tüm ölçümleri içeren son dosya, Google Colab'da bir Python komut dosyası yürütülerek oluşturulur. Bu dosya, çekirdek başına odak sayısı, odak MFI, homojenlik katsayısı (IC), nükleer MFI, nükleer merkezden odak mesafesi ve işgal edilen nükleer hacmin yüzdesi gibi ölçümleri içerir. Görsel kalite kontrol adımları da dahil olmak üzere görüntü başına yaklaşık 1 dakikalık bir işleme süresi tahmin ediyoruz. Bu işlem süresi, immünofloresan ayarlarının ve analiz parametrelerinin optimizasyonu yoluyla daha da hızlandırılabilir.
Boru hattı, hem iyi noktalı hem de dağınık desenler sergileyen nükleer proteinler için parametreleri hesaplayarak kullanıcıların en uygun parametreyi seçmesine olanak tanır. Protein odaklarının çözünmesini tanımlamak için homojen olmama katsayısını kullanmanızı öneririz.
Odakların doğru şekilde tanımlanmasını sağlamak için kritik bir adım, gerçek sinyal tepe noktalarını arka plan gürültüsünden ayırt etmek için parametrelerin ayarlanmasını içerir. İşlem hattını çalıştırmadan önce rastgele görüntüler üzerinde kapsamlı testler yapmak çok önemlidir. Çalışmamız öncelikle tek bir kanala odaklanırken, boru hattı, immünofloresan deneylerinde farklı proteinleri hedefleyen çeşitli boya kombinasyonlarının tanıtımını desteklemektedir. Birden çok kanalın aynı anda analizini desteklemez ve her kanal için ayrı ayrı çalıştırılmalıdır. Ayrıca, bu protokolde belirli ölçümler sunmayı tercih ettik; Bununla birlikte, floresan sinyalindeki mutlak değişiklikleri değerlendirmek için entegre yoğunluk veya dairesellik ve kompaktlık gibi fiziksel parametreler gibi ek ölçümler de Bölüm 3.3.2'deki boru hattına dahil edilebilir. Şu anda bu işlem hattına entegre edilmemiş metriklerin dahil edilmesi, bunları son metrik dosyasına entegre etmek için Python programlamada yeterlilik gerektirir; aksi takdirde, mekansal ("M_.csv") ve nicel ("Q_.csv") bilgi dosyalarına otomatik olarak dahil edilirler.
Bu işlem hattının zamanlaması, görüntü boyutu ve miktarı, analiz başına kanal sayısı ve mikroskop ayarları gibi faktörlere bağlıdır. Ek olarak, makine performansı yürütme süresini etkiler ve görsel adımlar süreci potansiyel olarak yavaşlatır.
Konfokal mikroskopi değerli bilgiler sunarken, gözlemlenen nükleer yapıların veya protein odaklarının hassasiyetini sınırlayabilecek kırınım sınırlı bir teknoloji olmaya devam ettiğini belirtmekte fayda var. Analizin sağlamlığını artırmak için, incelenen çekirdek ve biyolojik örneklerin sayısını artırmanızı öneririz. Analiz edilen çekirdek sayısı arttıkça, nükleer morfoloji veya protein dağılımındaki herhangi bir kayda değer değişiklik, çözünürlük sınırlamalarından bağımsız olarak daha belirgin hale gelir.
Gelecekteki potansiyel uygulamalar açısından, bu yöntem çok yönlülük ve çeşitli hücre tiplerine ve boyama protokollerine uyarlanabilirlik sergiler. Örneğin, iyonlaştırıcı radyasyonun neden olduğu odakların (IRIF'ler) doğru bir şekilde ölçülmesinin DNA hasarının32 doğrudan bir göstergesi olarak hizmet ettiği radyobiyoloji deneylerinde kolayca kullanılabilir. Birincil vurgumuz nükleer analize dayansa da, protokol bir sitoplazmik boya dahil edilerek ve senaryo içindeki nükleer kanal yerine ikame edilerek kolayca özelleştirilebilir. Ayrıca analiz, nükleer topoloji analizi için varsayılan parametrelerden yararlanarak DNA ve RNA FISH30,31 gibi diğer deneysel metodolojilere sorunsuz bir şekilde entegre edilebilir.
Açıklamalar
R.V., T-One Therapeutics Srl girişimi ile bilimsel bir işbirliğine sahiptir; B.B. ve F.M., T-One Therapeutics Srl adlı girişimin kurucu ortaklarıdır; E.P. şu anda T-One Therapeutics Srl'de çalışmaktadır; Diğer tüm yazarlar rekabet eden çıkarları olmadığını beyan ederler.
Teşekkürler
INGM Görüntüleme Tesisi'nin, özellikle C. Cordiglieri ve A. Fasciani'nin ve INGM FACS ayıklama tesisinin, özellikle M.C Crosti'nin (Istituto Nazionale di Genetica Molecolare 'Romeo ed Enrica Invernizzi' (INGM), Milano, İtalya) bilimsel ve teknik yardımına teşekkür ederiz. M. Giannaccari'ye teknik bilişim desteği için teşekkür ederiz. Bu çalışma aşağıdaki hibelerle finanse edilmiştir: Fondazione Cariplo (Bando Giovani, hibe nr 2018-0321) ve Fondazione AIRC (hibe nr MFAG 29165) F.M. Ricerca Finalizzata, (hibe nr GR-2018-12365280), Fondazione AIRC (hibe nr 2022 27066), Fondazione Cariplo (hibe nr 2019-3416), Fondazione Regionale per la Ricerca Biomedica (FRRB CP2_12/2018,) Piano Nazionale di Ripresa e Resilienza (PNRR) (hibe nr G43C22002620007) ve Progetti di Rilevante Interesse Nazionale (PRIN) (hibe nr 2022PKF9S) B'ye. B.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1.5 mL Safe-Lock Tubes | Eppendord | #0030121503 | Protocol section 1 |
| 10 mL Serological pipettes | VWR | #612-3700 | Protocol section 1 |
| 20 µL barrier pipette tip | Thermo Scientific | #2149P-HR | Protocol section 1 |
| 50 mL Polypropylene Conical Tube | Falcon | #352070 | Protocol section 1 |
| 200 µL barrier pipette tip | Thermo Scientific | #2069-HR | Protocol section 1 |
| antifade solution - ProlongGlass - mountingmedia | Invitrogen | #P36984 | Step 1.3.12 |
| BSA (Bovine Serum Albumin) | Sigma | #A7030 | Step 1.3.6., 1.3.8. |
| CD4+ T Cell Isolation Kit | Miltenyi Biotec | #130-096-533 | Step 1.1.2. |
| DAPI (4,6-diamidino-2-phenylindole) | Invitrogen | Cat#D1306 | Step 1.3.10. |
| Dry ice | Step 1.3.1. | ||
| Dynabeads Human T-activator anti-CD3/anti-CD28 bead | Life Technologies | #1131D | magnetic beads step 1.1.4. |
| EtOH | Carlo Erba | #4146320 | Step 1.2.1.1. |
| FACSAria SORP | BD Bioscences | Step 1.1.3. Equipped with BD FACSDiva Software version 8.0.3 | |
| FBS (Fetal Bovine Serum) | Life Technologies | #10270106 | Step 1.1.4 |
| FICOLL PAQUE PLUS | Euroclone | GEH17144003F32 | Step 1.1.1. |
| FIJI Version 2.14.0 | - | - | Protocol section 3 |
| Glass coverslip (10 mm, thickness 1.5 H) | Electron Microscopy Sciences | #72298-13 | Step 1.2.1. |
| Glycerol | Sigma | #G5516 | Step 1.2.7-1.3.1. |
| Goat anti-Rabbit AF568 secondary antibody | Invitrogen | A11036 | Step 1.3.8. |
| HCl | Sigma | #320331 | Step 1.3.4. |
| human neutralizing anti-IL-4 | Miltenyi Biotec | Cat#130-095-753 | Step 1.1.4. |
| human recombinant IL-12 | Miltenyi Biotec | Cat#130-096-704 | Step 1.1.4. |
| human recombinant IL-2 | Miltenyi Biotec | Cat#130-097-744 | Step 1.1.4. |
| Leica TCS SP5 Confocal microscope | Leica Microsystems | - | Protocol section 2, Equipped with HCX PL APO 63x, 1.40 NA oil immersion objective, with an additional 3x zoom. Pinhole size : 0.8 AU. Line average 2×. Frame size 1024×1024 pixel. |
| MEM Non-Essential Amino Acids Solution | Life Technologies | #11140035 | Step 1.1.4. |
| Microscope Slides | VWR | #631-1552 | Step 1.3.12. |
| Mouse monoclonal anti-Human CD4 APC-Cy7 (RPA-T4 clone) | BD Bioscience | #557871 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RA PECy5 (5H9 clone) | BD Bioscience | #552888 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RO APC (UCHL1 clone) | Miltenyi Biotec | #130-113-546 | Step 1.1.3. |
| Multiwell 24 well | Falcon | #353047 | Protocol section 1 |
| Normal Goat Serum | Invitrogen | PCN5000 | Step 1.3.6., 1.3.8. |
| PBS | Life Technologies | #14190094 | Protocol section 1 |
| Penicillin/Streptomycin solution | Life Technologies | #15070063 | Step 1.1.4. |
| PFA | Sigma | #P6148 | Step 1.2.4. |
| poly-L-lysine | Sigma | #P8920 | 1.2.1. |
| Primary antibody - BRD4 | Abcam | #ab128874 | Step 1.3.6. |
| Primary antibody - SUZ12 | Cel Signalling | mAb #3737 | Step 1.3.6. |
| RPMI 1640 W/GLUTAMAX-I | Life Technologies | #61870010 | Step 1.1.4. |
| Sodium Pyruvate | Life Technologies | #11360039 | Step 1.1.4. |
| Triton X-100 | Sigma | #T8787 | Step 1.2., 1.3. |
| TWEEN 20 | Sigma | #P9416 | Step 1.3. |
| Tweezers | - | - | Protocol section 1 |
Referanslar
- Aboelnour, E., Bonev, B. Decoding the organization, dynamics, and function of the 4D genome. Dev Cell. 56 (11), 1562-1573 (2021).
- Erdel, F., Rippe, K. Formation of chromatin subcompartments by phase separation. Biophys J. 114 (10), 2262-2270 (2018).
- Henninger, J. E., et al. RNA-mediated feedback control of transcriptional condensates. Cell. 184 (1), 207-225 (2021).
- Guo, Y. E., et al. Pol II phosphorylation regulates a switch between transcriptional and splicing condensates. Nature. 572 (7770), 543-548 (2019).
- Bhat, P., et al. 3D genome organization around nuclear speckles drives mRNA splicing efficiency. bioRxiv. , (2023).
- Spector, D. L., Lamond, A. I. Nuclear speckles. Cold Spring Harb Perspect Biol. 3 (2), a000646 (2011).
- Kilic, S., et al. Phase separation of 53BP1 determines liquid-like behavior of DNA repair compartments. EMBO J. 38 (16), e101379 (2019).
- Parker, M. W., et al. A new class of disordered elements controls DNA replication through initiator self-assembly. Elife. 8, e48562 (2019).
- Feric, M., et al. Coexisting liquid phases underlie nucleolar subcompartments. Cell. 165 (7), 1686-1697 (2016).
- Yoneda, M., Nakagawa, T., Hattori, N., Ito, T. The nucleolus from a liquid droplet perspective. J Biochem. 170 (2), 153-162 (2021).
- Alberti, S., Carra, S. Nucleolus: A liquid droplet compartment for misbehaving proteins. Curr Biol. 29 (19), R930-R932 (2019).
- Strom, A. R., et al. Phase separation drives heterochromatin domain formation. Nature. 547 (7662), 241-245 (2017).
- Sanulli, S., et al. HP1 reshapes nucleosome core to promote phase separation of heterochromatin. Nature. 575 (7782), 390-394 (2019).
- Hyman, A. A., Weber, C. A., Julicher, F. Liquid-liquid phase separation in biology. Annu Rev Cell Dev Biol. 30, 39-58 (2014).
- Feric, M., Misteli, T. Phase separation in genome organization across evolution. Trends Cell Biol. 31 (8), 671-685 (2021).
- Mitrea, D. M., et al. Methods for physical characterization of phase-separated bodies and membrane-less organelles. J Mol Biol. 430 (23), 4773-4805 (2018).
- Fetter, J., et al. Endogenous gene tagging with fluorescent proteins. Methods Mol Biol. 1239, 231-240 (2015).
- Alberti, S., Gladfelter, A., Mittag, T. Considerations and challenges in studying liquid-liquid phase separation and biomolecular condensates. Cell. 176 (3), 419-434 (2019).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. J Cell Sci. 124 (10), 1607-1611 (2011).
- Scalisi, S., Ahmad, A., D'Annunzio, S., Rousseau, D., Zippo, A. Quantitative analysis of PcG-associated condensates by stochastic optical reconstruction microscopy (STORM). Methods Mol Biol. 2655, 183-200 (2023).
- Shihan, M. H., Novo, S. G., Le Marchand, S. J., Wang, Y., Duncan, M. K. A simple method for quantitating confocal fluorescent images. Biochem Biophys Rep. 25, 100916 (2021).
- Herbert, A. D., Carr, A. M., Hoffmann, E. FindFoci: a focus detection algorithm with automated parameter training that closely matches human assignments, reduces human inconsistencies and increases speed of analysis. PLoS One. 9 (12), e114749 (2014).
- Ollion, J., Cochennec, J., Loll, F., Escude, C., Boudier, T. TANGO: a generic tool for high-throughput 3D image analysis for studying nuclear organization. Bioinformatics. 29 (14), 1840-1841 (2013).
- Sabari, B. R., et al. Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361 (6400), 3958 (2018).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19 (4), 523-534 (2005).
- Pasini, D., Bracken, A. P., Jensen, M. R., Denchi, E. L., Helin, K. Suz12 is essential for mouse development and for EZH2 histone methyltransferase activity. EMBO J. 23 (20), 4061-4071 (2004).
- Margueron, R., Reinberg, D. The Polycomb complex PRC2 and its mark in life. Nature. 469 (7330), 343-349 (2011).
- Peng, Z., et al. Brd4 regulates the homeostasis of CD8(+) T-lymphocytes and their proliferation in response to antigen stimulation. Front Immunol. 12, 728082 (2021).
- Marasca, F., et al. LINE1 are spliced in non-canonical transcript variants to regulate T cell quiescence and exhaustion. Nat Genet. 54 (2), 180-193 (2022).
- Marasca, F., Cortesi, A., Bodega, B. 3D COMBO chrRNA-DNA-ImmunoFISH. Methods Mol Biol. 2157, 281-297 (2021).
- Marasca, F., Cortesi, A., Manganaro, L., Bodega, B. 3D Multicolor DNA FISH tool to study nuclear architecture in human primary cells. J Vis Exp. (155), (2020).
- Jakob, B., Splinter, J., Durante, M., Taucher-Scholz, G. Live cell microscopy analysis of radiation-induced DNA double-strand break motion. Proc Natl Acad Sci U S A. 106 (9), 3172-3177 (2009).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır