Method Article
Una línea versátil para analizar los cambios dinámicos en los cuerpos nucleares en una variedad de tipos de células
En este artículo
Resumen
Este método describe un protocolo de inmunofluorescencia y un canal de cuantificación para evaluar la distribución de proteínas con patrones de organización nuclear variados en linfocitos T humanos. Este protocolo proporciona orientación paso a paso, comenzando con la preparación de la muestra y continuando con la ejecución de análisis semiautomatizados en Fiji, concluyendo con el manejo de datos por parte de un cuaderno de Google Colab.
Resumen
Varios procesos nucleares, como el control transcripcional, ocurren dentro de estructuras discretas conocidas como focos que se pueden discernir a través de la técnica de inmunofluorescencia. La investigación de la dinámica de estos focos en diversas condiciones celulares a través de la microscopía proporciona información valiosa sobre los mecanismos moleculares que gobiernan la identidad y las funciones celulares. Sin embargo, la realización de ensayos de inmunofluorescencia en diferentes tipos de células y la evaluación de las alteraciones en el ensamblaje, la difusión y la distribución de estos focos presentan numerosos desafíos. Estos desafíos abarcan complejidades en la preparación de muestras, la determinación de parámetros para analizar datos de imágenes y la gestión de volúmenes de datos sustanciales. Además, los flujos de trabajo de imágenes existentes a menudo se adaptan a usuarios competentes, lo que limita la accesibilidad a un público más amplio.
En este estudio, presentamos un protocolo de inmunofluorescencia optimizado diseñado para investigar proteínas nucleares en diferentes tipos de células T primarias humanas que se pueden personalizar para cualquier proteína de interés y tipo de célula. Además, presentamos un método para cuantificar de forma no sesgada la tinción de proteínas, ya sea que formen focos distintos o exhiban una distribución nuclear difusa.
Nuestro método propuesto ofrece una guía completa, desde la tinción celular hasta el análisis, aprovechando una tubería semiautomatizada desarrollada en Jython y ejecutable en Fiji. Además, proporcionamos un script de Python fácil de usar para agilizar la gestión de datos, de acceso público en un cuaderno de Google Colab. Nuestro enfoque ha demostrado eficacia en la producción de análisis de inmunofluorescencia altamente informativos para proteínas con diversos patrones de organización nuclear en diferentes contextos.
Introducción
La organización del genoma eucariota está gobernada por múltiples capas de modificaciones epigenéticas1, que coordinan varias funciones nucleares que pueden ocurrir dentro de compartimentos especializados llamados cuerpos nucleares o condensados2. Dentro de estas estructuras, tienen lugar procesos como la iniciación de la transcripción3, el procesamiento del ARN 4,5,6, la reparación del ADN 7,8, la biogénesis del ribosoma 9,10,11 y la regulación de la heterocromatina12,13. La regulación de los cuerpos nucleares se ajusta en las dimensiones espaciales y temporales para adaptarse a los requisitos celulares, guiada por los principios de separación de fases14,15. En consecuencia, estos cuerpos funcionan como fábricas transitorias donde los componentes funcionales se ensamblan y desensamblan, sufriendo cambios en el tamaño y la distribución espacial. Por lo tanto, la comprensión de las características de las proteínas nucleares mediante microscopía, incluida su propensión a formar cuerpos y su disposición espacial en diferentes condiciones celulares, ofrece información valiosa sobre sus roles funcionales. La microscopía de fluorescencia es un método ampliamente utilizado para el estudio de proteínas nucleares, permitiendo su detección a través de anticuerpos fluorescentes o expresando directamente dianas con un reportero de proteínas fluorescentes16,17.
En este contexto, los cuerpos nucleares aparecen como focos brillantes o puncta, con un notable grado de esfericidad, lo que los hace fácilmente distinguibles del ambiente circundante 16,18. Las técnicas de superresolución como STORM y PALM, al proporcionar una resolución mejorada (hasta 10 nm)19, permiten una caracterización más precisa de la estructura y composición de condensados específicos20. Sin embargo, su accesibilidad está limitada por los gastos de equipo y las habilidades especializadas necesarias para el análisis de datos. Por lo tanto, la microscopía confocal sigue siendo popular debido a su equilibrio favorable entre resolución y uso más amplio. Esta popularidad se ve facilitada por la eliminación inherente de la luz desenfocada, que disminuye la necesidad de extensos procedimientos de posprocesamiento para una segmentación precisa, su amplia disponibilidad en los institutos de investigación, su tiempo de adquisición efectivo y la preparación de muestras que suele ser eficiente. Sin embargo, la medición precisa de la distribución, el ensamblaje o la difusión de proteínas mediante ensayos de inmunofluorescencia en diversas condiciones celulares plantea desafíos, ya que muchos métodos existentes carecen de orientación sobre la selección de parámetros adecuados para proteínas con diferentes patrones de distribución21. Además, el manejo del gran volumen de datos resultante puede ser desalentador para los usuarios con experiencia limitada en el análisis de datos, lo que puede comprometer la importancia biológica de los resultados.
Para abordar estos desafíos, presentamos un protocolo detallado paso a paso para la preparación de inmunofluorescencia y el análisis de datos, con el objetivo de proporcionar un método imparcial para cuantificar la tinción de proteínas con varios patrones de organización (Figura 1). Esta tubería semiautomatizada está diseñada para usuarios con experiencia limitada en análisis computacional y de imágenes. Combina las funcionalidades de dos plugins establecidos de Fiji: FindFoci22 y 3D suite23. Al integrar la capacidad de identificación precisa de focos de FindFoci con las funciones de identificación y segmentación de objetos en el espacio 3D que ofrece 3D suite, nuestro enfoque genera dos archivos CSV por canal para cada campo de adquisición. Estos archivos contienen información complementaria que facilita el cálculo de métricas adecuadas para varios tipos de distribución de señales, como el recuento de focos por célula, la distancia de los focos al centroide nuclear y el coeficiente de inhomogeneidad (CI), que hemos introducido para la tinción difusa de proteínas. Además, reconocemos que la extrapolación de datos puede llevar mucho tiempo para los usuarios con habilidades limitadas de manejo de datos. Para agilizar este proceso, proporcionamos un script de Python que compila automáticamente todas las mediciones recopiladas en un solo archivo para cada experimento. Los usuarios pueden ejecutar este script sin necesidad de instalar ningún software de lenguaje de programación. Proporcionamos un código ejecutable en Google Colab, una plataforma basada en la nube que permite la escritura de scripts de Python directamente en el navegador. Esto garantiza que nuestro método sea intuitivo y fácilmente accesible para su uso inmediato.
Demostramos la efectividad de nuestro protocolo en el análisis y cuantificación de alteraciones en la distribución de señales de dos proteínas nucleares: la proteína 4 que contiene bromodominio (BRD4) y el supresor de zeste-12 (SUZ12). BRD4 es una proteína coactivadora bien documentada dentro del complejo mediador conocida por formar condensados asociados con la iniciación transcripcional dependiente de la polimerasa II24,25. SUZ12 es un componente proteico del Complejo Represivo Polycomb 2 (PRC2) responsable de regular la deposición de la modificación de histonas H3K27me326,27. Estas proteínas exhiben diferentes patrones dentro de dos tipos de células distintas: las células T humanas CD4+ naïve recién aisladas, que están inactivas y exhiben tasas lentas de actividad transcripcional, y las células TH1 CD4+ diferenciadas in vitro, que son células efectoras proliferantes especializadas que muestran una mayor transcripción28.
Protocolo
El uso de muestras humanas con fines de investigación fue aprobado por los Comités de Ética de la Fondazione Istituto di Ricovero e Cura a Carattere Scientifico (IRCCS) Cà Granda Ospedale Maggiore Policlinico (Milán), y se obtuvo el consentimiento informado de todos los sujetos (número de autorización: 708_2020). El protocolo se organiza en tres secciones principales: ejecución de inmunofluorescencia, adquisición de imágenes y análisis de imágenes. En promedio, requiere 4 días hábiles para completarse (Figura 1).
1. Preparación de inmunofluorescencia
NOTA: Este protocolo de inmunofluorescencia se puede personalizar fácilmente para varios tipos de células y objetivos proteicos ajustando las condiciones de fijación y permeabilización. La preparación de la inmunofluorescencia suele tardar menos de 3 días en completarse, y la duración de la incubación primaria de los anticuerpos varía en función de la calidad del anticuerpo y de la proteína diana (Figura 1).
- Preparación de la muestra
- Aislar células mononucleares de sangre periférica humana (PBMC) mediante una centrifugación en gradiente de densidad a través de un medio de aproximadamente 1,077 g/mL de densidad según las instrucciones del fabricante (Tabla de materiales).
- Aísle las células T CD4+ de las PBMC con perlas magnéticas, siguiendo las instrucciones del fabricante (Tabla de Materiales).
- Teñir las células con anticuerpos para CD4, CD45RA y CD45RO (Tabla de materiales) y proceder a la clasificación FACS de las células T CD4+ vírgenes como células CD4+/CD45RA+/CD45RO-, como se describe en otra parte29,30.
NOTA: Consulte la Tabla de Materiales para conocer las especificaciones del clasificador FACS utilizado en este protocolo. - Inducir la diferenciación de linfocitos T CD4+ vírgenes vírgenes en linfocitos T helper 1 (linfocitos TH1 CD4+ ) como se describe en el punto 29. En resumen, cultive 1,5 x 106 células/ml de células T CD4+ vírgenes clasificadas por FACS en medio TH1 estimulándolas con perlas magnéticas anti-CD3/anti-CD28 en una proporción de 1:1. Cuente las células y divídalas cuando alcancen 1,5 × 106 células/ml cada 2-3 días (ver Tabla 1 para la composición del medio TH1).
- Evaluar la secreción de citocinas de la función efectora después de 7 días de diferenciación, como se describe en 29.
- Fijación y permeabilización celular
NOTA: Todos estos procedimientos se describieron en 30,31 con pequeñas adaptaciones.- Para garantizar una adhesión celular óptima, trate los cubreobjetos de vidrio (10 mm, espesor 1,5 H) con una solución de recubrimiento (Tabla 1) de la siguiente manera:
- Limpie los cubreobjetos de vidrio lavándolos inicialmente con agua destilada (ddH2O), seguido de un enjuague con etanol al 70% (EtOH), y dejándolos secar al aire.
- Coloque los cubreobjetos lavados en una placa de 24 pocillos múltiples para los pasos 1.2.1.3-1.3.
- Aplique una gota de 200 μL de solución de recubrimiento sobre el cubreobjetos de vidrio. Después de 5 minutos, retira la gota y déjala secar al aire.
- Lavar los cubreobjetos aplicando una gota de 200 μL de ddH2O. Después de 5 minutos, retire la gota y seque al aire.
- Repita 3 pasos 1.2.1.3-1.2.1.4.
- Resuspenda las células T CD4+ naïve y las células TH1 CD4+ en solución salina tamponada con fosfato (PBS) 1x a una concentración de 2 × 106 células/mL.
NOTA: La concentración indicada se recomienda específicamente para células pequeñas, como los linfocitos T primarios humanos. Para las células adherentes, no es necesario el tratamiento de recubrimiento de vidrio. En su lugar, proceda directamente con el cultivo de las células en la superficie del vidrio utilizando el medio de cultivo celular adecuado. - Aplique una gota de 200 μL de la suspensión celular sobre el cubreobjetos de vidrio. Deje que las células se siembren a temperatura ambiente (RT) durante 30 min; A continuación, retira la gota.
- Fije las células con paraformaldehído al 3% recién filtrado (solución de PFA, Tabla 1) durante 10 min en RT.
- Lave el cubreobjetos de vidrio con TPBS (Tabla 1) durante 3 x 5 min en RT.
- Retire el TPBS y agregue la solución de permeabilización (Tabla 1) durante 10 min en RT.
- Deseche la solución de permeabilización e incube la muestra en una solución de almacenamiento (Tabla 1) desde 1 h hasta toda la noche (ON) a 4 °C.
NOTA: En esta etapa, el protocolo se puede detener de manera segura y los cubreobjetos de vidrio se pueden conservar en solución de almacenamiento en una placa de 24 pocillos múltiples durante 3-4 semanas.
- Para garantizar una adhesión celular óptima, trate los cubreobjetos de vidrio (10 mm, espesor 1,5 H) con una solución de recubrimiento (Tabla 1) de la siguiente manera:
- Inmunofluorescencia
- (Opcional) Retire el cubreobjetos de la placa de 24 pocillos múltiples y congele rápidamente en hielo seco durante 30 s, descongele a RT y luego lave el cubreobjetos de vidrio en un pocillo prellenado con solución de almacenamiento.
- (Opcional) Repita 3 x paso 1.3.1.
- Lavar en solución de permeabilización durante 5 min en RT. A continuación, lavar durante 2 x 5 min con TPBS en RT.
- Incubar en HCl de 0,1 N durante 12 min en RT.
- Realiza dos lavados rápidos en 1x PBS.
NOTA: Las condiciones de permeabilización celular especificadas, incluidos los pasos de congelación y descongelación y el tratamiento con HCl, son óptimas para teñir componentes nucleares en células caracterizadas por cromatina densamente empaquetada. Sin embargo, cuando se trata de células caracterizadas por una cromatina menos compactada, es aconsejable reducir o evitar estos pasos. Además, para dirigirse a los componentes citoplasmáticos, considere reducir o eliminar el tratamiento con HCl. - Incubar las células con el anticuerpo primario (BRD4, 1:500 o SUZ12, 1:100) diluido en tampón de dilución de anticuerpos (Tabla 1) (200 μL por cada cubreobjetos de vidrio) ON a 4 °C.
NOTA: La inmunofluorescencia se puede realizar mediante la multiplexación de más de un anticuerpo primario dependiendo de las necesidades experimentales, los anticuerpos secundarios y los sistemas de detección. - Lavar 3 x 5 min con PBS-T (Tabla 1) a RT con agitación suave.
- Incubar las células con anticuerpo secundario diluido en tampón de dilución de anticuerpos (200 μL por cada cubreobjetos de vidrio) durante 1 h a RT.
NOTA: Elegir el anticuerpo secundario que mejor se adapte a las necesidades experimentales y a los sistemas de detección disponibles. Asegúrese de que cada anticuerpo secundario se dirija a la especie de la que se deriva el anticuerpo primario. Además, seleccione fluoróforos que sean compatibles con los filtros y la fuente de luz del microscopio. Es crucial evitar la superposición espectral entre el fluoróforo elegido y el espectro de emisión de la mancha nuclear. - Lavar 3 x 5 min con PBS-T en RT con una leve agitación.
- Tinción con 1 ng/mL de 4',6-diamidino-2-fenilindol (DAPI) diluido en 1x PBS durante 5 min a RT.
NOTA: Se pueden utilizar colorantes alternativos para la tinción nuclear siempre que no coincidan con el espectro de emisión de los anticuerpos secundarios. - Realiza varios lavados rápidos en 1x PBS.
- Monte el cubreobjetos de vidrio en un portaobjetos de microscopía con medios de montaje antidecoloración.
2. Adquisición de imágenes
NOTA: La duración de la adquisición de la imagen depende del instrumento y de los ajustes seleccionados.
- Adquisición de microscopio confocal
- Capture imágenes 3D con un microscopio confocal, estableciendo un tamaño de paso de 0,25 μm en z y un tamaño de píxel de 0,1-0,2 μm.
NOTA: Para lograr una resolución óptima de difracción de luz limitada con nuestro objetivo de aceite de 1,4 NA de 63x, establecemos el tamaño del agujero de alfiler en 0,8 UA, un promedio de línea de 2x y un tamaño de fotograma de 1024 × 1024 píxeles. El láser de excitación, así como la secuencia de adquisición de canales, se seleccionaron deliberadamente para evitar interferencias o diafonías entre los fluoróforos utilizados. Sin embargo, el ajuste de los parámetros debe adaptarse a las características específicas del microscopio y de la muestra. Consulte la Tabla de materiales para conocer las especificaciones del microscopio confocal utilizado en este protocolo. - Adquiera un número constante de campos aleatorios para abarcar aproximadamente 50 células por réplica biológica.
- Capture imágenes 3D con un microscopio confocal, estableciendo un tamaño de paso de 0,25 μm en z y un tamaño de píxel de 0,1-0,2 μm.
3. Análisis de imágenes
- Instalación de software
- Descargue e instale la última versión disponible de Fiji desde la página oficial de descargas de Fiji (https://imagej.net/software/fiji/downloads).
- Instale 3D Suite a través del sitio de actualización de Fiji o manualmente siguiendo las instrucciones en el sitio web de 3D Suite (https://mcib3d.frama.io/3d-suite-imagej/#download).
- Instale GDSC (FindFoci) a través del sitio de actualización de Fiji o manualmente siguiendo las instrucciones en el repositorio de GitHub (https://github.com/aherbert/gdsc).
- Conversión TIFF
- Descargue el script "convert_to_TIFF.py" (Archivo complementario 1).
- Arrastre y suelte el script en Fiji y ejecute el código.
- En el panel que aparece, vaya a la ruta de acceso en la que se almacena el experimento. La subcarpeta que contiene los archivos TIFF convertidos se crea en la misma carpeta del experimento.
- Inicialice la configuración en 3D Manager.
- Abra el panel de opciones del Administrador 3D haciendo clic en Complementos | 3DSuite | Opciones del Administrador 3D.
- Dentro de la ventana de opciones del Administrador 3D, seleccione las casillas de verificación correspondientes a las siguientes mediciones: Volumen (unidad), Valor medio de gris, Cuadro delimitador (pix), Valor de gris estándar de desarrollo, Centroide (pix) y Centroide (unidad).
- Marque las siguientes opciones: Excluir objetos en los bordes XY y Excluir objetos en los bordes Z y haga clic en Aceptar.
NOTA: Este paso debe realizarse solo una vez para configurar inicialmente las métricas que se almacenarán en los archivos de información espacial y cuantitativa. Las métricas seleccionadas son esenciales para garantizar el correcto funcionamiento de la canalización. Se pueden incluir métricas adicionales opcionales en este paso, como se describe con más detalle en la sección Discusión.
- Establezca los parámetros en la GUI de FindFoci.
NOTA: Este paso debe realizarse solo una vez para configurar la tubería semiautomatizada con parámetros óptimos, que luego se incorporarán a los scripts.- Abra la imagen de prueba. Duplique el canal de proteína, incluida la pila (Ctrl + Shift + D), marque la casilla de verificación hyperstack y especifique el número de canal adecuado dentro del cuadro Canales (c) y cámbiele el nombre en consecuencia.
- Inicie la grabadora de macros Fiji navegando a Complementos | Macros | Grabar.
- Abra el complemento FindFoci (haga clic en Complementos | GDSC | FindFoci | FindFoci GUI) y seleccione la imagen a analizar en el menú desplegable "Imagen".
- Configure los parámetros de la siguiente manera (Figura 2A): Desenfoque gaussiano = 1,5; método de fondo = DE por encima de la media; parámetro de fondo = 9; método de búsqueda = fracción de pico - fondo; parámetro de búsqueda = 0,7; método de pico = relativo por encima del fondo; parámetro de pico = 0,2; tamaño mínimo = 5; Picos máximos = 1.000.000.
NOTA: Los parámetros indicados se han seleccionado en base a nuestros estudios de casos y pueden no ser adecuados para otros procedimientos de tinción. - Para mejorar la identificación de focos, ajuste los siguientes parámetros:
- El desenfoque gaussiano define el grado de suavizado para mejorar los focos de los segmentos. Manténgalo cerca del diámetro de los focos (píxel).
- El parámetro de fondo establece un umbral para distinguir la señal de fondo de la señal de foco. Aumente los valores para imponer umbrales más estrictos.
- El parámetro de búsqueda define el porcentaje de fluorescencia del pico que se incluye en el reconocimiento de señales. Disminuya los valores para incluir áreas más alejadas del pico de fluorescencia.
- El parámetro de pico determina el grado en que dos picos de señal se consideran continuos o separados. La disminución del valor dará como resultado la separación de picos.
- Picos máximos especifica el número máximo de focos identificables. Establezca números altos para incluir todos los focos de la imagen.
- Ejecute FindFoci y copie la cadena que aparece en la ventana de la grabadora (Paso 3.4.2, Figura 2B) que contiene los parámetros seleccionados, excluyendo las comillas. Para obtener más información relacionada con la configuración, consulte las instrucciones del manual del complemento 22.
- Línea de cuantificación de proteínas nucleares
- Descargue el script "nuclear_prot_q.py" (Archivo Complementario 2).
- Arrastre y suelte el script en Fiji y haga clic en ejecutar para ejecutar el código.
- Siga las instrucciones del cuadro de diálogo que se muestra para procesar las imágenes.
- Canal del núcleo: introduzca el número correspondiente al canal de DAPI (o cualquier tinción nuclear).
- Desenfoque gaussiano del núcleo: introduzca el valor de sigma necesario para desenfocar la imagen para la segmentación.
NOTA: Mantenga este parámetro más cerca del diámetro del núcleo (es decir, 5-6 μm). Los valores más altos de sigma están indicados para tinciones no homogéneas. - Canal de proteína: introduzca el número correspondiente al canal de la tinción de interés.
- Parámetro FindFoci: pega la cadena obtenida del paso de grabación de macros en el pasaje 3.4.6 (Figura 2B).
- (Opcional) Control de calidad: comprobar la calidad de los núcleos segmentados. Esto pausará el script y permitirá la verificación manual de cada región nuclear de interés (ROI) generada.
- Seleccione un directorio de imágenes: haga clic en el botón Examinar para navegar a la carpeta que contiene los archivos TIFF que se van a analizar.
- Una vez que se hayan compilado todos los cuadros, haga clic en Aceptar para continuar con la ejecución.
- Si un núcleo no está correctamente segmentado y no cumple con los requisitos de calidad, elimínelo o modifíquelo como se indica en la Figura 2C.
- Compruebe la lista de ROI en la ventana ROIManager3D; si la lista está vacía, seleccione la ventana Combinar y haga clic en Cuantificar 3D para actualizar el administrador. A continuación, cierre la tabla de resultados de Cuantificar 3D. Seleccione el canal de los núcleos y haga clic en Live-ROI para ON.
- Seleccione el ROI que pertenezca al mismo núcleo y presione Fusionar o presione Eliminar en el caso de núcleos no deseados.
- Haga clic en Seleccionar todo y continúe con el análisis haciendo clic en Aceptar en la ventana de comprobación de máscara .
- Para obtener resultados, busque en la carpeta Cuantificación dentro de la ruta del archivo indicada en el paso 3.5.3.6, que contiene un archivo txt con registros del parámetro utilizado para el análisis.
- Canalización en Google Colab
- Descargue el cuaderno "final_nuclear_protein_metrics.ipynb" (Archivo complementario 3).
- Abra el bloc de notas en Google Colab (https://colab.research.google.com/).
- Cargue todas las carpetas que contienen los archivos .csv de cada campo de imagen en una carpeta de preferencias en Google Drive.
- Indique en el cuaderno la ruta de la carpeta donde se almacenan las subcarpetas de resultados y ejecute todas las celdas. Cuando el código ha terminado de compilarse, el archivo final de la hoja de cálculo que contiene todos los datos compilados se crea en la misma carpeta donde se cargaron los archivos .csv.
Resultados
El protocolo descrito en este método facilita la visualización y cuantificación de las alteraciones en la tinción de proteínas nucleares dentro de las células T primarias humanas, y se puede personalizar para diversos tipos de células y objetivos proteicos. Como casos de estudio, realizamos y analizamos la tinción de BRD4 y SUZ12 en células naïve y TH1 CD4+ .
BRD4 muestra un patrón de tinción bien punteado tanto en las células TH1 CD4+ inactivas naïve como en las diferenciadas, lo que permite el uso de los parámetros diseñados para la identificación de focos para ambos tipos de células (Figura 3A). Nuestra tubería permite la cuantificación de varios parámetros, incluido el número, el volumen, la señal de fluorescencia (expresada como intensidad de fluorescencia media (MFI)), la cobertura y la disposición espacial de los focos BRD4 dentro del núcleo (Figura 3B-F). Además, hemos introducido un cálculo automático para determinar el porcentaje de los focos en el volumen nuclear, lo que facilita la comparación entre células con diferentes tamaños nucleares (Figura 3E).
Finalmente, hemos proporcionado la opción de mapear las distancias de los focos desde el centro nuclear, lo que ayuda a comprender su posición nuclear (Figura 3F). Los resultados obtenidos de la cuantificación ponen de manifiesto alteraciones notables en los focos BRD4 entre los linfocitos TH1 y los linfocitos T CD4+ en reposo, que abarcan un aumento en el número, tamaño, brillo y volumen de los focos, así como diferencias en su distribución y localización. Estos hallazgos son consistentes con el aumento de la actividad transcripcional de las células T activadas/proliferantes en comparación con su contraparte quiescente28.
Por el contrario, SUZ12 no participa en la formación de condensado. En nuestra investigación, observamos una disparidad significativa en el patrón de tinción de SUZ12 entre los dos estados celulares: un patrón punteado en las células T naïve pasa a un patrón difuso en las células TH1 CD4+ (Figura 4A). Este cambio significativo en el comportamiento de las proteínas dificulta la comparabilidad de las características de los focos utilizando parámetros idénticos, ya que la identificación de puntos en las células TH1 CD4+ es en gran medida infructuosa (Figura 4A). Si bien generalmente se recomienda mantener la consistencia en los parámetros de adquisición y análisis al comparar la tinción de inmunofluorescencia en distintas condiciones celulares, los ajustes se vuelven necesarios en casos de cambios sustanciales en el objetivo de interés.
En este escenario específico, como se ilustra en la Figura 4B, a pesar de experimentar con varias combinaciones de parámetros, incluido el ajuste de los parámetros de fondo, el desenfoque gaussiano y los parámetros de búsqueda (como se sugiere en el paso del protocolo 3.4.5), FindFoci encuentra dificultades para distinguir los picos de señal verdaderos de los insignificantes debido a la naturaleza de distribución inherente de la proteína. Además, enfatizamos que las mediciones convencionales como la MFI nuclear pueden ser engañosas (Figura 4C), ya que no tienen en cuenta las alteraciones en la distribución de la señal.
Por lo tanto, para medir dichos cambios, sugerimos el uso del coeficiente de variación, una métrica que hemos introducido en este protocolo para caracterizar la difusión de proteínas, conocida como coeficiente de inhomogeneidad (CI). El CI captura con precisión la difusión de los focos SUZ12 en las células TH1 CD4+ sin forzar su identificación con enfoques de segmentación incorrectos (ver Figura 4D). Finalmente, al utilizar DAPI como control, que permanece distribuido uniformemente en ambas condiciones celulares, validamos aún más la eficacia de este parámetro (Figura 4E).
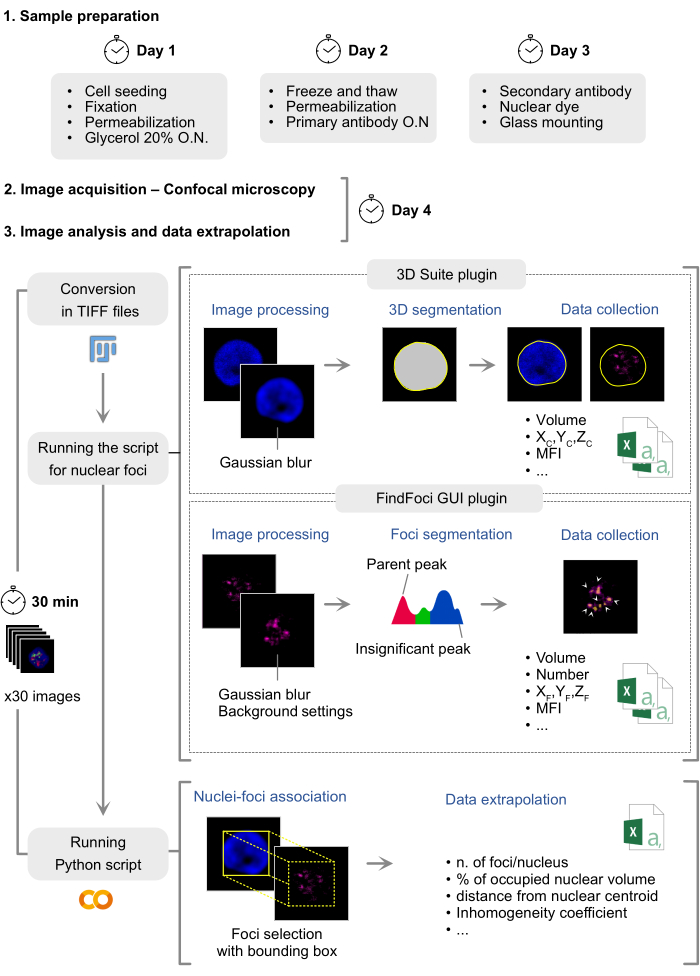
Figura 1: Representación esquemática de la preparación de inmunofluorescencia y análisis de datos. El protocolo para la tinción y el análisis de la inmunofluorescencia de proteínas nucleares consta de tres fases principales: preparación de la muestra, adquisición de imágenes y análisis, que abarcan aproximadamente 4 días hábiles. El script utiliza dos complementos, 3D Suite para realizar la segmentación de núcleos y FindFoci para procesar el canal de proteínas. La información espacial y cuantitativa sobre los focos y el núcleo se recopila en archivos csv separados. Un script ejecutable en Google Colab vincula las mediciones de focos con los núcleos correspondientes mediante el cuadro delimitador de núcleos y calcula las mediciones derivadas en función de los datos recopilados. Haga clic aquí para ver una versión más grande de esta figura.
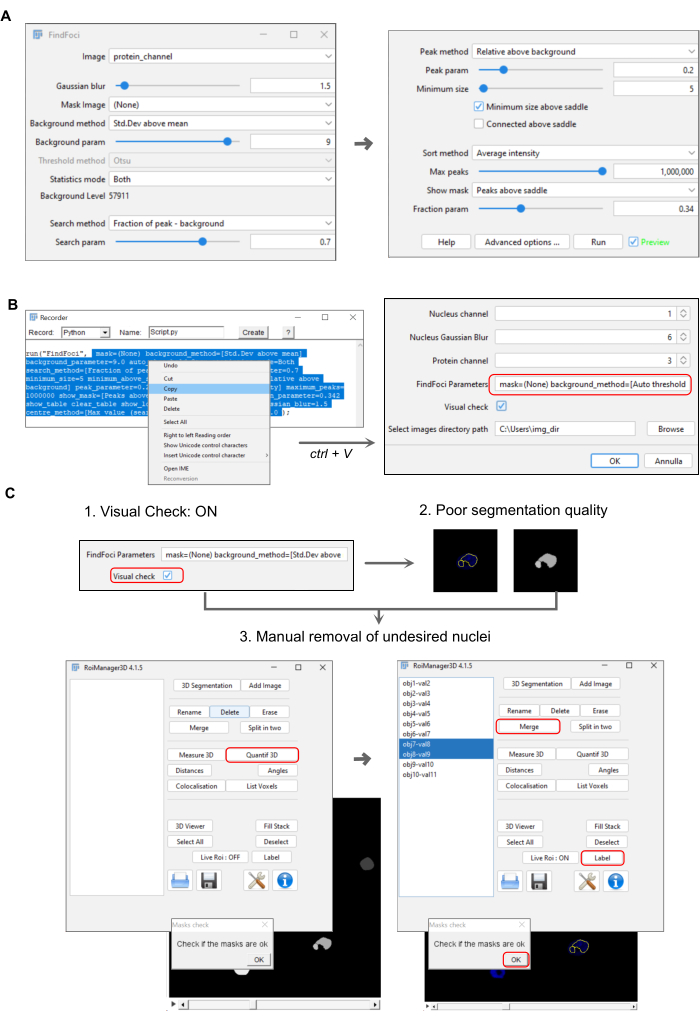
Figura 2: Representación de la configuración de la tubería de cuantificación de proteínas nucleares. (A) Ventana emergente de FindFoci que ilustra los parámetros para la identificación de focos (sección 3.4.3 del protocolo). (B) Ventana emergente que representa la macro que registra los parámetros seleccionados de FindFoci. La cadena copiada se pega en la ventana de inicio (pasos del protocolo 3.4.6-3.5.3.4). (C) Panel que muestra el paso de control de calidad de segmentación de núcleos para eliminar o modificar manualmente las regiones nucleares de interés utilizando el administrador 3D (pasos de protocolo 3.5.5.1 -3.5.5.3). Haga clic aquí para ver una versión más grande de esta figura.
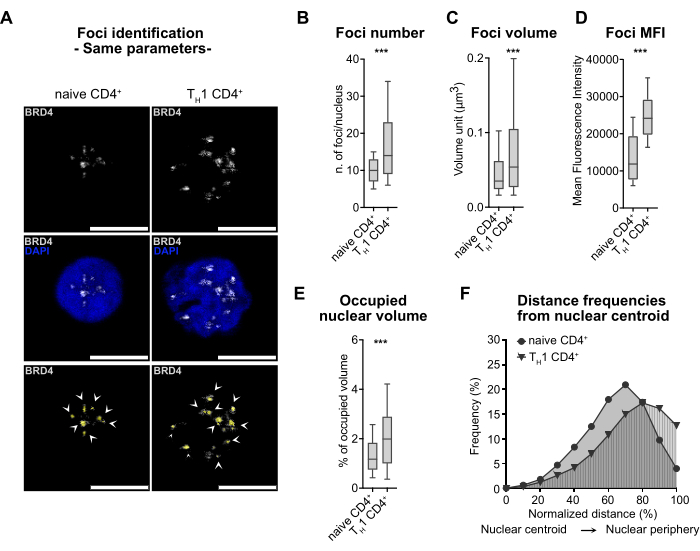
Figura 3: Comparaciones entre distintas condiciones celulares basadas en análisis de focos BRD4. (A) Imágenes representativas de microscopía de fluorescencia confocal de la tinción por inmunofluorescencia de BRD4 (gris) en células T CD4+ primarias humanas y células TH1 CD4+ . Los núcleos se contratiñen con DAPI (azul). Aumento original 63x; Barra de escala = 5 μm. En la parte inferior, los focos BRD4, identificados por la tubería, están marcados con puntas de flecha blancas y contrarrestados en amarillo (abajo). (B) Diagrama de caja que representa el número de focos/núcleo BRD4 en células naïve y TH1 CD4+ (n = 2 individuos). (C) Representación en diagrama de caja del volumen (μm3) de los focos BRD4 en células naïve y TH1 CD4+ (n = 2 individuos). (D) Representación en diagrama de caja de focos MFI de focos BRD4 en células naïve y TH1 CD4+ (n = 2 individuos). (E) Diagrama de caja que representa el porcentaje del volumen nuclear ocupado por los focos BRD4 en relación con el volumen total del núcleo (n = 2 individuos). (F) Representación de las frecuencias de distancia de los focos BRD4 desde el centroide nuclear hasta la periferia nuclear (n = 2 individuos). Haga clic aquí para ver una versión más grande de esta figura.
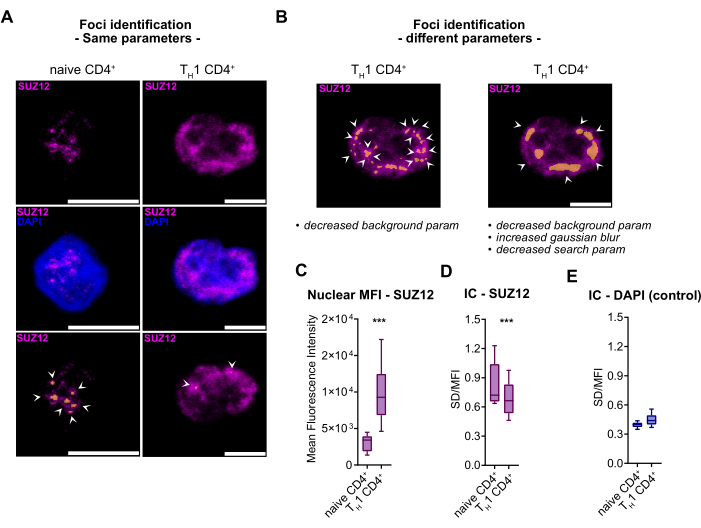
Figura 4: Coeficiente de inhomogeneidad, una métrica para cuantificar la transición de un patrón punteado a uno disperso en las señales de inmunofluorescencia SUZ12. (A) Imágenes representativas de microscopía de fluorescencia confocal de la tinción de inmunofluorescencia para SUZ12 (magenta) en células T CD4+ naïve primarias humanas y TH1 CD4+ células. Los núcleos se contratiñen con DAPI (azul). Aumento original 63x; Barra de escala = 5 μm. En la parte inferior, los focos SUZ12 identificados por la tubería están marcados con una punta de flecha blanca y contados en naranja (B). Ejemplos de identificación incorrecta de focos en células TH1 CD4+ que se muestran con flechas blancas y máscara naranja con dos configuraciones de parámetros diferentes. (C) Representación en diagrama de caja de MFI nuclear de SUZ12 en células naïve y TH1 CD4+ (n = 2 individuos). (D) Representación en diagrama de caja de SUZ12 IC en células naïve y TH1 CD4+ (n = 2 individuos). (E) Representación en diagrama de caja de DAPI IC en células naïve y TH1 CD4+ (n = 2 individuos). Abreviaturas: DAPI = 4',6-diamidino-2-fenilindol; MFI = intensidad media de fluorescencia; IC = coeficiente de inhomogeneidad. Haga clic aquí para ver una versión más grande de esta figura.
| Solución | Composición | Comentarios/Descripción | |
| TH1 medio | RPMI con GlutaMAX-I, 10% (v/v) de suero fetal bovino (FBS), 1% (v/v) de aminoácidos no esenciales, 1 mM de piruvato de sodio, 50 UI/mL de penicilina, 50 μg/mL de estreptomicina, 20 UI/mL de IL-2 recombinante, 10 ng/mL de IL-12 recombinante, 2 mg/mL neutralizante anti-IL-4. | Paso 1.1.4. | |
| Solución de recubrimiento | 0,1% de poli-L-lisina en ddH2O | Paso 1.2.1. | |
| PBS-T | 1x PBS/0.1% TWEEN 20 pH 7.0 | Paso 1.3. | |
| Solución de PFA para la fijación | 3% de paraformaldehído (PFA) diluido en 0,1% de PBS-TWEEN | Paso 1.2.4. | |
| TPBS | 0.05% Triton X-100 diluido en 1x PBS | Paso 1.2., 1.3. | |
| Solución de permeabilización | 0,5% TPBS diluido en 1x PBS | Paso 1.2., 1.3. | |
| Solución de almacenamiento | 20% glicerol/1x PBS | Paso 1.2.7., 1.3.1. | |
| Tampón de dilución de anticuerpos | 0.1% PBS-TWEEN/2% Suero de cabra/1% BSA | Paso 1.3.6., 1.3.8. | |
Tabla 1: Composición de los medios y búferes utilizados en este protocolo.
Legajo Complementario 1: "convert_to_TIFF.py". Este archivo contiene el script para convertir cualquier archivo de imagen (por ejemplo, ND2, LIF, etc.) en archivos TIFF necesarios para procesos de cuantificación precisos. Haga clic aquí para descargar este archivo.
Legajo Complementario 2: "nuclear_prot_q.py". Este archivo contiene el script que permite medir y cuantificar imágenes que contienen al menos dos canales (tinción de núcleos, tinción de proteínas nucleares). Haga clic aquí para descargar este archivo.
Archivo complementario 3: "final_nuclear_protein metrics.ipynb". Este archivo contiene un cuaderno de Jupyter Notebook que extrae y compila un resumen de todos los parámetros relevantes en un único archivo de Excel, utilizando la salida de "nuclear_prot_q.py" como entrada. Haga clic aquí para descargar este archivo.
Discusión
En este estudio, presentamos un método para realizar experimentos de inmunofluorescencia sobre proteínas nucleares en linfocitos T humanos. Este método ofrece flexibilidad para su uso con varios tipos de células a través de modificaciones menores en los pasos de fijación y permeabilización, como se describió anteriormente30,31.
Nuestro flujo de trabajo de imágenes se basa en técnicas establecidas descritas en la literatura, específicamente FindFoci y 3D Suite 22,23. A diferencia de publicaciones anteriores, proporcionamos un código Jython ejecutable integrado en Fiji para una tubería semiautomatizada, lo que permite un seguimiento meticuloso de los parámetros y controles de calidad visuales. En particular, introdujimos la capacidad opcional de regular la calidad de la segmentación de núcleos en la sección 3.5.3.5 del protocolo e implementamos un paso correctivo en la sección 3.5.5 del protocolo. Con el script ejecutable en Google Colab, los usuarios pueden recuperar mediciones completas y elegir las más adecuadas para sus escenarios biológicos específicos.
La canalización de análisis proporcionada está escrita en Jython (Java + Python) y requiere la instalación de Fiji para su ejecución, junto con dos complementos distintos: 3D Suite y FindFoci (Figura 1). La canalización comienza con la conversión de imágenes a formato TIFF y el preprocesamiento del canal que contiene los núcleos con un filtro de desenfoque gaussiano para mejorar la segmentación de los núcleos. Dirigimos 3D Suite para capturar i) información espacial, como el volumen nuclear, el centroide y las coordenadas del cuadro delimitador (que representa el cuadro más pequeño que encierra el objeto y se utiliza para asociar focos al núcleo derecho), y ii) información cuantitativa, incluida la MFI y la desviación estándar de la señal. Posteriormente, la canalización emplea FindFoci para identificar los máximos locales en la señal de los focos, eliminando la dependencia de los métodos de umbral globales (que a menudo dependen de la discreción del usuario y pueden carecer de precisión), y registra información espacial y cuantitativa relativa a los focos. Por lo tanto, la carpeta de salida contendrá dos archivos CSV por canal, que abarcan métricas espaciales ("M_.csv") y cuantitativas ("Q_.csv").
El archivo final, que contiene todas las mediciones recopiladas y derivadas, se genera mediante la ejecución de un script de Python en Google Colab. Este archivo incluye métricas como el número de focos por núcleo, MFI de focos, coeficiente de inhomogeneidad (IC), MFI nuclear, distancia de focos al centroide nuclear y porcentaje de volumen nuclear ocupado. Estimamos un tiempo de procesamiento de aproximadamente 1 minuto por imagen, incluidos los pasos de control de calidad visual. Este tiempo de procesamiento se puede acelerar aún más mediante la optimización de la configuración de inmunofluorescencia y los parámetros de análisis.
La tubería calcula los parámetros de las proteínas nucleares que exhiben patrones bien punteados y difusos, lo que permite a los usuarios seleccionar el parámetro más apropiado. Recomendamos utilizar el coeficiente de no homogeneidad para describir la disolución de los focos de proteínas.
Un paso crítico para garantizar la identificación adecuada de los focos implica el ajuste de parámetros para discriminar los picos de señal genuinos del ruido de fondo. Es crucial realizar pruebas exhaustivas en imágenes aleatorias antes de ejecutar la canalización. Si bien nuestro estudio se centra principalmente en un solo canal, la cartera apoya la introducción de varias combinaciones de colorantes dirigidas a diferentes proteínas en experimentos de inmunofluorescencia. No admite el análisis simultáneo de varios canales y debe ejecutarse por separado para cada canal. Además, hemos optado por presentar medidas específicas en este protocolo; Sin embargo, en la sección 3.3.2 también se pueden incorporar métricas adicionales, como la intensidad integrada para evaluar los cambios absolutos en la señal de fluorescencia o parámetros físicos como la circularidad y la compacidad. La inclusión de métricas que actualmente no están integradas en esta canalización requiere dominio de la programación de Python para integrarlas en el archivo de métricas final; en caso contrario, se incluirán automáticamente en los ficheros de información espacial ("M_.csv") y cuantitativa ("Q_.csv").
El tiempo de esta canalización depende de factores como el tamaño y la cantidad de imágenes, el número de canales por análisis y la configuración del microscopio. Además, el rendimiento de la máquina influye en el tiempo de ejecución, y los pasos visuales pueden ralentizar el proceso.
Vale la pena señalar que, si bien la microscopía confocal ofrece información valiosa, sigue siendo una tecnología limitada por la difracción que puede limitar la precisión de las estructuras nucleares o los focos de proteínas observados. Para mejorar la solidez del análisis, sugerimos aumentar el número de núcleos y muestras biológicas examinadas. A medida que aumenta el número de núcleos analizados, cualquier cambio notable en la morfología nuclear o la distribución de proteínas se hace más evidente, independientemente de las limitaciones de resolución.
En términos de posibles aplicaciones futuras, este método exhibe versatilidad y adaptabilidad a varios tipos de células y protocolos de tinción. Por ejemplo, puede emplearse fácilmente en experimentos de radiobiología, donde la cuantificación precisa de los focos inducidos por la radiación ionizante (IRIF) sirve como indicador directo del daño en el ADN32. Si bien nuestro énfasis principal radica en el análisis nuclear, el protocolo se puede personalizar fácilmente incorporando un tinte citoplasmático y sustituyéndolo por el canal nuclear dentro del script. Además, el análisis puede integrarse sin problemas en otras metodologías experimentales, como el ADN y el ARN FISH30,31, aprovechando los parámetros predeterminados para el análisis de topología nuclear.
Divulgaciones
R.V. tiene una colaboración científica con la startup T-One Therapeutics Srl; B.B. y F.M. son cofundadores de la startup T-One Therapeutics Srl; E.P. es actualmente empleado de T-One Therapeutics Srl; Todos los demás autores declaran que no tienen intereses contrapuestos.
Agradecimientos
Agradecemos la asistencia científica y técnica del INGM Imaging Facility, en particular, C. Cordiglieri y A. Fasciani, y de la instalación de clasificación INGM FACS en particular M.C Crosti (Istituto Nazionale di Genetica Molecolare 'Romeo ed Enrica Invernizzi' (INGM), Milán, Italia). Agradecemos al Sr. Giannaccari por su apoyo técnico informático. Este trabajo fue financiado por las siguientes subvenciones: Fondazione Cariplo (Bando Giovani, subvención n.º 2018-0321) y Fondazione AIRC (subvención n.º MFAG 29165) a F.M. Ricerca Finalizzata, (subvención n.º GR-2018-12365280), Fondazione AIRC (subvención n.º 2022 27066), Fondazione Cariplo (subvención n.º 2019-3416), Fondazione Regionale per la Ricerca Biomedica (FRRB CP2_12/2018), Piano Nazionale di Ripresa e Resilienza (PNRR) (subvención n.º G43C22002620007) y Progetti di Rilevante Interesse Nazionale (PRIN) (subvención n.º 2022PKF9S) a B. B.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL Safe-Lock Tubes | Eppendord | #0030121503 | Protocol section 1 |
| 10 mL Serological pipettes | VWR | #612-3700 | Protocol section 1 |
| 20 µL barrier pipette tip | Thermo Scientific | #2149P-HR | Protocol section 1 |
| 50 mL Polypropylene Conical Tube | Falcon | #352070 | Protocol section 1 |
| 200 µL barrier pipette tip | Thermo Scientific | #2069-HR | Protocol section 1 |
| antifade solution - ProlongGlass - mountingmedia | Invitrogen | #P36984 | Step 1.3.12 |
| BSA (Bovine Serum Albumin) | Sigma | #A7030 | Step 1.3.6., 1.3.8. |
| CD4+ T Cell Isolation Kit | Miltenyi Biotec | #130-096-533 | Step 1.1.2. |
| DAPI (4,6-diamidino-2-phenylindole) | Invitrogen | Cat#D1306 | Step 1.3.10. |
| Dry ice | Step 1.3.1. | ||
| Dynabeads Human T-activator anti-CD3/anti-CD28 bead | Life Technologies | #1131D | magnetic beads step 1.1.4. |
| EtOH | Carlo Erba | #4146320 | Step 1.2.1.1. |
| FACSAria SORP | BD Bioscences | Step 1.1.3. Equipped with BD FACSDiva Software version 8.0.3 | |
| FBS (Fetal Bovine Serum) | Life Technologies | #10270106 | Step 1.1.4 |
| FICOLL PAQUE PLUS | Euroclone | GEH17144003F32 | Step 1.1.1. |
| FIJI Version 2.14.0 | - | - | Protocol section 3 |
| Glass coverslip (10 mm, thickness 1.5 H) | Electron Microscopy Sciences | #72298-13 | Step 1.2.1. |
| Glycerol | Sigma | #G5516 | Step 1.2.7-1.3.1. |
| Goat anti-Rabbit AF568 secondary antibody | Invitrogen | A11036 | Step 1.3.8. |
| HCl | Sigma | #320331 | Step 1.3.4. |
| human neutralizing anti-IL-4 | Miltenyi Biotec | Cat#130-095-753 | Step 1.1.4. |
| human recombinant IL-12 | Miltenyi Biotec | Cat#130-096-704 | Step 1.1.4. |
| human recombinant IL-2 | Miltenyi Biotec | Cat#130-097-744 | Step 1.1.4. |
| Leica TCS SP5 Confocal microscope | Leica Microsystems | - | Protocol section 2, Equipped with HCX PL APO 63x, 1.40 NA oil immersion objective, with an additional 3x zoom. Pinhole size : 0.8 AU. Line average 2×. Frame size 1024×1024 pixel. |
| MEM Non-Essential Amino Acids Solution | Life Technologies | #11140035 | Step 1.1.4. |
| Microscope Slides | VWR | #631-1552 | Step 1.3.12. |
| Mouse monoclonal anti-Human CD4 APC-Cy7 (RPA-T4 clone) | BD Bioscience | #557871 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RA PECy5 (5H9 clone) | BD Bioscience | #552888 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RO APC (UCHL1 clone) | Miltenyi Biotec | #130-113-546 | Step 1.1.3. |
| Multiwell 24 well | Falcon | #353047 | Protocol section 1 |
| Normal Goat Serum | Invitrogen | PCN5000 | Step 1.3.6., 1.3.8. |
| PBS | Life Technologies | #14190094 | Protocol section 1 |
| Penicillin/Streptomycin solution | Life Technologies | #15070063 | Step 1.1.4. |
| PFA | Sigma | #P6148 | Step 1.2.4. |
| poly-L-lysine | Sigma | #P8920 | 1.2.1. |
| Primary antibody - BRD4 | Abcam | #ab128874 | Step 1.3.6. |
| Primary antibody - SUZ12 | Cel Signalling | mAb #3737 | Step 1.3.6. |
| RPMI 1640 W/GLUTAMAX-I | Life Technologies | #61870010 | Step 1.1.4. |
| Sodium Pyruvate | Life Technologies | #11360039 | Step 1.1.4. |
| Triton X-100 | Sigma | #T8787 | Step 1.2., 1.3. |
| TWEEN 20 | Sigma | #P9416 | Step 1.3. |
| Tweezers | - | - | Protocol section 1 |
Referencias
- Aboelnour, E., Bonev, B. Decoding the organization, dynamics, and function of the 4D genome. Dev Cell. 56 (11), 1562-1573 (2021).
- Erdel, F., Rippe, K. Formation of chromatin subcompartments by phase separation. Biophys J. 114 (10), 2262-2270 (2018).
- Henninger, J. E., et al. RNA-mediated feedback control of transcriptional condensates. Cell. 184 (1), 207-225 (2021).
- Guo, Y. E., et al. Pol II phosphorylation regulates a switch between transcriptional and splicing condensates. Nature. 572 (7770), 543-548 (2019).
- Bhat, P., et al. 3D genome organization around nuclear speckles drives mRNA splicing efficiency. bioRxiv. , (2023).
- Spector, D. L., Lamond, A. I. Nuclear speckles. Cold Spring Harb Perspect Biol. 3 (2), a000646 (2011).
- Kilic, S., et al. Phase separation of 53BP1 determines liquid-like behavior of DNA repair compartments. EMBO J. 38 (16), e101379 (2019).
- Parker, M. W., et al. A new class of disordered elements controls DNA replication through initiator self-assembly. Elife. 8, e48562 (2019).
- Feric, M., et al. Coexisting liquid phases underlie nucleolar subcompartments. Cell. 165 (7), 1686-1697 (2016).
- Yoneda, M., Nakagawa, T., Hattori, N., Ito, T. The nucleolus from a liquid droplet perspective. J Biochem. 170 (2), 153-162 (2021).
- Alberti, S., Carra, S. Nucleolus: A liquid droplet compartment for misbehaving proteins. Curr Biol. 29 (19), R930-R932 (2019).
- Strom, A. R., et al. Phase separation drives heterochromatin domain formation. Nature. 547 (7662), 241-245 (2017).
- Sanulli, S., et al. HP1 reshapes nucleosome core to promote phase separation of heterochromatin. Nature. 575 (7782), 390-394 (2019).
- Hyman, A. A., Weber, C. A., Julicher, F. Liquid-liquid phase separation in biology. Annu Rev Cell Dev Biol. 30, 39-58 (2014).
- Feric, M., Misteli, T. Phase separation in genome organization across evolution. Trends Cell Biol. 31 (8), 671-685 (2021).
- Mitrea, D. M., et al. Methods for physical characterization of phase-separated bodies and membrane-less organelles. J Mol Biol. 430 (23), 4773-4805 (2018).
- Fetter, J., et al. Endogenous gene tagging with fluorescent proteins. Methods Mol Biol. 1239, 231-240 (2015).
- Alberti, S., Gladfelter, A., Mittag, T. Considerations and challenges in studying liquid-liquid phase separation and biomolecular condensates. Cell. 176 (3), 419-434 (2019).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. J Cell Sci. 124 (10), 1607-1611 (2011).
- Scalisi, S., Ahmad, A., D'Annunzio, S., Rousseau, D., Zippo, A. Quantitative analysis of PcG-associated condensates by stochastic optical reconstruction microscopy (STORM). Methods Mol Biol. 2655, 183-200 (2023).
- Shihan, M. H., Novo, S. G., Le Marchand, S. J., Wang, Y., Duncan, M. K. A simple method for quantitating confocal fluorescent images. Biochem Biophys Rep. 25, 100916 (2021).
- Herbert, A. D., Carr, A. M., Hoffmann, E. FindFoci: a focus detection algorithm with automated parameter training that closely matches human assignments, reduces human inconsistencies and increases speed of analysis. PLoS One. 9 (12), e114749 (2014).
- Ollion, J., Cochennec, J., Loll, F., Escude, C., Boudier, T. TANGO: a generic tool for high-throughput 3D image analysis for studying nuclear organization. Bioinformatics. 29 (14), 1840-1841 (2013).
- Sabari, B. R., et al. Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361 (6400), 3958 (2018).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19 (4), 523-534 (2005).
- Pasini, D., Bracken, A. P., Jensen, M. R., Denchi, E. L., Helin, K. Suz12 is essential for mouse development and for EZH2 histone methyltransferase activity. EMBO J. 23 (20), 4061-4071 (2004).
- Margueron, R., Reinberg, D. The Polycomb complex PRC2 and its mark in life. Nature. 469 (7330), 343-349 (2011).
- Peng, Z., et al. Brd4 regulates the homeostasis of CD8(+) T-lymphocytes and their proliferation in response to antigen stimulation. Front Immunol. 12, 728082 (2021).
- Marasca, F., et al. LINE1 are spliced in non-canonical transcript variants to regulate T cell quiescence and exhaustion. Nat Genet. 54 (2), 180-193 (2022).
- Marasca, F., Cortesi, A., Bodega, B. 3D COMBO chrRNA-DNA-ImmunoFISH. Methods Mol Biol. 2157, 281-297 (2021).
- Marasca, F., Cortesi, A., Manganaro, L., Bodega, B. 3D Multicolor DNA FISH tool to study nuclear architecture in human primary cells. J Vis Exp. (155), (2020).
- Jakob, B., Splinter, J., Durante, M., Taucher-Scholz, G. Live cell microscopy analysis of radiation-induced DNA double-strand break motion. Proc Natl Acad Sci U S A. 106 (9), 3172-3177 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados