Method Article
Un pipeline polyvalent pour l’analyse des changements dynamiques dans les corps nucléaires dans une variété de types de cellules
Dans cet article
Résumé
Cette méthode décrit un protocole d’immunofluorescence et un pipeline de quantification pour évaluer la distribution des protéines avec des modèles d’organisation nucléaire variés dans les lymphocytes T humains. Ce protocole fournit des conseils étape par étape, en commençant par la préparation des échantillons et en continuant par l’exécution d’une analyse semi-automatisée aux Fidji, se terminant par le traitement des données par un ordinateur portable Google Colab.
Résumé
Divers processus nucléaires, tels que le contrôle transcriptionnel, se produisent dans des structures discrètes appelées foyers qui sont discernables grâce à la technique d’immunofluorescence. L’étude de la dynamique de ces foyers dans diverses conditions cellulaires par microscopie fournit des informations précieuses sur les mécanismes moléculaires qui régissent l’identité et les fonctions cellulaires. Cependant, la réalisation de tests d’immunofluorescence sur différents types de cellules et l’évaluation des altérations dans l’assemblage, la diffusion et la distribution de ces foyers présentent de nombreux défis. Ces défis englobent la complexité de la préparation des échantillons, de la détermination des paramètres d’analyse des données d’imagerie et de la gestion de volumes de données importants. De plus, les flux de travail d’imagerie existants sont souvent adaptés aux utilisateurs expérimentés, ce qui limite l’accessibilité à un public plus large.
Dans cette étude, nous présentons un protocole d’immunofluorescence optimisé conçu pour l’étude des protéines nucléaires dans différents types de lymphocytes T primaires humains qui peuvent être personnalisés pour n’importe quelle protéine d’intérêt et type de cellule. De plus, nous présentons une méthode pour quantifier de manière impartiale la coloration des protéines, qu’elles forment des foyers distincts ou qu’elles présentent une distribution nucléaire diffuse.
La méthode que nous proposons offre un guide complet, de la coloration cellulaire à l’analyse, en s’appuyant sur un pipeline semi-automatisé développé à Jython et exécutable aux Fidji. De plus, nous fournissons un script Python convivial pour rationaliser la gestion des données, accessible publiquement sur un cahier Google Colab. Notre approche a démontré son efficacité à produire des analyses d’immunofluorescence très informatives pour des protéines présentant divers modèles d’organisation nucléaire dans différents contextes.
Introduction
L’organisation du génome eucaryote est régie par de multiples couches de modifications épigénétiques1, coordonnant plusieurs fonctions nucléaires qui peuvent se produire dans des compartiments spécialisés appelés corps nucléaires ou condensats2. Au sein de ces structures, des processus tels que l’initiation de la transcription3, le traitement de l’ARN 4,5,6, la réparation de l’ADN 7,8, la biogenèse des ribosomes 9,10,11 et la régulation de l’hétérochromatine12,13 ont lieu. La régulation des corps nucléaires s’ajuste sur les dimensions spatiales et temporelles pour répondre aux besoins cellulaires, guidée par les principes de séparation de phase14,15. Par conséquent, ces corps fonctionnent comme des usines transitoires où les composants fonctionnels s’assemblent et se désassemblent, subissant des changements de taille et de distribution spatiale. Par conséquent, la compréhension des caractéristiques des protéines nucléaires par microscopie, y compris leur propension à former des corps et leur arrangement spatial dans différentes conditions cellulaires, offre des informations précieuses sur leurs rôles fonctionnels. La microscopie à fluorescence est une méthode largement utilisée pour étudier les protéines nucléaires, permettant leur détection à l’aide d’anticorps fluorescents ou exprimant directement des cibles avec un rapporteur de protéines fluorescentes16,17.
Dans ce contexte, les corps nucléaires apparaissent comme des foyers brillants ou puncta, avec un degré notable de sphéricité, ce qui les rend facilement distinguables de l’environnement environnant16,18. Les techniques de super-résolution telles que STORM et PALM, en offrant une résolution améliorée (jusqu’à 10 nm)19, permettent une caractérisation plus précise de la structure et de la composition de condensatsspécifiques20. Cependant, leur accessibilité est limitée par les dépenses d’équipement et les compétences spécialisées nécessaires à l’analyse des données. Par conséquent, la microscopie confocale reste populaire en raison de son équilibre favorable entre la résolution et une utilisation plus large. Cette popularité est facilitée par la suppression inhérente de la lumière floue, ce qui réduit la nécessité de procédures de post-traitement étendues pour une segmentation précise, sa disponibilité généralisée dans les instituts de recherche, son temps d’acquisition effectif et sa préparation d’échantillons qui est généralement efficace. Cependant, la mesure précise de la distribution, de l’assemblage ou de la diffusion des protéines à l’aide de tests d’immunofluorescence dans diverses conditions cellulaires pose des défis, car de nombreuses méthodes existantes manquent de conseils sur la sélection de paramètres appropriés pour les protéines ayant des modèles de distribution variables21. De plus, la gestion du grand volume de données qui en résulte peut être intimidante pour les utilisateurs ayant une expérience limitée de l’analyse des données, ce qui peut compromettre la signification biologique des résultats.
Pour relever ces défis, nous introduisons un protocole détaillé étape par étape pour la préparation de l’immunofluorescence et l’analyse des données, visant à fournir une méthode non biaisée pour quantifier la coloration des protéines avec divers modèles d’organisation (Figure 1). Ce pipeline semi-automatisé est conçu pour les utilisateurs ayant une expertise limitée en analyse informatique et d’imagerie. Il combine les fonctionnalités de deux plugins fidjiens établis : FindFoci22 et 3D suite23. En intégrant la capacité d’identification précise des foyers de FindFoci aux fonctionnalités d’identification et de segmentation d’objets dans l’espace 3D offertes par 3D suite, notre approche génère deux fichiers CSV par canal pour chaque champ d’acquisition. Ces fichiers contiennent des informations complémentaires qui facilitent le calcul de métriques adaptées à divers types de distribution de signaux, telles que le nombre de foyers par cellule, la distance des foyers par rapport au centroïde nucléaire et le coefficient d’inhomogénéité (CI), que nous avons introduit pour la coloration diffuse des protéines. En outre, nous reconnaissons que l’extrapolation des données peut prendre beaucoup de temps pour les utilisateurs ayant des compétences limitées en matière de traitement des données. Pour rationaliser ce processus, nous fournissons un script Python qui compile automatiquement toutes les mesures collectées dans un seul fichier pour chaque expérience. Les utilisateurs peuvent exécuter ce script sans avoir besoin d’installer un logiciel de langage de programmation. Nous fournissons un code exécutable sur Google Colab, une plate-forme basée sur le cloud qui permet d’écrire des scripts Python directement dans le navigateur. Cela garantit que notre méthode est intuitive et facilement accessible pour une utilisation immédiate.
Nous démontrons l’efficacité de notre protocole dans l’analyse et la quantification des altérations de la distribution du signal de deux protéines nucléaires : la protéine 4 contenant le bromodomaine (BRD4) et le suppresseur de zeste-12 (SUZ12). BRD4 est une protéine coactivatrice bien documentée au sein du complexe Mediator connue pour former des condensats associés à l’initiation transcriptionnelle dépendante de la polymérase II24,25. SUZ12 est un composant protéique du complexe répressif Polycomb 2 (PRC2) responsable de la régulation du dépôt de la modificationdes histones H3K27me3 26,27. Ces protéines présentent des motifs différents au sein de deux types de cellules distincts : les lymphocytes T humains CD4+ naïfs fraîchement isolés, qui sont quiescents et présentent des taux d’activité transcriptionnelle lents, et les cellules TH1 CD4+ différenciées in vitro, qui sont des cellules effectrices proliférantes spécialisées présentant une transcription accrue28.
Protocole
L’utilisation d’échantillons humains à des fins de recherche a été approuvée par les comités d’éthique de la Fondazione Istituto di Ricovero e Cura a Carattere Scientifico (IRCCS) Cà Granda Ospedale Maggiore Policlinico (Milan), et le consentement éclairé a été obtenu de tous les sujets (numéros d’autorisation : 708_2020). Le protocole est organisé en trois sections principales : l’exécution de l’immunofluorescence, l’acquisition d’images et l’analyse d’images. En moyenne, il faut 4 jours ouvrables pour le réaliser (Figure 1).
1. Préparation par immunofluorescence
REMARQUE : Ce protocole d’immunofluorescence peut être facilement personnalisé pour divers types de cellules et cibles protéiques en ajustant les conditions de fixation et de perméabilisation. La préparation de l’immunofluorescence prend généralement moins de 3 jours, la durée de l’incubation primaire de l’anticorps variant en fonction de la qualité de l’anticorps et de la protéine cible (Figure 1).
- Préparation des échantillons
- Isolez les cellules mononucléées du sang périphérique humain (PBMC) par centrifugation à gradient de densité via un milieu d’une densité d’environ 1,077 g/mL selon les instructions du fabricant (Table des matériaux).
- Isolez les lymphocytes T CD4+ des PBMC à l’aide de billes magnétiques, en suivant les instructions du fabricant (Table des matières).
- Colorer les cellules avec des anticorps pour CD4, CD45RA et CD45RO (Table des matériaux) et procéder au tri FACS des lymphocytes T CD4+ naïfs en tant que cellules CD4+/CD45RA+/CD45RO-, comme décrit ailleurs29,30.
REMARQUE : Voir la table des matériaux pour les spécifications du trieur FACS utilisé dans ce protocole. - Induire la différenciation des lymphocytes T CD4+ naïfs en lymphocytes T auxiliaires 1 (cellules T,H, 1, CD4+ ), comme décrit en 29. Brièvement, cultivez 1,5 x 106 cellules/mL de lymphocytes T CD4+ naïfs triés par FACS dans un milieu TH1 en les stimulant avec des billes magnétiques anti-CD3/anti-CD28 dans un rapport de 1:1. Comptez les cellules et divisez-les lorsqu’elles atteignent 1,5 × 106 cellules/ml tous les 2-3 jours (voir le tableau 1 pour la composition du milieu TH1).
- Évaluer la sécrétion de cytokines de la fonction effectrice après 7 jours de différenciation, comme décrit en 29.
- Fixation et perméabilisation des cellules
REMARQUE : Toutes ces procédures ont été décrites en 30 et 31 avec des adaptations mineures.- Pour assurer une adhérence optimale des cellules, traiter les lamelles de verre (10 mm, épaisseur 1,5 H) avec une solution de revêtement (tableau 1) comme suit :
- Nettoyez les lamelles en verre en les lavant d’abord à l’eau distillée (ddH2O), puis en les rinçant à l’éthanol à 70 % (EtOH), et en les laissant sécher à l’air.
- Placez les lamelles lavées dans une plaque à 24 puits multiples pour les étapes 1.2.1.3-1.3.
- Appliquez une goutte de 200 μL de solution de revêtement sur la lamelle de verre. Au bout de 5 min, retirez la goutte et laissez-la sécher à l’air libre.
- Lavez les lamelles en appliquant une goutte de 200 μL de ddH2O. Après 5 min, retirez la goutte et séchez à l’air.
- Répétez les 3 étapes 1.2.1.3-1.2.1.4.
- Remettre en suspension des lymphocytes T CD4+ naïfs et des lymphocytes CD4+ TH1 dans une solution saline tamponnée au phosphate (PBS) à une concentration de 2 × 106 cellules/mL.
REMARQUE : La concentration indiquée est conseillée spécifiquement pour les petites cellules telles que les lymphocytes T primaires humains. Pour les cellules adhérentes, un traitement de revêtement de verre n’est pas nécessaire. Au lieu de cela, procédez directement à la croissance des cellules sur la surface du verre à l’aide du milieu de culture cellulaire approprié. - Appliquez une goutte de 200 μL de la suspension de la cellule sur la lamelle de verre. Laisser les cellules s’ensemencer à température ambiante (RT) pendant 30 min ; Ensuite, retirez la goutte.
- Fixez les cellules avec du paraformaldéhyde à 3 % fraîchement filtré (solution PFA, tableau 1) pendant 10 min à RT.
- Lavez la lamelle en verre avec du TPBS (tableau 1) pendant 3 x 5 min à RT.
- Retirer le TPBS et ajouter la solution de perméabilisation (tableau 1) pendant 10 min à RT.
- Jeter la solution de perméabilisation et incuber l’échantillon dans une solution de stockage (tableau 1) de 1 h à toute la nuit (ON) à 4 °C.
REMARQUE : À ce stade, le protocole peut être arrêté en toute sécurité et les lamelles de verre peuvent être conservées dans une solution de stockage dans une plaque à 24 puits multiples pendant 3 à 4 semaines.
- Pour assurer une adhérence optimale des cellules, traiter les lamelles de verre (10 mm, épaisseur 1,5 H) avec une solution de revêtement (tableau 1) comme suit :
- Immunofluorescence
- (Facultatif) Retirez la lamelle de la plaque à 24 puits et congelez-la rapidement sur de la glace sèche pendant 30 s, décongelez-la à RT, puis lavez la lamelle en verre dans un puits prérempli de solution de stockage.
- (Facultatif) Répétez 3 fois l’étape 1.3.1.
- Laver dans une solution de perméabilisation pendant 5 min à RT. Ensuite, lavez pendant 2 x 5 min avec TPBS à RT.
- Incuber dans 0,1 N HCl pendant 12 min à RT.
- Effectuez deux lavages rapides en 1x PBS.
REMARQUE : Les conditions de perméabilisation cellulaire spécifiées, y compris les étapes de congélation et de décongélation et le traitement au HCl, sont optimales pour la coloration des composants nucléaires dans les cellules caractérisées par une chromatine densément emballée. Cependant, lorsqu’il s’agit de cellules caractérisées par une chromatine moins compactée, il est conseillé de réduire ou d’éviter ces étapes. De plus, pour cibler les composants cytoplasmiques, envisagez de réduire ou d’éliminer le traitement par HCl. - Incuber les cellules avec l’anticorps primaire (BRD4, 1:500 ou SUZ12, 1:100) dilué dans un tampon de dilution d’anticorps (tableau 1) (200 μL pour chaque lamelle de verre) à 4 °C.
REMARQUE : L’immunofluorescence peut être réalisée en multiplexant plus d’un anticorps primaire en fonction des besoins expérimentaux, des anticorps secondaires et des systèmes de détection. - Laver 3 x 5 min avec PBS-T (tableau 1) à RT en secouant doucement.
- Incuber les cellules avec l’anticorps secondaire dilué dans un tampon de dilution d’anticorps (200 μL pour chaque lamelle de verre) pendant 1 h à RT.
REMARQUE : Choisissez l’anticorps secondaire qui convient le mieux aux besoins expérimentaux et aux systèmes de détection disponibles. Assurez-vous que chaque anticorps secondaire cible l’espèce dont il est dérivé. De plus, choisissez des fluorophores compatibles avec les filtres et la source lumineuse du microscope. Il est crucial d’éviter le chevauchement spectral entre le fluorophore choisi et le spectre d’émission de la coloration nucléaire. - Laver 3 x 5 min avec PBS-T à RT en secouant doucement.
- Colorer avec 1 ng/mL de 4',6-diamidino-2-phénylindole (DAPI) dilué dans 1x PBS pendant 5 min à RT.
REMARQUE : Des colorants alternatifs peuvent être utilisés pour la coloration nucléaire tant qu’ils ne chevauchent pas le spectre d’émission des anticorps secondaires. - Effectuez plusieurs lavages rapides en 1x PBS.
- Montez la lamelle en verre sur une lame de microscopie avec un support de montage anti-évanouissement.
2. Acquisition d’images
REMARQUE : La durée d’acquisition de l’image dépend de l’instrument et des paramètres sélectionnés.
- Acquisition par microscope confocal
- Capturez des images 3D à l’aide d’un microscope confocal, en réglant une taille de pas de 0,25 μm en z et une taille de pixel de 0,1 à 0,2 μm.
REMARQUE : Pour obtenir une résolution optimale limitée par la diffraction de la lumière avec notre objectif à huile 63x 1,4 NA, nous avons défini la taille du sténopé à 0,8 AU, la moyenne de ligne 2x et la taille de l’image 1024 × 1024 pixels. Le laser d’excitation ainsi que la séquence d’acquisition de canal ont été délibérément sélectionnés pour éviter les interférences ou la diaphonie entre les fluorophores utilisés. Cependant, l’ajustement des paramètres doit être adapté aux caractéristiques spécifiques du microscope et de l’échantillon. Voir le tableau des matériaux pour les spécifications du microscope confocal utilisé dans ce protocole. - Acquérir un nombre constant de champs aléatoires pour englober environ 50 cellules par réplication biologique.
- Capturez des images 3D à l’aide d’un microscope confocal, en réglant une taille de pas de 0,25 μm en z et une taille de pixel de 0,1 à 0,2 μm.
3. Analyse d’images
- Installation du logiciel
- Téléchargez et installez la dernière version disponible de Fidji à partir de la page de téléchargement officielle de Fidji (https://imagej.net/software/fiji/downloads).
- Installez 3D Suite soit via le site de mise à jour Fiji, soit manuellement en suivant les instructions sur le site Web de 3D Suite (https://mcib3d.frama.io/3d-suite-imagej/#download).
- Installez GDSC (FindFoci) soit via le site de mise à jour des Fidji, soit manuellement en suivant les instructions sur le référentiel GitHub (https://github.com/aherbert/gdsc).
- Conversion TIFF
- Téléchargez le script « convert_to_TIFF.py » (Fichier supplémentaire 1).
- Faites glisser et déposez le script sur Fidji et exécutez le code.
- Dans le panneau qui s’affiche, accédez au chemin d’accès où l’expérience est stockée. Le sous-dossier contenant les fichiers TIFF convertis est créé dans le même dossier d’expérience.
- Initialisez les paramètres sur 3D Manager.
- Ouvrez le panneau d’options du Gestionnaire 3D en cliquant sur Plugins | 3DSuite | Options du Gestionnaire 3D.
- Dans la fenêtre d’options du Gestionnaire 3D, cochez les cases correspondant aux mesures suivantes : Volume (unité), Valeur de gris moyenne, Cadre de délimitation (pix), Valeur de gris de type standard, Centroïde (pix) et Centroïde (unité).
- Cochez les options suivantes : Exclure les objets sur les arêtes XY et Exclure les objets sur les arêtes Z et cliquez sur OK.
REMARQUE : Cette étape ne doit être effectuée qu’une seule fois pour configurer initialement les mesures qui seront stockées dans les fichiers d’informations spatiales et quantitatives. Les indicateurs sélectionnés sont essentiels pour assurer le bon fonctionnement du pipeline. Des mesures supplémentaires facultatives peuvent être incluses dans cette étape, comme décrit plus en détail dans la section Discussion.
- Définissez les paramètres sur l’interface graphique de FindFoci.
REMARQUE : Cette étape ne doit être effectuée qu’une seule fois pour configurer le pipeline semi-automatisé avec des paramètres optimaux, qui seront ensuite intégrés dans les scripts.- Ouvrez l’image de test. Dupliquez le canal protéique, y compris la pile (Ctrl + Maj + D), cochez la case hyperstack et spécifiez le numéro de canal approprié dans la case Canaux (c), puis renommez-le en conséquence.
- Lancez l’enregistreur de macros Fidji en accédant à Plugins | Macros | Enregistrer.
- Ouvrez le plugin FindFoci (cliquez sur Plugins | GDSC | FindFoci | FindFoci GUI) et sélectionnez l’image à analyser dans le menu déroulant « Image ».
- Réglez les paramètres comme suit (Figure 2A) : Flou gaussien = 1,5 ; méthode de fond = écart-type au-dessus de la moyenne ; paramètre d’arrière-plan = 9 ; méthode de recherche = fraction de pic - arrière-plan ; paramètre de recherche = 0,7 ; méthode du pic = relatif au-dessus du bruit de fond ; paramètre de crête = 0,2 ; taille minimale = 5 ; pics max = 1 000 000.
REMARQUE : Les paramètres indiqués ont été sélectionnés sur la base de nos études de cas et peuvent ne pas convenir à d’autres procédures de coloration. - Pour améliorer l’identification des foyers, ajustez les paramètres suivants :
- Le flou gaussien définit l’étendue du lissage pour mieux segmenter les foyers. Gardez-le près du diamètre des foyers (pixel).
- Le paramètre d’arrière-plan définit un seuil permettant de distinguer le signal d’arrière-plan du signal foci. Augmentez les valeurs pour imposer des seuils plus stricts.
- Le paramètre de recherche définit le pourcentage de fluorescence à partir du pic qui est inclus dans la reconnaissance du signal. Diminuez les valeurs pour inclure les zones plus éloignées du pic de fluorescence.
- Le paramètre de crête détermine le degré auquel deux crêtes de signal sont considérées comme continues ou séparées. La diminution de la valeur entraînera la séparation des pics.
- Max peaks spécifie le nombre maximal de foyers identifiables. Définissez des nombres élevés pour inclure tous les foyers de l’image.
- Exécutez FindFoci et copiez la chaîne qui apparaît dans la fenêtre de l’enregistreur (étape 3.4.2, Figure 2B) et qui contient les paramètres sélectionnés, à l’exclusion des guillemets. Pour plus d’informations sur les paramètres, reportez-vous à l’instruction 22 du manuel du plugin.
- Pipeline de quantification des protéines nucléaires
- Téléchargez le texte radiophonique « nuclear_prot_q.py » (Fichier supplémentaire 2).
- Faites glisser et déposez le script sur Fidji et cliquez sur Exécuter pour exécuter le code.
- Suivez les instructions de la boîte de dialogue affichée pour traiter les images.
- Canal du noyau : entrez le numéro correspondant au canal du DAPI (ou de toute coloration nucléaire).
- Flou gaussien du noyau : entrez la valeur de sigma nécessaire pour flouter l’image pour la segmentation.
REMARQUE : Maintenez ce paramètre plus près du diamètre du noyau ( c’est-à-dire 5-6 μm). Des valeurs sigma plus élevées sont indiquées pour les colorations non homogènes. - Canal protéique : entrez le numéro correspondant au canal de coloration d’intérêt.
- Paramètre FindFoci : collez la chaîne obtenue à partir de l’étape d’enregistrement de la macro dans le passage 3.4.6 (Figure 2B).
- (Facultatif) Contrôle de la qualité : vérifier la qualité des noyaux segmentés. Cela mettra le script en pause et permettra la vérification manuelle de chaque région nucléaire d’intérêt (ROI) générée.
- Sélectionnez un répertoire d’images : cliquez sur le bouton Parcourir pour accéder au dossier contenant les fichiers TIFF à analyser.
- Une fois toutes les cases compilées, cliquez sur OK pour poursuivre l’exécution.
- Si un noyau n’est pas correctement segmenté et ne répond pas aux exigences de qualité, supprimez-le ou modifiez-le comme indiqué à la figure 2C.
- Vérifiez la liste des ROI dans la fenêtre ROIManager3D ; si la liste est vide, sélectionnez la fenêtre de fusion et cliquez sur Quantifier 3D pour actualiser le gestionnaire. Fermez ensuite la table de résultats Quantifier 3D. Sélectionnez le canal des noyaux et cliquez sur Live-ROI sur ON.
- Sélectionnez le retour d’intérêt appartenant au même noyau et appuyez sur Fusionner ou appuyez sur Supprimer dans le cas de noyaux indésirables.
- Cliquez sur Sélectionner tout et poursuivez l’analyse en cliquant sur OK dans la fenêtre de vérification du masque .
- Pour les résultats, consultez le dossier Quantification dans le chemin d’accès au fichier indiqué à l’étape 3.5.3.6, contenant un fichier txt avec les enregistrements du paramètre utilisé pour l’analyse.
- Pipeline sur Google Colab
- Téléchargez le cahier « final_nuclear_protein_metrics.ipynb » (Fichier supplémentaire 3).
- Ouvrez le carnet sur Google Colab (https://colab.research.google.com/).
- Téléchargez tous les dossiers contenant les fichiers .csv de chaque champ d’image dans le dossier de votre choix dans Google Drive.
- Indiquez dans le notebook le chemin d’accès du dossier où sont stockés les sous-dossiers de résultats et exécutez toutes les cellules. Une fois la compilation du code terminée, le fichier final de la feuille de calcul contenant toutes les données compilées est créé dans le même dossier que celui où les fichiers .csv ont été téléchargés.
Résultats
Le protocole décrit dans cette méthode facilite la visualisation et la quantification des altérations de la coloration des protéines nucléaires dans les cellules T primaires humaines, et il peut être personnalisé pour divers types de cellules et cibles protéiques. À titre d’études de cas, nous avons mené et analysé la coloration de BRD4 et SUZ12 dans des cellules CD4+ naïves et TH1.
BRD4 présente un motif de coloration bien pointillé dans les cellules CD4+ TH1 naïves au repos et différenciées, ce qui permet l’utilisation de paramètres conçus pour l’identification des foyers pour les deux types de cellules (Figure 3A). Notre pipeline permet de quantifier divers paramètres, notamment le nombre, le volume, le signal de fluorescence (exprimé en intensité de fluorescence moyenne (MFI)), la couverture et la disposition spatiale des foyers BRD4 dans le noyau (Figure 3B-F). De plus, nous avons introduit un calcul automatique pour déterminer le pourcentage des foyers dans le volume nucléaire, facilitant ainsi la comparaison entre des cellules de différentes tailles nucléaires (Figure 3E).
Enfin, nous avons fourni la possibilité de cartographier les distances des foyers par rapport au centre nucléaire, ce qui aide à comprendre leur positionnement nucléaire (Figure 3F). Les résultats obtenus à partir de la quantification mettent en évidence des altérations notables des foyers BRD4 entre les lymphocytes TH1 et CD4+ quiescents, englobant une augmentation du nombre, de la taille, de la luminosité et du volume des foyers, ainsi que des différences dans leur distribution et leur localisation. Ces résultats sont cohérents avec l’activité transcriptionnelle accrue des lymphocytes T activés/proliférants par rapport à leurs homologues quiescents28.
En revanche, SUZ12 ne participe pas à la formation de condensats. Dans notre étude, nous avons observé une disparité significative dans le motif de coloration de SUZ12 entre les deux états cellulaires : un motif ponctué chez les lymphocytes T naïfs passe à un motif diffus dans les cellules TH1 CD4+ (Figure 4A). Ce changement significatif dans le comportement des protéines entrave la comparabilité des caractéristiques des foyers en utilisant des paramètres identiques, car l’identification des points dans les cellules TH1 CD4+ est largement infructueuse (Figure 4A). Bien qu’il soit généralement recommandé de maintenir la cohérence des paramètres d’acquisition et d’analyse lors de la comparaison de la coloration par immunofluorescence dans des conditions cellulaires distinctes, des ajustements deviennent nécessaires en cas de changements substantiels dans la cible d’intérêt.
Dans ce scénario spécifique, comme illustré à la figure 4B, malgré l’expérimentation de diverses combinaisons de paramètres, y compris l’ajustement aux paramètres d’arrière-plan, au flou gaussien et aux paramètres de recherche (comme suggéré à l’étape 3.4.5 du protocole), FindFoci rencontre des difficultés à distinguer les vrais pics de signal des pics non significatifs en raison de la nature de distribution inhérente de la protéine. De plus, nous soulignons que les mesures conventionnelles comme l’IFM nucléaire peuvent être trompeuses (Figure 4C), ne tenant pas compte des altérations de la distribution du signal.
Par conséquent, pour mesurer de tels changements, nous suggérons l’utilisation du coefficient de variation, une métrique que nous avons introduite dans ce protocole pour caractériser la diffusion des protéines, appelée coefficient d’inhomogénéité (CI). L’IC capture avec précision la diffusion des foyers SUZ12 dans les cellules CD4+ TH1 sans forcer leur identification avec des approches de segmentation incorrectes (voir Figure 4D). Enfin, en utilisant le DAPI comme contrôle, qui reste uniformément distribué dans les deux conditions cellulaires, nous validons davantage l’efficacité de ce paramètre (Figure 4E).
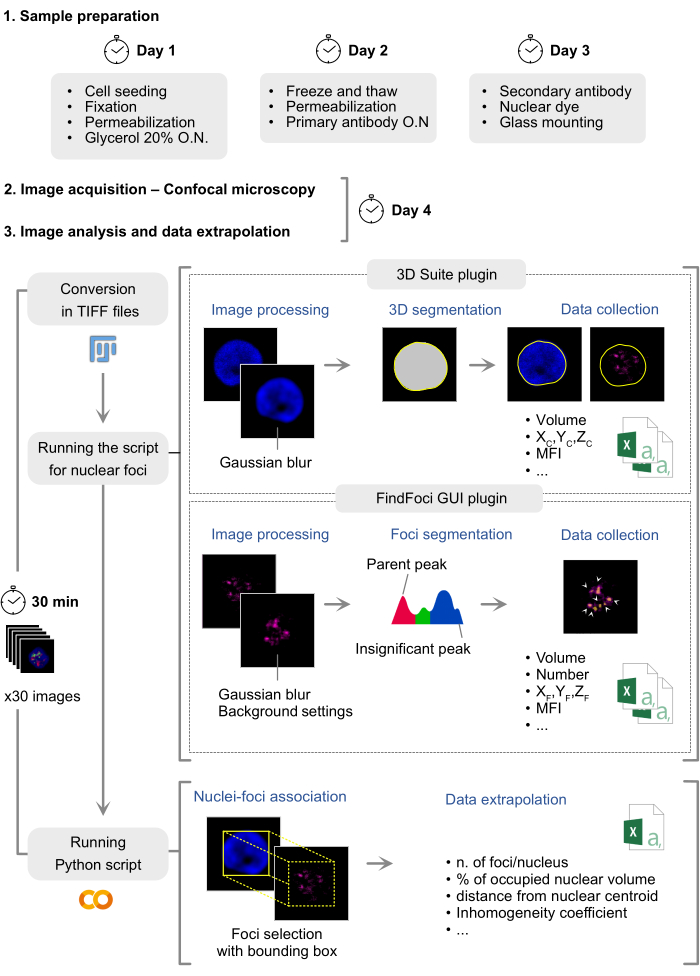
Figure 1 : Représentation schématique de la préparation de l’immunofluorescence et de l’analyse des données. Le protocole de coloration et d’analyse de l’immunofluorescence des protéines nucléaires se compose de trois phases principales : la préparation de l’échantillon, l’acquisition d’images et l’analyse, s’étendant sur environ 4 jours ouvrables. Le script utilise deux plugins, 3D Suite pour effectuer la segmentation des noyaux et FindFoci pour traiter le canal protéique. Les informations spatiales et quantitatives concernant les foyers et le noyau sont rassemblées dans des fichiers csv séparés. Un script exécutable sur Google Colab relie les mesures des foyers aux noyaux correspondants à l’aide de la boîte englobante du noyau et calcule les mesures dérivées en fonction des données collectées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
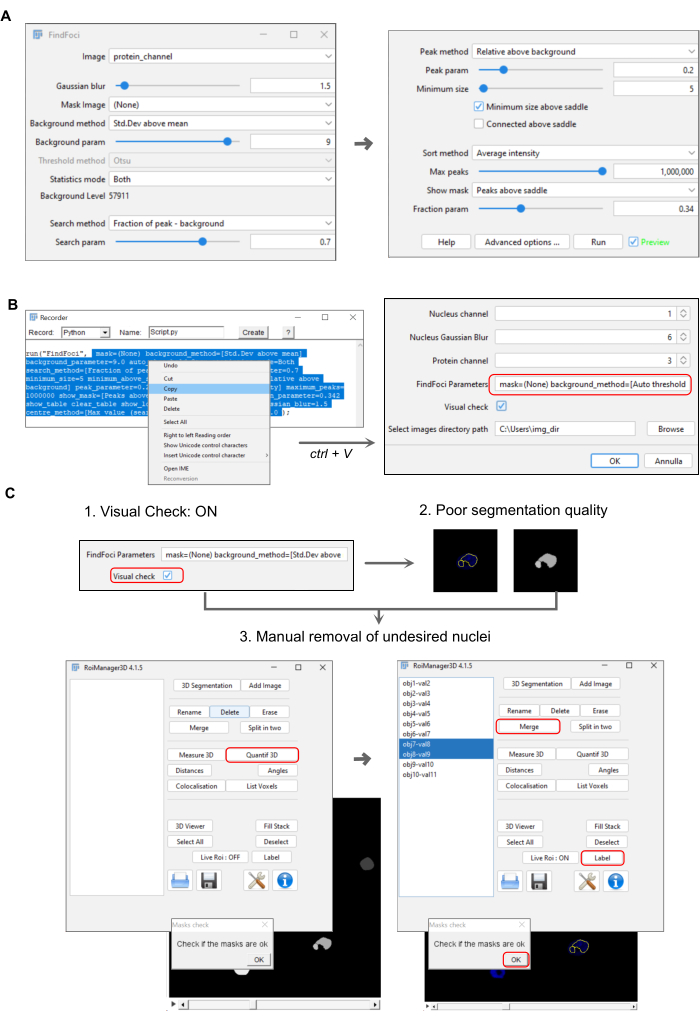
Figure 2 : Représentation des paramètres du pipeline de quantification des protéines nucléaires. (A) Fenêtre contextuelle FindFoci illustrant les paramètres d’identification des foyers (section 3.4.3 du protocole). (B) Fenêtre contextuelle représentant la macro enregistrant les paramètres sélectionnés de FindFoci. La chaîne copiée est collée dans la fenêtre de démarrage (étapes de protocole 3.4.6 à 3.5.3.4). (C) Panneau montrant l’étape de contrôle de la qualité de la segmentation des noyaux pour l’élimination ou la modification manuelle des régions nucléaires d’intérêt à l’aide du gestionnaire 3D (étapes de protocole 3.5.5.1 à 3.5.5.3). Veuillez cliquer ici pour voir une version agrandie de cette figure.
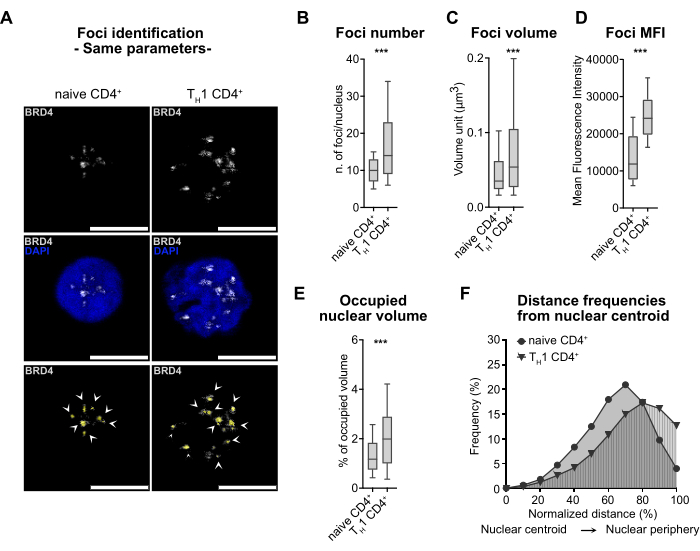
Figure 3 : Comparaisons entre des affections cellulaires distinctes basées sur l’analyse des foyers BRD4. (A) Images représentatives de la coloration par immunofluorescence de BRD4 (gris) dans les lymphocytes T CD4+ primaires naïfs humains et les lymphocytes TH1 CD4+. Les noyaux sont contre-colorés avec du DAPI (bleu). Grossissement d’origine 63x ; barre d’échelle = 5 μm. En bas, les foyers BRD4, identifiés par le pipeline, sont marqués par des pointes de flèches blanches et contrés en jaune (en bas). (B) Boîte à moustaches représentant le nombre/noyau de foyers BRD4 dans les cellules CD4+ naïves et TH1 (n = 2 individus). (C) Représentation en boîte à moustaches du volume (μm3) des foyers BRD4 dans les cellules CD4+ naïves et TH1 (n = 2 individus). (D) Représentation en boîte à moustaches des foyers MFI des foyers BRD4 dans les cellules CD4+ naïves et TH1 (n = 2 individus). (E) Boîte à moustaches représentant le pourcentage du volume nucléaire occupé par les foyers BRD4 par rapport au volume total du noyau (n = 2 individus). (F) Représentation des fréquences de distance des foyers BRD4 entre le centroïde nucléaire et la périphérie nucléaire (n = 2 individus). Veuillez cliquer ici pour voir une version agrandie de cette figure.
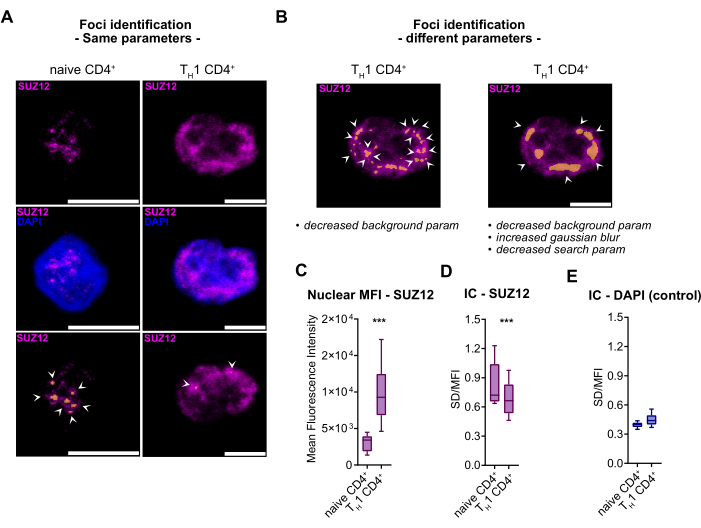
Figure 4 : Coefficient d’inhomogénéité, une métrique permettant de quantifier la transition d’un motif ponctué à un motif dispersé dans les signaux d’immunofluorescence SUZ12. (A) Images représentatives de la coloration par immunofluorescence pour SUZ12 (magenta) dans les lymphocytes T CD4+ primaires naïfs humains et les lymphocytes TH1 CD4+ Cellules. Les noyaux sont contre-colorés avec du DAPI (bleu). Grossissement d’origine 63x ; barre d’échelle = 5 μm. En bas, les foyers de SUZ12 identifiés par la canalisation sont marqués d’une pointe de flèche blanche et contrés en orange (B). Exemples d’identification erronée des foyers dans les cellules TH1 CD4+ illustrés par des flèches blanches et un masque orange avec deux réglages de paramètres différents. (C) Représentation en boîte à moustaches de l’IFM nucléaire de SUZ12 dans des cellules CD4+ naïves et TH1 (n = 2 individus). (D) Représentation en boîte à moustaches de SUZ12 IC dans les cellules CD4+ naïves et TH1 (n = 2 individus). (E) Représentation en boîte à moustaches de DAPI IC dans les cellules CD4+ naïves et TH1 (n = 2 individus). Abréviations : DAPI = 4',6-diamidino-2-phénylindole ; MFI = intensité moyenne de fluorescence ; IC = coefficient d’inhomogénéité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Solution | Composition | Commentaires/Description | |
| TH1 moyen | RPMI avec GlutaMAX-I, 10 % (v/v) de sérum de veau fœtal (FBS), 1 % (v/v) d’acides aminés non essentiels, 1 mM de pyruvate de sodium, 50 UI/mL de pénicilline, 50 μg/mL de streptomycine, 20 UI/mL d’IL-2 recombinante, 10 ng/mL d’IL-12 recombinante, 2 mg/mL d’anti-IL-4 neutralisant. | Étape 1.1.4. | |
| Solution de revêtement | 0,1% de poly-L-lysine dans ddH2O | Étape 1.2.1. | |
| PBS-T | 1x PBS/0,1% TWEEN 20 pH 7,0 | Étape 1.3. | |
| Solution PFA pour la fixation | 3 % de paraformaldéhyde (PFA) dilué dans 0,1 % de PBS-TWEEN | Étape 1.2.4. | |
| Le | 0,05% Triton X-100 dilué dans 1x PBS | Étape 1.2., 1.3. | |
| Solution de perméabilisation | 0,5 % de TPBS dilué dans 1x PBS | Étape 1.2., 1.3. | |
| Solution de stockage | 20% de glycérol/1x PBS | Étape 1.2.7., 1.3.1. | |
| Tampon de dilution d’anticorps | 0,1 % PBS-TWEEN/2 % sérum de chèvre/1 % BSA | Étapes 1.3.6., 1.3.8. | |
Tableau 1 : Composition des supports et des tampons utilisés dans ce protocole.
Dossier supplémentaire 1 : « convert_to_TIFF.py ». Ce fichier contient le script permettant de convertir n’importe quel fichier image (par exemple, ND2, LIF, etc.) en fichiers TIFF nécessaires à des processus de quantification précis. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 2 : « nuclear_prot_q.py ». Ce fichier contient le script qui permet de mesurer et de quantifier des images contenant au moins deux canaux (coloration nucléique, coloration des protéines nucléaires). Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : « final_nuclear_protein metrics.ipynb ». Ce fichier contient un bloc-notes Jupyter qui extrait et compile un résumé de tous les paramètres pertinents dans un seul fichier Excel, en utilisant la sortie « nuclear_prot_q.py » comme entrée. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans cette étude, nous présentons une méthode pour réaliser des expériences d’immunofluorescence sur des protéines nucléaires dans des lymphocytes T humains. Cette méthode offre une flexibilité d’utilisation avec différents types de cellules grâce à des modifications mineures dans les étapes de fixation et de perméabilisation, comme décrit précédemment30,31.
Notre flux de travail d’imagerie s’appuie sur des techniques établies décrites dans la littérature, en particulier FindFoci et 3D Suite22,23. Contrairement aux publications précédentes, nous fournissons un code Jython exécutable intégré aux Fidji pour un pipeline semi-automatisé, permettant un suivi méticuleux des paramètres et des contrôles visuels de la qualité. Notamment, nous avons introduit la capacité facultative de réguler la qualité de la segmentation des noyaux dans la section 3.5.3.5 du protocole et mis en œuvre une étape corrective dans la section 3.5.5 du protocole. Avec l’exécutable de script sur Google Colab, les utilisateurs peuvent récupérer des mesures complètes et choisir celles qui conviennent le mieux à leurs scénarios biologiques spécifiques.
Le pipeline d’analyse fourni est écrit en Jython (Java + Python) et nécessite l’installation de Fiji pour l’exécution, ainsi que deux plugins distincts : 3D Suite et FindFoci (Figure 1). Le pipeline commence par la conversion de l’image au format TIFF et le prétraitement du canal contenant les noyaux avec un filtre de flou gaussien pour améliorer la segmentation des noyaux. Nous demandons à 3D Suite de capturer à la fois i) des informations spatiales, telles que le volume nucléaire, le centroïde et les coordonnées de la boîte englobante (représentant la plus petite boîte entourant l’objet et utilisée pour associer des foyers au noyau droit), et ii) des informations quantitatives, y compris l’IFM et l’écart-type du signal. Par la suite, le pipeline utilise FindFoci pour identifier les maxima locaux dans le signal des foyers, éliminant ainsi la dépendance aux méthodes de seuillage global (qui reposent souvent sur la discrétion de l’utilisateur et peuvent manquer de précision), et enregistre à la fois des informations spatiales et quantitatives relatives aux foyers. Le dossier de sortie contiendra donc deux fichiers CSV par canal, englobant les métriques spatiales (« M_.csv ») et quantitatives (« Q_.csv »).
Le fichier final, contenant toutes les mesures collectées et dérivées, est généré en exécutant un script Python sur Google Colab. Ce fichier comprend des paramètres tels que le nombre de foyers par noyau, l’IFM des foyers, le coefficient d’inhomogénéité (IC), l’IFM nucléaire, la distance des foyers par rapport au centroïde nucléaire et le pourcentage du volume nucléaire occupé. Nous estimons un temps de traitement d’environ 1 minute par image, y compris les étapes de contrôle visuel de la qualité. Ce temps de traitement peut être encore accéléré grâce à l’optimisation des paramètres d’immunofluorescence et des paramètres d’analyse.
Le pipeline calcule les paramètres des protéines nucléaires présentant à la fois des motifs bien pointillés et diffus, ce qui permet aux utilisateurs de sélectionner le paramètre le plus approprié. Nous recommandons d’utiliser le coefficient d’inhomogénéité pour décrire la dissolution des foyers protéiques.
Une étape essentielle pour assurer une identification correcte des foyers implique le réglage de paramètres permettant de distinguer les pics de signal réels du bruit de fond. Il est crucial d’effectuer des tests approfondis sur des images aléatoires avant d’exécuter le pipeline. Bien que notre étude se concentre principalement sur un seul canal, le pipeline soutient l’introduction de diverses combinaisons de colorants ciblant différentes protéines dans les expériences d’immunofluorescence. Il ne prend pas en charge l’analyse simultanée de plusieurs canaux et doit être exécuté séparément pour chaque canal. De plus, nous avons choisi de présenter des mesures spécifiques dans ce protocole ; Cependant, des paramètres supplémentaires, tels que l’intensité intégrée pour évaluer les changements absolus du signal de fluorescence ou des paramètres physiques tels que la circularité et la compacité, peuvent également être intégrés au pipeline à la section 3.3.2. L’inclusion de métriques qui ne sont pas actuellement intégrées dans ce pipeline nécessite une maîtrise de la programmation Python pour les intégrer dans le fichier de métriques final ; Dans le cas contraire, ils seront automatiquement inclus dans les fichiers d’informations spatiales (« M_.csv ») et quantitatives (« Q_.csv »).
Le calendrier de ce pipeline dépend de facteurs tels que la taille et la quantité d’images, le nombre de canaux par analyse et les paramètres du microscope. De plus, les performances de la machine influencent le temps d’exécution, les étapes visuelles pouvant ralentir le processus.
Il convient de noter que si la microscopie confocale offre des informations précieuses, elle reste une technologie limitée par la diffraction qui peut limiter la précision des structures nucléaires observées ou des foyers protéiques. Pour renforcer la robustesse de l’analyse, nous suggérons d’augmenter le nombre de noyaux et d’échantillons biologiques examinés. À mesure que le nombre de noyaux analysés augmente, tout changement notable dans la morphologie nucléaire ou la distribution des protéines devient plus évident, quelles que soient les limites de résolution.
En termes d’applications futures potentielles, cette méthode fait preuve de polyvalence et d’adaptabilité à divers types de cellules et de protocoles de coloration. Par exemple, il peut être facilement utilisé dans les expériences de radiobiologie, où la quantification précise des foyers induits par les rayonnements ionisants (IRIF) sert d’indicateur direct des dommages à l’ADN32. Bien que notre objectif principal soit l’analyse nucléaire, le protocole peut être facilement personnalisé en incorporant un colorant cytoplasmique et en le remplaçant par le canal nucléaire dans le script. De plus, l’analyse peut s’intégrer de manière transparente dans d’autres méthodologies expérimentales telles que l’ADN et l’ARN FISH30,31, en exploitant les paramètres par défaut pour l’analyse de la topologie nucléaire.
Déclarations de divulgation
R.V. a une collaboration scientifique avec la startup T-One Therapeutics Srl ; B.B. et F.M. sont cofondateurs de la startup T-One Therapeutics Srl ; E.P. est actuellement employé par T-One Therapeutics Srl ; Tous les autres auteurs déclarent qu’ils n’ont pas d’intérêts concurrents.
Remerciements
Nous remercions l’assistance scientifique et technique de l’installation d’imagerie de l’INGM, en particulier C. Cordiglieri et A. Fasciani, et du centre de tri AGM FACS en particulier M.C Crosti (Istituto Nazionale di Genetica Molecolare 'Romeo ed Enrica Invernizzi' (INGM), Milan, Italie). Nous remercions M. Giannaccari pour son soutien technique en informatique. Ce travail a été financé par les subventions suivantes : Fondazione Cariplo (Bando Giovani, subvention n° 2018-0321) et Fondazione AIRC (subvention n° MFAG 29165) à F.M. Ricerca Finalizzata, (subvention n° GR-2018-12365280), Fondazione AIRC (subvention n° 2022 27066), Fondazione Cariplo (subvention n° 2019-3416), Fondazione Regionale per la Ricerca Biomedica (FRRB CP2_12/2018,), Piano Nazionale di Ripresa e Resilienza (PNRR) (subvention n° G43C22002620007) et Progetti di Rilevante Interesse Nazionale (PRIN) (subvention n° 2022PKF9S) à B. B.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL Safe-Lock Tubes | Eppendord | #0030121503 | Protocol section 1 |
| 10 mL Serological pipettes | VWR | #612-3700 | Protocol section 1 |
| 20 µL barrier pipette tip | Thermo Scientific | #2149P-HR | Protocol section 1 |
| 50 mL Polypropylene Conical Tube | Falcon | #352070 | Protocol section 1 |
| 200 µL barrier pipette tip | Thermo Scientific | #2069-HR | Protocol section 1 |
| antifade solution - ProlongGlass - mountingmedia | Invitrogen | #P36984 | Step 1.3.12 |
| BSA (Bovine Serum Albumin) | Sigma | #A7030 | Step 1.3.6., 1.3.8. |
| CD4+ T Cell Isolation Kit | Miltenyi Biotec | #130-096-533 | Step 1.1.2. |
| DAPI (4,6-diamidino-2-phenylindole) | Invitrogen | Cat#D1306 | Step 1.3.10. |
| Dry ice | Step 1.3.1. | ||
| Dynabeads Human T-activator anti-CD3/anti-CD28 bead | Life Technologies | #1131D | magnetic beads step 1.1.4. |
| EtOH | Carlo Erba | #4146320 | Step 1.2.1.1. |
| FACSAria SORP | BD Bioscences | Step 1.1.3. Equipped with BD FACSDiva Software version 8.0.3 | |
| FBS (Fetal Bovine Serum) | Life Technologies | #10270106 | Step 1.1.4 |
| FICOLL PAQUE PLUS | Euroclone | GEH17144003F32 | Step 1.1.1. |
| FIJI Version 2.14.0 | - | - | Protocol section 3 |
| Glass coverslip (10 mm, thickness 1.5 H) | Electron Microscopy Sciences | #72298-13 | Step 1.2.1. |
| Glycerol | Sigma | #G5516 | Step 1.2.7-1.3.1. |
| Goat anti-Rabbit AF568 secondary antibody | Invitrogen | A11036 | Step 1.3.8. |
| HCl | Sigma | #320331 | Step 1.3.4. |
| human neutralizing anti-IL-4 | Miltenyi Biotec | Cat#130-095-753 | Step 1.1.4. |
| human recombinant IL-12 | Miltenyi Biotec | Cat#130-096-704 | Step 1.1.4. |
| human recombinant IL-2 | Miltenyi Biotec | Cat#130-097-744 | Step 1.1.4. |
| Leica TCS SP5 Confocal microscope | Leica Microsystems | - | Protocol section 2, Equipped with HCX PL APO 63x, 1.40 NA oil immersion objective, with an additional 3x zoom. Pinhole size : 0.8 AU. Line average 2×. Frame size 1024×1024 pixel. |
| MEM Non-Essential Amino Acids Solution | Life Technologies | #11140035 | Step 1.1.4. |
| Microscope Slides | VWR | #631-1552 | Step 1.3.12. |
| Mouse monoclonal anti-Human CD4 APC-Cy7 (RPA-T4 clone) | BD Bioscience | #557871 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RA PECy5 (5H9 clone) | BD Bioscience | #552888 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RO APC (UCHL1 clone) | Miltenyi Biotec | #130-113-546 | Step 1.1.3. |
| Multiwell 24 well | Falcon | #353047 | Protocol section 1 |
| Normal Goat Serum | Invitrogen | PCN5000 | Step 1.3.6., 1.3.8. |
| PBS | Life Technologies | #14190094 | Protocol section 1 |
| Penicillin/Streptomycin solution | Life Technologies | #15070063 | Step 1.1.4. |
| PFA | Sigma | #P6148 | Step 1.2.4. |
| poly-L-lysine | Sigma | #P8920 | 1.2.1. |
| Primary antibody - BRD4 | Abcam | #ab128874 | Step 1.3.6. |
| Primary antibody - SUZ12 | Cel Signalling | mAb #3737 | Step 1.3.6. |
| RPMI 1640 W/GLUTAMAX-I | Life Technologies | #61870010 | Step 1.1.4. |
| Sodium Pyruvate | Life Technologies | #11360039 | Step 1.1.4. |
| Triton X-100 | Sigma | #T8787 | Step 1.2., 1.3. |
| TWEEN 20 | Sigma | #P9416 | Step 1.3. |
| Tweezers | - | - | Protocol section 1 |
Références
- Aboelnour, E., Bonev, B. Decoding the organization, dynamics, and function of the 4D genome. Dev Cell. 56 (11), 1562-1573 (2021).
- Erdel, F., Rippe, K. Formation of chromatin subcompartments by phase separation. Biophys J. 114 (10), 2262-2270 (2018).
- Henninger, J. E., et al. RNA-mediated feedback control of transcriptional condensates. Cell. 184 (1), 207-225 (2021).
- Guo, Y. E., et al. Pol II phosphorylation regulates a switch between transcriptional and splicing condensates. Nature. 572 (7770), 543-548 (2019).
- Bhat, P., et al. 3D genome organization around nuclear speckles drives mRNA splicing efficiency. bioRxiv. , (2023).
- Spector, D. L., Lamond, A. I. Nuclear speckles. Cold Spring Harb Perspect Biol. 3 (2), a000646 (2011).
- Kilic, S., et al. Phase separation of 53BP1 determines liquid-like behavior of DNA repair compartments. EMBO J. 38 (16), e101379 (2019).
- Parker, M. W., et al. A new class of disordered elements controls DNA replication through initiator self-assembly. Elife. 8, e48562 (2019).
- Feric, M., et al. Coexisting liquid phases underlie nucleolar subcompartments. Cell. 165 (7), 1686-1697 (2016).
- Yoneda, M., Nakagawa, T., Hattori, N., Ito, T. The nucleolus from a liquid droplet perspective. J Biochem. 170 (2), 153-162 (2021).
- Alberti, S., Carra, S. Nucleolus: A liquid droplet compartment for misbehaving proteins. Curr Biol. 29 (19), R930-R932 (2019).
- Strom, A. R., et al. Phase separation drives heterochromatin domain formation. Nature. 547 (7662), 241-245 (2017).
- Sanulli, S., et al. HP1 reshapes nucleosome core to promote phase separation of heterochromatin. Nature. 575 (7782), 390-394 (2019).
- Hyman, A. A., Weber, C. A., Julicher, F. Liquid-liquid phase separation in biology. Annu Rev Cell Dev Biol. 30, 39-58 (2014).
- Feric, M., Misteli, T. Phase separation in genome organization across evolution. Trends Cell Biol. 31 (8), 671-685 (2021).
- Mitrea, D. M., et al. Methods for physical characterization of phase-separated bodies and membrane-less organelles. J Mol Biol. 430 (23), 4773-4805 (2018).
- Fetter, J., et al. Endogenous gene tagging with fluorescent proteins. Methods Mol Biol. 1239, 231-240 (2015).
- Alberti, S., Gladfelter, A., Mittag, T. Considerations and challenges in studying liquid-liquid phase separation and biomolecular condensates. Cell. 176 (3), 419-434 (2019).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. J Cell Sci. 124 (10), 1607-1611 (2011).
- Scalisi, S., Ahmad, A., D'Annunzio, S., Rousseau, D., Zippo, A. Quantitative analysis of PcG-associated condensates by stochastic optical reconstruction microscopy (STORM). Methods Mol Biol. 2655, 183-200 (2023).
- Shihan, M. H., Novo, S. G., Le Marchand, S. J., Wang, Y., Duncan, M. K. A simple method for quantitating confocal fluorescent images. Biochem Biophys Rep. 25, 100916 (2021).
- Herbert, A. D., Carr, A. M., Hoffmann, E. FindFoci: a focus detection algorithm with automated parameter training that closely matches human assignments, reduces human inconsistencies and increases speed of analysis. PLoS One. 9 (12), e114749 (2014).
- Ollion, J., Cochennec, J., Loll, F., Escude, C., Boudier, T. TANGO: a generic tool for high-throughput 3D image analysis for studying nuclear organization. Bioinformatics. 29 (14), 1840-1841 (2013).
- Sabari, B. R., et al. Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361 (6400), 3958 (2018).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19 (4), 523-534 (2005).
- Pasini, D., Bracken, A. P., Jensen, M. R., Denchi, E. L., Helin, K. Suz12 is essential for mouse development and for EZH2 histone methyltransferase activity. EMBO J. 23 (20), 4061-4071 (2004).
- Margueron, R., Reinberg, D. The Polycomb complex PRC2 and its mark in life. Nature. 469 (7330), 343-349 (2011).
- Peng, Z., et al. Brd4 regulates the homeostasis of CD8(+) T-lymphocytes and their proliferation in response to antigen stimulation. Front Immunol. 12, 728082 (2021).
- Marasca, F., et al. LINE1 are spliced in non-canonical transcript variants to regulate T cell quiescence and exhaustion. Nat Genet. 54 (2), 180-193 (2022).
- Marasca, F., Cortesi, A., Bodega, B. 3D COMBO chrRNA-DNA-ImmunoFISH. Methods Mol Biol. 2157, 281-297 (2021).
- Marasca, F., Cortesi, A., Manganaro, L., Bodega, B. 3D Multicolor DNA FISH tool to study nuclear architecture in human primary cells. J Vis Exp. (155), (2020).
- Jakob, B., Splinter, J., Durante, M., Taucher-Scholz, G. Live cell microscopy analysis of radiation-induced DNA double-strand break motion. Proc Natl Acad Sci U S A. 106 (9), 3172-3177 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon