Method Article
Eine vielseitige Pipeline für die Analyse dynamischer Veränderungen in Kernkörpern in einer Vielzahl von Zelltypen
In diesem Artikel
Zusammenfassung
Diese Methode beschreibt ein Immunfluoreszenzprotokoll und eine Quantifizierungspipeline zur Bewertung der Proteinverteilung mit unterschiedlichen Kernorganisationsmustern in humanen T-Lymphozyten. Dieses Protokoll bietet eine Schritt-für-Schritt-Anleitung, beginnend bei der Probenvorbereitung über die Durchführung halbautomatischer Analysen in Fidschi bis hin zur Datenverarbeitung durch ein Google Colab-Notebook.
Zusammenfassung
Verschiedene nukleäre Prozesse, wie z. B. die transkriptionelle Kontrolle, finden in diskreten Strukturen statt, die als Foki bekannt sind und durch die Immunfluoreszenztechnik erkennbar sind. Die Untersuchung der Dynamik dieser Herde unter verschiedenen zellulären Bedingungen mittels Mikroskopie liefert wertvolle Einblicke in die molekularen Mechanismen, die die zelluläre Identität und Funktionen steuern. Die Durchführung von Immunfluoreszenz-Assays über verschiedene Zelltypen hinweg und die Beurteilung von Veränderungen in der Assemblierung, Diffusion und Verteilung dieser Herde stellen jedoch zahlreiche Herausforderungen dar. Diese Herausforderungen umfassen die Komplexität der Probenvorbereitung, die Bestimmung von Parametern für die Analyse von Bilddaten und das Management großer Datenmengen. Darüber hinaus sind bestehende Bildgebungs-Workflows oft auf erfahrene Benutzer zugeschnitten, wodurch der Zugang für ein breiteres Publikum eingeschränkt wird.
In dieser Studie stellen wir ein optimiertes Immunfluoreszenzprotokoll vor, das auf die Untersuchung von Kernproteinen in verschiedenen humanen primären T-Zelltypen zugeschnitten ist und für jedes Protein von Interesse und Zelltyp angepasst werden kann. Darüber hinaus stellen wir eine Methode zur unvoreingenommenen Quantifizierung der Proteinfärbung vor, unabhängig davon, ob sie unterschiedliche Foki bilden oder eine diffuse Kernverteilung aufweisen.
Die von uns vorgeschlagene Methode bietet einen umfassenden Leitfaden, von der zellulären Färbung bis zur Analyse, und nutzt dabei eine halbautomatische Pipeline, die in Jython entwickelt wurde und auf Fidschi ausgeführt werden kann. Darüber hinaus stellen wir ein benutzerfreundliches Python-Skript zur Verfügung, um die Datenverwaltung zu rationalisieren, das auf einem Google Colab-Notebook öffentlich zugänglich ist. Unser Ansatz hat sich als wirksam erwiesen, indem er hochinformative Immunfluoreszenzanalysen für Proteine mit unterschiedlichen Mustern der Kernorganisation in verschiedenen Kontexten liefert.
Einleitung
Die Organisation des eukaryotischen Genoms wird durch mehrere Schichten epigenetischer Modifikationen1 gesteuert, die mehrere Kernfunktionen koordinieren, die in spezialisierten Kompartimenten, den sogenannten Kernkörpern oder Kondensaten, ablaufen können2. Innerhalb dieser Strukturen finden Prozesse wie die Transkriptionsinitiierung3, die RNA-Prozessierung 4,5,6, die DNA-Reparatur 7,8, die Ribosomenbiogenese 9,10,11 und die Heterochromatinregulation12,13 statt. Die Regulation von Kernkörpern passt sich sowohl in räumlicher als auch in zeitlicher Hinsicht an die zellulären Anforderungen an, geleitet von den Prinzipien der Phasentrennung14,15. Folglich fungieren diese Körper als vergängliche Fabriken, in denen funktionale Komponenten zusammen- und zerlegt werden und sich in Größe und räumlicher Verteilung ändern. Daher bietet das Verständnis der Eigenschaften von Kernproteinen durch Mikroskopie, einschließlich ihrer Neigung zur Körperbildung und ihrer räumlichen Anordnung unter verschiedenen zellulären Bedingungen, wertvolle Einblicke in ihre funktionelle Rolle. Die Fluoreszenzmikroskopie ist eine weit verbreitete Methode zur Untersuchung von Kernproteinen, die ihren Nachweis durch fluoreszierende Antikörper oder die direkte Expression von Zielen mit einem fluoreszierenden Proteinreporter ermöglicht16,17.
In diesem Zusammenhang erscheinen Kernkörper als helle Brennpunkte oder Puncta mit einem bemerkenswerten Grad an Sphärizität, wodurch sie leicht von der umgebenden Umgebung unterscheidbar sind16,18. Superauflösungstechniken wie STORM und PALM ermöglichen durch ihre verbesserte Auflösung (bis zu 10 nm)19 eine genauere Charakterisierung der Struktur und Zusammensetzung spezifischer Kondensate20. Ihre Zugänglichkeit ist jedoch durch die Kosten für die Ausrüstung und die für die Datenanalyse erforderlichen Fachkenntnisse eingeschränkt. Daher ist die konfokale Mikroskopie aufgrund ihres günstigen Gleichgewichts zwischen Auflösung und breiterer Verwendung nach wie vor beliebt. Diese Popularität wird durch die inhärente Entfernung von unscharfem Licht erleichtert, wodurch der Bedarf an umfangreichen Nachbearbeitungsverfahren für eine genaue Segmentierung verringert wird, durch seine weit verbreitete Verfügbarkeit in Forschungsinstituten, durch seine effektive Erfassungszeit und durch eine in der Regel effiziente Probenvorbereitung. Die genaue Messung der Proteinverteilung, -assemblierung oder -diffusion mit Immunfluoreszenz-Assays unter verschiedenen zellulären Bedingungen stellt jedoch eine Herausforderung dar, da vielen bestehenden Methoden eine Anleitung zur Auswahl geeigneter Parameter für Proteine mit unterschiedlichen Verteilungsmustern fehlt21. Darüber hinaus kann der Umgang mit dem daraus resultierenden großen Datenvolumen für Benutzer mit begrenzter Erfahrung in der Datenanalyse entmutigend sein, was die biologische Bedeutung der Ergebnisse beeinträchtigen kann.
Um diesen Herausforderungen zu begegnen, stellen wir ein detailliertes Schritt-für-Schritt-Protokoll für die Immunfluoreszenzvorbereitung und Datenanalyse vor, das darauf abzielt, eine unvoreingenommene Methode zur Quantifizierung der Proteinfärbung mit verschiedenen Organisationsmustern bereitzustellen (Abbildung 1). Diese halbautomatische Pipeline wurde für Benutzer mit begrenztem Fachwissen in der Computer- und Bildgebungsanalyse entwickelt. Es kombiniert die Funktionalitäten von zwei etablierten Fiji-Plugins: FindFoci22 und 3D Suite23. Durch die Integration der präzisen Foki-Identifikationsfunktion von FindFoci mit den Objektidentifikations- und Segmentierungsfunktionen im 3D-Raum, die von der 3D Suite angeboten werden, generiert unser Ansatz zwei CSV-Dateien pro Kanal für jeden Erfassungsbereich. Diese Dateien enthalten ergänzende Informationen, die die Berechnung von Metriken erleichtern, die für verschiedene Arten der Signalverteilung geeignet sind, wie z. B. die Anzahl der Herde pro Zelle, den Abstand der Brennpunkte vom Kernschwerpunkt und den Inhomogenitätskoeffizienten (IC), den wir für die diffuse Proteinfärbung eingeführt haben. Darüber hinaus erkennen wir an, dass die Datenextrapolation für Benutzer mit begrenzten Fähigkeiten im Umgang mit Daten zeitaufwändig sein kann. Um diesen Prozess zu optimieren, stellen wir ein Python-Skript zur Verfügung, das alle gesammelten Messungen automatisch in einer einzigen Datei für jedes Experiment zusammenfasst. Benutzer können dieses Skript ausführen, ohne eine Programmiersprachensoftware installieren zu müssen. Wir stellen einen ausführbaren Code auf Google Colab zur Verfügung, einer Cloud-basierten Plattform, die das Schreiben von Python-Skripten direkt im Browser ermöglicht. Dadurch wird sichergestellt, dass unsere Methode intuitiv und für den sofortigen Einsatz leicht zugänglich ist.
Wir demonstrieren die Wirksamkeit unseres Protokolls bei der Analyse und Quantifizierung von Veränderungen in der Signalverteilung von zwei nukleären Proteinen: dem Bromodomänen-haltigen Protein 4 (BRD4) und dem Suppressor von Zeste-12 (SUZ12). BRD4 ist ein gut dokumentiertes Koaktivatorprotein innerhalb des Mediator-Komplexes, von dem bekannt ist, dass es Kondensate bildet, die mit der Polymerase II-abhängigen transkriptionellen Initiation assoziiert sind24,25. SUZ12 ist eine Proteinkomponente des Polycomb Repressive Complex 2 (PRC2), der für die Regulierung der Ablagerung der H3K27me3-Histonmodifikation verantwortlich ist26,27. Diese Proteine weisen unterschiedliche Muster innerhalb von zwei verschiedenen Zelltypen auf: frisch isolierte humane CD4+-naive T-Zellen, die ruhen und eine langsame Transkriptionsaktivität aufweisen, und in vitro differenzierte TH1 CD4+-Zellen, bei denen es sich um spezialisierte, proliferierende Effektorzellen handelt, die eine erhöhte Transkription aufweisen28.
Protokoll
Die Verwendung von menschlichen Proben zu Forschungszwecken wurde von den Ethikkommissionen der Fondazione Istituto di Ricovero e Cura a Carattere Scientifico (IRCCS) Cà Granda Ospedale Maggiore Policlinico (Mailand) genehmigt, und es wurde die Einverständniserklärung aller Probanden eingeholt (Zulassungsnummern: 708_2020). Das Protokoll ist in drei Hauptabschnitte unterteilt: Immunfluoreszenzausführung, Bildaufnahme und Bildanalyse. Im Durchschnitt dauert es 4 Arbeitstage, um fertig zu werden (Abbildung 1).
1. Immunfluoreszenz-Vorbereitung
HINWEIS: Dieses Immunfluoreszenzprotokoll kann leicht an verschiedene Zelltypen und Proteinziele angepasst werden, indem die Fixierungs- und Permeabilisierungsbedingungen angepasst werden. Die Immunfluoreszenzvorbereitung dauert in der Regel weniger als 3 Tage, wobei die Dauer der Inkubation des primären Antikörpers je nach Antikörperqualität und Zielprotein variiert (Abbildung 1).
- Probenvorbereitung
- Isolierung von humanen mononukleären Zellen des peripheren Blutes (PBMCs) durch eine Dichtegradientenzentrifugation über ein Medium mit einer Dichte von ca. 1,077 g/mL gemäß den Anweisungen des Herstellers (Materialtabelle).
- Isolieren Sie CD4+ T-Zellen aus PBMCs mit magnetischen Kügelchen gemäß den Anweisungen des Herstellers (Materialtabelle).
- Färbt Zellen mit Antikörpern für CD4, CD45RA und CD45RO ein (Table of Materials) und fährt mit der FACS-Sortierung von naiven CD4+ T-Zellen als CD4+/CD45RA+/CD45RO-Zellen fort, wie an anderer Stelle beschrieben 29,30.
HINWEIS: In der Materialtabelle finden Sie die Spezifikationen des FACS-Sortierers, der in diesem Protokoll verwendet wird. - Induzieren Sie die Differenzierung von naiven CD4+ T-Zellen in T-Helfer 1 (T, H,1, CD4+ Zellen), wie in 29 beschrieben. Kurz gesagt, kultivieren Sie 1,5 x 106 Zellen/ml FACS-sortierte naive CD4+ T-Zellen in TH1 Medium und stimulieren Sie sie mit anti-CD3/anti-CD28 magnetischen Kügelchen im Verhältnis 1:1. Zählen Sie die Zellen und teilen Sie sie, wenn sie 1,5 × 106 Zellen/ml alle 2-3 Tage erreichen (siehe Tabelle 1 für die mittlere Zusammensetzung von TH1).
- Bewerten Sie die Sekretion von Effektorfunktionszytokinen nach 7 Tagen der Differenzierung, wie in 29 beschrieben.
- Zellfixierung und Permeabilisierung
HINWEIS: Alle diese Verfahren wurden in 30,31 mit geringfügigen Anpassungen beschrieben.- Um eine optimale Zelladhäsion zu gewährleisten, behandeln Sie Glasdeckgläser (10 mm, Dicke 1,5 H) wie folgt mit einer Beschichtungslösung (Tabelle 1):
- Reinigen Sie die Deckgläser, indem Sie sie zunächst mit destilliertem Wasser (ddH2O) waschen, dann mit 70 % Ethanol (EtOH) abspülen und an der Luft trocknen lassen.
- Legen Sie die gewaschenen Deckgläser für die Schritte 1.2.1.3-1.3 in eine 24-Multiwell-Platte.
- Tragen Sie einen Tropfen mit 200 μl Beschichtungslösung auf das Glasdeckglas auf. Nach 5 Minuten den Tropfen herausnehmen und an der Luft trocknen lassen.
- Waschen Sie die Deckgläser, indem Sie einen Tropfen von 200 μL ddH2O auftragen. Nach 5 Minuten den Tropfen herausnehmen und an der Luft trocknen lassen.
- Wiederholen Sie 3 x Schritte 1.2.1.3-1.2.1.4.
- Resuspendieren Sie naive CD4+ T-Zellen und TH1 CD4+ Zellen in 1x phosphatgepufferter Kochsalzlösung (PBS) in einer Konzentration von 2 × 106 Zellen/ml.
HINWEIS: Die angegebene Konzentration wird speziell für kleine Zellen wie menschliche primäre T-Lymphozyten empfohlen. Für adhärente Zellen ist eine Behandlung mit Glasbeschichtung nicht erforderlich. Fahren Sie stattdessen direkt mit der Züchtung der Zellen auf der Glasoberfläche unter Verwendung des entsprechenden Zellkulturmediums fort. - Tragen Sie einen 200 μl Tropfen der Zellsuspension auf den Glasdeckglas auf. Lassen Sie die Zellen 30 Minuten lang bei Raumtemperatur (RT) aussäen; Entfernen Sie dann den Tropfen.
- Fixieren Sie die Zellen mit frisch filtriertem 3%igem Paraformaldehyd (PFA-Lösung, Tabelle 1) für 10 min bei RT.
- Waschen Sie das Glasdeckglas mit TPBS (Tabelle 1) für 3 x 5 min bei RT.
- Entfernen Sie TPBS und fügen Sie die Permeabilisierungslösung (Tabelle 1) für 10 min bei RT hinzu.
- Die Permeabilisierungslösung wird verworfen und die Probe in einer Lagerlösung (Tabelle 1) von 1 h bis über Nacht (EIN) bei 4 °C inkubiert.
HINWEIS: In diesem Stadium kann das Protokoll sicher gestoppt werden, und die Glasdeckgläser können in einer Lagerlösung in einer 24-Multiwell-Platte für 3-4 Wochen aufbewahrt werden.
- Um eine optimale Zelladhäsion zu gewährleisten, behandeln Sie Glasdeckgläser (10 mm, Dicke 1,5 H) wie folgt mit einer Beschichtungslösung (Tabelle 1):
- Immunfluoreszenz
- (Fakultativ) Entfernen Sie das Deckglas von der 24-Multiwell-Platte und frieren Sie es schnell 30 s lang auf Trockeneis ein, tauen Sie es bei RT auf und waschen Sie dann das Glasdeckglas in einer vorgefüllten Vertiefung mit Aufbewahrungslösung.
- (Fakultativ) Wiederholen Sie 3 x Schritt 1.3.1.
- 5 min bei RT in Permeabilisierungslösung waschen. Anschließend 2 x 5 min mit TPBS bei RT waschen.
- Inkubieren Sie in 0,1 N HCl für 12 min bei RT.
- Führen Sie zwei Schnellwäschen in 1x PBS durch.
HINWEIS: Die spezifizierten Zellpermeabilisierungsbedingungen, einschließlich Gefrier- und Auftauschritten und HCl-Behandlung, sind optimal für die Färbung von Kernkomponenten in Zellen, die durch dicht gepacktes Chromatin gekennzeichnet sind. Wenn es sich jedoch um Zellen handelt, die durch ein weniger kompaktes Chromatin gekennzeichnet sind, ist es ratsam, diese Schritte zu reduzieren oder zu vermeiden. Um auf zytoplasmatische Komponenten abzuzielen, sollten Sie außerdem in Betracht ziehen, die HCl-Behandlung zu reduzieren oder zu eliminieren. - Die Zellen werden mit einem Primärantikörper (BRD4, 1:500 oder SUZ12, 1:100) inkubiert, der in Antikörperverdünnungspuffer (Tabelle 1) (200 μl für jedes Glasdeckglas) bei 4 °C verdünnt ist.
HINWEIS: Die Immunfluoreszenz kann durch Multiplexen von mehr als einem primären Antikörper durchgeführt werden, abhängig von den experimentellen Anforderungen, den Sekundärantikörpern und den Nachweissystemen. - 3 x 5 min mit PBS-T (Tabelle 1) bei RT unter leichtem Schütteln waschen.
- Inkubieren Sie die Zellen mit einem Sekundärantikörper, der in Antikörperverdünnungspuffer (200 μl für jedes Glasdeckglas) verdünnt ist, für 1 h bei RT.
HINWEIS: Wählen Sie den Sekundärantikörper, der am besten zu den experimentellen Anforderungen und den verfügbaren Nachweissystemen passt. Stellen Sie sicher, dass jeder Sekundärantikörper auf die Spezies abzielt, von der der Primärantikörper abgeleitet ist. Wählen Sie außerdem Fluorophore aus, die mit den Filtern und der Lichtquelle des Mikroskops kompatibel sind. Es ist entscheidend, eine spektrale Überlappung zwischen dem gewählten Fluorophor und dem Emissionsspektrum der Kernfärbung zu vermeiden. - 3 x 5 min mit PBS-T bei RT unter leichtem Schütteln waschen.
- Färben Sie mit 1 ng/ml 4',6-Diamidino-2-phenylindol (DAPI), verdünnt in 1x PBS für 5 min bei RT.
HINWEIS: Alternative Farbstoffe können für die Kernfärbung verwendet werden, solange sie sich nicht mit dem Emissionsspektrum von Sekundärantikörpern überlappen. - Führen Sie mehrere Schnellwäschen in 1x PBS durch.
- Montieren Sie das Glasdeckglas mit Antifa-Eindeckmedium auf einen Objektträger.
2. Bilderfassung
HINWEIS: Die Dauer der Bildaufnahme hängt vom Instrument und den gewählten Einstellungen ab.
- Aufnahme eines konfokalen Mikroskops
- Nehmen Sie 3D-Bilder mit einem konfokalen Mikroskop auf, indem Sie eine Schrittweite von 0,25 μm in z und eine Pixelgröße von 0,1-0,2 μm einstellen.
HINWEIS: Um mit unserem 63x 1,4 NA Ölobjektiv eine optimale lichtbeugungsbegrenzte Auflösung zu erreichen, haben wir die Lochblendengröße auf 0,8 AE, den 2-fachen Liniendurchschnitt und die Bildgröße auf 1024 × 1024 Pixel eingestellt. Sowohl der Anregungslaser als auch die Kanalerfassungssequenz wurden bewusst gewählt, um Interferenzen oder Übersprechen zwischen den verwendeten Fluorophoren zu vermeiden. Die Einstellung der Parameter sollte jedoch auf die spezifischen Eigenschaften des Mikroskops und der Probe abgestimmt sein. In der Materialtabelle finden Sie die Spezifikationen des in diesem Protokoll verwendeten konfokalen Mikroskops. - Erfassen Sie eine konsistente Anzahl von Zufallsfeldern, um ca. 50 Zellen pro biologischem Replikat zu umfassen.
- Nehmen Sie 3D-Bilder mit einem konfokalen Mikroskop auf, indem Sie eine Schrittweite von 0,25 μm in z und eine Pixelgröße von 0,1-0,2 μm einstellen.
3. Bildanalyse
- Installation der Software
- Laden Sie die letzte verfügbare Version von Fiji von der offiziellen Download-Seite (https://imagej.net/software/fiji/downloads herunter, und installieren Sie sie.
- Installieren Sie die 3D Suite entweder über die Fiji-Update-Site oder folgen Sie manuell den Anweisungen auf der 3D Suite-Website (https://mcib3d.frama.io/3d-suite-imagej/#download).
- Installieren Sie GDSC (FindFoci) entweder über die Fiji-Update-Site oder manuell, indem Sie den Anweisungen im GitHub-Repository (https://github.com/aherbert/gdsc) folgen.
- Initialisieren Sie die Einstellungen im 3D-Manager.
- Öffnen Sie das Optionsfeld des 3D-Managers , indem Sie auf Plugins | 3DSuite | 3D-Manager-Optionen klicken.
- Aktivieren Sie im Optionsfenster des 3D-Managers die Kontrollkästchen für die folgenden Messwerte: Volumen (Einheit), Mittlerer Grauwert, Begrenzungsrahmen (pix), Std Dev Grauwert, Schwerpunkt (pix) und Schwerpunkt (Einheit).
- Aktivieren Sie die folgenden Optionen: Objekte auf Kanten XY ausschließen und Objekte auf Kanten Z ausschließen und klicken Sie auf OK.
HINWEIS: Dieser Schritt muss nur einmal ausgeführt werden, um die Metriken zu konfigurieren, die in den räumlichen und quantitativen Informationsdateien gespeichert werden. Die ausgewählten Metriken sind für die Sicherstellung einer ordnungsgemäßen Funktion der Pipeline unerlässlich. Optionale zusätzliche Metriken können in diesen Schritt einbezogen werden, wie im Abschnitt Diskussion näher beschrieben.
- Legen Sie die Parameter in der FindFoci-GUI fest.
HINWEIS: Dieser Schritt muss nur einmal ausgeführt werden, um die halbautomatische Pipeline mit optimalen Parametern zu konfigurieren, die dann in die Skripte integriert werden.- Öffnen Sie das Testbild. Duplizieren Sie den Proteinkanal einschließlich des Stacks (Strg + Umschalt + D), aktivieren Sie das Kontrollkästchen Hyperstack , geben Sie die entsprechende Kanalnummer im Feld Kanäle (c) an und benennen Sie ihn entsprechend um.
- Starten Sie den Fiji Macro Recorder, indem Sie zu Plugins | Makros | Aufzeichnen.
- Öffnen Sie das FindFoci-Plugin (klicken Sie auf Plugins | GDSC | FindFoci | FindFoci GUI) und wählen Sie das zu analysierende Bild aus dem Dropdown-Menü "Bild" aus.
- Stellen Sie die Parameter wie folgt ein (Abbildung 2A): Gaußsche Unschärfe = 1,5; background-Methode = SD über dem Mittelwert; Hintergrund Parameter = 9; Suchmethode = Bruchteil des Peaks - Hintergrund; Suchparameter = 0,7; peak-Methode = relativ über dem Hintergrund; Spitzenwert = 0,2; Mindestgröße = 5; Maximale Spitzen = 1.000.000.
HINWEIS: Die angegebenen Parameter wurden auf der Grundlage unserer Fallstudien ausgewählt und sind möglicherweise nicht für andere Färbeverfahren geeignet. - Um die Fokusidentifikation zu verbessern, passen Sie die folgenden Parameter an:
- Die Gaußsche Unschärfe definiert das Ausmaß der Glättung, um die Segmentfokus zu verbessern. Halten Sie es in der Nähe des Brennpunktdurchmessers (Pixel).
- Der Parameter background legt einen Schwellenwert fest, um den Hintergrund vom Fokussignal zu unterscheiden. Erhöhen Sie die Werte, um strengere Schwellenwerte festzulegen.
- Der Suchparameter definiert den prozentualen Anteil der Fluoreszenz vom Peak, der in die Signalerkennung einbezogen wird. Verringern Sie die Werte, um Bereiche einzubeziehen, die weiter vom Fluoreszenzpeak entfernt sind.
- Der Peak-Parameter bestimmt den Grad, in dem zwei Signalspitzen als kontinuierlich oder getrennt betrachtet werden. Eine Verringerung des Wertes führt zur Trennung der Peaks.
- Max peaks gibt die maximale Anzahl der identifizierbaren Brennpunkte an. Legen Sie hohe Zahlen fest, um alle Brennpunkte im Bild einzubeziehen.
- Führen Sie FindFoci aus , und kopieren Sie die Zeichenfolge, die im Aufzeichnungsfenster angezeigt wird (Schritt 3.4.2, Abbildung 2B), die die ausgewählten Parameter enthält, ohne Anführungszeichen. Weitere Informationen zu den Einstellungen finden Sie in der Anleitung des Plugins 22.
- Pipeline auf Google Colab
- Laden Sie das Notebook "final_nuclear_protein_metrics.ipynb" herunter (Ergänzende Datei 3).
- Öffnen Sie das Notizbuch in Google Colab (https://colab.research.google.com/).
- Laden Sie alle Ordner, die die .csv Dateien der einzelnen Bildfelder enthalten, in einen bevorzugten Ordner in Google Drive hoch.
- Geben Sie im Notebook den Pfad des Ordners an, in dem die Ergebnisunterordner gespeichert sind, und führen Sie alle Zellen aus. Wenn die Kompilierung des Codes abgeschlossen ist, wird die endgültige Tabellenkalkulationsdatei mit allen kompilierten Daten in demselben Ordner erstellt, in dem die .csv Dateien hochgeladen wurden.
Ergebnisse
Das in dieser Methode skizzierte Protokoll erleichtert die Visualisierung und Quantifizierung von Veränderungen in der nuklearen Proteinfärbung in humanen primären T-Zellen und kann für verschiedene Zelltypen und Proteinziele angepasst werden. Als Fallstudien haben wir die Färbung von BRD4 und SUZ12 in naiven und TH1 CD4+ Zellen durchgeführt und analysiert.
BRD4 zeigt ein gut punktiertes Färbemuster sowohl in ruhenden naiven als auch in differenzierten TH1 CD4+ Zellen, was die Verwendung von designten Parametern zur Foki-Identifizierung für beide Zelltypen ermöglicht (Abbildung 3A). Unsere Pipeline ermöglicht die Quantifizierung verschiedener Parameter, einschließlich der Anzahl, des Volumens, des Fluoreszenzsignals (ausgedrückt als mittlere Fluoreszenzintensität (MFI)), der Abdeckung und der räumlichen Anordnung von BRD4-Brennpunkten innerhalb des Zellkerns (Abbildung 3B-F). Zusätzlich haben wir eine automatische Berechnung eingeführt, um den prozentualen Anteil der Brennpunkte am Kernvolumen zu bestimmen, was den Vergleich zwischen Zellen mit unterschiedlichen Kerngrößen erleichtert (Abbildung 3E).
Schließlich haben wir die Möglichkeit geschaffen, die Entfernungen der Brennpunkte vom Kernzentrum zu kartieren, um ihre nukleare Positionierung zu verstehen (Abbildung 3F). Die Ergebnisse der Quantifizierung zeigen bemerkenswerte Veränderungen in den BRD4-Herden zwischen TH1 und ruhenden CD4+ T-Zellen, die eine Zunahme der Anzahl, Größe, Helligkeit und des Volumens sowie Unterschiede in ihrer Verteilung und Lokalisation umfassen. Diese Befunde stimmen mit der erhöhten transkriptionellen Aktivität von aktivierten/proliferierenden T-Zellen im Vergleich zu ihrem ruhenden Gegenstück überein28.
Im Gegensatz dazu ist SUZ12 nicht an der Kondensatbildung beteiligt. In unserer Untersuchung beobachteten wir einen signifikanten Unterschied im Färbemuster von SUZ12 zwischen den beiden zellulären Zuständen: Ein punktförmiges Muster in naiven T-Zellen geht in ein diffuses Muster in TH1 CD4+ Zellen über (Abbildung 4A). Diese signifikante Verschiebung des Proteinverhaltens erschwert die Vergleichbarkeit von Foki-Merkmalen bei identischen Parametern, da die Punktidentifikation in TH1 CD4+ Zellen weitgehend erfolglos ist (Abbildung 4A). Während es im Allgemeinen empfohlen wird, beim Vergleich der Immunfluoreszenzfärbung unter verschiedenen zellulären Bedingungen die Konsistenz sowohl der Erfassungs- als auch der Analyseparameter zu wahren, werden Anpassungen bei wesentlichen Änderungen des Ziels erforderlich.
In diesem speziellen Szenario, wie in Abbildung 4B dargestellt, stößt FindFoci trotz des Experimentierens mit verschiedenen Parameterkombinationen, einschließlich der Anpassung von Hintergrundparametern, Gaußscher Unschärfe und Suchparametern (wie in Protokollschritt 3.4.5 vorgeschlagen), aufgrund der inhärenten Verteilungsnatur des Proteins auf Schwierigkeiten bei der Unterscheidung von echten Signalpeaks. Darüber hinaus betonen wir, dass konventionelle Messungen wie die nukleare MFI irreführend sein können (Abbildung 4C), da sie Veränderungen in der Signalverteilung nicht berücksichtigen.
Um solche Veränderungen zu messen, schlagen wir daher die Verwendung des Variationskoeffizienten vor, einer Metrik, die wir in dieses Protokoll eingeführt haben, um die Proteindiffusion zu charakterisieren, die als Inhomogenitätskoeffizient (IC) bezeichnet wird. Das IC erfasst die Diffusion von SUZ12-Foki in TH1 CD4+ -Zellen genau, ohne deren Identifizierung mit falschen Segmentierungsansätzen zu erzwingen (siehe Abbildung 4D). Schließlich validieren wir durch die Verwendung von DAPI als Kontrolle, die gleichmäßig über beide Zellbedingungen verteilt bleibt, die Wirksamkeit dieses Parameters weiter (Abbildung 4E).
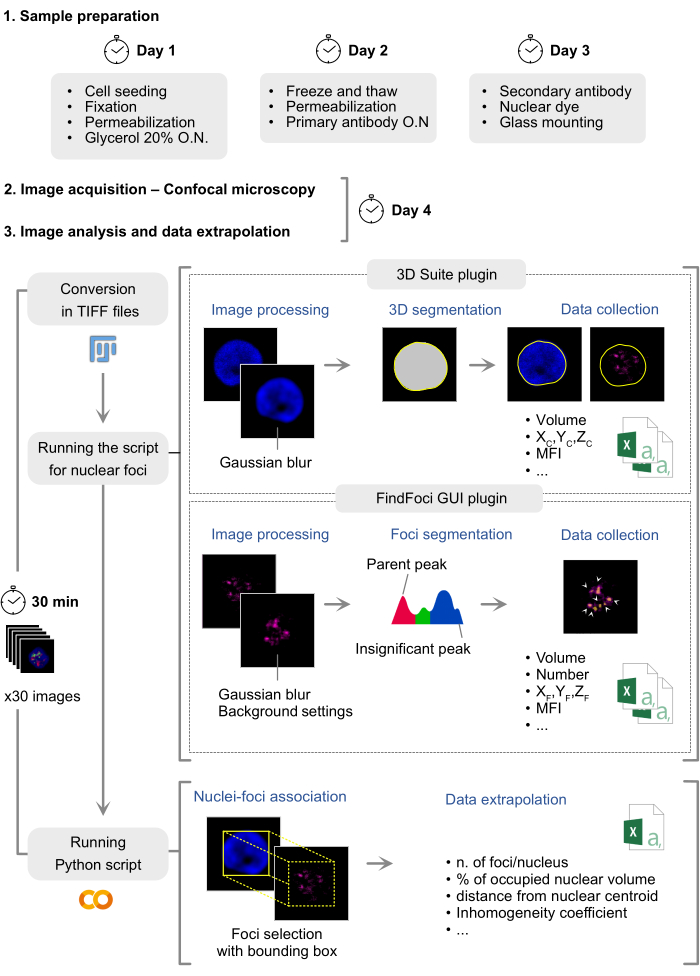
Abbildung 1: Schematische Darstellung der Immunfluoreszenzpräparation und Datenanalyse. Das Protokoll für die Färbung und Analyse der Immunfluoreszenz von Kernproteinen besteht aus drei Hauptphasen: Probenvorbereitung, Bildaufnahme und Analyse, die sich über etwa 4 Arbeitstage erstrecken. Das Skript verwendet zwei Plugins, die 3D Suite zur Durchführung der Zellkernsegmentierung und FindFoci zur Verarbeitung des Proteinkanals. Räumliche und quantitative Informationen zu den Brennpunkten und dem Zellkern werden in separaten csv-Dateien gesammelt. Ein auf Google Colab ausführbares Skript verknüpft Foki-Messungen mit den entsprechenden Kernen mithilfe des Kern-Begrenzungsrahmens und berechnet abgeleitete Messungen auf der Grundlage der gesammelten Daten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
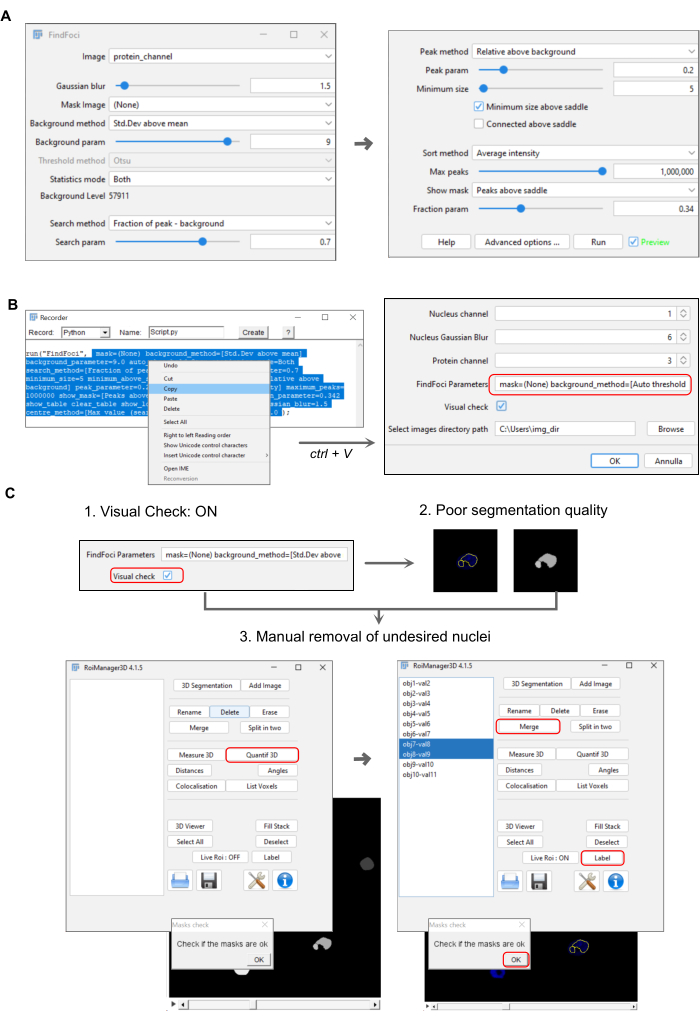
Abbildung 2: Darstellung der Einstellungen der Pipeline zur Quantifizierung von Kernproteinen. (A) FindFoci-Popup-Fenster, das die Parameter für die Foki-Identifizierung veranschaulicht (Protokollabschnitt 3.4.3). (B) Popup-Fenster, das das Makro darstellt, das die ausgewählten Parameter von FindFoci aufzeichnet. Der kopierte String wird im Startfenster eingefügt (Protokollschritte 3.4.6-3.5.3.4). (C) Panel, das den Schritt der Qualitätskontrolle der Kernsegmentierung zum manuellen Entfernen oder Modifizieren von Kernbereichen von Interesse mit dem 3D-Manager zeigt (Protokollschritte 3.5.5.1 -3.5.5.3). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
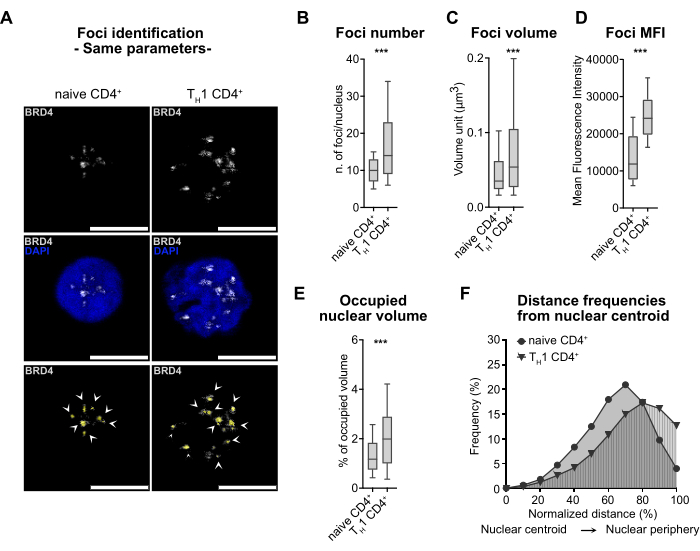
Abbildung 3: Vergleiche zwischen verschiedenen zellulären Bedingungen auf der Grundlage von BRD4-Foki-Analysen. (A) Repräsentative konfokale Fluoreszenzmikroskopie-Bilder der Immunfluoreszenzfärbung von BRD4 (grau) in humanen primär naiven CD4+ T-Zellen und TH1 CD4+ Zellen. Die Zellkerne werden mit DAPI (blau) gegengefärbt. Originalvergrößerung 63x; Maßstabsleiste = 5 μm. Unten, BRD4-Brennpunkte, die durch die Pipeline gekennzeichnet sind, sind mit weißen Pfeilspitzen markiert und gelb gekontert (unten). (B) Boxplot zur Darstellung der BRD4-Foki-Anzahl/des BRD4-Zellkerns in naiven und TH1 CD4+ Zellen (n = 2 Individuen). (C) Boxplot-Darstellung des Volumens (μm 3) von BRD4-Foki in naiven und TH1 CD4+ Zellen (n = 2 Individuen). (D) Boxplot-Darstellung der MFI-Foki von BRD4-Foci in naiven und TH1 CD4+ Zellen (n = 2 Individuen). (E) Boxplot, der den prozentualen Anteil des Kernvolumens an BRD4-Brennpunkten im Verhältnis zum Gesamtkernvolumen darstellt (n = 2 Individuen). (F) Darstellung der Abstandshäufigkeiten von BRD4-Brennpunkten vom Kernschwerpunkt zur Kernperipherie (n = 2 Individuen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
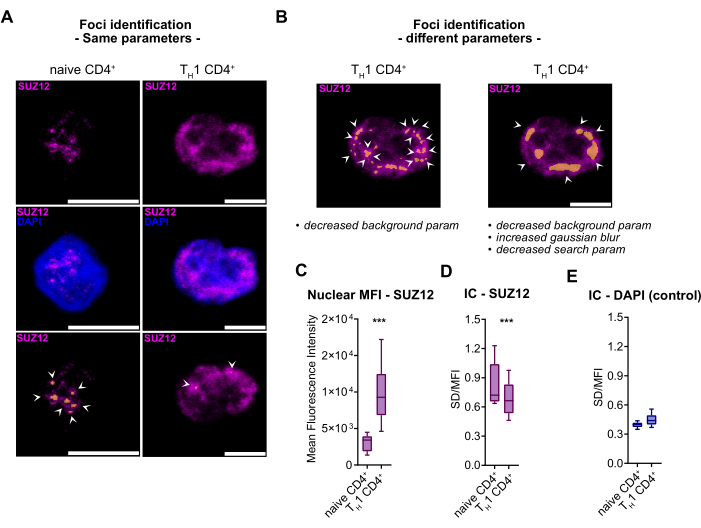
Abbildung 4: Inhomogenitätskoeffizient, eine Metrik zur Quantifizierung des Übergangs von einem punktförmigen zu einem dispergierten Muster in SUZ12-Immunfluoreszenzsignalen. (A) Repräsentative konfokale Fluoreszenzmikroskopiebilder der Immunfluoreszenzfärbung für SUZ12 (magenta) in humanen primären naiven CD4+ T-Zellen und TH1 CD4+ Zellen. Die Zellkerne werden mit DAPI (blau) gegengefärbt. Originalvergrößerung 63x; Maßstabsleiste = 5 μm. Unten sind die durch die Pipeline identifizierten SUZ12-Herde mit einer weißen Pfeilspitze markiert und orange (B) gekontert. Beispiele für eine falsche Foki-Identifizierung in TH1 CD4+ Zellen dargestellt durch weiße Pfeile und orangefarbene Maske mit zwei verschiedenen Parametereinstellungen. (C) Boxplot-Darstellung des nukleären MFI von SUZ12 in naiven und TH1 CD4+ Zellen (n = 2 Individuen). (D) Boxplot-Darstellung von SUZ12 IC in naiven und TH1 CD4+ Zellen (n = 2 Individuen). (E) Boxplot-Darstellung von DAPI IC in naiven und TH1 CD4+ Zellen (n = 2 Individuen). Abkürzungen: DAPI = 4',6-Diamidino-2-phenylindol; MFI = mittlere Fluoreszenzintensität; IC = Inhomogenitätskoeffizient. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Lösung | Zusammensetzung | Kommentare/Beschreibung | |
| TH1 mittel | RPMI mit GlutaMAX-I, 10 % (v/v) fötalem Kälberserum (FBS), 1 % (v/v) nicht-essentiellen Aminosäuren, 1 mM Natriumpyruvat, 50 IE/ml Penicillin, 50 μg/ml Streptomycin, 20 IE/ml rekombinantes IL-2, 10 ng/ml rekombinantes IL-12, 2 mg/ml neutralisierendes Anti-IL-4. | Schritt 1.1.4. | |
| Beschichtungslösung | 0,1 % Poly-L-Lysin in ddH2O | Schritt 1.2.1. | |
| PBS-T | 1x PBS/0,1% TWEEN 20 pH 7,0 | Schritt 1.3. | |
| PFA-Lösung zur Fixierung | 3 % Paraformaldehyd (PFA), verdünnt in 0,1 % PBS-TWEEN | Schritt 1.2.4. | |
| TPBS (Englisch) | 0,05 % Triton X-100 verwässert in 1x PBS | Schritt 1.2., 1.3. | |
| Lösung für die Permeabilisierung | 0,5 % TPBS verwässert in 1x PBS | Schritt 1.2., 1.3. | |
| Aufbewahrungslösung | 20% Glycerin/1x PBS | Schritt 1.2.7., 1.3.1. | |
| Antikörper-Verdünnungspuffer | 0,1 % PBS-TWEEN/2 % Ziegenserum/1 % BSA | Schritt 1.3.6., 1.3.8. | |
Tabelle 1: Zusammensetzung der in diesem Protokoll verwendeten Medien und Puffer.
Ergänzende Datei 1: "convert_to_TIFF.py". Diese Datei enthält das Skript zum Konvertieren beliebiger Bilddateien (z. B. ND2, LIF usw.) in TIFF-Dateien, die für genaue Quantifizierungsprozesse erforderlich sind. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Akte 2: "nuclear_prot_q.py". Diese Datei enthält das Skript, das es ermöglicht, Bilder mit mindestens zwei Kanälen (nukleöse Färbung, nukleare Proteinfärbung) zu messen und zu quantifizieren. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: "final_nuclear_protein metrics.ipynb". Diese Datei enthält ein Jupyter-Notebook, das eine Zusammenfassung aller relevanten Parameter extrahiert und in einer einzigen Excel-Datei zusammenstellt, wobei die Ausgabe von "nuclear_prot_q.py" als Eingabe verwendet wird. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Arbeit stellen wir eine Methode zur Durchführung von Immunfluoreszenzexperimenten an nukleären Proteinen in humanen T-Lymphozyten vor. Diese Methode bietet Flexibilität für den Einsatz mit verschiedenen Zelltypen durch geringfügige Modifikationen in den Fixierungs- und Permeabilisierungsschritten, wie zuvor beschrieben30,31.
Unser Imaging-Workflow baut auf etablierten Techniken auf, die in der Literatur beschrieben sind, insbesondere FindFoci und 3D Suite22,23. Im Gegensatz zu früheren Veröffentlichungen stellen wir einen ausführbaren, in Fidschi integrierten Jython-Code für eine halbautomatische Pipeline zur Verfügung, der eine akribische Parameterverfolgung und visuelle Qualitätsprüfungen ermöglicht. Insbesondere haben wir die optionale Möglichkeit, die Qualität der Kernsegmentierung im Protokollabschnitt 3.5.3.5 zu regulieren, eingeführt und einen Korrekturschritt im Protokollabschnitt 3.5.5 implementiert. Mit dem auf Google Colab ausführbaren Skript können Benutzer umfassende Messungen abrufen und die für ihre spezifischen biologischen Szenarien am besten geeigneten auswählen.
Die bereitgestellte Analyse-Pipeline ist in Jython (Java + Python) geschrieben und erfordert die Installation von Fiji für die Ausführung, zusammen mit zwei unterschiedlichen Plugins: 3D Suite und FindFoci (Abbildung 1). Die Pipeline beginnt mit der Bildkonvertierung in das TIFF-Format und der Vorverarbeitung des Kanals, der die Kerne enthält, mit einem Gaußschen Unschärfefilter, um die Segmentierung der Kerne zu verbessern. Wir weisen die 3D Suite an, sowohl i) räumliche Informationen wie Kernvolumen, Schwerpunkt- und Bounding-Box-Koordinaten (die das kleinste Quader darstellen, das das Objekt umschließt und zur Zuordnung von Brennpunkten zum rechten Kern verwendet wird) als auch quantitative Informationen, einschließlich MFI und Standardabweichung des Signals, zu erfassen. Anschließend verwendet die Pipeline FindFoci, um lokale Maxima im Foki-Signal zu identifizieren, wodurch die Abhängigkeit von globalen Schwellenwertmethoden (die oft auf der Diskretion des Benutzers beruhen und möglicherweise nicht präzise sind) beseitigt wird, und sowohl räumliche als auch quantitative Informationen relativ zu den Foci aufgezeichnet werden. Der Ausgabeordner enthält somit zwei CSV-Dateien pro Kanal, die räumliche ("M_.csv") und quantitative ("Q_.csv") Metriken umfassen.
Die endgültige Datei, die alle gesammelten und abgeleiteten Messungen enthält, wird durch Ausführen eines Python-Skripts auf Google Colab generiert. Diese Datei enthält Metriken wie die Anzahl der Brennpunkte pro Kern, den Brennpunkt-MFI, den Inhomogenitätskoeffizienten (IC), den Kern-MFI, den Brennpunktabstand vom Kernschwerpunkt und den Prozentsatz des belegten Kernvolumens. Wir schätzen eine Verarbeitungszeit von ca. 1 Minute pro Bild einschließlich visueller Qualitätskontrollschritte. Diese Verarbeitungszeit kann durch die Optimierung der Immunfluoreszenzeinstellungen und Analyseparameter weiter verkürzt werden.
Die Pipeline berechnet Parameter für Kernproteine, die sowohl gut gepunktete als auch diffuse Muster aufweisen, so dass der Benutzer den am besten geeigneten Parameter auswählen kann. Wir empfehlen, den Inhomogenitätskoeffizienten zu verwenden, um die Auflösung von Proteinherden zu beschreiben.
Ein kritischer Schritt, um eine korrekte Identifizierung von Brennpunkten zu gewährleisten, ist die Einstellung von Parametern, um echte Signalspitzen vom Hintergrundrauschen zu unterscheiden. Es ist wichtig, vor dem Ausführen der Pipeline gründliche Tests an zufälligen Bildern durchzuführen. Während sich unsere Studie in erster Linie auf einen einzigen Kanal konzentriert, unterstützt die Pipeline die Einführung verschiedener Farbstoffkombinationen, die auf verschiedene Proteine abzielen, in Immunfluoreszenzexperimenten. Die gleichzeitige Analyse mehrerer Kanäle wird nicht unterstützt und muss für jeden Kanal separat ausgeführt werden. Darüber hinaus haben wir uns dafür entschieden, spezifische Messungen in diesem Protokoll darzustellen. In Abschnitt 3.3.2 können jedoch auch zusätzliche Metriken wie die integrierte Intensität zur Bewertung absoluter Änderungen des Fluoreszenzsignals oder physikalische Parameter wie Zirkularität und Kompaktheit in die Rohrleitung integriert werden. Die Einbeziehung von Metriken, die derzeit nicht in diese Pipeline integriert sind, erfordert Kenntnisse in der Python-Programmierung, um sie in die endgültige Metrikdatei zu integrieren. Andernfalls werden sie automatisch in die räumlichen ("M_.csv") und quantitativen ("Q_.csv") Informationsdateien aufgenommen.
Der Zeitpunkt dieser Pipeline hängt von Faktoren wie Bildgröße und -menge, der Anzahl der Kanäle pro Analyse und den Mikroskopeinstellungen ab. Darüber hinaus beeinflusst die Maschinenleistung die Ausführungszeit, wobei visuelle Schritte den Prozess möglicherweise verlangsamen.
Es ist erwähnenswert, dass die konfokale Mikroskopie zwar wertvolle Einblicke bietet, aber eine beugungsbegrenzte Technologie bleibt, die die Präzision der beobachteten Kernstrukturen oder Proteinherde einschränken kann. Um die Robustheit der Analyse zu erhöhen, empfehlen wir, die Anzahl der untersuchten Zellkerne und biologischen Proben zu erhöhen. Mit zunehmender Anzahl der analysierten Kerne werden bemerkenswerte Veränderungen in der Kernmorphologie oder Proteinverteilung deutlicher, unabhängig von Auflösungsbeschränkungen.
Im Hinblick auf mögliche zukünftige Anwendungen weist diese Methode eine Vielseitigkeit und Anpassungsfähigkeit an verschiedene Zelltypen und Färbeprotokolle auf. Zum Beispiel kann es problemlos in radiobiologischen Experimenten eingesetzt werden, wo die genaue Quantifizierung von durch ionisierende Strahlung induzierten Herden (IRIFs) als direkter Indikator für DNA-Schäden dient32. Obwohl unser Hauptaugenmerk auf der nuklearen Analyse liegt, kann das Protokoll leicht angepasst werden, indem ein zytoplasmatischer Farbstoff eingebaut und dieser durch den Kernkanal innerhalb des Skripts ersetzt wird. Darüber hinaus kann die Analyse nahtlos in andere experimentelle Methoden wie DNA- und RNA-FISH30,31 integriert werden, wobei Standardparameter für die Kerntopologieanalyse genutzt werden.
Offenlegungen
R.V. arbeitet wissenschaftlich mit dem Start-up T-One Therapeutics Srl zusammen. B.B. und F.M. sind Mitbegründer des Startups T-One Therapeutics Srl; E.P. ist derzeit bei T-One Therapeutics Srl beschäftigt; Alle anderen Autoren erklären, dass sie keine konkurrierenden Interessen haben.
Danksagungen
Wir danken der INGM Imaging Facility, insbesondere C. Cordiglieri und A. Fasciani, und der INGM FACS-Sortieranlage, insbesondere M.C. Crosti (Istituto Nazionale di Genetica Molecolare 'Romeo ed Enrica Invernizzi' (INGM), Mailand, Italien, für ihre wissenschaftliche und technische Unterstützung. Wir danken M. Giannaccari für seine technische informatische Unterstützung. Diese Arbeit wurde durch die folgenden Stipendien finanziert: Fondazione Cariplo (Bando Giovani, Fördernummer 2018-0321) und Fondazione AIRC (Fördernummer MFAG 29165) an F.M. Ricerca Finalizzata, (Fördernummer GR-2018-12365280), Fondazione AIRC (Fördernummer 2022 27066), Fondazione Cariplo (Fördernummer 2019-3416), Fondazione Regionale per la Ricerca Biomedica (FRRB CP2_12/2018), Piano Nazionale di Ripresa e Resilienza (PNRR) (Fördernummer G43C22002620007) und Progetti di Rilevante Interesse Nazionale (PRIN) (Förderkennzeichen 2022PKF9S) an B. B.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL Safe-Lock Tubes | Eppendord | #0030121503 | Protocol section 1 |
| 10 mL Serological pipettes | VWR | #612-3700 | Protocol section 1 |
| 20 µL barrier pipette tip | Thermo Scientific | #2149P-HR | Protocol section 1 |
| 50 mL Polypropylene Conical Tube | Falcon | #352070 | Protocol section 1 |
| 200 µL barrier pipette tip | Thermo Scientific | #2069-HR | Protocol section 1 |
| antifade solution - ProlongGlass - mountingmedia | Invitrogen | #P36984 | Step 1.3.12 |
| BSA (Bovine Serum Albumin) | Sigma | #A7030 | Step 1.3.6., 1.3.8. |
| CD4+ T Cell Isolation Kit | Miltenyi Biotec | #130-096-533 | Step 1.1.2. |
| DAPI (4,6-diamidino-2-phenylindole) | Invitrogen | Cat#D1306 | Step 1.3.10. |
| Dry ice | Step 1.3.1. | ||
| Dynabeads Human T-activator anti-CD3/anti-CD28 bead | Life Technologies | #1131D | magnetic beads step 1.1.4. |
| EtOH | Carlo Erba | #4146320 | Step 1.2.1.1. |
| FACSAria SORP | BD Bioscences | Step 1.1.3. Equipped with BD FACSDiva Software version 8.0.3 | |
| FBS (Fetal Bovine Serum) | Life Technologies | #10270106 | Step 1.1.4 |
| FICOLL PAQUE PLUS | Euroclone | GEH17144003F32 | Step 1.1.1. |
| FIJI Version 2.14.0 | - | - | Protocol section 3 |
| Glass coverslip (10 mm, thickness 1.5 H) | Electron Microscopy Sciences | #72298-13 | Step 1.2.1. |
| Glycerol | Sigma | #G5516 | Step 1.2.7-1.3.1. |
| Goat anti-Rabbit AF568 secondary antibody | Invitrogen | A11036 | Step 1.3.8. |
| HCl | Sigma | #320331 | Step 1.3.4. |
| human neutralizing anti-IL-4 | Miltenyi Biotec | Cat#130-095-753 | Step 1.1.4. |
| human recombinant IL-12 | Miltenyi Biotec | Cat#130-096-704 | Step 1.1.4. |
| human recombinant IL-2 | Miltenyi Biotec | Cat#130-097-744 | Step 1.1.4. |
| Leica TCS SP5 Confocal microscope | Leica Microsystems | - | Protocol section 2, Equipped with HCX PL APO 63x, 1.40 NA oil immersion objective, with an additional 3x zoom. Pinhole size : 0.8 AU. Line average 2×. Frame size 1024×1024 pixel. |
| MEM Non-Essential Amino Acids Solution | Life Technologies | #11140035 | Step 1.1.4. |
| Microscope Slides | VWR | #631-1552 | Step 1.3.12. |
| Mouse monoclonal anti-Human CD4 APC-Cy7 (RPA-T4 clone) | BD Bioscience | #557871 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RA PECy5 (5H9 clone) | BD Bioscience | #552888 | Step 1.1.3. |
| Mouse monoclonal anti-Human CD45RO APC (UCHL1 clone) | Miltenyi Biotec | #130-113-546 | Step 1.1.3. |
| Multiwell 24 well | Falcon | #353047 | Protocol section 1 |
| Normal Goat Serum | Invitrogen | PCN5000 | Step 1.3.6., 1.3.8. |
| PBS | Life Technologies | #14190094 | Protocol section 1 |
| Penicillin/Streptomycin solution | Life Technologies | #15070063 | Step 1.1.4. |
| PFA | Sigma | #P6148 | Step 1.2.4. |
| poly-L-lysine | Sigma | #P8920 | 1.2.1. |
| Primary antibody - BRD4 | Abcam | #ab128874 | Step 1.3.6. |
| Primary antibody - SUZ12 | Cel Signalling | mAb #3737 | Step 1.3.6. |
| RPMI 1640 W/GLUTAMAX-I | Life Technologies | #61870010 | Step 1.1.4. |
| Sodium Pyruvate | Life Technologies | #11360039 | Step 1.1.4. |
| Triton X-100 | Sigma | #T8787 | Step 1.2., 1.3. |
| TWEEN 20 | Sigma | #P9416 | Step 1.3. |
| Tweezers | - | - | Protocol section 1 |
Referenzen
- Aboelnour, E., Bonev, B. Decoding the organization, dynamics, and function of the 4D genome. Dev Cell. 56 (11), 1562-1573 (2021).
- Erdel, F., Rippe, K. Formation of chromatin subcompartments by phase separation. Biophys J. 114 (10), 2262-2270 (2018).
- Henninger, J. E., et al. RNA-mediated feedback control of transcriptional condensates. Cell. 184 (1), 207-225 (2021).
- Guo, Y. E., et al. Pol II phosphorylation regulates a switch between transcriptional and splicing condensates. Nature. 572 (7770), 543-548 (2019).
- Bhat, P., et al. 3D genome organization around nuclear speckles drives mRNA splicing efficiency. bioRxiv. , (2023).
- Spector, D. L., Lamond, A. I. Nuclear speckles. Cold Spring Harb Perspect Biol. 3 (2), a000646(2011).
- Kilic, S., et al. Phase separation of 53BP1 determines liquid-like behavior of DNA repair compartments. EMBO J. 38 (16), e101379(2019).
- Parker, M. W., et al. A new class of disordered elements controls DNA replication through initiator self-assembly. Elife. 8, e48562(2019).
- Feric, M., et al. Coexisting liquid phases underlie nucleolar subcompartments. Cell. 165 (7), 1686-1697 (2016).
- Yoneda, M., Nakagawa, T., Hattori, N., Ito, T. The nucleolus from a liquid droplet perspective. J Biochem. 170 (2), 153-162 (2021).
- Alberti, S., Carra, S. Nucleolus: A liquid droplet compartment for misbehaving proteins. Curr Biol. 29 (19), R930-R932 (2019).
- Strom, A. R., et al. Phase separation drives heterochromatin domain formation. Nature. 547 (7662), 241-245 (2017).
- Sanulli, S., et al. HP1 reshapes nucleosome core to promote phase separation of heterochromatin. Nature. 575 (7782), 390-394 (2019).
- Hyman, A. A., Weber, C. A., Julicher, F. Liquid-liquid phase separation in biology. Annu Rev Cell Dev Biol. 30, 39-58 (2014).
- Feric, M., Misteli, T. Phase separation in genome organization across evolution. Trends Cell Biol. 31 (8), 671-685 (2021).
- Mitrea, D. M., et al. Methods for physical characterization of phase-separated bodies and membrane-less organelles. J Mol Biol. 430 (23), 4773-4805 (2018).
- Fetter, J., et al. Endogenous gene tagging with fluorescent proteins. Methods Mol Biol. 1239, 231-240 (2015).
- Alberti, S., Gladfelter, A., Mittag, T. Considerations and challenges in studying liquid-liquid phase separation and biomolecular condensates. Cell. 176 (3), 419-434 (2019).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. J Cell Sci. 124 (10), 1607-1611 (2011).
- Scalisi, S., Ahmad, A., D'Annunzio, S., Rousseau, D., Zippo, A. Quantitative analysis of PcG-associated condensates by stochastic optical reconstruction microscopy (STORM). Methods Mol Biol. 2655, 183-200 (2023).
- Shihan, M. H., Novo, S. G., Le Marchand, S. J., Wang, Y., Duncan, M. K. A simple method for quantitating confocal fluorescent images. Biochem Biophys Rep. 25, 100916(2021).
- Herbert, A. D., Carr, A. M., Hoffmann, E. FindFoci: a focus detection algorithm with automated parameter training that closely matches human assignments, reduces human inconsistencies and increases speed of analysis. PLoS One. 9 (12), e114749(2014).
- Ollion, J., Cochennec, J., Loll, F., Escude, C., Boudier, T. TANGO: a generic tool for high-throughput 3D image analysis for studying nuclear organization. Bioinformatics. 29 (14), 1840-1841 (2013).
- Sabari, B. R., et al. Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361 (6400), 3958(2018).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19 (4), 523-534 (2005).
- Pasini, D., Bracken, A. P., Jensen, M. R., Denchi, E. L., Helin, K. Suz12 is essential for mouse development and for EZH2 histone methyltransferase activity. EMBO J. 23 (20), 4061-4071 (2004).
- Margueron, R., Reinberg, D. The Polycomb complex PRC2 and its mark in life. Nature. 469 (7330), 343-349 (2011).
- Peng, Z., et al. Brd4 regulates the homeostasis of CD8(+) T-lymphocytes and their proliferation in response to antigen stimulation. Front Immunol. 12, 728082(2021).
- Marasca, F., et al. LINE1 are spliced in non-canonical transcript variants to regulate T cell quiescence and exhaustion. Nat Genet. 54 (2), 180-193 (2022).
- Marasca, F., Cortesi, A., Bodega, B. 3D COMBO chrRNA-DNA-ImmunoFISH. Methods Mol Biol. 2157, 281-297 (2021).
- Marasca, F., Cortesi, A., Manganaro, L., Bodega, B. 3D Multicolor DNA FISH tool to study nuclear architecture in human primary cells. J Vis Exp. (155), (2020).
- Jakob, B., Splinter, J., Durante, M., Taucher-Scholz, G. Live cell microscopy analysis of radiation-induced DNA double-strand break motion. Proc Natl Acad Sci U S A. 106 (9), 3172-3177 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
ISSN 2578-6326
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.