Method Article
Damlacık tek hücre barkodlama tabanlı Transcriptomics yetişkin memeli dokuların
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu iletişim kuralı genel işlemlerini açıklar ve kalite kontrol sağlıklı yetişkin memeli tek hücreleri damlacık-esaslı, yüksek üretilen iş tek hücre RNA-Seq hazırlıkları hazırlanması için gerekli denetler. Sıralama parametreleri, okuma hizalama ve aşağı akım tek hücreli bioinformatic analiz da sağlanır.
Özet
Tek hücre gen ekspresyonu doku veya microenvironment içindeki tek tek hücreleri binlerce genelinde analizidir tanımlayıcı hücre kompozisyon, ayrımcılık işlevsel Devletler ve moleküler yolları gözlenen doku temel için değerli bir araç işlevler ve hayvan davranışları. Ancak, sonraki aşağı akım tek hücre moleküler analiz için yetişkin memeli doku sağlam, sağlıklı tek hücre izolasyon zor olabilir. Bu protokol genel işlemlerini açıklar ve yüksek kalite yetişkin tek hücre hazırlıklar sinir sisteminden elde etmek veya bu cilt için gereken sonraki tarafsız tek hücre RNA sıralama ve analiz etkin kalite kontrol eder. Aşağı akım bioinformatic analiz için yönergeler de temin edilmektedir.
Giriş
Yüksek işlem hacmi tek hücre teknolojisi1,2 ve Kullanıcı dostu biyoinformatik araçlarını son on yılda3üzerinde gelişmeler geliştirilmesi ile yüksek çözünürlüklü gen ifade analizi yeni bir alan ortaya çıkmıştır- tek hücreli RNA sıralama (scRNA-Seq). Tek hücre gen ekspresyonu çalışmanın ilk kök hücre veya kanser hücreleri, heterojen tanımlı hücre popülasyonlarının içinde tanımlamak için ya da ulaşılamaz kullanarak olan nadir hücreleri4,5, olasılığını belirlemek için geliştirilmiştir geleneksel toplu RNA sıralama teknikleri. Bioinformatic araçları kimliği roman alt nüfus (Seurat)2, görselleştirme hücreleri bir psuedotime alanı (tek gözlük)6, tanımı içinde veya arasında nüfus (etkin sinyal ağların boyunca sipariş etkinleştirdiyseniz Doğa Manzaralı)7, tek-hücreleri bir yapay 3B alanda (Seurat ve daha fazlası) Meclisi tahmin8. Bu yeni ve heyecan verici analizler ile bilimsel topluluk için kullanılabilir, scRNA-Seq fast-gen ifade analizi için yeni standart yaklaşım haline geliyor.
ScRNA-Seq büyük potansiyel rağmen temiz bir veri kümesi üretmek ve doğru sonuçlar yorumlamak için gerekli teknik skillsets yeni gelenler için zor olabilir. Burada, temel, ancak kapsamlı bir iletişim kuralı, Bütün birincil dokulara görselleştirme tek hücre izolasyon ve yayın için verilerin sunumunu başlayarak (Şekil 1) sunulmuştur. Farklı dokuların enzimatik sindirim ve sonraki mekanik ayrışma duyarlılık onların derecesine göre değişiklik gibi ilk olarak, sağlıklı tek hücre izolasyon zorlu, kabul edilebilir. Bu iletişim kuralı aşağıdaki yalıtım adımları sağlar ve denetim sürecinde önemli kalite kontrol noktaları tanımlar. İkinci olarak, uyumluluk ve tek hücre teknolojisi ve yeni nesil sıralamanın arasında gereksinimleri anlama kafa karıştırıcı olabilir. Bu iletişim kuralı bir Kullanıcı dostu, damlacık tabanlı tek hücreli barkodlama platform uygulamak ve sıralama gerçekleştirmek için yönergeler sağlar. Son olarak, bilgisayar programlama tek hücreli transcriptomic veri analiz etmek için önemli bir önkoşuldur. Bu iletişim kuralı R programlama dili ile Başlarken kaynaklar sağlar ve iki popüler scRNA Seq özel R paketleri uygulama yönergeler sağlar. Birlikte, bu protokol yapılan açık, yorumlanabilir sonuçları elde etmek için scRNA-Seq analiz işlemlerinde yeni gelenler rehberlik eder. Bu protokolü çoğu dokulara fare ayarlanabilir ve önemlisi insan dokusu da dahil olmak üzere diğer organizmaların kullanılmak üzere değiştirilmiş olabilir. Ayarlamalar bağlı olarak doku ve kullanıcı-ecek var olmak gerekli.
Bu iletişim kuralı aşağıdaki sırada akılda tutulması gereken birkaç önemli noktalar vardır; dahil olmak üzere, 1) tüm kalite kontrol yönergeleri adım 1 ve 2 Bu protokol aşağıdaki doğru Toplam hücresini sayı sayar ( Şekil 2 özetlenen sağlarken kalıcı tek hücre süspansiyon ilgi örnek içindeki tüm hücreleri sağlamak için tavsiye edilir ). Bir kez bu sağlanır ve en iyi duruma getirilmiş koşulların tümü yerine getirilirse, kalite kontrol adımları kesilmesine (için zamandan tasarruf - RNA kalite koruyarak ve azaltılması hücre kaybı). Yüksek canlılık tek hücre ilgi dokusundan başarılı yalıtım onaylayan son derece tavsiye herhangi önce aşağı işliyor. 2) aşırı ayrışma teknikleri bazı hücre tipleri strese diğerlerinden daha duyarlı olduğundan, bu nedenle aşağı akım Analizi semptomlarıdır nüfus, farkında olmadan önyargı. Nazik ayrılma gereksiz hücresel kesme ve sindirim olmadan yüksek hücresel verimleri ve doku kompozisyon bir doğru bir şekilde temsil ulaşmak için önemlidir. Kesme kuvvetleri sırasında toz, FACS ve resuspension adımlar gerçekleşir. 3) olarak herhangi bir RNA çalışma ile bu hazırlık sırasında içine mümkün olduğunca örnek küçük ek RNase tanıtmak en iyisidir. Bu yüksek kaliteli RNA korumak yardımcı olacaktır. Temiz araçlar ve değil herhangi bir donanım için durulama ile ribonükleaz inhibitör çözümleri kullanır RNase free ama DEPC tedavi ürünler kaçının. 4) ürünleri mümkün olduğunca hızlı bir şekilde gerçekleştirmek. Bu yüksek kaliteli RNA korumak ve hücre ölümü azaltmak yardımcı olacaktır. Doku diseksiyon uzunluğu ve hayvan sayısı bağlı olarak, aynı anda birden fazla diseksiyonlarının/hazırlıklar başlangıç düşünün. 5) hücreleri buz üzerinde mümkün yüksek kaliteli RNA korumak, hücre ölümü azaltmak ve yavaş hücre zaman sinyal gönderme ve transkripsiyon etkinlik hazırlamak. Gerçi, buz gibi işleme çoğu hücre tipleri için idealdir, bazı hücre türleri (Örneğin, nötrofiller) Oda sıcaklığında işlendiğinde daha iyi performans. 6) kalsiyum, magnezyum, EDTA ve DEPC tedavi ürünleri hücre hazırlık sırasında kaçının.
Protokol
Burada açıklanan tüm iletişim kuralları'e göre vardır ve Calgary Üniversitesi hayvan bakımı Komitesi tarafından onaylanmış.
1. dissociating doku (1 gün)
- Sodyum Fentobarbital (IP, 50 mg/kg) veya hayvan etik protokolüne göre uygun olarak aşırı doz ile fareler ötenazi. O zaman sırt ve bacak fare istenmeyen tüylerden ve etanol-diseksiyon bölgenin sterilize.
- Doku veya microenvironment ilgi incelemek. Bu iletişim kuralı için damlacık tek hücre barkodlama tabanlı transcriptomics yetişkin doku ayrılma takip generalizability göstermek için cilt ve sinir dokuları kullanıyoruz.
- Siyatik sinir için Stratton vd. bulundu detaylı iletişim kuralı kullanın 9. kısa bir süre, fare geri/bacaklar arka bölge uzak deriyi kesme. Uyluk boyunca bir kesi ile steril neşter bıçak yapmak. İyi forseps ve makas maruz ve siyatik sinir kaldırmak için kullanın.
- Arka cilt için Biernaskie vd. buldum detaylı iletişim kuralını kullanan 10. kısaca, dorsal arka deri omuz omuza insizyon yaparak, kıç genelinde ve iyi forseps ve makas kullanarak geri incelemek. Steril neşter bıçak kullanarak cilt (0.5 cm kalınlık) ince dilimler halinde kesin.
- Doku 2 kez ile buz gibi HBSS yıkama ve istenmeyen bag doku, yağ mevduat veya diseksiyon mikroskop altında enkaz kaldırma.
- Dispase (5 mg/mL, 5 U/mL) HBSS olarak 37 ° C'de 30-40 dk içinde dilimleri deri dermiste için yalnızca, float Cerrahi olarak dermis epidermis ayırın. Epidermis atmak ya da daha fazla ilgi eğer tripsin kullanarak ayırmak.
- Örnek 1-2 mm parçalara steril neşter bıçakları kullanarak kıyma ve taze çözdürülen 2 mg/mL soğuk collagenase-IV enzim (2 mg/mL, 125 CDU/mg, F12 medya) içine koyun.
- Sinir için 2 x siyatik sinir başına ~ 500 µL kullanın. Cilt için ~ 8 mL 1 x fare geri cilt başına kullanın.
Not: Dokuları tam olarak collagenase-IV çözümde batık. Herhangi bir sindirim enzimleri işlenmiş, depolanan ve uygun şekilde hazır olmadığını çok önemlidir. Enzimler oda sıcaklığında uzun süre kalması durumunda, tek hücreli yalıtım aşırı mekanik toz gerektirir ve hücre canlılığı azaltmak. Collagenase-IV Ayrıca hücre kültür hücre canlılığı en uygun nerede medyada kadar yapılabilir. Ancak, bu enzim etkinliği değiştirmek veya transkripsiyon imza kadar Kullanıcı tarafından optimize edilmelidir.
- Sinir için 2 x siyatik sinir başına ~ 500 µL kullanın. Cilt için ~ 8 mL 1 x fare geri cilt başına kullanın.
- Enzim bir 37 ° C banyoda nazik her 10 min sallayarak ile 30 dk için örnekte kuluçkaya. 37 ° C'de yerleştirilen bir shaker da uygun bir alternatiftir.
- P1000 pipettor 20 - 30 kez, 30 dk sonrası enzim eklenmesi ile triturate.
- Toz her 30 dakikada çözüm bulutlu görünür ve doku parçaları büyük ölçüde ayrışmış kadar yineleyin.
Not: hücre (Şekil 2b, 2 c) dolu serbest bırakmak olun. Tam sürüm onaylamak için plaka hücreleri Nuc mavi (2 damla 1 mL başına) ve 20 dk sonra tüm çekirdeklerin enkaz yerine tek hücreleri ile ilişkili olduğundan emin olmak için mikroskop altında kontrol edin. Hücre yayını verilen deney her doku türü veya koşul içinde derecesini kontrol etmek önemlidir. Fibrotik doku (yani, kronik yaralanma) veya zarar görmemiş yetişkin doku, hücre yayın akut yaralanma veya embriyonik doku önemli ölçüde değişir. Bazı hücre tipleri böylece tercihen bu hücreleri aşağı akım Analizi hariç--dan diğer, dokusundan serbest bırakmak daha az olasıdır çünkü bu özellikle önemlidir.- Sinir doku 0,5-1,5 için ayırmak saat toplam. Cilt için doku ayırmak için 2 saat Toplam (son bir saatte, kuluçka, DNaz (1 mg/mL) deri örneği ekleyin).
- İki kez 40 µm filtre ile filtre. Buz gibi %1 filtresiyle durulama BSA/HBSS.
- 260 x g 8 min için de santrifüj kapasitesi. Sonra süpernatant kaldırın.
- Hücre pelet % 1 BSA wide-geçişli ucu kullanarak içeren HBSS resuspend ve buza koyun. Resuspension cilt doku birimde dayanmaktadır (800 mg ıslak ağırlık cilt için 800 µL = birim; 10 kilo sinirler için ıslak mg 100 µL hacmi =).
- İsteğe bağlı olarak, düşük resuspension birimi ile başlamak ve sonra gerektiği şekilde akış hızı (saniye başına olaylar) FACS sıralayıcıda göre ayarlayın. En etkin yoğunluğuna Sırala (zaman minimalizing sırasında toplanan hücre sayısını en üst düzeye çıkarmak) koleksiyonları için 3000-7000 olaylar/saniyedir.
- Canlılığı boya kullanıyorsanız, bir subaliquot günahı bir kontrol için dışarı taşır. Sonra 1:15,000 canlılığı boya ekleyin (stok: 20.000 nM/µL) kesme azaltmak için geniş-geçişli ucu kullanarak örnek (1.3 nM/µL son konsantrasyonu) için.
Not: Hücre ölümü her doku türü veya koşul için verilen bir deney içinde derecesini kontrol etmek önemlidir. Bazı hücre tipleri bir örnek içinde--dan diğer, böylece tercihen aşağı akım Analizi hariç ölmek olasılığı daha yüksektir.- Örnek 5-10 dk içinde belgili tanımlık karanlık buzda canlılığı boya ile kuluçkaya. Buz gibi %1 4 mL ekleyin BSA/HBSS örnek için. Aşırı canlılığı boya kaldırmak 8 min için 260 x g, santrifüj kapasitesi. Tedavi subaliquot yok canlılığı ile aynı şekilde günahı denetim boya.
2. ayırma kalıcı ve sağlıklı hücreleri (1 gün)
- FACS tesis sıralama (FACS) parametreleri uygun floresan aktif hücre olduğundan emin olun.
- Önceden adım 1'deki son santrifüj işlemi tamamlandıktan sonra hazır olduğundan emin olmak için FACS makineyi hazırlamak ve koleksiyonu bölmenin soğuk tutulmasını sağlamak buz blokları kullanarak.
- Aşağıdaki parametreleri kullanın: akış hızı: 1.0 (karşılık gelen yaklaşık 10 µL/dak); Filtre: 1.5 ND; Başlık boyutu: 100 µm; İleri dağılım: 80-180 V (olayların boyutu ayırmak için gereken değiştir); Yan dağılım: 150-220 V (değişiklik taneciklik/olaylar şeklinde ayırmak için gerekli olarak); Lazer: 100-400 V (değiştir canlılığı boya olumlu vs negatif olayları ayırt & bu canlılığı boya kontrol karşı kontrol için gerekli); Gates: tüm hücreleri toplanan emin olmak için gereken değişiklikleri yapın. Şekil 2B-2 gbkz.
Not: FACS parametreleri hücre tipleri üzerinde son derece bağımlıdır ve Sıralayıcı istihdam ve bu nedenle Kullanıcı tarafından optimize edilmiş olması gerekiyor.
- 15 mL buz gibi %1 8 mL dar alt tüplerini hazırlayın BSA/HBSS örnek koleksiyonları için. Statik tüp ve yüzey gerilimi içinde toplama verimliliği etkiler. Tüpler koleksiyonları arasında sıvı yüzeyinin arabirim sağlamak için önce ters çevir ve tüp iç nemli olduğunu.
Not: Eğer çok düşük hücre sayıları ile çalışma, küçük toplama beden uygun olarak ayarlayın. - Bir kez tüm hücreler toplanır, örneği ' 260 x g 8 dk santrifüj kapasitesi.
Not: Santrifüjü önce % 1 eklemek yıkama/hücreleri itmek için BSA/HBSS aşağı yan yüzeyden ve ters çevir'i / tüp hemen FACS sonra karışımı. - Hücre pelet %1 BSA/HBSS ve buz üzerinde tutun resuspend. Adım 3 işleme ile uyumlu olan örnek başına maksimum ses 33,8 µL, yani son hücre seyreltme/resuspension birim 33,8 µL ideal cep numarası almak uygun olduğundan emin olun. Bu adım için diğer seyreltme ortam seçenekleri (ve tüm önceki dilutions %1 BSA/HBSS) DMEM, içerir ve yukarı % 40 serum için ancak kalsiyum, magnezyum veya EDTA içeren reaktifler uzak.
- Hücreler buz üzerinde en az bir süre bırakın. İdeal iş arkadaşı tüm ekipmanları ve Kimyasalları (adım 3) için aşağıdaki adım adım 2 son adımları sırasında hazır olmalıyız.
- Hücre hazırlık kritik kontrol
- Tahminleri FACS elde edilen hücre sayı onaylayın. Doku tipi ve ayrılma bağlı olarak uzunlukları, enkaz ve hücre boyutu ve şekli çok benzer olabilir. Böylece, floresan muhabir kullanılmadığı sürece, FACS tüm enkaz dışlayamazsınız. Bu FACS koleksiyonu olayları (FACS göre) yüzde anlamaya gerçekleştirildikten sonra son hücre sayısı aslında verilen hazırlık (Şekil 2 g) için hücreler vardır tavsiye edilir. Bir hemasitometre veya otomatik hücre sayacı (iki kez tekrar) kullanan hücre sayısı gerçekleştirmek ve FACS makine göre toplanan toplam olaylar tarafından temsil edilen hücrelerin yüzdesini hesaplamak.
- Hücre hazırlık doğrulayın. Bunu doğrulamak hiçbir büyük parçacıklar (> 100 µm) mevcut ekipman aşağı akım adımda yapışmasına neden şunlardır. Enkaz yetersiz kaldırma tek hücreli mikrosıvısal çip tıkanma riski. Kalan hücreleri ile Nuc mavi (yukarıdaki gibi) hiçbir büyük enkaz parçaları mevcut olduğundan emin olmak için plaka. Bu-ecek da bırakmak hücrelere tekil onay için (yani, birbirine yapışmasını değil) o aşağı akım tek hücre genetik analiz güven veren temsil eden birden çok hücre yerine tek hücre.
- Hücre sayı sırası için karar: erişkin doku kaynaklı hücre sayı içine belgili tanımlık sistem aynı anda çalıştırabilirsiniz 8 örnekleri ile yüklenen örnek başına büyük bir aralıktır. Yazarlar herhangi bir yerde 500 – 50.000 hücreleri örnek başına yüklenen ve kaliteli scRNA Seq veri kümeleri elde. Yüklemek için en uygun cep numaraları ile ilgili olarak daha fazla tartışma tartışma bölümünde bulunabilir. Sıralı hücre sayıları son çıktı ağır tek hücreleri izole kalitesine bağlı. 10.000 yetişkin doku kaynaklı hücreler yükleme herhangi bir yerde 1000 4000 sıralı hücrelere (10-%40 dönüş) dönebilirsiniz. Yüksek hücre sayıları (~ 10.000 hücreleri, bu sistem için önerilen en yüksek) sıralama ilgilenen varsa, 25.000-100,000 hücreleri yükleme gerekli olacaktır.
3. GEM (emülsiyon jel boncuk) üretimi ve barkodlama (1 gün)
Not: Adım 3-6 Bu iletişim platformu ile birlikte 10 X Genomics tarafından üretilen en yaygın microdroplet tabanlı tek hücreli, kullanılmak üzere tasarlanmıştır. Ayrıntılı yönergeleri için adım 3 ve 4 üreticinin özetlenen (Krom tek hücre 3' protokolü bakın)11,12 Protokolü ve bu protokolü ile birlikte izlenmesi gerekir. En iyi sonuçlar için adım 3 ayrılma (adım 1) ve hücre izolasyon (adım 2) adımları hemen sonra bu protokolün 1 gün tamamlanmalıdır.
- Üreticinin iletişim kuralı 11,12göre çip hazırlamak. Bu tek hücreli platformu microdroplet dayalı ayrı ayrı her hücrenin transcriptome dizin için bu örnekleri ~ 750.000 barkod teknolojisini kullanır. Bu jel boncuk nerede oluşturulan cDNA paylaşmak ortak bir barkod emülsiyonlar (taşlar) içinde içine hücreleri bölümleme tarafından sağlanır. Mücevher üretimi sırasında oluşturulan taşlar çoğunluğu (% 90-99) kalan, süre hiçbir hücre çoğunlukla içerir böylece hücreleri teslim edilir, tek bir hücre içerir.
- Çip çip tutucuya yerleştirin.
- Hücre ana mix buz üzerinde hazırlamak.
- % 50 gliserol kullanılmayan wells için ekleyin ve iyi 1, 90 µL hücre ana karışımı ekleyin jel boncuklar petrol için de 3 bölümleme de 2 ve 270 µL için 90 µL.
- Çip ile conta kapak.
- Belgili tanımlık küçük parça yük ve bir tek hücreli denetleyicisi çalıştırın.
- Tepsi dışarı atmak, çip tepsisine yerleştirin, tepsiyi geri çek ve Playtuşuna basın. Astar (1 sıralama astar okuyun), kısmi bir Illumina R1 sıra içeren bir tek hücre 3' jel boncuk içinde bir mücevher içerir 16 nükleotit (nt) 10 x Barcode, bir 10 nt benzersiz moleküler tanımlayıcı (UMI) ve poli-dT astar sıralaması. Çalışma sırasında jel boncuklar denetleyicisi serbest ve hücre lysate ve ana karışımı ile karışık.
- Örnek ve PCR tüp yerde 100 µL toplamak.
- Önceden ayarlanmış PCR yer PCR tüpleri makine ve PCR kiti göre çalıştırın. Kuluçka, mücevher içerir tam uzunlukta, barkodlu cDNA poli-adenylated mRNA üzerinden.
- Çalıştır sonraki adıma önceki önce 1 hafta-20 ° C'de gecede yerleştirin.
4. temizlik, güçlendirme, Kütüphane İnşaat ve Kütüphane miktar (2 günden itibaren)
Not: Ayrıntılı yönergeleri adım 4 için üreticinin iletişim kuralı 11,12, içinde açıklanmıştır ve bu iletişim kuralı ile birlikte gelmelidir.
- Silane manyetik boncuklar artık biyokimyasal reaktifler/astar GEM tepki karışımı kaldırmak için kullanın.
- Yükseltmek tam uzunlukta, barkodlu cDNA Kütüphanesi yapımı için yeterli yığın üretmek için.
- DNA verim değerlendirmek. Kütüphane inşaat önce DNA verim örnek değerlendirmek. Bu aşağı akım PCR adım (örnek dizin PCR Kütüphane inşaat sırasında) kullanmak için kaç devir belirler. Harekete geçirmek Birleşik bağlı olarak (Örneğin, yaralı denetim vs, vs.), hücre tipi ve hücre verim değişebilir, belirli bir örnek RNA içeriğe bağlı olarak önerilen döngüsü sayısı değişebilir.
- Sıralama ~ 3.000 doku kaynaklı hücreler için (harekete geçirmek Amerika'ya alakasız), yazarlar 14 döngüleri bulduk (örnekler: ~ 10-100 ng DNA) standarttır.
- Bir Bioanalyzer DNA analizi için kullanın. Başvurmak için Kullanım Kılavuzu13.
- Örnek parçası ve DNA boyutunu seçin. Kütüphane inşaat önce uygun cDNA amplicon boyutu elde etmek için enzimatik parçalanma ve boyut seçimi iletişim kurallarını kullanın.
- Örnek kütüphane yapımı için hazır olun. R1 (1 astar sıra okuyun) molekülleri için GEM kuluçka sırasında eklenirken; P5, P7 (bir örnek dizini) ve R2 kitaplık inşaat sırasında eklenir (2 astar sıra okuyun).
- DNA verim değerlendirmek. Çoğu sıralama imkanları DNA verim ve kalite bilgileri içeren son kütüphaneleri sunulması gerekir. Böylece, tüm protokol ve sıralama tesisine taşınması önce tamamlanmasının ardından bioanalyzer çalıştırın.
- Örnekleri-80 ° C'de 2 aya kadar saklayın.
- Sıralama önce bir DNA miktar kit kullanarak örnekleri ölçmek. Bu sıralama tesisinde yapılabilir.
5. Kütüphane (3 günden itibaren) sıralama
Not: tek hücreli transcriptome barkod platformu bu protokol için kullanılan başlangıç ve bitiş P5 ve P7 dizileri ile Illumina uyumlu eşleştirilmiş uç kütüphaneleri oluşturur. Hücre türü kimlik 10.000-50.000 okuma/hücre15,16az olabilir çözmek Minimum Derinlik gerekli, ancak ~ 100.000 okur/hücre (bazı hücre göz önünde bulundurarak yetişkin içinde vivo hücreler için bir en uygun maliyet-kapsama ticaret-off olarak önerilir türleri veya en az aktif hücre Birleşik doygunluk 30.000-50.000 okuma/hücre ulaşacak).
- Kuru buz uygun bir Illumina sequencer ile donatılmış bir sıralama tesise cDNA arşivini taşıma.
- Sıralama tesis için aşağıdaki bilgileri sağlayın:
- Örnek ayrıntıları: örnek her kitaplığa; karşılık gelen dizin kimliği türler; genomik veritabanı için birincil derleme (yani, GRCm38 fare için); parça boyutları bioanalyzer (bp) 200 ve 9.000 arasında; gösterilen electropherogram cDNA konsantrasyon (ng/µL) ve toplam Kütüphane konsantrasyonu (200-1400 ng toplam verimi arasındadır); örnek (µL) hacmi.
- Sıralama istekleri sağlar: bir DNA miktar kiti; kullanarak örnekleri ölçmek Bağdaştırıcı/Dizin türü (TruSeq DNA); Plaka tipi (Eppendorf twin.tec, DNA için önerilen etek tam -); teknoloji/kitaplık türü (10 x, tam sıralama yönergeleri ve döngüsü öneriler)17sıralanıyor.
- Sığ sıralama (isteğe bağlı) çalıştırın: birden fazla biyolojik örnekleri analiz çalışmaları yararına tüm örneklerini gelen verileri içeren bir tek gen-barkod matris oluşturmak için örnekleri (toplama) Havuzu. Havuzu oluşturma zaman örnekleri arasında toplu iş etkileri en aza indirmek için farklı kitaplıklar arasında okuma derinlik standart olacak. Bunu yapabilmek için tek hücre sayıları doğru bir yaklaşım gereklidir. MiSeq sequencer sığ sıralama sağlayacak ve doğru hücre tahminleri elde etmek için bir düşük maliyetli, pratik bir yoldur.
Not: bir çalışma MiSeq SR50 sequencer kullanarak doğru yaklaşık 20.000 hücreleri tahmin etmek için yeterli kapsama alanı sağlar. Bu çalıştırmak için benzersiz her barkod kurtarılan UMI sayısı yaklaşık. Şekil 3a, bir örnek (örnek 1.6) çıktı (.csv) başlığı, Bar kodları listesi gösterilir ve onun karşılık gelen UMI güvenle eşlenen okuma tarafından belirlenen sayar.- Programlama dili R ile bilgi sahibi olmak için bioinformatician başvurun. DataCamp eğitim için daha fazla bilgi18bakın.
- Ham veri bir şablon19sağlanan R komut dosyasını kullanarak sequencer elde edilen değerlendirmek. Ham veri her benzersiz hücre Barkod olarak eşlenen UMIs sayısını ifade eder. Komut dosyası bir .csv dosyası nerede ilk sütun barkodlar listesidir ve ikinci sütun karşılık gelen onun UMI sayar okur. Bu komut dosyası bir arsa (Şekil 3b) yanı sıra barkodlu hücreleri her örnek içinde tahmini sayısını sağlar. UMI sayıları belirli bir örnek için girilen sayısı ilk dik damla üçte bir noktada olması için komut dosyası ayarlayın. Şekil 3b', bu dirsek 3,480 barkodlu hücrelere karşılık gelen yaklaşık 225 UMIs düşüyor.
- HiSeq kullanarak tam kapsamlı sıralama karşılaştırılabilir (3,516 hücreleri başarıyla neredeydin sıralı, Şekil 3 c), sığ sıralama tahminleri 3,480 hücreleri tahmin.
- Kullanım hücre kurtarma yaklaşımları (Kimden adım 5.3) veya kullanım kurtarma grafik lane dağıtım için daha derin sıralama planlamak için üreticinin iletişim kuralı20 bulundu. Orada hücrelerinin (Bu kez durum) her örnek farklı sayılar sığ sıralama ortaya çıkarırsa o zaman lane dağıtım buna göre hesaplanması gerektiğini bu yüzden her örnek karşılaştırılabilir kapsamı, almanız gerekir. (Hangi 8 yolları oluşur) bir HiSeq akışı hücre 2,4 milyar özel eşleştirilmiş son okuma sıra. Örnek akış hücre set-up şekil 3dsunulur.
6. işleme dosyaları okumak
Not: tek bir hücre 3' Bu iletişim kuralını kullanan kütüphane sıralama ham veri ikili temel arama (BCL) biçiminde oluşturur. Hücre Ranger paketi BCL dosyalarından metin tabanlı FASTQ dosyaları oluşturmak, genomik gerçekleştirmek için kullanılır ve transcriptomic hizalamaları, gen sayar, azaltma ve toplama örnekleri. Bu bölümde, kullanıcıların bir sıralama tesisten ham BCL veri indirme ve filtre uygulanmış gen-barkod matrisler için aşağı akım Biyoinformatik hazır oluşturmak önemli adımlar sunulmaktadır.
- Merkezi bir sunucu program çalıştırmak için kullanın. BCL dosya, FASTQ dosyaları ve işleme akış aşağı Biyoinformatik çoğunu önemli işlem gücü gerektirir.
- Sunucusu (veya varsa FASTQ dosyaları) tüm ham okuma dosyalarını indirin.
- Bir merkezi sunucu veya küme üzerindeki bir hesap oluşturmak ve UNIX21ile tanımak için sunucu yöneticinize başvurun.
- Tüm dosyaları sıralama tesisin sunucusundan yüklemek için sunucunun işletim sisteminiz için uygun bir getirme komutunu kullanın.
- Çoğu sıralama özellikleri komut satırından çalıştırmak güvenli bir yol dosyaları indirmek için bir komut sağlar (aşağıdaki örneğe bakın).
- "< >" Eski yerine koymak username ve sağlanan kimlik bilgileri ile komut satırında "< parola >" yer tutucular.
wget - O - "https://your_sequencing_facilitys_server.com/path_to_raw_read_files/--no-kurabiye--Hayır onay sertifikası--sonrası veri ' j_username = Kullanıcı adı & j_password = parola ' | wget--no-kurabiye--Hayır onay sertifikası--sonrası veri ' j_username = Kullanıcı adı & j_password = parola ' - ci -
- Yalnızca dosyaları mutlak bir yol (yani https://your_sequencing_facilitys_server.com/path_to_raw_read_files/) sağlanır, bu yolu getirme komut ekle.
- Dosyaları unzip: ".gz" uzantılı dosyaları son karşıdan yüklediyseniz, bu "gzip" komutunu kullanarak sıkıştırılmış. Açmak için Çalıştır unzip komutu komut satırında (aşağıdaki örneğe bakın).
gunzip raw_read_files.gz - Download cep Ranger geç yorum sunucusuna kendi kendine yeten .tar22.
-
Kritik: Download önce Linux sistem minimum gereksinimleri23karşıladığından olun. En az 8 çekirdekli Intel işlemci 64 GB RAM ve boş disk alanı 1 TB ile olun.
Not: Hücre Ranger önceden inşa edilmiş insan ve kemirgen başvuru transcriptomes sağlar. Bunlar genler GFP24gibi algılamaya cellranger mkref komutu kullanılarak değiştirilebilir.
-
Kritik: Download önce Linux sistem minimum gereksinimleri23karşıladığından olun. En az 8 çekirdekli Intel işlemci 64 GB RAM ve boş disk alanı 1 TB ile olun.
- FASTQ dosyaları sequencer'ın temel arama dosyaları (BCLs) cellranger mkfastq komutunu kullanarak oluşturmak.
Not: Program ham okuma yapması (FASTQ dosyaları) için başvuru genom hizalayın ve aşağı akım analizi için gen-hücre matrisler oluşturmak. Uçbirleştirme kullanan bir başvuru genom okuyan hizalamasını yapar STAR aligner kullanır. Sadece güvenle eşlenen okuma (yani, okuma bir tek gen ek açıklama ile uyumlu) kullanılan UMI sayımı için.- Örneğin, cellranger mkfastq komutu kullanın:
cellranger mkfastq --numarası sample_name = \
--= / yol/için/örneği çalıştırmak \
--csv=csv_file_containing_lane_sample_index.csv
- Örneğin, cellranger mkfastq komutu kullanın:
- Cellranger sayısı tek hücreli gen sayıları üretmek için mkfastq kullanarak oluşturulan FASTQ dosyaları üzerinde çalıştırın.
- Örneğin, cellranger sayısı komutu kullanın:
cellranger sayısı --numarası sample_name = \
--transcriptome refdata-cellranger-mm10-1.2.0 = \
-fastqs = / mutlak/yolu / / fastq/dosyaları \
--örnek same_sample_name_supplied_to_cellranger_mkfastq = \
-localcores = 30
- Örneğin, cellranger sayısı komutu kullanın:
- Multi kitaplığın toplama (isteğe bağlı): örnekleri birleştirmek, cellranger sayısı çıkışlarını cellranger aggr kullanarak havuz için. Birden fazla kitaplık havuza alınmış verileri içeren bir tek gen-barkod matris sonuçlanır. Örnek cellranger aggr komut:
cellranger aggr--numarası sample_name = \
--csv csv_with_libraryID_ & _path_to_molecule_h5.csv = \
--normalize = eşlenen
Not: Kütüphaneler üç normalleştirme modu kullanılarak toplanan (eşlenen, ham, hiçbiri). Eşlenen tüm kitaplıkları eşit sıralama derinlik25kadar derinlik daha yüksek kütüphaneler subsamples önerilir. - Hemen görselleştirme/için veri analizi, 10 x büyüteç hücre tarayıcı26(cellranger sayısı veya cellranger aggr kullanılarak oluşturulan) .cloupe çıktı dosyasını alın.
7. gelişmiş analiz scRNA Seq veri kümeleri
Not: ScRNA-Araçlar3,27, tam scRNA-Seq araçları veritabanı bulunabilir. Aşağıda denetimsiz hücre kümeleme Seurat2 kullanan ve pseudotemporal için bir çerçeve tek gözlük6kullanarak emrediyor. Her ne kadar çok bu iş-ebilmek kılınmak üstünde a yerel bilgisayar, aşağıdaki adımları hesaplama kurumsal bir sunucu kullanarak tamamlanmış olacaktır varsayalım.
- Linux platformu28kullanarak sunucu hesabı üzerine Miniconda en son sürümünü karşıdan yükleyin.
- R conda29kullanarak en son sürümünü yükleyin.
- Bir şablon30sağlanan Seurat R komut dosyasını kullanarak verileri çizmek.
Not: Seurat kalite kontrol denetimleri, kümeleme, fark gen ifade analizi, marker gen tanımlama, dimensionality azaltma ve scRNA-Seq veri görüntülenmesini sağlayan bir R tabanlı araç takımıdır. Satija Lab web sitesi31tarihinde Seurat kodlama ve Rehberler kapsamlı bir açıklaması bulunur. - Bir şablon32sağlanan tek gözlük R komut dosyasını kullanarak verileri çizmek.
Not: Tek gözlük pseudotime üzerinden ifade değişiklikleri görselleştirme sağlar ve hücre kaderi karar altında yatan genlerin tanımlayan R tabanlı başka bir araç takımıdır. Monokl kodlama ve Rehberler kapsamlı bir açıklaması monokl Web sitesi33' te bulunabilir. - R-paketleri kBET gibi test ve veri kümeleri34havuzu oluşturma sonucu olarak toplu iş etkileri düzeltmek için istihdam edilebilir.
8. NCBI'ın coğrafi ve SRA gönderimler
Not: tekrarlanabilirlik ve reanalysis yana ham sıralama dosyalara kolay erişim sağlamak, accessioned çıkanlar online halk için elde edilebilir depoları için önerilen veya makale gönderme öncesinde gerekli. Ulusal Merkezi biyoteknoloji bilgi'nın (NCBI) için gen ifade Omnibus (GEO) ve sıra okuma Arşiv (SRA) yüksek üretilen iş sıralama veri35,36için genel olarak erişilebilen veri depoları vardır.
- NCBI'ın GEO Submitter hesap37için kayıt.
- Üç bileşenden bir müdür/ağıl (GEO submitter'ın Kullanıcı adı olarak başlıklı) içine derlenmiş içeren tam GEO gönderme: 1) meta veri kaydı (bir elektronik tablo proje teslim başına); 2) ham veri dosyaları; 3) işlenmiş veri dosyaları.
- Download ve meta verileri elektronik tablo38tamamlamak. Aşağıdaki genel coğrafi başvuru kılavuzu (GSE100320)39kullanılabilir. Elektronik tablo dizine yerleştirin.
- Yer ham veri dosyaları dizine tüm kitaplıkları için cellranger sayısı komut dosyasından oluşturulan.
- Yer işlenen veri içine belgili tanımlık müdür tüm kitaplıkları için cellranger sayısı komut dosyasından oluşturulan dosyaları (filtre uygulanmış barcodes.tsv, genes.tsv ve matrix.mtx dosyaları).
- GEO submitter'ın FTP sunucusu kimlik bilgileri tüm üç bileşenleri içeren dizin aktarmak için kullanın. Linux/Unix kullanıcıları için: ncftp, lftp, ftp, sftp ve ncftpput kullanılabilir.
- GEO için tüm transferler38bildir.
Sonuçlar
ScRNA-Seq veri analiz etmek için tasarlanmış açık kaynak paketlerini repertuar40 bu paketleri Kullanım R dayalı dillerde3çoğunluğu ile önemli ölçüde artmıştır. Burada, iki bu paketleri kullanarak temsilcisi sonuçları sunulmuştur: denetimsiz gruplandırması değerlendirirken tek gen ifadeye dayalı hücreleri ve hücre heterojenite ve biyolojik deconstruct sorunu çözmek için tek hücreleri bir yörünge boyunca sipariş işler.
Şekil 4 kalite kontrolleri ve aşağı akım Biyoinformatik Analizi ön işleme için Seurat kullanımını göstermektedir. İlk olarak, filtrasyon ve analiz sapkın hücrelerden kaldırılması esastır kalite kontrol etmek için. Bu yapıldı mitokondrial genler, genler (nGene) sayısı ve hücre birini ve aykırı tanımlamak için UMI (nUMI) sayısı yüzdesi görselleştirmek için keman (Şekil 4a) ve dağılım kullanarak çizer (4b rakam). Herhangi bir hücreyi açıkça aykırı birkaç genler, UMI veya mitokondriyal genlerinin yüzde Seurat'ın FilterCells işlevini kullanarak kaldırıldı. Seurat asıl bileşeni (PC) kullandığından analiz puanları dahil etmek için istatistiksel olarak anlamlı PC'ler belirleme kümeleri hücrelere önemli bir adım olduğunu. Dirsek araziler (Şekil 4 c) eksen hangi PC'ler Yaylası 'standart sapma' in PC ötesinde hariç PC seçimi için kullanılmıştır. Kümeleme çözünürlük de 4 (yüksek hücre kümeleri, Şekil 4e için önde gelen yüksek çözünürlük için küme sayısı, 0,4 (düşük çözünürlükte daha az sayıda hücre kümeleri, Şekil 4 diçin lider) arasında değişen değiştirilebilir olduğunu gösteren manipüle edildi ). Yüksek çözünürlükte bu da alt türlerinden veya geçici Birleşik bir hücre nüfusunun gösterebilir, ancak düşük çözünürlükte, bu her küme tanımlanmış hücre tipi temsil ettiğini muhtemeldir. Bu durumda, düşük çözünürlüklü küme ayarlarını daha fazla ifade heatmaps (Seurat'ın DoHeatmap işlevini kullanarak) çözümlemek için verilen küme (Şekil 4f) en çok ifade genlerinde tanımlamak için kullanılmıştır. Bu durumda, en yüksek ifade genlerin her küme benzersiz olarak tanımlanan genler tarafından temsil edildi gösteren kombine, diğer tüm kümeleri karşı belirli bir kümedeki farklı ifade değerlendirirken tarafından tespit edilmiştir. Ayrıca, tek tek aday genler Seurat'ın FeaturePlot işlevini (Şekil 4 g) kullanarak tSNE araziler üzerinde görüntülenir. Bu deşifre için makrofajlar temsil kümeleri olup izin verdi. FeaturePlot kullanarak, bulduğumuz her ikisi de 2 küme ve 4 Cd68 - pan-makrofaj marker ifade.
Tek gözlük paketi Seurat içinde tanımlanan hücre kümeleri corroborating ve hücre yörüngeleri veya pseudotemporal siparişi, oluşturma biyolojik süreçlerin (Şekil 5) özetlemek için kullanıldı. Pseudotemporal sipariş için numuneleri nerede tek hücreli ifade profilleri bir biyolojik saat ders izleyin bekleniyor kullanılabilir. Hücreleri pseudotemporal bir süreklilik ara Birleşik, iki alternatif hücre kaderi, çatallanma noktaları gidermek için sipariş edilebilir ve gen imzalar her kader edinimi temel tanımlar. Öncelikle, Seurat'ın filtrasyon, benzer kalitesiz hücreleri mRNA dağıtım tüm hücreler üzerinde günlük normal ve Şekil 5a' tanımlandığı gibi alt ve üst sınırları arasında düştü böyle çıkarıldı. Sonra tek gözlük'ın newCellTypeHierarchy işlevini kullanarak, tek hücreler sınıflandırıldı ve bilinen sülale marker gen (Şekil 5b, 5 c) kullanarak sayılır. Örneğin, PDGF reseptör Alfa veya Fibroblast belirli Protein 1 ifade hücreleri fibroblastlar tanımlamak için bir ölçüt oluşturmak için hücre türü #1 olarak atanmıştır. Daha sonra bu nüfus (hücre türü #1) fibroblast yörüngeleri deşifre değerlendirildi. Bunu yapmak için tek gözlük'ın farklı GeneTest işlev, hangi aşırı Birleşik Devletleri nüfus içinde temsil eden hücreler karşılaştırıldığında ve kalan hücreleri nüfus (Şekil 5 d) sipariş vermek için farklı genler bulundu kullanılmıştır. Bütün hücreler arasında manifoldu öğrenme yöntemleri (doğrusal olmayan dimensionality azaltma bir tür) uygulayarak, pseudotemporal bir yol boyunca bir koordinat atandı. Bu yörünge sonra hücre devlet (Şekil 5e) ve pseudotime (Şekil 5f) tarafından görüntülenmiştir.

Şekil 1: akış şeması. Tek analiz için birkaç adım mesafede bütün hayvan hazırlık genel kullanıma sunulan bir depo için son veri gönderme için RNA-Seq veri hücre. Emülsiyon (taşlar) jel boncuklar boncuk ile binlerce tek hücre saklayan barkodlu oligonucleotides bakın. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
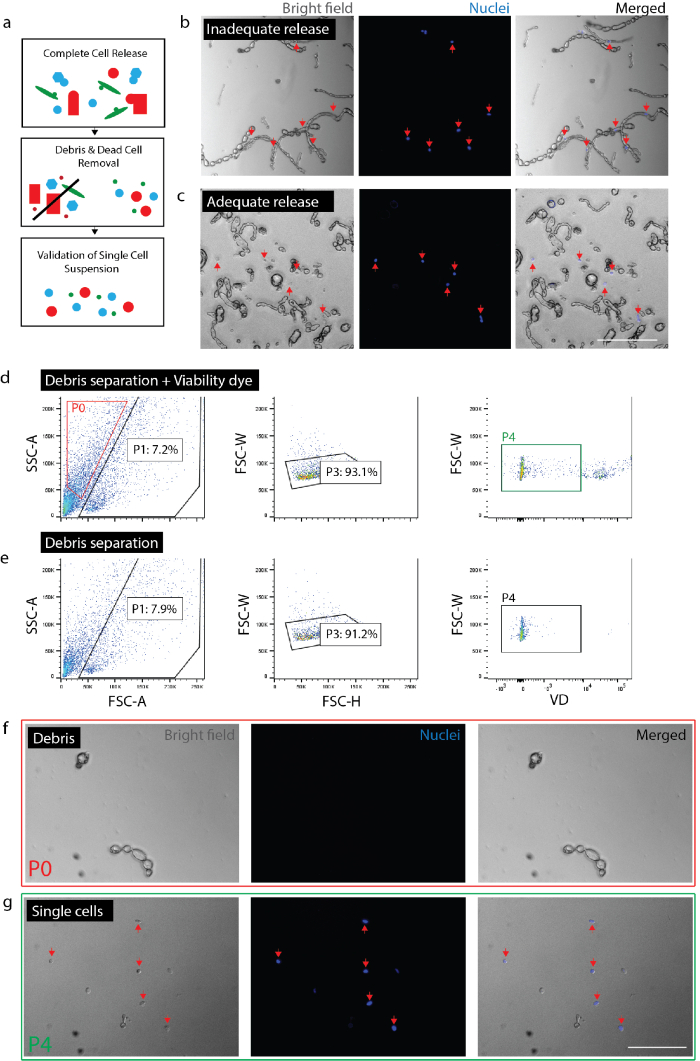
Şekil 2: uygun tek hücre süspansiyon sinir dokusu oluşturma. kalite kontrol (a) karikatür bakış denetler. (b) hücreleri ve enkaz hücrelerle hala enkaz (kırmızı oklar) dahil. (c) enkaz (kırmızı oklar) serbest bırakmak hücrelere. (d) hücre izolasyon FACS tarafından. P0: enkaz kesir; P1: hücre benzeri kesir; P3: duplets dışlanması; P4: canlılık boya (Sytox turuncu) negatif kesir. (e) canlılık boya kontrol. (f) görüntü izole enkaz temsil eden P0 kesir. (g) görüntü izole hücrelerin (kırmızı oklar) temsil eden P4 kesir. (b) (c) (f) ve (g) nükleer boya görüntüleme önce 20 dakika eklendi. Ölçek çubukları: 80 µm. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 3: sığ sıralama 10 X işlenmiş örnekleri kurtarılan hücre sayısı öngörür. (a) MiSeq tarafından oluşturulan csv cep barkod ve onun karşılık gelen UMI listeleme bir örnek (örnek 1.6) güvenle eşlenen okuma tarafından belirlenen sayar. (b) Barkod rütbe Arsa örnek 1,6 için hücre barkodlar bir fonksiyonu olarak UMI sayıma bir düşüş gösterir. Kesik çizgili ve düz çizgiler kesme hücreleri görsel denetim tarafından belirlendiği gibi arka plan arasında gösterir. (c) hücre barkod cep Ranger boru hattı post-HiSeq kullanarak sığ sıralama doğru örnek 1,6 için hücre sayısı yaklaşık olarak ortaya koymaktadır görülmektedir. (d) bir örnek üzerine sığ dayandırılmış bir akış hücreli tuzak hücre tahminleri elde. Sığ sıralama 3480 hücreleri, tahmin beri örnek 1,6 için 1.17 yolları sağlamak için ayrıldı > hücre sıralama kapsamında HiSeq başına 100.000 defa okundu. Not: %100 için tüm yolları eklemeniz gerekir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
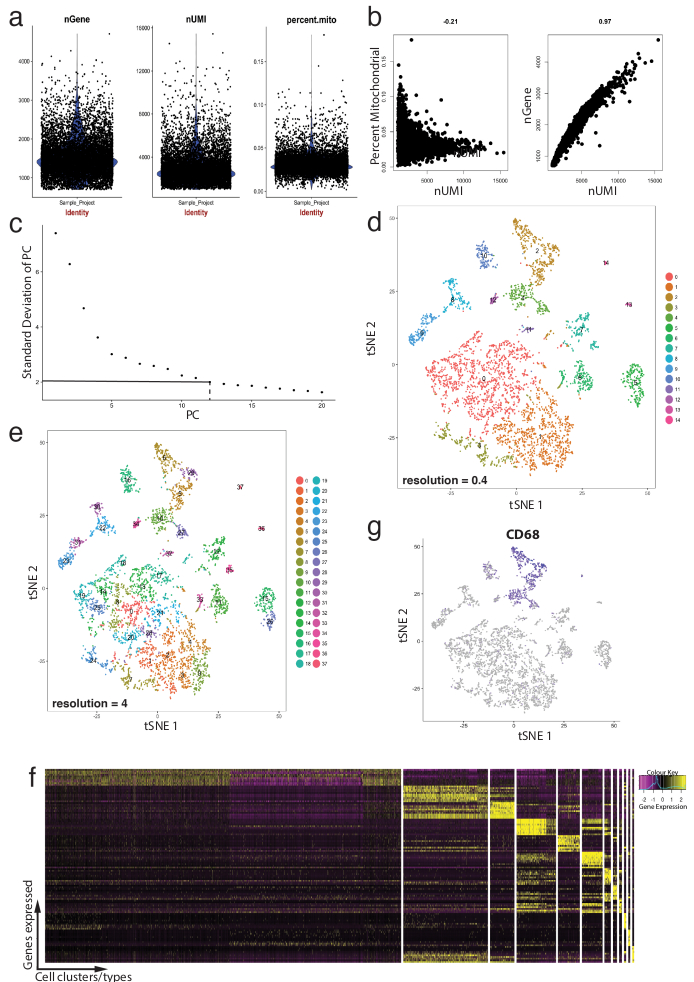
Şekil 4: kalite kontrol ve Biyoinformatik Seurat R paketi kullanarak tek hücreli RNA-Seq DataSet. (a) gen sayısı, benzersiz moleküler tanımlayıcıları (UMIs) sayısı ve mitokondrial genom eşleme transkript yüzdesi kapsayan kalite kontrol ölçümleri ve araziler. (b) örnek gen algılama hücreleri sapkın düzeyde mitokondrial transkript ve UMIs ile çizer. (c) örnek dirsek Arsa istatistiksel olarak anlamlı adet geçici belirlenmesi için kullanılan. Kesik ve nokta kesik çizgiler nerede net bir "dirsek" grafikte ortaya çıkıyor kesme gösterir. PC boyutları bu dirsek önce aşağı akım Analize dahil. (d, e) Grafik tabanlı hücre kümeleri tSNE arsa kullanarak düşük boyutlu uzayda iki farklı çözünürlüklerde görüntülenir. (f) Seurat'ın DoHeatmap işlevini kullanarak bir ifade heatmap görüntülenir üstteki işaretleyiciyi genler (sarı) her küme için. (g) ifade işareti, örneğin, makrofajlar (mor Seurat'ın FeaturePlot işlevini kullanarak) temsil eden Cd68 gen görselleştirme. Bu o küme 2 ve 4 (panel d) Bu veri kümesi temsil eden makrofajlar göstermektedir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 5: hücre kategori ve tek gözlük araç setini kullanarak peudotemporal yörünge sipariş. (a) (UMI sayar anlaşılmaktadır) mRNA dağıtım bir örnek tüm hücreler boyunca teftiş. Sadece hücreleri arasında 0 - mRNA ile ~ 20.000 aşağı akım analiz için kullanıldı. (b, c) Atama ve bilinen sülale hücre işaretleri üzerinde temel hücre tipleri devam ediyor. Örneğin, PDGF reseptör Alfa veya Fibroblast belirli Protein 1 ifade hücreleri pan-fibroblastlar temsil eden hücre türü # 1'e atanmış monokl'ın newCellTypeHierarchy işlevini kullanarak. Farklı hücre türleri bir pasta grafiği (b) ve (c) bir tablo olarak görüntülenmeyecektir. (d) hücre kullanarak bir örnek olarak, hücre görselleştirildiği gen dağılım vs kötü ifade gösterir bir dağılım çizim kullanarak sipariş vermek için kullanılan genlerin türü 1 (fibroblastlar). Kırmızı eğri kesim monokl'ın estimateDispersions işlevini kullanarak ortalama varyans modeli tarafından hesaplanan sipariş vermek için kullanılan genler için gösterir. Bu kesim karşılamak genler aşağı akım pseudotime sipariş vermek için kullanıldı. (e, f) Hücre yörüngeleri azaltılmış bir iki boyutlu uzayda hücrenin "durumu" (e) ve "Pseudotime" (f) tek gözlük tarafından atanan renkli görselleştirme. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
Bu protokolü nasıl tek hücreler uygun hazırlanması tek hücreleri binlerce transkripsiyon heterojen ortaya çıkarmak ve işlevsel devletler veya bir doku içinde benzersiz hücresel kimlikler ayırımcılık gösterir. Protokol does değil istemek floresan muhabir proteinler veya transgenik araçları ve tek hücre izolasyon için ilgi insanlar de dahil olmak üzere çeşitli dokularda uygulanabilir; Her doku göz önünde bulundurarak benzersizdir ve bu protokol ayarlama/değiştirme bir dereceye gerektirir.
Hücre içinde farklı ve son derece dinamik transkripsiyon programlar tek hücreli genomik değerini vurgulamıştık. Yüksek kaliteli RNA izole bir yana, kaliteli veri kümeleri için gerekli kritik örnek hazırlama adım hücreleri tamamen dokudan serbest bırakılır ve hücreleri sağlıklı ve sağlam olduğunu garanti etmektedir. Bu nispeten düz ön kolayca olan hücreleri toplama, nereye hücreler gevşek korunur, dokularda veya hücreleri dolaşan gibi lenfoid doku gibi serbest bırakmak için olduğunu. Ama bu büyük mesafeler, hücre dışı matriks ve genellikle katı hücre iskeleti proteinleri hücre yapısını korumak dahil çevreleyen kapsayan çok gelişmiş hücresel mimarisi nedeniyle diğer yetişkin dokular için zor olabilir. Hücre dolu serbest bırakmak için bile uygun ayrışma teknikleri ile gerekli titiz ve sık sık uzun işleme mRNA kalite ve hücre bütünlüğü değiştirecek potansiyel yoktur. Buna ek olarak, enzim destekli ayrılma için kullanılan yüksek sıcaklıklar da transkripsiyon imzalar29,30etkiler. İletişim kuralı kalite kontrol sunmak için nasıl en iyi duruma getirme bu engelleri aşmak için yardımcı olabilir göstermek için dokulara myelinated yetişkin sinir ve ekstraselüler matriks zengini yetişkin cilt gibi kullanarak denetler kararlıdır.
Herhangi bir scRNA-Seq deneme tasarlarken büyük göz önünde bulundurarak bir sıralama derinlik seçimdir. Sıralama olabilir son derece multiplexed ve okumak derinliği değişir damla-Seq2 5 milyon kadar okuma/hücre14 akıllı-devamı gibi tam uzunlukta bir RNA-Seq yöntemiyle kullanarak çok düşük olması Çoğu scRNA-Seq deneyler ile sıralama 10.000 okuma/hücre, düşük orta yüksek ifade tutanakları hücre tipi sınıflandırma41,42için genellikle yeterli olduğu algılayabilir. Nerede hücreleri binlerce güvenle nadir nüfus atfetmek için gerekli olabilecek karmaşık dokular arasında nadir hücre popülasyonlarının algılamaya çalışırken sıralama maliyetlerinde tasarruf değerinin sığ sıralama derinliğidir. Ama gen ekspresyonu ve ince transkripsiyon imzaları ile ilişkili işlemler hakkında ayrıntılı bilgi gerekli olduğunda sığ derinlik sıralama yeterli değildir. Şu anda, bu genlerin hücrede büyük çoğunluğu 500.000 okuma/hücre ile tespit edilir ama bu protokol bağlı olarak değişebilir ve doku yazın43,44tahmin edilmektedir. Tam uzunlukta transkript sıralama derleme ihtiyacını kaçınmanızı sağlar ve bu nedenle, roman ya da nadir splice türevleri, tespit edebilen sıralama maliyeti genellikle karmaşık doku sistem oluşan hücreler binlerce incelemek için bu tür yaklaşımlar ölçekleme sınırlayın. Bu protokol için tipik olarak açıklanan olanlar daha düşük karmaşıklık ve sığ sıralama gereksinimi gibi buna ek olarak, 3' tek hücreli kitaplıkları öğesini. Açıklanan protokolü kullanılarak oluşturulan kitaplıkları beş desteklenen sıralayıcılar birinde sıralı unutmamak gerekir: 1) NovaSeq, 2) HiSeq 3000/4000, 3) HiSeq 2500 hızlı koşmak ve yüksek çıkış, 4) NextSeq 500/550 ve 5) MiSeq.
Narin doku ve hücre henüz işleme azaltacak alternatif bir yaklaşım tek hücre RNA-Seq, tek hücre RNA-Seq yararlarından bazıları tutar, RNA tek çekirdek45' den analizidir. Bu yaklaşım daha hızlı işleme RNA bozulması ve yeterli çekirdek sürümü sağlamak için daha fazla aşırı önlemler azaltmak sağlar ve böylece büyük olasılıkla belirli bir doku içinde tüm hücreleri temsil eden transkripsiyon profilleri daha güvenli bir yakalama sağlar. Bu tabii ki, sadece transkripsiyon etkinlik böylece bu yaklaşım olabilir veya uygun olmayabilir ilgi deneysel hedefleri nelerdir bağlı olarak belirli bir hücrenin içinde mevcut bir bölümünü sağlar.
Belirli bir doku içinde hücresel kimlikleri tam karakterizasyonu yanı sıra, scRNA-Seq veri kümeleri için en değerli analizleri ara Transkripsiyon Birleşik değerlendirilmesi 'tanımlanmış' hücre popülasyonlarının biridir. Bu ara devletlerin geleneksel toplu RNA-Seq yaklaşımlar mümkün değildi tanımlanan nüfus içindeki hücreleri arasında soy ilişkileri anlayışlar aktarabilir. Birkaç scRNA Seq bioinformatic araç şimdi bu aydınlatmak için geliştirilmiştir. Gibi araçlar, örneğin, kök hücre içine çeşitli terminal kader ya da bağışıklık hücreleri aktif ve durgun durumlar arasında mekik olgunlaşması bir oncogenic/metastatik durumuna geçiş kanser hücrelerinin dahil süreçleri değerlendirebilirsiniz. Hücreleri ince transcriptome farklılıkları da son zamanlarda geliştirilen bioinformatic araçlar FateID gibi sonucuna göre47lineage önyargıları göstergesi olabilir. Hücreleri geçiş arasında farklar zor olabilir bu yana transkripsiyon farklılıklar tespit için ince olabilir, daha derin sıralama gerekli46olabilir. Neyse ki, kapsamı, derin sıralı bir Kütüphane Kütüphane başka bir akışı cep telefonundan yeniden çalıştırarak veri kümesi daha fazla sondalama ilgilenen varsa artırılabilir.
Birlikte ele alındığında, bu iletişim kuralı transcriptionally tek-hücreleri bir deney içinde binlerce yüzlerce profil kullanıcılarının kullanmasına izin veren bir kolay uyum iş akışı sağlar. ScRNA-Seq dataset nesnesinin son kalite en iyi duruma getirilmiş hücre izolasyon, akış sitometresi, cDNA Kütüphanesi nesil ve ham gen-barkod matrisler yorumlanması dayanmaktadır. Bu amaçla, bu protokol çalışmaları çeşitli doku tipleri etkinleştirmek için kolayca değiştirilebilir tüm anahtar adımların kapsamlı bir genel bakış sağlar.
Açıklamalar
Hiçbir açıklamalar
Teşekkürler
Destek personeli UCDNA Hizmetleri tesisinde, hem de hayvan bakım tesisi personeli University of Calgary anıyoruz. Biz onun Biyoinformatik desteği için Matt Workentine ve Jens Durruthy onun teknik destek için teşekkür ederiz. Bu eser yapıldı (RM ve JB), yanında bir CIHR vermek bir CIHR yeni araştırmacı Ödülü JB ve bir Alberta çocuk sağlığı Araştırma Enstitüsü Bursu (js) finanse.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Products | |||
| RNAse out | Biosciences | 786-70 | |
| Pentobarbital sodium | Euthanyl | 50mg/kg | |
| HBSS | Gibco | 14175-095 | |
| Dispase 5U/ml | StemCell Technologies | 7913 | 5 mg/ml |
| Collagenase-4 125 CDU/mg | Sigma-Aldrich | C5138 | 2 mg/ml |
| DNAse | Sigma-Aldrich | DN25 | 10mg/ml |
| BSA | Sigma-Aldrich | A7906 | |
| 15 ml Narrow bottom tube VWR® High-Performance Centrifuge Tubes | VWR | 89039-666 | |
| Sytox Orange Viability Dye | Molecular Probes | 11320972 | 1.3 nM/µl |
| Nuc Blue Live ReadyProbes | Invitrogen | R37605 | |
| Agilent 2100 Bioanalyzer High senitivity DNA Reagents | Agilent | 5067-4626 | |
| Kapa DNA Quantification Kit | Kapa Biosystems | KK4844 | |
| Chromium Single Cell 3' reagents | 10x Genomics | ||
| Equipment | |||
| BD FACSAria III | BD Biosciences | ||
| Agilent 2100 Bioanalyzer Platform | Agilent | ||
| Illumina® HiSeq 4000 | Illumina | ||
| Illumina® MiSeq SR50 | Illumina | ||
| 10X Controller + accessories | 10x Genomics | ||
| Software | |||
| The Cell Ranger | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/overview/welcome | |
| Loupe Cell Browser | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest | |
| R | https://anaconda.org/r/r |
Referanslar
- Shalek, A. K., et al. Single-cell RNA-seq reveals dynamic paracrine control for cellular variation. Nature. 510, 363-369 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161, 1202-1214 (2015).
- Zappia, L., Phipson, B., Oshlack, A. Exploring the single-cell RNA-seq analysis landscape with the scRNA-tools database. bioRxiv:206573. , (2018).
- Dulken, B. W., Leeman, D. S., Boutet, S. C., Hebestreit, K., Brunet, A. Single cell transcriptomic analysis defines heterogeneity and transcriptional dynamics in the adult neural stem cell lineage. Cell Reports. 18 (3), 777-790 (2017).
- Llorens-Bobadilla, E., et al. Single-Cell Transcriptomics Reveals a Population of Dormant Neural Stem Cells that Become Activated upon Brain Injury. Cell Stem Cell. 17 (3), 329-340 (2015).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32, 381-386 (2014).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14, 1083-1086 (2017).
- Mayer, C., et al. Developmental diversification of cortical inhibitory interneurons. Nature. 555 (7697), 457-462 (2018).
- Stratton, J. A., et al. Purification and Characterization of Schwann Cells from Adult Human Skin and Nerve. eNeuro. 4 (3), (2017).
- Biernaskie, J. A., McKenzie, I. A., Toma, J. G., Miller, F. D. Isolation of skin-derived precursors (SKPs) and differentiation and enrichment of their Schwann cell progeny. Nature Protocols. 1 (6), 2803-2812 (2007).
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Agilent Available from: https://www.agilent.com/en-us/library/usermanuals?N=135 (2018)
- Kolodziejczyk, A. A. Single Cell RNA-Sequencing of Pluripotent States Unlocks Modular Transcriptional Variation. Cell Stem Cell. 17, 471-485 (2015).
- Jaitin, D. A., et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 343, 776-779 (2014).
- Pollen, A. A., et al. Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex. Nature Biotechnology. 32, 1053-1058 (2014).
- . Sequencing Requirements for Single Cell 3' Available from: https://support.10xgenomics.com/single-cell-gene-expression/sequencing/doc/specifications-sequencing-requirements-for-single-cell-3 (2018)
- . Introduction to R Available from: https://www.datacamp.com/courses/free-introduction-to-r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics#39; Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Creating a Reference Package with cellranger mkref Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/advanced/references (2018)
- . System Requirements Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/overview/system-requirements (2018)
- . Software Downloads Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest (2018)
- . Aggregating Multiple Libraries with cellranger aggr Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/using/aggregate#depth_normalization (2018)
- . Loupe Cell Browser Gene Expression Tutorial Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/tutorial (2018)
- . A table of tools for the analysis of single-cell RNA-seq data Available from: https://www.scrna-tools.org/ (2018)
- . Downloading conda Available from: https://conda.io/docs/user-guide/install/download.html (2018)
- . r / packages / r 3.5.1 Available from: https://anaconda.org/r/r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . Seurat - Guided Clustering Tutorial Available from: https://satijalab.org/seurat/pbmc3k_tutorial.html (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- Github. . An R package to test for batch effects in high-dimensional single-cell RNA sequencing data. , (2018).
- Edgar, R. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30, 207-210 (2002).
- Leinonen, R., Sugawara, H., Shumway, M. The sequence read archive. Nucleic Acids Research. 39, D19-D21 (2011).
- . GenBank Submission Portal Wizards Available from: https://www.ncbi.nlm.nih.gov/account/register/?back_url=/geo/submitter/ (2018)
- . Submitting data Available from: https://submit.ncbi.nlm.nih.gov/geo/submission/ (2018)
- Shah, P. T., et al. Single-Cell Transcriptomics and Fate Mapping of Ependymal Cells Reveals an Absence of Neural Stem Cell Function. Cell. 173, 1045-1057 (2018).
- Anon, Method of the Year 2013. Nature Methods. 11, 1 (2013).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144, 3625-3632 (2017).
- Wu, Y. E., Pan, L., Zuo, Y., Li, X., Hong, W. Detecting activated cell populations using single-cell RNA-seq. Neuron. 96, 313-329 (2017).
- Zeigenhain, C., et al. Comparative Analysis of Single-Cell RNA Sequencing Methods. Molecular Cell. 65 (4), 631-643 (2017).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nature Methods. 11 (1), 41-46 (2014).
- Habib, N., et al. Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science. 353 (6302), 925-928 (2016).
- Janes, K. A. Single-cell states versus single-cell atlases - two classes of heterogeneity that differ in meaning and method. Current Opinions in Biotechnology. 39, 120-125 (2016).
- Herman, J. S., Sagar, D., Grün, FateID infers cell fate bias in multipotent progenitors from single-cell RNA-seq data. Nature Methods. 15 (5), 379-386 (2018).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır