Method Article
Transcriptómica de célula basada en código de barras gotita de tejidos mamíferos adultos
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe el proceso general y control de calidad controles necesarios para la preparación de células sanas de mamíferas adultos solo para preparaciones de RNA-Seq de celulares solo basada en gota, alto rendimiento. Secuenciación parámetros, lea alineación y análisis Bioinformático unicelular aguas abajo también se proporcionan.
Resumen
El análisis de la expresión génica de células individuales a través de miles de células individuales dentro de un tejido o microambiente es una herramienta valiosa para identificar composición celular, discriminación de Estados funcionales y vías moleculares subyacentes tejido observado funciones y comportamiento de los animales. Sin embargo, el aislamiento de células intactas, sanas de tejidos mamíferos adultos para análisis molecular de la célula descendente posterior puede ser difícil. Este protocolo describe el proceso general y control de calidad verifica necesarios para obtener preparaciones celulares solo adultos de alta calidad desde el sistema nervioso o la piel activa análisis y secuenciación posterior imparcial unicelular RNA. También se ofrecen pautas para el análisis Bioinformático aguas abajo.
Introducción
Con el desarrollo del alto rendimiento unicelular tecnología1,2 y los avances en Bioinformática fácil de usar herramientas en la última década3, ha surgido un nuevo campo de análisis de expresión génica de alta resolución – secuencia de RNA unicelular (scRNA-Seq). El estudio de la expresión génica de células solo primero fue desarrollado para identificar heterogeneidad en las poblaciones de celda definida, como en las células madre o células cancerosas, o para identificar raras poblaciones de células4,5, que eran inalcanzables utilizando técnicas de secuenciación de RNA a granel tradicionales. Herramientas bioinformáticas han permitido la identificación de subpoblaciones novela (Seurat)2, visualización del orden de las células a lo largo de un psuedotime espacio (monóculo)6, definición de redes de señalización activadas dentro o entre poblaciones ( SCENIC)7, predicción de la Asamblea de solo las células en un espacio 3D artificial (Seurat y más)8. Con estos análisis nuevos y emocionantes para la comunidad científica, scRNA-Seq es rápido-convertirse en el nuevo enfoque estándar para análisis de expresión génica.
A pesar del gran potencial de scRNA-Seq, los conjuntos de habilidades técnicas necesarios para producir un conjunto de datos limpia y a interpretar con precisión los resultados pueden ser difícil para los recién llegados. Aquí, un protocolo básico, pero completo, a partir del aislamiento de las células de tejidos toda primarias a la visualización y presentación de datos para la publicación se presenta (figura 1). En primer lugar, el aislamiento de las células sanas se puede considerar un reto, como distintos tejidos varían en su grado de sensibilidad a la digestión enzimática y posterior disociación mecánica. Este protocolo proporciona una guía en estos pasos de aislamiento e identifica puntos importantes de control de calidad durante todo el proceso. En segundo lugar, puede ser confuso entender la compatibilidad y requerimientos entre la tecnología de la célula y secuenciación de próxima generación. Este protocolo proporciona las directrices para implantar una plataforma fácil de usar, basado en la gotita sola celda el código de barras y realizar la secuencia. Finalmente, la programación es un prerrequisito importante para el análisis de conjuntos de datos transcriptómicos unicelular. Este protocolo proporciona los recursos para empezar con el lenguaje de programación R y proporciona orientación sobre la aplicación de dos populares paquetes de R scRNA-Seq-específicos. Juntos, este protocolo puede guiar a los recién llegados en la realización de análisis scRNA-Seq para la obtención de resultados claramente interpretables. Este protocolo se puede ajustar a la mayoría de los tejidos en el ratón y lo importante es que podría modificarse para su uso con otros organismos, incluyendo el tejido humano. Se requerirá de ajustes según el tejido y el usuario.
Hay varias consideraciones a tener en cuenta siguiendo este protocolo; incluyendo 1) siguiendo todas las pautas de control de calidad en los pasos 1 y 2 del presente Protocolo se recomienda para asegurar una suspensión unicelular viable de todas las células dentro de la muestra de interés asegurando precisión total número recuento (resumido en figura 2 ). Una vez que esto se consigue, y si se siguen todas las condiciones optimizadas, las medidas de control de calidad pueden ser bajadas (para ahorrar tiempo - preservando calidad de RNA y la reducción de la célula pérdida). Confirmando el aislamiento exitoso de células alta viabilidad de los tejidos de interés es muy aguas abajo se recomienda antes de cualquier proceso. 2) ya que algunos tipos de células son más sensibles que otras al estrés, técnicas de disociación excesivas inadvertidamente pueden sesgar a la población, por lo tanto confundir análisis aguas abajo. Disociación suave sin corte celulares innecesarios y la digestión es fundamental para el logro de altos rendimientos celulares y una representación exacta de la composición tisular. Las fuerzas de corte se producen durante las etapas de trituración, FACS y resuspensión. 3) como con cualquier trabajo del RNA, es mejor introducir como poco Rnasa adicional en la muestra como sea posible durante la preparación. Esto le ayudará a mantener la alta calidad RNA. Utilice soluciones de inhibidor de la ribonucleasa con enjuague para limpiarlos herramientas y cualquier equipo que no es libre de Rnasa, pero evitar los productos tratada con DEPC. 4) realizar los preparativos lo antes posible. Esto ayudará a mantener la alta calidad RNA y reducir la muerte celular. Dependiendo de la longitud de la disección del tejido y el número de animales, tener en cuenta a partir de preparaciones de disecciones múltiples al mismo tiempo. 5) preparar las células en hielo cuando sea posible para mantener la alta calidad RNA, reducir la muerte celular y el retraso de la célula actividad transcripcional y señalización. Aunque helada es ideal para la mayoría de los tipos de la célula, algunos tipos de células (e.g., neutrófilos) se desempeñan mejor cuando se procesa a temperatura ambiente. 6) Evite productos de tratada con DEPC, EDTA, magnesio y calcio durante la preparación de la célula.
Protocolo
Todos los protocolos descritos aquí están de acuerdo con y aprobaron por el Comité de cuidado Animal de la Universidad de Calgary.
1. disociación del tejido (día 1)
- Eutanasia a ratones con una sobredosis de pentobarbital sódico (i.p., 50 mg/kg) o en su caso según protocolo de ética animal. Luego quitar el pelo indeseado de la espalda y las piernas del ratón y etanol-esterilizar la región de la disección.
- Disecar el tejido o microentorno de interés. De este protocolo, utilizamos tejidos de la piel y los nervios para demostrar la generalización de gota transcriptómica de célula basada en código de barras después de disociación del tejido adulto.
- Para el nervio ciático, use el protocolo detallado en Stratton et al. 9. brevemente, cortar la piel de la región trasera de las espalda, piernas de ratón. Hacer una incisión a lo largo de la longitud del muslo con una hoja de bisturí estéril. Use finas pinzas y tijeras para exponer y eliminar el nervio ciático.
- Para la piel de la espalda, use el protocolo detallado en Biernaskie et al. 10. brevemente, disecar la piel espalda dorsal haciendo incisiones de hombro a hombro, a través de la grupa y hacia atrás utilizando pinza fina y tijeras. Cortar la piel en rodajas finas (0,5 cm de espesor) utilizando una hoja de bisturí estéril.
- Lavar 2 veces con HBSS helada del tejido y retirar tejido conectivo no deseado, los depósitos de grasa o residuos debajo de un microscopio de disección.
- Para la dermis piel flotar las rebanadas en dispase (5 mg/mL, 5 U/mL) en HBSS por 30-40 min a 37 ° C. Quirúrgico, separar la epidermis de la dermis. Deshágase de la epidermis o disociar más utilizando tripsina de interés.
- Moler la muestra en trozos de 1-2 mm con una hojas de bisturí estériles y en enzima colagenasa IV fría de recién descongelada 2 mg/mL (2 mg/mL, 125 CDU/mg, en medios de F12).
- Para el nervio, use ~ 500 μl 2 nervios ciáticos de x. Para la piel, use ~ 8 mL por ratón 1 x nuevo piel.
Nota: Los tejidos deben ser completamente sumergidos en la solución de la colagenasa IV. Es fundamental que cualquier enzimas de digestión son manejadas, almacenadas y preparadas apropiadamente. Si las enzimas se dejan a temperatura ambiente durante largos periodos de tiempo, unicelular aislamiento requieren trituración mecánica excesiva y reducir la viabilidad de las células. Colagenasa IV también puede ser formado en medios de cultivo celular donde la viabilidad de las células es más óptimo. Sin embargo, esto puede alterar la actividad enzimática o transcripcional firma tan debe ser optimizado por el usuario.
- Para el nervio, use ~ 500 μl 2 nervios ciáticos de x. Para la piel, use ~ 8 mL por ratón 1 x nuevo piel.
- Incubar la muestra en la enzima en un baño de 37 ° C durante 30 min con suave agitación cada 10 minutos. Una coctelera colocada a 37 ° C es también una alternativa adecuada.
- Triturate con una pipeta P1000 20 - 30 veces en 30 min adición de la enzima.
- Repita trituración cada 30 minutos hasta que la solución aparece nublado y trozos de tejido en gran parte son disociados.
Nota: Asegúrese de que versión completa de las células (figura 2b, 2C). Para confirmar el comunicado completo, placa de células con Nuc azul (2 gotas por 1 mL) y después de 20 minutos, bajo el microscopio para asegurarse de todos los núcleos están asociados con las células en lugar de desechos. Es fundamental para comprobar el grado de liberación de la célula dentro de un experimento determinado para cada tipo de tejido o condición. En tejido fibrótico (es decir, lesiones crónicas) o tejido adulto ileso, la liberación de las células varía dramáticamente de lesión aguda o tejido embrionario. Esto es especialmente importante debido a que ciertos tipos de células son menos propensos a liberar de los tejidos que otras, así preferencial excepto ésos células de análisis aguas abajo.- Para el nervio, disociar tejido de 0.5-1.5 horas en total. Para la piel, disociar tejido total 2 horas (en la última hora de incubación, agregar ADNasa (1 mg/mL) a la muestra de la piel).
- Filtro doble con un filtro de 40 μm. Enjuague el filtro con helada 1% BSA/HBSS.
- Centrifugue a 260 x g por 8 min. Luego retire el sobrenadante.
- Resuspender el precipitado de células en HBSS conteniendo 1% de BSA con una punta ancha y coloque en hielo. El volumen de resuspensión se basa en el volumen de tejido (peso húmedo 800 mg para la piel = 800 μl volumen; 10 mg húmedo peso para nervios = 100 μl volumen).
- Opcionalmente, empiece con un volumen de resuspensión bajo y ajuste como sea necesario basado en flujo (eventos por segundo) en el clasificador de FACS. La densidad de la especie más eficiente (máximo número de células recogidas mientras minimizar tiempo) para las colecciones es 3.000-7.000 eventos por segundo.
- Si utiliza tinte viabilidad, sacar un subaliquot para un control sin manchas. Añada colorante de viabilidad 1:15,000 (stock: 20.000 nM/μL) muestra (concentración final de 1.3 nM/μL) con una punta ancha para reducir el corte.
Nota: Es fundamental para comprobar el grado de muerte celular dentro de un experimento determinado para cada tipo de tejido o condición. Algunos tipos de células dentro de una muestra son más propensos a morir que otros, por lo tanto preferencial excluidos de posteriores análisis.- Incubar la muestra con el tinte de viabilidad durante 5-10 min en hielo en la oscuridad. Luego agregar 4 mL de helado 1% BSA/HBSS a muestra. Centrifugue a 260 x g por 8 min eliminar el tinte sobrante viabilidad. Tratar la subaliquot con la no viabilidad del tinte sin manchas control de la misma manera.
2. aislamiento de células viables y saludables (día 1)
- Asegúrese de que la instalación de FACS sigue celular apropiado fluorescencia activada clasificación parámetros (FACS).
- Preparar la máquina de FACS por adelantado para asegurarse de que está listo una vez que la centrífuga final en el paso 1 y asegúrese de que el compartimiento de la colección frío utilizando bloques de hielo.
- Utilice los siguientes parámetros: tasa de flujo: 1.0 (corresponde aproximadamente a 10 μl/min); Filtro: 1.5 ND; Tamaño de la boquilla: 100 μm; Dispersión hacia adelante: 80-180 V (cambiar si es necesario para distinguir el tamaño de eventos); Dispersión lateral: 150-220 V (cambiar si es necesario para distinguir granularidad/forma de eventos); Laser: 100-400 V (cambiar si es necesario con el fin de distinguir los acontecimientos negativos de viabilidad tinte positivo vs y cotejar esto con ningún control de tinte de viabilidad); Puertas: Cambiar como sea necesario para asegurar que todas las células se recogen. Ver Figura 2d-2 g.
Nota: Los parámetros FACS son altamente dependientes de los tipos de células y el clasificador empleado y por lo tanto necesita ser optimizado por el usuario.
- Preparar 15 mL fondo estrecho tubos con 8 mL de helado 1% BSA/HBSS para las colecciones de muestra. Estática dentro de tubo y la tensión superficial puede afectar la eficacia de la colección. Invierta los tubos antes de colecciones para interfaz entre la superficie del líquido y el interior del tubo es húmedo.
Nota: Si trabaja con números muy bajos de células, ajuste al recipiente de colección pequeña según sea el caso. - Una vez que se recogen todas las células, centrifugar la muestra a 260 x g por 8 min.
Nota: Antes de centrifugación, añadir 1% BSA/HBSS lavado/empuja las células desde la superficie lateral y mezcla/invertir el tubo inmediatamente después de la FACS. - Resuspender el precipitado de células en 1% BSA/HBSS y mantener en hielo. El volumen máximo por ejemplo que es compatible con el proceso del paso 3 μl 33.8, así que asegurarse de que volumen de dilución/resuspensión celular final es apropiado para obtener el número celular ideal en 33.8 μl. Otras opciones de medios de dilución para este paso (y todas las diluciones anteriores en 1% BSA/HBSS) incluyen DMEM y hasta 40% de suero, pero evitar el calcio, el magnesio o el EDTA que contiene reactivos.
- Dejar las células en hielo para una cantidad mínima de tiempo. Ideal compañero de trabajo debe preparar todos los equipos y reactivos para el paso siguiente (paso 3) durante los últimos pasos del paso 2.
- Celular preparación controles críticos
- Confirman las estimaciones del número de células obtenida de FACS. Dependiendo del tipo de tejido y disociación longitudes, desechos y células pueden ser muy similares en tamaño y forma. Por lo tanto, salvo que un reportero fluorescente, FACS no puede excluir toda la suciedad. Se recomienda que un conteo final después de que colección de FACS se realiza para entender qué porcentaje de los eventos (según FACS) son en realidad células de una determinada preparación (figura 2 g). Realizar recuento de células usando un hemocitómetro o un contador de células automatizado (repetir dos veces) y calcular el porcentaje de células viables que está representado por el total de eventos recogido según máquina de FACS.
- Validar la preparación de la célula. Validar que no hay partículas grandes (> 100 μm) están presentes como que pueden atascar al equipo en pasos posteriores. Inadecuada eliminación de residuos corre el riesgo de obstrucción el chip microfluídico unicelular. Placa de células restantes con Nuc azul (como arriba) para asegurarse de que no fragmentos de basura grande están presentes. Esto también permitirá la confirmación de que las células son singular (es decir, no pegadas) dar confianza que análisis genético de la célula abajo representa las células en lugar de varias celdas.
- Decidir sobre el número de células a la secuencia: hay una amplia gama de números de células adultas derivadas de tejido por cada muestra que se puede cargar en el sistema con hasta 8 muestras que se pueden ejecutar al mismo tiempo. Autores han cargado en cualquier lugar de 500 – 50.000 células por muestra y obtiene conjuntos de datos de buena calidad scRNA Seq. Más discusión sobre el número de células más apropiado para cargar se puede encontrar en la sección de discusión. El resultado final de un número secuencial de células depende fuertemente de la calidad de la solo-células aisladas. Carga 10.000 células adultas derivadas de tejido puede devolver en cualquier lugar de 1.000 a 4.000 células secuenciadas (10-40% de retorno). Si está interesado en secuencia un número alto de células (~ 10.000 células, el número máximo recomendado para este sistema), luego 25.000-100.000 células de carga se requiere.
3. gema (Gel grano en emulsión) generación y código de barras (día 1)
Nota: Pasos 3-6 de este protocolo están diseñados para utilizarse en conjunto con la plataforma más común del unicelular microdroplet base, fabricada por 10 X Genomics. Directrices detalladas para pasos 3 y 4 se describen en el fabricante (véase el protocolo de célula cromo 3')11,12 de protocolo y deben seguirse junto con este protocolo. Paso 3 para obtener mejores resultados, debe completarse inmediatamente después de la disociación (paso 1) y medidas de aislamiento (paso 2) de la célula el día 1 del presente Protocolo.
- Preparar el chip según protocolo 11,12 el fabricante. Esta plataforma unicelular microdroplet utiliza tecnología códigos de muestras ~ 750.000 por separado índice transcriptoma de cada célula. Esto se consigue dividiendo las células en grano de Gel en emulsiones (GEMs) donde cDNA generada comparten un común código de barras. Durante la generación de la joya, las células se entregan para que la mayoría (90-99%) de ahorro generado no contiene las células, mientras que el resto, en su mayor parte, contienen una sola célula.
- Coloque el chip chip.
- Preparar mezcla maestro celular en hielo.
- Añadir glicerol al 50% en los pocillos no utilizados y agregar 90 μl de la mezcla principal de la célula al pozo 1, 90 μl de perlas de gel bien 2 y 270 μl de partición aceite bien 3.
- El chip con la Junta de la cubierta.
- El chip de cargar y ejecutar en un controlador de unicelulares.
- Abrir la bandeja, colocar el chip en bandeja, replegar la bandeja y pulse Play. Un sola célula 3' gel grano en una joya incluye cartillas que contienen una secuencia parcial de Illumina R1 (leer 1 cartilla de secuenciación), un 16 nucleótidos (nt) 10 x Barcode, 10 nt identificador Molecular única (UMI) y una secuencia de cartilla de poli-dT. Durante el funcionamiento, perlas de gel en el controlador son liberados y mezclados con mezcla de lisado y maestro de la célula.
- Recoger 100 μl de la muestra y colocarla en un tubo PCR.
- Tubos de PCR de lugar en PCR preestablecido la máquina y ejecutan la PCR según el kit. Tras la incubación, se incluyen gemas larga duración, con código de barras cDNA de mRNA de poli-adenylated.
- Tras la carrera, lugar a-20 ° C durante la noche para hasta 1 semana antes precede al paso siguiente.
4. limpiar, amplificación, construcción de biblioteca y biblioteca cuantificación (día 2)
Nota: Directrices detalladas para 4 pasos se describen en protocolo 11,12 el fabricante y deben seguirse junto con este protocolo.
- Utilizar granos magnéticos de silano para quitar restos reactivos bioquímicos/cartillas de mezcla de reacción de la joya.
- Amplificar cDNA del larga duración, con código de barras para generar la suficiente masa para la construcción de la biblioteca.
- Evaluar el rendimiento de ADN. Antes de la construcción de la biblioteca, evaluar el rendimiento de la DNA de la muestra. Esto determinará cuántos ciclos para utilizar en el paso descendente de PCR (muestra índice de PCR durante la construcción de la biblioteca). Dependiendo del contenido de RNA de una muestra dada, que puede variar dependiendo de Estados de activación (p. ej., control vs heridos, etcetera), tipo de células y producción de la célula, puede cambiar el número de ciclo recomendado.
- Para la secuencia ~ 3.000 tejido células derivadas (irrelevantes para los Estados de activación), los autores han encontrado que 14 ciclos (muestras: ~ 10-100 ng de ADN) es estándar.
- Utilice un equipo Bioanalyzer para análisis de ADN. Consulte al usuario guía13.
- Fragmento de la muestra y seleccionar el tamaño del ADN. Antes de la construcción de la biblioteca, utilizar protocolos de selección de tamaño y fragmentación enzimática para obtener tamaño de amplicón de cDNA correspondientes.
- Preparar la muestra para la construcción de la biblioteca. Mientras que R1 (leer 1 secuencia de cartilla) se agrega a las moléculas durante la incubación de la joya; P5, P7 (un índice de la muestra), y R2 (leer secuencia de primer 2) se agregan durante la construcción de la biblioteca.
- Evaluar el rendimiento de ADN. Más servicios de secuenciación exigen presentación de finales bibliotecas que incluyen información de rendimiento y calidad de ADN. Por lo tanto, ejecute el equipo bioanalyzer tras la conclusión del protocolo completo y antes de transportar a un centro de secuenciación.
- Almacenar las muestras a-80 ° C hasta por 2 meses.
- Antes de la secuencia, cuantificar las muestras usando un kit de cuantificación de ADN. Esto puede hacerse en el establecimiento de la secuencia.
5. Biblioteca de secuenciación (día 3)
Nota: La plataforma de código de barras de transcriptoma unicelular utilizada en este protocolo genera bibliotecas de extremo apareado Illumina-compatible comenzando y terminando con secuencias de P5 y P7. Aunque la profundidad mínima necesaria para resolver la identidad de tipo celular puede ser tan sólo 10.000 – 50.000 lecturas/celular15,16, ~ 100.000 celular Lee se recomienda como un óptimo equilibrio de cobertura costo para adultos células in vivo (teniendo en cuenta algunas células tipos o Estados mínimamente activada de la célula llega a saturación en 30.000-50.000 lecturas/celular).
- Las bibliotecas de cDNA en hielo seco a un centro de secuenciación equipado con un secuenciador de Illumina apropiado de transporte.
- Proporcione la siguiente información al centro de la secuencia:
- Proporcionar los datos de muestra: muestra el ID de índice correspondiente a cada biblioteca; especies; base de datos genómica para ensamblado primario (es decir, GRCm38 para ratón); electroferograma mostrando tamaños de fragmento del equipo bioanalyzer (entre 200 y 9.000 bp); cDNA concentración (ng/μL) y biblioteca total (total rendimientos entre 200 – 1400 ng); volumen (μL) de muestra.
- Proporcionar las peticiones de la secuencia: cuantificar las muestras usando un kit de cuantificación de ADN; tipo de adaptador/índice (DNA TruSeq); tipo de placa (Eppendorf twin.tec, falda - recomendado para ADN); secuenciación/la biblioteca de tecnología tipo (10 x, secuenciación completa instrucciones y recomendaciones de ciclo)17.
- Ejecutar la secuencia superficial (opcional): estudios de análisis de múltiples muestras biológicas podrán aprovechar la agrupación de las muestras (agregación) para generar una matriz de gen único-código de barras que contiene datos de todas las muestras. Para minimizar efectos de lote entre muestras al agrupamiento, debe estandarizarse la profundidad de lectura entre diferentes bibliotecas. Para ello, es necesaria una exacta aproximación de números de la célula. El secuenciador de MiSeq permitirá secuencia superficial y es una manera rentable y práctica para obtener estimaciones exactas de la célula.
Nota: Una carrera con un secuenciador MiSeq SR50 proporciona cobertura suficiente para estimar con exactitud aproximadamente 20.000 células. Esta carrera será aproximado el número de UMI recuperado para cada código de barras único. En la figura 3a, se muestra el encabezado de una salida de ejemplo (muestra 1.6) (.csv), listado de códigos de barras y su correspondiente UMI cuenta según lecturas asignadas con confianza.- Consulte con un Bioinformático para familiarizarse con el lenguaje de programación de R. Consulte los tutoriales de DataCamp para más información18.
- Evaluar datos obtenidos desde el secuenciador usando el script proporcionado de R como una plantilla de19. Datos en bruto se refieren a número de UMIs asignada a cada código de barras único de la célula. La secuencia de comandos Lee un archivo .csv donde la primera columna es una lista de códigos de barras y la segunda columna son su correspondiente UMI cuenta. Este script proporciona un diagrama (figura 3b) así como el número de células con código de barras en cada muestra. Ajustar el script para que sea el número entrado de UMI cuentas para una muestra determinada en el punto de una tercera parte de la primera gota escarpada. En la figura 3b, este codo cae alrededor 225 UMIs correspondiente a 3.480 células con código de barras.
- Comparable a la secuenciación de toda profundidad usando HiSeq (donde las 3.516 células fueron correctamente ordenados, figura 3 c), secuencias someras estimaciones predicen las 3.480 células.
- Uso celular recuperación aproximaciones (desde paso 5.3) o use la tabla de recuperación se encuentra en protocolo20 al plan de distribución de carril para la secuencia más profunda el fabricante. Cada muestra debe recibir cobertura comparable, por lo que si secuencia superficial revela que existen diferentes números de células en cada muestra (que suele ser el caso) luego distribución de carriles se debe calcular en consecuencia. Una célula de flujo de HiSeq (que consta de 8 carriles) puede secuencia lecturas de medida final emparejados hasta 2,4 billones. Configuración de célula de flujo de ejemplo se presenta en la Figura 3d.
6. proceso lee archivos
Nota: Secuenciación de una sola célula 3' Biblioteca mediante este protocolo genera datos sin procesar en formato binario llamado base (BCL). El Cell Ranger paquete se utiliza para generar archivos FASTQ basado en texto desde archivos BCL, realizar genómica y transcriptómicos alineaciones, gene cuentas, demultiplexado y agregación de las muestras. En esta sección, se presenta los pasos clave que permiten a los usuarios descargar datos sin procesar de BCL de un centro de secuenciación y generar matrices de filtrado gene-código de barras listos para bioinformática aguas abajo.
- Utilizar un servidor centralizado para correr el programa. Archivos BCL, FASTQ archivos y la mayoría de la bioinformática downstream procesamiento exige potencia de procesamiento significativo.
- Descargar todos los archivos leemos raws en el servidor (o FASTQ archivos si están disponibles).
- Consulte con el administrador del servidor para configurar una cuenta en un servidor centralizado o cluster y para familiarizarse con Unix21.
- Utilice un comando fetch apropiado para el sistema operativo del servidor para descargar todos los archivos del servidor de la secuencia de la instalación.
- Más servicios de secuenciación proporcionan un comando para descargar archivos desde un camino seguro que se puede ejecutar desde la línea de comandos (ver ejemplo abajo).
- Reemplazar el "< nombre de usuario >" y "< contraseña >" marcadores de posición en la línea de comandos con las credenciales proporcionadas.
wget - O - "https://your_sequencing_facilitys_server.com/path_to_raw_read_files/--no-cookies-certificado de no retención--datos post ' j_username = nombre de usuario y j_password = password' | wget--no-cookies-certificado de no retención--datos post ' j_username = nombre de usuario y j_password = password' - ci -
- Si sólo una ruta absoluta a los archivos se proporciona (por ejemplo, https://your_sequencing_facilitys_server.com/path_to_raw_read_files/), introduzca este camino en un comando fetch.
- Descomprimir archivos: si descargaron archivos terminen con la extensión ".gz", se ha comprimido usando el comando "gzip". Para descomprimir, ejecutar descomprimir comando en la línea de comandos (ver ejemplo abajo).
gunzip raw_read_files.gz - Descargar la última versión de Cell Ranger en el servidor como un.tar independientes22.
-
Crítica: Antes de descargar, asegúrese de que el sistema Linux cumple con requisitos mínimos23. Garantizar un mínimo de procesador de Intel de 8 núcleos con 64 GB de RAM y 1 TB de espacio libre en disco.
Nota: Cell Ranger proporciona transcriptomas referencia preconstruidos de humanos y roedores. Estos pueden ser modificados utilizando cellranger mkref comando para detectar genes como GFP24.
-
Crítica: Antes de descargar, asegúrese de que el sistema Linux cumple con requisitos mínimos23. Garantizar un mínimo de procesador de Intel de 8 núcleos con 64 GB de RAM y 1 TB de espacio libre en disco.
- Generar FASTQ archivos desde archivos de llamada base del secuenciador (BCLs) utilizando el comando de mkfastq de cellranger.
Nota: El programa alinear raw Lee (desde archivos FASTQ) un genoma de referencia y generar matrices de célula del gene para análisis posteriores. Utiliza el alineador de la estrella que realiza alineación consciente de empalme de Lee para un genoma de referencia. Sólo lee con confianza asignada (es decir, Lee compatible con la anotación de un solo gen) se utiliza para el recuento de UMI.- Por ejemplo, utilice el comando de mkfastq de cellranger:
cellranger mkfastq - id = sample_name \
--ejecutar = / ruta/a/muestra \
--csv=csv_file_containing_lane_sample_index.csv
- Por ejemplo, utilice el comando de mkfastq de cellranger:
- Ejecute cellranger Conde FASTQ archivos generados usando mkfastq para generar cuentas sola célula gene.
- Por ejemplo, utilice el comando de la cuenta de cellranger:
Conde de cellranger - id = sample_name \
--transcriptoma = refdata-cellranger-mm10-1.2.0 \
--fastqs = / absoluta/ruta/a/fastq/archivos \
--muestra = same_sample_name_supplied_to_cellranger_mkfastq \
--localcores = 30
- Por ejemplo, utilice el comando de la cuenta de cellranger:
- Multi-biblioteca agregación (opcional): al combinar las muestras, salidas de la cuenta de cellranger usando cellranger aggr en la piscina. Esto resulta en una matriz de gen único-código de barras que contiene los datos de múltiples bibliotecas. Ejemplo cellranger aggr de comando:
cellranger aggr - id = sample_name \
--csv = csv_with_libraryID_ & _path_to_molecule_h5.csv \
-normalizar = asignado
Nota: Las bibliotecas pueden agruparse utilizando tres modos de normalización (asignadas, crudo, ninguno). Asignado se recomienda como sub-muestras bibliotecas de profundidad más alta hasta que todas las bibliotecas tienen igual secuencia profundidad25. - Para la inmediato visualización y análisis de datos, importar el archivo de salida .cloupe (generado mediante la cuenta cellranger o cellranger aggr) en 10 x lupa celular navegador26.
7. avanzado análisis de conjuntos de datos scRNA-Seq
Nota: Una base de datos de herramientas de scRNA-Seq completa puede encontrarse en scRNA herramientas3,27. A continuación es un marco para células sin supervisión clustering con Seurat2 y pseudotemporal pedidos con monóculo6. Aunque mucho de este trabajo puede realizarse en un equipo local, los siguientes pasos suponen que el cómputo se completará utilizando un servidor institucional.
- Descargar la versión más reciente de Miniconda a cuenta de servidor usando la plataforma de Linux28.
- Instale la última versión de R con conda29.
- Trama de datos usando el script proporcionado de Seurat R como una plantilla de30.
Nota: Seurat es un conjunto de herramientas basadas en R que permite controles de calidad, clustering, diferencial Análisis de expresión génica, identificación de genes marcadores, reducción de la dimensionalidad y visualización de datos scRNA-Seq. Una descripción completa de Seurat codificación y tutoriales puede encontrarse en el sitio web de laboratorio Satija31. - Trama de datos usando el script proporcionado de monóculo R como una plantilla de32.
Nota: Monóculo es otra base de R que permite la visualización de los cambios de expresión sobre pseudotime e identifica genes subyacentes a las decisiones de destino celular. Una descripción completa de codificación de monóculo y tutoriales puede encontrarse en la Página Web de monóculo33. - Paquetes de R como kBET pueden emplearse para probar y corregir efectos de lote como resultado de la agrupación de conjuntos de datos34.
8. NCBI GEO y las presentaciones de la SRA
Nota: Ya que el fácil acceso a los archivos de secuencia de la materia prima garantizar la reproducibilidad y reanálisis, asentado en repositorios públicamente disponibles online se recomienda o necesaria antes de la presentación de manuscritos. Centro Nacional de información biotecnológica (NCBI) Gene expresión Omnibus (GEO) y secuencia leer archivo (SRA) son repositorios de datos públicamente accesible de alto rendimiento de secuenciación datos35,36.
- Registro de NCBI GEO remitente cuenta37.
- Presentación de GEO completa que incluye tres componentes que compila en un directorio (titulado como nombre de usuario del remitente GEO): 1) registro de metadatos (una hoja de cálculo por presentación de proyecto); Archivos de datos raw 2); 3) procesa los archivos de datos.
- Descargar y completar la hoja de cálculo metadatos del38. La siguiente presentación de GEO pública puede utilizarse como una guía (GSE100320)39. Coloque la hoja de cálculo en el directorio.
- Lugar archivos datos Raw generado por script cellranger cuenta para todas las bibliotecas en el directorio.
- Lugar procesa datos archivos (filtrado barcodes.tsv, genes.tsv y matrix.mtx) generado por script cellranger cuenta para todas las bibliotecas en el directorio.
- Utilizar credenciales de servidor FTP del remitente GEO para transferir el directorio que contiene los tres componentes. Para los usuarios de Linux/Unix: ncftp, lftp, ftp, sftp y ncftpput pueden ser utilizados.
- Notificar a GEO para todas las transferencias38.
Resultados
El repertorio de paquetes de código abierto diseñado para analizar conjuntos de datos scRNA-Seq ha disparado40 con la mayoría de estos uso de paquetes de lenguajes basados en R3. Aquí, se presentan resultados representativos usando dos de estos paquetes: evaluar agrupamiento no supervisado de basado en la expresión génica de células y ordenar a las células a lo largo de una trayectoria para resolver heterogeneidad de células y deconstruir biológico procesos.
Figura 4 ilustra el uso de Seurat para el procesamiento de los controles de calidad y análisis bioinformáticos posteriores. En primer lugar, filtración y eliminación de las células desviadas del análisis es esencial para el control de calidad. Esto se hizo utilizando la violín (figura 4a) y la dispersión de parcelas (Figura 4b) para visualizar el porcentaje de genes mitocondriales, número de genes (nGene) y número de UMI (nUMI) para identificar los dobletes de la célula y afloramientos. Cualquier célula con un número claro aislados de genes, UMI o el porcentaje de genes mitocondriales fue quitada usando la función FilterCells de Seurat. Puesto que Seurat utiliza componentes principales (PC) resultados de análisis a racimos de células, determinar estadísticamente significativa PC incluir es un paso crítico. Parcelas de codo (figura 4 c) fueron utilizados para la selección de PC, en que PC más allá de la meseta de la desviación estándar de la PC, eje se excluyeron. La resolución de la agrupación también fue manipulada demostrando que puede cambiarse el número de racimos, que van desde 0.4 (baja resolución a menos racimos de célula, figura 4 d) a 4 (de alta resolución a racimos de célula mayor, Figura 4e ). En baja resolución, es probable que cada grupo representa un tipo de celda definida, mientras que en alta resolución esto puede representar también subtipos o Estados transicionales de una población celular. En este caso, se utilizaba cluster baja resolución configuración para el análisis adicional de expresión heatmaps (usando la función DoHeatmap de Seurat) para identificar los genes más altamente expresados en un conjunto dado (figura 4f). En este caso, los genes más altamente expresados fueron identificados mediante la evaluación de expresión diferencial en un clúster determinado frente a todos los demás grupos combinados, demostrando que cada cluster estuvo representada únicamente por genes definidos. Además, se pueden visualizar genes candidatos individuales en parcelas de tSNE función de FeaturePlot de Seurat (figura 4 g). Esto permitió descifrar si hubo grupos que representan a los macrófagos. Usando el FeaturePlot, encontramos que ambos cluster 2 y 4 se expresan Cd68 - un marcador de pan-macrófago.
El paquete de monóculo se utilizó para corroborar racimos de célula identificados en Seurat y para la construcción de trayectorias de la célula, u ordenar pseudotemporal, recapitular procesos biológicos (figura 5). Ordenar pseudotemporal puede utilizarse para muestras donde perfiles de expresión unicelular se esperan que siga un curso de tiempo biológico. Las células pueden ordenarse a lo largo de un continuum pseudotemporal para resolver Estados intermedios, puntos de bifurcación de dos destinos alternativos de la célula e identifican firmas de genes subyacentes de adquisición de cada destino. En primer lugar, similar a la filtración de Seurat, células de mala calidad se retiraron tal que la distribución de mRNA a través de todas las células fue la log normal y cayó entre los límites superiores e inferiores identificadas en la figura 5a. Luego, mediante de monóculo newCellTypeHierarchy, las células fueron clasificadas y contaron utilizando genes marcadores de linaje conocido (figura 5b, 5C). Por ejemplo, las células alfa del receptor PDGF o fibroblastos específicos proteína 1 fueron asignadas a células tipo #1 para crear un criterio para la definición de fibroblastos. A continuación, esta población (célula tipo #1) se evaluó para descifrar trayectorias del fibroblasto. Para ello, se utilizó función diferencial de GeneTest de monóculo, que en comparación con las células que representan los Estados extremos dentro de la población y encontrar genes diferencial para ordenar las celdas restantes de la población (figura 5 d). Mediante la aplicación de métodos de aprendizaje múltiple (un tipo de reducción no lineal de la dimensionalidad) a través de todas las células, fue asignada una coordenada a lo largo de un camino pseudotemporal. Esta trayectoria se visualizó entonces por estado de la célula (figura 5e) y pseudotime (figura 5f).

Figura 1: Diagrama de flujo. Pasos de la preparación de todo animal a analizar solo células RNA-Seq datasets a presentar final conjuntos de datos en un repositorio público. Perlas de gel en emulsión (GEMs) se refieren a cuentas con oligonucleótidos con código de barras que encapsulan miles de células individuales. Haga clic aquí para ver una versión más grande de esta figura.
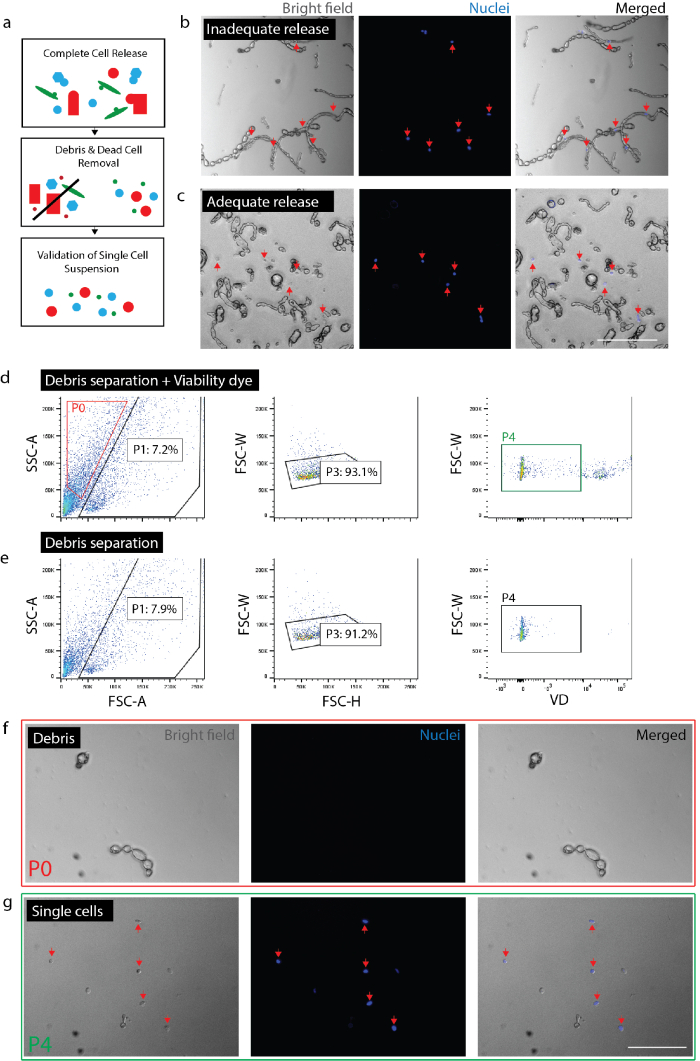
Figura 2: creación de suspensión unicelular viable del tejido nervioso. (a) Resumen de controles de calidad de la historieta. (b) células y desechos con las células todavía incorporan en escombros (flechas rojas). (c) células de escombros (flechas rojas). (d) célula de aislamiento por FACS. P0: fracción de escombros; P1: célula-como fracción; P3: exclusión de duplas; P4: fracción negativa de viabilidad tinte (Sytox Orange). (e) ningún control de tinte de viabilidad. (f) imagen de P0 fracción que representan restos aislados. (g) imagen de P4 fracción representar aislado células viables (flechas rojas). (b) .co (f) y (g) tenía tinte nuclear agrega 20 minutos antes de la proyección de imagen. Barras de escala: 80 μm. haga clic aquí para ver una versión más grande de esta figura.

Figura 3: secuenciación superficial predice el número de células recuperadas en 10 X muestras procesadas. un ejemplo (muestra 1.6) de csv generado MiSeq lista de códigos de barras de la célula y su correspondiente UMI cuenta según lecturas asignadas con confianza. (b) código de barras fila para 1.6 muestra muestra una gota significativa en el conteo de la UMI en función de códigos de barras de la célula. Las líneas sólidas y punteadas representan el límite entre las células y fondo según lo determinado por inspección visual. (c) códigos de barras célula observan usando el Cell Ranger tubería post-HiSeq revela secuencia superficial con precisión aproximada el número de células por ejemplo 1.6. (d) un ejemplo de una configuración de celda de flujo basado en la secuencia baja deriva las estimaciones de la célula. Para muestra 1.6, desde secuencias someras predijeron 3480 células, 1,17 carriles fueron asignados para garantizar > 100.000 lecturas por cobertura de la secuencia de células en HiSeq. Nota: Todos los carriles se deben agregar a 100%. Haga clic aquí para ver una versión más grande de esta figura.
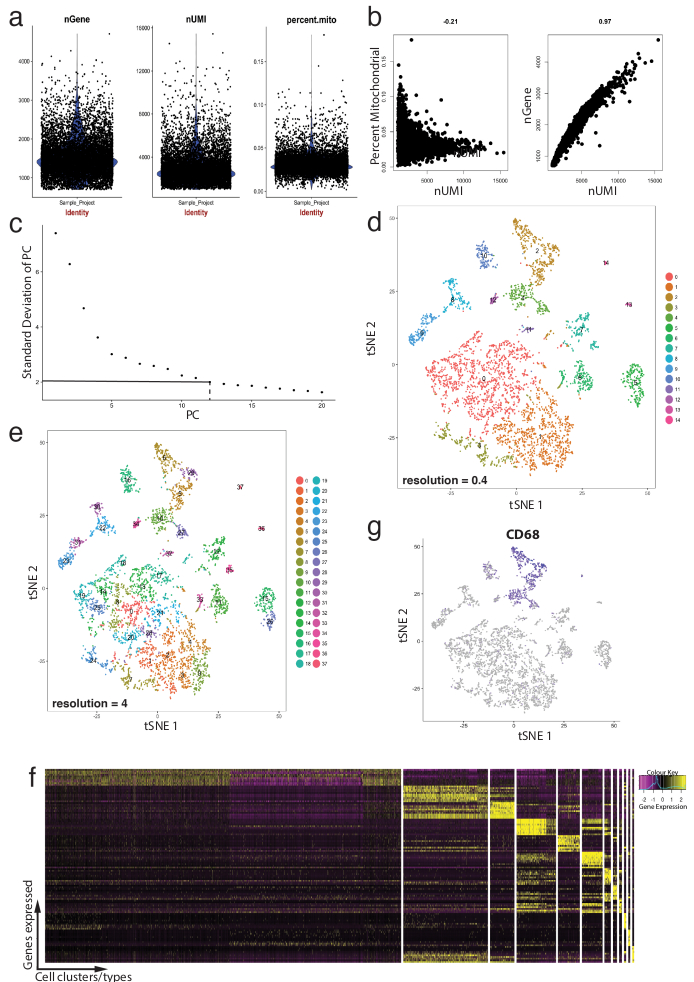
Figura 4: control de calidad y bioinformática de unicelular RNA-Seq dataset mediante paquete Seurat R. (a) parcelas de métricas de control de calidad que incluyen el número de genes, número de identificadores únicos de moleculares (UMIs) y el porcentaje de las transcripciones con el genoma mitocondrial. (b) gen muestra parcelas detectar células con niveles desviadas de las transcripciones mitochondrial y UMIs. (c) parcela de codo de la muestra utilizado para la determinación especial de PC estadísticamente significativa. Las líneas discontinuas y discontinua de punto representan el límite donde un "codo" de claro se hace evidente en el gráfico. Dimensiones del PC antes de este codo se incluyen en análisis posteriores. (d, e) Racimos de célula basada en gráfico visualizados en dos resoluciones diferentes en un espacio de baja dimensión con una parcela de tSNE. (f) mejores genes marcadores (amarillos) para cada cluster visualizados en un mapa de calor de expresión usando la función DoHeatmap de Seurat. g visualizar la expresión de marcadores de, por ejemplo, Cd68 gen representando macrófagos (morados) con función de FeaturePlot de Seurat. Esto sugiere que cluster 2 y 4 (panel d) de este conjunto de datos representa los macrófagos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: célula de categorización y ordenamiento a lo largo de la trayectoria de peudotemporal usan monóculo herramientas. (a) inspeccionar la distribución de mRNA (deducido de la cuenta UMI) a través de todas las células en una muestra. Sólo las células con el mRNA entre 0 - ~ 20.000 fueron utilizados para el análisis de aguas abajo. (b, c) Asignar y contando los tipos de la célula basados en marcadores de la célula de linaje conocido. Por ejemplo, las células alfa del receptor PDGF o fibroblastos específicos proteína 1 fueron asignadas a células tipo #1 que representan fibroblastos de pan usando la función newCellTypeHierarchy de monóculo. Número de diferentes tipos de células se puede visualizar como un gráfico de sectores (b) y como una tabla (c). (d) con célula tipo #1 (fibroblastos), por ejemplo, los genes utilizados para ordenar las células pueden ser visualizadas usando un terreno de dispersión que demuestra el dispersión génica vs expresión media. La curva roja muestra el atajo de genes utilizados para ordenar calculado por el modelo de media varianza usando la función estimateDispersions de monóculo. Genes que cumplen con este atajo se utilizaron para ordenar pseudotime aguas abajo. (e, f) Visualización de trayectorias de la célula en un reducido espacio bidimensional de color "Estado" de la celda (e) y monóculo asignado "Pseudotime" (f). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este protocolo demuestra cómo la preparación adecuada de las células puede revelar la heterogeneidad transcripcional de miles de células individuales y discriminar Estados funcionales o identidades únicas celulares dentro de un tejido. El Protocolo no requiere proteínas reportero fluorescente o herramientas de transgénicos y se puede aplicar al aislamiento de las células de diversos tejidos, incluyendo los de seres humanos; teniendo en cuenta cada tejido es único y este protocolo requiere cierto grado de ajuste o la modificación.
Los programas transcripcionales diversos y dinámicos dentro de las células han hecho hincapié en el valor de la sola célula genómica. Además de aislar el RNA de alta calidad, un paso de preparación de muestra crítica necesario para conjuntos de datos de alta calidad es asegurar que las células se liberan completamente de tejido y que las células son sanos e intactos. Esto es relativamente directo para la recolección de células que son fácilmente liberados, como la circulación de las células o en los tejidos donde las células se mantienen libremente, como en los tejidos linfoides. Pero esto puede ser un reto para otros tejidos adultos, debido a la arquitectura celular altamente desarrolladas que abarcan largas distancias, que rodea la matriz extracelular y las proteínas citoesqueléticas rígido a menudo involucrados en mantener la estructura de la célula. Incluso con técnicas de disociación apropiado para la versión completa de las células, es posible que la rigurosa y a menudo largas elaboraciones alteraría integridad calidad y celular mRNA. Además, las altas temperaturas usadas para ayuda de enzima disociación también afectan a firmas transcripcional29,30. El propósito del protocolo es presentar control de calidad verifica, utilizando tejidos como el nervio myelinated adulto y la piel de adultos de ricos en matriz extracelular, para demostrar cómo la optimización puede ayudar a superar estos obstáculos.
Una consideración importante al diseñar cualquier experimento de scRNA-Seq es la elección de la profundidad de la secuencia. La secuencia puede ser altamente multiplexada y leer profundidad puede variar de ser muy bajo con Seq gota2 a hasta 5 millones de lecturas/celular14 utilizando un método integral de RNA-Seq como Smart-SS. Mayoría de los experimentos scRNA-Seq puede detectar transcripciones de moderada a alta expresión con secuencias tan bajo como 10.000 lecturas/célula, que es generalmente suficiente para celular tipo clasificación41,42. Profundidad de la secuencia superficial es de valor para ahorrar en costos de secuenciación para detectar poblaciones de raros de la célula a través de tejidos complejos que pueden ser necesarios miles de células atribuir con confianza las poblaciones raras. Pero la secuencia de la profundidad no es adecuada cuando es necesaria obtener información detallada sobre expresión de genes y procesos asociados con firmas transcripcionales sutiles. Actualmente, se estima que la gran mayoría de los genes en una célula se detecta con 500.000 lecturas/cell, pero esto puede variar dependiendo del protocolo y tejido tipo43,44. Mientras que secuencia de transcripción larga duración evita la necesidad de Asamblea y, por lo tanto, detectar novela o variantes raras de empalme, los costes de la secuenciación a menudo limitan escala estos enfoques para examinar miles de células que comprende un sistema complejo tejido. En contraste, 3' etiquetado bibliotecas unicelular como las descriptas en este protocolo por lo general tienen menor complejidad y requieren menor secuencia. Es importante tener en cuenta que pueden ser secuenciadas de bibliotecas mediante el protocolo descrito en uno de los secuenciadores compatibles cinco: 1) NovaSeq, 2) HiSeq 3000/4000, 3) HiSeq 2500 ejecutar rápida y alto rendimiento, 4) NextSeq 500/550 y 5) MiSeq.
Un enfoque alternativo a celular solo RNA-Seq, que reduce la necesidad de delicados tejidos y células manejo todavía mantiene algunos de los beneficios de célula única RNA-Seq, es el análisis de ARN de núcleos solo45. Este enfoque permite la tramitación más rápida reducción de la degradación del RNA y medidas más extremas para asegurar la adecuada liberación de núcleos y así probablemente permite una captura de más confianza de los perfiles transcripcionales que representa todas las celdas dentro de un determinado tejido. Esto, por supuesto, sólo daría una parte de la actividad transcripcional presente dentro de una célula dada, así dependiendo de lo que son objetivos experimentales de interés este enfoque puede o puede no ser apropiado.
Además de la caracterización completa de identidades celulares en un tejido dado, uno de los más valiosos análisis para conjuntos de datos scRNA-Seq es la evaluación de Estados transcripcionales intermedias en poblaciones celulares 'definido'. Estos estados intermedios pueden impartir conocimientos sobre las relaciones de linaje entre las células dentro de las poblaciones identificadas, que no era posible con a granel tradicional que acerca de RNA-Seq. Han desarrollado ahora varias herramientas bioinformáticas de scRNA-Seq para aclarar esto. Tales herramientas pueden evaluar los procesos involucrados, por ejemplo, las células cancerosas la transición a un estado oncogénico, metastásico, las células madre maduran en diversos destinos terminal o las células inmunes, transporte entre los Estados activados y quietos. Transcriptoma sutiles diferencias en las células también pueden ser indicativas de sesgos de linaje que, herramientas bioinformáticas desarrolladas recientemente como FateID, puede inferir47. Puesto que las diferencias entre las células de transición pueden ser difíciles determinar dadas las diferencias transcripcionales puede ser sutil, la secuencia más profunda puede ser necesario46. Afortunadamente, la cobertura de una biblioteca bajo secuenciada puede aumentar si está interesado en sondear el conjunto de datos más de nuevo ejecutando la biblioteca en otro célula de flujo.
Tomados en conjunto, este protocolo proporciona un flujo de trabajo fácil adaptación que permite a los usuarios de perfil transcripcionalmente cientos o miles de células individuales dentro de un experimento. La calidad final de un conjunto de datos de scRNA-Seq se basa en el aislamiento optimizada de la célula, citometría de flujo, generación de biblioteca de cDNA e interpretación de matrices de crudo gene-código de barras. Para ello, este protocolo proporciona una descripción comprensiva de todos los pasos claves que puede ser fácilmente modificado para permitir estudios de tipos de tejidos diversos.
Divulgaciones
No hay revelaciones
Agradecimientos
Reconocemos que el personal de apoyo en la instalación de servicios de UCDNA, así como el personal de la institución de cuidado Animal de la Universidad de Calgary. Agradecemos a Matt Workentine por su apoyo de Bioinformática y Jens Durruthy por su apoyo técnico. Este trabajo fue financiado por una donación CIHR (R.M. y J.B.), un premio de investigador nuevo de CIHR y J.B., salud investigación Instituto beca (J.S. un infantil de Alberta).
Materiales
| Name | Company | Catalog Number | Comments |
| Products | |||
| RNAse out | Biosciences | 786-70 | |
| Pentobarbital sodium | Euthanyl | 50mg/kg | |
| HBSS | Gibco | 14175-095 | |
| Dispase 5U/ml | StemCell Technologies | 7913 | 5 mg/ml |
| Collagenase-4 125 CDU/mg | Sigma-Aldrich | C5138 | 2 mg/ml |
| DNAse | Sigma-Aldrich | DN25 | 10mg/ml |
| BSA | Sigma-Aldrich | A7906 | |
| 15 ml Narrow bottom tube VWR® High-Performance Centrifuge Tubes | VWR | 89039-666 | |
| Sytox Orange Viability Dye | Molecular Probes | 11320972 | 1.3 nM/µl |
| Nuc Blue Live ReadyProbes | Invitrogen | R37605 | |
| Agilent 2100 Bioanalyzer High senitivity DNA Reagents | Agilent | 5067-4626 | |
| Kapa DNA Quantification Kit | Kapa Biosystems | KK4844 | |
| Chromium Single Cell 3' reagents | 10x Genomics | ||
| Equipment | |||
| BD FACSAria III | BD Biosciences | ||
| Agilent 2100 Bioanalyzer Platform | Agilent | ||
| Illumina® HiSeq 4000 | Illumina | ||
| Illumina® MiSeq SR50 | Illumina | ||
| 10X Controller + accessories | 10x Genomics | ||
| Software | |||
| The Cell Ranger | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/overview/welcome | |
| Loupe Cell Browser | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest | |
| R | https://anaconda.org/r/r |
Referencias
- Shalek, A. K., et al. Single-cell RNA-seq reveals dynamic paracrine control for cellular variation. Nature. 510, 363-369 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161, 1202-1214 (2015).
- Zappia, L., Phipson, B., Oshlack, A. Exploring the single-cell RNA-seq analysis landscape with the scRNA-tools database. bioRxiv:206573. , (2018).
- Dulken, B. W., Leeman, D. S., Boutet, S. C., Hebestreit, K., Brunet, A. Single cell transcriptomic analysis defines heterogeneity and transcriptional dynamics in the adult neural stem cell lineage. Cell Reports. 18 (3), 777-790 (2017).
- Llorens-Bobadilla, E., et al. Single-Cell Transcriptomics Reveals a Population of Dormant Neural Stem Cells that Become Activated upon Brain Injury. Cell Stem Cell. 17 (3), 329-340 (2015).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32, 381-386 (2014).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14, 1083-1086 (2017).
- Mayer, C., et al. Developmental diversification of cortical inhibitory interneurons. Nature. 555 (7697), 457-462 (2018).
- Stratton, J. A., et al. Purification and Characterization of Schwann Cells from Adult Human Skin and Nerve. eNeuro. 4 (3), (2017).
- Biernaskie, J. A., McKenzie, I. A., Toma, J. G., Miller, F. D. Isolation of skin-derived precursors (SKPs) and differentiation and enrichment of their Schwann cell progeny. Nature Protocols. 1 (6), 2803-2812 (2007).
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Agilent Available from: https://www.agilent.com/en-us/library/usermanuals?N=135 (2018)
- Kolodziejczyk, A. A. Single Cell RNA-Sequencing of Pluripotent States Unlocks Modular Transcriptional Variation. Cell Stem Cell. 17, 471-485 (2015).
- Jaitin, D. A., et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 343, 776-779 (2014).
- Pollen, A. A., et al. Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex. Nature Biotechnology. 32, 1053-1058 (2014).
- . Sequencing Requirements for Single Cell 3' Available from: https://support.10xgenomics.com/single-cell-gene-expression/sequencing/doc/specifications-sequencing-requirements-for-single-cell-3 (2018)
- . Introduction to R Available from: https://www.datacamp.com/courses/free-introduction-to-r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics#39; Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Creating a Reference Package with cellranger mkref Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/advanced/references (2018)
- . System Requirements Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/overview/system-requirements (2018)
- . Software Downloads Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest (2018)
- . Aggregating Multiple Libraries with cellranger aggr Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/using/aggregate#depth_normalization (2018)
- . Loupe Cell Browser Gene Expression Tutorial Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/tutorial (2018)
- . A table of tools for the analysis of single-cell RNA-seq data Available from: https://www.scrna-tools.org/ (2018)
- . Downloading conda Available from: https://conda.io/docs/user-guide/install/download.html (2018)
- . r / packages / r 3.5.1 Available from: https://anaconda.org/r/r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . Seurat - Guided Clustering Tutorial Available from: https://satijalab.org/seurat/pbmc3k_tutorial.html (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- Github. . An R package to test for batch effects in high-dimensional single-cell RNA sequencing data. , (2018).
- Edgar, R. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30, 207-210 (2002).
- Leinonen, R., Sugawara, H., Shumway, M. The sequence read archive. Nucleic Acids Research. 39, D19-D21 (2011).
- . GenBank Submission Portal Wizards Available from: https://www.ncbi.nlm.nih.gov/account/register/?back_url=/geo/submitter/ (2018)
- . Submitting data Available from: https://submit.ncbi.nlm.nih.gov/geo/submission/ (2018)
- Shah, P. T., et al. Single-Cell Transcriptomics and Fate Mapping of Ependymal Cells Reveals an Absence of Neural Stem Cell Function. Cell. 173, 1045-1057 (2018).
- Anon, Method of the Year 2013. Nature Methods. 11, 1 (2013).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144, 3625-3632 (2017).
- Wu, Y. E., Pan, L., Zuo, Y., Li, X., Hong, W. Detecting activated cell populations using single-cell RNA-seq. Neuron. 96, 313-329 (2017).
- Zeigenhain, C., et al. Comparative Analysis of Single-Cell RNA Sequencing Methods. Molecular Cell. 65 (4), 631-643 (2017).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nature Methods. 11 (1), 41-46 (2014).
- Habib, N., et al. Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science. 353 (6302), 925-928 (2016).
- Janes, K. A. Single-cell states versus single-cell atlases - two classes of heterogeneity that differ in meaning and method. Current Opinions in Biotechnology. 39, 120-125 (2016).
- Herman, J. S., Sagar, D., Grün, FateID infers cell fate bias in multipotent progenitors from single-cell RNA-seq data. Nature Methods. 15 (5), 379-386 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados