Method Article
哺乳類の成体の液滴バーコード ベースの単一細胞トランスクリプトーム
* これらの著者は同等に貢献しました
要約
このプロトコルを記述する一般的なプロセスと品質管理チェック液滴ベース、高スループット単一細胞 RNA シーケンス準備のため健康な成体哺乳類単一細胞を準備するために必要です。パラメーターのシーケンス、読み取りの配置、およびダウン ストリームの単一細胞バイオ情報解析されています。
要約
組織や微小環境の内で個々 の細胞の何千もの単一細胞遺伝子発現の解析は識別の細胞組成、機能状態と分子経路観測組織の差別のための貴重なツール関数は、動物の行動。ただし、以降の下流の単一細胞分子解析成体哺乳類組織からそのままに、健康的な単一セルの隔離は挑戦することができます。このプロトコルは、一般的なプロセスを説明し、品質管理は神経系から高品質大人の単一細胞製剤を入手するか、皮膚に必要な有効その後公平な単一細胞 RNA シーケンスと解析をチェックします。下流のバイオ情報解析のためのガイドラインも提供されます。
概要
高スループット単一のセル技術1,2の最後の十年3以上のユーザーフレンドリーなバイオインフォマティクス ツールの進歩発展に伴い、高解像度遺伝子発現解析の新しい分野を浮上している-単一細胞 RNA シーケンス (Seq-scRNA)。単一細胞の遺伝子発現の研究を幹細胞やがん細胞のように定義された細胞集団内の不均一性を識別するためにまたは使用不可能であったセル4、5、特殊な集団を識別するために最初に開発されました従来のばら積み RNA の配列解析技術。Bioinformatic ツールが新規のサブ集団 (スーラ)2, psuedotime スペース (モノクル)6、アクティブのシグナリング ネットワーク内または集団 (間の定義に沿って細胞の可視化の識別を有効にしています。風光明媚な)7、人工 3 D 空間 (スーラなど) の単一セルのアセンブリの予測8。新しくて刺激的な分析科学のコミュニティに利用可能、scRNA Seq は高速-なっている遺伝子発現解析のための新しい標準的なアプローチ。
ScRNA Seq の広大な潜在性にもかかわらずきれいなデータセットを生成し、結果を正確に解釈するために必要な技術的なスキルセットは新人に挑戦することができます。ここでは、基本的なしかし、包括的なプロトコル、可視化する全体主組織から単一セルの隔離およびパブリケーションのデータの提示から始まってされます (図 1)。まず、健康な単一細胞の分離とみなすことが困難な酵素消化とその後の機械的解離に対する感度の度合いで変わるさまざまな組織として。このプロトコルは分離手順のガイダンスを提供し、プロセス全体を通して品質管理の重要なチェックポイントを識別します。第二に、互換性および単一のセル技術と次世代シーケンサーと要件を理解することは混乱することができます。このプロトコルは、ユーザーフレンドリーな液滴を用いた単一細胞のバーコード プラットフォームを実装して、シーケンス処理の実行するためのガイドラインを提供します。最後に、コンピュータ ・ プログラミングは単一細胞トランスクリプトームのデータセットを分析するための重要な前提条件です。このプロトコルは、R のプログラミング言語を使うにあたってのリソースを提供し、2 つの人気 scRNA-Seq 固有の R パッケージの実装について説明します。一緒に、このプロトコルは、分析 scRNA Seq クリア、解釈の結果を取得するために初心者を導くことができます。このプロトコルはマウスのほとんどの組織に調整することができます、重要なの組織の人間を含む他の有機体で使用するために変更することが。組織とユーザーに応じて調整が必要になります。
この議定書に従いながら、留意すべきいくつかの考慮事項があります。正確な合計のセル番号のカウント (図 2 に要約を確保しながら興味のサンプル内のすべてのセルの実行可能な単一細胞懸濁液を確実に推奨は 1) 手順 1 ですべての品質管理の指針とこのプロトコルの 2 次など).これが実現すると、最適化のすべての条件に従っている場合品質管理手順で削除できます (に短縮 - RNA の品質を維持し、細胞の損失)。確認する興味の組織から高い生存率単一細胞の分離を成功させる高い下流より前に、お勧め処理しています。2) ある細胞のタイプはストレスを他の人よりも敏感なので、過剰解離手法が人口、したがって下流解析を交絡バイアスは誤って。不要な携帯電話せんと消化せず穏やかな解離が高利回り細胞および組織組成の正確な表現を実現するため重要です。巻き上がり、FACS 製粉段階でせん断力が発生します。3) として任意の RNA の作品とはそれの準備中にできるだけサンプルに少し追加 RNase として取り入れるが最善。高品質の RNA を維持に役立ちます。リボヌクレアーゼ阻害剤のソリューションを使用しクリーン ツールとではない機器を洗浄 RNase フリーが、DEPC 処理製品を避けてください。4) できるだけ早く準備を実行します。これは高品質の RNA を維持し、細胞死を削減を助けます。組織郭清の長さと動物の数、に応じて、同時に複数の解剖/準備を開始を検討します。5) 高品質 RNA を維持、細胞死を軽減し低速可能な細胞シグナル伝達と転写活性とき氷の上のセルを準備します。とはいえ、冷たい処理は、ほとんどの種類の細胞に最適です、室温で処理されるときは、ある細胞のタイプ (例えば、好中球) の方がパフォーマンスが向上します。6) セルの準備中にカルシウム、マグネシウム、EDTA、DEPC 処理製品を避けてください。
プロトコル
ここで説明したすべてのプロトコルに従い、カルガリー大学動物ケア委員会によって承認されました。
1. 分離組織 (1 日目)
- ペントバルビ タール ナトリウム (i. p.、50 mg/kg) または動物の倫理のプロトコルに従って適切な過量を持つマウスを安楽死させます。マウスの足の裏から不要な毛を削除し、郭清領域をエタノール消毒します。
- 組織または関心の微小環境を分析します。このプロトコルでは、次の成体組織解離液滴バーコード ベースの単一細胞トランスクリプトームの一般化の可能性を実証するのに皮膚と神経組織を使用します。
- 坐骨神経・ ストラトンらは、詳細なプロトコルを使用します。9. 簡単に言えば、マウスの背中/脚の後部の地域から皮膚をカットします。生殖不能のメスの刃で太ももの長さに沿って切開を行います。微細鉗子、はさみを使用して公開および坐骨神経を削除します。
- 背中の皮膚 Biernaskieらは、詳細なプロトコルを使用します。10. 微細鉗子、はさみを使用してバックの上下左右臀部から肩を切開することによって簡単に、背部皮膚を解剖。生殖不能のメス刃を使用して薄くスライス (厚さ 0.5 cm) に皮膚をカットします。
- 冷たい HBSS で 2 回組織を洗浄し、不要な結合組織、脂肪や解剖顕微鏡の下でゴミを取り除きます。
- 皮膚の真皮のみに 37 ° C で 30 ~ 40 分の HBSS で当期 (5 mg/mL、5 U/mL) でスライスをフロートします。真皮から表皮を分離手術。表皮を破棄またはさらにトリプシンを使用して興味のある場合の関連付けを解除します。
- 生殖不能のメス刃を使用して 1-2 mm の部分にサンプルをミンチし、新鮮解凍 2 mg/mL 冷たいコラゲナーゼ IV 酵素 (2 mg/mL、F12 メディアで 125 CDU/mg) に入れた。
- 神経、2 x 坐骨神経あたり ~ 500 μ L を使用します。肌の 1 x マウスの背部の皮膚ごと ~ 8 mL を使用します。
注: の組織は、コラゲナーゼ IV ソリューションで完全に水没する必要があります。任意の消化酵素、処理、格納され、適切に準備が重要です。酵素は、時間の長い期間の室温で残っている単一セルの分離は過度の機械製粉を必要とされ、細胞生存率を減らします。コラゲナーゼ IV は、細胞生存率は最適な細胞培養媒体でまた構成ことができます。ただし、これは酵素活性を変えることができるまたは転写署名ですのでユーザーが最適化する必要があります。
- 神経、2 x 坐骨神経あたり ~ 500 μ L を使用します。肌の 1 x マウスの背部の皮膚ごと ~ 8 mL を使用します。
- 穏やかな揺れ 10 分と 30 分のための 37 ° C バースの酵素でサンプルをインキュベートします。シェーカーの 37 ° C で配置も適切な代替案です。
- 30 分後酵素剤添加で 20-30 回 P1000 pipettor とカップ刻んだ。
- ソリューションは、曇りが表示され、ティッシュの塊が大きく解離まで製粉 30 分ごとを繰り返します。
注: セル (図 2 b, 2 c) の完全なリリースを確保します。完全なリリースを確認する Nuc 青 (1 mL につき 2 滴) で細胞をプレートと 20 分後にすべての核が破片ではなく、単一のセルに関連付けられていることを確認を顕微鏡下でチェックします。組織の種類または条件ごとに指定された実験内セル リリースの度をチェックする重要です。線維組織 (すなわち、慢性的な損傷) または無傷の成体組織、細胞のリリースは外傷急性期または萌芽期ティッシュから劇的に変わる。特定のセルタイプ可能性が低いので、他よりも組織から解放するため下流の分析からそれらの細胞を優先的に排除、これは特に重要です。- 0.5 〜 1.5 のための組織を切り離して考える神経の合計時間。肌に計 2 時間の組織を分離 (インキュベーションの最後の時間では、DNase (1 mg/mL) に追加皮膚サンプル)。
- 二度 40 μ m のフィルターをフィルター処理します。冷たい 1% が付いているフィルターを洗う BSA/HBSS。
- 8 分の 260 × g で遠心分離機します。上清を削除します。
- ワイドボアの探針を用いた 1 %bsa を含む HBSS で細胞ペレットを再懸濁し、氷の場所します。再懸濁量は組織の数量に基づきます (800 mg 皮膚の湿重量 800 μ L = ボリューム; 神経の太さをウェット 10 mg = 100 μ L のボリューム)。
- 必要に応じて、低巻き上がりボリュームで始まる、FACS の並べ替えアイコン フロー レート (1 秒あたりのイベント) に基づいて必要に応じて調整します。最も効率的な密度の並べ替え (minimalizing 時間中に収集されたセルの数を最大化) のコレクションは 3,000-7,000 のイベント/秒。
- 性染料を使用している場合は、無染色の制御のための subaliquot を取る。1:15,000 生存率染料を追加 (在庫: 20,000 nM/μ L) サンプル (1.3 nM/μ L 最終濃度) せん断を抑えるワイドボア チップを使用します。
注: 組織の種類または条件ごとに指定された実験内細胞死の程度をチェックする重要です。サンプルには、いくつかの細胞の種類、このように優先的に下流の分析から除外されている他の人に比べて死亡するらしい。- 暗闇の中で氷の上の 5-10 分の生存率染付サンプルをインキュベートします。4 mL の冷たい 1% 追加 BSA/HBSS サンプルに。余分な性の色素を削除する 8 分の 260 × g で遠心分離機します。治療なしの生存率と subaliquot は、同じ方法で無染色のコントロールを染めます。
2. 健康な実行可能な細胞 (1 日目) を分離します。
- FACS 施設続く適切な蛍光活性化セル (FACS) パラメーターの並べ替えであることを確認します。
- ステップ 1 で最終的なの遠心分離機が完了したら、準備がように事前に FACS マシンを準備し、コレクション コンパートメントが冷たい保たれることを確保する氷のブロックを使用して。
- 次のパラメーターを使用する: 流量: 1.0 (10 μ L/分にほぼ相当);フィルター: 1.5 ND;ノズルのサイズ: 100 μ m;前方散乱: 80-180 V (イベントのサイズを区別するために必要に応じて変更);側方散乱: 150-220 V (イベントの粒度・形状を区別するために必要に応じて変更);レーザー: 100-400 V (生存色素正対否定的なイベントを区別・色素・ コントロールなし生存率に対してこれをチェックするために必要に応じて変更);ゲート: をすべての細胞を収集するには、必要に応じて変更します。図 2 d-2 gを参照してください。
注: FACS パラメーターが細胞の種類に大きく依存、ソーターを採用、したがってユーザーに最適化する必要があります。
- 8 ml の冷たい 1% の狭い下チューブ 15 mL を準備 BSA/HBSS サンプル コレクション。管と表面張力の中の静電気はコレクションの効率に影響を与えます。液体の表面との間のインターフェイスを確保するためのコレクションの前にチューブを反転し、管の中はしっとり。
注: 非常に低いセル番号を使用して場合、に応じて小さなコレクション容器に調整します。 - すべてのセルが収集されると、8 分の 260 x g でサンプルを遠心します。
注: 遠心分離の前に追加 1 %bsa/HBSS 洗浄/プッシュ細胞に側面から下がると反転/ミックス チューブ FACS の直後後。 - 1 %bsa/HBSS し氷の上で細胞ペレットを再懸濁します。ステップ 3 処理と互換性のあるサンプルごとの最大数量は 33.8 μ L、だから最終希釈/再懸濁量 33.8 μ L で理想的なセル番号を取得する適切な使用します。この手順の他の希釈メディア オプション (および 1% のすべての前の希薄 BSA/HBSS)、DMEM を含めると 40% 血清がカルシウム、マグネシウム、または EDTA 試薬を含むを避けるため。
- 時間の最小限の量の氷に細胞を残します。理想的に同僚はステップ 2 の最後の手順の中に次のステップ (ステップ 3) のすべての機器・試薬を準備必要があります。
- セル準備の重要なチェック
- FACS から得られた細胞数の推定値を確認します。組織の種類や解離によって長さ、破片およびセル サイズと形状に非常に似ていることができます。したがって、蛍光レポーターを使用しない限り、FACS はすべての残骸を除外できません。(FACS) によるとイベントの何パーセントを理解する FACS コレクションが実行された後の最終的な細胞数が実際に与えられた準備 (図 2 g) のセルであることをお勧めします。検定または自動化された細胞カウンター (2 回繰り返し) を使用してセル数を実行し、FACS マシンによって収集したイベントの合計数によって表される実行可能なセルのパーセンテージを計算します。
- セル準備を検証します。検証なしの大きな粒子 (> 100 μ m)、後工程の機器を詰まらせる可能性があります彼らが存在。破片の不十分な取り外しは、単一細胞マイクロ流体チップの目詰まりを危険性があります。大きな破片の断片が存在しないことを確認する (上記) Nuc 青で残りのセルをプレートします。これは細胞が単一であることの確認のため、また (すなわち、一緒に付着されていない) 複数のセルではなく、単一のセルを表すその下流の単一細胞遺伝学的解析に信用を与えます。
- シーケンスに細胞数を決定する: 成人組織由来細胞を一度に実行することができます最大 8 サンプルをシステムに読み込むことができるサンプル数の大きい範囲があります。著者はサンプルあたり 500-50,000 セルからどこでもロードし、良質 scRNA Seq データセットを取得します。読み込みに最も適切なセル番号に関する詳細については、ディスカッション セクションで見つけることができます。シーケンスされた細胞の数の最終的な出力は、分離した単一細胞の品質に大きく依存します。10,000 成体組織由来細胞を読み込んで返すことがどこでも 1,000 から 4,000 シーケンスされたセル (10-40% のリターン)。(~ 10,000 セル、このシステムの推奨最大数) の高い携帯番号を配列に興味がある場合、25,000 100,000 セルを読み込みが必要となります。
3. 宝石 (エマルジョン、ゲル ビーズ) の生成とバーコード (1 日目)
注: このプロトコルの手順 3-6 が最も一般的な液滴を用いた単一細胞プラットフォーム、10 X Genomics 製と組み合わせて使用する設計されています。詳細なガイドラインの製造元の手順 3 と 4 のとおりプロトコル (クロム単一セル 3' プロトコル参照)11,12とと共にこのプロトコルに従う必要があります。最高の結果を得るには、このプロトコルの 1 日の解離 (ステップ 1) および細胞分離 (ステップ 2) の手順を実行後すぐにステップ 3 を完了する必要があります。
- 製造元のプロトコル11,12によるとチップを準備します。この液滴を用いた単一細胞プラットフォームは、個別に各セルのトランスクリプトームをインデックスにそのサンプル 〜 750,000 バーコード技術を使用します。これは、エマルジョン (宝石) 生成された cDNA が一般的なバーコードを共有でゲルビーズにセルを分割することにより達成されます。宝石の生成中に生成される宝石の大半 (90-99%) は、ほとんどの部分、残りのセルを含まないので、細胞が配信される、単一のセルを含みます。
- チップ ホルダーにチップを置きます。
- 氷の上携帯マスター ミックスを準備します。
- 未使用の井戸に 50% のグリセロールを追加し、よく 1 セル マスター ミックスの 90 μ L を追加のオイルも 3 分割も 2 と 270 μ L をゲル粒子の 90 μ L。
- ガスケットにチップをカバーしてください。
- チップを読み込み、単一セルのコント ローラーで実行します。
- トレイを取り出し、トレイにチップを置き、トレイを撤回、再生を押します。宝石の 1 つのセル 3' ゲル ビーズを含むプライマー (1 配列のプライマーを読む) 部分的な R1 イルミナ シーケンスを含む 16 のヌクレオチド (nt) 10 x Barcode、10 nt ユニークな分子識別子 (UMI) とポリ dT プライマー シーケンス。、実行中にコント ローラーのゲル粒子を公開して細胞ライセートとマスター ミックスと混合します。
- 100 μ L のサンプルと PCR チューブの場所を収集します。
- 事前に設定の PCR の場所 PCR チューブ マシン キットによると PCR を実行してください。次の孵化、宝石が含まれます、バーコード cDNA ポリ adenylated mRNA から。
- 次の実行では、一晩-20 ° C で次のステップへ先行する前に最大 1 週間。
4. クリーンアップ、増幅、ライブラリの構築、ライブラリ定量化 (2 日目以降)
注: 手順 4 の詳細なガイドラインは製造元のプロトコル、11,12に記載されて、と共にこのプロトコルに従う必要があります。
- 宝石反応混合物から残りの生化学試薬/プライマーを削除するのにシラン磁気ビーズを使用します。
- 増幅、バーコード cDNA ライブラリ構築のための十分な質量を生成します。
- DNA の収穫を評価します。図書館の建設の前に、サンプルの DNA の収穫を評価します。これは下流の PCR のステップ (図書館の建設中にサンプル インデックス PCR) で使用するどのように多くのサイクルが決定されます。セルの種類、および細胞のアクティブ化の状態(例えば制御対負傷等)、によって異なります、特定のサンプルの RNA 含量により推奨サイクル数が異なります。
- シーケンス 〜 3,000 組織由来細胞 (アクティブ化の状態とは無関係)、著者ことを発見 14 サイクル (サンプル: 10 ~ 100 ng DNA) 標準です。
- DNA 分析のため、バイオアナライザーを使用します。参照してくださいユーザー ガイド13。
- サンプルのフラグメントし、DNA のサイズを選択します。図書館の建設前に酵素断片化とサイズ選択プロトコルを使用適切な cDNA 増幅サイズを取得します。
- 図書館建設のためサンプルを準備します。R1 の宝石の孵化; 中に分子に追加 (1 のプライマー シーケンスを読む) 間P5、P7 (サンプル インデックス)、R2 (2 プライマー シーケンスを読む) ライブラリの構築時に追加されます。
- DNA の収穫を評価します。ほとんどのシーケンス処理設備は、DNA の収量・品質の情報を含む最終的なライブラリの提出を必要とします。したがって、シーケンス処理施設に輸送する前に、プロトコル全体の完了に続くバイオアナライザーを実行します。
- 最大 2 ヶ月間-80 ° c のサンプルを格納します。
- シーケンス処理の前に DNA 定量キットを使用してサンプルを定量化します。これは、シーケンスの施設で実行できます。
5. ライブラリ シーケンス (3 日以降)
注: このプロトコルで使用する単一細胞トランスクリプトーム バーコード プラットフォームは、イルミナ互換ペアエンド ライブラリ P5 と P7 のシーケンスで終わるを生成します。最小深さセル型 id は 10,000-50,000 読み取り/セル15,16ほどすることができますを解決する必要がある 〜 100,000 ヒット/セルは成人の体内細胞 (念頭に置いていくつかのセルの最適なコスト カバレッジ トレードオフとしてお勧めします型または最小活性化細胞の状態が飽和に到達で 30,000-50,000 の読み取り/セル)。
- 適切なイルミナ シーケンサー搭載シーケンス処理施設にドライアイスの cDNA ライブラリを転送します。
- シーケンス機能に次の情報を提供します。
- サンプルの詳細を提供する: 各ライブラリに対応するインデックス Id のサンプル種;ゲノムのデータベースのプライマリ アセンブリ (つまりマウスの GRCm38);レーザービーム (間 200 および 9,000 bp); バイオアナライザーからフラグメント サイズを表示cDNA 濃度 (ng/μ L) と合計ライブラリ濃度 (200-1400 ng の合計利回り範囲);サンプルの量 (μ L)。
- シーケンス要求を提供する: DNA の定量キット; を使用してサンプルを定量化アダプター/インデックス型 (TruSeq DNA);プレート型 (エッペン twin.tec、フルスカート - DNA のためをお勧めします)。技術/ライブラリ型 (10 x、完全シーケンス指令およびサイクル推奨事項)17のシーケンス。
- 浅い配列 (省略可能) を実行: 複数試料の分析研究にお越しの方にもすべてのサンプルからのデータを含む単一遺伝子バーコード マトリックスを生成するサンプル (集計) をプールから。バッチをプールする場合のサンプルの間の効果を最小限に抑えるための異なるライブラリの読み取りの深さを標準化しなければなりません。これを行うには、単一のセル番号の正確な近似が必要です。MiSeq シーケンサーが浅い配列、セルの正確な見積もりを取得するコスト効果の高い、実用的な方法です。
注: MiSeq SR50 シーケンサーを使用して 1 つの実行は、約 20,000 細胞を正確に推定する十分なカバレッジを提供します。この実行は海各一意のバーコードの回復の数を概算します。図 3 a出力例 (サンプル 1.6) (.csv) のヘッダーが表示されているバーコードのリストとその対応する海数自信を持ってマップリードによって決定されます。- R のプログラミング言語を理解する bioinformatician を参照してください。詳細情報18DataCamp チュートリアルを参照してください。
- テンプレート19として提供されている R のスクリプトを使用してシーケンサーから得られる生データを評価します。Raw データは、行政の各一意のセル バーコードにマップの数を指します。このスクリプトでは、.csv ファイルでバーコードのリストは、最初の列と 2 番目の列は、対応する海は、カウントを読み込みます。このスクリプトでは、各サンプルのバーコード細胞数の推定だけでなく、プロット (図 3 b) を提供します。ある特定のサンプルの海の入力数を最初の急な低下の 3 分の 1 の時点で確実にスクリプトを調整します。図 3 b、この肘滝約 225 Umi 3,480 バーコード セルに対応します。
- HiSeq を使用して全深さ配列に匹敵する (3,516 細胞が正常にされたシーケンス、図 3 c)、浅いシーケンスの見積もり予測 3,480 細胞。
- 使用細胞回復近似 (ステップ 5.3) からまたは使用回復チャートは、製造元のプロトコル20レーン分布より深いシーケンスを計画します。各サンプルは、浅いシーケンス (これは多くの場合) 各サンプル内のセル数が異なるがあることを明らかにする場合、レーン分布がそれに応じて計算する必要があるので対等な適用範囲を受け取るはずです。1 つ HiSeq のフロー ・ セル (8 レーンを占める) を最大 24 億カスタム ペア終了読み取りシーケンス処理できます。例フロー セル設定は3 d 図で提示されます。
6. 処理ファイルの読み取り
注: 単一のセル 3' このプロトコルを使用してライブラリをシーケンス バイナリ ベース呼び出し (BCL) 形式で raw データを生成します。セル レンジャー パッケージを使用して BCL ファイルからテキスト ベース FASTQ ファイルを生成、ゲノムとトランスクリプトーム線形、遺伝子数、逆多重化、およびサンプルの集計。このセクションでは、シーケンス処理施設から生の BCL データをダウンロードし、フィルター処理された遺伝子バーコード行列の生成下流バイオインフォマティクスのため準備ができてするユーザーを有効にする主要な手順が表示されます。
- プログラムを実行する集中管理サーバーを使用します。BCL ファイル、FASTQ ファイルおよび下流のバイオインフォマティクスの処理のほとんどは、大量の処理能力を要求します。
- サーバー (または FASTQ ファイル彼らが利用可能な場合) にすべての raw 読み取りファイルをダウンロードします。
- 集中化されたサーバーまたはクラスター上のアカウントを設定して Unix21に精通して取得するサーバー管理者に問い合わせてください。
- シーケンス機能のサーバーからすべてのファイルをダウンロードするのにサーバーのオペレーティング システムの適切な fetch コマンドを使用します。
- ほとんどのシーケンスの施設提供、コマンドラインから実行することができますセキュリティで保護されたパスからファイルをダウンロードするためのコマンド (下の例を参照してください)。
- 「< ユーザー名 >」を交換し、提供された資格情報を持つコマンド ラインに「< パスワード >」プレース ホルダー。
wget - O -"https://your_sequencing_facilitys_server.com/path_to_raw_read_files/- は-クッキー-- チェック-証明書 ― 後データ 'j_username = ユーザ名 & j_password = パスワード' |wget - は-クッキー-- チェック-証明書 ― 後データ 'j_username = ユーザ名 & j_password = パスワード' - ci -
- のみを (すなわちhttps://your_sequencing_facilitys_server.com/path_to_raw_read_files/) ファイルへの絶対パスを指定すると、このパスを fetch コマンドに挿入します。
- 解凍ファイル:".gz"拡張子を持つファイルの終わりをダウンロードした場合それは"gzip"コマンドを使用して圧縮されています。解凍、実行はコマンドラインでコマンドを解凍 (以下の例を参照してください)。
gunzip raw_read_files.gz - 自己完結型 .tar22としてサーバーにセル レンジャーの最新バージョンをダウンロードします。
-
重要な:ダウンロードする前に、Linux システムが23の最小要件を満たしているを確認します。8 コア インテル プロセッサと 64 GB の RAM と 1 TB のディスクの空き容量の最小値を確認します。
注: セル レンジャーは、構築済みの人間と齧歯動物の参照トランスクリプトームを提供します。これらは、GFP の24のような遺伝子を検出するcellranger mkrefコマンドを使用して変更できます。
-
重要な:ダウンロードする前に、Linux システムが23の最小要件を満たしているを確認します。8 コア インテル プロセッサと 64 GB の RAM と 1 TB のディスクの空き容量の最小値を確認します。
- シーケンサーの基本呼び出しファイル (Bcl) cellranger mkfastq コマンドを使用してから FASTQ ファイルを生成します。
注: プログラムが参照ゲノム (FASTQ ファイル) から raw 読み取りを合わせ、下流解析のための遺伝子細胞行列を生成します。参照ゲノムに読み取りの融着接続に対応の配置を実行星アライナを使用します。自信を持って割り当てられた読み取り (すなわち、単一の遺伝子アノテーションと互換性の読み取り) のみが使用される海を数えるため。- たとえば、cellranger mkfastq コマンドを使用します。
cellranger mkfastq - id = sample_name \
-=/パス/を/サンプルを実行 \
-csv=csv_file_containing_lane_sample_index.csv
- たとえば、cellranger mkfastq コマンドを使用します。
- Mkfastq を使用して単一細胞遺伝子数を生成する生成された FASTQ ファイルの cellranger カウントを実行します。
- たとえば、cellranger カウント コマンドを使用します。
cellranger カウント--id = sample_name \
トランスクリプトーム-refdata-cellranger-mm10-1.2.0 を = \
-fastqs =/絶対/パス/を/fastq/ファイル \
-サンプル same_sample_name_supplied_to_cellranger_mkfastq を = \
-localcores = 30
- たとえば、cellranger カウント コマンドを使用します。
- (省略可能) 複数のライブラリの集計: プールの cellranger aggr を使用した cellranger カウント出力サンプルを結合します。データを複数のライブラリからプールを含む単一遺伝子バーコード行列でこの結果します。Cellranger aggr コマンドの例:
cellranger aggr - id = sample_name \
-csv = csv_with_libraryID_ & _path_to_molecule_h5.csv \
-正規化 = マップ
注: ライブラリは正規化の 3 つのモードを使用してを集約することができます (マップされた、生のどれも)。マップされているすべてのライブラリは、同じシーケンス深さ25を持っているまで、それ検体より高い深さのライブラリとしてお勧めします。 - データの即時の可視化/解析、ルーペ携帯ブラウザー26x 10 に (cellranger カウントまたは cellranger aggr を使用して生成された) .cloupe 出力ファイルをインポートします。
7 scRNA Seq データセットの高度な分析
注: 完全な scRNA Seq ツール データベースは、scRNA ツール3,27で見つけることが。教師なしセル スーラ2を使用してクラスタ リングおよび pseudotemporal のためのフレームワークは、以下、モノクル6を使用してを注文します。この作業の多くは、ローカル コンピューターで行うことができます、ただし、次の手順は、機関サーバーを使用して、計算が完了したと仮定します。
- Linux プラットフォーム28を使用したサーバー アカウントに Miniconda の最新バージョンをダウンロードします。
- Conda29を使用して R の最新バージョンをインストールします。
- 30のテンプレートとして提供されているスーラ R スクリプトを使用してデータをプロットします。
注: スーラは、品質管理チェック、クラスタ リング、差動遺伝子発現解析、マーカー遺伝子の同定, 次元削減と scRNA シーケンス データの可視化を可能にする (R) ベースのツールキットです。Satija ラボ ウェブサイト31コーディング スーラとチュートリアルの包括的な説明を見つけることができます。 - テンプレート32として提供されているモノクル R スクリプトを使用してデータをプロットします。
注: モノクルは pseudotime 式変更の可視化と細胞運命決定を根底にある遺伝子を識別する別の R ベースのツールキットです。モノクル ウェブサイト33モノクル コーディングとチュートリアルの包括的な説明を見つけることができます。 - テストし、正しいデータセット34をプールの結果バッチ効果 kBET など R パッケージを使用できます。
8. NCBI GEO と SRA のサブミッション
注: ので再現性と再生シーケンス ファイルに簡単にアクセスを確保する、オンライン公開リポジトリに accessioned サブミッションが推奨または原稿提出する前に必要な。センター生物工学情報 (NCBI) の高スループット シーケンス データ35,36公開データ リポジトリは、シーケンス読み取りアーカイブ (SRA) 遺伝子表現オムニバス (GEO)。
- NCBI の GEO 投稿者アカウント37に登録します。
- (GEO 送信者のユーザー名とタイトル) のディレクトリ/フォルダーにコンパイルされる 3 つのコンポーネントを含む完全なジオ送信: 1) メタデータ レコード (プロジェクト提出につき 1 つのスプレッドシート);2) 生データ ファイル。3) データのファイルを処理します。
- ダウンロードして、メタデータ スプレッドシート38を完了します。次のパブリック GEO 提出はガイド (GSE100320)39として使用できます。ディレクトリにスプレッドシートを配置します。
- 場所生ディレクトリに、すべてのライブラリcellranger カウントスクリプトから生成されたデータ ファイル。
- 場所処理データ (フィルター処理された barcodes.tsv、genes.tsv、および matrix.mtx ファイル) から生成されたファイルすべてのライブラリのcellranger カウントスクリプト ディレクトリに。
- ジオ送信者の FTP サーバーの資格情報を使用すると、すべての 3 つのコンポーネントを含むディレクトリを転送します。Linux/Unix のユーザー: ncftp、lftp、ftp、sftp、および ncftpput を使用できます。
- すべての転送38の地域を通知します。
結果
ScRNA Seq のデータセットを分析するために設計されたオープン ソース パッケージのレパートリーは40 R ベースの言語3使用のこれらのパッケージの多数で劇的に増加しています。ここでは、これらのパッケージの 2 つを使用して代表的な結果が表示されます: の教師のグループを評価する単一の遺伝子発現に基づくセルとセルの不均一性を解決するために軌道に沿って単一セルを順序し生物を分解処理します。
図 4は、スーラの処理前の品質チェックおよび下流のバイオインフォマティクス解析の使用法を示します。まず、分析から逸脱した細胞をろ過して除去は、品質チェックは欠かせません。これは、ミトコンドリア遺伝子、遺伝子 (nGene) の数と海 (nUMI) 細胞ダブレットと外れ値を特定の数の割合を可視化する (図 4 b) をプロット ヴァイオリン (図 4 a) と散布図を使用します。明確な外れ値数の遺伝子、海、またはミトコンドリア遺伝子の割合で任意のセルは、スーラの FilterCells 関数を使用して削除されました。スーラは、主成分 (PC) を使用するので、クラスター細胞、統計的に有意な Pc を含めることを決定する分析スコアは重要なステップです。肘プロット (図 4 c) は、PC の選択、' PC の標準偏差」の高原を越えて Pc で軸が除外されたため使用されました。クラスタ リングの解像度は 4 (高解像度高い細胞塊、図 4e につながるに 0.4 (低解像度が少ない細胞塊、図 4 dにつながる) に至る、クラスター数することができます変更することを示すことを操作しても).低解像度、高解像度でまたサブタイプまたはセル人口の過渡的状態を表す場合がありますこれに対し各クラスターが定義済みのセルの種類を表す可能性が高いです。このインスタンスで低解像度のクラスターの設定は特定のクラスター (図 4 階) で最も高い発現遺伝子を識別する (スーラの DoHeatmap 関数を使用して) 式ヒートマップなどをさらに分析するために使用されました。このインスタンスで最も高い発現遺伝子を各クラスターが定義された遺伝子で一意に表されたことを示す、他のすべてのクラスターとクラスターの差分式を評価することによって同定しました。さらに、スーラの FeaturePlot 関数 (図 4 g) を使用して恒常的なプロットで個々 の候補者の遺伝子を視覚化できます。これにより解読するマクロファージを表すクラスターがあったかどうか。FeaturePlot を使用すると、両方クラスター 2 であることがわかった 4 Cd68 - パン ・ マクロファージ マーカーを表現していたと。
モノクル パッケージ、スーラで識別される細胞塊を裏付けに使用されたセル軌道や pseudotemporal の順序を構築するため生物学的プロセス (図 5) を要約します。Pseudotemporal 注文使用できますのサンプル コースを時間生物学的単一細胞発現されます。セルは中間の状態、2 つの代替セル運命の分岐ポイントを解決する pseudotemporal 連続体に沿って注文することができます、各運命の取得の基礎となる遺伝子の署名を識別します。まずスーラのろ過と同様、すべてのセルの mRNA の分布は通常、図 5 aで指定された上限と下限の間落ちたログするように質の悪い細胞が削除されました。モノクルの newCellTypeHierarchy 関数を使用して、単一のセル分類されたし、知られている系統マーカー遺伝子 (図 5 b、 5 c) を使用してカウントします。たとえば、PDGF の受容器のアルファや線維芽細胞特定蛋白質 1 を発現する細胞は、線維芽細胞を定義するための条件を作成する細胞の種類 #1 に割り当てられました。次に、この人口 (携帯型 #1) が評価され、線維芽細胞の軌跡を解読します。これを行うは、集団内で極端な状態を表すセルを比較し、(図 5 d) 人口の残りのセルの順序の差動遺伝子を発見、モノクルの差分 GeneTest 関数が利用されました。多様体学習法 (非線形次元削減の種類) を適用するとすべてのセルの間で、pseudotemporal のパスに沿った座標が割り当てられていた。この軌道は、セル状態 (図 5e) と pseudotime (図 5 階) で可視化しました。

図 1: フローチャート。全体の動物の準備から単一の分析へのステップは細胞 RNA Seq データセットの公開リポジトリへのデータセットの最後の送信です。ゲルビーズ エマルジョン (宝石) では数千の単一のセルをカプセル化するバーコード オリゴヌクレオチドとビーズを参照してください。この図の拡大版を表示するのにはここをクリックしてください。
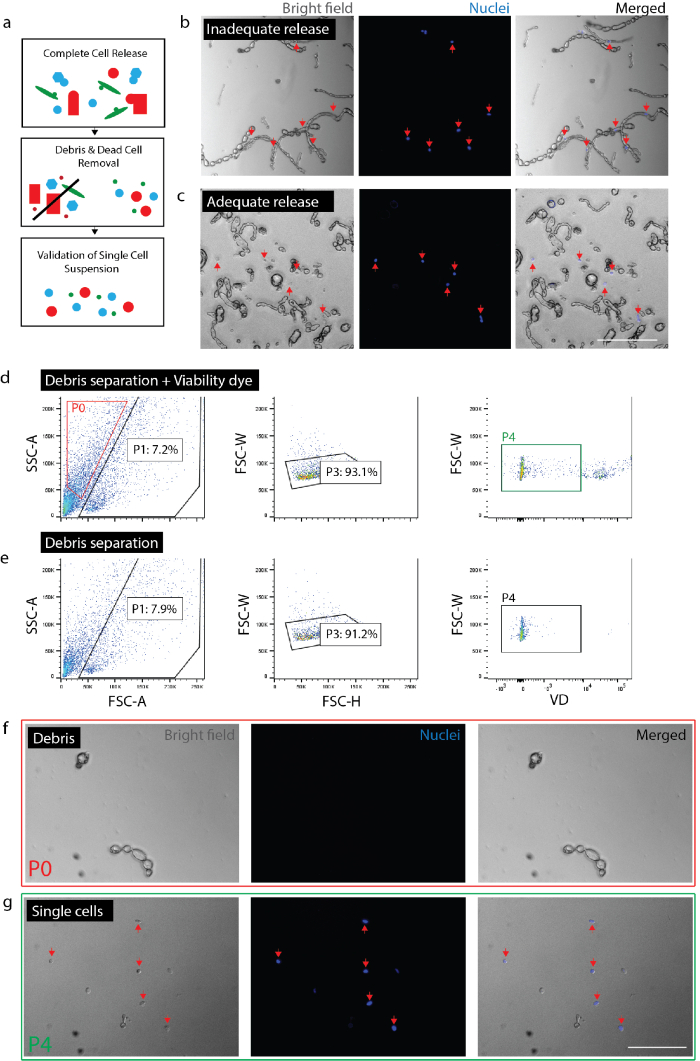
図 2: 神経組織から実行可能な単一細胞懸濁液を作成します。(a) 品質管理チェックの概要を漫画します。(b) 細胞および細胞残骸まだ残骸 (赤矢印) に組み込まれています。(c) 細胞残骸 (赤矢印) からリリースされました。(d) セル FACS により分離。P0: 破片端数P1: 細胞のような端数Duplets; の P3: 除外P4: 生存率色素 (Sytox オレンジ) 否定的な割合です。(e) の生存率色素コントロール。(f) P0 分数を表すのイメージは、破片を分離しました。(g) 分離可能な細胞 (赤矢印) を表す P4 分数のイメージ。(b)(c)(f)、(g) あった核色素イメージングの前に 20 分を追加しました。スケール バー: 80 μ m.この図の拡大版を表示するのにはここをクリックしてください。

図 3: 浅いシーケンス処理サンプル X 10 の回復されたセルの数を予測する。(自信を持ってマップリードにより MiSeq によって生成された csv リスト携帯バーコードとその対応する海の a) の例 (サンプル 1.6) をカウントします。(サンプル 1.6 b) バーコード ランク プロットは、携帯バーコードの機能として、海の数に 1 つの大幅な低下を示しています。破線と固体の線は、セルと背景ビジュアル検査によって決定されるカットオフを表しています。(c) 携帯バーコードは、浅いシーケンスは、サンプル 1.6 のセルの数を正確に近似明らかにセル レンジャー パイプラインのポスト HiSeq を使用して観察。(d) 浅いシーケンスに基づくフロー セル設定の例は、セルの見積もりを派生します。サンプル 1.6 の浅いシーケンス予測 3480 細胞 1.17 レーンに割り当てられたことを確認 > HiSeq のセル配列範囲あたり 100,000 の読み取り。注: すべてのレーンは、100% を追加する必要があります。この図の拡大版を表示するのにはここをクリックしてください。
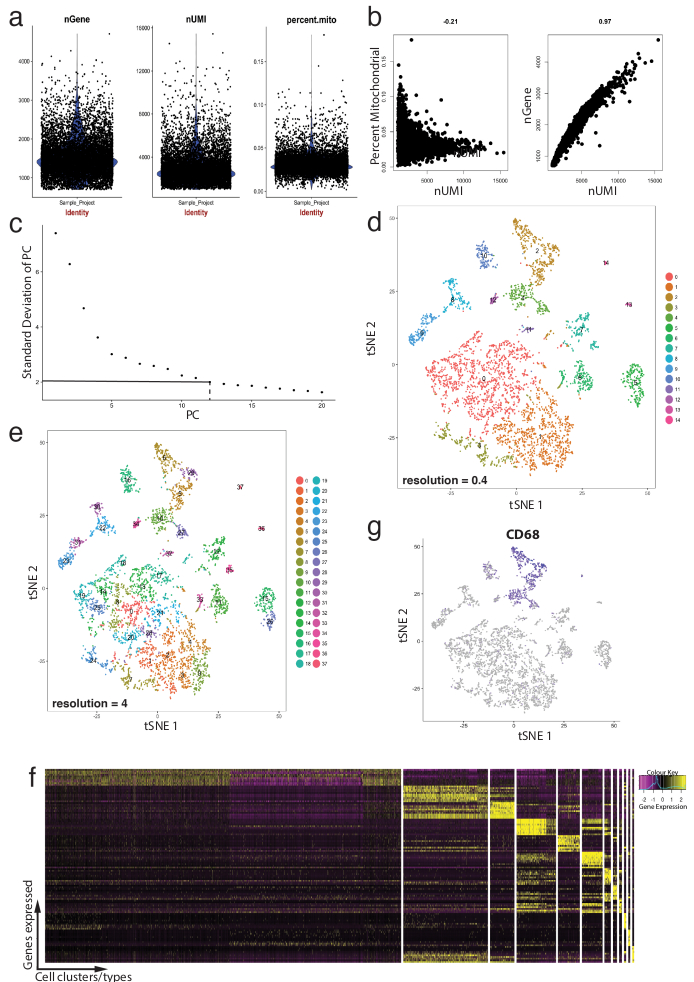
図 4: 品質管理とスーラ R パッケージを使用して単一細胞 RNA Seq データセットのバイオインフォマティクス。(a) 品質管理指標遺伝子の数、数分子の一意の識別子 (Umi) のミトコンドリアのゲノムへのマッピングの成績証明書の割合などのプロット。(b) サンプルの遺伝子は、ミトコンドリアの転写産物と Umi の常軌を逸したレベルで検出セルがプロットされます。(c) サンプル肘のプロットが統計的に有意なパソコンのアドホックの決定に使用されます。破線や一点鎖線は、明確な「肘」がグラフに明らかになるカットオフを表しています。この肘の前に PC の寸法は、下流の分析に含まれます。(d, e)恒常的なプロットを使用して低次元空間における 2 つの異なる解像度で可視化グラフ ベースの細胞塊。(f) 上のマーカー遺伝子 (黄) 各クラスターのスーラの DoHeatmap 関数を使用して式ヒートマップで視覚化。(g) 可視化マーカー遺伝子の発現、たとえば、Cd68 マクロファージ スーラの FeaturePlot 関数を使用して (紫) を表します。2、このデータセットのパネル (ニ) 4 クラスターを表しますマクロファージが示唆されました。この図の拡大版を表示するのにはここをクリックしてください。

図 5: 細胞分類とモノクル ツールキットを使用して peudotemporal 軌道に沿って順序。(a) サンプルのすべてのセルに (海カウントから推論) mRNA の分布を調べること。のみ細胞 mRNA 0 ~ ~ 20,000 下流解析に使用されました。(b, c)割り当てることと知られている系統の細胞マーカーに基づく細胞の種類を数えます。たとえば、PDGF の受容器のアルファや線維芽細胞特定蛋白質 1 を発現する細胞に割り当てられていた携帯型 #1 パン線維芽細胞を表すモノクルの newCellTypeHierarchy 関数を使用します。円グラフ (b) と (c) のテーブルとして、異なる種類の細胞の数を視覚化できます。(d) セルを使用してたとえば、平均式対遺伝子分散を示す散布を使用してセルを視覚化できる並べ替えに使用する遺伝子型 #1 (繊維芽細胞)。赤色の曲線は、モノクルの estimateDispersions 関数を使用して平均分散モデルによる計算順序のための遺伝子のためのカットオフを示しています。このカットオフを満たす遺伝子は、下流の pseudotime 順序に使用されました。(e, f)色のセルの「状態」(e)、モノクルに割り当てられた"Pseudotime"(f) 減らされた次元セル軌跡の可視化。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
このプロトコルは、どのように単一細胞の適切な準備できる単一細胞の何千もの転写の不均一性を明らかにし、差別の機能の状態や、組織内で一意の携帯 id を示します。プロトコル蛍光レポーター蛋白質や遺伝子組換えのツールは必要ありませんし、人間からのそれらを含むさまざまな組織の単一細胞の分離に適用できます。各組織を念頭、ユニークで、このプロトコルの調整/修正の程度が必要になります。
細胞内の多様な非常にダイナミックな転写プログラムは、ゲノムの単一セルの値を強調しています。別に高品質の RNA を隔離する、質の高いデータセットに必要な重要な試料前処理は確実に細胞が組織から完全に解放されるおよび細胞、健康かつそのままであります。これは容易に細胞を収集公開、循環細胞など、細胞の保持は、組織でリンパ組織などでは比較的まっすぐ進む。しかし、これは大規模な距離、周辺の細胞外マトリックスと細胞構造を維持することに関与する多くの場合堅い細胞骨格タンパク質にまたがる高度細胞アーキテクチャのための他の成体組織のために挑戦することができます。セルの完全なリリースの技法の適切な解離とも必要な厳格なと多くの場合時間のかかる処理が mRNA の品質とセルの整合性を変更可能性があります。さらに、酵素による分解に使用される高温転写署名29,30にも影響します。プロトコルの目的は、品質管理にチェック、最適化はこれらの障害を克服するために助けることができる方法を実証する有髄の大人の神経や細胞外マトリックスの豊富な大人の皮膚などの組織を使用です。
ScRNA Seq 実験を設計する際の主要な考慮事項は、シーケンスの深さの選択です。シーケンスを高度に多重化して読み取ることがされて非常に低いドロップ Seq2最大 500 万読み取り/セル14スマート シーケンスなどフルレングス RNA シーケンスのメソッドを使用してを使用してから深さがありますほとんどの scRNA Seq 実験は、細胞型分類41,42通常十分であるシーケンス 10,000 読み取り/セル、低中程度の高式成績証明書を検出できます。浅いシーケンスの深さは、細胞の何千も特殊な集団のせいにする自信を持ってする必要があります複雑な組織で、稀な細胞集団を検出しようとしたときに、シーケンシング コストを保存する値のことです。しかし、遺伝子発現と微妙な転写署名に関連付けられたプロセスに関する詳細な情報が必要な場合、浅い深さシーケンスが不十分。現在、500,000 の読み取り/セルでセルに遺伝子の大多数が検出されたが、プロトコルによって異なりますが、組織型43,44と推定されます。フルレングスのトラン スクリプトのシーケンスがアセンブリの必要性を回避、小説や珍しいスプライスバリアントを検出できます、したがって、シーケンス コストは多くの場合複雑な組織システムを構成する細胞の数を調べて、このようなアプローチをスケーリングを制限します。対照的に、3' タグ付きの単一セル ライブラリ通常このプロトコルで記述されているものより低い複雑さと浅くシーケンスを必要とします。5 つのサポートされているシーケンサーの 1 つで説明されているプロトコルを使用して生成されたライブラリをシーケンス処理できることに注意してくださいすることが重要です: NovaSeq 1)、2) HiSeq 3000/4000、3) HiSeq 2500 急速な実行と高出力、4) NextSeq 500/550、MiSeq 5)。
繊細な組織とまだ処理セルの必要性を削減する単一細胞 RNA シーケンスへの代替アプローチは単一細胞 RNA シーケンスの利点のいくつかを維持、単一核45から RNA の解析です。このアプローチは、RNA の劣化と核の適切なリリースを確保するためより多くの極端な措置の削減より迅速な処理を可能に、したがって可能性が高い特定の組織内のすべてのセルを表す転写プロファイルのもっと自信を持って捕獲のためことができます。もちろん、転写活性の実験的な目的がこのアプローチ可能性がありますまたは適さない場合があります興味あるものによって従って特定のセル内の一部のみ提供だろう、これ。
特定組織内の細胞のアイデンティティの完全な特性評価、ほか scRNA Seq データセットの最も貴重な分析の 1 つは '定義' 細胞集団間で中間転写状態の評価です。これらの中間の状態は、伝統的な一括 RNA シーケンスのアプローチでは不可能でした特定の集団内のセル間の系列リレーションシップへの洞察力を伝えることができます。これを明らかにするいくつかの scRNA Seq bioinformatic ツールを開発されている今。このようなツールは、たとえば、がん細胞の多様な端末の運命や、静止状態との間の往復の免疫細胞に成熟幹細胞の発癌性/転移状態に移行に関与するプロセスを評価できます。細胞のトランスクリプトームの微妙な違いは、FateID のような最近開発された bioinformatic ツールが47を推論できること、血統バイアスを示す場合もあります。移行細胞の区別が難しいことができますので微妙かもしれない特定の転写の違いを確認するため、深いシーケンスが必要な46あります。幸いなことに、別のフロー ・ セルでライブラリを再実行してさらにデータセットを調査に興味がある場合、浅くシーケンスされたライブラリをカバーは増やすことができます。
一緒に取られて、このプロトコルは、発現プロファイルの 1 つの実験内の単一セルの何千もの何百もすることができます簡単に適応するためのワークフローを提供します。ScRNA Seq データセットの最終的な品質は、最適化された細胞の隔離、フローサイトメトリー、cDNA ライブラリを生成、および生遺伝子バーコード行列の解釈に依存しています。この目的のためには、このプロトコルは、多様な組織の種類の研究を有効にする簡単に変更することができますすべての重要なステップの包括的な概観を提供します。
開示事項
開示
謝辞
我々 はカルガリーの大学で動物実験施設スタッフだけでなく、UCDNA サービス施設でのサポート スタッフを認めます。バイオインフォマティクスの彼のサポートのためのマットの Workentine と彼のテクニカル サポートのイェンス Durruthy に感謝しますこの作品はバトンとアルバータ子供の健康研究所フェローシップ (バッハ) 機構の新しい賞機構助成金 (r. m. と j. b.) によって資金を供給します。
資料
| Name | Company | Catalog Number | Comments |
| Products | |||
| RNAse out | Biosciences | 786-70 | |
| Pentobarbital sodium | Euthanyl | 50mg/kg | |
| HBSS | Gibco | 14175-095 | |
| Dispase 5U/ml | StemCell Technologies | 7913 | 5 mg/ml |
| Collagenase-4 125 CDU/mg | Sigma-Aldrich | C5138 | 2 mg/ml |
| DNAse | Sigma-Aldrich | DN25 | 10mg/ml |
| BSA | Sigma-Aldrich | A7906 | |
| 15 ml Narrow bottom tube VWR® High-Performance Centrifuge Tubes | VWR | 89039-666 | |
| Sytox Orange Viability Dye | Molecular Probes | 11320972 | 1.3 nM/µl |
| Nuc Blue Live ReadyProbes | Invitrogen | R37605 | |
| Agilent 2100 Bioanalyzer High senitivity DNA Reagents | Agilent | 5067-4626 | |
| Kapa DNA Quantification Kit | Kapa Biosystems | KK4844 | |
| Chromium Single Cell 3' reagents | 10x Genomics | ||
| Equipment | |||
| BD FACSAria III | BD Biosciences | ||
| Agilent 2100 Bioanalyzer Platform | Agilent | ||
| Illumina® HiSeq 4000 | Illumina | ||
| Illumina® MiSeq SR50 | Illumina | ||
| 10X Controller + accessories | 10x Genomics | ||
| Software | |||
| The Cell Ranger | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/overview/welcome | |
| Loupe Cell Browser | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest | |
| R | https://anaconda.org/r/r |
参考文献
- Shalek, A. K., et al. Single-cell RNA-seq reveals dynamic paracrine control for cellular variation. Nature. 510, 363-369 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161, 1202-1214 (2015).
- Zappia, L., Phipson, B., Oshlack, A. Exploring the single-cell RNA-seq analysis landscape with the scRNA-tools database. bioRxiv:206573. , (2018).
- Dulken, B. W., Leeman, D. S., Boutet, S. C., Hebestreit, K., Brunet, A. Single cell transcriptomic analysis defines heterogeneity and transcriptional dynamics in the adult neural stem cell lineage. Cell Reports. 18 (3), 777-790 (2017).
- Llorens-Bobadilla, E., et al. Single-Cell Transcriptomics Reveals a Population of Dormant Neural Stem Cells that Become Activated upon Brain Injury. Cell Stem Cell. 17 (3), 329-340 (2015).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32, 381-386 (2014).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14, 1083-1086 (2017).
- Mayer, C., et al. Developmental diversification of cortical inhibitory interneurons. Nature. 555 (7697), 457-462 (2018).
- Stratton, J. A., et al. Purification and Characterization of Schwann Cells from Adult Human Skin and Nerve. eNeuro. 4 (3), (2017).
- Biernaskie, J. A., McKenzie, I. A., Toma, J. G., Miller, F. D. Isolation of skin-derived precursors (SKPs) and differentiation and enrichment of their Schwann cell progeny. Nature Protocols. 1 (6), 2803-2812 (2007).
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Agilent Available from: https://www.agilent.com/en-us/library/usermanuals?N=135 (2018)
- Kolodziejczyk, A. A. Single Cell RNA-Sequencing of Pluripotent States Unlocks Modular Transcriptional Variation. Cell Stem Cell. 17, 471-485 (2015).
- Jaitin, D. A., et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 343, 776-779 (2014).
- Pollen, A. A., et al. Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex. Nature Biotechnology. 32, 1053-1058 (2014).
- . Sequencing Requirements for Single Cell 3' Available from: https://support.10xgenomics.com/single-cell-gene-expression/sequencing/doc/specifications-sequencing-requirements-for-single-cell-3 (2018)
- . Introduction to R Available from: https://www.datacamp.com/courses/free-introduction-to-r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics#39; Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Creating a Reference Package with cellranger mkref Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/advanced/references (2018)
- . System Requirements Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/overview/system-requirements (2018)
- . Software Downloads Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest (2018)
- . Aggregating Multiple Libraries with cellranger aggr Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/using/aggregate#depth_normalization (2018)
- . Loupe Cell Browser Gene Expression Tutorial Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/tutorial (2018)
- . A table of tools for the analysis of single-cell RNA-seq data Available from: https://www.scrna-tools.org/ (2018)
- . Downloading conda Available from: https://conda.io/docs/user-guide/install/download.html (2018)
- . r / packages / r 3.5.1 Available from: https://anaconda.org/r/r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . Seurat - Guided Clustering Tutorial Available from: https://satijalab.org/seurat/pbmc3k_tutorial.html (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- Github. . An R package to test for batch effects in high-dimensional single-cell RNA sequencing data. , (2018).
- Edgar, R. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30, 207-210 (2002).
- Leinonen, R., Sugawara, H., Shumway, M. The sequence read archive. Nucleic Acids Research. 39, D19-D21 (2011).
- . GenBank Submission Portal Wizards Available from: https://www.ncbi.nlm.nih.gov/account/register/?back_url=/geo/submitter/ (2018)
- . Submitting data Available from: https://submit.ncbi.nlm.nih.gov/geo/submission/ (2018)
- Shah, P. T., et al. Single-Cell Transcriptomics and Fate Mapping of Ependymal Cells Reveals an Absence of Neural Stem Cell Function. Cell. 173, 1045-1057 (2018).
- Anon, Method of the Year 2013. Nature Methods. 11, 1 (2013).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144, 3625-3632 (2017).
- Wu, Y. E., Pan, L., Zuo, Y., Li, X., Hong, W. Detecting activated cell populations using single-cell RNA-seq. Neuron. 96, 313-329 (2017).
- Zeigenhain, C., et al. Comparative Analysis of Single-Cell RNA Sequencing Methods. Molecular Cell. 65 (4), 631-643 (2017).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nature Methods. 11 (1), 41-46 (2014).
- Habib, N., et al. Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science. 353 (6302), 925-928 (2016).
- Janes, K. A. Single-cell states versus single-cell atlases - two classes of heterogeneity that differ in meaning and method. Current Opinions in Biotechnology. 39, 120-125 (2016).
- Herman, J. S., Sagar, D., Grün, FateID infers cell fate bias in multipotent progenitors from single-cell RNA-seq data. Nature Methods. 15 (5), 379-386 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved