Method Article
ترانسكريبتوميكس خلية واحدة على أساس المتوازية الحبرية من أنسجة الثدييات الكبار
* These authors contributed equally
In This Article
Summary
هذا البروتوكول يصف العمليات العامة ومراقبة الجودة الشيكات اللازمة لإعداد الخلايا المفردة الثدييات الكبار صحية التحضيرات تسلسل الحمض النووي الريبي في خلية مفردة المستندة إلى الحبرية، وارتفاع الإنتاجية. ترتيب المعلمات، محاذاة قراءة وتحليل المصب خلية واحدة bioinformatic تتوفر أيضا.
Abstract
تحليل التعبير الجيني خلية مفردة عبر آلاف خلايا الفردية داخل الأنسجة أو المكروية أداة قيمة لتحديد تكوين الخلية، والتمييز الدول الوظيفية، والمسارات الجزيئية الكامنة وراء الأنسجة الملاحظة وظائف والسلوك الحيواني. ومع ذلك، يمكن أن يكون تحديا عزل الخلايا المفردة سليمة وصحية من أنسجة الثدييات الكبار للتحليل الجزيئي خلية مفردة المصب اللاحقة. هذا البروتوكول يصف العمليات العامة ومراقبة جودة التدقيق اللازم للحصول على الأعمال التحضيرية خلية مفردة الكبار عالية الجودة من على الجهاز العصبي أو الجلد التي تم تمكين تسلسل خلية مفردة غير متحيزة اللاحقة الجيش الملكي النيبالي والتحليل. كما يتم توفير مبادئ توجيهية لتحليل bioinformatic المتلقين للمعلومات.
Introduction
مع تطور الإنتاجية العالية التكنولوجيا خلية مفردة1،2 والتقدم في أدوات سهلة الاستعمال المعلوماتية الحيوية على مدى العقد الماضي3، برز حقل جديد لتحليل التعبير الجيني عالية الدقة – خلية واحدة تسلسل الحمض النووي الريبي (سرنا-Seq). ووضعت دراسة التعبير الجيني خلية مفردة أولاً لتحديد عدم التجانس داخل خلية محددة بالسكان، كما هو الحال في الخلايا الجذعية أو الخلايا السرطانية، أو لتحديد السكان نادرة من الخلايا4،5، الذي قد لا يمكن تحقيقه باستخدام تقنيات تسلسل الحمض النووي الريبي السائبة التقليدية. ومكنت الأدوات بيوينفورماتيك تحديد هوية السكان الفرعية رواية (سورا)2، التصور وسام الخلايا على طول بسويدوتيمي الفضاء (مونوكل)6، تعريف الشبكات إشارات نشطة داخل أو بين السكان ( المناظر الطبيعية الخلابة)7، والتنبؤ بجمعية خلايا مفردة في مساحة ثلاثية الأبعاد اصطناعية (سورا، وأكثر)8. مع هذه التحليلات الجديدة والمثيرة المتاحة للمجتمع العلمي، سرنا Seq سريعة-أصبح نهج قياسية جديدة لتحليل التعبير الجيني.
على الرغم من الإمكانات الهائلة ل Seq سرنا، skillsets التقنية المطلوبة لإنتاج مجموعة بيانات نظيفة وتفسير النتائج بدقة يمكن أن يكون تحديا للقادمين الجدد. هنا، بدءاً من عزل الخلايا المفردة من الأنسجة الابتدائية كاملة للتصور وعرض البيانات للنشر يقدم بروتوكول أساسي، ولكنه شامل، (الشكل 1). أولاً، عزل الخلايا المفردة صحية يمكن أن يعتبر تحديا، كما أنسجة مختلفة تتفاوت في درجة حساسية الهضم الأنزيمي والتفكك الميكانيكية اللاحقة. هذا البروتوكول يوفر الإرشاد في هذه الخطوات العزلة، ويحدد نقاط التفتيش مهمة مراقبة الجودة في جميع مراحل العملية. وثانيا، فهم التوافق ومتطلبات بين تكنولوجيا خلية مفردة وتسلسل الجيل القادم يمكن أن يكون مربكاً. هذا البروتوكول يوفر مبادئ توجيهية لتنفيذ منصة المتوازية خلية واحدة سهلة الاستخدام المستندة إلى الحبرية، وإجراء تسلسل. وأخيراً، برمجة الحاسوب شرطا أساسيا هاما لتحليل مجموعات البيانات ترانسكريبتوميك خلية واحدة. هذا البروتوكول يوفر الموارد للشروع في العمل مع لغة البرمجة R، ويقدم توجيهات بشأن تنفيذ مجموعتي R سرنا Seq-الخاصة بشعبية. معا، هذا البروتوكول يمكن توجيه الوافدين الجدد في أداء تحليل Seq سرنا للحصول على نتائج واضحة، والتفسير. هذا البروتوكول يمكن أن يتكيف مع معظم الأنسجة في الماوس، والأهم من ذلك يمكن تعديلها للاستخدام مع الكائنات الأخرى، بما في ذلك الأنسجة البشرية. وسوف يلزم إدخال تعديلات اعتماداً على الأنسجة والمستخدم.
وهناك عدة اعتبارات يجب وضعها في الاعتبار حين عقب هذا البروتوكول؛ 1) بعد جميع المبادئ التوجيهية لمراقبة الجودة في الخطوات 1 و 2 من هذا البروتوكول يوصي بما في ذلك، لضمان تعليق خلية واحدة فقط قابلة للتطبيق لكافة الخلايا داخل عينة مصلحة مع ضمان دقيقة خلية الإجمالي عدد من التهم الموجهة إليه (الملخصة في رقم 2 ). حالما يتحقق ذلك، وإذا ما اتبعت جميع الشروط الأمثل، يمكن أن يكون إسقاط خطوات مراقبة الجودة (لتوفير الوقت--الحفاظ على جودة الجيش الملكي النيبالي، والحد من خلية الخسارة). تؤكد عزلة ناجحة من استمرارية ارتفاع الخلايا المفردة من الأنسجة لمصلحة هو الغاية يوصي قبل أي المصب تجهيز. 2) تقنيات تفارق المفرطة دون قصد نظراً لبعض أنواع الخلايا أكثر حساسية من غيرها التأكيد، يمكن أن التحيز السكان، ولذلك الخلط تحليل المتلقين للمعلومات. الانفصال لطيف دون قص الخلوية لا لزوم لها والهضم أمر بالغ الأهمية لتحقيق ارتفاع غلة الخلوية وتمثيل دقيق لتكوين الأنسجة. قوي القص تحدث خلال الخطوات تريتوريشن، ونظام مراقبة الأصول الميدانية، واستثارة. 3) كما مع أي عمل الجيش الملكي النيبالي، من الأفضل أن إدخال رناسي إضافية صغيرة في العينة قدر الإمكان أثناء الإعداد. وهذا سيساعد في الحفاظ على الحمض النووي الريبي عالية الجودة. استخدام حلول المانع ribonuclease مع الشطف بأدوات نظيفة وأي معدات غير خالية من رناسي ولكن تجنب المنتجات المعالجة ديبك. 4) أداء الأعمال التحضيرية، في أسرع وقت ممكن. وهذا سيساعد على الحفاظ على الحمض النووي الريبي عالية الجودة، والحد من موت الخلايا. اعتماداً على طول تشريح الأنسجة وعدد الحيوانات، النظر في بدء تشريح/استعدادات متعددة في نفس الوقت. 5) إعداد الخلايا على الجليد عند الخلية من الممكن الحفاظ على الحمض النووي الريبي عالية الجودة، والحد من موت الخلايا، وبطء النشاط إرسال الإشارات والنسخي. بعض أنواع الخلايا (مثلاً، العَدلات) أن كان تجهيز المثلج المثل الأعلى لمعظم أنواع الخلايا، أداء أفضل عند معالجة في درجة حرارة الغرفة. 6) تجنب الكالسيوم والمغنيسيوم، يدتا والمنتجات المعالجة ديبك أثناء إعداد الخلية.
Protocol
جميع البروتوكولات المذكورة هنا هي وفقا وأقرته "لجنة رعاية الحيوان" جامعة كالغاري.
1-الناي بالانسجة (1 يوم)
- Euthanize الفئران مع جرعة بينتوباربيتال الصوديوم (القائمة، 50 مغ/كغ) أو حسب الاقتضاء وفقا للبروتوكول الأخلاق الحيوانية. إزالة الشعر غير المرغوب فيه من الظهر والساقين من الماوس، ثم الإيثانول-تعقيم منطقة التشريح.
- تشريح الأنسجة أو المكروية للفائدة. لهذا البروتوكول، نستخدم أنسجة الجلد والأعصاب لإثبات جينيراليزابيليتي ترانسكريبتوميكس خلية واحدة على أساس المتوازية الحبرية عقب تفكك النسيج الكبار.
- للعصب الوركي، استخدام بروتوكول مفصلة تبين ستراتون et al. 9-باختصار، قطع الجلد بعيداً عن منطقة هند من الخلف/الساقين بالماوس. جعل شق على طول الفخذ مع شفرة مشرط معقم. استخدام الملقط غرامة ومقص لكشف وإزالة العصب الوركي.
- للجلد مرة أخرى، واستخدام بروتوكول مفصلة تبين بيرناسكي et al. 10-بإيجاز، تشريح الجلد الخلفي الظهرية شقوق من كتفا إلى كتف، وعبر الردف وأسفل الظهر باستخدام الملقط غرامة والمقص. قص الجلد إلى شرائح رقيقة (سمك 0.5 سم) باستخدام شفرة مشرط معقم.
- تغسل الأنسجة 2 مرات مع حبس المثلج، وإزالة النسيج الضام غير المرغوب فيها، ورواسب الدهون أو الحطام تحت مجهر تشريح.
- الأدمة الجلد فقط، تطفو الشرائح في ديسباسي (5 ملغ/مل، 5 U/mL) في حبس لمدة 30-40 دقيقة عند 37 درجة مئوية. جراحيا يفصل البشرة عن الأدمة. تجاهل البشرة أو ننأى بزيادة استخدام التربسين إذا للفائدة.
- فرم العينة إلى أجزاء 1-2 مم باستخدام شفرات مشرط عقيم ووضعها في إنزيم كولاجيناز الباردة-رابعا طازج مذاب 2 مغ/مل (2 ملغ/مل، الاتحاد الديمقراطي المسيحي/mg 125، في وسائل الإعلام F12).
- للأعصاب، استخدم ~ 500 ميليلتر كل الأعصاب الوركي x 2. للجلد، واستخدام مل ~ 8 في الماوس 1 x الجلد مرة أخرى.
ملاحظة: الأنسجة وينبغي أن يكون تماما غارقة في الحل الرابع كولاجيناز. من الأهمية بمكان أن إنزيمات الهضم أي معالجة وتخزين واعدت على نحو ملائم. إذا تركت الإنزيمات في درجة حرارة الغرفة لفترات طويلة من الزمن، عزل الخلايا المفردة ستتطلب المفرطة تريتوريشن الميكانيكية والتقليل من بقاء الخلية. يمكن أن تتكون كولاجيناز-رابعا أيضا في خلية ثقافة وسائل الإعلام فيها بقاء الخلية المثلى. ومع ذلك، هذا يمكن أن يغير نشاط إنزيم أو التوقيع النسخي ينبغي أن يكون الأمثل لذلك المستخدم.
- للأعصاب، استخدم ~ 500 ميليلتر كل الأعصاب الوركي x 2. للجلد، واستخدام مل ~ 8 في الماوس 1 x الجلد مرة أخرى.
- احتضان العينة في الإنزيم في حمام 37 درجة مئوية لمدة 30 دقيقة مع لطيف تهز كل 10 دقيقة. شاكر وضعها في 37 درجة مئوية أيضا بديلاً مناسباً.
- تريتوراتي مع بيبيتور P1000 20-30 مرة في إضافة الإنزيم بعد 30 دقيقة.
- كرر تريتوريشن كل 30 دقيقة حتى يظهر الحل غائم وقطع الأنسجة يتم فصلها إلى حد كبير.
ملاحظة: تأكد من الإصدار الكامل من الخلايا (الشكل 2، ج 2). لتأكيد الإفراج الكامل، لوحة الخلايا مع نشاط الأزرق (2 قطرات كل 1 مل) وبعد 20 دقيقة، فحص تحت المجهر التأكد من نوى جميع المرتبطة بالخلايا المفردة بدلاً من الحطام. من المهم أن تحقق الدرجة من الإفراج عن الخلية داخل تجربة معينة لكل نوع الأنسجة أو شرط. في أنسجة تليفية (أي، الإصابة المزمنة) أو أنسجة البالغين يصب، سوف تختلف خلايا هائلا من الإصابات الحادة أو الأنسجة الجنينية. هذا مهم خاصة لبعض أنواع الخلايا أقل احتمالاً لإطلاق سراح من الأنسجة من غيرها، وهكذا ترتبط بشكل تفضيلي باستثناء تلك الخلايا من تحليل المتلقين للمعلومات.- للعصب، ننأى بالانسجة ل 0.5-1.5 ساعة المجموع. للجلد، وينأى بالانسجة لمجموع 2 ساعة (في آخر ساعة من الحضانة، إضافة الدناز (1 ملغ/مل) للعينة الجلد).
- تصفية مرتين مع عامل تصفية 40 ميكرومتر. شطف عامل التصفية مع المثلج 1% جيش صرب البوسنة/حبس.
- الطرد المركزي في 260 غ س ل 8 دقيقة. قم بإزالة المادة طافية.
- ريسوسبيند بيليه الخلية في حبس المحتوية على جيش صرب البوسنة 1% باستخدام تلميح تحمل على نطاق، ووضع على الجليد. حجم استثارة يستند حجم الأنسجة (800 ملغ من الوزن الرطب للجلد = ميليلتر 800 وحدة التخزين؛ 10 ملغ الرطب الوزن للأعصاب = 100 ميليلتر حجم).
- بشكل اختياري، ابدأ مع حجم استثارة منخفضة وثم ضبط حسب الضرورة استناداً إلى معدل التدفق (الأحداث في الثانية الواحدة) على أرز نظام مراقبة الأصول الميدانية. كثافة النوع الأكثر كفاءة (زيادة عدد الخلايا التي تم جمعها في حين قلل الوقت) لمجموعات هو 3,000-7,000 الأحداث في الثانية.
- إذا كان استخدام صبغة الجدوى، تأخذ بها سوباليقوت لعنصر تحكم أونستاينيد. ثم قم بإضافة 1:15,000 صلاحية صبغ (الأوراق المالية: 20,000 شمال البحر الأبيض المتوسط/ميليلتر) للعينة (1.3 تركيز شمال البحر الأبيض المتوسط/ميليلتر النهائي) استخدام تلميح تحمل على صعيد الحد من القص.
ملاحظة: من المهم للتحقق من درجة موت الخلية داخل تجربة معينة لكل نوع الأنسجة أو الشرط. بعض أنواع الخلايا داخل عينة هم أكثر عرضه للوفاة من غيرهم، وبالتالي استبعادهم تفضيلي من تحليل المتلقين للمعلومات.- تبني نموذج مع صلاحية صبغ لمدة 5-10 دقائق على الجليد في الظلام. قم بإضافة 4 مل من المثلج 1% جيش صرب البوسنة/حبس بعينه. الطرد المركزي في 260 غ س لمدة 8 دقائق لإزالة الصبغة الزائدة صلاحية. علاج سوباليقوت مع لا صلاحية صبغ التحكم أونستينيد بنفس الطريقة.
2-عزل خلايا سليمة وصحية (1 يوم)
- تأكد من أن يتبع مرفق نظام مراقبة الأصول الميدانية المناسبة fluorescence تنشيط الخلية فرز المعلمات (نظام مراقبة الأصول الميدانية).
- إعداد الجهاز نظام مراقبة الأصول الميدانية في وقت مبكر للتأكد من أنها جاهزة بمجرد اكتمال الطرد النهائي في الخطوة 1، وضمان أن تظل مقصورة جمع الباردة استخدام كتل الجليد.
- استخدم المعلمات التالية: معدل التدفق: 1.0 (يتوافق مع تقريبا 10 ميليلتر/دقيقة)؛ عامل التصفية: 1.5 ND؛ حجم الفوهة: 100 ميكرومتر؛ مبعثر إلى الأمام: 80-180 الخامس (التغيير حسب الضرورة من أجل التمييز بين حجم الأحداث)؛ مبعثر الجانب: 150-220 V (التغيير حسب الضرورة من أجل التمييز بين تقسيمات/شكل الأحداث)؛ الليزر: 100-400 الخامس (التغيير حسب الضرورة من أجل التمييز بين صلاحية صبغ مقابل إيجابية الأحداث السلبية وهذا الاختيار ضد أي سيطرة صبغ صلاحية)؛ غيتس: تغيير حسب الاقتضاء لضمان جمع كافة الخلايا. انظر الشكل 2dز-2.
ملاحظة: معلمات نظام مراقبة الأصول الميدانية تعتمد اعتماداً كبيرا على أنواع الخلايا المستخدمة فارز ولذلك ينبغي أن يكون الأمثل من قبل المستخدم.
- إعداد 15 مل أنابيب ضيقة لأسفل مع 8 مل من المثلج 1% جيش صرب البوسنة/حبس لمجموعات العينة. ثابت داخل الأنبوب والتوتر السطحي يمكن أن تؤثر على كفاءة جمع. عكس هذه الأنابيب من قبل مجموعات لضمان التفاعل بين سطح السائل وداخل أنبوب رطبة.
ملاحظة: إذا كان يعمل مع أرقام الخلية منخفضة جداً، ضبط السفينة مجموعة صغيرة حسب الاقتضاء. - حالما يتم تجميع كافة الخلايا، الطرد المركزي عينة في 260 غ س ل 8 دقيقة.
ملاحظة: قبل الطرد المركزي، بإضافة 1% إلى أسفل من سطح الجانب جيش صرب البوسنة/هبس المياه والصرف الصحي/دفع الخلايا وعكس/مزيج الأنبوب فورا بعد نظام مراقبة الأصول الميدانية. - ريسوسبيند بيليه الخلية في 1% جيش صرب البوسنة/حبس والحفاظ على الجليد. الحد الأقصى لحجم كل عينة متوافق مع تجهيز الخطوة 3 ميليلتر 33.8، حتى ضمان أن حجم تمييع/استثارة الخلية النهائية المناسبة للحصول على عدد الخلايا مثالية في 33.8 ميليلتر. خيارات الوسائط الأخرى تمييع لهذه الخطوة (وجميع تخفيف السابقة في 1% جيش صرب البوسنة/حبس) تشمل دميم، وتصل إلى 40% المصل، ولكن تجنب الكالسيوم أو المغنيسيوم يدتا المحتوية على المواد الكاشفة.
- ترك الخلايا على الجليد للحد أدنى من الوقت. ومن الناحية المثالية العامل المشترك ينبغي إعداد جميع المعدات والمواد الكاشفة للخطوة التالية (الخطوة 3) خلال الخطوات النهائية من الخطوة 2.
- خلية إعداد الشيكات الحرجة
- وتؤكد تقديرات أرقام الخلية التي تم الحصول عليها من نظام مراقبة الأصول الميدانية. تبعاً لنوع النسيج وتفكك الأطوال والحطام والخلايا يمكن أن تكون مشابهة جداً في الحجم والشكل. وهكذا، ما لم يتم استخدام مراسل فلورسنت، نظام مراقبة الأصول الميدانية لا يمكن استبعاد جميع الأنقاض. من المستحسن أن عدد خلايا نهائية بعد أن يتم تنفيذ نظام مراقبة الأصول الميدانية جمع لفهم ما هي النسبة المئوية للأحداث (وفقا لنظام مراقبة الأصول الميدانية) في الواقع خلايا لإعداد معين (الشكل 2 ز). القيام بعدد الخلايا باستخدام هيموسيتوميتير أو العداد الآلي الخلية (كرر مرتين) وحساب النسبة المئوية لخلايا قابلة للحياة التي يمثلها مجموع الأحداث التي تم جمعها وفقا لنظام مراقبة الأصول الميدانية آلة.
- التحقق من صحة إعداد الخلية. التحقق من أن لا الجزيئات الكبيرة (> 100 ميكرومتر) موجودة كما أنها قد تسد معدات في الخطوات النهائية. قد تواجه خطر عدم كفاية إزالة الحطام انسداد رقاقة موائع جزيئية خلية واحدة. لوحة الخلايا المتبقية مع نشاط الأزرق (على النحو الوارد أعلاه) التأكد من لا شظايا الحطام الكبيرة الحالية. هذا سوف يسمح أيضا لتأكيد أن الخلايا صيغة المفرد (أيعدم الالتصاق معا) إعطاء الثقة هذا التحليل الجيني المصب خلية مفردة تمثل الخلايا المفردة بدلاً من خلايا متعددة.
- اتخاذ قرار بشأن أرقام الخلية إلى تسلسل: هناك مجموعة كبيرة من أرقام الخلية المستمدة من أنسجة البالغين كل عينة يمكن تحميله في النظام مع عينات يصل إلى 8 التي يمكن تشغيلها في وقت واحد. قد تم تحميله في أي مكان من 500 – 50,000 الخلايا كل عينة المؤلفين والحصول على مجموعات بيانات Seq سرنا ذات نوعية جيدة. يمكن الاطلاع على مزيد من المناقشة بشأن أرقام الخلية الأكثر ملاءمة لتحميل في قسم النقاش. الناتج النهائي لأرقام متسلسلة الخلية يعتمد اعتماداً كبيرا على نوعية خلايا مفردة معزولة. تحميل خلايا البالغين 10,000 المستمدة من الأنسجة يمكن العودة في أي مكان من 000 1 إلى الخلايا المتتابعة 4,000 (عودة 10-40 ٪). إذا كانت مهتمة في تسلسل أرقام ارتفاع الخلية (الخلايا ~ 10,000، الحد الأقصى لعدد الموصى بها لهذا النظام)، ثم تحميل خلايا 25,000 100,000 سيكون مطلوباً.
3-جوهره جيل (هلام حبة في مستحلب) والمتوازية (1 يوم)
ملاحظة: الخطوات من 3 إلى 6 من هذا البروتوكول صممت لاستخدامها بالاقتران مع منصة خلية واحدة على أساس ميكرودروبليت الأكثر شيوعاً، المصنعة من قبل 10 X Genomics. مبادئ توجيهية مفصلة للخطوتين 3 و 4 وترد في الشركة المصنعة البروتوكول (انظر البروتوكول 3 ' خلية مفردة الكروم)11،12 ويجب أن يتبع بالتزامن مع هذا البروتوكول. للحصول على أفضل النتائج، يجب إكمال الخطوة 3 مباشرة بعد الانفصال (الخطوة 1) وخطوات العزل (الخطوة 2) خلية في يوم 1 من هذا البروتوكول.
- إعداد شرائح وفقا للبروتوكول الخاص بالشركة المصنعة 11,12. يستخدم هذا المنبر خلية واحدة على أساس ميكرودروبليت التكنولوجيا تلك الرموز الشريطية عينات ~ 750,000 مؤشر الترنسكربيتوم كل خلية على حدة. ويتحقق ذلك بتقسيم الخلايا إلى "حبة جل" في مستحلبات (GEMs) حيث كدنا ولدت حصة باركود مشترك. خلال الجيل جوهره، يتم تسليم الخلايا حيث أن الأغلبية (90-99%) من الأحجار الكريمة التي تم إنشاؤها تحتوي على أي من الخلايا، بينما تبقى، معظم الأحيان، تحتوي على خلية واحدة.
- ضع الرقاقة في حامل الشريحة.
- إعداد مزيج الرئيسية الخلية على الجليد.
- إضافة الجلسرين 50% إلى الآبار غير المستعملة وإضافة ميليلتر 90 من هذا المزيج الرئيسية الخلية إلى 1 جيدا، ميليلتر 90 من حبات جل إلى ميليلتر 2 جيدا، و 270 من تقسيم النفط بشكل جيد 3.
- تغطي الرقاقة مع طوقا.
- تحميل الرقاقة وتشغيلها في وحدة تحكم خلية واحدة.
- إخراج العلبة ووضع الرقاقة في علبة، وسحب الدرج واضغط على تشغيل. خلية مفردة 3 ' حبة جل في جوهره يشمل الإشعال تحتوي على تسلسل R1 إيلومينا جزئي (اقرأ 1 تسلسل التمهيدي)، النيوكليوتيدات 16 (nt) 10 x Barcode، 10 nt المعرف الفريد الجزيئية (أومي)، وتسلسل التمهيدي بولي-dT. أثناء التشغيل، إطلاق سراح جل الخرز في وحدة تحكم ومختلطة مع مزيج ليستي والرئيسية الخلية.
- جمع 100 ميليلتر بعينه ومكان في أنبوب PCR.
- آلة أنابيب PCR مكان في PCR المحددة مسبقاً وتشغيل الاسترداد وفقا لعدة. بعد الحضانة، سوف تشمل الأحجار الكريمة كاملة الطول، كدنا المراقب من مرناً بولي-أدينيلاتيد.
- بعد التشغيل، ضع في-20 درجة مئوية بين عشية وضحاها ليصل إلى 1 أسبوع قبل السابقة إلى الخطوة التالية.
4-تنظيف، التضخيم، وبناء مكتبة ومكتبة القياس الكمي (اليوم الثاني فصاعدا)
ملاحظة: مبادئ توجيهية مفصلة للخطوات 4 ترد في البروتوكول الخاص بالشركة المصنعة 11,12، ويجب أن يتبع بالتزامن مع هذا البروتوكول.
- استخدم الخرز المغناطيسي سيلاني لإزالة الكواشف البيوكيميائية بقايا/كبسولة تفجير من جوهره رد فعل الخليط.
- تضخيم كاملة الطول، كدنا المراقب لتوليد كتلة كافية لبناء مكتبة.
- تقييم العائد الحمض النووي. قبل بناء مكتبة، تقييم العائد الحمض النووي للعينة. هذا وسوف يحدد كم عدد دورات لاستخدام في المصب PCR الخطوة (عينة فهرس PCR أثناء تشييد المكتبة). اعتماداً على محتوى الحمض النووي الريبي لنموذج معين، التي يمكن أن تختلف باختلاف الدول التنشيط (مثلاً، مقابل التحكم بجروح، إلخ.)، نوع الخلية، والخلية الغلة، قد يتغير عدد دورة الموصى بها.
- لتسلسل ~ 3,000 المستمدة من أنسجة خلايا (لا علاقة لها بالدول التنشيط)، الكتاب وجدت أن دورات 14 (عينات: ~ 10-100 نانوغرام الحمض النووي) هو المعيار.
- استخدام بيواناليزير لتحليل الحمض النووي. راجع دليل المستخدم13.
- تجزئة العينة وتحديد حجم الحمض النووي. قبل بناء مكتبة، استخدام تجزئة الانزيمية وبروتوكولات تحديد حجم للحصول على حجم amplicon كدنا المناسبة.
- إعداد نموذج لبناء مكتبة. وفي حين R1 (قراءة تسلسل التمهيدي 1) إضافة إلى الجزيئات أثناء حضانة جوهره؛ P5، P7 (عينة فهرس)، و (قراءة تسلسل التمهيدي 2) يتم إضافتها R2 أثناء تشييد مكتبة.
- تقييم العائد الحمض النووي. تتطلب معظم تسلسل مرافق تقديم مكتبات النهائية التي تتضمن معلومات الحمض النووي الغلة والجودة. وبالتالي، قم بتشغيل بيواناليزير بعد الانتهاء من البروتوكول بأكملها وقبل نقلهم إلى مرفق التسلسل.
- تخزين العينات في-80 درجة مئوية لمدة تصل إلى شهرين.
- قبل التسلسل، تحديد حجم العينات باستخدام مجموعة أدوات القياس الكمي الحمض النووي. يمكن أن يتم ذلك في مرفق التسلسل.
5-مكتبة التسلسل (3 اليوم فصاعدا)
ملاحظة: ينشئ منصة المتوازية الترنسكربيتوم خلية واحدة استخدمت في هذا البروتوكول متوافق مع إيلومينا نهاية إقران مكتبات تبدأ وتنتهي مع تسلسل P5 و P7. على الرغم من أن الحد الأدنى للعمق اللازم لحل نوع الخلية الهوية يمكن أن يكون عدد قليل من 10,000 – 50,000 القراءات/خلية15،16، يوصي بما يلي: ~ 100,000/خلية مفاضلة تغطية تكاليف أمثل للخلايا الحية في الكبار (مع الأخذ في الاعتبار بعض الخلايا أنواع أو الدول خلية تنشيط الحد الأدنى سوف تصل إلى التشبع في 30,000-50,000 القراءات/الخلية).
- النقل مكتبات كدنا في الثلج الجاف إلى منشأة تسلسل مزودة التسلسل مناسب إيلومينا.
- توفير المعلومات التالية إلى مرفق التسلسل:
- تقديم تفاصيل العينة: عينة معرفات الفهرس المطابق لكل مكتبة؛ الأنواع؛ قاعدة بيانات الجينوم للجمعية الأساسية (أي، GRCm38 للماوس)؛ اليكتروفيروجرام عرض أحجام جزء من بيواناليزير (بين 200 و 9,000 bp)؛ تركيز كدنا (نانوغرام/ميليلتر) وتركيز مكتبة الكلية (مجموع عائدات تتراوح بين 200-1400 نانوغرام)؛ حجم العينة (ميليلتر).
- تقديم طلبات التسلسل: تحديد حجم العينات باستخدام مجموعة أدوات القياس الكمي الحمض النووي؛ نوع المحول/الفهرس (تراسك الحمض النووي)؛ نوع اللوحة (twin.tec ايبندورف، "تنورة الكامل"--أوصت للحمض النووي)؛ التسلسل التكنولوجيا/مكتبة النوع (10 x وتسلسل كامل تعليمات وتوصيات دورة)17.
- تشغيل تسلسل الضحلة (اختياري): دراسات تحليل العينات البيولوجية متعددة سوف تستفيد من تجميع عينات (تجميع) لإنشاء مصفوفة باركود مورثة واحدة تحتوي على البيانات من جميع العينات. للتقليل من آثار دفعة بين العينات عند تجميع، ينبغي أن تكون موحدة عمق القراءة بين المكتبات المختلفة. ولكي نفعل هذا، ضروري تقريبي دقيق لأرقام خلية مفردة. التسلسل مسك تسمح التسلسل الضحلة، وهي طريقة عملية وفعالة من حيث التكلفة للحصول على تقديرات دقيقة خلية.
ملاحظة: تشغيل واحد استخدام المنظم SR50 مسك توفر تغطية كافية لتقدير دقة ما يقرب من 20,000 الخلايا. سوف التقريبي هذا التشغيل عدد أومي المستردة لكل الباركود فريدة من نوعها. في الشكل 3 ألف، يظهر رأس ناتج المثال (العينة 1.6) (.csv)، إدراج الرموز الشريطية وتحسب لها أومي المقابلة حسبما يقرره ثقة المعينة ما يلي:.- التشاور مع بيوينفورماتيسيان أن تصبح مألوفة مع لغة البرمجة R. الرجوع إلى دروس داتاكامب لمزيد من المعلومات18.
- تقييم البيانات الأولية التي تم الحصول عليها من جهاز التسلسل باستخدام البرنامج النصي ص المقدمة ك قالب19. البيانات الأولية تشير إلى عدد أميس المعينة لكل رمز شريطي خلية فريدة من نوعها. البرنامج النصي يقرأ ملف.csv حيث العمود الأول قائمة بالرموز الشريطية والعمود الثاني أومي المقابلة لها التهم. وسيوفر هذا البرنامج النصي أرض (الشكل 3b) فضلا عن العدد المقدر للخلايا المراقب في كل عينة. تعديل البرنامج النصي للتأكد من أن عدد التهم أومي لنموذج معين تستقر في النقطة الثالثة وواحد من أول هبوط حاد. في الشكل 3b، يندرج هذا الكوع حوالي 225 أميس تناظر 3,480 المراقب الخلايا.
- قابلة للمقارنة للتسلسل الكامل والعمق باستخدام هيسيق (حيث كانت خلايا 3,516 بنجاح متسلسلة، الشكل 3 جيم)، توقعت تقديرات التسلسل الضحلة الخلايا 3,480.
- أما استخدام خلية الإنعاش تقريبية (من الخطوة 5، 3) أو استخدام مخطط الإنعاش وجدت في البروتوكول الخاص بالشركة المصنعة20 خطة التوزيع لين لتسلسل أعمق. يجب أن تتلقى كل عينة تغطية قابلة للمقارنة، وحتى إذا كان التسلسل الضحلة يكشف أن هناك أعدادا متباينة من الخلايا في كل عينة (الذي هو الحال غالباً) ثم توزيع لين أن تحسب وفقا لذلك. يمكن تسلسل واحد خلية تدفق هيسيق (التي تضم 8 حارات) يصل إلى 2.4 بیلیون نهاية الاقتران المخصص ما يلي:. ويرد مثال تدفق إنشاء خلية في 3d الشكل.
6-تجهيز قراءة الملفات
ملاحظة: تسلسل خلية واحدة 3 ' مكتبة باستخدام هذا البروتوكول إنشاء البيانات الخام في شكل استدعاء قاعدة ثنائي (BCL). "خلية الحارس" حزمة يستخدم لإنشاء ملفات فستق المستندة إلى النصوص من ملفات BCL، أداء الجينوم والتحالفات ترانسكريبتوميك، التهم الجينات، demultiplexing، وتجميع عينات. ويرد في هذا القسم، الخطوات الرئيسية التي تمكن المستخدمين من تحميل البيانات الخام BCL من منشأة لتسلسل وتوليد الباركود الجينات التي تم تصفيتها المصفوفات جاهزة للمعلوماتية الحيوية المتلقين للمعلومات.
- استخدام خادم مركزي لتشغيل البرنامج. BCL الملفات وملفات فستق ومعظم المعلوماتية المصب تجهيز يتطلب طاقة معالجة كبيرة.
- تحميل جميع ملفات raw القراءة على الملقم (أو فستق الملفات إذا كانت متوفرة).
- استشر مسؤول الخادم إعداد حساب على خادم مركزي أو الكتلة، والحصول على دراية يونكس21.
- استخدام أمر إحضار مناسبة لنظام التشغيل الخاص بالخادم لتحميل كافة الملفات من الخادم التسلسل للمرفق.
- وتوفر معظم تسلسل المرافق أمر لتحميل الملفات من مسار آمنة التي يمكن تشغيلها من سطر الأوامر (انظر المثال أدناه).
- استبدال "< اسم المستخدم >" و "كلمة المرور < >" العناصر النائبة في سطر الأوامر باستخدام بيانات الاعتماد المتوفرة.
wget-O-"https://your_sequencing_facilitys_server.com/path_to_raw_read_files/-لا-الكوكيز-لا-الاختيار-شهادة-البيانات بعد انتهاء' j_username = اسم المستخدم & j_password = كلمة المرور ' | wget-لا-الكوكيز-لا-الاختيار-شهادة-البيانات بعد انتهاء 'j_username = اسم المستخدم & j_password = كلمة المرور'-ci-
- إلا إذا كان مسار مطلق إلى الملفات يتم توفيرها (أي https://your_sequencing_facilitys_server.com/path_to_raw_read_files/)، أدخل هذا المسار في أمر إحضار.
- فك ضغط الملفات: إذا تم تحميل نهاية ملفات بملحق ".gz"، فإنه قد تم ضغطه باستخدام الأمر "gzip". بفك، فك تشغيل الأوامر في سطر الأوامر (انظر المثال أدناه).
gunzip raw_read_files.gz - تحميل أحدث إصدار من "الحارس خلية" على الملقم ك.tar مكتفية ذاتيا22.
-
الحاسمة: قبل تنزيل، وضمان نظام لينكس يلبي الحد الأدنى من متطلبات23. ضمان حد أدنى معالج إنتل كور 8 مع 64 جيجابايت من ذاكرة الوصول العشوائي و 1 تيرابايت من مساحة القرص الحرة.
ملاحظة: الحارس الخلية يوفر مرجع الإنسان والقوارض بنيت قبل ترانسكريبتوميس. وهذه يمكن تعديلها باستخدام الأمر مكريف سيلرانجير للكشف عن الجينات مثل بروتينات فلورية خضراء24.
-
الحاسمة: قبل تنزيل، وضمان نظام لينكس يلبي الحد الأدنى من متطلبات23. ضمان حد أدنى معالج إنتل كور 8 مع 64 جيجابايت من ذاكرة الوصول العشوائي و 1 تيرابايت من مساحة القرص الحرة.
- إنشاء ملفات فاستق من استدعاء قاعدة الملفات للتسلسل (BCLs) باستخدام الأمر مكفاستق سيلرانجير.
ملاحظة: سيقوم البرنامج محاذاة الخام على ما يلي (من ملفات فستق) لجينوم مرجع وتوليد مصفوفات الخلايا الجينات لتحليل المتلقين للمعلومات. ويستخدم راصفة النجم الذي يقوم بمحاذاة الربط-علم القراءات لجينوم إشارة. يقرأ ثقة المعينة فقط (أي، يقرأ متوافقة مع تعليق توضيحي واحد جينات) تستخدم للعد أومي.- على سبيل المثال، استخدم الأمر مكفاستق سيلرانجير:
مكفاستق سيلرانجير -معرف = sample_name \
-تشغيل =/المسار/إلى/عينة \
-csv=csv_file_containing_lane_sample_index.csv
- على سبيل المثال، استخدم الأمر مكفاستق سيلرانجير:
- تشغيل عدد سيلرانجير على فستق الملفات التي تم إنشاؤها باستخدام مكفاستق لتوليد الجينات خلية واحدة من التهم الموجهة إليه.
- على سبيل المثال، استخدام الأمر count سيلرانجير:
الكونت سيلرانجير -معرف = sample_name \
-الترنسكربيتوم ريفداتا-سيلرانجير-mm10-1.2.0 = \
-فاستقس =/المطلقة/المسار/إلى/فستق/ملفات \
-عينة = same_sample_name_supplied_to_cellranger_mkfastq \
-لوكالكوريس = 30
- على سبيل المثال، استخدام الأمر count سيلرانجير:
- تجميع مكتبة متعددة (اختياري): جمع العينات، وتجمع سيلرانجير عدد النواتج باستخدام أجر سيلرانجير. هذه النتائج في مصفوفة باركود مورثة واحدة تحتوي على البيانات المجمعة من مكتبات متعددة. مثال سيلرانجير أجر الأمر:
أجر سيلرانجير-معرف = sample_name \
-csv = csv_with_libraryID_ & _path_to_molecule_h5.csv \
-تطبيع = تم تعيينها
ملاحظة: مكتبات يمكن تجميعها باستخدام ثلاثة تطبيع أوضاع (المعين، الخام، لا شيء). تعيين المستحسن كما أنها عنها مكتبات عمق أعلى حتى يصبح جميع المكتبات تسلسل تساوي عمق25. - للتصور فوري/تحليل البيانات، استيراد ملف الإخراج.cloupe (التي تم إنشاؤها باستخدام العد سيلرانجير أو أجر سيلرانجير) إلى 10 × "العدسة اللاصقة خلية مستعرض"26.
7-متقدمة تحليل مجموعات البيانات Seq سرنا
ملاحظة: يمكن الاطلاع على قاعدة بيانات أدوات Seq سرنا كاملة في أدوات سرنا-3،27. أدناه هو يأمر إطارا للخلية دون إشراف التجميع باستخدام سورا2 وبسيودوتيمبورال باستخدام مونوكل6. على الرغم من أن جزءا كبيرا من هذا العمل يمكن أن يتم على كمبيوتر محلي، تفترض الخطوات التالية أن تكتمل عملية حسابية استخدام ملقم مؤسسية.
- تحميل أحدث إصدار من مينيكوندا على حساب الملقم باستخدام منصة لينكس28.
- قم بتثبيت أحدث إصدار من البحث والتطوير باستخدام كندا29.
- رسم البيانات باستخدام البرنامج النصي R سورا المقدمة ك قالب30.
ملاحظة: سورا هو مجموعة أدوات تمكين تدقيق مراقبة الجودة وتجميع والتفاضلية تحليل التعبير الجيني، علامة تحديد الجينات، والحد من أبعاد والتصور البيانات Seq سرنا القائم على البحث والتطوير. يمكن الاطلاع على وصف شامل لسورا الترميز ودروس على موقع مختبر ساتيجا31. - رسم البيانات باستخدام البرنامج النصي R مونوكل المقدمة ك قالب32.
ملاحظة: مونوكل هو آخر مجموعة الأدوات على أساس البحث والتطوير يمكن تصور تغييرات التعبير عبر بسيودوتيمي ويحدد الجينات التي تستند إليها قرارات مصير الخلية. يمكن الاطلاع على وصف شامل للترميز مونوكل ودروس على الموقع مونوكل33. - R-مجموعات مثل كبة يمكن أن تستخدم لاختبار وتصحيح الآثار دفعة نتيجة لتجميع مجموعات البيانات34.
8-نكبي توقعات البيئة العالمية والتقارير SRA
ملاحظة: نظراً لسهولة الوصول إلى الملفات الخام تسلسل ضمان إمكانية تكرار نتائج وتحليل، عروض أكسيسيونيد إلى مستودعات متاحة للجمهور على شبكة الإنترنت هي الموصى بها أو المطلوبة قبل تقديم المخطوط. المركز الوطني لمعلومات التكنولوجيا الحيوية (نكبي) الجامع التعبير الجيني (GEO) وتسلسل قراءة الأرشيف (SRA) مستودعات البيانات متاحة للجمهور للتسلسل الفائق البيانات35،36.
- التسجيل ل حساب "المرسل جيو" نكبي في37.
- تقديم "كامل توقعات البيئة العالمية" الذي يتضمن ثلاثة مكونات تحويلها برمجياً إلى دليل/مجلد (بعنوان كاسم المستخدم المرسل GEO): 1) سجل بيانات التعريف (جدول واحد لتقديم المشروع)؛ ملفات البيانات الخام 2)؛ 3) معالجة ملفات البيانات.
- قم بتحميل واستكمال جدول البيانات الفوقية38. تقديم توقعات البيئة العالمية العامة التالية يمكن استخدامها ك دليل (GSE100320)39. ضع جدول البيانات في الدليل.
- مكان ملفات البيانات الخام الناتجة من البرنامج النصي عد سيلرانجير لجميع المكتبات إلى الدليل.
- مكان معالجة البيانات (ملفات تمت تصفيتها barcodes.tsv و genes.tsv و matrix.mtx) المتولدة من البرنامج النصي عد سيلرانجير لجميع المكتبات في الدليل.
- استخدام بيانات اعتماد خادم FTP جيو المرسل لنقل الدليل الذي يحتوي على كافة المكونات الثلاثة. لمستخدمي لينكس/يونكس: يمكن استخدام ncftp lftp، وبروتوكول نقل الملفات، سفتب ونكفتبوت.
- إعلام الجغرافية لجميع التحويلات38.
النتائج
مرجع حزم مفتوحة المصدر مصممة لتحليل مجموعات البيانات Seq سرنا ازداد كبيرة40 مع غالبية هذه استخدام حزم اللغات المستندة إلى ص3. هنا، يتم عرض نتائج تمثيلية باستخدام اثنين من هذه الطرود: تقييم التجميع دون إشراف من واحد الخلايا استناداً إلى التعبير الجيني، ويأمر الخلايا المفردة على طول مسار من أجل حل الخلية التغايرية وتفكيك البيولوجية العمليات.
ويبين الشكل 4 استخدام سورا لتجهيز ما قبل عمليات التحقق من النوعية وتحليل المعلوماتية المتلقين للمعلومات. أولاً، الترشيح وإزالة الخلايا المنحرف من التحليل ضروري للتحقق من الجودة. وقد تم ذلك باستخدام الكمان (الشكل 4a) ومبعثر مؤامرات (الشكل 4 باء) تصور النسبة المئوية لجينات الميتوكوندريا وعدد الجينات (نجني) وعدد من أومي (نومي) لتحديد الخلية دوبليتس والقيم المتطرفة. تمت إزالة أي خلية مع عدد واضح الخارجة من الجينات، أومي، أو النسبة المئوية من جينات الميتوكوندريا باستخدام الدالة فيلتيرسيلس لسورا. منذ سورا يستخدم المكون الرئيسي (PC) من عشرات التحليل لخلايا المجموعات وتحديد أجهزة الكمبيوتر ذات دلالة إحصائية تشمل خطوة حاسمة. واستخدمت قطع الكوع (الشكل 4 ج) لاختيار جهاز كمبيوتر، استبعدت في أجهزة الكمبيوتر التي وراء الهضبة 'الانحراف المعياري للكمبيوتر' المحور. قرار التكتل أيضا التلاعب بها مما يدل على أنه يمكن تغيير عدد الكتل، تتراوح بين 0.4 (دقة منخفضة مما يؤدي إلى مجموعات خلية أقل، الرقم 4 د) إلى 4 (دقة عالية مما أدى إلى أعلى الخلية مجموعات، 4e الشكل ). بدقة منخفضة، فمن المحتمل أن يمثل كل مجموعة نوع خلية محددة، بينما بدقة عالية وهذا قد يمثل أيضا الأنواع الفرعية أو الدول الانتقالية لمحتوى الخلية. في هذه الحالة، استخدمت إعدادات المجموعة منخفضة الدقة لمواصلة تحليل التعبير heatmaps (باستخدام الدالة دوهيتماب لسورا) لتحديد الجينات الأكثر شدة المعرب عنها في مجموعة معينة (الشكل 4f). وفي هذه الحالة، حددت الجينات الأكثر شدة أعرب تقييم التعبير التفاضلية في مجموعة معينة مقابل المجموعات الأخرى جنبا إلى جنب، مما يدل على أن كل مجموعة مثلت فريد بجينات محددة. بالإضافة إلى ذلك، يمكن تصور الجينات المرشحة الفردية في مؤامرات تسن استخدام الدالة فيتوريبلوت لسورا (الشكل 4 ز). وهذا يسمح لفك رموز عما إذا كانت هناك مجموعات تمثل الضامة. باستخدام فيتوريبلوت، وجدنا أن كل المجموعة 2 وتم الإعراب عن 4 Cd68-علامة عموم بلعم.
حزمة مونوكل تم استخدامه لمساندة مجموعات الخلية المحددة في سورا، وبناء مسارات الخلية، أو يأمر بسيودوتيمبورال، بإيجاز العمليات البيولوجية (الشكل 5). يمكن استخدام الأمر بسيودوتيمبورال لعينات حيث من المتوقع ملامح التعبير خلية واحدة لمتابعة دورة الساعة بيولوجية. الخلايا يمكن أن يؤمر على طول سلسلة متصلة بسيودوتيمبورال لحل الدول المتوسطة، نقاط التشعب اثنين خلية البديلة الأقدار، والتعرف على التواقيع الجينات الكامنة وراء اقتناء كل مصير. أولاً، على غرار الترشيح لسورا، وأزيلت الخلايا ذات النوعية الرديئة أن كان توزيع مرناً عبر جميع خلايا السجل العادي وسقطت بين حدود العلوية والسفلية كما تم تحديدها في الشكل 5a. ثم، باستخدام الدالة نيوسيلتيبهيراركهي في مونوكل، صنفت الخلايا المفردة وتحسب باستخدام الجينات علامة النسب المعروفة (الشكل 5b، ج 5). على سبيل المثال، عين نوع الخلية #1 الخلايا معربا عن PDGF مستقبلات ألفا أو تنتجها الخلايا الليفية المحددة البروتين 1 لإنشاء معيار لتحديد الخلايا الليفية. بعد ذلك، تم تقييم هذه الفئة من السكان (خلية نوع #1) فك المسارات تنتجها الخلايا الليفية. للقيام بذلك، استخدم الدالة جينيتيست التفاضلية في مونوكل، الذي مقارنة الخلايا تمثل الدول المتطرفة داخل السكان والعثور على جينات التفاضلية لترتيب الخلايا المتبقية في السكان (الشكل 5 د). عن طريق تطبيق أساليب التعلم المتعددة (وهو نوع من الحد من أبعاد غير الخطية) عبر كافة الخلايا، كلف تنسيق على طول مسار بسيودوتيمبورال. تم تصور هذا المسار ثم بالدولة الخلية (الشكل 5e) وبسيودوتيمي (5f الشكل).

رقم 1: الرسم البياني- الخطوات من إعداد الحيوانات كاملة لتحليل واحد الخلية مجموعات بيانات تسلسل الحمض النووي الريبي لتقديم مجموعات البيانات النهائية لمستودع متاحة للجمهور. هلام الخرز في شكل مستحلب (GEMs) تشير إلى الخرز مع النوكليوتيد المراقب التي تغلف آلاف خلايا المفردة. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
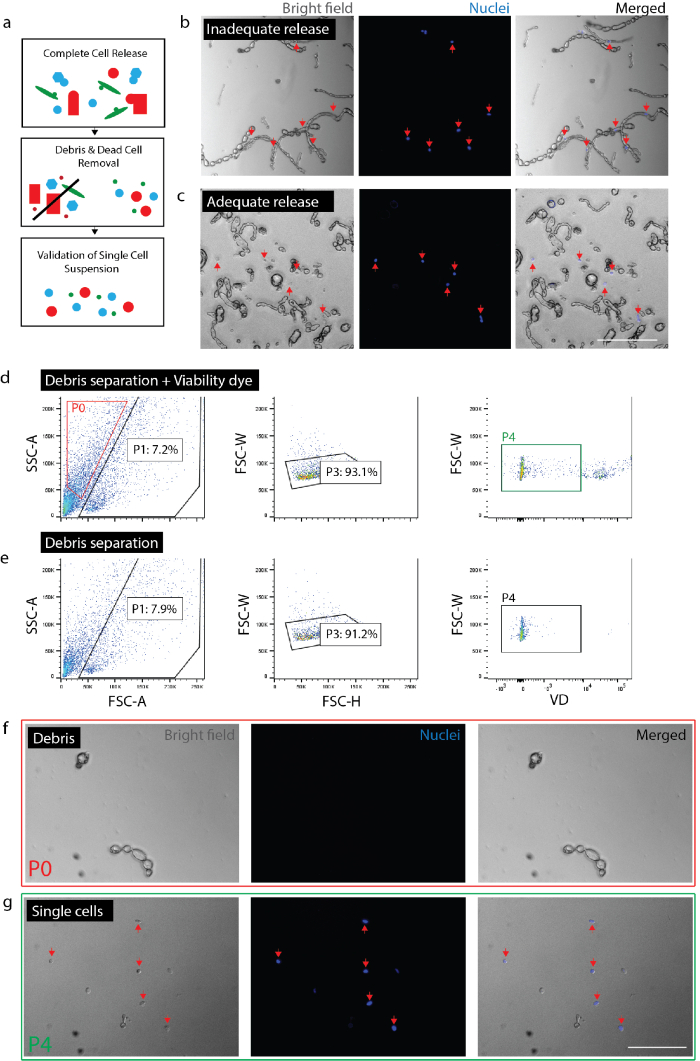
رقم 2: إنشاء تعليق خلية واحدة فقط قابلة للاستمرار من أنسجة العصب. (أ) نظرة عامة على الرسوم متحركة لمراقبة الجودة بالتحقق. لا يزال إدراج الخلايا (ب) والحطام مع الخلايا في الحطام (الأسهم الحمراء). (ج) الخلايا صدر من الحطام (الأسهم الحمراء). (د) خلية العزل بواسطة نظام مراقبة الأصول الميدانية. P0: كسر الحطام; P1: خلية مثل الكسر؛ P3: استبعاد duplets؛ P4: صلاحية صبغ (سيتوكس البرتقالي) كسر السلبية. (ﻫ) أي سيطرة صبغ البقاء. (و) الصورة لكسر P0 يمثل الحطام معزولة. (ز) صورة لكسر P4 يمثل خلايا قابلة للحياة المعزولة (الأسهم الحمراء). (ب) (ج) (و (و (ز) وقد صبغ النووية إضافة 20 دقيقة قبل التصوير. أشرطة الحجم: 80 ميكرومتر. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-

الشكل 3: تسلسل الضحلة تتوقع عدد الخلايا المستردة في 10 × العينات المجهزة. (أ) مثال (العينة 1.6) csv الذي تم إنشاؤه ميسيق إدراج الرموز الشريطية الخلية ولها أومي المقابلة التهم كما يحددها ثقة المعينة ما يلي:. (ب) ارسم الرمز الشريطي رتبة للعينة 1.6 يظهر قطره كبيرة واحدة في العد أومي كدالة للخلية الرموز الشريطية. وتمثل الخطوط المتقطعة والصلبة قطع بين الخلايا والخلفية كما يحدده الفحص البصري. (ج) خلية الرموز الشريطية لاحظ استخدام "الحارس الخلية" خط أنابيب آخر-هيسيق يكشف تسلسل الضحلة دقة تقريب عدد الخلايا للعينة 1.6. (د) مثال لهيكل الخلية تدفق استناداً إلى تسلسل الضحلة اشتقاق تقديرات الخلية. للعينة 1.6، نظراً لتوقع تسلسل الضحلة الخلايا 3480، تم تعيين ممرات 1.17 لضمان > 100,000 يقرأ كل خلية تتابع التغطية في هيسيق. ملاحظة: يجب إضافة جميع الممرات إلى 100%. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
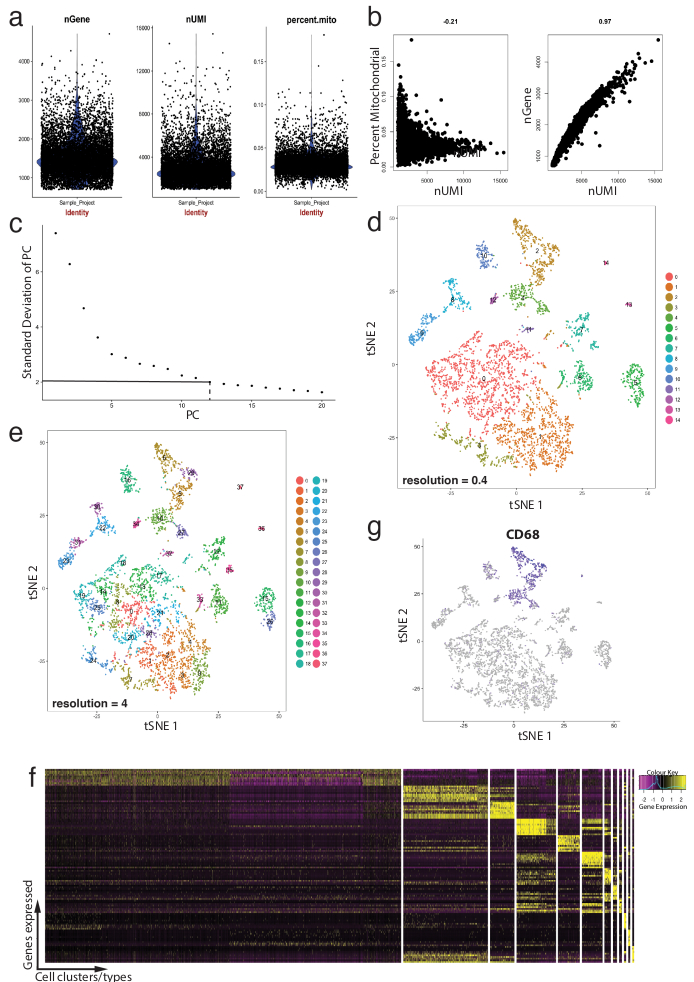
الشكل 4: مراقبة الجودة والمعلوماتية الحيوية من خلية واحدة تسلسل الحمض النووي الريبي dataset باستخدام حزمة "آر سورا". (أ) قطع مقاييس الجودة التي تشمل عدد الجينات وعدد معرفات فريدة الجزيئية (أميس)، والنسبة المئوية للنصوص رسم خرائط الجينوم المتقدرية. (ب) الجينات عينة يرسم الكشف عن الخلايا مع مستويات المنحرف المستنسخات mitochondrial واميس. (ج) عينة الكوع الأرض المستخدمة لتحديد المخصص لأجهزة الكمبيوتر ذات دلالة إحصائية. وتمثل الخطوط المتقطعة ومتقطع دوت قطع حيث يصبح واضحا "كوع" الظاهر في الرسم البياني. يتم تضمين الأبعاد PC قبل هذا الكوع في تحليل المتلقين للمعلومات. (د، ه) الخلية على أساس الرسم البياني الكتل تصور في قرارين المختلفة في مساحة منخفضة ثلاثي الأبعاد باستخدام قطعة تسنى. (و (أعلى علامة الجينات (أصفر) لكل مجموعة تصور في heatmap تعبير استخدام الدالة دوهيتماب لسورا. (ز) وضع تصور لها علامة التعبير عن، على سبيل المثال، Cd68 الجينات تمثل الضامة (الأرجواني) باستخدام الدالة فيتوريبلوت لسورا. وهذا يوحي بأن الكتلة 2 و 4 (في لوحة d) من dataset هذه تمثل الضامة. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-

الرقم 5: الخلية تصنيف وترتيب على طول مسار بيودوتيمبورال باستخدام أدوات مونوكل. (أ) تفتيش توزيع مرناً (الاستدلال من التهم أومي) عبر كافة الخلايا في عينة. فقط الخلايا مع مرناً بين 0-~ 20,000 كانت تستخدم لتحليل المتلقين للمعلومات. (ب، ج) تعيين والعد أنواع الخلايا استناداً إلى علامات خلية النسب المعروفة. على سبيل المثال، خصصت الخلايا معربا عن PDGF مستقبلات ألفا أو تنتجها الخلايا الليفية المحددة البروتين 1 خلية نوع # 1 تمثل عموم الليفية باستخدام الدالة نيوسيلتيبهيراركهي في مونوكل. يمكن تصور عدد من أنواع مختلفة من الخلايا كمخطط دائري (ب) وجدول (ج). (د) استخدام الخلية نوع #1 (الليفية) على سبيل مثال، الجينات المستخدمة لترتيب الخلايا يمكن تصور استخدام مؤامرة مبعثر الذي يوضح التشتت الجينات مقابل التعبير يعني. ويبين المنحنى الأحمر قطع للجينات المستخدمة لترتيب محسوب بالطراز يعني الفرق باستخدام الدالة استيماتيديسبيرسيونس في مونوكل. واستخدمت الجينات التي تفي بهذا قطع لترتيب بسيودوتيمي المتلقين للمعلومات. (ه، و) التصور لمسارات الخلية في الفضاء ثنائي الأبعاد تقليص الملونة "الدولة للخلية" (ه)، وتعيين مونوكل "بسيودوتيمي" (و). الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
Discussion
هذا البروتوكول يوضح كيف يمكن الكشف عن عدم تجانس النسخي الآلاف من الخلايا المفردة المناسبة إعداد الخلايا المفردة وتميز الدول الوظيفية أو هويات الخلوية فريدة داخل أنسجة. البروتوكول لا يتطلب البروتينات الفلورية مراسل أو الأدوات المعدلة وراثيا ويمكن تطبيقها على عزل الخلايا المفردة من أنسجة مختلفة من الاهتمام بما فيها من البشر؛ مع الأخذ في الاعتبار كل الأنسجة فريدة من نوعها وهذا البروتوكول سوف تتطلب قدرا من التكيف/التعديل.
وأكدت البرامج النسخي متنوعة وديناميكية للغاية داخل الخلايا قيمة الجينوميات خلية واحدة. وبصرف النظر عن عزل الحمض النووي الريبي عالية الجودة، خطوة إعداد عينة حاسمة اللازمة لمجموعات بيانات عالية الجودة هو ضمان أن الخلايا تصدر تماما من الأنسجة وأن خلايا صحية وسليمة. هذا نسبيا على التوالي إلى الأمام لجمع الخلايا بسهولة إصدارها، مثل تعميم خلايا أو أنسجة خلايا فضفاضة استبقي فيها، مثلما حدث في الأنسجة اللمفاوية. ولكن هذا يمكن أن يكون تحديا للأنسجة الأخرى الكبار، بسبب البنية الخلوية متطورة جداً التي تمتد على مسافات كبيرة، المحيطة بالمصفوفة خارج الخلية وبروتينات cytoskeletal جامدة في كثير من الأحيان المشاركة في الحفاظ على هيكل الخلية. حتى مع تقنيات الانفصال المناسبة للإصدار الكامل من الخلايا، هناك إمكانية أن معالجة دقيقة وطويلة وكثيراً ما تتطلب أن يغير سلامة الجودة وخلية مرناً. وبالإضافة إلى ذلك، تؤثر درجات الحرارة العالية المستخدمة للانفصال بمساعدة إنزيم أيضا التوقيعات النسخي29،30. أن قصد البروتوكول تقديم مراقبة الجودة يتحقق، استخدام أنسجة مثل العصب الكبار ميليناتيد والجلد الكبار الغنية بالمصفوفة خارج الخلية، لإظهار كيف يمكن أن تساعد الأمثل للتغلب على هذه العقبات.
أحد الاعتبارات رئيسية عند تصميم أي تجربة Seq سرنا هو اختيار عمق التسلسل. يمكن جداً متعدد التسلسل وقراءة العمق يمكن أن تختلف من كونها منخفضة جداً باستخدام Seq قطره2 ليصل إلى 5 مليون خلية يقرأ/14 استخدام أسلوب تسلسل الحمض النووي الريبي كاملة طول مثل ما يليها الذكية يمكن اكتشاف معظم Seq سكرنا تجارب النصوص التعبير معتدلة إلى عالية مع تسلسل منخفضة قدر ما يلي 10,000/الخلية، وعادة ما تكون كافية للخلية نوع التصنيف41،42. عمق ضحل التسلسل القيمة لإنقاذ على تكاليف التسلسل عند محاولة الكشف عن خلية نادرة السكان عبر الأنسجة المعقدة حيث آلاف خلايا قد تكون مطلوبة لثقة تنسب نادرة السكان. ولكن تسلسل عمق ضحل لا يكفي عند معلومات مفصلة عن التعبير الجيني والعمليات المرتبطة بالتوقيعات النسخي خفية ضروري. ويقدر حاليا، أن يتم الكشف عن الغالبية العظمى من المورثات في خلية ما يلي 500,000/في الخلية، ولكن هذا يمكن أن تختلف تبعاً للبروتوكول والأنسجة اكتب43،44. تكاليف التسلسل بينما تعاقب نسخة كاملة الطول تلتف الحاجة إلى الجمعية، ويمكن، لذلك، الكشف عن رواية أو المتغيرات لصق نادرة، كثيرا ما تحد من توسيع نطاق هذا النهج لدراسة آلاف خلايا التي تتألف من نظام معقد من أنسجة. وفي المقابل، 3 ' معلم مكتبات خلية واحدة مثل تلك المبينة في هذا البروتوكول عادة أقل من التعقيد وتتطلب تسلسل ضحالة. من المهم أن نلاحظ أن المكتبات التي تم إنشاؤها باستخدام بروتوكول وصف يمكن أن تكون متسلسلة على واحد من خمسة التعاقب معتمدة: 1) نوفاسيق, 2) هيسيق 3000/4000، 2500 3) هيسيق التشغيل السريع وإخراج عالي، 4) نيكستسيق 500/550 و 5) مسك.
نهج بديل لخلية مفردة الجيش الملكي النيبالي-Seq، أن يقلل من الحاجة إلى الأنسجة الحساسة وخلية معالجة بعد يحافظ على بعض فوائد خلية مفردة تسلسل الحمض النووي الريبي، وإجراء تحليل للحمض النووي الريبي من نواة واحدة45. هذا النهج يسمح أكثر سرعة معالجة الحد من تدهور الجيش الملكي النيبالي، والمزيد من الإجراءات المتطرفة لضمان الإفراج كافية من نوى، والمرجح أن يسمح بالتالي لالتقاط أكثر ثقة من الملامح النسخي يمثلون كافة الخلايا داخل نسيج معين. وبطبيعة الحال، وهذا سيوفر فقط جزء من النشاط الترانسكربتي موجودة داخل خلية معينة، وبالتالي اعتماداً على ما أهداف تجريبية لفائدة هذا النهج قد أو قد لا يكون مناسباً.
بالإضافة إلى توصيف كامل للهويات الخلوية داخل نسيج معين، واحدة من التحليلات الأكثر قيمة لمجموعات البيانات سكرنا Seq هو أنصبة الدول النسخي المتوسطة عبر السكان خلية 'المعرفة'. يمكن نقل هذه الدول الوسيطة ثاقبة علاقات النسب بين الخلايا داخل السكان التي تم تحديدها، والتي لم يكن من الممكن مع الجملة التقليدية نهج تسلسل الحمض النووي الريبي. الآن وضعت عدة أدوات bioinformatic Seq سرنا لتوضيح هذا. يمكن تقييم هذه الأدوات في عمليات المشاركة في، على سبيل المثال، الخلايا السرطانية الانتقال إلى حالة النمطان/المنتشر، الخلايا الجذعية الناضجة في مصائر المحطة الطرفية المتنوعة أو الخلايا المناعية يتنقلون بين الدول النشطة وهادئة. قد تكون الخلافات الترنسكربيتوم خفية في الخلايا أيضا يدل على التحيزات القائمة على النسب، أدوات bioinformatic وضعت مؤخرا مثل فاتيد، يمكن الاستدلال47. نظراً للفروق بين الانتقال من الخلايا يمكن أن يكون صعباً للتأكد من نظراً للاختلافات النسخي قد تكون خفية، تسلسل أعمق قد يكون ضروريا46. لحسن الحظ، يمكن زيادة التغطية لمكتبة شالوولي التسلسل إذا كان مهتما بسبر dataset المزيد عن طريق إعادة تشغيل المكتبة في آخر تدفق الخلية.
أخذت معا، يوفر هذا البروتوكول سهلة للتكيف مع سير عمل التي تمكن المستخدمين من ترانسكريبتيونالي الشخصية مئات آلاف خلايا مفردة ضمن تجربة واحدة. الجودة النهائية من dataset Seq سرنا يعتمد على التدفق الخلوي وكدنا مكتبة الجيل، عزل الخلية الأمثل وتفسير لمصفوفات الباركود الجين الخام. وتحقيقا لهذه الغاية، يوفر هذا البروتوكول استعراضاً شاملا لجميع الخطوات الرئيسية التي يمكن تعديلها بسهولة لتمكين الدراسات أنواع الأنسجة المتنوعة.
Disclosures
لا توجد عمليات الكشف
Acknowledgements
نعترف بموظفي الدعم في "مرفق خدمات أوكدنا"، وكذلك موظفي مرفق "رعاية الحيوان" في جامعة كالغاري. ونشكر ووركينتيني مات لدعمه المعلوماتية الحيوية وينس دوروثي لدعمه التقني. وكان هذا العمل ممول بمنحه استوفوا (جمهورية مقدونيا وج. ب.)، "جائزة المحقق استوفوا جديدة" إلى ج. ب.، والصحة بحوث المعهد زمالة (ج. س. الطفولة ألبرتا).
Materials
| Name | Company | Catalog Number | Comments |
| Products | |||
| RNAse out | Biosciences | 786-70 | |
| Pentobarbital sodium | Euthanyl | 50mg/kg | |
| HBSS | Gibco | 14175-095 | |
| Dispase 5U/ml | StemCell Technologies | 7913 | 5 mg/ml |
| Collagenase-4 125 CDU/mg | Sigma-Aldrich | C5138 | 2 mg/ml |
| DNAse | Sigma-Aldrich | DN25 | 10mg/ml |
| BSA | Sigma-Aldrich | A7906 | |
| 15 ml Narrow bottom tube VWR® High-Performance Centrifuge Tubes | VWR | 89039-666 | |
| Sytox Orange Viability Dye | Molecular Probes | 11320972 | 1.3 nM/µl |
| Nuc Blue Live ReadyProbes | Invitrogen | R37605 | |
| Agilent 2100 Bioanalyzer High senitivity DNA Reagents | Agilent | 5067-4626 | |
| Kapa DNA Quantification Kit | Kapa Biosystems | KK4844 | |
| Chromium Single Cell 3' reagents | 10x Genomics | ||
| Equipment | |||
| BD FACSAria III | BD Biosciences | ||
| Agilent 2100 Bioanalyzer Platform | Agilent | ||
| Illumina® HiSeq 4000 | Illumina | ||
| Illumina® MiSeq SR50 | Illumina | ||
| 10X Controller + accessories | 10x Genomics | ||
| Software | |||
| The Cell Ranger | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/overview/welcome | |
| Loupe Cell Browser | 10x GENOMICS | support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest | |
| R | https://anaconda.org/r/r |
References
- Shalek, A. K., et al. Single-cell RNA-seq reveals dynamic paracrine control for cellular variation. Nature. 510, 363-369 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161, 1202-1214 (2015).
- Zappia, L., Phipson, B., Oshlack, A. Exploring the single-cell RNA-seq analysis landscape with the scRNA-tools database. bioRxiv:206573. , (2018).
- Dulken, B. W., Leeman, D. S., Boutet, S. C., Hebestreit, K., Brunet, A. Single cell transcriptomic analysis defines heterogeneity and transcriptional dynamics in the adult neural stem cell lineage. Cell Reports. 18 (3), 777-790 (2017).
- Llorens-Bobadilla, E., et al. Single-Cell Transcriptomics Reveals a Population of Dormant Neural Stem Cells that Become Activated upon Brain Injury. Cell Stem Cell. 17 (3), 329-340 (2015).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32, 381-386 (2014).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14, 1083-1086 (2017).
- Mayer, C., et al. Developmental diversification of cortical inhibitory interneurons. Nature. 555 (7697), 457-462 (2018).
- Stratton, J. A., et al. Purification and Characterization of Schwann Cells from Adult Human Skin and Nerve. eNeuro. 4 (3), (2017).
- Biernaskie, J. A., McKenzie, I. A., Toma, J. G., Miller, F. D. Isolation of skin-derived precursors (SKPs) and differentiation and enrichment of their Schwann cell progeny. Nature Protocols. 1 (6), 2803-2812 (2007).
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Agilent Available from: https://www.agilent.com/en-us/library/usermanuals?N=135 (2018)
- Kolodziejczyk, A. A. Single Cell RNA-Sequencing of Pluripotent States Unlocks Modular Transcriptional Variation. Cell Stem Cell. 17, 471-485 (2015).
- Jaitin, D. A., et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 343, 776-779 (2014).
- Pollen, A. A., et al. Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex. Nature Biotechnology. 32, 1053-1058 (2014).
- . Sequencing Requirements for Single Cell 3' Available from: https://support.10xgenomics.com/single-cell-gene-expression/sequencing/doc/specifications-sequencing-requirements-for-single-cell-3 (2018)
- . Introduction to R Available from: https://www.datacamp.com/courses/free-introduction-to-r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics#39; Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . User Guides Available from: https://www.10xgenomics.com/resources/user-guides/ (2018)
- . Creating a Reference Package with cellranger mkref Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/advanced/references (2018)
- . System Requirements Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/overview/system-requirements (2018)
- . Software Downloads Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest (2018)
- . Aggregating Multiple Libraries with cellranger aggr Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/using/aggregate#depth_normalization (2018)
- . Loupe Cell Browser Gene Expression Tutorial Available from: https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/tutorial (2018)
- . A table of tools for the analysis of single-cell RNA-seq data Available from: https://www.scrna-tools.org/ (2018)
- . Downloading conda Available from: https://conda.io/docs/user-guide/install/download.html (2018)
- . r / packages / r 3.5.1 Available from: https://anaconda.org/r/r (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- . Seurat - Guided Clustering Tutorial Available from: https://satijalab.org/seurat/pbmc3k_tutorial.html (2018)
- . Droplet-based, high-throughput single cell transcriptional analysis of adult mouse tissue using 10X Genomics' Chromium Single Cell 3' (v2) system: From tissue preparation to bioinformatic analysis Available from: https://figshare.com/s/97b83e649e5eefd01357 (2018)
- Github. . An R package to test for batch effects in high-dimensional single-cell RNA sequencing data. , (2018).
- Edgar, R. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30, 207-210 (2002).
- Leinonen, R., Sugawara, H., Shumway, M. The sequence read archive. Nucleic Acids Research. 39, D19-D21 (2011).
- . GenBank Submission Portal Wizards Available from: https://www.ncbi.nlm.nih.gov/account/register/?back_url=/geo/submitter/ (2018)
- . Submitting data Available from: https://submit.ncbi.nlm.nih.gov/geo/submission/ (2018)
- Shah, P. T., et al. Single-Cell Transcriptomics and Fate Mapping of Ependymal Cells Reveals an Absence of Neural Stem Cell Function. Cell. 173, 1045-1057 (2018).
- Anon, Method of the Year 2013. Nature Methods. 11, 1 (2013).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144, 3625-3632 (2017).
- Wu, Y. E., Pan, L., Zuo, Y., Li, X., Hong, W. Detecting activated cell populations using single-cell RNA-seq. Neuron. 96, 313-329 (2017).
- Zeigenhain, C., et al. Comparative Analysis of Single-Cell RNA Sequencing Methods. Molecular Cell. 65 (4), 631-643 (2017).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nature Methods. 11 (1), 41-46 (2014).
- Habib, N., et al. Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science. 353 (6302), 925-928 (2016).
- Janes, K. A. Single-cell states versus single-cell atlases - two classes of heterogeneity that differ in meaning and method. Current Opinions in Biotechnology. 39, 120-125 (2016).
- Herman, J. S., Sagar, D., Grün, FateID infers cell fate bias in multipotent progenitors from single-cell RNA-seq data. Nature Methods. 15 (5), 379-386 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved