Method Article
TChIP-Seq: Hücre türüne özgü Epigenome profil oluşturma
Bu Makalede
Özet
Biz tandem kromatin için adım adım bir protokol tarif immunoprecipitation sıralama (tChIP-Seq) hücre türüne özgü genom çapında histon değişiklik çözümleme sağlar.
Özet
Epigenetik yönetmelik gen ifadesinde merkezi rol oynar. Histon değişiklik 1960'larda keşfetti, fizyolojik ve patolojik fonksiyonları kapsamlı bir şekilde incelenmiştir. Gerçekten de, yeni nesil derin sıralama ve kromatin immunoprecipitation (ChIP) belirli histon değişiklik antikorlar ile bizim görünümü epigenetik Yönetmeliğin genom arasında devrim yarattı. Diğer taraftan, dokular genellikle farklı hücre türleri oluşur ve onların karmaşık karışımı belirli bir hücreye epigenome soruşturma için analitik sorunlar teşkil etmektedir. Biz son zamanlarda kromatin seçici arıtma tagged çekirdek histon proteinleri hücre tarafından dayanmaktadır tandem kromatin immunoprecipitation sıralama (tChIP-Seq), hücre türüne özgü kromatin devlet bir genom geniş şekilde yönelik olarak geliştirilen faiz, ChIP-Seq. tarafından takip türleri Bu iletişim kuralının amacı tChIP-devamı en iyi uygulamalar giriştir Bu teknik için çeşitli histon modifikasyonları ve model organizmalar doku özel epigenome soruşturma çok yönlü bir araç sağlar.
Giriş
Hayvan dokuları farklı hücre türleri oluşur. Gen düzenlemesi her hücrede hücre türü tanımlar. Kromatin değişiklikler - DNA metilasyonu ve histon değişikliği - gen ekspresyonu hücre tipi özgüllük altında yatan. Bu nedenle, her hücre tipi epigenetik yönetmelikle ölçülmesi istenen ancak teknik bir mücadele vardı.
Tandem kromatin immunoprecipitation sıralama (tChIP-Seq) belirli bir hücreye epigenetik araştırmak için son zamanlarda geliştirilen (resim 1)1yapıldı. TChIP içinde bir hücre türüne özgü organizatörü epitope öğesini çekirdek histon protein H2B ifade edilir. Malzeme çeşitli hücre türlerinin bir karışımını başlar, ancak bu özellik chromatins ilgi, hücrelerden yalıtım sağlar. Aşağıdaki ChIP-Seq — kromatin arınma yolu ile bir değişikliğin histon işareti ve yeni nesil derin sıralama, izole DNA — bir genom geniş şekilde hedeflenen hücre tipi epigenetik durumunu izleyebilirsiniz.
Bu tekniği kullanarak, biz son zamanlarda araştırdık histon H3 protein lizin 4 (H3K4me3), nöron özel trimethylation işaretleri. Bu çalışmada, geliştirdiğimiz bir çakma fare hangi C-ölümcül içinde bayrak öğesini H2B protein Cre-aracılı rekombinasyon (Rosa26CAG floxed-pA H2B-bayrak) ifade edildi. Cre-endoplasmic retikulum (ER) gen CamK2a Düzenleyicinin kontrolü altında sahip bir fare ile geçiş tarafından elde edilen fare çizgi üzerine tamoxifen enjeksiyon (Camk2aH2B-bayrak)1aktif nöron H2B-bayrak indüklenen. Kurulan fare çizgi beyinden başlayarak, biz tChIP-Seq anti-H3K4me3 antikor ile gerçekleştirilen. Bu yana H3K4me3 işaretleri çoğunlukla organizatörü bölgelere karşılık, özellikle nöronlar1' ifade mRNA'ların yüzlerce keşfetmek olabilir.
Burada, doku diseksiyon adımlarına Kütüphane inşaat ()Şekil 1)kapsayan bir tipik tChIP-Seq yöntemi açıklanmaktadır. Bu iletişim kuralı son tChIP-Seq performansını ve diğer hücre tipleri ve histon değişiklikler bu yöntemin uygulama gelecek için bizim en iyi uygulamaları paylaşmak için hedeftir.
Protokol
Burada açıklanan tüm yöntemleri RIKEN (H27-EP071) Emanet bölümü tarafından onaylanmış ve ilgili kurallar ve düzenlemeler yapılmıştır.
1. doku diseksiyon
- Küçük parçalar halinde faiz dokuların teşrih (yaklaşık < 3 mm2) güzel bahar makas kullanarak.
Not: Daha büyük doku parçaları dondurmak için daha uzun sürer ve daha küçük parçalar arabellek, ikisi de sonuçları etkileyebilecek daha büyük birimleri üzerinde taşıyacak. - Sıvı azot ile dolu bir temiz kapsayıcı disseke doku parçaları ekleyin ve onların 2 mL tüpler (tüp başına 1 adet) içine sıvı azot ile dolu toplamak.
Not: Tüpler hidrofilik yüzey ile bu adım için tavsiye edilir. - -80 ° c > 5 dakika kalan sıvı azot buharlaşır için aç kapaklı tüpler yerleştirin.
Not: kapağı kapatıldıktan sonra doku parçaları-80 ° C'de kullanmadan önce bir ay depolanabilir.
2. hücre fiksasyon
Not: Biz bu adımı için kullanışlı bir kriyojenik değirmeni kullanılır. Alternatif bir aparat kullanılabilir.
- Yer 2 mL-protein düşük bağlayıcı tüpler bir metal buz raf üzerinde doku örnekleri içeren sıvı azot soğutulmuş.
- Metal bir kurşun bir 1,5 mL tüp ve chill sıvı azot ile yerleştirin. Metal buz rafa yerleştirin.
- 2 mL tüp açın ve prechilled metal mermi tüpün içine yerleştirin. Kapağı kapatın, kullanışlı bir kriyojenik değirmeni tüp yuvasına 2 mL tüpler ayarla ve hemen 1 dk. için sıvı azot içinde birleştirilmiş tüp sahibinin daldırma.
- Donmuş tüp sahibi kullanışlı kriyojenik değirmeni dış kaset yerleştirin ve şiddetle çalkalanır 60 kez için 30 s.
- Boru tutucu sökmeye metal kurşunu çıkarıp yer 2 mL-tüp bir örneği soğutucu-20 ° C'de prechilled
- Örnek bir sonraki adımda sabitleştirici dondurulması önlemek 15 dakika-20 ° C'de soğutucu yerleştirin.
- Tezgah için örnek soğutucu getirmek, tüp kapağını açın ve hemen 900 µL % 1 formaldehit içine damla. Birkaç kez pipetting sonra süspansiyon yeni 2 mL tüp 900 µL % 1 formaldehit ve 10 dk 23 ° C'de ayarla bir kuluçka nazik döndürme için düzeltme içeren transfer.
Dikkat: Formaldehit zehirli ve zararlı olduğunu. - Fiksasyon reaksiyonu durdurmak için 2.5 M glisin 100 µL her tüp ve santrifüj 3000 x g 4 ° C'de 5 dakika için ekleyin Süpernatant atmak.
- Buz gibi fosfat tamponlu tuz (PBS), santrifüj, 3000 x g 4 ° C'de 5 min için 1 mL ekleyin ve süpernatant atın. Bu iki kez (Toplam 3 yıkar) tekrarlayın.
Not: Flash donmuş sıvı nitrojen tarafından ve saklı-80 ° C'de sabit örnekleri olabilir - Lizis arabellek 1 500 µL ekleyin (50 mM 4-(2-hydroxyethyl)-1-piperazineethanesulfonic asit (HEPES)-KOH (pH 7.5), 140 mM NaCl, 1 mM ethylenediaminetetraacetic asit (EDTA) (pH 8.0), % 10 w/v gliserol, % 0,5 w/v NP-40, % 0,25 w/v Triton X-100 ve 0.1 x proteaz inhibitörü kokteyl) için Pelet, damlalıklı birkaç kez ve Döndür'ün 4 ° C'de 10 dakika
Not: Lizis arabellek 1 filtre ve kullanımdan önce 4 ° C'de depolanan gerekir. - 3000 x g 4 ° c de 5 dk santrifüj kapasitesi ve süpernatant atın.
- Lizis tampon 2 1 mL ekleyin (10 mM Tris-HCl (pH 8.0), 200 mM NaCl, 1 mM EDTA (pH 8.0), 0,5 mM EGTA ve proteaz inhibitörü kokteyl x 0.1) için Pelet, girdap ve Döndür'ün 4 ° C'de 10 dakika
Not: Lizis arabellek 2 filtre ve kullanımdan önce 4 ° C'de depolanan gerekir. - 3000 x g 4 ° c de 5 dk santrifüj kapasitesi ve süpernatant atın.
- Radioimmunoprecipitation tahlil (RIPA) arabelleği 1 x proteaz inhibitörü kokteyl ile 800 µL için Pelet ekleyin ve Pelet pipetting tarafından resuspend.
- 3000 x g 4 ° c de 5 dk santrifüj kapasitesi ve süpernatant atın.
- RIPA arabelleği 1 x proteaz inhibitörü kokteyl ile 500 µL ekleyin.
- 3000 x g 4 ° c de 5 dk santrifüj kapasitesi ve süpernatant atın.
- 1 mL 1 x proteaz inhibitörü kokteyl ile RIPA tampon ekleyin. Hemen bir sonraki adıma geçin.
3. Kromatin yamultma
- Lysate bir sonicator tüp aktarmak ve tüp üzerinde bir ultrasonicator yerleştirin.
- Kromatin aşağıdaki ayarlarla kesme: sıcaklık: 4 ° C; tepe olay gücü: 175 W; görev faktörü: % 10; devir/seri çekim: 200; ve Saat: 2400 s.
- Örnek bir 1,5 mL-protein düşük bağlama tüp ve santrifüj 20.000 x g 4 ° C'de, 5 min için transfer
- Süpernatant yeni bir protein düşük bağlama tüp içine toplamak.
Not: Örnek olabilir flaş donmuş sıvı azot ve-80 ° C'de depolanan
4. kalite kontrol DNA (ters çapraz)
- Mix 20 µL, lysate ve 180 µL çip elüsyon arabelleği (10 mM Tris-HCl (pH 8.0), 300 mM NaCl, 5 mM EDTA (pH 8.0) ve % 1 w/v Sodyum Lauryl Sülfat (SDS)) bir polimeraz zincir reaksiyonu (PCR) tüp. Örnek bir termal cycler açık termal cycler kapaklı 6 h 65 ° C'de kuluçkaya. Aşırı denatürasyon önlemek için termal cycler aç ağzını tut.
Not: ChIP elüsyon arabellek filtre ve kullanımdan önce oda sıcaklığında saklanan gerekir. Bu kuluçka gece için genişletilebilir. - 10 mg/ml RNase 1 µL eklemek bir, girdap ve 30 dk 37 ° C'de kuluçkaya.
- 15 mg/ml 6.5 µL eklemek İndinavir K, girdap ve 60 dk 55 ° C'de kuluçkaya.
- Bir DNA düşük bağlama tüp için tepki aktarmak, 5 mg/mL glikojen ve girdap 4 µL ekleyin. Daha sonra fenol/kloroform/izoamil alkol (25:24:1) ve 5 min için santrifüj 200 µL 18.000 x g oda sıcaklığında de ekleyin.
- Süpernatant yeni bir DNA düşük bağlama tüp aktarın. Tris-EDTA-NaCl arabelleği 200 µL ekleyin (10 mM Tris-HCl (pH 8.0), 1 mM EDTA (pH 8.0) ve 200 mM NaCl) kalan fenol/kloroform/izoamil alkol çözümü ve santrifüj 18.000 x g oda sıcaklığında, 5 min için. Süpernatant toplamak ve ilk süpernatant ile karıştırın.
- Buz gibi soğuk etanol 900 µL ekleyin ve 1 h-20 ° C'de için kuluçkaya
Not: Bu kuluçka 4 - 6 için s uzatılabilir. - 18.000 x g 4 ° C'de de 30 dk santrifüj
- Süpernatant atmak. Pelet ve 18.000 x g 4 ° C'de de 30 dk santrifüj buz gibi % 75 etanol 1 mL ekleyin (Toplam iki yıkar) bu adımı yineleyin.
- İyice süpernatant atın ve oda sıcaklığında 1 dakika kuruması.
- Tris-EDTA (TE) arabelleği 50 µL ekleyin ve DNA bu 30 dakika oda sıcaklığında veya 4 ° C'de gecede kuluçka tarafından çözülür. Vortexing veya pipetting kaçının. Bu DNA'yı "giriş" olarak tutmak ve gerekirse Kütüphane hazırlık için kullanabilirsiniz.
- DNA toplama bir yer üreticinin yönerge göre ölçü ( tablo malzemelerigörmek) ve boyut dağılımı ile bir mikrosıvısal Elektroforez makine kontrol edin (bkz: Şekil gösterilen temsilcisi verileri 2A). kullanım 10 ng veya daha az DNA bu tahlil için.
Not: 100-500 baz çifti (bp) parçaları % 50 veya daha fazla DNA'ın yukarı yapmak gerekir.
5. benzeşme arıtma Anti-bayrak antikor tarafından
Not: nöron tChIP-Seq için 8 fareler dokulardan beyin genellikle tChIP-Seq bir örneği için 2 hazırlamak için kullanılan ng tandem bağışıklık-arıtma tarafından saf DNA'ın (bkz. Adım 7.1). Bizim ellerde 0.1 ng sağlanan DNA hala yüksek kaliteli DNA kitaplıkları için ChIP-Seq (Kadota, yayınlanmamış). Böylece, teorik olarak, tek bir fare nöron tChIP-Seq. gerçekleştirmek için yeterli olabilir Gerekli miktar faiz doku ve hücre türü için optimize edilmelidir. İmumuno-arıtma deney negatif kontrol için beyin dokusu lysate herhangi bir H2B-bayrak ifade olmadan fare üzerinden hazırlanan ve kullanılan.
- Koyun Anti-fare immünoglobulin G (IgG) bir protein düşük bağlama tüp içine Birleşik manyetik boncuklar 200 µL pipet.
- Tüp manyetik bir stand üzerine yerleştirin ve netlik süpernatant 1 dakika bekleyin. Süpernatant atmak, buz gibi PBS ve girdap 1 mL ekleyin. (Toplam iki yıkar) bu adımı yineleyin.
- 1 mg/mL Anti-bayrak antikor 20 µL ekleyin ve 4 ° C'de 5 h için döndürün
Not: Bu kuluçka gece için genişletilebilir. - Adım 5.2 takip boncuk yıkayın. Boncuk 15 mL tüp içine yerleştirin.
- Boncuk RIPA arabellek 1110 µL içinde resuspend ve 10 engelleme reaktif, 180 Denhardt'ın çözüm x 50 µL, proteaz inhibitörü kokteyl x 100 10 µL ve adım 3'te hazırlanan lysate 6.8 mL x 900 µL ekleyin. Bu karışımı 5 protein düşük bağlayıcı tüpler içine bölünmüş. 4 ° C'de 6 h için döndürme
Not: Bu kuluçka gece için genişletilebilir. - Tüp manyetik stand üzerine yerleştirin ve netlik süpernatant 1 dakika bekleyin. Süpernatant atmak, RIPA arabellek 1 mL ekleyin ve karışımı yavaşça. Bu adım 5 kez tekrarlayın (Toplam 6 yıkar). Tüm boncuklar bir tek protein düşük bağlama tüpüne havuz.
- ChIP elüsyon arabelleği 200 µL ekleyin ve 15 dakika oda sıcaklığında döndürün. DNA'ın kalite kontrol edin (bkz. Şekil 2B ve Cgösterilen temsilcisi veriler) 4.11. adımda açıklandığı gibi. Hemen bir sonraki adıma geçin.
6. benzeşme arıtma Anti-H3K4me3 antikor ve ters çapraz
Not: Adımları (6.1-6.4)-meli var olmak kılınmak önce adım 5,7 aşağıdaki boncuk hazırlık.
- Manyetik boncuklar 2 mL-protein düşük bağlama tüp içine koyun Anti-fare IgG için Birleşik 40 µL pipet.
- Tüp manyetik stand üzerine yerleştirin ve netlik süpernatant 1 dakika bekleyin. Süpernatant atmak, buz gibi PBS 1 mL ekleyin ve karışımı yavaşça. (Toplam iki yıkar) bu adımı yineleyin.
- 1 mg/mL anti-H3K4me3 antikor 4 µL ekleyin ve 4 ° C'de 6 h için döndürün
- Adım 6.2 takip boncuk yıkayın. Boncuk 2 mL-protein düşük bağlama tüp içine yerleştirin.
- Boncuk RIPA arabellek 1558 µL içinde resuspend ve reaktif, 50 Denhardt'ın çözüm x 40 µL, 2 µL proteaz inhibitörü kokteyl x 100 ve üzerinde adım 5.7 hazırlanan eluate 200 µL engelleme x 10 200 µL ekleyin. Gecede 4 ° C'de döndürmek
Not: SDS çip elüsyon arabelleğinde 10 kereden daha fazla bu kuluçka düşürülmüştü. - Tüp manyetik stand üzerine yerleştirin ve netlik süpernatant 1 dakika bekleyin. Süpernatant atmak, RIPA arabellek 1 mL ekleyin ve karışımı yavaşça. 4 kez bu işlemi tekrarlayın (Toplam 5 yıkar). Son yıkama sonra boncuklar bir DNA düşük bağlama tüp içine aktarmak ve süpernatant atın.
- ChIP elüsyon arabelleği 200 µL ekleyin ve 15 dakika oda sıcaklığında döndürün. Eluate yeni bir DNA düşük bindingTube aktarın.
Not: Eluate-80 ° C'de saklanabilir - Adım 4 DNA'ın decrosslinking için izleyin. Saf DNA TE arabellek 20 µL içinde resuspend.
- DNA'ın kalite kontrol edin (bkz. Şekil 2Bolarak gösterilen temsilcisi veriler) 4.11. adımda açıklandığı gibi. (isteğe bağlı) Kantitatif PCR ile zenginleştirme analiz gerçekleştirmek2daha önce açıklandığı gibi. Panolarında veya büyük zenginleştirme gözlenen.
7. Kütüphane İnşaat
- Her girdi ve çip DNA örneği için DNA mikrosıvısal Elektroforez makine için yazılım smear analiz işlevini kullanarak 100-500-bp bölgedeki oranı hesaplar. 2'yi edinmek için gerekli numune hacmi tahmin ng 100-500 BP DNA. 20 ng DNA 100-500-bp aralığında "giriş" örnek olarak kullanın.
- DNA örnekleri 8-şerit PCR tüpler içine pipet ve DNA hacmi 10 µL, orantılı olarak ölçek-up aşarsa, toplam hacim 10 µL. için (isteğe bağlı) getirmek için H2O aşağıdaki bitiş-onarım tepki ve boyut seçimi ekleyin.
- Son onarım ana karışım hazırlamak ( Tablo malzemelerigörmek). Son onarım ana mix 4 µL DNA'yı ekleyin ve 20 ° C'de 30 dk için kuluçkaya
- 10 mm Tris-Cl, pH 8,5 36 µL ekleyin ve tersinir immobilizasyon (SPRI) manyetik boncuklar faz ve oda sıcaklığında 5 min için kuluçkaya 0.6 x Ses (30 µL) katı.
- Tüp manyetik bir stand üzerine yerleştirin ve süpernatant temiz hale gelinceye kadar bekleyin.
- Yeni bir tüp süpernatant transfer 1.2 x (60 µL) hacimleri SPRI manyetik boncuklar eklemek ve oda sıcaklığında 5 dk kuluçkaya.
- Tüp manyetik bir stand üzerine yerleştirin ve süpernatant temiz hale gelinceye kadar bekleyin.
- Boncuk 200 µL % 80 ile iki kez yıkayın alkol, tüp mıknatıs hala üzerinde tutarak.
- Kısa bir süre kalan alkol alt toplamak ve geri üzerinde mıknatıs yerleştirmek için tüp santrifüj kapasitesi. Artık alkol iyice kaldır.
- Tüp kapak alkol buharlaşır izin vermek 1-2 min için açık bırakın.
- Kapağı kapatın ve tüp buz üzerinde tutun.
Not: Şimdi boyutu seçili DNA 100-500 BP boncukları üzerinde almış. Çift boyut seçimi hakkında daha fazla bilgi (www.beckman.com) üreticinin Web sitesinde bulunabilir. - A-takip ana hazırla ( Tablo malzemelerigörmek) karıştırın.
- Boncuk (Kimden adım 7,10) A-takip ana mix 10 µL ile resuspend ve 30 ° C'de 30 dk için kuluçkaya
- 1.8 x birim (18 µL) Polietilen glikol (PEG) eklemek / NaCl çözüm ve oda sıcaklığında 5 min için kuluçkaya.
- DNA arındırmak için 7.7-7,11 adımları izleyin.
- Tüp ligasyonu arabellek karışım hazırlamak ( Tablo malzemelerigörmek).
- Tüp ligasyonu arabellek Mix (Kimden adım 7,14) boncuk üzerine 8 µL pipet, 1 µM bağdaştırıcısının 1 µL eklemek ve boncuk resuspend. 5 µM bağdaştırıcısının 1 µL giriş örneği için kullanmak. Çoğullama için her örnek için farklı bağdaştırıcıları kullanmayı düşünün.
- DNA ligaz 1 µL ekleyin ve 20 ° C'de 15 dakika kuluçkaya
- Boncuk resuspend ve oda sıcaklığında 5 min için kuluçkaya 1.0 x birim (10 µL) PEG/NaCl çözeltisi ekleyin.
- DNA arındırmak için 7.7-7.10 adımları izleyin.
- 10 mm Tris-Cl, pH 8,5 tüp içine 25 µL pipet ve boncuk resuspend.
- 1.0 x (25 µL) hacmi PEG/NaCl çözüm ekleyin ve oda sıcaklığında 5 min için kuluçkaya.
- DNA arındırmak için 7.7-7.10 adımları izleyin.
- 10 mm Tris-Cl, pH 8,5 tüp içine 11 µL pipet ve boncuk resuspend.
- Tüp manyetik bir stand üzerine yerleştirin ve süpernatant temiz hale gelinceye kadar bekleyin.
- Süpernatant yeni 8-şerit PCR tüp içine toplamak.
- Gerçek zamanlı PCR ana karışım hazırlamak (malzeme Ayrıntılar için bakınız).
- Gerçek zamanlı PCR ana Mix 384-şey kantitatif PCR (qPCR) plaka 8,5 µL pipet ve bağdaştırıcının 1,5 µL DNA'dan (adım 7.26) tane ekleyin.
- Boş Wells floresan her standart 10 µL pipet.
- Aşağıdaki programı ile gerçek zamanlı PCR gerçekleştirmek: (98 ° C 45 için s) 1 döngüsü, x (98 ° C 15 s, 30 60 ° C s, 30 72 ° C s) x 20 döngüleri, ve (72 ° C 60 s) x 1 döngüsü.
- PCR döngüleri floresan Standart 2 floresan yoğunluğu ulaşmak için gereken sayısını belirleyin.
- PCR ana hazırla ( Tablo malzemelerigörmek) karıştırın.
- 11,5 µL PCR ana karışımı içine (Kimden adım 7.26) kalan bağdaştırıcısı bakmaksızın DNA pipet ve PCR adım 7.31 belirlenen PCR çevrimleri ile adım 7,30 içinde belirtildiği şekilde gerçekleştirin.
- 1.0 x (20 µL) hacmi PEG/NaCl çözüm ekleyin ve oda sıcaklığında 5 min için kuluçkaya.
- DNA arındırmak için 7.7-7.10 adımları izleyin.
- 10 mm Tris-Cl, pH 8,5 tüp içine 20 µL pipet ve boncuk resuspend.
- Tüp manyetik bir stand üzerine yerleştirin ve süpernatant temiz hale gelinceye kadar bekleyin.
- Süpernatant yeni 1,5 mL-DNA düşük bağlama tüp içine toplamak.
- Kütüphanede kalitesini kontrol edin (bkz: 2E ve F rakamgösterilen temsilcisi veri) 4.11 adımda açıklandığı gibi. Kütüphane DNA örneği 1 µL kullanın. (isteğe bağlı) 2daha önce açıklandığı gibi qPCR tarafından zenginleştirme çözümlemesi gerçekleştirin. Panolarında veya büyük zenginleştirme gözlenen.
8. sıralama
- Yeni nesil sekanslama kitaplıklarda sıra (bkz: Şekil 3' te gösterilen temsilcisi veri).
Not: Veri analizi için yeterli sıra derinliği organizma3genom boyutuna bağlı olarak değişir. İnsan ve fare, biz tavsiye almak en az 20-40 milyon tek taraflı kenar defa okundu. Çoğullama okuma verimini göre uygun maliyetli olabilir.
Sonuçlar
Burada, tandem arıtma kromatin ve DNA Kütüphane hazırlık yeni nesil sıralayıcılar için doku diseksiyon, fiksasyon, hücre lizis tarif. İşlemler sırasında bir birden çok adımı (resim 2), başarılı sıralama anahtarıdır DNA kalitesini test edebilirsiniz. Bir tek nucleosome genellikle 147 bp DNA 4tarafından çevrili beri yamultulmuş DNA o boyutundan daha kısa olmamalıdır. Hemen ultrasonication sonra DNA izole ve bir mikrosıvısal Elektroforez makinede (Şekil 2A) çalıştırın. Büyüklük sıra optimize etmek için çabalarımıza rağmen hala DNA'sı (yaklaşık 2 kbp) unsheared kaldı nüfusu vardı. Bu adımda, kesir 100-500 bp değişen DNA'ın % 50 veya daha fazla nüfus olmalıdır. Anti-bayrak antikor (Şekil 2B) ve daha sonra anti-H3K4me3 antikor (Şekil 2B) tarafından benzeşme arıtma sonra tekrar mikrosıvısal Elektroforez makine ve DNA arasında değişen miktarda kullanarak izole DNA kalitesini kontrol edildi 100-500 bp tahmin edilmektedir. Her ne kadar biz kez daha güçlü bir önyargı bu aşamada 2 kbp parçaları doğru gözlenen, SPRI manyetik boncuklar çift boyutu seçerek bu kesir kaldırıldı.
Bağışıklık-arıtma DNA H2B bayrağı özgüllüğü hangi beyin lysate fareler H2B-bayrak olmadan üzerinden ifade kullanıldı negatif kontrol örnekleri ile teyit edildi (Şekil 2C). Genellikle önemsiz miktarda DNA negatif kontrol örneklerinden tespit ettik.
A-takip, bağdaştırıcı ligasyonu ve PCR aşağıdaki sıralama kitaplığı kalitesini doğrulanmadı (Şekil 2E). Tipik olarak, 250-600 bp DNA elde edildi. Başarılı normal ChIP ve tChIP göre zenginleştirme analiz organizatörü bölge GAPDH gen (Şekil 2F) hedefleme primerler kullanılarak qPCR2 ile kanıtlanan.
Temsilcisi dağıtımları nöron genler (Camk2a, Slc17a7 ve Gria1) genom tarayıcı tarafından boyunca Şekil 3' te gösterilmektedir okuyun. Okuma genler tarafından nöron tChIP-Seq tüm beyin tChIP-Seq üzerinde 5'sonunda zenginleştirme gözlendi.

Şekil 1: tChIP-Seq. şematik gösterimi Bu rakam Mito, değiştirilmiş olan M. ve ark. 1. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
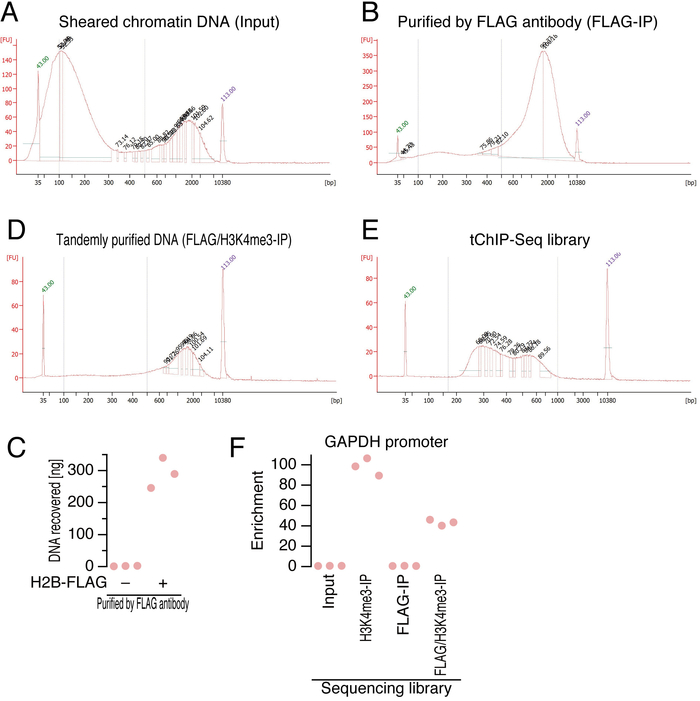
Şekil 2: DNA kalite kontrolü. (A, B, Dve E) Yamultulmuş Kromatin DNA(a)Anti-bayrak antikor (B) ve sonra anti-H3K4me3 antikor (C) ve (D) son Kütüphane ile saf DNA için bir mikrosıvısal Elektroforez makine kullanılarak elde Electropherogram. Yeşil ve mor numaraları ile vurgulanan doruklarına iç boyutu standartları temsil eder. Aşağı akım analiz için kabul DNA boyutu bölgeler mavi kesik çizgilerle gösterilir. FU: floresan birimleri.
(C) DNA miktarı Anti-bayrak bağışıklık-arıtma tarafından kurtarıldı. Her nokta bir bağımsız çoğaltma temsil eder. (F) GAPDH hedefleme qPCR göre zenginleştirme analiz. Her nokta bir bağımsız çoğaltma temsil eder. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 3: dağıtımları nöron tChIP-Seq. okumak (A-C) Okuma dağıtımları nöron tChIP-Seq ve kontrol tüm beyin tChIP-Seq temsilcisi nöron genlerinde boyunca gösterilir. RPM: okuma başına milyon defa okundu. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
Bizim iletişim kuralı H2B bayrak öğesini ifade tamoxifen enjeksiyonu ile indüklenen olduğu fare beynin nöronlar için optimize edildi. H2B ifade, başlangıç doku malzeme ve doku miktarı için kullanılan Rehberleri başarılı tChIP-devamı çok önemli parametreleridir Böylece, bu faktörlerin optimizasyonu ilgi her hücre türü için düşünülmelidir.
Bu protokol için kullanılan yordamlar arasında önemli bir adım 100-500 bp5kromatin uzunluğunu elde etmek için kesme DNA'sı. Genel olarak, çok çeşitli faktörlere bağlıdır beri ultrasonication adım standardizasyon meydan okuyor; fiksasyon koşulları, kullanılan dokular, ultra-sonication için kullanılan araç ve cihazın ayarı, diğerleri arasında. Bu nedenle, deneme-yanılma optimizasyonu bu DNA kesme adımın bireysel deneyler için gerekli olacaktır. Ultra-sonication yerine micrococcal nükleaz tedavi olarak kullanılan saf çip Yöntem6' nucleosome boyutlu tek DNA izole etmek için bir seçenek olmalıdır. Her ne kadar homojen boyutu dağıtım DNA'ın idealdir güdülmesini DNA'lar Şekil 2Agösterildiği gibi birden fazla nüfus gösterebilir. Çift boyut seçim SPRI manyetik boncuklar, yukarıda açıklandığı gibi daha uzun boy, DNA parçacıkları kaldırmak için yardımcı olur.
Tipik olarak, 1 Kütüphane hazırlık gerektirir ng 100-500 BP DNA. Bu şartlarına göre nöronlar için beyin dokusunun malzeme ilk yukarı ölçekli. Ancak, bu yordamı nadir hücre tipleri son derece küçük örnekleri için zor olabilir. Bu gibi durumlarda, hangi Tn5 transposase ligaz yerine temel alır ve daha az malzeme7gerektirir, ChIP okunmasının iyi bir alternatif olabilir.
Yine de, bu geçerli protokol malzeme büyük hücre türleriyle sınırlıdır. Bizim ellerimizde özgün hücre nüfus hedeflenen hücrelerin % 10 doluluk kromatin hedef hücreleri1adil arıtma sağlanan. Daha nadir hücre tipleri hedeflenir, ancak, daha sonra immunoprecipitation epitope etikete göre daha hassas optimizasyonu olmayan hedef hücrelerden kirlenmiş DNA izole DNA ayırmak için garanti kapsamında olacaktır. İçin en iyi uygulamalar veri analizi, veri kontrol tChIP-Seq analizi için deneme başlatmak için kullanılan tüm dokudan karşılaştırma öneririz. Exogenously ifade H2B-bayrak protein üzerinden önemli önyargıları bulduğumuzdan beri analizi karşılaştırıldığında ve zenginleştirme (veya tükenmesi) için arka plan1analiz gerekir. Bu denetim için gerçekten bir fare çizgi ubiquitously ifade H2B ile-bayrak (RosaH2B-bayrak) oluşturulur ve tChIP-Seq1yapılır. Verileri daha ayrıntılı bir yorum oldu daha önce tartışılan1.
Bizim çözümleme bağlı olarak, H3K4me3 tChIP-Seq nöronlar dan karşılaştırılabilir ve/veya daha iyi algılama RNA-Seq FACS sıralanmış nöronlar üzerinden daha bilinen nöron özel genlerin8 ve çeviri ribozom benzeşme arıtma (tuzak) sağlanan-Seq9. Örneğin, bilinen axon/dendrite-lokalize genler verimli nöron tChIP Seq1kullanarak ele geçirildi. Dikkat çekici, bizim tChIP Seq organizatörü ilişkili histon mark için sınırlı değildir ama antikor yoksa Ayrıca diğer çeşitli histon değişiklikler10 için geçerlidir. Buna ek olarak, kromatin tagged çekirdek histon proteinleri tarafından izole strateji diğer canlılar arasında kullanılabilir. Burada biz çekirdek histon protein etiketlemek için bayrak epitope kullanılmış olmasına rağmen diğer Etiketler uygulanabilir. Formaldehit fiksasyon DNA için en sık bir protein11lizin artıkları oluşur beri birçok lisin içeren bir etiketi önyargıları ortadan kaldırmak için epitope etiketleyerek kaçınılmalıdır. Böylece, tChIP-Seq yaklaşım pek çok bağlamlarda epigenetik çalışmalar boyunca çeşitli dokularda çok yönlü olmalıdır.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Iwasaki laboratuvar şekilde alt alta yazılmalıdır eleştirel okuma için tüm üyeleri teşekkür ediyoruz. Bu eser kısmen yenilikçi alanları (#26113005); S.N. ve JP17H05679 sı için bilimsel araştırma için bir Grant-in-Aid tarafından desteklenmiştir Genç bilim adamları için bir Grant-in-Aid (A) sı için (JP17H04998) Milli Eğitim Bakanlığı, bilim, spor ve kültür Japonya (MEXT); ve Pioneering "Hücresel evrim" ve tüm RIKEN proje "Hastalığı ve Epigenome" RIKEN üzerinden (için S.N. ve sı) projeleri.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Protein LoBind tube, 2 mL | Eppendorf | No. 0030108132 | For cell lysis |
| Protein LoBind tube, 1.5 mL | Eppendorf | No. 0030108116 | For ChIP and library preparation |
| DNA LoBind tube, 1.5 mL | Eppendorf | No. 0030108051 | For ChIP and library preparation |
| 8-strip PCR tube | BIO-BIK | 3247-00 | For ChIP and library preparation |
| SK Mill | TOKKEN | SK-200 | Handy cryogenic grinder to make cell powder for fixation |
| Metal bullet | TOKKEN | SK-100-DLC10 | Accessory of SK Mill |
| 2 mL stainless steel tube | TOKKEN | TK-AM5-SUS | An option for cell lysis |
| 2 mL stainless steel tube holder | TOKKEN | SK-100-TL | An option for cell lysis |
| 16% formaldehyde (w/v), methanol-free | Pierce | 28906 | To fix cells. Prepare 1% solution before use. |
| Glycine | Nacalai Tesque | 17109-35 | Prepare 2.5 M stock |
| D-PBS (-)(1x) | Nacalai Tesque | 14249-24 | For washing lysate and purified DNA |
| HEPES | Nacalai Tesque | 02443-05 | For Lysis buffer 1. Prepare 1 M, pH 7.5 stock. |
| 5 M NaCl, molecular biology grade | Nacalai Tesque | 06900-14 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| 0.5 M EDTA, molecular biology grade | Wako Pure Chemical Industries, Ltd. | 311-90075 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| Glycerol | Wako Pure Chemical Industries, Ltd. | 072-04945 | For lysis buffer 1 |
| NP-40 | Nacalai Tesque | 25223-75 | For lysis buffer 1 |
| Triton X-100, molecular biology grade | Nacalai Tesque | 12967-32 | For Lysis buffer 1 |
| Tris | Nacalai Tesque | 35406-91 | For Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer. Prepare 1 M, pH 8.0 stock. |
| 0.1 M EGTA pH neutral | Nacalai Tesque | 08947-35 | For Lysis Buffer 2 |
| Protease inhibitor cocktail (100x) | Nacalai Tesque | 25955-24 | To block degradation of protein |
| RIPA buffer | Thermo Fisher Scientific | 89900 | For cell lysis and washing |
| milliTUBE 1 mL AFA Fiber | Covaris | 520130 | Sonicator tube. Accessory of Focused-ultrasonicator |
| Focused-ultrasonicator | Covaris | S220 or E220 | To digest DNA into adequate size for ChIP-Seq |
| UltraPure 10% SDS | Thermo Fisher Scientific | 15553-027 | For ChIP Elution Buffer |
| RNase A | Nacalai Tesque | 30141-14 | To purify DNA from lysate |
| Proteinase K, recombinant, PCR Grade | Sigma-Aldrich | 3115887001 | To purify DNA from lysate |
| Ethanol | Wako Pure Chemical Industries, Ltd. | 054-07225 | Make 70% solution |
| Monoclonal anti-FLAG M2 antibody produced in mouse | Sigma-Aldrich | F1804 | To purify chromatin expressed in cells of interest |
| Dynabead M-280 Sheep Anti-Mouse IgG | Thermo Fisher Scientific | 11201D | This can be used for anti-FLAG IP and anti-H3K4me3 IP |
| Anti-tri-methyl histone H3 (K4), mouse monoclonal antibody | Wako Pure Chemical Industries, Ltd. | 301-34811 | Any other antibody that works for ChIP analysis will work |
| 10x Blocking Reagent | Sigma-Aldrich | 11096176001 | For blocking during affinity purification |
| Denhardt’s solution | Nacalai Tesque | 10727-74 | For blocking during affinity purification |
| Glycogen (5 mg/ml) | Thermo Fisher Scientific | AM9510 | To purify DNA from lysate |
| Qubit 2.0 Fluorometer | Thermo Fisher Scientific | Q32866 | For quantification of isolated DNA |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | For quantification of isolated DNA |
| 0.5 mL tube | Axygen | 10011-830 | For quantification by Qubit |
| Phenol/chloroform/isoamyl alcohol (25:24:1) | Nacalai Tesque | 25970-56 | To purify DNA from lysate |
| AMPure XP beads | Beckman Coulter | A63881 | SPRI magnetic beads for library preparation |
| Metal ice rack | Funakoshi | IR-1 | To keep the cell lysate frozen |
| Sample Cooler | New England Biolabs | T7771S | Helps fix cells with minimal damage |
| 2100 Bioanalyzer | Agilent Technologies | G2939BA | To check the quality of isolated DNA fragments. Another fragment analyzer can be used. |
| Bioanalyzer 2100 Expert Software | Agilent Technologies | G2946CA | Supplied with the Bioanalyzer |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | To check the quality of the isolated DNA fragments |
| KAPA LTP Library Preparation Kit | Roche | 07961898001 | Supplied with 10x KAPA End Repair Buffer, KAPA End Repair Enzyme Mix, KAPA A-Tailing Buffer, KAPA A-Tailing Enzyme, KAPA Ligation Buffer, KAPA DNA Ligase, and PEG/NaCl solution |
| NEXTflex DNA Barcodes | BIOO Scientific | NOVA-514101 | Adapter for library preparation. Supplied with DNA Barcode Adapters and Primer Mix. |
| KAPA Real-Time Library Amplification Kit | Roche | 07959028001 | Supplied with 2x KAPA HiFi HS real-time PCR Master Mix, PCR Primer Mix, and Fluorescent Standards |
| 2x KAPA HiFi HotStart ReadyMix | Roche | KM2602 | For library preparation. Additionally, this enzyme may be required for the KAPA Real-Time Library Amplification Kit |
| Buffer EB | Qiagen | 19086 | 10 mM Tris-Cl, pH 8.5 for elution of DNA |
| 386-well qPCR plate | Thermo Fisher Scientific | 4309849 | For real-time PCR |
| QuantStudio 7 Flex Real-Time PCR System | Thermo Fisher Scientific | 4485701 | To quantify DNA |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 4311971 | For real-time PCR |
| MicroAmp Clear Adhesive Film | Thermo Fisher Scientific | 4306311 | For plate sealing |
| End-repair master mix | Combine 1.4 µL of 10x KAPA End Repair Buffer, 1 µL of KAPA End Repair Enzyme Mix, and 1.6 µL of H2O | ||
| A-taling master mix | Combine 1 µL of KAPA A-Tailing Buffer, 0.6 µL of KAPA A-Tailing Enzyme, and 8.4 µL of H2O | ||
| Ligation buffer mix | Combine 2 µL of KAPA ligation buffer and 6 µL of H2O | ||
| Real-time PCR master mix | Combine 5 µL of 2x KAPA HiFi HS real-time PCR Master Mix, 0.35 µL of PCR Primer Mix (10 µM each of forward primer AATGATACGGCGACCACCGAG and reverse primer CAAGCAGAAGACGGCATACGAG), and 3.15 µL of H2O | ||
| PCR master mix | Combine 10 µL of 2x KAPA HiFi Ready Mix, 0.9 µL of PCR Primer Mix, and 0.6 µL of H2O | ||
| Integrative Genomics Viewer | Broad Institute | IGV_2.3.88 | Genome browser to visualize sequencing data |
| DNA olgionucleotide: 5′-GCCTACGCAGGTCTTGCTGAC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| DNA olgionucleotide: 5′-CGAGCGCTGACCTTGAGGTC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| SYBR Premix Ex Taq | Takara | RR420L | To quantify the DNA corresponding to the GADPH promoter region |
| Thermal Cycler Dice | Takara | TP870 | To quantify the DNA corresponding to the GADPH promoter region |
Referanslar
- Mito, M., et al. Cell type-specific survey of epigenetic modifications by tandem chromatin immunoprecipitation sequencing. Scientific Reports. 8 (1), 1143 (2018).
- Kadota, M., et al. CTCF binding landscape in jawless fish with reference to Hox cluster evolution. Scientific Reports. 7 (1), 4957 (2017).
- Jung, Y. L., et al. Impact of sequencing depth in ChIP-seq experiments. Nucleic Acids Research. 42 (9), (2014).
- Becker, P. B., Workman, J. L. Nucleosome remodeling and epigenetics. Cold Spring Harbor Perspectives in Biology. 5 (9), a017905 (2013).
- Kidder, B. L., Zhao, K. Efficient library preparation for next-generation sequencing analysis of genome-wide epigenetic and transcriptional landscapes in embryonic stem cells. Methods in Molecular Biology. , 3-20 (2014).
- Gilfillan, G. D., et al. Limitations and possibilities of low cell number ChIP-seq. BMC Genomics. 13, 645 (2012).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Zhang, Y., et al. An RNA-sequencing transcriptome and splicing database of glia, neurons, and vascular cells of the cerebral cortex. Journal of Neuroscience. 34 (36), 11929-11947 (2014).
- Hornstein, N., et al. Ligation-free ribosome profiling of cell type-specific translation in the brain. Genome Biology. 17 (1), 149 (2016).
- Zhao, Y., Garcia, B. A. Comprehensive catalog of currently documented histone modifications. Cold Spring Harbor Perspectives in Biology. 7 (9), a025064 (2015).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: a tool for the study of chromatin complexes. The Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır