Method Article
TChIP-Seq: יצירת פרופילים ייחודיים לסוג התא Epigenome
In This Article
Summary
אנו מתארים פרוטוקול צעד אחר צעד עבור טנדם כרומטין רצף immunoprecipitation (tChIP-Seq) המאפשרת הניתוח של שינוי היסטון הגנום כולו ייחודיים לסוג התא.
Abstract
תקנה epigenetic ממלא תפקיד מרכזי ביטוי גנים. מאז שינוי היסטון התגלה בשנות השישים, תפקידיה פיזיולוגיים ופתולוגיים נחקרו בהרחבה. אכן, כניסתו של רצף עמוק הדור הבא, immunoprecipitation כרומטין (ChIP) באמצעות נוגדנים שינוי היסטון ספציפי יש מהפכה התפיסה שלנו לגבי רגולציה epigenetic ברחבי הגנום. לעומת זאת, רקמות בדרך כלל מורכבים של סוגי תאים שונים, תערובת מורכבת שלהם מציב אתגרים אנליטית כדי לחקור את epigenome בסוג לתא מסוים. כדי לטפל המדינה כרומטין ייחודיים לסוג התא באופן הגנום כולו, שפיתחנו לאחרונה טנדם כרומטין immunoprecipitation רצף (tChIP-Seq), אשר מבוססת על הטיהור סלקטיבי של כרומטין על-ידי הליבה מתויג חלבוני היסטון מתא סוגי ריבית, ואחריו שבב-תת סעיף. המטרה של פרוטוקול זה הוא ההקדמה של שיטות עבודה מומלצות של tChIP-תת סעיף. טכניקה זו מספקת כלי רב-תכליתי לחקירה רקמות ספציפיות epigenome שינויים היסטון מגוונים, מודל אורגניזמים.
Introduction
ברקמות של בעלי חיים מורכבים של סוגי תאים שונים. הכונה בכל תא מגדיר את סוג התא. כרומטין שינויים - DNA מתילציה של היסטון-שינוי - ביסוד יחודיות תא-סוג של ביטוי גנים. לפיכך, המדידה של תקנה epigenetic בסוג התא כל רצונך בכך, אבל זה כבר אתגר טכני.
כדי לחקור את אפיגנטיקה בסוג לתא מסוים, היה רצף immunoprecipitation טנדם כרומטין (tChIP-Seq) פיתחה לאחרונה (איור 1)1. TChIP, מבוטא מתויג epitope הליבה היסטון חלבון ויזת עבודה H2B מ מקדם ייחודיים לסוג התא. תכונה זו מאפשרת את ניתוקה של chromatins את התאים של עניין, למרות החומר מתחיל תערובת של סוגי תאים שונים. השבב הבא-Seq — כרומטין טיהור באמצעות שינוי היסטון מארק בעלת רצף עמוק הדור הבא של מבודדים DNA – נוכל לעקוב אחר המצב epigenetic סוג התא יישוב באופן הגנום כולו.
בעזרת טכניקה זו, חקרנו לאחרונה נוירון ספציפי trimethylation של היסטון H3 חלבון-ליזין 4 (H3K4me3) סימני. במחקר זה, פיתחנו בטוק עכבר ב אילו C-סופני מתויג דגל ויזת עבודה H2B חלבון בא לידי ביטוי על רקומבינציה בתיווך Cre (Rosa26חטיבתי floxed-pA ויזת עבודה H2B-דגל...). על ידי מעבר עם עכבר אחזקת הגן רשת (ER) Cre-endoplasmic תחת השליטה של האמרגן CamK2a, הקו העכבר שהושג המושרה ויזת עבודה H2B-דגל בנוירונים פעיל עם טמוקסיפן הזרקה (Camk2aויזת עבודה H2B-דגל)1. החל מהמוח של הקו העכבר הוקמה, ביצענו tChIP-Seq עם נוגדן anti-H3K4me3. מאז H3K4me3 סימני לעיתים קרובות להתאים לאזורים יזם, נוכל לגלות מאות mRNAs במיוחד לידי ביטוי נוירונים1.
כאן, אנו מתארים שיטה tChIP טיפוסי-Seq שמכסה את השלבים של רקמות לנתיחה לבנייה הספרייה (איור 1). המטרה הסופית של פרוטוקול זה היא לשתף שלנו מומלצות עבור הביצועים של tChIP-Seq לבין היישום העתידי של שיטה זו על סוגי תאים אחרים והשינויים היסטון.
Protocol
כל השיטות המתוארות במסמך זה מאושר על ידי מחלקת הבטיחות של RIKEN (H27-EP071), שנערך עם רלוונטיות הנחיות ותקנות.
1. רקמות לנתיחה
- לנתח את רקמות עניין לחתיכות קטנות (כ < 3 מ מ2) באמצעות מספריים אביבי נאה.
הערה: שברים גדולים יותר של רקמת להתארך להקפיא, חתיכות קטנות תישא על אחסון גדולים יותר מאגר, אשר שניהם עשויים להשפיע על התוצאות. - להוסיף שברי רקמת ביתור במיכל נקי מלא עם חנקן נוזלי ולאסוף אותם לתוך צינורות 2 מ ל (1 חתיכה למחזור) מלא עם חנקן נוזלי.
הערה: צינורות עם משטח הידרופילית מומלץ בשלב זה. - הצב הצינורות ב-80 מעלות צלזיוס עבור > חמש דקות עם הכיפה פתוח מתמוססות החנקן הנוזלי הנותרים.
הערה: לאחר סגירת הכיפה, שברי רקמת שניתן לאחסן ב-80 מעלות צלזיוס למשך חודש אחד לפני השימוש.
2. התא קיבוע
הערה: השתמשנו מטחנה קריוגני שימושי עבור שלב זה. מנגנון חלופי יכול לשמש.
- מקום 2 מ"ל-חלבון נמוכה מחייב צינורות המכיל את דגימות רקמה על מתלה מתכת קרח צונן של חנקן נוזלי.
- מניחים כדור מתכת 1.5 mL-שפופרת, תירגע עם חנקן נוזלי. למקם אותו על המדף קרח מתכת.
- פתח הצינור-mL 2 ולמקם את כדור מתכת prechilled לתוך הצינור. לסגור את הפקק, להגדיר 2 מ"ל-הצינורות לתוך צינור בעל מטחנה קריוגני בהישג יד, מיד לטבול בעל שפופרת שהורכב בחנקן נוזלי עבור 1 דקות.
- הכנס בעל שפופרת קפוא בקלטת החיצוני של מטחנת קריוגני בהישג יד ומנערים נמרצות 60 פעמים ב-30 s.
- לפרק את מחזיק שפופרת, להוציא את הקליע מתכת, מקום 2 מ"ל-שפופרת על מדגם קריר יותר prechilled ב-20 ° C.
- מקם את הדגימה קריר יותר ב-20 מעלות צלזיוס למשך 15 דקות למניעת קיפאון של מקבע בשלב הבא.
- להביא את הדגימה קריר הספסל, פותחים את המכסה של הצינור, מיד לטפטף µL 900 של 1% פורמלדהיד לתוכו. לאחר pipetting מספר פעמים, להעביר את המתלים לתוך חדש 2 מ"ל-שפופרת המכילה µL 900 של 1% פורמלדהיד, תיקון עבור 10 דקות עם סיבוב עדין בתוך אינקובטור שוכן בגובה 23 ° C.
התראה: פורמלדהיד הוא רעילים ואינם מזיקים. - כדי להפסיק את התגובה קיבעון, להוסיף 100 µL של גליצין 2.5 מטר כל שפופרת של צנטריפוגה למשך 5 דקות ב 3000 g x-4 מעלות צלזיוס. למחוק את תגובת שיקוע.
- להוסיף 1 מ"ל תמיסת מלח קרה כקרח באגירה פוספט (PBS), צנטריפוגה למשך 5 דקות ב 3000 g x-4 ° C, ולמחוק את תגובת שיקוע. חזור על שלב זה פעמיים (3 מנקי סה כ).
הערה: דגימות קבוע יכול להיות קפואה על-ידי חנקן נוזלי, המאוחסנת ב- 80 ° c - להוסיף 500 µL של פירוק מאגר 1 (50 מ מ 4-(2-hydroxyethyl)-1-piperazineethanesulfonic חומצה (HEPES)-קו (pH 7.5), 140 מ מ NaCl, 1 מ ethylenediaminetetraacetic חומצה (EDTA) (pH 8.0), 10% w/v גליצרול, 0.5% w/v NP-40, 0.25% w/v טריטון X-100, ו- 0.1 x מעכב פרוטאז קוקטייל) בגדר, pipet מספר פעמים, וסיבוב 10 דקות ב 4 º C.
הערה: פירוק מאגר 1 צריך להיות מסונן ומאוחסנים ב 4 ° C לפני השימוש. - צנטריפוגה למשך 5 דקות ב 3000 g x ב 4 ° C וזורקים את תגובת שיקוע.
- להוסיף 1 מ"ל של פירוק מאגר 2 (10 מ מ טריס-HCl (pH 8.0), 200 מ"מ NaCl, 1 מ"מ EDTA (pH 8.0), 0.5 מ מ EGTA, ו- 0.1 x מעכב פרוטאז קוקטייל) כדי בגדר, מערבולת, וסיבוב 10 דקות ב 4 º C.
הערה: פירוק מאגר 2 צריכה להיות מסונן ומאוחסנים ב 4 ° C לפני השימוש. - צנטריפוגה למשך 5 דקות ב 3000 g x ב 4 ° C וזורקים את תגובת שיקוע.
- להוסיף µL 800 radioimmunoprecipitation assay (ריפה) מאגר עם מעכב פרוטאז 1 x קוקטייל בגדר, resuspend בגדר על-ידי pipetting.
- צנטריפוגה למשך 5 דקות ב 3000 g x ב 4 ° C וזורקים את תגובת שיקוע.
- להוסיף 500 µL ריפה מאגר עם מעכב פרוטאז x 1 קוקטייל.
- צנטריפוגה למשך 5 דקות ב 3000 g x ב 4 ° C וזורקים את תגובת שיקוע.
- להוסיף 1 מ"ל ריפה מאגר עם מעכב פרוטאז x 1 קוקטייל. מיד להמשיך לשלב הבא.
3. כרומטין הטיה
- להעביר את lysate צינור sonicator ולאחר מכן מקם את הצינורית על ultrasonicator.
- הטיה של כרומטין עם ההגדרות הבאות: טמפרטורה: 4 ° C; שיא האירוע כוח: 175 וואט; פקטור חובה: 10%; מחזורים/פרץ: 200; זמן: 2,400 s.
- להעביר את הדגימה mL-חלבון נמוכה איגוד צינור 1.5 ו צנטריפוגה למשך 5 דקות ב 20,000 g x-4 מעלות צלזיוס.
- לאסוף את תגובת שיקוע לתוך צינור נמוך מחייב חלבון חדש.
הערה: המדגם יכול להיות קפוא בחנקן נוזלי ומאוחסנים ב-80 מעלות צלזיוס.
4. איכות הסימון של ה-DNA (Crosslinking הפוך)
- מיקס 20 µL של lysate ו- 180 µL שבב • תנאי מאגר (10 מ מ טריס-HCl (pH 8.0), 300 מ"מ NaCl, 5 מ מ EDTA (pH 8.0), ו 1% w/v dodecyl נתרן גופרתי (מרחביות)) צינור תגובת שרשרת (PCR) פולימראז. דגירה המדגם-65 מעלות צלזיוס הצנטרפוגה תרמית עבור 6-אייץ ' עם המכסה הצנטרפוגה תרמי הפתוחים. שמור את מכסה פתח הצנטרפוגה תרמית כדי למנוע דנטורציה יתר.
הערה: המאגר • תנאי שבב צריך להיות מסונן, בטמפרטורת החדר לפני השימוש. ניתן להאריך את הדגירה ללון. - להוסיף 1 µL של 10 מ"ג/מ"ל RNase, מערבולת, דגירה ב 37 מעלות צלזיוס למשך 30 דקות.
- להוסיף µL 6.5 של 15 מ"ג/מ"ל proteinase K, מערבולת, דגירה ב 55 מעלות צלזיוס למשך 60 דקות.
- להעביר את התגובה צינור DNA קשירה נמוכה, להוסיף 4 µL של 5 מ"ג/מ"ל ו מערבולת. לאחר מכן, הוסף 200 µL של אלכוהול פנול/כלורופורם/isoamyl (25:24:1), צנטריפוגה במשך 5 דקות ב g x 18,000 בטמפרטורת החדר.
- להעביר את תגובת שיקוע צינור נמוך איגוד ה-DNA. להוסיף 200 µL מאגר טריס-EDTA-NaCl (10 מ מ טריס-HCl (pH 8.0), 1 מ"מ EDTA (pH 8.0) ו-200 מ מ NaCl) תמיסת כוהל פנול/כלורופורם/isoamyl הנותרים, צנטריפוגה למשך 5 דקות ב g x 18,000 בטמפרטורת החדר. לאסוף את תגובת שיקוע ומערבבים עם תגובת שיקוע הראשון.
- להוסיף µL 900 של אתנול קר כקרח, תקופת דגירה של h 1 ב-20 ° C.
הערה: הדגירה זו ניתן להרחיב ל 4 - 6 שעות. - צנטריפוגה למשך 30 דקות ב 18,000 g x-4 מעלות צלזיוס.
- למחוק את תגובת שיקוע. להוסיף 1 מ"ל אתנול 75% קר כקרח צניפה, צנטריפוגה למשך 30 דקות ב 18,000 g x-4 מעלות צלזיוס. חזור על שלב זה (שני מנקי סה כ).
- למחוק את תגובת שיקוע ביסודיות, מילה נהדרת עבור 1 דקות בטמפרטורת החדר.
- הוסף µL 50 מאגר טריס-EDTA (TE), לפזר את ה-DNA על ידי המקננת זה בטמפרטורת החדר למשך 30 דקות או ב 4 מעלות צלזיוס למשך הלילה. להימנע vortexing או pipetting. לשמור את הדנ א "קלט" ולהשתמש בו עבור ספריית הכנה במידת הצורך.
- למדוד את ריכוז הדנ א עם fluorometer לפי הוראות היצרן (ראה טבלה של חומרים) לבדוק את התפלגות גודל עם מכונה אלקטרופורזה microfluidic (ראה נציג נתונים המוצגים איור 2A)-שימוש 10 ng או פחות דנ א זה וזמינותו.
הערה: קטעים של 100-500 בסיסים (bp) שתתפייס 50% או יותר של ה-DNA.
5. הזיקה טיהור על ידי נוגדנים אנטי-דגל
הערה: עבור נוירון tChIP-Seq, ברקמות המוח של עכברים 8 משמשים בדרך כלל עבור מדגם אחד של tChIP-Seq להכין 2 ng של DNA מטוהרים מאת טנדם החיסון-טיהור (ראה צעד 7.1). בידיים שלנו, ספריות DNA באיכות גבוהה ng של DNA עדיין מסופקים 0.1 עבור שבב-Seq (Kadota, לא פורסם). לפיכך, באופן תיאורטי, עכבר אחת עשוי להיות מספיק לבצע נוירון tChIP-תת סעיף צריך להיות מוטבת הכמות הדרושה עבור סוג הרקמה והתא עניין. עבור הפקד שלילי של הניסוי imumuno-טיהור, רקמת המוח lysate של עכברים ללא כל ביטוי ויזת עבודה H2B-דגל צריך להיות מוכן ולא להשתמש בו.
- פיפטה µL 200 של חרוזים מגנטי מצומדת כדי כבשים אנטי-העכבר אימונוגלובולין G (IgG) לתוך צינור חלבון נמוכה מחייב.
- מקם את הצינורית אל דוכן מגנטי ולחכות 1דקות. עבור תגובת שיקוע להתבהר. למחוק את תגובת שיקוע, להוסיף 1 מ"ל של PBS קר כקרח, מערבולת. חזור על שלב זה (שני מנקי סה כ).
- הוסף 20 µL של נוגדן אנטי-דגל 1 מ"ג/מ"ל ולסובב עבור 5 שעות-4 מעלות צלזיוס.
הערה: ניתן להרחיב את הדגירה ללון. - לשטוף את החרוזים הבאים 5.2 שלב. מקם את החרוזים לתוך צינור 15-mL.
- Resuspend את החרוזים בשנת 1110 µL ריפה המאגר ולאחר מכן להוסיף µL 900 של 10 x חסימת ריאגנט 180 µL של 50 x הפתרון של Denhardt, µL 10 של 100 x מעכב פרוטאז קוקטייל, 6.8 מ ל lysate המוכנים בשלב 3. פצל תערובת זו 5 צינורות קשירה נמוכה של חלבון. לסובב עבור 6-אייץ '-4 מעלות צלזיוס.
הערה: ניתן להרחיב את הדגירה ללון. - מקם את הצינורית על גבי המעמד מגנטי ולחכות 1דקות. עבור תגובת שיקוע להתבהר. למחוק את תגובת שיקוע, מוסיפים 1 מ"ל של מאגר ריפה, ומערבבים בעדינות. חזור על שלב זה 5 פעמים (6 מנקי סה כ). מאגר כל החרוזים לתוך צינור נמוך מחייב חלבון יחיד.
- להוסיף 200 µL שבב • תנאי מאגר וסובב למשך 15 דקות בטמפרטורת החדר. בדוק את איכות ה-dna כפי שמתואר בשלב 4.11 (ראה נציג נתונים המוצגים איור 2B ו- C). מיד להמשיך לשלב הבא.
6. זיקה טיהור ע י נוגדן Anti-H3K4me3 Crosslinking הפוכה
הערה: הבאים חרוז הכנת שהשלבים (6.1 – 6.4) צריכה להתבצע לפני שלב 5.7.
- פיפטה µL 40 של beads מגנטי מצומדת כבשים העכבר אנטי איג לתוך צינור 2 מ"ל-חלבון נמוכה מחייב.
- מקם את הצינורית על גבי המעמד מגנטי ולחכות 1דקות. עבור תגובת שיקוע להתבהר. למחוק את תגובת שיקוע, מוסיפים 1 מ"ל של PBS קר כקרח, ומערבבים בעדינות. חזור על שלב זה (שני מנקי סה כ).
- להוסיף 4 µL של נוגדן anti-H3K4me3 1 מ"ג/מ"ל ולסובב עבור 6-אייץ '-4 מעלות צלזיוס.
- לשטוף את החרוזים בעקבות צעד 6.2. מקם את החרוזים לתוך צינור 2 מ"ל-חלבון נמוכה מחייב.
- Resuspend את החרוזים ב- 1558 µL ריפה המאגר ולאחר מכן להוסיף 200 µL של 10 x חסימת ריאגנט, µL 40 של 50 x הפתרון של Denhardt, 2 µL של 100 x מעכב פרוטאז קוקטיילים ו 200 µL של eluate המבושלות שלב 5.7. סובב בין לילה ב 4 º C.
הערה: זמנים תוססים במאגר • תנאי השבב היה מדולל יותר מ 10 פעמים ב הזה הדגירה. - מקם את הצינורית על גבי המעמד מגנטי ולחכות 1דקות. עבור תגובת שיקוע להתבהר. למחוק את תגובת שיקוע, מוסיפים 1 מ"ל של מאגר ריפה, ומערבבים בעדינות. חזור על שלב זה 4 פעמים (5 מנקי סה כ). לאחר השטיפה האחרונה להעביר את החרוזים לתוך צינור נמוך איגוד ה-DNA, ולמחוק את תגובת שיקוע.
- להוסיף 200 µL שבב • תנאי מאגר וסובב למשך 15 דקות בטמפרטורת החדר. להעביר את eluate bindingTube נמוך החדש ה-DNA.
הערה: eluate שניתן לאחסן ב- 80 ° c - בצע את שלב 4 עבור decrosslinking של ה-DNA. Resuspend ה-DNA מטוהרים ב 20 µL טה המאגר.
- בדוק את איכות ה-dna כפי שמתואר בשלב 4.11 (ראה נציג נתונים המוצגים באיור 2D). (אופציונלי) ביצוע ניתוח העשרה על ידי ה-PCR כמותי כמתואר קודם לכן2. Ten-fold או העשרה יותר להתייחס.
7. ספריית בנייה
- לכל קלט או שבב דנ א, לחשב את שיעור ה-DNA באזור 100-500-bp באמצעות הפונקציה ניתוח השמצות של התוכנה עבור המכונה אלקטרופורזה microfluidic. מעריכים את אמצעי האחסון מדגם הנדרשים על מנת לקבל 2 ng של 100-500 bp הדנ א. להשתמש 20 ng DNA בטווח של 100-500-bp המדגם "קלט".
- פיפטה דגימות די אן איי לתוך חשפנות-8 המבחנות ולהוסיף H2O כדי להביא את הנפח הכולל 10 µL. (אופציונלי) אם אמצעי האחסון של ה-DNA חורג µL 10, באופן פרופורציונלי הסולם התגובה הבאה סוף-תיקון בחירת גודל.
- להכין מיקס מאסטר סוף-תיקון (ראה טבלה של חומרים). להוסיף 4 µL של מיקס מאסטר סוף-תיקון ה-DNA, תקופת דגירה של 30 דקות ב- 20 ° C.
- להוסיף µL 36 10 מ מ טריס-קלרנית, pH 8.5 ו- 0.6 x נפח (30 µL) של מוצק שלב beads מגנטי הפיך הנייח (אלוהים אדירים) תקופת דגירה של 5 דקות בטמפרטורת החדר.
- מניחים את הצינורית על דוכן מגנטי והמתן עד תגובת שיקוע נעשה ברור.
- להעביר את תגובת שיקוע צינור, להוסיף כרכים x 1.2 (60 µL) של אלוהים אדירים beads מגנטי, דגירה 5 דקות בטמפרטורת החדר.
- מניחים את הצינורית על דוכן מגנטי והמתן עד תגובת שיקוע נעשה ברור.
- לשטוף את החרוזים פעמיים עם 200 µL של 80% EtOH, מחזיק את הצינור עדיין על המגנט.
- בקצרה centrifuge צינור כדי לאסוף את EtOH שיורית בתחתית ולמקם אותו בחזרה על המגנט. הסר את EtOH שיורית ביסודיות.
- השאר את המכסה צינור פתוח למשך 1-2 דקות לאפשר את EtOH מתמוססות.
- סגור את המכסה ולשמור את הצינורית על קרח.
הערה: עכשיו אתה השגת DNA נבחר גודל של 100-500 bp על החרוזים. פרטים נוספים אודות בחירת גודל כפול ניתן למצוא באתר האינטרנט של היצרן (www.beckman.com). - הכנת אב א-עוקב לערבב (ראה טבלה של חומרים).
- Resuspend את החרוזים (מתוך שלב 7.10) עם µL 10 של מיקס מאסטר א-עוקב, תקופת דגירה של 30 דקות ב- 30 ° C.
- להוסיף x 1.8 נפח (18 µL) של פוליאתילן גליקול (PEG) / פתרון NaCl דגירה למשך 5 דקות בטמפרטורת החדר.
- בצע שלבים 7.7-7.11 כדי לטהר את ה-DNA.
- להכין תערובת מאגר מצדו (ראה טבלה של חומרים).
- פיפטה 8 µL של שילוב מאגר מצדו על גבי החרוזים (מתוך שלב 7.14), להוסיף 1 µL 1 מיקרומטר מתאם, resuspend את החרוזים. השתמש µL 1 של 5 מיקרומטר מתאם עבור המדגם קלט. שקול שימוש במתאמים שונים עבור כל דגימה עבור ריבוב.
- להוסיף 1 µL של ה-DNA ליגאז ולאחר תקופת דגירה של 15 דקות ב 20 º C.
- להוסיף נפח x 1.0 (10 µL) של פג/NaCl פתרון, resuspend את החרוזים, תקופת דגירה של 5 דקות בטמפרטורת החדר.
- בצע שלבים 7.7-7.10 כדי לטהר את ה-DNA.
- פיפטה 25 µL 10 מ מ טריס-קלרנית, pH 8.5 לתוך הצינור, resuspend את החרוזים.
- להוסיף נפח x 1.0 (25 µL) של פג/NaCl פתרון, תקופת דגירה של 5 דקות בטמפרטורת החדר.
- בצע שלבים 7.7-7.10 כדי לטהר את ה-DNA.
- פיפטה µL 11 10 מ מ טריס-קלרנית, pH 8.5 לתוך הצינור, resuspend את החרוזים.
- מניחים את הצינורית על דוכן מגנטי והמתן עד תגובת שיקוע נעשה ברור.
- לאסוף את תגובת שיקוע לתוך צינור 8-רצועת ה-PCR.
- להכין בזמן אמת PCR מיקס מאסטר (ראה חומרים לפרטים).
- פיפטה 8.5 µL של בזמן אמת PCR מיקס מאסטר בצלחת (qPCR) PCR כמותי 384-ובכן, להוסיף 1.5 µL של המתאם מאתרים דנ א (מתוך שלב 7.26).
- פיפטה 10 µL של כל תקן פלורסנט בבארות ריק.
- לבצע PCR בזמן אמת עם programm הבאים: (98 ° C עבור 45 s) x מחזור 1, (98 ° C 15 s, 60 ° C ל 30 s, 72 ° C ל 30 s) x 20 מחזורים, ו (72 ° C 60 s) x מחזור 1.
- לקבוע את מספר מחזורי PCR צריכה להגיע עוצמת קרינה פלואורסצנטית של 2 רגיל פלורסנט.
- הכנת הבסיס PCR לערבב (ראה טבלה של חומרים).
- פיפטה 11.5 µL של המיקס מאסטר PCR לתוך הדי ובין אם-לא מתאם הנותרים (מתוך שלב 7.26) ולבצע PCR כמצוין ב 7.30 צעד עם המחזורים PCR נקבע שלב 7.31.
- להוסיף נפח x 1.0 (20 µL) של פג/NaCl פתרון, תקופת דגירה של 5 דקות בטמפרטורת החדר.
- בצע שלבים 7.7-7.10 כדי לטהר את ה-DNA.
- פיפטה 20 µL 10 מ מ טריס-קלרנית, pH 8.5 לתוך הצינור, resuspend את החרוזים.
- מניחים את הצינורית על דוכן מגנטי והמתן עד תגובת שיקוע נעשה ברור.
- לאסוף את תגובת שיקוע לתוך צינור 1.5 מ ל- DNA קשירה נמוכה.
- בדוק את האיכות של הספרייה כפי שמתואר בשלב 4.11 (ראה נציג נתונים המוצגים איור 2E ו- F). השתמש µL 1 של דגימת DNA הספרייה. (אופציונלי) לבצע ניתוח העשרה על ידי qPCR כפי שתואר לעיל 2. Ten-fold או העשרה יותר להתייחס.
8. רצף
- רצף של הספריות ב ברצף הדור הבא (ראה נציג נתונים בתרשים 3).
הערה: העומק רצף מספיק עבור ניתוח הנתונים משתנה בהתאם לגודל הגנום של האורגניזם3. עבור האדם העכבר, ואנו ממליצים קבלת קריאות יחיד-end לפחות 20-40 מיליון. על פי התשואה הקריאות, ריבוב עשוי להיות חסכוני.
תוצאות
כאן, אנו מתארים את רקמת לנתיחה, קיבוע, פירוק התא, טנדם טיהור של כרומטין, ספריית DNA הכנה סקוונסרים הדור הבא. במהלך ההליכים, אחד יכול לבדוק את האיכות של ה-DNA, אשר היא המפתח רצף מוצלח, על מספר השלבים (איור 2). מאז נוקלאוזום בודד מוקף בדרך כלל bp 147 דנ א 4, הוטו DNA לא צריך להיות קצר יותר בגודל הזה. מיד לאחר ultrasonication, ה-DNA היה מבודד, לפעול על מחשב אלקטרופורזה microfluidic (איור 2 א). למרות מאמצינו כדי למטב את הטווח בגודל, ועדיין יש אוכלוסיה של DNA (כ 2 kbp) נשארו unsheared. בשלב זה, השבר של DNA הנע בין 100-500 bp צריך להיות 50% או יותר של האוכלוסייה. לאחר טיהור זיקה נוגדן אנטי-דגל (איור 2B) ולאחר מכן נוגדן anti-H3K4me3 (איור דו-ממדי), האיכות של ה-DNA מבודדות נבדקה שוב באמצעות המכונה אלקטרופורזה microfluidic, ואת כמות הדנ א החל bp 100-500 הוערך. אמנם לעתים קרובות הבחנו דעה קדומה חזקה לכיוון 2 שברי kbp בשלב זה, כפול גודל הבחירה על ידי אלוהים אדירים beads מגנטי הסיר הזה שבר.
ייחודה של החיסון-טיהור של ה-DNA על ויזת עבודה H2B-דגל אושר עם הדגימות בקרה שלילית, lysate של עכברים ללא ויזת עבודה H2B-דגל במוח אשר שימש הביטוי (איור 2C). אנחנו בדרך כלל זוהה כמויות זניח של ה-DNA בין דגימות שליטה שלילי.
בעקבות A-עוקב, מתאם מצדו, PCR, אומתה האיכות של הספרייה רצף (2E איור). בדרך כלל, 250-600 bp DNA הושג. שבב רגילים מוצלחת ואת tChIP היו שמעידים הניתוח העשרה עם qPCR2 באמצעות צבעי בסיס פילוח אזור המקדם של הגן GAPDH (איור 2F).
נציג לקרוא הפצות לאורך הגנים נוירון (Camk2a, Slc17a7 ו Gria1) על-ידי הגנום-דפדפן מוצגים באיור3. העשרת הקריאות בסוף יונקות גנים על ידי נוירון tChIP-Seq מעל כל המוח tChIP-Seq נצפתה.

איור 1: ייצוג סכמטי של tChIP-תת סעיף. איור זה שונה מ- Mito, מ' ואח. 1. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
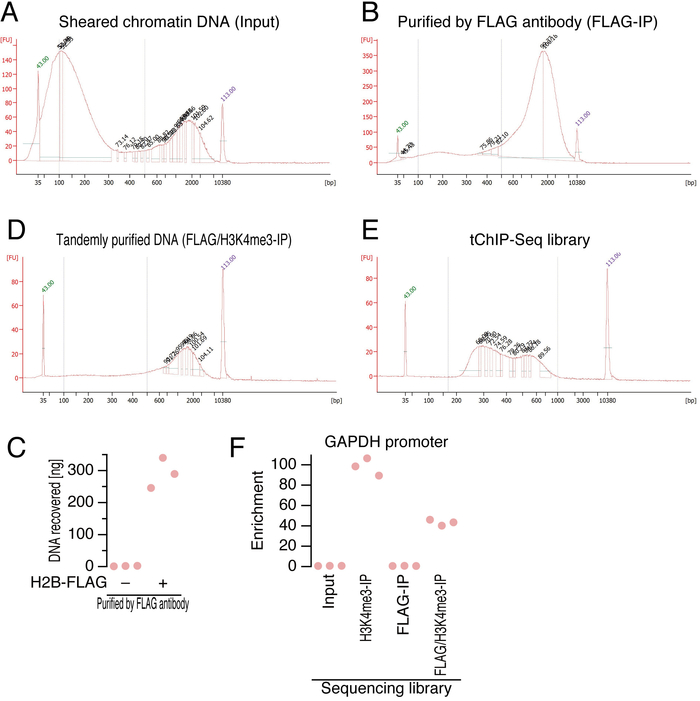
איור 2: בדיקת האיכות דנ א. (A, B, Dו- E) Electropherogram שהושג באמצעות מכונת אלקטרופורזה microfluidic עבור הוטו כרומטין דנ א (א), דנ א טהור עם נוגדן אנטי-דגל (B) נוגדן anti-H3K4me3 (ג), ואת הספרייה הסופי (D). הפסגות מסומנות במספרים ירוק וסגול מייצגים פנימי בגודל תקני. כחול קווים מקווקווים מציינים את האזורים גודל ה-DNA נחשב לניתוח במורד הזרם. פו: יחידות קרינה פלואורסצנטית.
(ג) כמות ה-DNA התאושש על ידי דגל נגד החיסון-טיהור. כל נקודה מייצג של שכפול עצמאי. (F) ניתוח העשרה לפי qPCR מיקוד GAPDH. כל נקודה מייצג של שכפול עצמאי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3: לקרוא התפלגויות של נוירון tChIP-תת סעיף (A-C) הפצות לקריאה לאורך הנוירון נציג גנים נוירון tChIP-Seq, שליטה כל המוח tChIP-Seq מוצגים. סל"ד: קורא לכל קורא מיליון. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
פרוטוקול שלנו היה ממוטב הנוירונים במוח העכבר, שבו הביטוי של ויזת עבודה H2B מתויג דגל הנגרמת על ידי הזרקת טמוקסיפן. היזמים להשתמש בביטוי ויזת עבודה H2B, חומרים רקמת המוצא של הסכום של הרקמות הם פרמטרים מרכזי עבור מוצלחת tChIP-תת סעיף. לפיכך, אופטימיזציה של גורמים אלה להתייחס לכל סוג התא של ריבית.
שלב קריטי בין ההליכים בשימוש פרוטוקול זה הוא ה-DNA הטיה כדי להשיג באורך כרומטין של bp 100-5005. באופן כללי, סטנדרטיזציה של השלב ultrasonication הוא מאתגר מאז זה מאוד תלוי במגוון גורמים; קיבוע תנאים, הרקמות בשימוש, הציוד המשמש אולטרה-sonication וציוד את הגדרת, בין היתר. לכן, משפט--שגיאה אופטימיזציה של שלב חיתוך הדנ א יידרש לניסויים בודדים. במקום אולטרה-sonication, נוקלאז micrococcal הטיפול צריך להיות אפשרות לבודד יחיד בגודל נוקלאוזום ה-DNA, המשמשים ב שיטת שבב6תמים. למרות הומוגנית התפלגות גודל של ה-DNA הוא אידיאלי, לכסנתם DNAs עשוי להציג את אוכלוסיות מרובות, כפי שמוצג באיור 2A. בחירת גודל כפול beads מגנטי אלוהים אדירים, כמתואר לעיל, מסייע להסיר את יותר בגודל של קטעי DNA.
בדרך כלל, ספריית הכנה דורש 1 ng של 100-500 bp הדנ א. בהתבסס על דרישה זו, אנחנו יורה את החומר הראשוני של רקמת המוח לתאי העצב. עם זאת, הליך זה יכול להיות מאתגר עבור מדגמים קטנים מאוד של סוגי תאים נדיר. במקרים כאלה, שבב mentation, אשר מבוסס על Tn5 transposase במקום ליגאז ודורש פחות גשמי7, יכול להיות חלופה טובה יותר.
למרות זאת, פרוטוקול הנוכחי זה מוגבל לסוגי תאים עיקריים בחומר. בידיים שלנו, 10% תפוסה של תאים ממוקדות באוכלוסייה התא המקורי שסופק טיהור הוגן של כרומטין היעד תאים1. עם זאת, אם סוגי תאים נדירים יותר הם מטרה, ואז אופטימיזציה עדינה יותר של immunoprecipitation על ידי התג epitope תינתן לבידול ה-DNA מבודדים מזוהמים ה-DNA של תאים שאינם במיקוד. על שיטות עבודה מומלצות של ניתוח הנתונים, אנו ממליצים השוואת הנתונים לניתוח tChIP-Seq שליטה מתוך הרקמה כל להשתמש כדי ליזום את הניסוי. מאז שמצאנו הטיות משמעותית מ exogenously ביטוי חלבון ויזת עבודה H2B-דגל, הניתוח צריך להיות בהשוואה ולא נותחו על ידי העשרה (או דלדול) הרקע1. עבור פקד זה, אנחנו אכן נוצר קו עכבר עם ויזת עבודה H2B ubiquitously ביטוי-דגל (רוזהויזת עבודה H2B-דגל) וביצע tChIP Seq1. פרשנות מפורטת יותר של הנתונים כבר דנו בעבר1.
על סמך הניתוח שלנו, H3K4me3 tChIP-Seq של נוירונים מספק זיהוי דומות או יותר של גנים ספציפיים נוירון ידוע מ- RNA-Seq של נוירונים ממוין FACS8 ו מתרגם ריבוזום זיקה טיהור (השמנה)-Seq9. לדוגמה, גנים האקסון/דנדריט-מקומי ידוע נמצאו ביעילות באמצעות neuron tChIP Seq1. למרבה הפלא, tChIP-Seq שלנו אינה מוגבלת בסימן היסטון יזם-הקשורים אך חל גם שינוי היסטון שונים10 אם הנוגדנים זמינים. בנוסף, ניתן להשתמש את האסטרטגיה של בידוד כרומטין על ידי חלבוני היסטון הליבה מתויג אורגניזמים אחרים מודל. למרות כאן השתמשנו epitope את הדגל כדי לתייג את החלבון היסטון ליבה, ניתן להחיל תגים אחרים. מאז פורמלדהיד קיבוע ל- DNA מתרחשת לרוב ליזין שאריות חלבון11, יש להימנע תג עם ליזין רבים כדי למנוע הטיות על-ידי תיוג epitope. לפיכך, הגישה tChIP-Seq צריך להיות תכליתי ברקמות מגוונות לאורך סוגים רבים של הקשרים במחקרים epigenetic.
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנו מודים לכל חברי המעבדה איוואסאקי לקריאה ביקורתית של כתב היד. עבודה זו נתמכה בחלקו על ידי מענק הסיוע למחקר מדעי בתחומים חדשניים (#26113005 כיהן ו JP17H05679 ל ס. י); מענק הסיוע מדענים ומפתחים צעירים (א) (JP17H04998 ל ס. י) מ משרד החינוך, המדע, הספורט והתרבות של יפן (MEXT); והחלוץ פרויקטים "אבולוציה סלולרית" ו RIKEN כל פרוייקט "המחלה, Epigenome" מ RIKEN (כדי כיהן, ס. י).
Materials
| Name | Company | Catalog Number | Comments |
| Protein LoBind tube, 2 mL | Eppendorf | No. 0030108132 | For cell lysis |
| Protein LoBind tube, 1.5 mL | Eppendorf | No. 0030108116 | For ChIP and library preparation |
| DNA LoBind tube, 1.5 mL | Eppendorf | No. 0030108051 | For ChIP and library preparation |
| 8-strip PCR tube | BIO-BIK | 3247-00 | For ChIP and library preparation |
| SK Mill | TOKKEN | SK-200 | Handy cryogenic grinder to make cell powder for fixation |
| Metal bullet | TOKKEN | SK-100-DLC10 | Accessory of SK Mill |
| 2 mL stainless steel tube | TOKKEN | TK-AM5-SUS | An option for cell lysis |
| 2 mL stainless steel tube holder | TOKKEN | SK-100-TL | An option for cell lysis |
| 16% formaldehyde (w/v), methanol-free | Pierce | 28906 | To fix cells. Prepare 1% solution before use. |
| Glycine | Nacalai Tesque | 17109-35 | Prepare 2.5 M stock |
| D-PBS (-)(1x) | Nacalai Tesque | 14249-24 | For washing lysate and purified DNA |
| HEPES | Nacalai Tesque | 02443-05 | For Lysis buffer 1. Prepare 1 M, pH 7.5 stock. |
| 5 M NaCl, molecular biology grade | Nacalai Tesque | 06900-14 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| 0.5 M EDTA, molecular biology grade | Wako Pure Chemical Industries, Ltd. | 311-90075 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| Glycerol | Wako Pure Chemical Industries, Ltd. | 072-04945 | For lysis buffer 1 |
| NP-40 | Nacalai Tesque | 25223-75 | For lysis buffer 1 |
| Triton X-100, molecular biology grade | Nacalai Tesque | 12967-32 | For Lysis buffer 1 |
| Tris | Nacalai Tesque | 35406-91 | For Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer. Prepare 1 M, pH 8.0 stock. |
| 0.1 M EGTA pH neutral | Nacalai Tesque | 08947-35 | For Lysis Buffer 2 |
| Protease inhibitor cocktail (100x) | Nacalai Tesque | 25955-24 | To block degradation of protein |
| RIPA buffer | Thermo Fisher Scientific | 89900 | For cell lysis and washing |
| milliTUBE 1 mL AFA Fiber | Covaris | 520130 | Sonicator tube. Accessory of Focused-ultrasonicator |
| Focused-ultrasonicator | Covaris | S220 or E220 | To digest DNA into adequate size for ChIP-Seq |
| UltraPure 10% SDS | Thermo Fisher Scientific | 15553-027 | For ChIP Elution Buffer |
| RNase A | Nacalai Tesque | 30141-14 | To purify DNA from lysate |
| Proteinase K, recombinant, PCR Grade | Sigma-Aldrich | 3115887001 | To purify DNA from lysate |
| Ethanol | Wako Pure Chemical Industries, Ltd. | 054-07225 | Make 70% solution |
| Monoclonal anti-FLAG M2 antibody produced in mouse | Sigma-Aldrich | F1804 | To purify chromatin expressed in cells of interest |
| Dynabead M-280 Sheep Anti-Mouse IgG | Thermo Fisher Scientific | 11201D | This can be used for anti-FLAG IP and anti-H3K4me3 IP |
| Anti-tri-methyl histone H3 (K4), mouse monoclonal antibody | Wako Pure Chemical Industries, Ltd. | 301-34811 | Any other antibody that works for ChIP analysis will work |
| 10x Blocking Reagent | Sigma-Aldrich | 11096176001 | For blocking during affinity purification |
| Denhardt’s solution | Nacalai Tesque | 10727-74 | For blocking during affinity purification |
| Glycogen (5 mg/ml) | Thermo Fisher Scientific | AM9510 | To purify DNA from lysate |
| Qubit 2.0 Fluorometer | Thermo Fisher Scientific | Q32866 | For quantification of isolated DNA |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | For quantification of isolated DNA |
| 0.5 mL tube | Axygen | 10011-830 | For quantification by Qubit |
| Phenol/chloroform/isoamyl alcohol (25:24:1) | Nacalai Tesque | 25970-56 | To purify DNA from lysate |
| AMPure XP beads | Beckman Coulter | A63881 | SPRI magnetic beads for library preparation |
| Metal ice rack | Funakoshi | IR-1 | To keep the cell lysate frozen |
| Sample Cooler | New England Biolabs | T7771S | Helps fix cells with minimal damage |
| 2100 Bioanalyzer | Agilent Technologies | G2939BA | To check the quality of isolated DNA fragments. Another fragment analyzer can be used. |
| Bioanalyzer 2100 Expert Software | Agilent Technologies | G2946CA | Supplied with the Bioanalyzer |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | To check the quality of the isolated DNA fragments |
| KAPA LTP Library Preparation Kit | Roche | 07961898001 | Supplied with 10x KAPA End Repair Buffer, KAPA End Repair Enzyme Mix, KAPA A-Tailing Buffer, KAPA A-Tailing Enzyme, KAPA Ligation Buffer, KAPA DNA Ligase, and PEG/NaCl solution |
| NEXTflex DNA Barcodes | BIOO Scientific | NOVA-514101 | Adapter for library preparation. Supplied with DNA Barcode Adapters and Primer Mix. |
| KAPA Real-Time Library Amplification Kit | Roche | 07959028001 | Supplied with 2x KAPA HiFi HS real-time PCR Master Mix, PCR Primer Mix, and Fluorescent Standards |
| 2x KAPA HiFi HotStart ReadyMix | Roche | KM2602 | For library preparation. Additionally, this enzyme may be required for the KAPA Real-Time Library Amplification Kit |
| Buffer EB | Qiagen | 19086 | 10 mM Tris-Cl, pH 8.5 for elution of DNA |
| 386-well qPCR plate | Thermo Fisher Scientific | 4309849 | For real-time PCR |
| QuantStudio 7 Flex Real-Time PCR System | Thermo Fisher Scientific | 4485701 | To quantify DNA |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 4311971 | For real-time PCR |
| MicroAmp Clear Adhesive Film | Thermo Fisher Scientific | 4306311 | For plate sealing |
| End-repair master mix | Combine 1.4 µL of 10x KAPA End Repair Buffer, 1 µL of KAPA End Repair Enzyme Mix, and 1.6 µL of H2O | ||
| A-taling master mix | Combine 1 µL of KAPA A-Tailing Buffer, 0.6 µL of KAPA A-Tailing Enzyme, and 8.4 µL of H2O | ||
| Ligation buffer mix | Combine 2 µL of KAPA ligation buffer and 6 µL of H2O | ||
| Real-time PCR master mix | Combine 5 µL of 2x KAPA HiFi HS real-time PCR Master Mix, 0.35 µL of PCR Primer Mix (10 µM each of forward primer AATGATACGGCGACCACCGAG and reverse primer CAAGCAGAAGACGGCATACGAG), and 3.15 µL of H2O | ||
| PCR master mix | Combine 10 µL of 2x KAPA HiFi Ready Mix, 0.9 µL of PCR Primer Mix, and 0.6 µL of H2O | ||
| Integrative Genomics Viewer | Broad Institute | IGV_2.3.88 | Genome browser to visualize sequencing data |
| DNA olgionucleotide: 5′-GCCTACGCAGGTCTTGCTGAC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| DNA olgionucleotide: 5′-CGAGCGCTGACCTTGAGGTC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| SYBR Premix Ex Taq | Takara | RR420L | To quantify the DNA corresponding to the GADPH promoter region |
| Thermal Cycler Dice | Takara | TP870 | To quantify the DNA corresponding to the GADPH promoter region |
References
- Mito, M., et al. Cell type-specific survey of epigenetic modifications by tandem chromatin immunoprecipitation sequencing. Scientific Reports. 8 (1), 1143 (2018).
- Kadota, M., et al. CTCF binding landscape in jawless fish with reference to Hox cluster evolution. Scientific Reports. 7 (1), 4957 (2017).
- Jung, Y. L., et al. Impact of sequencing depth in ChIP-seq experiments. Nucleic Acids Research. 42 (9), (2014).
- Becker, P. B., Workman, J. L. Nucleosome remodeling and epigenetics. Cold Spring Harbor Perspectives in Biology. 5 (9), a017905 (2013).
- Kidder, B. L., Zhao, K. Efficient library preparation for next-generation sequencing analysis of genome-wide epigenetic and transcriptional landscapes in embryonic stem cells. Methods in Molecular Biology. , 3-20 (2014).
- Gilfillan, G. D., et al. Limitations and possibilities of low cell number ChIP-seq. BMC Genomics. 13, 645 (2012).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Zhang, Y., et al. An RNA-sequencing transcriptome and splicing database of glia, neurons, and vascular cells of the cerebral cortex. Journal of Neuroscience. 34 (36), 11929-11947 (2014).
- Hornstein, N., et al. Ligation-free ribosome profiling of cell type-specific translation in the brain. Genome Biology. 17 (1), 149 (2016).
- Zhao, Y., Garcia, B. A. Comprehensive catalog of currently documented histone modifications. Cold Spring Harbor Perspectives in Biology. 7 (9), a025064 (2015).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: a tool for the study of chromatin complexes. The Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved