Method Article
تشيب-Seq: التنميط Epigenome الخلية-نوع معين
In This Article
Summary
يمكننا وصف بروتوكول خطوة بخطوة للترادف الكروماتين إيمونوبريسيبيتيشن التسلسل (تشيب-Seq) التي تمكن من تحليل الخلية-نوع معين على نطاق الجينوم هستون تعديل.
Abstract
تنظيم جينية يلعب الأدوار المركزية في التعبير الجيني. منذ اكتشفت هستون تعديل في الستينات، بوظائفها الفسيولوجية والمرضية وقد درست على نطاق واسع. والواقع أن ثورة ظهور الجيل التالي تسلسل العميق وإيمونوبريسيبيتيشن الكروماتين (رقاقة) عن طريق محددة هستون تعديل الأجسام المضادة رأينا التنظيم جينية عبر الجينوم. على العكس من ذلك، الأنسجة وعادة ما تتألف من أنواع الخلايا المختلفة، وعن خليط معقد تحديات تحليلية للتحقيق في ابيجينومي في نوع خلية معينة. لمعالجة حالة الكروماتين الخاصة بنوع الخلية في طريقة على نطاق الجينوم، وضعنا مؤخرا جنبا إلى جنب الكروماتين إيمونوبريسيبيتيشن التسلسل (تشيب-Seq)، الذي يرتكز على تنقية انتقائية من الكروماتين بالبروتينات هستون المعلمة الأساسية من الخلية أنواع المصالح، متبوعاً بما يليها رقاقة والهدف من هذا البروتوكول هو الأخذ بممارسات أفضل لما يليها تشيب هذا الأسلوب يوفر أداة متعددة الاستخدامات للتحقيق ابيجينومي الخاصة بالانسجة في التعديلات هيستون المتنوعة ونموذج الكائنات الحية.
Introduction
تتكون أنسجة حيوانات من أنواع مختلفة من الخلايا. تنظيم الجينات في كل خلية يعرف نوع الخلية. التعديلات الكروماتين-الحمض النووي مثلايشن وهستون تعديل-تكمن وراء خصوصية خلية من نوع التعبير الجيني. وهكذا، قد تم المطلوب قياس التنظيم جينية في كل نوع من أنواع الخلايا، ولكن كان تحديا تقنيا.
للتحقيق في التخلق في نوع خلية معينة، كان جنبا إلى جنب الكروماتين إيمونوبريسيبيتيشن التسلسل (تشيب-Seq) وضعت مؤخرا (رقم 1)1. في تشيب، يعبر عن معلم حانمه الأساسية هيستون البروتين H2B من مروج الخلية-نوع معين. تسمح هذه الميزة لعزل تشروماتينس من الخلايا ذات الاهتمام، على الرغم من أن المواد يبدأ من خليط أنواع مختلفة من الخلايا. رقاقة-Seq التالية — تنقية الكروماتين عبر مارك هستون تعديل والجيل التالي تسلسل العميق لعزل الحمض النووي – يمكننا رصد حالة جينية من نوع الخلية المستهدفة بطريقة المجين على نطاق المنظومة.
باستخدام هذا الأسلوب، ونحن مؤخرا التحقيق تريميثيليشن الخاصة بالخلايا العصبية من هستون البروتين H3 على يسين 4 (H3K4me3) علامات. في تلك الدراسة، قمنا بتطوير تدق بماوس في أي ج-الميؤوس من شفائهم H2B معلم العلم وأعرب البروتين عند اقترانها بوساطة لجنة المساواة العرقية (Rosa26كاج فلوكسيد-السلطة الفلسطينية H2B العلم). بالعبور بماوس حيازة الجينات الشبكة (ER) Cre اندوبلاسميك تحت سيطرة المروج CamK2a، المستحث السطر الماوس التي يتم الحصول عليها العلم H2B في الخلايا العصبية نشطة على تاموكسيفن حقن (Camk2aالعلم H2B)1. بدءاً من دماغ السطر الماوس ثابتة، أجرينا Seq تشيب مع الأجسام المضادة H3K4me3. إذ كثيرا ما تتوافق مع علامات H3K4me3 إلى مناطق المروج، يمكن أن نكتشف مئات مرناس أعرب على وجه التحديد في الخلايا العصبية1.
هنا، يمكننا وصف أسلوب Seq تشيب نموذجية التي تغطي الخطوات من تشريح الأنسجة لبناء مكتبة (رقم 1). والهدف النهائي من هذا البروتوكول حصة لدينا أفضل الممارسات من أجل أداء Seq تشيب وتطبيق هذا الأسلوب مستقبلا للتعديلات هيستون وأنواع الخلايا الأخرى.
Protocol
وقد وافقت عليه شعبة السلامة بتبريد (H27-EP071) جميع الأساليب الموضحة هنا وأجريت مع اللوائح والمبادئ التوجيهية ذات الصلة.
1. تشريح الأنسجة
- تشريح أنسجة المصلحة إلى قطع صغيرة (تقريبا < 3 مم2) استخدام مقص الربيع الجميلة.
ملاحظة: أكبر الأنسجة شظايا يستغرق وقتاً أطول لتجميد، وسيحمل قطع صغيرة على كميات أكبر من المخزن المؤقت، والتي قد تؤثر على النتائج. - إضافة أجزاء تشريح الأنسجة إلى حاوية نظيفة مليئة بسائل النيتروجين وجمعها في أنابيب 2 مل (1 قطعة كل أنبوب) مملوءة بالنتروجين السائل.
ملاحظة: ينصح أنابيب مع سطح ماء لهذه الخطوة. - ضع الأنابيب في-80 درجة مئوية > 5 دقائق مع سقف مفتوح يتبخر النيتروجين السائل المتبقية.
ملاحظة: بعد إغلاق الغطاء، شظايا الأنسجة يمكن تخزينها في-80 درجة مئوية لمدة شهر واحد قبل الاستخدام.
2. تثبيت الخلية
ملاحظة: نحن نستخدم طاحونة مبردة مفيد لهذه الخطوة. ويمكن استخدام جهاز بديل.
- أنابيب المكان 2 مل من البروتين المنخفض ملزمة تحتوي على عينات الأنسجة على رف معدني جليد مبردة في نيتروجين سائل.
- ضع عيار 1.5 مل-الأنبوبة والبرد مع النتروجين السائل. وضعه على رفوف معدنية الجليد.
- فتح مل 2-الأنبوب ووضع رصاصة معدنية بريشيليد في الأنبوب. إغلاق الغطاء وتعيين مل 2-الأنابيب في حامل أنبوب من طاحونة المبردة مفيد وتراجع فورا صاحب الأنبوبة المجمعة في النتروجين السائل لمدة 1 دقيقة.
- إدراج حامل أنبوب المجمدة في الكاسيت الخارجي من طاحونة المبردة مفيد واهتز بشدة 60 مرة لمدة 30 ثانية.
- تفكيك حامل الأنبوب وإزالة رصاصة معدنية بريشيليد مكان 2 مل-الأنبوبة على عينة أكثر برودة في-20 درجة مئوية.
- ضع العينة برودة في-20 درجة مئوية لمدة 15 دقيقة لمنع تجميد مثبت في الخطوة التالية.
- إحضار العينة برودة على مقاعد البدلاء وفتح غطاء الأنبوب التنقيط ميليلتر 900 من 1% فورمالدهايد فورا في ذلك. وبعد بيبيتينج عدة مرات، نقل التعليق إلى جديد 2 مل-الأنبوب الذي يحتوي على 900 ميليلتر من 1% فورمالدهايد والإصلاح لمدة 10 دقيقة مع تناوب لطيف في حاضنة في 23 درجة مئوية.
تنبيه: الفورمالدهيد السامة والضارة. - للتوقف عن رد فعل التثبيت، إضافة 100 ميليلتر من 2.5 م جليكاين إلى كل أنبوب وجهاز الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية. تجاهل المادة طافية.
- أضف 1 مل من المثلج مخزنة الفوسفات المالحة (PBS)، أجهزة الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية، وتجاهل المادة طافية. كرر هذه الخطوة مرتين (يغسل 3 في المجموع).
ملاحظة: يمكن عينات ثابتة فلاش المجمدة بالنتروجين السائل والمخزنة في-80 درجة مئوية. - إضافة 500 ميليلتر من تحلل المخزن المؤقت 1 (حمض-(2-hydroxyethyl)-1-بيبيرازينيثانيسولفونيك 50 ملم 4 (حبيس)-كوه (درجة الحموضة 7.5)، 140 ملم كلوريد الصوديوم، 1 مم الإيثيلين حمض (يدتا) (pH 8.0)، 10% w/v والغليسيرول، 0.5% w/v NP-40، 0.25% w/v تريتون X-100، و 0.1 x مثبط البروتياز كوكتيل) لبيليه، بيبيت عدة مرات، واستدارة لمدة 10 دقائق في 4 درجات مئوية.
ملاحظة: ينبغي تصفيتها تحلل المخزن المؤقت 1 والمخزنة في 4 درجات مئوية قبل الاستخدام. - الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية وتجاهل المادة طافية.
- أضف 1 مل من تحلل المخزن المؤقت 2 (10 ملم تريس-HCl (pH 8.0)، 200 ملم كلوريد الصوديوم، 1 مم يدتا (pH 8.0)، 0.5 مم عطا، و 0.1 × مثبطات البروتياز كوكتيل) لبيليه، دوامة، واستدارة لمدة 10 دقائق في 4 درجات مئوية.
ملاحظة: ينبغي تصفيتها تحلل العازلة 2 والمخزنة في 4 درجات مئوية قبل الاستخدام. - الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية وتجاهل المادة طافية.
- إضافة 800 ميليلتر راديويمونوبريسيبيتيشن المقايسة (ريبا) المخزن المؤقت مع مثبط البروتياز 1 x كوكتيل لبيليه وريسوسبيند بيليه بيبيتينج.
- الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية وتجاهل المادة طافية.
- إضافة 500 ميليلتر من المخزن المؤقت ريبا مع مثبط البروتياز x 1 كوكتيل.
- الطرد المركزي لمدة 5 دقائق في 3,000 ز x عند 4 درجة مئوية وتجاهل المادة طافية.
- أضف 1 مل من المخزن المؤقت ريبا مع مثبط البروتياز x 1 كوكتيل. فورا للانتقال إلى الخطوة التالية.
3-الكروماتين القص
- نقل إلى أنبوب سونيكاتور وثم ضع الأنبوبة في أولتراسونيكاتور.
- إمالة الكروماتين بالإعدادات التالية: درجة الحرارة: 4 درجة مئوية؛ ذروة السلطة الحادث: 175 ث؛ عامل واجب: 10%؛ دورات/انفجار: 200؛ والوقت: 2,400 s.
- نقل العينة إلى 1.5 مل من البروتين ملزمة منخفضة أنبوب وجهاز الطرد المركزي لمدة 5 دقائق في 20,000 ز x عند 4 درجة مئوية.
- جمع المادة طافية في أنبوب ملزمة منخفضة بروتين جديد.
ملاحظة: العينة يمكن تجميد فلاش في النتروجين السائل وتخزينها في-80 درجة مئوية.
4-نوعية فحص للحمض النووي (عكس Crosslinking)
- ميكس 20 ميليلتر من وميليلتر 180 شرائح شطف المخزن المؤقت (10 ملم تريس-HCl (pH 8.0)، 300 مم كلوريد الصوديوم، 5 ملم يدتا (pH 8.0)، و 1% w/v دوديسيل كبريتات الصوديوم (SDS)) في أنبوب بوليميريز سلسلة من ردود فعل (PCR). احتضان العينة عند 65 درجة مئوية في cycler حرارية ح 6 مع غطاء cycler الحرارية مفتوحة. الحفاظ على غطاء فتح cycler الحرارية لتجنب الإفراط تمسخ.
ملاحظة: يجب تصفية المخزن المؤقت شطف رقاقة وتخزينها في درجة حرارة الغرفة قبل الاستعمال. ويمكن تمديد هذه الحضانة بين عشية وضحاها. - أضف 1 ميليلتر من 10 ملغ/مل رناسي، دوامة، واحتضان في 37 درجة مئوية لمدة 30 دقيقة.
- إضافة ميليلتر 6.5 من 15 ملغ/مل بروتيناز ك، ودوامه، واحتضان في 55 درجة مئوية لمدة 60 دقيقة.
- نقل رد فعل على أنبوب ربط منخفضا الحمض النووي، وإضافة 4 ميليلتر من الجليكوجين 5 ملغ/مل، ودوامه. ثم إضافة 200 ميليلتر من الكحول الفينول/كلوروفورم/إيسواميل (25:24:1) وجهاز الطرد المركزي لمدة 5 دقائق في 18,000 س ز في درجة حرارة الغرفة.
- نقل المادة طافية إلى أنبوب ملزمة منخفضا الحمض النووي جديد. إضافة 200 ميليلتر من المخزن المؤقت تريس-يدتا-كلوريد الصوديوم (10 ملم تريس-HCl (pH 8.0)، 1 يدتا (pH 8.0)، و 200 ملم كلوريد الصوديوم) المتبقية الكحول الفينول/كلوروفورم/إيسواميل الحل والطرد المركزي لمدة 5 دقائق في 18,000 س ز في درجة حرارة الغرفة. جمع المادة طافية وخلط مع المادة الأولى طافية.
- إضافة 900 ميليلتر من الإيثانول المثلج واحتضانها ح 1 في-20 درجة مئوية.
ملاحظة: يمكن تمديد هذه الحضانة إلى 4-6 ح. - أجهزة الطرد المركزي لمدة 30 دقيقة في 18,000 س ز في 4 درجات مئوية.
- تجاهل المادة طافية. أضف 1 مل من المثلج 75% إيثانول بيليه والطرد المركزي لمدة 30 دقيقة في 18,000 س ز في 4 درجات مئوية. كرر هذه الخطوة (يغسل اثنين في المجموع).
- تجاهل المادة طافية دقة وأيردري لمدة 1 دقيقة في درجة حرارة الغرفة.
- إضافة 50 ميليلتر من المخزن المؤقت تريس يدتا (TE) ويحل الحمض النووي بحضانة في درجة حرارة الغرفة لمدة 30 دقيقة، أو عند 4 درجة مئوية بين عشية وضحاها. تجنب فورتيكسينج أو بيبيتينج. إبقاء هذا الحمض النووي "المدخلات" واستخدامه لإعداد مكتبة إذا لزم الأمر.
- قياس تركيز الحمض النووي مع فلوروميتير وفقا لتعليمات الشركة المصنعة (انظر الجدول المواد)، والتحقق من توزيع حجم مع جهاز التفريد موائع جزيئية (انظر بيانات تمثيلية هو مبين في الشكل 2A)-10 استخدام الحرس الوطني أو أقل الحمض النووي لهذا التحليل.
ملاحظة: ينبغي أن تشكل أجزاء من 100 – 500 زوج قاعدي (bp) في 50% أو أكثر من الحمض النووي.
5-انجذاب تطهير بالأجسام المضادة العلم
ملاحظة: للعصبية تشيب-Seq، أنسجة المخ من الفئران 8 يتم استخدامها عادة عينة واحدة من تشيب Seq لإعداد 2 نانوغرام لتنقية الحمض النووي بتنقية المناعي جنبا إلى جنب (راجع الخطوة 7، 1). في أيدينا، 0.1 نانوغرام من الحمض النووي لا تزال تقدم عالية الجودة مكتبات الحمض النووي لرقاقة-Seq (كادوتا، غير منشورة). وهكذا، من الناحية النظرية، ماوس واحدة قد تكون كافية لأداء الخلايا العصبية تشيب--ما يليها المبلغ المطلوب ينبغي أن يكون الأمثل لنوع الأنسجة والخلايا للفائدة. لمراقبة إيمومونو-تنقية التجربة السلبية، ينبغي إعداد أنسجة المخ ليساتي من الفئران دون أي تعبير H2B-العلم واستخدامها.
- "الماصة؛" 200 ميليلتر من الخرز المغناطيسي مترافق للأغنام الماوس المضادة غلوبيولين مناعي G (IgG) في أنبوب ملزمة منخفضة بروتين.
- ضع الأنبوب على موقف مغناطيسية والانتظار لمدة 1 دقيقة للمادة طافية لتصبح واضحة. تجاهل المادة طافية، وإضافة 1 مل من برنامج تلفزيوني المثلج، ودوامه. كرر هذه الخطوة (يغسل اثنين في المجموع).
- إضافة 20 ميليلتر من الأجسام المضادة العلم 1 ملغ/مل وتدوير ح 5 في 4 درجات مئوية.
ملاحظة: يمكن تمديد هذه الحضانة بين عشية وضحاها. - أغسل حبات بعد "الخطوة 5، 2". ضع الخرز في أنبوب 15 مل.
- ريسوسبيند الخرز في ميليلتر 1110 ريبا المخزن المؤقت ثم قم بإضافة ميليلتر 900 10 × حجب الكاشف، 180 ميليلتر من 50 × الحل دينهارت، 10 ميليلتر من 100 × مثبطات البروتياز كوكتيل و 6.8 مل من إعدادها في الخطوة 3. تقسيم هذا الخليط في أنابيب ملزمة منخفضة البروتين 5. تدوير ح 6 في 4 درجات مئوية.
ملاحظة: يمكن تمديد هذه الحضانة بين عشية وضحاها. - ضع الأنبوب على موقف المغناطيسية والانتظار لمدة 1 دقيقة للمادة طافية لتصبح واضحة. تجاهل المادة طافية وإضافة 1 مل من المخزن المؤقت للمعهد الملكي، وتخلط برفق. كرر هذه الخطوة 5 مرات (6 يغسل في المجموع). تجمع جميع الخرز في أنبوب ملزمة منخفضة بروتين وحيد.
- إضافة 200 ميليلتر من "الرقائق العازلة شطف" وتدوير لمدة 15 دقيقة في درجة حرارة الغرفة. التحقق من جودة الحمض النووي كما هو موضح في الخطوة 4، 11 (انظر بيانات تمثيلية هو مبين في الشكل 2 وج). فورا للانتقال إلى الخطوة التالية.
6-انجذاب تطهير الأجسام المضادة H3K4me3 وعكس كروسلينكينج
ملاحظة: التالي حبة إعداد الخطوات (6، 1 – 6، 4) ينبغي أن يقوم قبل 5.7 خطوة.
- "الماصة؛" ميليلتر 40 من حبات مغناطيسية مترافق للأغنام الماوس المضادة مفتش في أنبوب 2 مل-بروتين ملزمة منخفضة.
- ضع الأنبوب على موقف المغناطيسية والانتظار لمدة 1 دقيقة للمادة طافية لتصبح واضحة. تجاهل المادة طافية وإضافة 1 مل من برنامج تلفزيوني المثلج وتخلط برفق. كرر هذه الخطوة (يغسل اثنين في المجموع).
- إضافة 4 ميليلتر من الأجسام المضادة-H3K4me3 1 ملغ/مل وتدوير ح 6 في 4 درجات مئوية.
- أغسل حبات بعد "الخطوة 6، 2". ضع الخرز في أنبوب 2 مل من بروتين ملزمة منخفضة.
- ريسوسبيند الخرز في ميليلتر 1558 ريبا المخزن المؤقت ثم قم بإضافة 200 ميليلتر من 10 × حجب كاشف ميليلتر 40 50 × الحل دينهارت، 2 ميليلتر من 100 × مثبطات البروتياز كوكتيل و 200 ميليلتر من النذرة إعدادها في "الخطوة 5، 7". تدوير بين عشية وضحاها في 4 درجات مئوية.
ملاحظة: الحزب الديمقراطي الصربي في المخزن المؤقت شطف رقاقة قد تضعف أكثر من 10 مرات في هذه الحضانة. - ضع الأنبوب على موقف المغناطيسية والانتظار لمدة 1 دقيقة للمادة طافية لتصبح واضحة. تجاهل المادة طافية وإضافة 1 مل من المخزن المؤقت للمعهد الملكي، وتخلط برفق. كرر هذه الخطوة 4 مرات (يغسل 5 في المجموع). بعد يغسل الماضي، نقل الخرز في أنبوب ملزمة منخفضا الحمض النووي، وتجاهل المادة طافية.
- إضافة 200 ميليلتر من "الرقائق العازلة شطف" وتدوير لمدة 15 دقيقة في درجة حرارة الغرفة. نقل النذرة بيندينجتوبي منخفضا الحمض النووي جديد.
ملاحظة: يمكن تخزين النذرة في-80 درجة مئوية. - اتبع الخطوة 4 ديكروسلينكينج من الحمض النووي. ريسوسبيند الحمض النووي المنقاة في 20 ميليلتر من المخزن المؤقت للشركة المصرية للاتصالات.
- التحقق من جودة الحمض النووي كما هو موضح في الخطوة 4.11 (انظر بيانات تمثيلية هو موضح في الشكل 2D). (اختياري) إجراء تحليل لتخصيب اليورانيوم قبل PCR كمي كما هو موضح سابقا2. عشرة إضعاف أو ينبغي التقيد بتخصيب أكبر.
7-مكتبة البناء
- لكل المدخلات وعينه "رقاقة الحمض النووي"، بحساب نسبة الحمض النووي في المنطقة 100 – 500-بي بي باستخدام وظيفة البرنامج تحليل لطاخة للجهاز الكهربي موائع جزيئية. تقدير حجم العينة المطلوبة للحصول على 2 نانوغرام من 100 – 500 bp الحمض النووي. استخدام 20 نانوغرام الدنا في نطاق 100-500-بي بي كالعينة "الإدخال".
- "الماصة؛" عينات من الحمض النووي في أنابيب PCR 8-قطاع وإضافة ح2س لتحقيق الحجم الإجمالي إلى 10 ميليلتر. (اختياري) إذا كان حجم الحمض النووي يتجاوز 10 ميليلتر، الارتقاء بشكل متناسب رد فعل الإصلاح نهاية التالية واختيار حجم.
- إعداد الإصلاح نهاية ميكس الرئيسية (انظر الجدول للمواد). إضافة 4 ميليلتر من مزيج الرئيسي الإصلاح نهاية للحمض النووي، واحتضان لمدة 30 دقيقة عند 20 درجة مئوية.
- إضافة 36 ميليلتر من 10 ملم تريس-Cl، الأس الهيدروجيني 8.5 و 0.6 x حجم (30 ميليلتر) الصلبة المرحلة تجميد عكسها (سبري) المغناطيسي الخرز واحتضان لمدة 5 دقائق في درجة حرارة الغرفة.
- ضع الأنبوب على الوقوف مغناطيسية والانتظار حتى يصبح من الواضح المادة طافية.
- نقل المادة طافية إلى أنبوب جديد وإضافة 1.2 x أحجام (60 ميليلتر) سبري المغناطيسي الخرز، واحتضان 5 دقائق في درجة حرارة الغرفة.
- ضع الأنبوب على الوقوف مغناطيسية والانتظار حتى يصبح من الواضح المادة طافية.
- أغسل حبات مرتين مع 200 ميليلتر من 80% EtOH، عقد الأنبوب لا يزال على المغناطيس.
- باختصار الطرد المركزي على أنبوب لجمع EtOH المتبقية في الجزء السفلي ووضعه مرة أخرى على المغناطيس. إزالة EtOH المتبقية دقة.
- اترك غطاء أنبوب مفتوحة لمدة 1-2 دقيقة للسماح EtOH تتبخر.
- إغلاق الغطاء وإبقاء الأنبوب على الجليد.
ملاحظة: الآن كنت قد حصلت على اختيار حجم الحمض النووي من 100 – 500 bp في الخرز. يمكن الاطلاع على مزيد من التفاصيل حول تحديد حجم مزدوج على موقع الويب الخاص بالشركة المصنعة (www.beckman.com). - إعداد ماجستير أ-تراجع مزيج (انظر الجدول للمواد).
- ريسوسبيند الخرز (من الخطوة 7.10) مع 10 ميليلتر من مزيج الرئيسي A-تراجع واحتضان لمدة 30 دقيقة في 30 درجة مئوية.
- إضافة إلى 1.8 x حجم (18 ميليلتر) من البولي إيثيلين غليكول (شماعة)/محلول كلوريد الصوديوم واحتضان لمدة 5 دقائق في درجة حرارة الغرفة.
- اتبع الخطوات 7.7 7.11 لتنقية الحمض النووي.
- إعداد مزيج ربط المخزن المؤقت (انظر الجدول للمواد).
- "الماصة؛" 8 ميليلتر من ربط المخزن المؤقت ميكس على الخرز (من 7.14 خطوة) وإضافة 1 ميليلتر من 1 ميكرومتر محول ريسوسبيند الخرز. استخدم 1 ميليلتر من 5 ميكرومتر المحول للحصول على نموذج الإدخال. النظر في استخدام محولات مختلفة لكل عينة للإرسال المتعدد.
- أضف 1 ميليلتر من "ليجاسى الحمض النووي" واحتضان لمدة 15 دقيقة في 20 درجة مئوية.
- إضافة المجلد x 1.0 (10 ميليلتر) حل الوتد/كلوريد الصوديوم، ريسوسبيند الخرز، واحتضان لمدة 5 دقائق في درجة حرارة الغرفة.
- اتبع الخطوات 7.7 7.10 لتنقية الحمض النووي.
- "الماصة؛" 25 ميليلتر من 10 ملم تريس-Cl، الأس الهيدروجيني 8.5 في الأنبوب والخرز ريسوسبيند.
- إضافة إلى حجم x 1.0 (25 ميليلتر) حل الوتد/كلوريد الصوديوم، واحتضان لمدة 5 دقائق في درجة حرارة الغرفة.
- اتبع الخطوات 7.7 7.10 لتنقية الحمض النووي.
- "الماصة؛" ميليلتر 11 مم 10 تريس-Cl، الأس الهيدروجيني 8.5 في الأنبوب والخرز ريسوسبيند.
- ضع الأنبوب على الوقوف مغناطيسية والانتظار حتى يصبح من الواضح المادة طافية.
- جمع المادة طافية في أنبوب بكر 8-قطاع جديد.
- إعداد مزيج الرئيسي PCR الوقت الحقيقي (انظر المواد للحصول على التفاصيل).
- "الماصة؛" ميليلتر 8.5 في الوقت الحقيقي PCR مزيج الرئيسي في صفيحة بكر (qPCR) كمية 384-جيدا وإضافة 1.5 ميليلتر محول متصلة الحمض النووي (من 7.26 الخطوة).
- "الماصة؛" 10 ميليلتر لكل معيار الفلورسنت في الآبار فارغة.
- إجراء PCR الوقت الحقيقي مع البرنامج التالي: (98 درجة مئوية ل 45 s) × 1 دورة، (98 درجة مئوية لمدة 15 ثانية، 60 درجة مئوية لمدة 30 s، 72 درجة مئوية لمدة 30 s) × 20 دورات، و (72 درجة مئوية مقابل 60 s) × 1 دورة.
- تحديد عدد دورات PCR اللازمة للوصول إلى كثافة الأسفار 2 القياسية الفلورسنت.
- إعداد سيد بكر مزيج (انظر الجدول للمواد).
- "الماصة؛" 11.5 ميليلتر من مزيج PCR الرئيسي في الدنا متصلة محول المتبقية (من الخطوة 7.26) وأداء بكر كما هو مبين في "الخطوة 7، 30" مع دورات بكر مصممة في 7.31 خطوة.
- إضافة إلى حجم x 1.0 (20 ميليلتر) حل الوتد/كلوريد الصوديوم واحتضان لمدة 5 دقائق في درجة حرارة الغرفة.
- اتبع الخطوات 7.7 7.10 لتنقية الحمض النووي.
- "الماصة؛" 20 ميليلتر من 10 ملم تريس-Cl، الأس الهيدروجيني 8.5 في الأنبوب والخرز ريسوسبيند.
- ضع الأنبوب على الوقوف مغناطيسية والانتظار حتى يصبح من الواضح المادة طافية.
- جمع المادة طافية في أنبوب 1.5 مل-الحمض النووي منخفض ملزمة جديدة.
- التحقق من جودة المكتبة كما هو موضح في الخطوة 4، 11 (انظر بيانات تمثيلية هو مبين في الشكل 2 هاء وواو). استخدم 1 ميليلتر من عينات الحمض النووي مكتبة. (اختياري) إجراء تحليل لتخصيب اليورانيوم قبل قبكر كما هو موضح سابقا 2. عشرة إضعاف أو ينبغي التقيد بتخصيب أكبر.
8-تسلسل
- تسلسل المكتبات في التسلسل الجيل التالي (انظر بيانات تمثيلية هو موضح في الشكل 3).
ملاحظة: تسلسل عمق كاف لتحليل البيانات سوف تختلف تبعاً لحجم جينوم الكائن الحي3. لحقوق الإنسان، والماوس، نوصي بالحصول على مالا يقل عن 20 مليون واحد في نهاية القراءة. وفقا لحصيلة ما يلي، الإرسال المتعدد قد تكون فعالة من حيث التكلفة.
النتائج
هنا، نحن تصف تشريح الأنسجة، تثبيت، وتحلل الخلية، تنقية جنبا إلى جنب من الكروماتين، وإعداد مكتبة الحمض النووي للتعاقب الجيل القادم. أثناء الإجراءات، يمكن اختبار واحد نوعية الحمض النووي، الذي هو مفتاح لتسلسل النجاح، في خطوات متعددة (الشكل 2). منذ نوكليوسومي واحد عادة محاط ب الحمض النووي bp 147 4، يجب أن لا أقصر من أن الحجم الحمض النووي المنفصمة. فور ultrasonication، عزل الحمض النووي وتشغيلها على جهاز التفريد موائع جزيئية (الشكل 2A). وعلى الرغم من أفضل جهودنا لتحسين نطاق الحجم، كان يزال عدد سكان من الحمض النووي (حوالي 2 kbp) التي ظلت أونشيريد. في هذه الخطوة، ينبغي أن يكون الجزء من الحمض النووي تتراوح ما بين 100-500 bp 50% أو أكثر من السكان. بعد انجذاب تطهير الأجسام المضادة العلم (الشكل 2)، ومن ثم الأجسام المضادة H3K4me3 (الشكل 2D)، تم فحص جودة عزل الحمض النووي مرة أخرى باستخدام آلة التفريد موائع جزيئية، وكمية تتراوح بين الحمض النووي وقدرت شركة bp 100-500. على الرغم من أن كثيرا ما لاحظنا انحياز الشظايا kbp 2 في هذه الخطوة أقوى، ضعف اختيار حجم الخرز المغناطيسي سبري إزالة هذا الكسر.
أكدت خصوصية المناعة-تنقية الحمض النووي في علم H2B مع عينات المراقبة السلبية، في الدماغ التي ليساتي من الفئران دون علم H2B استخدمت التعبير (الشكل 2). ونحن عادة الكشف عن مبالغ ضئيلة من الحمض النووي من عينات المراقبة السلبية.
في أعقاب تراجع A، ربط المحول، وبكر، وتم التحقق من نوعية المكتبة التسلسل (الشكل 2E). وبشكل نموذجي, bp 250-600 الحمض النووي تم الحصول عليها. رقاقة العادية الناجحة وتشيب اتضحت بتحليل تخصيب اليورانيوم مع قبكر2 باستخدام كبسولة تفجير استهدفت منطقة المروج الجينات جابده (الشكل 2 واو).
وتلا ممثل التوزيعات على طول الجينات العصبية (Camk2a و Slc17a7 و Gria1) بمتصفح الجينوم وترد في الشكل 3. وقد لوحظ في إثراء ما يلي في نهاية 5 ' الجينات بالخلايا العصبية تشيب-Seq على الدماغ كله تشيب-Seq.

رقم 1: التمثيل التخطيطي لما يليها تشيب وقد تم تعديل هذا الرقم من ميتو، وآخرون. 1- الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
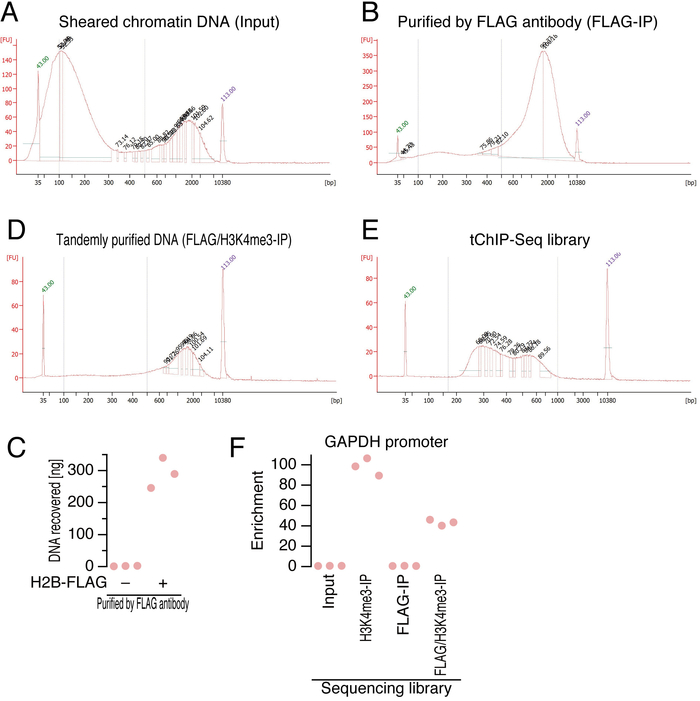
رقم 2: فحص الحمض النووي الجودة- (أ وب، د وه) اليكتروفيروجرام التي تم الحصول عليها باستخدام جهاز التفريد موائع جزيئية للونين المنفصمة الحمض النووي (A)، الحمض النووي تنقية مع الأجسام المضادة العلم (ب) ثم الأجسام المضادة H3K4me3 (ج)، والمكتبة النهائية (د). قمم وأبرزت مع أرقام خضراء والأرجواني تمثل معايير المساحة الداخلية. تشير الخطوط المتقطعة الأزرق إلى مناطق حجم الحمض النووي لتحليل المتلقين للمعلومات. فو: fluorescence الوحدات.
(ج) استرداد كمية الحمض النووي بتنقية المناعة المضادة العلم. وتمثل كل نقطة تكرار مستقل. (و) تحليل الإثراء باستهداف جابده qPCR. وتمثل كل نقطة تكرار مستقل. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-

الشكل 3: قراءة توزيعات في الخلايا العصبية تشيب--ما بعدها (أ-ج) قراءة التوزيعات على طول الممثل العصبية تظهر الجينات في الخلايا العصبية تشيب-Seq والتحكم الكامل للعقل تشيب-Seq. دورة في الدقيقة: يقرأ كل القراءات مليون. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
Discussion
وكان لدينا بروتوكول الأمثل للخلايا العصبية للدماغ الماوس، وفيه التعبير عن معلم العلم H2B هو الناجم عن الحقن بعقار تاموكسيفين. المروجين المستخدمة للتعبير H2B وانطلاق مواد الأنسجة وكمية الأنسجة معلمات محورية لنجاح تشيب--ما يليها وبالتالي، ينبغي النظر في الاستفادة المثلى من هذه العوامل لكل خلية نوع من الفائدة.
خطوة حاسمة بين الإجراءات المستخدمة في هذا البروتوكول هو الحمض النووي القص لتحقيق طول الكروماتين من 100-500 bp5. بشكل عام، هو التحدي بتوحيد هذه الخطوة ultrasonication حيث أنه يعتمد إلى حد كبير على مجموعة متنوعة من العوامل؛ شروط التثبيت، والأنسجة المستخدمة، والمعدات المستخدمة الترا--سونيكيشن، ومعدات الإعداد، من بين أمور أخرى. ولذلك، سيلزم الأمثل التجربة والخطأ لهذه الخطوة قص الحمض النووي للتجارب الفردية. بدلاً من الترا-سونيكيشن، معاملة نوكلاس ميكروكوككال يجب أن تكون خياراً لعزل واحد الحجم nucleosome الحمض النووي، كما تستخدم في الأسلوب رقاقة ساذجة6. على الرغم من أن توزيع حجم الحمض النووي مثالي، المنفصمة متجانسة السلطات الوطنية المعينة قد تظهر على السكان متعددة، كما هو مبين في الشكل 2 ألف. اختيار حجم مزدوج سبري الخرز المغناطيسي، كما هو موضح أعلاه، يساعد على إزالة أطول الحجم شظايا من الحمض النووي.
عادة، يتطلب إعداد مكتبة 1 نانوغرام من 100-500 bp الحمض النووي. وبناء على هذا الشرط، نحن الارتقاء بالمواد الأولية من أنسجة المخ للخلايا العصبية. ومع ذلك، قد يكون هذا الإجراء تحديا لعينات صغيرة للغاية من أنواع نادرة من الخلية. وفي مثل هذه الحالات، قد تكون رقاقة الوثائق، الذي يستند إلى ترانسبوساسي Tn5 بدلاً من ليجاسى ويتطلب أقل المواد7، بديل أفضل.
ومع ذلك، يقتصر هذا البروتوكول الحالي لأنواع الخلايا الرئيسية في المواد. في أيدينا، قدمت شغل 10% من الخلايا المستهدفة في السكان الخلية الأصلي تنقية عادلة من الكروماتين من خلايا الهدف1. بيد إذا كانت تستهدف أكثر أنواع الخلايا نادرة، ثم التحسين أدق من إيمونوبريسيبيتيشن بالعلامة حانمه سوف يكون له ما يبرره للتمييز بين الحمض النووي المعزولة من الدنا الملوثة من الخلايا غير المستهدفة. لأفضل الممارسات لتحليل البيانات، نوصي بشدة بمقارنة البيانات لتحليل Seq تشيب التحكم من الأنسجة كلها تستخدم للبدء في التجربة. منذ وجدنا تحيز كبير من مناشئ أعرب العلم H2B البروتين، ينبغي التحليل مقارنة وتحليلها بواسطة تخصيب (أو نضوب) إلى الخلفية1. لعنصر التحكم هذا، نحن في الواقع إنشاء خط ماوس مع العلم H2B أعرب أوبيكويتوسلي (روزاالعلم H2B) وأداء تشيب Seq1. بتفسير أكثر تفصيلاً للبيانات قد تم مناقشتها سابقا1.
استناداً إلى تحليلنا، H3K4me3 تشيب Seq من الخلايا العصبية المقدمة في الكشف عن الجينات الخاصة بالخلايا العصبية المعروفة من تسلسل الحمض النووي الريبي من نظام مراقبة الأصول الميدانية فرز الخلايا العصبية مماثلة و/أو أفضل8 وترجمة الريبوسوم انجذاب تطهير (TRAP)-Seq9. على سبيل المثال، تم انتشال الجينات المعروفة إكسون/تغصن-مترجمة كفاءة استخدام الخلايا العصبية تشيب Seq1. بشكل ملحوظ، لدينا تشيب-Seq ليست محدودة بالنسبة لعلامة هيستون المرتبطة بالمروج لكن ينطبق أيضا على التعديلات هيستون الأخرى المختلفة10 إذا كانت تتوفر الأجسام المضادة. وبالإضافة إلى ذلك، يمكن استخدام استراتيجية عزل الكروماتين بالبروتينات هستون المعلمة الأساسية في الكائنات الحية الأخرى في النموذج. على الرغم من أن هنا استخدمنا حانمه العلم للعلامة البروتين هيستون الأساسية، يمكن تطبيق العلامات الأخرى. منذ التثبيت فورمالدهايد للحمض النووي يحدث الأكثر شيوعاً في المخلفات يسين البروتين11، ينبغي تجنب علامة مع يسين كثيرة للقضاء على التحيز بعلامات حانمه. وبالتالي، ينبغي أن يكون النهج Seq تشيب تنوعاً في أنسجة متنوعة على امتداد العديد من أنواع من السياقات في دراسات جينية.
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
ونشكر جميع أعضاء المختبر ايواساكي لقراءة نقدية من المخطوطة. هذا العمل كان تدعمها جزئيا معونات "البحث العلمي" في "مجالات مبتكرة" (26113005 # للتعطيل و JP17H05679 للصك القانوني)؛ معونات للعلماء الشباب (أ) (JP17H04998 للصك القانوني) من وزارة التربية والتعليم والعلوم، والرياضة، والثقافة من اليابان (يأمرون)؛ والمشاريع بيونيرينج "تطور الهاتف الخلوي"، وجميع المشاريع بتبريد "المرض وابيجينومي" من بتبريد (للتعطيل والصك القانوني).
Materials
| Name | Company | Catalog Number | Comments |
| Protein LoBind tube, 2 mL | Eppendorf | No. 0030108132 | For cell lysis |
| Protein LoBind tube, 1.5 mL | Eppendorf | No. 0030108116 | For ChIP and library preparation |
| DNA LoBind tube, 1.5 mL | Eppendorf | No. 0030108051 | For ChIP and library preparation |
| 8-strip PCR tube | BIO-BIK | 3247-00 | For ChIP and library preparation |
| SK Mill | TOKKEN | SK-200 | Handy cryogenic grinder to make cell powder for fixation |
| Metal bullet | TOKKEN | SK-100-DLC10 | Accessory of SK Mill |
| 2 mL stainless steel tube | TOKKEN | TK-AM5-SUS | An option for cell lysis |
| 2 mL stainless steel tube holder | TOKKEN | SK-100-TL | An option for cell lysis |
| 16% formaldehyde (w/v), methanol-free | Pierce | 28906 | To fix cells. Prepare 1% solution before use. |
| Glycine | Nacalai Tesque | 17109-35 | Prepare 2.5 M stock |
| D-PBS (-)(1x) | Nacalai Tesque | 14249-24 | For washing lysate and purified DNA |
| HEPES | Nacalai Tesque | 02443-05 | For Lysis buffer 1. Prepare 1 M, pH 7.5 stock. |
| 5 M NaCl, molecular biology grade | Nacalai Tesque | 06900-14 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| 0.5 M EDTA, molecular biology grade | Wako Pure Chemical Industries, Ltd. | 311-90075 | For Lysis buffer 1, Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer |
| Glycerol | Wako Pure Chemical Industries, Ltd. | 072-04945 | For lysis buffer 1 |
| NP-40 | Nacalai Tesque | 25223-75 | For lysis buffer 1 |
| Triton X-100, molecular biology grade | Nacalai Tesque | 12967-32 | For Lysis buffer 1 |
| Tris | Nacalai Tesque | 35406-91 | For Lysis buffer 2, ChIP Elution Buffer, and Tris-EDTA-NaCl Buffer. Prepare 1 M, pH 8.0 stock. |
| 0.1 M EGTA pH neutral | Nacalai Tesque | 08947-35 | For Lysis Buffer 2 |
| Protease inhibitor cocktail (100x) | Nacalai Tesque | 25955-24 | To block degradation of protein |
| RIPA buffer | Thermo Fisher Scientific | 89900 | For cell lysis and washing |
| milliTUBE 1 mL AFA Fiber | Covaris | 520130 | Sonicator tube. Accessory of Focused-ultrasonicator |
| Focused-ultrasonicator | Covaris | S220 or E220 | To digest DNA into adequate size for ChIP-Seq |
| UltraPure 10% SDS | Thermo Fisher Scientific | 15553-027 | For ChIP Elution Buffer |
| RNase A | Nacalai Tesque | 30141-14 | To purify DNA from lysate |
| Proteinase K, recombinant, PCR Grade | Sigma-Aldrich | 3115887001 | To purify DNA from lysate |
| Ethanol | Wako Pure Chemical Industries, Ltd. | 054-07225 | Make 70% solution |
| Monoclonal anti-FLAG M2 antibody produced in mouse | Sigma-Aldrich | F1804 | To purify chromatin expressed in cells of interest |
| Dynabead M-280 Sheep Anti-Mouse IgG | Thermo Fisher Scientific | 11201D | This can be used for anti-FLAG IP and anti-H3K4me3 IP |
| Anti-tri-methyl histone H3 (K4), mouse monoclonal antibody | Wako Pure Chemical Industries, Ltd. | 301-34811 | Any other antibody that works for ChIP analysis will work |
| 10x Blocking Reagent | Sigma-Aldrich | 11096176001 | For blocking during affinity purification |
| Denhardt’s solution | Nacalai Tesque | 10727-74 | For blocking during affinity purification |
| Glycogen (5 mg/ml) | Thermo Fisher Scientific | AM9510 | To purify DNA from lysate |
| Qubit 2.0 Fluorometer | Thermo Fisher Scientific | Q32866 | For quantification of isolated DNA |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | For quantification of isolated DNA |
| 0.5 mL tube | Axygen | 10011-830 | For quantification by Qubit |
| Phenol/chloroform/isoamyl alcohol (25:24:1) | Nacalai Tesque | 25970-56 | To purify DNA from lysate |
| AMPure XP beads | Beckman Coulter | A63881 | SPRI magnetic beads for library preparation |
| Metal ice rack | Funakoshi | IR-1 | To keep the cell lysate frozen |
| Sample Cooler | New England Biolabs | T7771S | Helps fix cells with minimal damage |
| 2100 Bioanalyzer | Agilent Technologies | G2939BA | To check the quality of isolated DNA fragments. Another fragment analyzer can be used. |
| Bioanalyzer 2100 Expert Software | Agilent Technologies | G2946CA | Supplied with the Bioanalyzer |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | To check the quality of the isolated DNA fragments |
| KAPA LTP Library Preparation Kit | Roche | 07961898001 | Supplied with 10x KAPA End Repair Buffer, KAPA End Repair Enzyme Mix, KAPA A-Tailing Buffer, KAPA A-Tailing Enzyme, KAPA Ligation Buffer, KAPA DNA Ligase, and PEG/NaCl solution |
| NEXTflex DNA Barcodes | BIOO Scientific | NOVA-514101 | Adapter for library preparation. Supplied with DNA Barcode Adapters and Primer Mix. |
| KAPA Real-Time Library Amplification Kit | Roche | 07959028001 | Supplied with 2x KAPA HiFi HS real-time PCR Master Mix, PCR Primer Mix, and Fluorescent Standards |
| 2x KAPA HiFi HotStart ReadyMix | Roche | KM2602 | For library preparation. Additionally, this enzyme may be required for the KAPA Real-Time Library Amplification Kit |
| Buffer EB | Qiagen | 19086 | 10 mM Tris-Cl, pH 8.5 for elution of DNA |
| 386-well qPCR plate | Thermo Fisher Scientific | 4309849 | For real-time PCR |
| QuantStudio 7 Flex Real-Time PCR System | Thermo Fisher Scientific | 4485701 | To quantify DNA |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 4311971 | For real-time PCR |
| MicroAmp Clear Adhesive Film | Thermo Fisher Scientific | 4306311 | For plate sealing |
| End-repair master mix | Combine 1.4 µL of 10x KAPA End Repair Buffer, 1 µL of KAPA End Repair Enzyme Mix, and 1.6 µL of H2O | ||
| A-taling master mix | Combine 1 µL of KAPA A-Tailing Buffer, 0.6 µL of KAPA A-Tailing Enzyme, and 8.4 µL of H2O | ||
| Ligation buffer mix | Combine 2 µL of KAPA ligation buffer and 6 µL of H2O | ||
| Real-time PCR master mix | Combine 5 µL of 2x KAPA HiFi HS real-time PCR Master Mix, 0.35 µL of PCR Primer Mix (10 µM each of forward primer AATGATACGGCGACCACCGAG and reverse primer CAAGCAGAAGACGGCATACGAG), and 3.15 µL of H2O | ||
| PCR master mix | Combine 10 µL of 2x KAPA HiFi Ready Mix, 0.9 µL of PCR Primer Mix, and 0.6 µL of H2O | ||
| Integrative Genomics Viewer | Broad Institute | IGV_2.3.88 | Genome browser to visualize sequencing data |
| DNA olgionucleotide: 5′-GCCTACGCAGGTCTTGCTGAC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| DNA olgionucleotide: 5′-CGAGCGCTGACCTTGAGGTC-3′ | Eurofins Genomics | A primer to amplify the promoter region of GAPDH | |
| SYBR Premix Ex Taq | Takara | RR420L | To quantify the DNA corresponding to the GADPH promoter region |
| Thermal Cycler Dice | Takara | TP870 | To quantify the DNA corresponding to the GADPH promoter region |
References
- Mito, M., et al. Cell type-specific survey of epigenetic modifications by tandem chromatin immunoprecipitation sequencing. Scientific Reports. 8 (1), 1143 (2018).
- Kadota, M., et al. CTCF binding landscape in jawless fish with reference to Hox cluster evolution. Scientific Reports. 7 (1), 4957 (2017).
- Jung, Y. L., et al. Impact of sequencing depth in ChIP-seq experiments. Nucleic Acids Research. 42 (9), (2014).
- Becker, P. B., Workman, J. L. Nucleosome remodeling and epigenetics. Cold Spring Harbor Perspectives in Biology. 5 (9), a017905 (2013).
- Kidder, B. L., Zhao, K. Efficient library preparation for next-generation sequencing analysis of genome-wide epigenetic and transcriptional landscapes in embryonic stem cells. Methods in Molecular Biology. , 3-20 (2014).
- Gilfillan, G. D., et al. Limitations and possibilities of low cell number ChIP-seq. BMC Genomics. 13, 645 (2012).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Zhang, Y., et al. An RNA-sequencing transcriptome and splicing database of glia, neurons, and vascular cells of the cerebral cortex. Journal of Neuroscience. 34 (36), 11929-11947 (2014).
- Hornstein, N., et al. Ligation-free ribosome profiling of cell type-specific translation in the brain. Genome Biology. 17 (1), 149 (2016).
- Zhao, Y., Garcia, B. A. Comprehensive catalog of currently documented histone modifications. Cold Spring Harbor Perspectives in Biology. 7 (9), a025064 (2015).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: a tool for the study of chromatin complexes. The Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved