Method Article
Количественная флуоресценция в ситу Гибридизации (FISH) и иммунофлуоресценции (ИФ) специфических генных продуктов в KSHV-зараженных клетках
В этой статье
Резюме
Мы описываем протокол с использованием флуоресценции на месте гибридизации (FISH) для визуализации нескольких герпесвиральных РНК в литически инфицированных человеческих клеток, либо в подвеске или приверженцев. Этот протокол включает в себя количественную оценку флуоресценции, производящей нуклеоцитоплазмическое соотношение и может быть расширен для одновременной визуализации принимающих и вирусных белков с иммунофлуоресценцией (ИФ).
Аннотация
Механистический анализ приходит из тщательного изучения и количественной оценки конкретных РНК и белков. Относительное расположение этих биомолекул по всей клетке в определенное время может быть захвачено с флуоресценцией на месте гибридизации (FISH) и иммунофлуоресценции (IF). Во время литической герпесвирусной инфекции вирус захватывает клетку-хозяина, чтобы преимущественно выражать вирусные гены, вызывая изменения в морфологии клеток и поведении биомолекул. Литические деятельности сосредоточены на ядерных заводах, называется вирусных репликации отсеков, которые заметны только с FISH и IF. Здесь мы описываем адаптируемый протокол РНК FISH и IF методы для Саркомы Апоши связанных герпесвирус (KSHV) инфицированных клеток, как приверженцев и в подвеске. Метод включает в себя шаги по разработке специфических антисмысловых олигонуклеотидов, двойной РНК FISH, РНК FISH с IF, а также количественные расчеты интенсивности флуоресценции. Этот протокол был успешно применен к нескольким типам клеток, неинфицированных клеток, скрытых клеток, литических клеток, временных курсов и клеток, обработанных ингибиторами для анализа пространственно-временной деятельности конкретных РНК и белков как от хозяина человека, так и КШВ.
Введение
В их литической (активной) фазе, герпесвирусы захватывают клетку-хозяина, вызывая изменения в морфологии клеток и локализации биологических молекул, для производства вирионов. Основой операций является ядро, где двухцепочечный геном ДНК вирусного реплицируется и упакован в белковую оболочку, называемую капсидом1. Для начала вирус выражает свои собственные белки, угон принимающей машины и предотвращения экспрессии несущественных генов хозяина, процесс, называемый эффект выключения хозяина. Большая часть этой деятельности локализована на конкретные 4 ",6-диамидино-2-фенилиндинол (DAPI) свободных ядерных регионов, называемых вирусных репликации отсеков, состоящий из как принимающих и вирусных белков, РНК, и вирусной ДНК2. Ячейка капитально отремонтирована, чтобы обеспечить пространство и ресурсы для репликации отсеков и, таким образом, сборки вирусных капсидов. После того, как капсид выходит из ядра, как капсид окутан цитоплазмой для производства мембраны связанных вирусной частицы, также известный как вирион, неясно. Понимание локализации и пространственных сдвигов как хоста, так и вирусных биомолекул во время литической фазы обеспечивает более глубокое механистическое понимание расположения отсека репликации, эффекта выключения хозяина, пути выхода из virиона и других процессов, связанных с герпесвирусной инфекцией и репликацией.
В настоящее время лучшим методом обнаружения и изучения этих изменений является визуализация белков и РНК в инфицированных клетках с иммунофлуоресценцией (ИФ) и флуоресцентной гибридизации на месте (FISH), соответственно. Использование временного курса с помощью этих методов показывает локализацию биомолекул в ключевых точках литической фазы или просто пространственно-временных данных. FISH и IF дополняют другие биохимические методы, такие как ингибирование клеточного процесса (например, ингибирование репликации вирусной ДНК), RT-qPCR (цепная реакция полимеразы в реальном времени), секвенирование РНК, северные помарки, масс-спектрометрия, западная блоттинг и анализ производства вирусной ДНК, который может обеспечить более глобальную картину клеточной деятельности.
Мы разработали стратегии RNA FISH для изучения продуктов РНК из конкретных генов и вычислительного анализа, который количественно вычисляет нуклеоцитоплазмическое соотношение конкретного генного продукта. Подготовка образца, измененная из предыдущих публикаций Steitz и коллегами3,4,относительно проста и может быть использована как для приверженцев, так и для подвесных клеток. Протокол также адаптируется для одновременного использования нескольких стратегий РНК FISH (двойная РНК FISH) или RNA FISH со стратегиями IF. Разработка конкретной стратегии FISH является сложной задачей, но предложения по улучшению успеха изложены. Анализ данных, описанный здесь, является количественным, если используются флуоресцентные бусы и сильные маркеры границ отсека, и дает дополнительное представление о микрографах, понимание, которое устраняет предвзятость наблюдения. Подробный протокол предназначен как для скрытых, так и для литических клеток, инфицированных саркома-ассоциированным герпесвирусом Капоши (KSHV) и может быть использован с неинфицированными клетками или клетками, инфицированными другими герпесвирусами5. Методы количественной оценки применимы к исследованиям по нуклеоцитоплазматическим сдвигам или перелокализации между субклеточными отсеками в большинстве клеток.
протокол
1. Дизайн флуоресценции на месте (FISH) анти-чувство олигонуклеотидов для обнаружения конкретных герпесвиральных стенограммы
- Выберите от 25 до 40 nt сегментов из последовательности РНК интерес и конвертировать в анти-чувство. Успешная стратегия FISH может содержать от одного до десяти или более различных анти-смысл олигонуклеотидов. При выборе последовательностей следует учитывать следующее:
- Если РНК интереса содержит уникальный регион повторения, а затем извлечь выгоду из этой функции и разработать анти-чувство олигонуклеотида для целевой повторяют последовательность.
ПРИМЕЧАНИЕ: Tycowski и коллеги5 представить пример этой стратегии с rhesus rhadinovirus (RRV) полиаденелаповой ядерной (PAN) РНК. - Если РНК, интересующаяся содержит известный белково-связывающий участок или структуру стволовой петли, проектируйте олигонуклеотиды, которые избегают этих регионов.
- В зависимости от целей экспериментов, рассмотреть интронную последовательность и следует ли разрабатывать анти-чувство олигонуклеотида для него.
- Если РНК интереса содержит уникальный регион повторения, а затем извлечь выгоду из этой функции и разработать анти-чувство олигонуклеотида для целевой повторяют последовательность.
- Выполните простой вычислительный анализ выбранных антисмысловых последовательностей для обеспечения связывающей специфичности и уменьшения агрегации античувствеонного олигонуклеотида.
- Последовательности должны быть примерно 50% GC-богатых (высокое содержание гуанина и цитозина) и имеют температуру плавления в диапазоне от 60 до 70 градусов по Цельсию.
- Используйте инструмент анализа последовательности, чтобы выбрать последовательности, которые не самодимеризируют сяочку или не образуют шпильки с температурой плавления выше 37 градусов по Цельсию, температурой гибридизации.
- Выполните NCBI BLASTn (Национальный центр биотехнологии информации Основные местные выравнивания Search Tool для нуклеотидов выравнивания) поиск выбранных последовательностей против как хозяина и вирусных транскриптомов, используя "несколько аналогичных" настройки. Этот поиск позволит определить уникальные анти-чувство олигонуклеотидов, которые, вероятно, не связываются с другими хостом или вирусных стенограмм.
ПРИМЕЧАНИЕ: Если транскриптомы недоступны, выполните поиск BLAST с геномными последовательностями. Идеально, если поиск выполняется по последовательностям от вируса, изолированного от инфицированных клеток, используемых в эксперименте, потому что дикие штаммы, как правило, диверсифицируют и содержат комбинацию последовательностей из различных лабораторных штаммов.
- Закажите очищенные ДНК-олигонуклеотиды, соответствующие антисмысленной последовательности, которая была проверена вычислительно, чтобы быть уникальной и, вероятно, связываться с целевой РНК. Никаких специальных модификаций в олигонуклеотиды вводить не нужно.
- Испытайте разработанные антисмысловые олигонуклеотиды на обязательную специфичность FISH и Northern blot.
- Используя неинфицированную клеточную линию от одного и того же вида-хозяина (например, 293T) и в идеале одного и того же типа клеток, проведите трансфекцию с плазмидом, выражающий интерес РНК со стороны надежного промотора (CMV, цитомегаловирус) и с пустым вектором (например, pcDNA3). Используйте положительный контроль для трансфекции, такие как со-трансфекция с ГФП (зеленый флуоресцентный белок) плазмид (например, pmaxGFP) или вектор, содержащий GFP.
ПРИМЕЧАНИЕ: Важно, чтобы избежать клеточных линий, которые были увековечены с помощью вируса эппесина-Барра (EBV), поскольку Есть последовательность сходства между герпесвирусами. - Выполните FISH, как описано в разделе 3 на обоих наборах клеток с анти-чувство олигонуклеотидов, как описано в этом протоколе. Вывести успешных кандидатов, сравнивая FISH эксперименты с отдельными, пары, или наборы анти-чувство олигонуклеотидов. Используйте положительный контроль для протокола FISH, таких как U2 snRNA (малая ядерная РНК) FISH, присутствующих на 500000 копий на ядро клетки человека6 (Таблица 1).
- Флуоресцентный сигнал должен быть специфическим и сильным в клетке, содержащей РНК интереса. Дизайн дополнительных анти-чувство олигонуклеотидов для укрепления сигнала и удалить анти-чувство олигонуклеотиды, которые связывают неспецифически от рассмотрения. Сила сигнала должна быть выше фона и автофлюоресценции.
- Испытание связывающей специфичности по Северному помету.
- Используя неинфицированную клеточную линию от одного и того же вида-хозяина (например, 293T) и в идеале одного и того же типа клеток, проведите трансфекцию с плазмидом, выражающий интерес РНК со стороны надежного промотора (CMV, цитомегаловирус) и с пустым вектором (например, pcDNA3). Используйте положительный контроль для трансфекции, такие как со-трансфекция с ГФП (зеленый флуоресцентный белок) плазмид (например, pmaxGFP) или вектор, содержащий GFP.
2. Олигонуклеотид и клеточная подготовка
- Следуя инструкциям производителя, используйте терминальной трансферазы для обозначения антисмысловых олигонуклеотидов диоксигенином (DIG)-DUTP или, если сильно связывающими, непосредственно с флуоресцентным нуклеотидом, таким как Alexa Fluor 594-5-dUTP. После маркировки дополнительная очистка не требуется. Магазин помечены олигонуклеотиды на -20 градусов по Цельсию до нескольких лет и в фольге, если непосредственно помечены для предотвращения фотоотбелекирования.
ВНИМАНИЕ: Решение для маркировки содержит токсичный материал, калий какодилат. Ручка маркировки реакции с перчатками.
ПРИМЕЧАНИЕ: Для сохранения ресурсов, несколько различных анти-чувство олигонуклеотиды могут быть помечены в одной реакции. Этот протокол использует 3'конец маркировки. Внутренняя маркировка является сложной задачей, поскольку химическая группа (например, DIG или фторфор) попадается или не может войти в активный участок полимераза ДНК. Эксперимент с двумя различными РНК может быть выполнен с использованием прямо йогонуклеотидов и анти-DIG иммунофлуоресценции с различными флюорофорами (например, FITC (флуоресцеин) или Alexa Fluor 488 с Alexa Fluor 594). - Придерживайтесь ячеек к восьмикамерным горок.
ПРИМЕЧАНИЕ: Восьмикамерные слайды позволяют несколько одновременных экспериментов при минимизации драгоценных ресурсов, таких как антитела. Альтернативой восьмикамерным слайдам является шестикомнатная пластина культуры тканей со стандартными крышками (22 мм х 22 мм), которые являются стерильными. Аналогичное расположение возможно с круговыми крышками и 24-хорошо пластины культуры ткани. Для обоих увеличить объемы, упомянутые в этом протоколе, на 10-15 x (например, 1,75 мл решения гибридизации) и 4x (например, 600 л решения гибридизации) соответственно.- Для приверженных литические клетки, используйте 1x трипсин / PBS на 37 КС и 5% CO2 в течение 10 минут, чтобы приостановить клетки и разбавить до 60% выпуклости.
ПРИМЕЧАНИЕ: Приверженные клеточные линии, используемые в экспериментах FISH, включали 293T, iSLK.2197и iSLK-BAC36 ячеек8. - Нанесите 200 л клеточной подвески на каждую камеру стерильных восьмикамерных слайдов и допускайте ростсемян в течение 12-24 ч при 37 градусах Цельсия и 5% CO 2. Отрегулируйте по мере необходимости для медленно- или быстрорастущих клеток и для клеток, которые легко повреждаются трипсином.
ПРИМЕЧАНИЕ: Цель состоит в том, чтобы иметь равномерно расположенные ячейки, прочно прикрепленные к слайду. Рассмотрим индуцирование литической фазы после сливки, если литические клетки являются хрупкими. Выводы, сделанные из экспериментов с клетками iSLK, ограничены9. - Для литик-клеток подвески, предварительно лечить восьмикамерные слайды с 1:10 поли L-лизин в течение 5 минут под капотом культуры ткани. Затем оставьте горки высохнуть на ночь при комнатной температуре или 1 ч при температуре 65 градусов по Цельсию. Инкубировать 800 л литических клеток в концентрации 1 х 106 клеток/мл с камерными горками в течение 30 мин до 1 ч при 37 КС и 5% CO2.
ПРИМЕЧАНИЕ: Подвесные клетки оседают в монослой, прилипая к поли L-лизин и, таким образом, избыточные клетки не являются проблемой по сравнению с клетками адептов. Литические клетки, образующие виноградные скопления, по возможности, должны быть разделены нежным вихрем или химическими средствами. Правда, авторы не имели большого успеха с такими рекомендациями в случае литические BJAB-RRV-GFP клеток. Если подвесные клетки не прилипают хорошо, рассмотреть вопрос об увеличении времени или концентрации поли L-лизин инкубации.
- Для приверженных литические клетки, используйте 1x трипсин / PBS на 37 КС и 5% CO2 в течение 10 минут, чтобы приостановить клетки и разбавить до 60% выпуклости.
3. Фиксация, иммунофлуоресценция (необязательно), гибридизация и визуализация вирусных РНК
- Удалите носители и избыточные ячейки. На протяжении всего этого протокола, использовать вакуум всасывания для удаления решений и нежный микропиптинг, чтобы добавить решения.
ПРИМЕЧАНИЕ: Прочность вакуума может быть уменьшена путем размещения 200 йл микропайпет анаосии над стеклом Пастер пипетка. Замените наконечник micropipette между шагами мытья для предотвращения загрязнения. Каждый шаг мытья должен быть выполнен быстро, потому что крайне важно, чтобы клетки никогда не высыхают. - Немедленно, исправить клетки с предварительно охлажденной 4% формальдегида / PBS (фосфат-буферный солевым раствором) на льду в течение 30 мин. Вымойте клетки три раза с 200 Л Л 1x PBS охлаждается до 4 градусов по Цельсию и инкубировать в течение 5 минут при комнатной температуре или на льду.
- Пермяки фиксированная клетка с 200 qL предварительно охлажденного 0,5% Triton-X/PBS (фосфат буферный солен) в течение 10 мин на льду или 750 л предварительно охлажденного 70% этанола при 4 кс на 1 ч (мин) до 7 d (максимум).
ПРИМЕЧАНИЕ: Сбор белка, общей РНК, и геномных образцов ДНК в точке фиксации для обеспечения согласованности между изображениями и биохимических анализов. Все смывы на протяжении всего протокола выполняются одинаково, если не указано иное. 70% этанола ослабляет клей между камерами и слайдом, что облегчает последующее разделение, а также обеспечивает значительную паузу в протоколе. Тем не менее, используйте парафина пленка вокруг камеры слайд, чтобы уменьшить испарение и проверить уровень этанола в каждой камере около каждые 8 ч. 70% этанола также уплощает клетки, делая четкое изображение, в то время как Triton-X не обезвоживает клетки и изменить размеры ячейки.
- Пермяки фиксированная клетка с 200 qL предварительно охлажденного 0,5% Triton-X/PBS (фосфат буферный солен) в течение 10 мин на льду или 750 л предварительно охлажденного 70% этанола при 4 кс на 1 ч (мин) до 7 d (максимум).
- Тщательно удалите камеры, чтобы предотвратить растрескивание слайда. Если эксперимент включает в себя иммунофлуоресценцию (IF) вирусного или принимающего белка с поликлональным первичным антителом, выполните IF, как описано ниже, прежде чем приступить к РНК FISH. Если иммунофлуоресценция использует моноклональные первичные антитела, то выполнять иммунофлуоресценции, как описано в шаге 3.3.1 после шага 3.11.
ПРИМЕЧАНИЕ: Используйте новое устройство удаления или один с очень мало остатков клея, предоставляемых производителем и мягко облегчить камеры, чтобы предотвратить слайд от растрескивания. Использование 70% этанола в качестве пермякового реагента на 4 ч значительно снижает вероятность растрескивания. В случае трещины продолжайте протокол на камерах, не затронутых трещиной, и помните о более высокой скорости окисления несовершенно запечатанных слайдов (т.е. уменьшенного срока хранения).- Промыть клетки с предварительно охлажденной 1x PBS и блок с предварительно охлажденной 4% BSA (бувин сыворотки альбумина) /1x PBS в течение 30 минут при 4 градусах Цельсия.
ПРИМЕЧАНИЕ: Использование BSA на протяжении всего этого протокола ограничивает неспецифическую маркировку. - Удалить блокирующий раствор и инкубировать клетки с 1:200 или другой поликлональных первичных антител в 0,1% BSA/1x PBS для 1 ч при 4 c. Затем промыть три раза с 1x PBS.
ПРИМЕЧАНИЕ: Антитело10 для обнаружения SSB/ORF6 (вирусный одноцепочечный связывающий белок ДНК) было использовано в 1:200 разбавления. - Инкубировать клетки вторичным антителом с фторфором, совместимым с FISH-обнаруживающим антителом в течение 1 ч при 4 градусах Цельсия. Вымойте три раза с 1x PBS. Затем исправить с 4% формальдегида / 1x PBS в течение 10-15 мин и permeabilize либо Тритон-X или 70% этанола, как ранее описано, прежде чем приступить к FISH. Крышка слайд с фольгой оберморка для сохранения флуоресцентного сигнала и предотвращения фотоотбеления.
- Промыть клетки с предварительно охлажденной 1x PBS и блок с предварительно охлажденной 4% BSA (бувин сыворотки альбумина) /1x PBS в течение 30 минут при 4 градусах Цельсия.
- Вымойте клетки с 2x SSC (соленовый цитрат натрия) один раз, а затем применить 45 мл раствора гибридизации, состоящий из 50% формамида, 10% декекрон сульфат, 2x SSC, 0,1% BSA, 500 мкг/мл лосося ДНК, 125 мкг/мл E. coli tRNA, и 1 mM Комплексов. Инкубировать в течение 1 ч при 37 градусах Цельсия в камере влажности, которая может быть 150 мм чашкой Петри с увлажненные стерильные салфетки.
ПРИМЕЧАНИЕ: Подготовьте свежее решение гибридизации по крайней мере за час до использования. Растворите декстран сульфат в воде во-первых, вихрь часто и инкубации в 37 градусов по Цельсию водяной бане. - Рассчитайте, чтобы иметь рекомендуемую концентрацию 25 мкм олигонуклеотидов в 35-uL гибридизации раствора на камеру. Отрегулируйте концентрацию античувствеонного олигонуклеотида по мере необходимости. Добавьте дистиллированную воду в олигонуклеотиды, чтобы довести объем денатурации до 10 зл.
ПРИМЕЧАНИЕ: После реакции маркировки, олигонуклеотиды хранятся в утоленном растворе, содержащем 0,18 М калийный какодилат, 23 мм Трис-ХЛ, 0,23 мг/мл БСА, 4,5 мМ CoCl2,18 мМ EDTA, 2,7 мМ К-фосфат, и 6,8 мм КЛ, 45 ММ 2-Меркапана 0.02% Тритон X-100, и 2% глицерола. Концентрации достаточно высоки, что разбавление водой приведет к денатурации раствор концентрации вблизи 1x TE (10 мм Tris-HCl и 1 мм EDTA), стандартный буфер денатурации олигонуклеотида. - Денатурировать DIG- и / или Alexa Fluor 594-маркированные олигонуклеотиды при 95 кв кс в течение 5 мин. Затем добавьте 35 йл свежего раствора гибридизации на предназначенную камеру к денатурированным олигонуклеотидам. При выполнении двойной FISH, оба набора анти-чувство олигонуклеотиды могут быть денатурированы и гибридизированы вместе.
- Удалите решение предварительной гибридизации, а затем добавьте в клетки раствор гибридизации, содержащий помеченные олигонуклеотиды. Инкубировать ночь в камере влажности при 37 градусов по Цельсию с оловяной фольгой для защиты флурофора помечены олигонуклеотидов.
ПРИМЕЧАНИЕ: Инкубация должна быть не менее 10 ч и не более 24 ч. - На следующий день, мыть клетки дважды с 2x SSC в течение 10 минут при 37 градусов по Цельсию, а затем дважды с 1x SSC в течение 10 минут при 25 градусов по Цельсию.
- Зафиксировать клетки с предварительно охлажденным 4% формальдегидом/1x PBS в течение 10-15 мин на льду. Затем мыть клетки с PBS три раза и permeabilize в течение 1 ч с предварительно охлажденной 70% этанола или в течение 10 минут с предварительно охлажденной 0,5% Triton-X/1x PBS при 4 c.
- Инкубировать клетки с 1:200 анти-DIG FITC в предварительно охлажденной 0,1% BSA/1x PBS для 1 ч при 4 C. Удалить раствор антитела и промыть три раза с 1x PBS.
- Исправить с предварительно охлажденным 4% формальдегида/1x PBS в течение 10-15 мин при 4 C, а затем мыть три раза с 1x PBS. При выполнении иммунофлюоресценции для хозяина или вирусного белка с моноклональным первичным антителом, permeabilize клеток, а затем выполнить протокол IF, изложенные в шаге 3.3.1. В противном случае перейти к DAPI окрашивания.
- Инкубировать клетки с 0,4 мкг/мл DAPI в предварительно охлажденных 0,5% Triton-X/1x PBS в течение 15 минут на льду, а затем мыть три раза с 1x PBS.
- Горные горки с флуоресцентными бусинами (по желанию) и монтажной средой. Затем запечатайте крышку на слайдс с прозрачным лаком для ногтей.
- Используя конфокальный микроскоп, собирайте изображения образцов в течение часа до недели после выполнения протокола при увеличении 630x. Нанесите несколько слоев лака для ногтей, чтобы запечатать крышку скольжения и продлить жизнь фторфора за счет снижения скорости окисления.
ПРИМЕЧАНИЕ: Не используйте dAPI-содержащий монтажную среду. При сборе изображений включите панель масштаба на каждом изображении для последующей количественной оценки. Флуоресцентные бусы служат в качестве контроля интенсивности флуоресценции между слайдами и образцом препаратов11. Приобретайте изображения в средней части ячейки для двумерной (2D) количественной оценки в шаге 4.
4. Количественная оценка изображений FISH и IF для выделения субклеточной локализации и определения нуклеоцитоплазмического соотношения флуоресценции
- Выполните анализ изображений на собранном стеке различных флуоресцентных окрашенных и слитых изображений для обеспечения согласованности. Установите масштаб программного обеспечения для анализа изображений с помощью панели шкалы, включенной при сборе изображений.
- Для количественной оценки интенсивности флуоресценции по нескольким каналам и в отношении ядерного пятна DAPI используйте линейный инструмент и функцию сюжетного профиля. Затем укажите строку постоянно на копии изображения с помощью маркеров, которые не препятствуют или влияют на суждения зрителя.
- Установить критерии, чтобы направлять, где линия обращается, например, след, который захватывает разнообразие топографических особенностей, пиков и долин, вдоль центральной оси или линии, которая не пересекает перенасыщенных областях.
ПРИМЕЧАНИЕ: Эти следы линии изображают сырую флуоресценцию в клетке и, таким образом, ограничиваются сравнениями местоположения пятна, а не интенсивности. Чтобы сравнить интенсивность одного и того же пятна между слайдами, процедурами или препаратами, добавьте флуоресцентную бисна к слайду в качестве внутреннего контроля во время шага 3.13. Флуоресцентный бусиной должен быть добавлен во время процесса монтажа и обнаружен с теми же настройками на возбуждающей лазерной и фотомультипликаторной трубке (конфокальной).
- Установить критерии, чтобы направлять, где линия обращается, например, след, который захватывает разнообразие топографических особенностей, пиков и долин, вдоль центральной оси или линии, которая не пересекает перенасыщенных областях.
- Чтобы количественно определить сдвиг в субклеточной локализации, вычислить нуклеоцитоплазматические соотношения клеток, проходящих различные процедуры.
- Измерьте площадь и интенсивность сырой флуоресценции как ядра, так и цитоплазмы, используя ядерное пятно DAPI для установки внутренней границы. Включите элементы элемента ядерного и цитоплазмического контроля, такие как ядерная РНК (например, КШВ ПАН РНК) и цитоплазматическая РНК (например, хост GAPDH mRNA). Кроме того, рассчитать интенсивность фона для трех клеточных областей и усреднение значений на пиксель или мкм2.
ПРИМЕЧАНИЕ: Значения интенсивности, как правило, не хватает единиц и поэтому термин "единицы" используется. - Нормализовать как ядерные, так и клеточные значения интенсивности сырья, сначала определив средний фон для одной и той же области, а затем вычитая это индивидуализированное значение из необработанной интенсивности области.
- Например, ядро литической В-клетки имеет площадь 133,4 мкм2 и сырую интенсивность 75976 единиц, в то время как фоновая интенсивность для того же флуоресцентного сигнала была определена в 0,67 единиц на мкм2. Нормализованная ядерная интенсивность будет

- Например, ядро литической В-клетки имеет площадь 133,4 мкм2 и сырую интенсивность 75976 единиц, в то время как фоновая интенсивность для того же флуоресцентного сигнала была определена в 0,67 единиц на мкм2. Нормализованная ядерная интенсивность будет
- Введите значения в следующем уравнении.

ПРИМЕЧАНИЕ: Этот расчет контролирует изменения в субклеточной области. Литическая индукция и медикаментозное лечение может увеличить ядро или изменить размер клетки, соответственно. - Чтобы интерпретировать результаты, создайте участок усов коробки. Равное распределение флуоресцентного сигнала будет близко к нулю, в то время как ядерное распределение будет способствовать положительному значению соотношения, а цитоплазмическое распределение будет направлено в сторону значения отрицательного соотношения.
- Измерьте площадь и интенсивность сырой флуоресценции как ядра, так и цитоплазмы, используя ядерное пятно DAPI для установки внутренней границы. Включите элементы элемента ядерного и цитоплазмического контроля, такие как ядерная РНК (например, КШВ ПАН РНК) и цитоплазматическая РНК (например, хост GAPDH mRNA). Кроме того, рассчитать интенсивность фона для трех клеточных областей и усреднение значений на пиксель или мкм2.
Результаты
Методы FISH и IF, описанные в данной рукописи, показаны на рисунке 1 наряду с количественной оценкой результатов по линейным следам флуоресцентной интенсивности. Представленные здесь результаты являются полуколичественными и дают представление о локализации, а не в сравнении интенсивности различных флуоресцентных пятен, поскольку эксперименты не включали флуоресцентную биса в подготовку слайда. На рисунке 1 также указывается, что цитоплазматические и ядерные области и их соотношения различны для скрытых и литических кШВ-инфицированных клеток. Таким образом, область контролируется в нуклеоцитоплазматических соотношении продемонстрированы на рисунке 2. Рисунок 2 подтверждает расчет, описанный в данной рукописи, на нуклеоцитоплазмическое соотношение с использованием ядерного контроля, вирусной полиаденилатной ядерной (PAN) РНК и цитоплазмического контроля, принимающей GAPDH mRNA. Рисунок 3 показывает, что, когда репликация ДНК KSHV ингибируется в литической фазе с помощью либо фосфоноацетической кислоты (Doxy и PAA) или цидофовира (Doxy и Cido), ранняя транскрипт ORF59-58 смещается в преимущественно цитоплазматическую локализацию. Микрографы и два метода количественной оценки на рисунке 3 поддерживают этот результат и показывают, что PAN РНК локализуется на конкретных ядерных объектах, несмотря на ингибирование репликации вирусной ДНК и изменения, наблюдаемые для ранней стенограммы ORF59-58.

Рисунок 1: Линии следы флуоресцентной интенсивности выявить тонкости флуоресценции в situ гибридизации (FISH) KSHV стенограммы и иммунофлуоресценции (IF) КШВ репликации отсеков. (A-B) Конфокальные изображения TREx RTA (тетрациклина индуцируемых вирусной репликации и транскрипции активатор белка) BCBL-1 клетки12, которые были индуцированы в литическую фазу в течение 24 ч с доксициклина (Докси). Шкала бар указывает 10 мкм. (A) Флуоресценция на месте гибридизации (FISH) для вирусных РНК (зеленый) и иммунофлуоресценции (IF) для вирусного одноцепочечного днк связывающего белка (ORF6/SSB) (красный), компонент KSHV реплики отсеков, показывают, что вирусные транскрипты локализуются в цитоплазме, ядре, и в ядерных очагах за пределами обогащенных ORF6/SSB областей, также известных как отсеки репликации. Анти-SSB антитела10 был разбавлен до 1:200 в 0,4% BSA/1x PBS и обнаружены с 1:500 анти-кролик Alexa Fluor 594 вторичного антитела в 0,4% BSA/1x PBS. Все анти-чувство олигонуклеотиды, используемые в течение этого исследования, приведены в таблице 1. Обнаружение ORF59-58 mRNA включает в себя как бицистроник, так и моноцистронные транскрипты. Однако, в KSHV-инфицированных АО-1 клеток, моноцистроника мРНК, по крайней мере в 18 раз менее обильные, чем бистроник стенограммы и, вероятно, способствует лишь незначительную часть общего флуоресцентного сигнала наблюдается13. Кроме того, один из PAN РНК олигонуклеотиды (SB88) может также обнаружить вирусный транскрипт для K7. Сигнал от обнаружения K7 не будет столь значительным по сравнению с сигналом обнаружения KSHV PAN РНК, который присутствует на почти 80% всех полиаденилатных РНК в литическом KSHV-инфицированной ячейке14. Кроме того, один из четырех антисмысловых олигонуклеотидов (tkv13) при обнаружении K8.1 mRNA способен связываться с несколькими изоформами K8.1 и другими изоформами близлежащих открытых кадров чтения (ORF). Сигнал FISH только от олигонуклеотида tkv13 недостаточен (данные не показаны). Комбинированная гибридизация четырех олигонуклеотидов и их связывание на одной и той же транскрипте, вероятно, обеспечивает наблюдаемый сильный сигнал. Белые линии фланговых ячеек в (A) изображают линейный путь интенсивности флуоресценции для FISH и IF сигналов, построенных в (C). (B) Цифровое увеличенное изображение клеток в (A) в окружении белых линий. Для простоты синий канал DAPI опущен. (C) Сюжеты показывают относительную флуоресцентную интенсивность для каждого пятна вдоль той же линии: SSB (красный), вирусные стенограммы (зеленый; стенограмма, указанная на участке), и DAPI (синий). Затененные области указывают на области, уменьшенные DAPI, которые соответствуют отсекам репликации вируса или обогащенным SSB/ORF6 участкам. (D) Отношение ядерной области к клеточной области изменения и, таким образом, соотношение интенсивности флуоресценции используется во всем была нормализована для области. (E) Ядерные и клеточные области, измеренные для TREx RTA BCBL-1 клеток с и без прохождения литической активации. Статистически значимые изменения наблюдаются по сравнению с неиндуцированными клетками. Участки коробки и усы представляют 10 и 90 процентилей. Рисунок перепечатан с небольшими изменениями из Vallery, Уизерс, и коллеги15 под лицензией Creative Commons Атрибуция. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
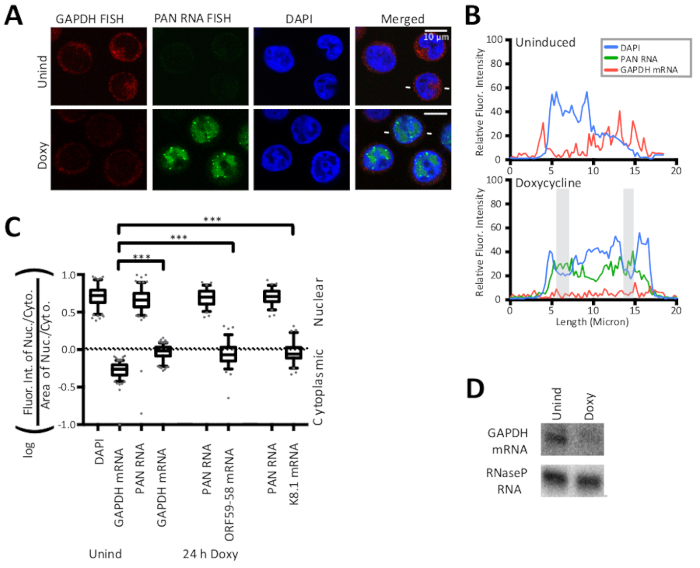
Рисунок 2 : Управление ядерными и цитоплазматическими стратегиями FISH подтверждают метод расчета нуклеоцитоплазмического соотношения. (A) FISH для хозяина GAPDH мРНК (красный) и для вирусных полиаденилаядерных (PAN) lncRNA (зеленый) и DAPI ядерного окрашивания (синий) являются положительными FISH контроля для расчета метода определения нуклеоцитоплазматического соотношения. Host GAPDH mRNA является канонической целью эффекта выключения хоста KSHV и деградирует при литической индукции, как показано здесь. (B) Интенсивность флуоресценции вдоль линии, указанной белыми линиями, фланговых литические клетки в (A). DAPI (синий), PAN РНК (зеленый), и GAPDH мРНК (красный). Затененные области определены на рисунке 1. (C) Количественная оценка интенсивности флуоресценции клеток, представленных (A) (n No 150 для каждого образца GAPDH, n No 75 для образцов ORF59-58 или K8.1) была выполнена для трех биологических репликаций клеток, показанных на рисунке 2 и рисунке 3 . P-значения: :gt;0.05 (нс), lt;0.05 (яп.), «lt;0.005» (яп.) и «lt;0.0005»). (D) Представитель Северной подьеРН от TREx RTA BCBL-1 клетки 24 ч после Докси. Участки коробки и усов представляют 10 и 90 процентиль. Рисунок перепечатан с небольшими изменениями из Vallery, Уизерс, и коллеги15 под лицензией Creative Commons Атрибуция. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
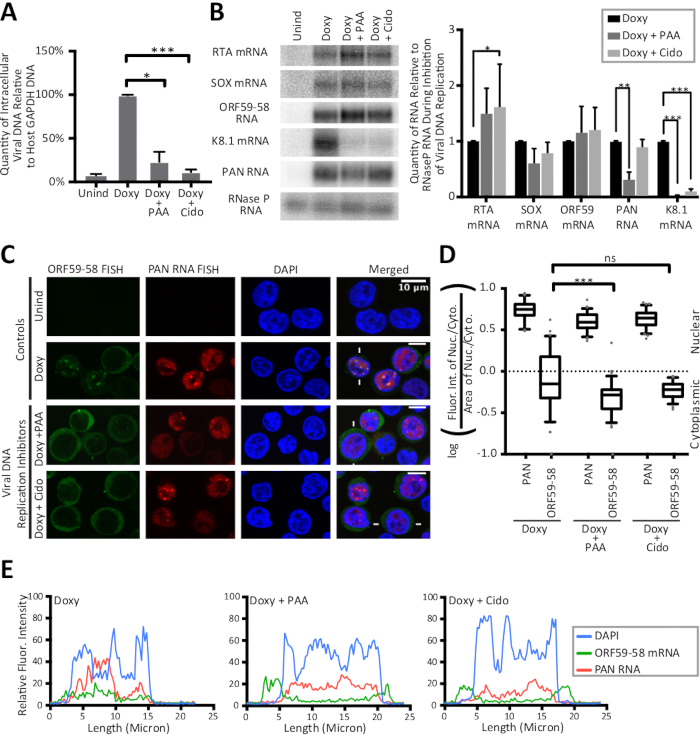
Рисунок 3: Следы линии и вычисление нуклеоцитоплазмических соотношений показывают сильный сдвиг к цитоплазме для раннего литического транскрипта ORF59-58 при ингибировании репликации вирусной ДНК. Клетки TREx RTA BCBL-1 лечились от 24 ч без лекарств (Unind), доксициклина только (Doxy), или с доксициклином и одним ингибитором репликации герпесвирусной ДНК, фосфоноацетической кислоты (Doxy) или сидофовир (Doxy и Cido). Панели(A-C) показывают данные из образцов, собранных из трех биологических репликаций. (A) значения qPCR для вирусной внутриклеточной ДНК во время ингибирования репликации вирусной ДНК были нормализованы к количеству промоторной ДНК гена ГАПДГ. (B) Северная поместье (слева) и количественная (справа) показывают общий уровень РНК во время ингибирования репликации вирусной ДНК. Неиндуцированные уровни всех РНК были необнаружимы. (C) Представитель FISH изображения для вирусных ORF59-58 стенограммы (зеленый) и PAN РНК (красный) при ингибировании вирусной репликации ДНК. DAPI (синий) был ядерного пятна. (D) Количественная оценка интенсивности флуоресценции клеток, представленных (C) (n и 75 каждый) была сделана на биологических триплицикатах. (E) Интенсивность флуоресценции вдоль линий, нарисованных между клетками, указанными белыми линиями в (C) показаны: DAPI (синий), PAN RNA (красный) и ORF59-58 mRNA (зеленый). P-значения: :gt;0.05 (нс), lt;0.05 (яп.), «lt;0.005» (яп.) и «lt;0.0005»). Последовательности всех олигонуклеотидов в данном исследовании приведены в таблице 1. Участки коробки и усы представляют 10 и 90 процентилей. Рисунок перепечатан из Vallery, Уизерс, и коллеги15 под Creative Commons Атрибуция лицензии. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Северный Олигос | |||||||
| Олиго Но. | Ген | Последовательности | Позиция внутри гена | Позиция с эталонным геномом NC009333.1 (числа не отражают направление или нить) | |||
| KORF50 | KSHV RTA/ORF50 | CGCATTGCGGTGGTGAAATTGCTGG | от 1284 до 1309 | от 73936 до 73961 | |||
| JBW249 | KSHV SOX/ORF37 | ТААККТАКТАККАКАКАКАКАКЕТЦКАК | от 262 до 291 | от 57633 до 57662 | |||
| tkv379 | KSHV ORF59-58 | TGGAGTCCTATAGAATCGGGAACCT | от 941 до 967 (ORF59 ORF) | от 95879 до 95905 | |||
| tkv13 | KSHV K8.1 | ААГГКАСТАСТАСТАСТАСТАСТАГАГКККАКАГГАТТКГГГГААТГГААТ | от 16 до 57 | от 76029 до 76070 | |||
| SB2 | КШВ ПАН РНК | АКАААТККККККАКТТГТГГКККК | от 664 до 687 | от 29496 до 29519 | |||
| Рразе P | Человек RNase P | TGGGCGGAGGAGAGTCTG | от 319 до 339 | N/A | |||
| Зонды FISH | |||||||
| Олиго Но. | Ген | Последовательности | |||||
| SB2 | КШВ ПАН РНК | АКАААТККККККАКТТГТГГКККК | от 664 до 687 | от 29496 до 29519 | |||
| SB85 | КШВ ПАН РНК | CGCTGCTTTTTTTCACATT | от 373 до 392 | от 29205 до 29224 | |||
| SB88 | КШВ ПАН РНК | GTGAAGCGGCAGCCAAGGTGTACTGG | От 1 до 22 | от 28830 до 28854 | |||
| tkv13 | KSHV K8.1 | ААГГКАСТАСТАСТАСТАСТАСТАГАГКККАКАГГАТТКГГГГААТГГААТ | от 16 до 57 | от 76029 до 76070 | |||
| tkv14 | KSHV K8.1 | TGATATTAGGCGGTCAGTCTGTGTGTGTGTGTGGGGGGGGGGA | от 377 до 414 | от 76390 до 76427 | |||
| tkv15 | KSHV K8.1 | GTAAGGTTACGCTTTCCCACACCGACGGTTTACCC | от 461 до 500 | от 76474 до 76513 | |||
| tkv16 | KSHV K8.1 | GGACAAGTCCCАГАГААТААААККАККАПКАПКАТТАТАТГГ | от 688 до 725 | от 76701 до 76738 | |||
| tkv376 | KSHV ORF59-58 | ТААТГГТКАТКАТТГАТКТКТГАТТГАТТГАТТ | от 54 до 79 | от 96767 до 96792 | |||
| tkv377 | KSHV ORF59-58 | ГККГГГГККККАКТАКТККГТГТТТТТ | от 93 до 122 | от 96724 до 96753 | |||
| tkv378 | KSHV ORF59-58 | AAGGCTATGCGCGCGTCGAGTACATTCGCA | от 300 до 329 | от 96517 до 96546 | |||
| tkv379 | KSHV ORF59-58 | TGGAGTCCTATAGAATCGGGAACCT | от 941 до 967 | от 95879 до 95905 | |||
| tkv380 | KSHV ORF59-58 | АААГАГТГТГААКТАКАГГКЦКТТ | от 1289 до 1315 | от 95531 до 95557 | |||
| tkv381 | KSHV ORF59-58 | АААКАКТГЦКГКГКАГКАТКАТЦК | от 1423 до 1450 | от 95396 до 95423 | |||
| tkv382 | KSHV ORF59-58 | TACCTGTGTACTTGGGGGCGCCTGATACAC | от 1571 до 1602 | от 95244 до 95275 | |||
| tkv383 | KSHV ORF59-58 | ГГГГТЦАГЦАГЦЦААТАСТАГГАААААКТКАКАКАГЯГГ | от 2136 до 2173 | от 94673 до 94710 | |||
| Стелларис | GAPDH | Сделано Stellaris | N/A | N/A | |||
| qPCR праймеры | |||||||
| tkv458 | Промоутер GAPDH | КГАККАККАККККТЦТТАГТАГТАГТАГТ | N/A | N/A | |||
| tkv459 | Промоутер GAPDH | ГТКТТГГГГГКГАГГГГГАТГГАТ | N/A | N/A | |||
| tkv319 | KSHV ORF39 (gM) | GTGAGGTGTTCGCTGAGTT | N/A | от 60075 до 60094 | |||
| tkv320 | KSHV ORF39 (gM) | CCTGGGTCAAGCTGTGTGTGTTTTT | N/A | от 60218 до 60237 | |||
| Праймеры RT-qPCR | |||||||
| tkv 455 | К8/К-БЗИП Вперед РТ qPCR Праймер | КГААААГКАААГГАГАГАГАГАКГ | от 655 до 673 | от 75603 до 75621 | |||
| tkv 456 | K8/K-bZIP Обратный RT qPCR праймер для unspliced | GCCATGTTCCCATTGAGTGAGT | от 755 до 774 | от 75703 до 75722 | |||
| tkv 457 | K8/K-bZIP Обратный RT qPCR праймер для сращивания | CATCAGCATGTCGCGAAG | от 871 до 888 | от 75819 до 75836 | |||
| JBW479 | Человек RNase P Вперед | АГКТТГГАААГАРАКККЕГ | от 238 до 257 | N/A | |||
| JBW480 | Человек RNase P Обратный | GCGGAGGAGAGAGTAGTCTGAAA | от 317 до 336 | N/A | |||
Таблица 1: Все олигонуклеотиды, используемые в анализе данной публикации. Таблица 1 была воспроизведена с разрешения Американского общества микробиологии под лицензией Creative Commons Attribution от Vallery et al.15.
Обсуждение
Протокол, описанный в настоящем докладе, может быть адаптирован к различным типам клеток и включает в себя шаги для двойной РНК FISH и RNA FISH с IF с использованием как моноклональных, так и поликлональных первичных антител. Хотя подготовленные слайды, как правило, изображены с конфокальный микроскоп, изображение может быть выполнено с STED (стимулируемое истощение выбросов) микроскоп после изменения повышенной концентрации антител и другой монтаж среды. Для расширенного анализа отдельных клеток, образцы, подготовленные с этим протоколом, также могут быть отсортированы, изображены и проанализированы сортатором клеток или цитометром потока со скромными изменениями, как показали Бора и его коллеги16. Этот протокол, однако, не может быть принят для визуализации живых клеток.
Методы количественной оценки, детализированные, устраняют предвзятость наблюдений и служат для проверки потенциального моклеоцитоплазмического сдвига. Соотношение нуклеоцитоплазматов также указывает, когда биомолекула приспосабливается от равномерного рассеиваемости к локализации в определенном субклеточном отсеке. Представленные здесь результаты являются полуколичественными, в то время как в протоколе излагаются пути укрепления количественной оценки. Сила нуклеоцитоплазмического соотношения и следов линии (шаг 4) зависит от использования флуоресцентных бусин в качестве контроля интенсивности (шаг 3.13) и использования четких субклеточных маркеров, таких как один для ядерного Lamin A/C. В настоящее время для отсеков репликации вируса KSHV не существует четкого пограничного маркера. Несмотря на это, этот расчет может быть распространен на другие субклеточные отсеки с использованием соответствующих маркеров.
Основным препятствием для протокола, подробно изложенного в настоящем докладе, является разработка стратегий FISH для конкретных стенограмм (шаг 1). Успех зависит от изобилия и связывания силы антисмысловых олигонуклеотидов. Специфика конкретных стенограмм еще более осложняется наличием перекрывающихся открытых кадров для чтения (ORF) в вирусных геномах. Таким образом, вирусные транскрипты часто имеют сходство последовательности17 с другими вирусными транскриптами из той же геномной области, особенно в случае герпесвирусов. Часто разработка стратегии FISH должна использовать более обильные стенограммы. Чтобы устранить неполадки в отсутствии сигнала FISH, пользователи должны выполнить протокол FISH со стратегией U2 snRNA FISH, чтобы подтвердить, что методы в клетках человека и подготовка реагентов являются адекватными. Аналогичным образом, стратегия KSHV PAN RNA FISH может подтвердить литические активации в кШВ-инфицированных клетках. Для устранения неполадок, связывающих антисмысловые олигонуклеотиды, авторы рекомендуют разработать несколько антисмысловых олигонуклеотидов. Если все не удается, коммерческий вариант доступен, как показано на использовании стратегии GAPDH FISH на рисунке 2 и Vallery, Уизерс, и коллеги15.
Более сильные алгоритмы определения клеточных и субклеточных границ еще больше устранили бы предвзятость количественной оценки. Некоторые аналитические программы обработки изображений могут установить границы для ячейки, ядра и многое другое, но требуют окончательных маркеров. Необычные морфологии клеток, такие как вирусные отсеки репликации трудно для такого программного обеспечения - задача для будущего развития. Кроме того, описанные здесь методы количественной оценки ограничиваются одним оптическим фрагментом ячейки (2D-анализ изображений). В то время как приобретение 3D-изображений возможно18,будущая разработка количественного анализа 3D изображений может обеспечить дальнейшее понимание пространственно-временной регуляции отсеков репликации вирусов.
Раскрытие информации
Авторы не имеют конфликта интересов раскрыть.
Благодарности
Мы благодарим Джонатана Роденфельса, Казимира Тычовски и Джоанну Б. Уизерс за советы по анализу данных. Мы также благодарим Г. Хейворд за анти-SSB антитела. Эта работа была поддержана грантами T32GM007223 и T32AI055403 от Национальных институтов здравоохранения (для ТКВ) и грантом NIH (CA16038) (для JAS). JAS является исследователем Медицинского института Говарда Хьюза. Цифры 1-3 и таблица 1 были воспроизведены с разрешения Американского общества микробиологии под лицензией Creative Commons Attribution из следующей публикации: Валлери, Т. К., Уизерс, Дж.Б., Андо, Дж.А., Штейтц, Саркома-Ассоциированные Дж.А. Капоши Накопление герпесвируса мРНК в ядерной foci зависит от вирусной репликации ДНК и вирусного некодирования полиаденинатной ядерной РНК. В журнале вирусологии. 92 (13), doi:10.1128/JVI.00220-18, (2018).
Материалы
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Ссылки
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81(2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300(2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены