Method Article
KSHV感染细胞中特定基因产物的定量原位荧光杂交(FISH)和免疫荧光(IF)
摘要
我们描述了一种利用荧光原位杂交(FISH)来可视化被神经感染的人体细胞中的多种疱疹病毒RNA的协议,无论是悬浮的还是粘附的。该协议包括荧光的定量,产生核细胞质比,并可扩展用于同时可视化具有免疫荧光(IF)的宿主和病毒蛋白。
摘要
机械洞察力来自对特定RNA和蛋白质的仔细研究和定量。这些生物分子在特定时间在整个细胞中的相对位置可以通过荧光原位杂交(FISH)和免疫荧光(IF)捕获。在溶性疱疹病毒感染过程中,病毒劫持宿主细胞以优先表达病毒基因,导致细胞形态和生物分子行为发生变化。Lytic 活动以核工厂为中心,称为病毒复制隔间,仅与 FISH 和 IF 一起可辨别。在这里,我们描述了一个适应RNA FISH和IF技术卡波西的肉瘤相关疱疹病毒(KSHV)感染细胞,无论是粘附的还是悬浮的。该方法包括特定抗感寡核苷酸、双RNA FISH、带IF的RNA FISH和荧光强度定量计算的步骤。该协议已成功应用于多种细胞类型、未感染细胞、潜伏细胞、溶酶细胞、时间过程和用抑制剂处理的细胞,以分析来自人类宿主和KSHV.
引言
在其溶性(活性)阶段,疱疹病毒劫持宿主细胞,导致细胞形态和生物分子的定位变化,以产生病毒。手术的基础是细胞核,其中双链DNA病毒基因组被复制并包装成一个蛋白质外壳,称为辣椒1。首先,病毒表达自己的蛋白质,劫持宿主机制,防止非必需宿主基因的表达,这个过程称为宿主关闭效应。这种活动的大部分被局部化为特定的4°,6-二酰胺-2-phenyinole(DAPI)无核区域称为病毒复制隔间,由宿主和病毒蛋白、RNA和病毒DNA2组成。细胞经过大修,为复制舱提供空间和资源,从而组装病毒辣椒。一旦辣椒离开细胞核,细胞质中如何包裹辣椒,产生膜结合的病毒颗粒,也称为病毒,目前还不清楚。了解宿主和病毒生物分子在溶质阶段中的定位和空间变化,有助于深入了解复制舱的排列、宿主关闭效应、病毒-出口通路和其他与疱疹病毒感染和复制相关的过程。
目前,检测和研究这些变化的最佳方法是分别对免疫荧光(IF)和荧光原位杂交(FISH)受感染细胞中的蛋白质和RNA进行可视化。使用这些技术的时间过程揭示了生物分子在lytic相的关键点或简单的时空数据的定位。FISH 和 IF 补充了其他生化技术,例如抑制细胞过程(例如抑制病毒DNA复制)、RT-qPCR(实时聚合酶链反应)、RNA 测序、北方印迹、质谱法、西方印迹和分析病毒DNA的产生,这可能提供一个更全球性的细胞活动图景。
我们开发了RNA FISH策略来检查来自特定基因的RNA产物,并进行了定量计算特定基因产物核细胞质比的计算分析。从Steitz和同事3,4的早期出版物中修改的样品制备相对容易,可用于粘附细胞和悬浮细胞。该协议还适用于同时使用多个RNA FISH策略(双RNA FISH)或RNA FISH与IF策略。制定具体的 FISH 战略具有挑战性,但概述了提高成功性的建议。如果使用荧光珠和隔间边界的强标记,并且提供对显微图的更多洞察,从而消除观测偏差,则此处描述的数据分析是定量的。详细方案是专为受卡波西肉瘤相关疱疹病毒(KSHV)感染的潜伏细胞和溶血细胞而设计的,可用于未感染的细胞或其他疱疹病毒感染的细胞5。定量方法适用于大多数细胞中核细胞质转移或亚细胞间重新定位的研究。
研究方案
1. 原位荧光(FISH)抗感寡核苷酸,以检测特定的疱疹病毒记录仪
- 从感兴趣的RNA序列中选择25到40个nt段,并转换为抗感。成功的FISH策略可能包含多达十种或更多不同的反义寡核苷酸。选择序列时,请考虑以下事项:
- 如果感兴趣的RNA包含一个独特的重复区域,则利用此功能并设计一个抗感寡核苷酸以靶向重复序列。
注:Tycowski 和同事5提供了此策略的示例,包括河套病毒 (RRV) 多核核 (PAN) RNA。 - 如果感兴趣的RNA含有已知的蛋白质结合位点或茎环结构,则设计避免这些区域的寡核苷酸。
- 根据实验的目标,考虑电子序列以及是否为其设计抗感寡核苷酸。
- 如果感兴趣的RNA包含一个独特的重复区域,则利用此功能并设计一个抗感寡核苷酸以靶向重复序列。
- 对选定的抗感序列执行简单的计算分析,以确保结合特异性并减少抗感寡核苷酸的聚合。
- 序列必须大约 50% GC 富集(高鸟氨酸和细胞氨酸含量),并且熔融温度在 60 到 70°C 之间。
- 使用序列分析工具选择不自混化或形成熔融温度高于 37°C(杂交温度)的发夹的序列。
- 使用"有点相似"的设置,针对宿主和病毒转录子对所选序列进行NCBI BLASTn(国家生物技术信息中心基本局部对齐搜索工具)搜索所选序列。此搜索将识别独特的抗感寡核苷酸,这些核苷酸不太可能绑定到其他宿主或病毒记录。
注:如果转录组不可用,则使用基因组序列执行 BLAST 搜索。如果从实验中使用的受感染细胞中分离出的病毒序列进行搜索,这是理想的选择,因为野生菌株往往多样化,并包含来自不同实验室菌株的序列组合。
- 订购纯化的DNA寡核苷酸对应于经过计算验证的抗感序列,该序列是唯一的,并有可能与目标RNA结合。寡核苷酸无需引入特殊修饰。
- 测试所设计的抗感寡核苷酸,以检测FISH和北方斑点的结合特异性。
- 使用来自同一宿主物种(例如,293T)和理想情况下相同细胞类型的未受感染细胞系,使用质粒进行转染,该质粒表达来自强健的启动子(CMV,细胞巨病毒)和空载体(例如PCDNA3)的RNA。使用正对照转染,例如与GFP(绿色荧光蛋白)质粒(例如,pmaxGFP)或含有GFP的载体共转染。
注意:重要的是要避免使用疱疹病毒爱泼斯坦-巴尔病毒(EBV)永生的细胞系,因为疱疹病毒之间有序列相似性。 - 如第 3 节所述,对具有抗感寡核苷酸的两组细胞执行 FISH,如本协议所述。通过将 FISH 实验与抗感寡核苷酸的单个、对或组进行比较,推断出成功的候选者。对FISH协议使用正对照,如U2snRNA(小核RNA)FISH,每人类细胞核6为500,000份(表1)。
- 荧光信号在含有感兴趣的RNA的细胞中应该是特异性和强的。设计额外的抗感寡核苷酸,以加强信号,并去除非特定结合的抗感寡核苷酸。信号强度必须高于背景和自荧光。
- 测试由北方斑点结合特异性。
- 使用来自同一宿主物种(例如,293T)和理想情况下相同细胞类型的未受感染细胞系,使用质粒进行转染,该质粒表达来自强健的启动子(CMV,细胞巨病毒)和空载体(例如PCDNA3)的RNA。使用正对照转染,例如与GFP(绿色荧光蛋白)质粒(例如,pmaxGFP)或含有GFP的载体共转染。
2. 寡核苷酸和细胞制备
- 按照制造商的说明,使用终端转移酶将抗感寡核苷酸与二恶英(DIG)-dUTP进行标记,或者,如果强结合,则直接与像Alexa Fluor 594-5-dUTP那样的荧光核苷酸结合。标记后,无需额外纯化。在-20°C下储存标有寡核苷酸,并储存在锡箔中(如果直接贴有,以防止光漂白)。
注意:标签溶液含有有毒物质,钾卡科迪酸盐。用手套处理标签反应。
注:为了节约资源,可以在一个反应中标记几个不同的抗感寡核苷酸。该协议使用 3'端标签。内部标记具有挑战性,因为化学组(例如 DIG 或荧光团)被捕获或无法进入 DNA 聚合酶的活性位点。使用直接标记的抗感寡核苷酸和具有不同荧光(例如,FITC(荧光素)或 Alexa Fluor 488 与 Alexa Fluor 594)的抗 DIG 免疫荧光,可对两种不同的 RNA 进行实验。 - 将单元格粘附到八室幻灯片上。
注:八室幻灯片允许同时进行多次实验,同时最大限度地减少抗体等宝贵资源。八室幻灯片的替代方案是六孔组织培养板,其标准盖玻片(22 mm x 22 mm)均无菌。与圆形盖玻片和 24 孔组织培养板类似安排是可能的。对于这两种方案,将分别将该协议中提到的体积增加10-15倍(例如,1.75 mL杂交溶液)和4倍(例如600 μL杂交溶液)。- 对于粘附性溶酶细胞,在37°C下使用1x胰蛋白酶/PBS,在5%CO2下10分钟暂停细胞,并稀释至60%的汇合。
注:FISH实验中使用的粘附细胞系包括293T、iSLK.2197和iSLK-BAC36细胞8。 - 在无菌八室幻灯片的每个腔室上涂抹200 μL的细胞悬浮液,在37°C和5%CO2下,使种子生长12-24小时。根据需要调整缓慢或快速生长的细胞和容易被胰蛋白酶损坏的细胞。
注: 目标是将均匀间距的单元格牢固地连接到幻灯片上。如果裂解细胞易碎,请考虑在粘附后诱导裂解阶段。从iSLK细胞实验中得出的结论有限9。 - 对于溶性悬浮细胞,在组织培养罩下用1:10聚L-流碱预处理8腔幻灯片5分钟。然后,让幻灯片在室温下干燥过夜,或在 65°C 下保持 1 小时干燥。在37°C和5%CO2下,用室形幻灯片孵育800 μL的溶血细胞,浓度为1 x 106细胞/mL。
注:悬浮细胞会稳定在单层中,粘附在多L-莱氨酸上,因此与附着细胞相比,多余的细胞不为人所关注。如果可能,形成葡萄簇的溶血细胞应通过温和的涡旋或化学手段分离。无可否认,在溶性BJAB-RRV-GFP细胞的情况下,作者在这些建议方面没有取得多大成功。如果悬浮细胞粘附性不好,请考虑增加多L-莱辛孵育的时间或浓度。
- 对于粘附性溶酶细胞,在37°C下使用1x胰蛋白酶/PBS,在5%CO2下10分钟暂停细胞,并稀释至60%的汇合。
3. 病毒RNA的固定、免疫荧光(可选)、杂交和可视化
- 移除介质和多余的单元格。在整个协议中,使用真空吸气去除溶液和温和的微移液来添加解决方案。
注:通过在玻璃巴斯德移液器上放置 200 μL 微移液器,可以降低真空强度。在洗涤步骤之间更换微移液器尖端,以防止污染。每个洗涤步骤必须快速执行,因为细胞必须永不干燥。 - 立即将预冷冻的4%甲醛/PBS(磷酸盐缓冲盐水)固定在冰上30分钟。用200μL 1x PBS冷却至4°C,在室温或冰上孵育5分钟,将细胞清洗三次。
- 用200μL预冷冻0.5%Triton-X/PBS(磷酸盐缓冲盐水)在冰上渗透10分钟,或4°C时750μL预冷冻70%乙醇,1小时(分钟)至7天(最大)。
注:在固定点收集蛋白质、总RNA和基因组DNA样本,以确保图像和生化检测之间的一致性。除非另有说明,否则此协议中的所有处理均以相同的方式执行。70% 乙醇使腔室和滑轨之间的胶水松动,从而简化了以后的分离,并在协议中提供了显著的暂停。尽管如此,在腔室滑动处使用石蜡薄膜来减少蒸发,并检查每个腔室中大约每 8 小时 70% 乙醇的乙醇水平,使细胞变平,使图像更加清晰,而 Triton-X 不会使细胞脱水并改变单元格的尺寸。
- 用200μL预冷冻0.5%Triton-X/PBS(磷酸盐缓冲盐水)在冰上渗透10分钟,或4°C时750μL预冷冻70%乙醇,1小时(分钟)至7天(最大)。
- 小心地拆下腔室,以防止滑动开裂。如果实验包括具有多克隆原抗体的病毒蛋白或宿主蛋白的免疫荧光 (IF),则按照下文所述执行 IF,然后再进入 RNA FISH。如果免疫荧光使用单克隆原抗体,则执行免疫荧光,如步骤 3.11 之后的步骤 3.3.1 所述。
注:使用新的拆卸装置或制造商提供的很少的剩胶的去除装置,轻轻将腔室关闭以防止滑片开裂。使用 70% 乙醇作为渗透试剂 4 小时可大大降低开裂的可能性。在裂纹的情况下,在不受裂纹影响的腔室上继续执行协议,并注意不完全密封的幻灯片的氧化率较高(即存储寿命缩短)。- 用预冷冻的1x PBS冲洗细胞,用预冷冻的4%BSA(牛血清白蛋白)/1xPBS在4°C下进行30分钟的冷冻。
注: 在整个协议中使用 BSA 会限制非特定标签。 - 去除阻断溶液,在4°C下用1:200或其他多克隆原抗体在0.1%BSA/1x PBS中孵育细胞1小时。然后用1x PBS洗三次。
注:在1:200稀释时,使用抗体10检测SSB/ORF6(病毒单链DNA结合蛋白)。 - 用与FISH检测抗体兼容的二级抗体孵育细胞,在4°C下孵育1小时。用 1x PBS 洗涤三次。然后用4%甲醛/1x PBS固定10-15分钟,然后像前面描述的那样用Triton-X或70%乙醇渗透,然后再继续FISH。用锡箔盖住幻灯片,以保留荧光信号并防止光漂白。
- 用预冷冻的1x PBS冲洗细胞,用预冷冻的4%BSA(牛血清白蛋白)/1xPBS在4°C下进行30分钟的冷冻。
- 用2x SSC(柠酸盐钠)清洗细胞,然后应用45μL的杂交溶液,包括50%形式酰胺、10%硫酸盐、2x SSC、0.1%BSA、500μg/mL鲑鱼DNA、125微克/mL大肠杆菌tRNA和1mMM vanadyl肋骨苷配合 物。在 37°C 下在湿度室中孵育 1 小时,该湿度室可以是 150 mm 培养皿,带湿润无菌湿巾。
注:在使用前至少一小时准备新的杂交溶液。先将硫酸盐溶解在水中,频繁涡旋,在37°C水浴中孵育。 - 计算每个腔室35-uL杂交溶液中建议浓度为25μM寡核苷酸。根据需要调整抗感寡核苷酸的浓度。将蒸馏水加入寡核苷酸,使变性体积至10μL。
注:在标记反应后,寡核苷酸储存在含有0.18M钾钙化、23 mM Tris-HCl、0.23 mg/mL BSA、4.5 mM CoCl 2、18mM EDTA、2.7 mM K-磷酸盐和6.8 mM KCl、45 μM 2-Mercaptoto酸的淬火溶液中。0.02% Triton X-100 和 2% 甘油。浓度足够高,与水稀释将使变性溶液的浓度接近1x TE(10 mM Tris-HCl和1 mM EDTA),一个标准的寡核苷酸变性缓冲液。 - 在 95°C 下使 DIG 和/或 Alexa Fluor 594 标记寡核苷酸变性 5 分钟。然后,将每个预期腔室的35μL新杂交溶液添加到变性寡核苷酸中。如果执行双FISH,两组抗感寡核苷酸可以变性和杂交在一起。
- 去除预杂交溶液,然后将含有标记的寡核苷酸的杂交溶液添加到细胞中。用锡箔在37°C的湿度室中孵育过夜,以保护含氟量标记的寡核苷酸。
注:孵育应至少为10小时,不超过24小时。 - 第二天,用2xSSC在37°C下洗涤细胞两次10分钟,然后用1xSSC在25°C下洗涤10分钟。
- 将细胞与预冷冻的4%甲醛/1x PBS固定在冰上10-15分钟。然后用PBS清洗细胞三次,用预冷冻的70%乙醇渗透1小时,或在4°C下用预冷冻0.5%Triton-X/1x PBS清洗10分钟。
- 在预冷0.1%BSA/1x PBS中孵育1:200抗DIG FITC的细胞,在4°C下孵育1小时。取出抗体溶液,用1x PBS洗涤三次。
- 在4°C下用预冷冻的4%甲醛/1x PBS修复10-15分钟,然后用1x PBS洗涤三次。如果使用单克隆原抗体对宿主或病毒蛋白进行免疫荧光,则渗透细胞,然后执行步骤 3.3.1 中概述的 IF 协议。否则,继续 DAPI 染色。
- 用0.4μg/mL DAPI在预冷冻0.5%Triton-X/1x PBS中孵育细胞15分钟,然后用1x PBS洗涤三次。
- 使用荧光珠子(可选)和安装介质安装幻灯片。然后用透明指甲油将盖玻片密封到滑轨上。
- 使用共聚焦显微镜,在以 630 倍放大率执行协议后一小时到一周内收集样本图像。应用多层指甲油密封盖滑,并通过降低氧化速率延长荧光酸寿命。
注:请勿使用包含 DAPI 的安装介质。收集图像时,请在每个图像上包括比例尺,以便以后进行量化。荧光珠子作为幻灯片和样品制剂之间的荧光强度的控制11。在步骤 4 中获取单元格中段的图像,进行二维 (2D) 定量。
4. FISH和IF图像的量化,以突出亚细胞定位并确定荧光的核细胞质比
- 对各种荧光染色和合并图像的组装堆栈执行图像分析,以确保一致性。使用收集图像时包括的比例尺设置图像分析软件的比例。
- 要量化多个通道的荧光强度,并参照核 DAPI 染色,请使用线工具和图谱图式函数。然后,使用不会妨碍或影响观看者判断的标记在图像副本上永久指示线条。
- 建立引导绘制直线的标准,例如沿中轴或不遍历超饱和区域的曲线捕获地形要素、峰谷多样性的轨迹。
注:这些线迹描绘了细胞中的原荧光,因此仅限于比较污渍的位置,而不是强度。要比较幻灯片、处理或制剂之间相同污渍的强度,请在第 3.13 步期间将荧光珠作为内部控制添加到幻灯片中。荧光珠必须在安装过程中添加,并在激励激光和光电倍增管(共聚焦)上使用相同的设置进行检测。
- 建立引导绘制直线的标准,例如沿中轴或不遍历超饱和区域的曲线捕获地形要素、峰谷多样性的轨迹。
- 要量化亚细胞定位的变化,计算接受不同治疗的细胞的核细胞质比。
- 使用核 DAPI 染色设置内部边界,测量细胞核和细胞质的面积和原始荧光强度。包括核和细胞质控制,如核RNA(如KSHV PAN RNA)和细胞质RNA(例如宿主GAPDH mRNA)。此外,计算三个类似像像细胞区域的背景强度,并平均每个像素或 μm2的值。
注: 强度值往往缺少单位,因此使用术语"单位"。 - 首先确定同一区域的平均背景,然后从区域的原始强度中减去该个性化值,从而使核强度和蜂窝原始强度值标准化。
- 例如,溶血B细胞的细胞核的面积为133.4μm2,原始强度为75976单位,而同一荧光信号的背景强度被确定为每μm20.67个单位。正常化的核强度将是

- 例如,溶血B细胞的细胞核的面积为133.4μm2,原始强度为75976单位,而同一荧光信号的背景强度被确定为每μm20.67个单位。正常化的核强度将是
- 输入以下公式中的值。

注: 此计算控制亚细胞区域的变化。溶质诱导和药物治疗可以分别扩大细胞核或改变细胞的大小。 - 要解释结果,请创建一个框胡须图。荧光信号的等值分布将接近于零,而核分布将倾向于正比值,而细胞质分布将趋向于负比值。
- 使用核 DAPI 染色设置内部边界,测量细胞核和细胞质的面积和原始荧光强度。包括核和细胞质控制,如核RNA(如KSHV PAN RNA)和细胞质RNA(例如宿主GAPDH mRNA)。此外,计算三个类似像像细胞区域的背景强度,并平均每个像素或 μm2的值。
结果
本手稿中详述的 FISH 和 IF 方法如图1所示,以及按荧光强度线痕迹对结果进行量化。这里给出的结果是半定量的,提供了对定位的洞察,而不是不同荧光污渍的强度的比较,因为实验在幻灯片制备中不包括荧光珠。图1还显示,细胞质和核区域及其比率对于潜伏和溶性KSHV感染细胞是不同的。因此,面积在图2所示的核质比中控制。图2验证了本手稿中详细计算的核细胞质比,使用核控制、病毒聚化核(PAN)RNA和细胞质控制,宿主GAPDH mRNA。图 3显示,当 KSHV DNA 复制在裂解阶段被抑制使用磷酸( + PAA )或西多福韦( + Cido ),早期的 ORF59 - 58 转录转成主要细胞质定位。图3中的显微图和两种定量方法支持这一结果,并表明PANRNA在抑制病毒DNA复制和早期ORF59-58转录本形变化的情况下,仍对特定的核位点进行本地化。

图1:荧光强度线迹揭示KSHV转录本的荧光原位杂交(FISH)和KSHV复制舱的免疫荧光(IF)的细微之处。(A-B)TREx RTA (四环素诱导病毒复制和转录活化蛋白)的共聚焦图像 BCBL - 1 细胞 12 ,诱导多氧环素()进入溶酶相 24 小时。刻度条表示 10 μm . (A) 病毒RNA(绿色)和免疫荧光(IF)的病毒单链DNA结合蛋白(ORF6/SSB)(红色)的原位杂交(FISH),这是KSHV复制隔间的组成部分,表明病毒转录本在细胞质、核和核核心体外ORF6/SSB富集区(也称为复制室)中本地化。抗SSB抗体10在0.4%BSA/1x PBS中稀释至1:200,并在0.4%BSA/1x PBS中检测出1:500抗兔子Alexa Fluor 594二次抗体。本研究中使用的所有抗感寡核苷酸均在表1中提供。ORF59-58 mRNA的检测包括双电子和单细胞文字。然而,在KSHV感染的JSC-1细胞中,单细胞mRNA的丰度至少比双核细胞低18倍,并且可能只占观测到的13个荧光信号总量的一小部分。此外,PAN RNA寡核苷酸(SB88)之一也可以检测K7的病毒记录。与检测KSHV PAN RNA的信号相比,K7检测产生的信号没有那么显著,KSHV PAN RNA在溶性KSHV感染细胞14中几乎占所有聚化RNA的80%。此外,在检测K8.1 mRNA的四个抗感寡核苷酸(tkv13)中,有一个能够结合到K8.1的多种等体以及附近开放阅读帧(ORF)的其他等形。仅来自寡核苷酸 tkv13 的 FISH 信号不足(未显示数据)。四个寡核苷酸的组合杂交和它们在同一转录本上的结合可能提供观察到的强烈信号。(A) 中的白线侧翼单元描绘了 FISH 和 IF 信号的荧光强度的线路径,绘制在 (C) 中。(B) 以白线为两侧的 (A) 单元格的数字缩放图像。为简单起见,省略了蓝色 DAPI 通道。(C) 图显示了沿同一行每个污渍的相对荧光强度:*SSB(红色)、病毒转录本(绿色;图中指示的脚本)和 DAPI(蓝色)。带沙区域表示 DAPI 减少的区域对应于病毒复制隔间或 SSB/ORF6 富集区域。(D) 核区与细胞面积的比例变化,因此整个区域使用的荧光强度比是标准化的。(E) 为TREx RTA BCBL-1细胞测量的核和细胞区域,无论是否进行溶精活化。与未诱导的细胞相比,可以看到具有统计学意义的变化。框和胡须图表示 10 和 90 百分位数。图重印,从瓦莱里,莱瑟斯和同事15在知识共享归因许可证略有修改。请点击此处查看此图的较大版本。
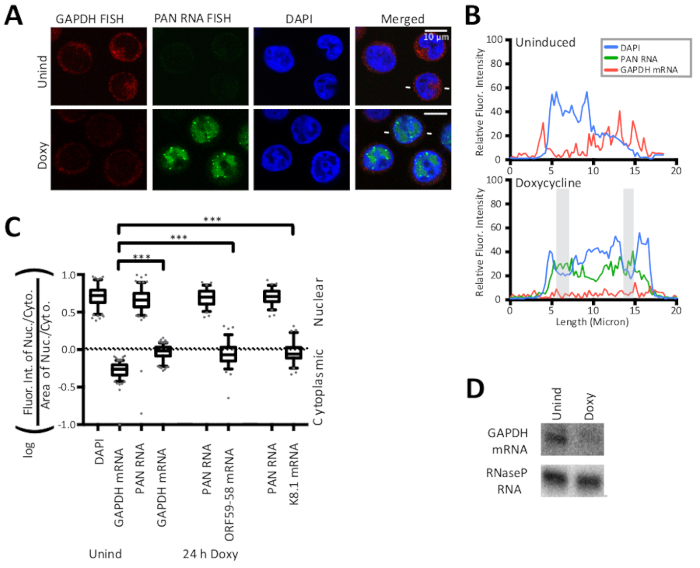
图 2:控制核质和细胞质FISH策略验证核细胞质比的计算方法。(A) FISH 用于宿主 GAPDH mRNA(红色)和病毒聚化核 (PAN) lncRNA (绿色) 和 DAPI 核染色 (蓝色) 是确定核细胞质比的计算方法的正 FISH 对照。宿主 GAPDH mRNA 是 KSHV 主机关闭效应的规范目标,在裂解诱导时降解,如下所示。(B) 沿(A)中白线向溶于流解细胞指示的线的荧光强度。DAPI(蓝色)、PAN RNA(绿色)和GAPDH mRNA(红色)。被调整的区域如图1中定义的那样。(C) 对图2和图3所示的三种细胞的荧光强度进行了量化,其中(A)(n = 150,每个GAPDH样本为150,ORF59-58或K8.1样本为75)。 .P 值: > 0.05 (ns), <0.05 (*), <0.005 (*) 和 <0.0005 (*)。(D) 在多氧之后24小时,TREx RTA BCBL-1细胞中具有代表性的RNA北方斑点。框和胡须图表示 10 和 90 百分位数。图重印,从瓦莱里,莱瑟斯和同事15在知识共享归因许可证略有修改。请点击此处查看此图的较大版本。
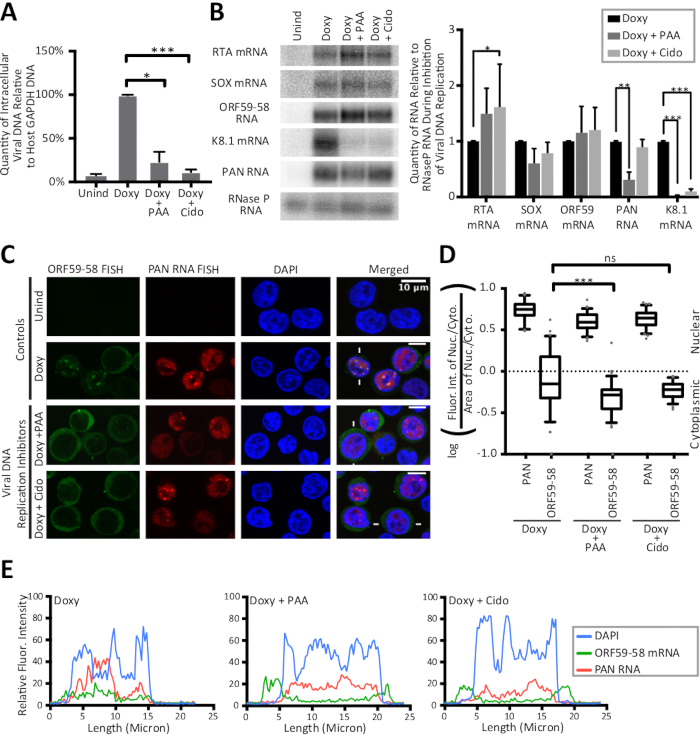
图3:线痕和核细胞质比的计算显示,早期裂解ORF59-58转录在抑制病毒DNA复制时,细胞质有很强的转移。TREx RTA BCBL-1细胞治疗24小时,无药物(Unind),仅多氧环素(多氧),或使用多氧环素和疱疹DNA复制的抑制剂之一,磷酸(多氧+PAA)或西多福韦(多氧+Cido)。面板 (A-C) 显示从三个生物复制中收集的样本的数据.(A) 病毒细胞内DNA在病毒DNA复制抑制过程中的qPCR值归一化为宿主细胞GAPDH基因的启动DNA数量。(B) 北方斑点(左)和定量(右)显示病毒DNA复制抑制期间的总RNA水平。所有RNA的未诱导水平均无法检测。(C) 病毒ORF59-58转录本(绿色)和PANRNA(红色)在抑制病毒DNA复制时具有代表性的FISH图像。DAPI(蓝色)是核污点。(D) 对生物三元细胞进行(c)(n = 75)代表的细胞荧光强度的定量。(E) 沿(C)中白线指示的细胞绘制的线的荧光强度显示:DAPI(蓝色)、PAN RNA(红色)和ORF59-58 mRNA(绿色)。P 值: > 0.05 (ns), <0.05 (*), <0.005 (*) 和 <0.0005 (*)。本研究中所有寡核苷酸的序列在表1中提供。框和胡须图表示 10 和 90 百分位数。图为瓦莱里、瓦瑟斯和同事在知识共享归因许可证下转载。请点击此处查看此图的较大版本。
| 北奥利戈斯 | |||||||
| 奥利戈号 | 基因 | 序列 | 在基因内的位置 | 参考 NC009333.1 基因组的位置(数字不反映方向或链) | |||
| KORF50 | KSHV RTA/ORF50 | CGCATTGGGTATATTGGGG | 1284 到 1309 | 73936 到 73961 | |||
| JBW249 | KSHV SOX/ORF37 | 塔阿加特加卡卡卡卡加中协 | 262 到 291 | 57633 到 57662 | |||
| tkv379 | KSHV ORF59-58 | TgggtCCGGATATGGGAATATGGGAACCT | 941 到 967 (ORF59 ORF) | 95879 到 95905 | |||
| tkv13 | KSHV K8.1 | AAGGATAGGATGGGCCGGCTCTGGGGAT | 16 到 57 | 76029 到 76070 | |||
| SB2 | KSHV PAN RNA | ACAATGCCACCTCCGC | 664 到 687 | 29496 到 29519 | |||
| Rnase P | 人类RNase P | TGGGGGGGGTCTG | 319 到 339 | 不适用 | |||
| FISH 探头 | |||||||
| 奥利戈号 | 基因 | 序列 | |||||
| SB2 | KSHV PAN RNA | ACAATGCCACCTCCGC | 664 到 687 | 29496 到 29519 | |||
| SB85 | KSHV PAN RNA | 中联委会 | 373 到 392 | 29205 到 29224 | |||
| SB88 | KSHV PAN RNA | GTAAGCGCAGCCAAGGGGACTGGGGAGG | 1 到 22 | 28830 到 28854 | |||
| tkv13 | KSHV K8.1 | AAGGATAGGATGGGCCGGCTCTGGGGAT | 16 到 57 | 76029 到 76070 | |||
| tkv14 | KSHV K8.1 | TGATATTAAGGCTGTGTGTGTGGGGGGGGGGGA | 377 到 414 | 76390 到 76427 | |||
| tkv15 | KSHV K8.1 | 格塔格塔格塔塔塔塔ACACACCGTTCTCTCTCTCTCTCTCTCTCCC | 461 到 500 | 76474 到 76513 | |||
| tkv16 | KSHV K8.1 | GGACAAGCCAAACACGCCTATG | 688 到 725 | 76701 到 76738 | |||
| tkv376 | KSHV ORF59-58 | 塔加特加特加特加特加特克加特 | 54 到 79 | 96767 到 96792 | |||
| tkv377 | KSHV ORF59-58 | GCCGATCCCCACTACTACTCCATATCCATATCCATATCCATATATCCGTT | 93 到 122 | 96724 到 96753 | |||
| tkv378 | KSHV ORF59-58 | AAGGCTATGCCAGGAGAGATCTATTTTTA | 300 到 329 | 96517 到 96546 | |||
| tkv379 | KSHV ORF59-58 | TgggtCCGGATATGGGAATATGGGAACCT | 941 到 967 | 95879 到 95905 | |||
| tkv380 | KSHV ORF59-58 | AAAAGGGAACGAGTACAGGGGGGGCCTT | 1289 到 1315 | 95531 到 95557 | |||
| tkv381 | KSHV ORF59-58 | AAACACTGCCGCGGATATCCCCCC | 1423 到 1450 | 95396 到 95423 | |||
| tkv382 | KSHV ORF59-58 | TACCTGTATATATGGCGCGATATATACAC | 1571 到 1602 | 95244 到 95275 | |||
| tkv383 | KSHV ORF59-58 | GGGGGGA加特加格加特加阿阿塔卡克塔格 | 2136 到 2173 | 94673 到 94710 | |||
| 斯特拉里斯 | 加普德 | 由斯特拉里斯预制 | 不适用 | 不适用 | |||
| qPCR 引基器 | |||||||
| tkv458 | GAPDH 发起人 | CTGCACCACACGCTTAG | 不适用 | 不适用 | |||
| tkv459 | GAPDH 发起人 | GTCTCTGGGGGGAT | 不适用 | 不适用 | |||
| tkv319 | KSHV ORF39 (gM) | GTGAGGTGCGCTGGGGGGGGGGGGGGGGGGGGGGG | 不适用 | 60075 到 60094 | |||
| tkv320 | KSHV ORF39 (gM) | CCTGGGTCTGTTTTTT | 不适用 | 60218 到 60237 | |||
| RT-qPCR 引基器 | |||||||
| tkv 455 | K8/K-bZIP 转发 RT qPCR 底漆 | CGAAGAGAGAGAGAGAGAGAGAGAGAGAGAGCG | 655 到 673 | 75603 到 75621 | |||
| tkv 456 | K8/K-bZIP 反向 RT qPCR 底漆,用于未切片 | GCCATTTCCCATATATATATATATATATATATATATATATATATATATATATATATATATATAT | 755 到 774 | 75703 到 75722 | |||
| tkv 457 | K8/K-bZIP 反向 RT qPCR 底漆,用于拼接 | CATCAGCATGCGAG | 871 到 888 | 75819 到 75836 | |||
| JBW479 | 人类RNase P前进 | AGCTTAAACAACTACTAGGGGGGG | 238 到 257 | 不适用 | |||
| JBW480 | 人类 RNase P 反向 | GCGG加加GTAGTTA | 317 到 336 | 不适用 | |||
表1:本出版物分析中使用的所有寡核苷酸。表1经美国微生物学会许可,根据瓦莱里等人的"知识共享归因"许可转载。
讨论
本报告中描述的方案可以适应不同的细胞类型,包括使用单克隆和多克隆原抗体的IF双RNA FISH和RNA FISH的步骤。虽然制备的幻灯片通常使用共聚焦显微镜进行成像,但经过抗体浓度增加和不同的安装介质的修改后,可以使用 STED(刺激发射消耗)显微镜进行成像。为了增强对单个细胞的分析,使用该协议制备的样品也可以由细胞分拣器或流动细胞仪进行分类、成像和分析,变化不大,如Borah及其同事16所示。但是,此协议不能用于活细胞成像。
详细的定量方法消除了观测偏差,并用于验证潜在的核质转移。当生物分子从均匀分散到特定亚细胞室的定位时,核质比也精确定位。这里介绍的结果是半定量的,而协议概述了加强量化的方法。核细胞质比和线痕的强度(步骤4)取决于使用荧光珠作为强度控制(步骤3.13)和使用清晰的亚细胞标记,如核拉明A/C的标记。此时,KSHV 病毒复制隔间不存在清晰的边界标记。无论如何,使用适当的标记,此计算可以扩展到其他亚细胞室。
本报告详述的协议的主要障碍是制定特定成绩单的FISH战略(步骤1)。成功依赖于抗感寡核苷酸的丰度和结合力。由于病毒基因组中存在重叠的开放阅读帧 (ORF),特定转录本的特异性变得更加困难。因此,病毒转录本通常具有序列相似性17与其他病毒转录本从同一基因组区域,特别是在疱疹病毒的情况下。通常,开发 FISH 战略必须利用更丰富的成绩单。为了排除FISH信号的缺失,用户应该使用U2 snRNA FISH策略执行FISH协议,以确认人体细胞和试剂制备中的技术和制备是足够的。同样,KSHV PAN RNA FISH 策略可以确认 KSHV 感染细胞中的溶性激活。为了排除抗感寡核苷酸结合的问题,作者建议开发几种抗感寡核苷酸。如果全部失败,一个商业选项是可用的,如使用GAPDH FISH策略在图2和瓦莱里,维瑟斯和同事15。
更强有力的算法来定义细胞和亚细胞边界将进一步消除量化偏差。某些分析图像处理软件可以为细胞、细胞核等设置边界,但需要明确的标记。对于此类软件而言,病毒复制隔间等异常细胞形态很难,这是未来发展的一个挑战。此外,此处描述的量化方法仅限于一个单元的一个光学切片(2D 图像分析)。虽然3D图像采集是可能的18,但未来定量3D图像分析的发展可能进一步洞察病毒复制隔间的时空调节。
披露声明
提交人没有利益冲突可披露。
致谢
我们感谢乔纳森·罗登费尔斯、卡齐米尔兹·泰科夫斯基和约翰娜·威瑟斯就数据分析提供建议。我们还感谢G.海沃德的抗SSB抗体。这项工作得到了国家卫生研究院(TKV)和NIH赠款(CA16038)(JAS)的T32GM007223和T32AI055403的资助。JAS是霍华德·休斯医学研究所的研究员。图1-3和表1经美国微生物学会许可转载,从以下出版物获得知识共享归因许可:瓦莱里、T.K.、威瑟斯、J.B.、安多、斯蒂茨、J.A.卡波西的《萨科马联系》核病毒中的疱疹病毒mRNA积累受病毒DNA复制和病毒非编码多基因化核RNA的影响。病毒学杂志。92 (13), doi:10.1128/JVI.00220-18, (2018)。
材料
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
参考文献
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81(2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300(2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。