Method Article
KSHV感染細胞における特定遺伝子産物の高量蛍光ハイブリダイゼーション(FISH)と免疫蛍光(IF)における定量蛍光
要約
我々は、その場ハイブリダイゼーション(FISH)における蛍光を利用して、凍結感染したヒト細胞内の複数のヘルペスウイルスRNAを懸濁または付着性のいずれかで可視化するプロトコルについて説明する。このプロトコルは、核細胞質比を産生する蛍光の定量を含み、免疫蛍光(IF)を有する宿主およびウイルスタンパク質の同時可視化のために拡張することができる。
要約
機械的な洞察は、特定のRNAおよびタンパク質の慎重な研究と定量から到着します。特定の時間における細胞全体のこれらの生体分子の相対的な位置は、その場ハイブリダイゼーション(FISH)および免疫蛍光(IF)における蛍光で捕捉することができる。溶解ヘルペスウイルス感染の間、ウイルスはウイルス遺伝子を優先的に発現するために宿主細胞をハイジャックし、細胞形態および生体分子の挙動に変化を引き起こす。溶解活性は、FISHとIFでのみ識別可能なウイルス複製コンパートメントと呼ぶ原子力工場を中心としています。ここでは、カポジ肉腫関連ヘルペスウイルス(KSHV)感染細胞に対するRNA FISHおよびIF技術の適応可能なプロトコルについて説明する。この方法には、特異的な抗感オリゴヌクレオチドの開発のためのステップ、ダブルRNA FISH、IFを有するRNA FISH、および蛍光強度の定量計算が含まれる。このプロトコルは、複数の細胞タイプ、未感染細胞、潜伏細胞、溶解細胞、時間コース、および阻害剤で処理された細胞に正常に適用され、ヒト宿主およびタンパク質の両方から特定のRNAおよびタンパク質の時空間的活性を分析し、KSHV
概要
その溶解性(活性)相において、ヘルペスウイルスは宿主細胞をハイジャックし、細胞形態の変化および生体分子の局在化を引き起こし、ビリオンを産生する。操作のベースは核で、二本鎖DNAウイルスゲノムが複製され、カプシド1と呼ばれるタンパク質シェルに包まれます。まず、ウイルスは独自のタンパク質を発現し、宿主機構を乗っ取り、非必須の宿主遺伝子の発現を防止し、宿主の遮断効果と呼んだプロセスである。この活性の大部分は、宿主およびウイルスタンパク質、RNA、およびウイルスDNA2の両方からなるウイルス複製コンパートメントと呼ばれる特定の4'、6-ジアミド-2-フェニリンドール(DAPI)フリー核領域に局在する。セルは、複製コンパートメントのスペースとリソースを提供し、ウイルスカプシドの組み立てに対して、オーバーホールされます。いったんカプシドが核から出ると、細胞質にカプセル化して膜結合型ウイルス粒子(ビリオンとも呼ばれる)を作り出す方法は不明である。溶解期における宿主とウイルス生体分子の両方の局在化と空間シフトの理解は、複製コンパートメントの配置、宿主の遮断効果、ビリオン出口経路、およびその他の機械的洞察を提供します。ヘルペスウイルス感染および複製に関連するプロセス。
現在、これらの変化を検出して研究する最良の方法は、免疫蛍光(IF)と蛍光を有する感染細胞におけるタンパク質およびRNAの可視化(FISH)である。これらの技術を用いたタイムコースの使用は、溶解相の重要な点または単に時空間的なデータにおける生体分子の局在を明らかにする。FISHおよびIFは、細胞プロセスの阻害(例えば、ウイルスDNA複製の阻害)、RT-qPCR(リアルタイムポリメラーゼ連鎖反応)、RNAシーケンシング、ノーザンブロット、質量分析、ウェスタンブロッティングなどの他の生化学的技術を補完する。ウイルスDNA産生の分析は、細胞活動のよりグローバルな画像を提供する可能性があります。
特定の遺伝子からRNA産物を調べるRNA FISH戦略と、特定遺伝子産物の核細胞質比を定量的に計算する計算解析を開発した。Steitzたちは、Steitzたちは以前の出版物3,4から改変したサンプル調製物は比較的容易であり、付着細胞および懸濁細胞の両方に使用することができる。プロトコルはまたIFの戦略が付いている複数のRNA FISHの戦略(二重RNA FISH)またはRNA FISHの同時使用のために適応可能である。特定のFISH戦略の開発は困難ですが、成功を改善するための提案が概説されています。ここで説明するデータ分析は、蛍光ビーズとコンパートメント境界の強いマーカーを使用する場合に定量的であり、顕微鏡写真に関するさらなる洞察、観察バイアスを除去する洞察を提供します。詳細なプロトコルは、カポジ肉腫関連ヘルペスウイルス(KSHV)に感染した潜伏細胞および溶解細胞の両方のために設計されており、他のヘルペスウイルス5に感染した未感染細胞または細胞で使用することができる。定量の方法は、ほとんどの細胞の細胞下のコンパートメント間の核細胞シフトまたは再局在化に関する研究に適用可能である。
プロトコル
1. 特定のヘルペスウイルス転写物を検出するためのその場所(FISH)抗感覚オリゴヌクレオチドにおける蛍光の設計
- 目的のRNAの配列から25~40ntセグメントを選択し、アンチセンスに変換します。成功したFISH戦略は、1つから1つ以上の異なる反感覚オリゴヌクレオチドを含むことがあります。シーケンスを選択するときは、次の点を考慮してください。
- 対象のRNAに固有のリピート領域が含まれている場合は、この特徴を利用し、反復配列を標的とするアンチセンスオリゴヌクレオチドを設計する。
注:Tycowskiたちは、レゲスラディノウイルス(RRV)ポリアデデン化核(PAN)RNAを使用したこの戦略の例を示す。 - 目的のRNAが既知のタンパク質結合部位またはステムループ構造を含む場合は、これらの領域を回避するオリゴヌクレオチドを設計する。
- 実験の目的に応じて、イントロニック配列とそれに対する反感覚オリゴヌクレオチドを設計するかどうかを検討する。
- 対象のRNAに固有のリピート領域が含まれている場合は、この特徴を利用し、反復配列を標的とするアンチセンスオリゴヌクレオチドを設計する。
- 選択したアンチセンス配列に対して簡単な計算解析を実行して、結合特異性を確保し、反感オリゴヌクレオチドの凝集を減らします。
- 配列は約50%GCリッチ(高グアニンおよびシトシン含有量)でなければならず、60〜70°Cの範囲で溶融温度を有する。
- シーケンスアナライザツールを使用して、ハイブリダイゼーション温度が37°Cを超える溶融温度でヘアピンを自己薄暗化または形成しないシーケンスを選択します。
- 「やや類似した」設定を使用して、ホストとウイルスのトランスクリプトームの両方に対して選択された配列のNCBI BLASTn(国立バイオテクノロジー情報基本局所アライメント検索ツール)を実行します。この検索は、他の宿主またはウイルス転写物に結合しない可能性が高いユニークな反感オリゴヌクレオチドを同定する。
注: トランスクリプトームが使用できない場合は、ゲノム配列を使用して BLAST 検索を実行します。野生株は多様化し、異なるラボ株からの配列の組み合わせを含む傾向があるので、実験で使用される感染細胞から単離されたウイルスからの配列に対して検索を行う場合に理想的です。
- 計算上検証されたアンチセンス配列に対応する精製DNAオリゴヌクレオチドを一意かつ標的RNAに結合する可能性が高い順に並べ替える。特別な修飾をオリゴヌクレオチドに導入する必要はありません。
- FISHおよびノーザンブロットによる結合特異性のために設計されたアンチセンスオリゴヌクレオチドをテストします。
- 同じ宿主種(例えば、293T)および理想的には同じ細胞型からの未感染細胞株を用いて、堅牢なプロモーター(CMV、サイトメガロウイルス)および空のベクター(例えば、pcDNA3)から目的のRNAを発現するプラスミドを用いてトランスフェクションを行う。GFP(緑色蛍光タンパク質)プラスミド(例えば、pmaxGFP)またはGFPを含むベクターとの共トランスフェクションなどのトランスフェクションに正の制御を使用します。
注:ヘルペスウイルス間に配列の類似性があるため、ヘルペスウイルスエプスタインバーウイルス(EBV)を使用して不死化された細胞株を避けることは重要です。 - このプロトコルに記載されているように、反感オリゴヌクレオチドを持つ細胞の両方のセットに関するセクション3に記載されているようにFISHを実行する。FISH実験を個人、ペア、またはアンチセンスオリゴヌクレオチドのセットと比較して、成功した候補を推測します。このようなU2 snRNA(小型核RNA)FISHなどのFISHプロトコルに対して正の対照を使用し、ヒト細胞核6当たり50万部に存在する(表1)。
- 蛍光シグナルは、目的のRNAを含む細胞において特異的かつ強いものでなければならない。信号を強化し、考慮から非特異的に結合するアンチセンスオリゴヌクレオチドを除去するために、追加のアンチセンスオリゴヌクレオチドを設計します。信号強度は、バックグラウンドおよび自己蛍光を上回る必要があります。
- ノーザンブロットによるテストバインディング特異性。
- 同じ宿主種(例えば、293T)および理想的には同じ細胞型からの未感染細胞株を用いて、堅牢なプロモーター(CMV、サイトメガロウイルス)および空のベクター(例えば、pcDNA3)から目的のRNAを発現するプラスミドを用いてトランスフェクションを行う。GFP(緑色蛍光タンパク質)プラスミド(例えば、pmaxGFP)またはGFPを含むベクターとの共トランスフェクションなどのトランスフェクションに正の制御を使用します。
2. オリゴヌクレオチドおよび細胞製剤
- メーカーの指示に従って、端子トランスフェラーゼを使用して、反感オリゴヌクレオチドにジオキシゲニン(DIG)-dUTPを付加するか、強く結合する場合は、Alexa Fluor 594-5-dUTPのような蛍光ヌクレオチドと直接付け加えます。ラベリング後、追加の精製は必要ありません。光漂白を防ぐために直接ラベル付けされた場合は、-20°Cで-20°C、スズ箔にラベル付けされたオリゴヌクレオチドを保管してください。
注意:標識溶液には、有毒物質、カコジ酸カリウムが含まれています。手袋でラベリング反応を処理します。
注:資源を節約するために、いくつかの異なる反感オリゴヌクレオチドは、1つの反応で標識することができる。このプロトコルは、3'エンドラベリングを利用します。内部標識は、化学グループ(例えば、DIGまたは蛍光素)が捕捉されるか、またはDNAポリメラーゼの活性部位に入ることができないため、困難である。2つの異なるRNAを用いた実験は、直接標識された抗感オリゴヌクレオチドおよび異なる蛍光を用いた抗DIG免疫蛍光(例えば、FITC(フルオレセイン)またはAlexa Fluor 594によるAlexa Fluor 488)を用いて行うことができる。 - 細胞を8チャンバースライドに付着させます。
注:8チャンバースライドは、抗体のような貴重な資源を最小限に抑えながら、いくつかの同時実験を可能にします。8チャンバースライドの代替は、両方とも無菌である標準的なカバースリップ(22 mm x 22 mm)を備えた6ウェル組織培養プレートです。円形カバースリップおよび24ウェルティッシュ培養板と同様の配置は可能である。いずれの場合も、このプロトコルで言及されている体積をそれぞれ10~15倍(例えば、1.75mLのハイブリダイゼーションソリューション)、および4倍(例えば、600μLハイブリダイゼーション溶液)ずつ増加させる。- 付着溶解細胞の場合は、37°Cで1xトリプシン/PBSを使用し、10分間5%のCO2を使用して細胞を中断し、60%の合流性に希釈します。
注:FISH実験で使用される付着細胞株は、293T、iSLK.2197、およびiSLK-BAC36細胞8を含む。 - 無菌8チャンバースライドの各チャンバに200 μLの細胞懸濁液を塗布し、37°Cおよび5%CO2で12-24 hの種子増殖を可能にする。遅い細胞や急速に成長する細胞やトリプシンによって損傷しやすい細胞に対して必要に応じて調整します。
注: 目的は、スライドに均等な間隔のセルをしっかりと取り付けることです。溶解細胞が壊れやすい場合は、接着後に溶解相を誘導することを検討してください。iSLK細胞を用いた実験から導き出された結論は9. - 溶解性懸濁細胞の場合、組織培養フードの下で5分間1:10ポリL-リジンを用いた8チャンバースライドを前処理する。その後、スライドを室温で一晩乾燥させるか、65°Cで1時間乾燥させます。1 x 106細胞/mLの濃度で800 μLの溶解細胞を37°Cおよび5%CO2で30分から1時間のチャンバリングスライドでインキュベートする。
注:懸濁細胞は単層に定着し、ポリL-リジンに付着するため、過剰な細胞は付着細胞と比較して問題にならない。ブドウクラスターを形成する溶解細胞は、可能であれば、穏やかな渦または化学的手段によって分離されるべきである。確かに、著者は、溶解性BJAB-RRV-GFP細胞の場合、このような推奨事項で多くの成功を収めていない。懸濁細胞がよく付着しない場合は、ポリL-リジンインキュベーションの時間または濃度を増加させることを検討してください。
- 付着溶解細胞の場合は、37°Cで1xトリプシン/PBSを使用し、10分間5%のCO2を使用して細胞を中断し、60%の合流性に希釈します。
3. ウイルスRNAの固定、免疫蛍光(オプション)、ハイブリダイゼーション、可視化
- メディアと余分なセルを削除します。このプロトコル全体を通して、真空吸引を使用して溶液を除去し、穏やかなマイクロピペッティングを使用して溶液を追加します。
注:ガラスパスツールピペットの上に200 μLマイクロピペットチップを置くことによって真空の強さを減らすことができます。汚染を防ぐために洗浄ステップの間にマイクロピペット先端を交換してください。細胞が決して乾燥しないことが不可欠であるため、各洗浄ステップは迅速に実行する必要があります。 - 直ちに、予め冷やされた4%ホルムアルデヒド/PBS(リン酸緩衝生理食べ物)を氷上に30分間固定し、200μL 1x PBSを4°Cに冷却して3回洗浄し、室温または氷上で5分間インキュベートします。
- 予め冷やされた0.5%のトリトンX/PBS(リン酸緩衝生理食べ物)の200 μLで固定細胞を透過化し、氷上で10分間、または4°Cで70%エタノールを750μLで1h(分)~7d(最大)で冷やします。
注:固定の時点でタンパク質、総RNA、およびゲノムDNAサンプルを収集し、画像と生化学アッセイの間の一貫性を確保します。特に指定がない限り、このプロトコル全体のすべての洗い出しは同じ方法で実行されます。70%のエタノールは、チャンバーとスライドの間の接着剤を緩め、後で分離を容易にし、またプロトコルの重要な一時停止を提供する。それにもかかわらず、蒸発を減らすためにチャンバースライドの周りにパラフィンフィルムを使用し、約8時間70%のエタノールも細胞を平坦化し、トリトン-Xは細胞を脱水し、変化させません。セルの寸法。
- 予め冷やされた0.5%のトリトンX/PBS(リン酸緩衝生理食べ物)の200 μLで固定細胞を透過化し、氷上で10分間、または4°Cで70%エタノールを750μLで1h(分)~7d(最大)で冷やします。
- スライドのひび割れを防ぐために慎重にチャンバーを取り外します。実験にポリクローナル一次抗体を有するウイルスまたは宿主タンパク質の免疫蛍光(IF)が含まれる場合は、RNA FISHに進む前に以下に説明するようにIFを実行する。免疫蛍光がモノクローナル一次抗体を使用する場合は、ステップ3.11の後にステップ3.3.1に記載されているように免疫蛍光を行う。
注:新鮮な取り外し装置またはメーカーが提供するほとんど残りの接着剤を使用し、スライドが割れるのを防ぐためにチャンバーを静かに緩和します。70%エタノールをパーメバイジング試薬として4時間使用すると、割れの可能性が大幅に低下します。亀裂の場合は、亀裂の影響を受けないチャンバー上のプロトコルを継続し、不完全に密封されたスライドの高い酸化速度(すなわち貯蔵寿命の減少)に注意してください。- 予め冷やされた1x PBSを持つ細胞をすすり、4%BSA(ウシ血清アルブミン)/1x PBSを4°Cで30分間冷やした。
注: このプロトコル全体で BSA を使用すると、非特異的ラベリングが制限されます。 - ブロッキング溶液を除去し、1:200または別のポリクローナル一次抗体で細胞を0.1%BSA/1x PBSで4°Cで1時間インキュベートします。その後、1x PBSで3回洗浄します。
注:SSB/ORF6(ウイルス一本鎖DNA結合タンパク質)を検出するための抗体10を1:200希釈で使用した。 - 4°Cで1時間のFISH検出抗体と互換性のあるフッ素を持つ二次抗体で細胞をインキュベートします。1x PBSで3回洗います。その後、4%ホルムアルデヒド/1x PBSを10〜15分間固定し、FISHに進む前に前述のトリトンXまたは70%エタノールのいずれかで透過させます。蛍光信号を保持し、光の漂白を防ぐために錫箔でスライドをカバーします。
- 予め冷やされた1x PBSを持つ細胞をすすり、4%BSA(ウシ血清アルブミン)/1x PBSを4°Cで30分間冷やした。
- 細胞を2x SSC(クエン酸生理塩水)で一度洗浄し、50%のホルムアミド、10%のデキストラン硫酸塩、2x SSC、0.1%BSA、500 μg/mLサーモン精子DNA、125 μg/mL E. Coli tRNA、および1Mmvad複合 体。湿った滅菌ワイプで150 mmペトリ皿にすることができる湿度室で37 °Cで1時間インキュベートします。
注:使用前に少なくとも1時間前に新鮮なハイブリダイゼーションソリューションを準備してください。デキストラン硫酸塩を最初に水に溶かし、頻繁に渦を吐き、37°Cの水浴でインキュベートします。 - チャンバー当たり35uLハイブリダイゼーション溶液中の25μMオリゴヌクレオチドの推奨濃度を有するように計算する。必要に応じてアンチセンスオリゴヌクレオチドの濃度を調整します。オリゴヌクレオチドに蒸留水を加え、変色量を10μLにします。
注:標識反応に続いて、 オリゴヌクレオチドは、0.18Mカコジウ酸カコジリン酸カコジウ酸カコジウ酸カコジウ酸カコジウ酸カコジウ酸、23mMトリス-HCl、0.23 mg/mL BSA、4.5mM CoCl 2、18mM EDTA、2.7 mM K-リン酸塩、および6.8mMKCl、および6.8mMKClを含むクエンチ溶液中に貯蔵される。0.02% トリトンX-100, グリセロール 2% .濃度は、水との希釈が1x TE(10 mMトリス-HClおよび1 mM EDTA)、標準的なオリゴヌクレオチド変変飽和バッファーに近い濃度に変色溶液をもたらすのに十分高いです。 - DIGおよび/またはアレクサ・フルオール594標識オリゴヌクレオチドを95°Cで5分間変性する。次に、変性オリゴヌクレオチドに意図したチャンバーあたり35μLの新鮮なハイブリダイゼーション溶液を添加する。二重FISHを行う場合、反感オリゴヌクレオチドの両方のセットは、一緒に変性し、ハイブリダイズされてもよい。
- ハイブリダイゼーション前溶液を除去し、標識されたオリゴヌクレオチドを含むハイブリダイゼーション溶液を細胞に添加する。蛍眼レベレータオリゴヌクレオチドを保護するために、スズ箔で37°Cの湿度室で一晩インキュベートします。
注:インキュベーションは、少なくとも10時間と24時間以下でなければなりません。 - 翌日、細胞を2x SSCで2回37°Cで10分間洗浄し、次に25°Cで10分間1x SSCで2回洗浄します。
- 氷の上で10-15分間、予め冷やされた4%ホルムアルデヒド/1x PBSで細胞を固定します。その後、PBSで細胞を3回洗浄し、予冷70%エタノールで1時間、または4°Cで0.5%のトリトンX/1x PBSを予冷させた10分間透過させます。
- 予め冷やされた0.1%BSA/1x PBSで1:200抗DIG FITCで細胞をインキュベートし、4°Cで1時間。抗体溶液を取り出し、1x PBSで3回洗浄する。
- 4°Cで10〜15分間、予め冷やされた4%ホルムアルデヒド/1x PBSで固定し、1x PBSで3回洗浄します。モノクローナル一次抗体を用いて宿主またはウイルスタンパク質に対して免疫蛍光を行う場合は、細胞を透過化し、ステップ3.3.1で概説するIFプロトコルを実行する。それ以外の場合は、DAPI 染色に進みます。
- 0.4 μg/mL DAPIで細胞を予冷0.5%のトリトンX/1x PBSで15分間氷上でインキュベートし、1x PBSで3回洗浄します。
- 蛍光ビーズ(オプション)と取り付け媒体を備えたスライドを取り付けます。その後、明確なマニキュアでスライドにカバースリップをシールします。
- 共焦点顕微鏡を使用して、630倍の倍率でプロトコルを実行する1時間以内にサンプルの画像を収集します。カバースリップを密封し、酸化率を下げることによって蛍舗の寿命を延ばすために、マニキュアの複数のコートを適用します。
注: DAPI を含む取り付け媒体は使用しないでください。画像を収集するときは、後で定量化するために各画像にスケールバーを含めます。蛍光ビーズは、スライドと試料調製物11との間の蛍光強度の対照として機能する。ステップ 4 で 2 次元(2D)定量化のために、セルの中間部分で画像を取得します。
4. 細胞内局在を強調し、蛍光の核細胞質比を決定するFISH画像とIF画像の定量化
- さまざまな蛍光染色およびマージされた画像の組み立てられたスタック上で画像解析を実行し、一貫性を確保します。画像の収集時に含まれるスケールバーを使用して、画像解析ソフトウェアのスケールを設定します。
- 複数のチャネルにわたって蛍光強度を定量し、核DAPI染色を参照するには、ラインツールとプロットプロファイル関数を使用します。次に、視聴者の判断を妨げたり影響を与えたりしないマーカーを使用して、画像のコピーに対して行を永続的に示します。
- 中心軸に沿って地形フィーチャ、ピーク、谷の多様性をキャプチャするトレースや、過飽和領域を横断しない線など、線が描画される場所をガイドする基準を確立します。
注:これらの線の痕跡は、細胞内の生の蛍光を示すので、強度ではなく、汚れの位置の比較に限定されます。スライド、トリートメント、または調製物の間で同じ汚れの強度を比較するには、ステップ3.13の間に内部制御としてスライドに蛍光ビーズを追加します。蛍光ビーズは、取り付けプロセス中に追加し、励起レーザーおよび光増倍管(共焦点)の同じ設定で検出する必要があります。
- 中心軸に沿って地形フィーチャ、ピーク、谷の多様性をキャプチャするトレースや、過飽和領域を横断しない線など、線が描画される場所をガイドする基準を確立します。
- 細胞内局在のシフトを定量化するには、異なる治療を受けている細胞のヌクレオシトプラスマズム比を計算する。
- 核DAPI染色を用いて核と細胞質の両方の面積と生蛍光強度を測定し、内側の境界を設定します。核RNA(例えば、KSHV PAN RNA)および細胞質RNA(例えば、宿主GAPDH mRNA)などの核および細胞質制御を含む。さらに、3 つのセルのような領域の背景強度を計算し、ピクセルあたりまたは μm2あたりの値を平均します。
注: 強度値には単位がない傾向があるので、"単位" という用語が使用されます。 - 最初に同じ領域の平均背景を決定し、その個別化値を領域の生の強度から差し引くことによって、核強度とセルラーの生の強度値の両方を正規化します。
- 例えば、溶解B細胞の核は面積が133.4 μm2、生強度が75976単位であるのに対し、同じ蛍光信号の背景強度はμm2当たり0.67単位であると判定された。正規化された核強度は

- 例えば、溶解B細胞の核は面積が133.4 μm2、生強度が75976単位であるのに対し、同じ蛍光信号の背景強度はμm2当たり0.67単位であると判定された。正規化された核強度は
- 次の式に値を入力します。

注: この計算は、セルラー領域の変化を制御します。溶解性誘導および薬物治療は、それぞれ核を拡大したり、細胞の大きさを変更することができます。 - 結果を解釈するには、ボックスウィスカープロットを作成します。蛍光シグナルの均等な分布はゼロに近いが、核分布は正の比率値を支持し、細胞質分布は負の比率値に向かう傾向にある。
- 核DAPI染色を用いて核と細胞質の両方の面積と生蛍光強度を測定し、内側の境界を設定します。核RNA(例えば、KSHV PAN RNA)および細胞質RNA(例えば、宿主GAPDH mRNA)などの核および細胞質制御を含む。さらに、3 つのセルのような領域の背景強度を計算し、ピクセルあたりまたは μm2あたりの値を平均します。
結果
この原稿に詳述されているFISHおよびIF法は、蛍光強度の線トレースによる結果の定量化と共に図1に示す。ここで提示される結果は半定量的であり、実験はスライド調製に蛍光ビーズを含んでいなかったため、異なる蛍光染色の強度間の比較ではなく、局在化に関する洞察を提供する。図1はまた、細胞質および核領域とその比率が潜伏細胞および溶解性KSHV感染細胞に対して異なることを明らかにする。したがって、面積は、図2に示す核細胞質比で制御される。図2は、核制御、ウイルスポリアデニル化核(PAN)RNA、および細胞質制御、宿主GAPDH mRNAを用いた核細胞質比について、この原稿に詳述された計算を検証する。図3は、ホスホノ酢酸(ドキシ+PAA)またはシドフォビル(ドキシ+シド)のいずれかを使用してKSHV DNA複製が溶解期で阻害されると、初期のORF59-58転写物が主に細胞質局在化にシフトすることを明らかにする。図3の顕微鏡写真と2つの定量法は、この結果を支持し、PAN RNAがウイルスDNA複製の阻害および初期ORF59-58転写物に見られる変化にもかかわらず、特定の核位に局所化することを明らかにした。

図1:KSHV転写物のハイブリダイゼーション(FISH)における蛍光強度の線跡は、KSHV複製コンパートメントの免疫蛍光(IF)の微妙性を明らかにする。(A-B)TREx RTAの共焦点画像(テトラサイクリン誘導性ウイルス複製および転写活性化タンパク質)BCBL-1細胞12は、ドキシサイクリン(Doxy)を用いて24時間の溶解相に誘導された。スケールバーは、ウイルスRNA(緑色)およびウイルス単一鎖DNA結合タンパク質(ORF6/SSB)(赤色)の上のハイブリダイゼーションにおける10μm(A)蛍光(S)を示し、KSHV複製コンパートメントの構成要素であることを明らかにする。ウイルス転写物は、細胞質、核、およびORF6/SSB濃縮領域外の核病巣に局所化し、複製コンパートメントとも呼ばれる。抗SSB抗体10を0.4%BSA/1x PBSで1:200に希釈し、0.4%BSA/1x PBSで1:500抗ウサギAlexa Fluor 594二次抗体で検出した。本研究全体で使用されるすべてのアンチセンスオリゴヌクレオチドは、表1に提供される。ORF59-58 mRNAの検出には、二次性および単一分裂性転写物の両方が含まれる。しかしながら、KSHV感染JSC-1細胞において、単一対化物mRNAは、ビシトロニクス転写物よりも少なくとも18倍少なく、観察された全蛍光シグナルのわずかな部分しか寄与しない可能性が高い13。さらにPAN RNAオリゴヌクレオチド(SB88)の1つはまた、K7のウイルス転写物を検出することができる。K7の検出からの信号は、溶解性KSHV感染細胞14内のすべてのポリアデデン化RNAのほぼ80%に存在するKSHV PAN RNAを検出するシグナルと比較して有意ではない。さらに、K8.1 mRNAの検出における4つのアンチセンスオリゴヌクレオチド(tkv13)のうちの1つは、K8.1および近くの開いている読書フレーム(ORF)の他のアイソフォームの複数のアイソフォームに結合することができる。オリゴヌクレオチドtkv13のみからのFISH信号が不十分である(データは示さない)。4つのオリゴヌクレオチドの結合と同じ転写物上のそれらの結合は、観察された強いシグナルを提供する可能性が高い。(A)中の白線横糸は、FISHおよびIFシグナルに対する蛍光強度の線経路を表し、(C)にプロットした。(B) 白い線で横たわる(A)のセルの画像をデジタルズームした。わかりやすくするために、青い DAPI チャネルは省略されています。(C) プロットは、同じ線に沿った各染色に対する相対的な蛍光強度を示します:αSSB(赤)、ウイルス転写物(緑色、プロットに示された転写物)、およびDAPI(青)。シェーディングされた領域は、ウイルスレプリケーション コンパートメントまたは SSB/ORF6 エンリッチ領域に対応する DAPI 削減領域を示します。(D) 細胞領域に対する核領域の比率が変化し、従って使用される蛍光強度比は領域について正規化された。(E) TREx RTA BCBL-1細胞について、溶解性活性化を受けた有無にかかわらず測定された核および細胞領域。非誘導細胞と比較して統計的に有意な変化が見られる。ボックスとウィスカーのプロットは、10 パーセンティルと 90 パーセンティルを表します。フィギュアは、クリエイティブ・コモンズ・アトリビューション・ライセンスの下で、ヴァレリー、ウィズーズ、および同僚15からのわずかな変更で転載しました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
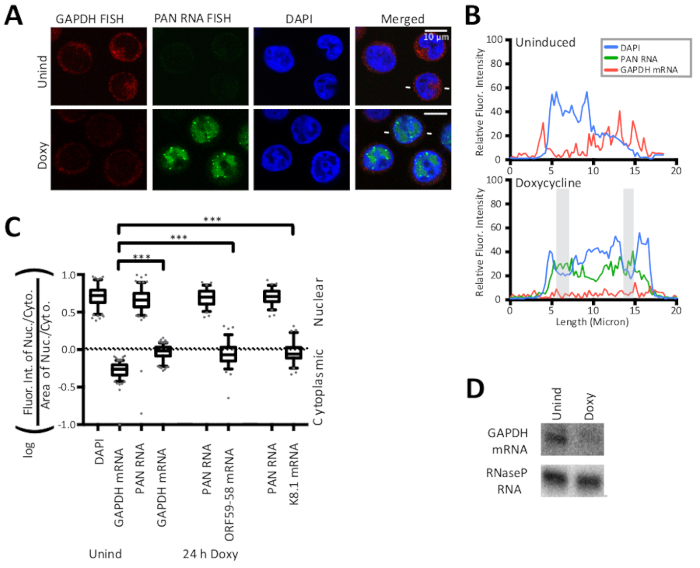
図 2:制御核および細胞質魚戦略は、核細胞質比の計算方法を検証する。(A) 宿主GAPDH mRNA(赤色)及びウイルスポリアデデン化核(PAN)用の魚(緑色)及びDAPI核染色(青色)は、核細胞質比を決定する計算方法に対する正のFISH対照である。宿主GAPDH mRNAは、KSHVの宿主シャットオフ効果の正規目標であり、ここに示すように溶解誘導時に分解される。(B)(A)の溶解細胞を横切る白線で示される線に沿った蛍光強度。DAPI(青)、パンRNA(緑)、GAPDH mRNA(赤)。シェード領域は、図 1で定義されているとおりです。(C) (A) (GAPDH サンプルごとに n = 150、 ORF59-58 または K8.1 サンプルの n = 75) で表される細胞の蛍光強度の定量を、図 2および図3 に示す 3 つの生物学的複製物について行った。.P 値: >0.05 (ns)、<0.05 (*)、<0.005 (**)、および <0.0005 (***)。(D)トレックスRTA BCBL-1細胞からのRNAの代表的なノーザンブロットは、ドキシー後24h。ボックスとウィスカーのプロットは、10 パーセンタイルと 90 パーセンタイルを表します。フィギュアは、クリエイティブ・コモンズ・アトリビューション・ライセンスの下で、ヴァレリー、ウィズーズ、および同僚15からのわずかな変更で転載しました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
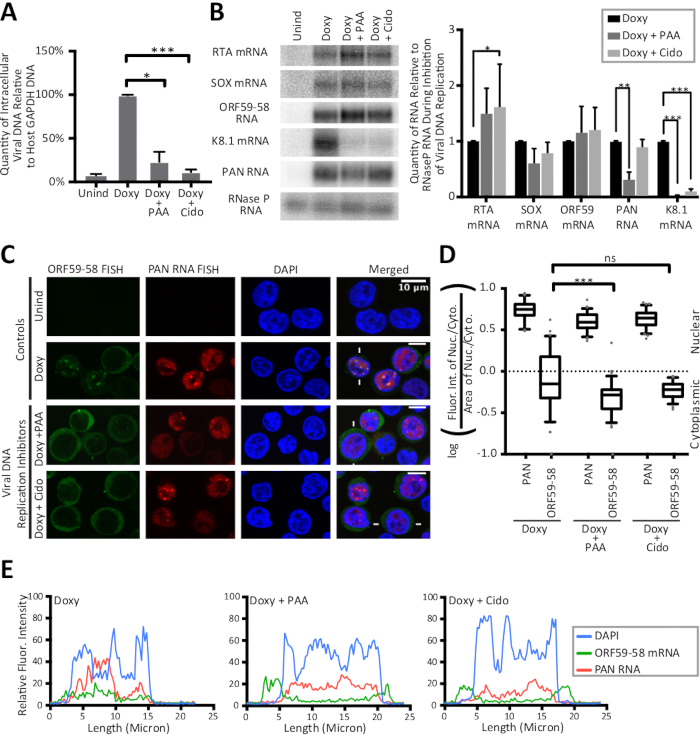
図3:ヌクレオ細胞質比の線の痕跡と計算は、ウイルスDNA複製の阻害時に早期溶解性ORF59-58転写物の細胞質への強いシフトを明らかにする。TREx RTA BCBL-1細胞を薬物なしで24時間治療した(Unind)、ドキシサイクリンのみ(ドキシ)、またはドキシサイクリンおよびヘルペスウイルスDNA複製の1阻害剤、ホスホノ酢酸(ドキシ+PAA)またはシドフォビル(ドキシ+シド)。パネル(A-C)は、3つの生物学的反復から収集されたサンプルからのデータを示す。(A)ウイルスDNA複製の阻害中のウイルス細胞内DNAのqPCR値を、宿主細胞GAPDH遺伝子のプロモーターDNAの量に正規化した。(B) ノーザンブロット(左)および定量(右)は、ウイルスDNA複製の阻害中の総RNAレベルを示す。すべてのRNAの誘導されていないレベルは検出できなかった。(C) ウイルスDNA複製の阻害時にウイルスORF59-58転写物(緑色)およびPAN RNA(赤色)の代表的なFISH画像。DAPI(青)は核染色でした。(D)(C)で表される細胞の蛍光強度の定量化(n=75各)を生物学的三量子で行った。(E) (C) で白線で示される細胞間で描かれた線に沿った蛍光強度が示されている:DAPI(青)、PAN RNA(赤)、およびORF59-58 mRNA(緑色)。P 値: >0.05 (ns)、<0.05 (*)、<0.005 (**)、および <0.0005 (***)。本研究におけるすべてのオリゴヌクレオチドの配列は表1に示されている。ボックスとウィスカーのプロットは、10 パーセンティルと 90 パーセンティルを表します。フィギュアは、クリエイティブ・コモンズ・アトリビューション・ライセンスの下で、ヴァレリー、ウィズーズ、および同僚15から転載したものです。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| 北オリゴス | |||||||
| オリゴいいえ | 遺伝子 | シーケンス | 遺伝子内の位置 | リファレンスNC009333.1ゲノムを持つ位置(数値は方向または鎖を反映しない) | |||
| コルフ50 | KSHV RTA/ORF50 | CGCATTGCGGTTTGTGTTTTTTGG | 1284 から 1309 | 73936 から 73961 | |||
| JBW249 | KSHVソックス/ORF37 | タクトガッカッカアカカカカッカッカッカッカッカッカック | 262 から 291 | 57633 から 57662 | |||
| tkv379 | KSHV ORF59-58 | TGGAGTCCGGGGッタガトグアッガット | 941 から 967 (ORF59 ORF) | 95879 から 95905 | |||
| tkv13 | KSHV K8.1 | アガガッタッタガグクカガッガッガットGTGCGAAT | 16~57 | 76029 から 76070 | |||
| SB2 | KSHV パン RNA | アカアアトックカククトTGTGC | 664 から 687 | 29496 から 29519 | |||
| ナース P | ヒューマン ルナス E | TGGGCGGAGGAggTAGTCTG | 319 から 339 | N/a | |||
| フィッシュプローブ | |||||||
| オリゴいいえ | 遺伝子 | シーケンス | |||||
| SB2 | KSHV パン RNA | アカアアトックカククトTGTGC | 664 から 687 | 29496 から 29519 | |||
| SB85 | KSHV パン RNA | CGCTCTTTTTTTカツットカツット | 373 から 392 | 29205 から 29224 | |||
| SB88 | KSHV パン RNA | GTGAAGCGGCAGCCAAGGTGACTGG | 1~22 | 28830 から 28854 | |||
| tkv13 | KSHV K8.1 | アガガッタッタガグクカガッガッガットGTGCGAAT | 16~57 | 76029 から 76070 | |||
| tkv14 | KSHV K8.1 | タガタッタグッガグトグトグトグトッグ | 377 から 414 | 76390 から 76427 | |||
| tkv15 | KSHV K8.1 | GTAAGGTTACGCTTTATCCカッカッカッカッカッカッカッガグタタック | 461~500 | 76474 から 76513 | |||
| tkv16 | KSHV K8.1 | ガカカグッカッカッカッカッカッカッカッカッカッタグ | 688 から 725 | 76701 から 76738 | |||
| tkv376 | KSHV ORF59-58 | TAATGTGTTCATTGCCCCCTTT | 54~79 | 96767 から 96792 | |||
| tkv377 | KSHV ORF59-58 | GCCGATCCGTGCACTACTCCCCGGTTTT | 93 から 122 | 96724 から 96753 | |||
| tkv378 | KSHV ORF59-58 | アグクタットGCGCGCGCTACTACTCGCA | 300~329 | 96517 から 96546 | |||
| tkv379 | KSHV ORF59-58 | TGGAGTCCGGGGッタガトグアッガット | 941 から 967 | 95879 から 95905 | |||
| tkv380 | KSHV ORF59-58 | アアガグトガッタグタグクト | 1289 から 1315 | 95531 から 95557 | |||
| tkv381 | KSHV ORF59-58 | AAACACTGCTGACGCGカガットック | 1423 から 1450 | 95396 から 95423 | |||
| tkv382 | KSHV ORF59-58 | CTGTGTGTGTACTATGGCGGCGCGCGC | 1571 から 1602 | 95244 から 95275 | |||
| tkv383 | KSHV ORF59-58 | ググッチャゲットカグスタッタグガアアクチャッグ | 2136 から 2173 | 94673 から 94710 | |||
| ステラリス | ギャップド | ステラリスによるプレメイド | N/a | N/a | |||
| qPCR プライマー | |||||||
| tkv458 | GAPDHプロモーター | CTGCACCAACTGCTTタグ | N/a | N/a | |||
| tkv459 | GAPDHプロモーター | GTCTCTGGGGGGGTGGガット | N/a | N/a | |||
| tkv319 | KSHV ORF39 (グラム) | GTGAGGTGCTTCGCTGAGTT | N/a | 60075 から 60094 | |||
| tkv320 | KSHV ORF39 (グラム) | CCTGGGTCAAGGTGTTTTTTTTTTT | N/a | 60218 から 60237 | |||
| RT-qPCR プライマー | |||||||
| tkv 455 | K8/K-bZIP フォワード RT qPCR プライマー | CGAAAgcaaggcagataCG | 655 から 673 | 75603 から 75621 | |||
| tkv 456 | K8/K-bZIP リバース RT qPCR プライマー | GCCATTGTTCCTTTTTTT | 755 から 774 | 75703 から 75722 | |||
| tkv 457 | K8/K-bZIP リバース RT qPCR プライマー用スプライス | カタグストグCGAAG | 871 から 888 | 75819 から 75836 | |||
| JBW479 | ヒューマン RNase P フォワード | アグットガアカガクタクカッグ | 238 から 257 | N/a | |||
| JBW480 | ヒューマン RNase P リバース | GCAGGAggGaGAGTAGTCTGAA | 317 から 336 | N/a | |||
表1:本書の分析で使用されるすべてのオリゴヌクレオチド。表1は、米国微生物学会の許可を受けて、Vallery et al. 15.のクリエイティブ・コモンズ・アトリビューション・ライセンスに基づいて複製された。
ディスカッション
このレポートに記載されているプロトコルは、異なる細胞タイプに適応することができ、モノクローナルおよびポリクローナル一次抗体の両方を使用してIFを用いた二重RNA FISHおよびRNA FISHのステップが含まれる。調製されたスライドは、通常、共焦点顕微鏡で画像化されるが、画像化は、増加した抗体濃度および異なる取り付け媒体の修飾後にSTED(刺激放出枯渇)顕微鏡で行うことができる。個々の細胞の増強分析のために、このプロトコルで調製されたサンプルは、Borahおよび同僚16によって示されているように、適度な変化を伴う細胞選別器またはフローサイトメーターによってソート、画像化、および分析され得る。ただし、このプロトコルは生細胞イメージングには採用できません。
詳細な定量法は、観察バイアスを排除し、潜在的な核細胞質シフトを検証するのに役立ちます。核細胞質比はまた、生体分子が特定の細胞内の局所化に均等に分散することから局所化するように調整する場合にも特定する。ここで示す結果は半定量であり、プロトコルは定量を強化する方法を概説します。核細胞質比と線跡の強さ(ステップ4)は、蛍光ビーズを強度制御(ステップ3.13)として使用し、核ラミンA/C用のような明確な細胞マーカーの使用に依存する。現時点では、KSHV ウイルス複製コンパートメントには明確な境界マーカーは存在しません。いずれにせよ、この計算は、適切なマーカーを使用して他の細胞下コンパートメントに拡張することができます。
このレポートで詳述されているプロトコルの主なハードルは、特定のトランスクリプトに対する FISH 戦略の開発です (ステップ 1)。成功は、反感覚オリゴヌクレオチドの豊富さと結合強度に依存しています。特定の転写物に対する特異性は、ウイルスゲノム中にオーバーラップする開いた読み取りフレーム(ORF)の存在によってさらに困難になる。したがって、ウイルス転写物は、多くの場合、特にヘルペスウイルスの場合には、同じゲノム領域からの他のウイルス転写物と配列類似性17を有する。多くの場合、FISH戦略の開発は、より豊富なトランスクリプトを利用する必要があります。FISH信号の欠如をトラブルシューティングするには、ユーザーはU2 snRNA FISH戦略を使用してFISHプロトコルを実行し、ヒト細胞および試薬の調製技術が適切であることを確認する必要があります。同様に、KSHV PAN RNA FISH戦略は、KSHV感染細胞における溶解活性化を確認することができる。反感オリゴヌクレオチドによる結合をトラブルシューティングするために、著者らはいくつかの反感オリゴヌクレオチドを開発することを推奨する。すべてが失敗した場合、図 2の GAPDH FISH 戦略と、Vallery、Withers、および同僚15の使用によって示されているように、商用オプションが利用可能です。
細胞と細胞内の境界を定義するより強力なアルゴリズムは、さらに定量バイアスを排除します。一部の分析画像処理ソフトウェアでは、セル、核などの境界を設定できますが、決定的なマーカーが必要です。ウイルス複製コンパートメントなどの異常な細胞形態は、このようなソフトウェアでは困難であり、将来の開発に向けての課題です。さらに、ここで説明する定量方法は、セルの1つの光学スライス(2D画像解析)に限定される。3D画像取得は18可能であるが、定量的な3D画像解析の将来的な開発は、ウイルス複製コンパートメントの時空間調節に関するさらなる洞察を提供するかもしれない。
開示事項
著者は、開示する利益相反を持っていません。
謝辞
データ分析に関するアドバイスをいただいたジョナサン・ローデンフェルス、カジミエシュ・ティコウスキー、ジョアンナ・B・ウィーザーズに感謝します。また、G.ヘイワードの抗SSB抗体に感謝します。この研究は、国立衛生研究所(TKV)およびNIH助成金(CA16038)(JAS)からの助成金T32GM007223およびT32AI055403によって支援されました。JASはハワード・ヒューズ医学研究所の研究者です。図1-3および表1は、次の出版物からクリエイティブ・コモンズ・アトリビューション・ライセンスの下で米国微生物学会の許可を受けて複製されました:ヴァレリー、T.K.、ブリザーズ、J.B.、アンドー、J.A.、スタイツ、J.A.カポジの肉腫関連核病巣におけるヘルペスウイルスmRNA蓄積は、ウイルスDNA複製およびウイルス非コードポリアデデン化核RNAの影響を受ける。ウイルス学のジャーナル。92 (13), doi:10.1128/JVI.00220-18, (2018)
資料
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
参考文献
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81(2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300(2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved