Method Article
Изучение рибонуклеотид включение: Обнаружение Strand конкретных Ribonucleotides в геноме дрожжей и измерения рибонуклеотид индуцированного мутагенеза
В этой статье
Резюме
Ribonucleotides относятся к числу наиболее распространенных неканонических нуклеотидов, включения в Геном эукариот ядерных репликации ДНК. Если удалены не должным образом, ribonucleotides может вызвать повреждение ДНК и мутагенез. Здесь мы представляем две экспериментальные подходы, которые используются для оценки обилие рибонуклеотид включения в ДНК и мутагенных эффектов.
Аннотация
Наличие ribonucleotides в ядерной ДНК было показано, быть источником геномной нестабильности. Степень включения рибонуклеотид могут быть оценены щелочной гидролиз и электрофорез геля, как РНК, является очень восприимчивы к гидролизу в щелочных условиях. Это, в сочетании с юга блот анализ может использоваться для определения местоположения и прядь в который были включены ribonucleotides. Однако эта процедура является только полуколичественного и не может быть достаточно чувствительным, чтобы обнаружить небольшие изменения в содержании рибонуклеотид, хотя Южная помарка стренги конкретных зондирующего улучшает чувствительность. Как мера одного из самых ярких биологических последствий ribonucleotides в ДНК спонтанного мутагенеза могут быть проанализированы с использованием пробирного вперед мутации. Используя соответствующие репортер генов, редкой мутации, которые приводит к потере функции может быть отдельных и общих и конкретных мутаций ставки может измеряться путем объединения данных из колебаний эксперименты с секвенирования ДНК гена репортера. Assay колебания применимо рассмотреть широкий спектр мутагенных процессов в конкретных генетический фон или условий роста.
Введение
Во время эукариотических ядерных репликации ДНК ribonucleotides включены в геноме всех трех основных replicases ДНК, ДНК полимеразы (Pols) α, ε и δ1,2. РНКазы H2-зависимых рибонуклеотид иссечение ремонт (3RER) удаляет большинство этих встроенных ribonucleotides.
Рибонуклеотид в ДНК подвержен гидролизу, как 2' гидроксильной группы части молекулы сахара может атаковать прилегающих Фосфодиэфирная связь, выпустив один конец с 2' - 3' циклических фосфата и другой с 5'-OH-4. Щелочная среда может значительно ускорить эту реакцию. Таким образом гидролиз встраиваемых ribonucleotides во время инкубации в основной раствор вызывает фрагментацию геномной ДНК, которые могут быть визуализированы щелочной агарозы электрофорез5. ДНК может быть передана мембраны и исследован, Южная помарка анализа с помощью зондов нити конкретные, которые позволяют выявлять щелочно чувствительных участков, вызванные ribonucleotides, включены в зарождающейся ведущих - или теплоизоляции strand ДНК, соответственно.
В клетках дрожжей не хватает активности РНКазы H2 удаление ribonucleotides может быть инициировано когда Топоизомераза I (Top1) Никс ДНК на встраиваемых рибонуклеотид6,7. Однако когда Top1 расщепляет на стороне 3' рибонуклеотид, это генерирует 5'-OH и 2' - 3' циклических фосфат ДНК концы которые огнеупоров для religation. Неспособность ремонт, или аномальным обработка этих «грязные концы» может привести к геномной нестабильности. Кроме того если разрез происходит внутри повторяющихся последовательностей ДНК, процесс восстановления может привести к удаления мутации. Это особенно проблематично для тандемные повторы, где короткие удаления (из между 2 и 5 пар оснований) обычно наблюдается в клетках РНКазы H2-недостаточным. Top1-зависимых пагубные последствия в отсутствие дрожжей РНКазы H2 активности усиливаются в прослушивающем для включения рибонуклеотид во время зарождающейся ведущих стренги синтеза мутант ε ДНК-полимеразы (pol2-M644G).
Обработка ribonucleotides в ДНК приводит к спонтанной мутации и этот мутагенеза можно обнаружить с помощью соответствующих репортер генов, выбрав для сопровождающих фенотипические изменения. Колебания тест или Лурия и Дельбрюк эксперимент является одним из наиболее часто используемых методов для измерения скорости спонтанной мутации, с помощью выбираемых репортер генов8,9. В дрожжей URA3 и CAN1 генов может использоваться как журналистам в assay вперед мутации, которая позволяет для обнаружения всех типов мутаций, приводящих к потере функции гена. Уровень спонтанной мутации оценивается как средний показатель наблюдается для нескольких параллельных культур, начал с одной колонии без мутации в гене репортер целевой. Дрожжи, RNase H2-недостаточным напрягаться, такие, как rnh201Δ имеет умеренно повышенной скоростью целом спонтанной мутации, во многом вызванные повышенной заболеваемости 2-5 bp удалений в тандеме повторить последовательности. Таким образом чтобы полностью характеризуют мутагенных эффектов ribonucleotides в геноме, скорости конкретной мутации должны определяться. В этом случае URA3 или CAN1 репортер генов может быть усиливается и последовательности для определения типа и местоположения мутаций, и скорости конкретной мутации могут быть рассчитаны. Компиляция мутации, определены в нескольких независимых URA3 или CAN1 мутантов, затем может использоваться для создания спектра мутаций.
протокол
1. щелочной гидролиз и специфичные для Strand Южная помарка (рис. 1)
- Геномная ДНК изоляции и рост клеток
- Изолируйте дрожжи геномной ДНК с помощью дрожжей ДНК очистки комплект следуя инструкциям производителя. Использование культуры, которые находятся в экспоненциальной фазе роста между (оптической плотностью 0,5) и 1. Ресуспензируйте окончательный Пелле ДНК в 35 мл 1 x TE буфера (10 мм трис-HCl, pH 7.5, 1 мм Этилендиаминтетрауксусная кислота (ЭДТА)).
- Добавьте 1 mL 5 мг/мл РНКазы A ДНК и Инкубируйте 30 мин при температуре 37 ° C.
- Осадок ДНК, с использованием 0.1 объемов ацетат натрия 3 M (рН 5.2) и 2,5 томов 100% этанола для 20 мин на льду.
- Центрифуга на 16000 x g 20 мин при 4 ° C. Удалите с помощью пипетки супернатант.
- Помойте лепешка дважды с 500 мл 70% этанола. Удалите с помощью пипетки супернатант.
- Сухие гранулы на скамейке и Ресуспензируйте 35 мл 1 x TE буфера.
- Quantitate ДНК, используя dsDNA BR Assay комплект fluorimeter (Таблица материалов).
- Осадок 5 мг от каждого образца, используя 0.1 томов 3 M ацетат натрия, рН 5.2 и 2.5 тома 100% ДНК EtOH для суммарный объем 200 мл (добавить H2O, если необходимо, дополнительные 5 мг ДНК необходима, если необходим контроль нейтральных гель). Инкубируйте на льду за 20 мин.
- Центрифуга на 16000 x g 20 мин при 4 ° C. Удалите с помощью пипетки супернатант.
- Сухие гранулы на скамейке. Ресуспензируйте ДНК в 14 мл H2O.
- Щелочной гидролиз и гель электрофорез
- Добавить 6 мл 1 м Кох и Инкубируйте на 55 ° C за 2 ч.
- Проинкубируйте образцы на льду на 5 мин.
- Добавить 4 мл 6 x щелочная ДНК загрузки буфера: 300 мм Кох, 6 мм ЭДТА, рН 8,0, 18% polysucrose (Таблица материалов), 0,15% бромкрезоловый зеленый и 0,25% ксилола cyanol FF.
- Литой щелочного гель, который состоит из агарозы 1%, 50 мм NaOH, ЭДТА 1 мм, pH 8.0. Используйте расческу, что позволяет быть загружены на переулок 24 мл образца ДНК.
- Побегите гель при комнатной температуре в 1 x буфер электрофореза щелочной: 50 мм NaOH, 1 ЭДТА, рН 8.0. Включать соответствующий маркер размера ДНК.
- Спин вниз трубы кратко и загрузить весь 24 мл образца в щелочной агарозном геле. Electrophorese 30 мин в 30 V, а затем 18-20 h 10 V.
- Нейтрализовать гель для 2 инкубаций 45 мин при комнатной температуре с нежным агитации в нейтрализации буфера I: 1 М трис-HCl, 1,5 М NaCl.
- Пятно ДНК путем замачивания нейтрализованы гель в 200 мл воды, содержащие 1/10000 разрежения золото SYBR пятно втечение 2 ч при комнатной температуре с нежным встряхивания.
- Визуализация с помощью УФ transilluminator ДНК. Увеличение щелочно чувствительность приводит к появлению небольших фрагментов ДНК, указывающее плотность неустраненный ribonucleotides в ДНК5.
- Южный передачи
- Передать ДНК из щелочного гель мембрана положительно заряженный нейлон капиллярность10. Пример того, как можно настроить этот перевод предоставляется в Sambrook и Рассела ручной10.
- Замочите щелочного гель для 15 мин при комнатной температуре в щелочной передачи буфера: 0.4 М NaOH, 1 M NaCl.
- Влажный нейлон мембраны (нарезанные по размеру геля) кратко с водой, а затем замочить на 5 минут в щелочной передачи буфера.
- Настройка трансферной платформе, инвертирование 3 стеклянные стаканы (100 мл) в стакан для выпечки. Это блюдо должно быть немного больше, чем размер геля агарозы. Установите стеклянную пластину поверх них.
- Драпировка 2 больших бумажки 3 мм ЦПЧ хроматографии (нарезанные по размеру ширины гель плюс больше сторон, которые могут драпировка над краями в буфер водохранилище) над стеклянной пластиной.
- Залить щелочного буфера передачи бумажной хроматографии и наполовину заполнить стеклянную посуду. Зачищают пузыри с помощью пипетки стеклянные 5 мл.
- Гель агарозы место на вершине, снова разглаживания пузырьков. Окружают всем краям геля с парафина. Налейте небольшое количество щелочного буфера передачи на геле.
- Место предварительно замоченные нейлон мембраны поверх геля. Зачищают пузыри.
- Мокрые 2 бумажки нарезанные по размеру геля агарозы. Разместите их на вершине мембраны и сгладить любые пузыри.
- Место большой стек бумажных полотенец (вырезать по размеру чуть больше, чем геля агарозы) поверх передачи сэндвич. Осторожно поместите стеклянную пластину поверх бумажные полотенца. Добавьте объект взвешенный на вершину (книга с массой 400 g работает хорошо).
- Пусть передачи перейти на ночь при комнатной температуре.
- Тщательно разобрать стек передачи и замочить мембрана для 15 мин при комнатной температуре в нейтрализации буфера II: 0.5 М трис-HCl, рН 7,2, 1 M NaCl.
- Crosslink ДНК заряженных нейлон мембраны с помощью УФ сшивателя. Мембрана в крест-компоновщик и использовать параметр autocrosslink.
- Подготовка Южной зонд
Примечание: Подход, описанный здесь, чтобы обнаружить щелочно чувствительных участков в зарождающейся ведущих-против отстает нити ДНК на основе протокола ранее опубликованы11. Для нашего эксперимента зондирование выполняется на URA3 Репортер ген, который был вставлен в одном из двух направлений (или1 или OR2) рядом с ARS306 репликации происхождения на хромосоме III (рис. 3A). Присутствие щелочно чувствительных участков в геномной ДНК дрожжей является показателем рибонуклеотид включения, что позволяет использовать ribonucleotides как биомаркеров ДНК-полимеразной активности12,13,14. Этот подход, с помощью целевого объекта последовательности ДНК, прилегающих к эффективной ARS306 происхождение репликации, должны поддаваться других геномной мест и генов-мишеней.- ПЦР усиливают 520-база сегмент пара гена URA3 репортер, используя следующие пары праймера: URA3-F1 (5´-GCTACATATAAGGAACGTGCTGC) и URA3-R1 (5´-CTTTGTCGCTCTTCGCAATGTC).
- Выполнить с помощью 10-20 нг дрожжей геномной ДНК как шаблон, 4 мкл каждого праймера (10 мкм бульон), 10 мкл 10 x Ex Taq буфера (мг2 + плюс), 8 мкл dNTP смеси (по 2,5 мм), 0.5 мкл из Ex полимераза дна Taq реакции PCR (5 U/мкл) и окончательного громкость до 100 мкл с H2O.
- Запустите следующую программу ПЦР: 95 ° C за 5 мин; 30 циклов 95 ° C за 1 мин, 55 ° C за 1 мин., 72 ° C на 2 мин; 72 ° C для 10 мин и удерживайте при 4 ° C.
- Очистить продукт PCR, используя комплект для очистки ПЦР и элюировать ДНК, используя 50 мл H2O. Это теперь может использоваться как шаблон в radiolabeling реакции.
- Используйте следующие Праймеры для нити конкретных radiolabeling: использование URA3-A (5´-CTCATCCTAGTCCTGTTGCTGCC) для визуализации ДНК, которая anneals к зонду A. Это соответствует зарождающегося ведущих нити ДНК при URA3 в OR2 и tnascent, отставание нити ДНК, когда URA3 в или1. Использовать URA3-B (5´-CAGTACCCTTAGTATATTCTCCAG) для визуализации зарождающейся ДНК, которые anneals к зонду б. Это соответствует зарождающегося ведущих нити ДНК, когда URA3 в или1 и зарождающейся отстающих нити ДНК при URA3 в OR2 (рис. 3).
- Выполнить radiolabeling реакции, с помощью 10-20 нг усиленный URA3 фрагмент как шаблон ДНК, 2 мкл грунтовка (10 мкм бульон), 5 мкл 10 x Ex Taq буфера (мг2 + плюс), 4 мкл дНТФ 2,5 мм (минус дЦТФ), 5 мкл (50 МККИ) -32P-дЦТФ , 0.5 мкл ExTaq ДНК-полимеразы (5 U/мкл) и окончательного громкость до 50 мкл с H2O. Run следующие программы для radiolabeling: 94 ° C за 5 мин; 25 циклов 94 ° C за 1 мин, 55 ° C за 30 s, 72 ° C в течение 1 мин; 72 ° C за 5 мин и удерживайте при 4 ° C.
- Для удаления неинкорпорированных радиоактивных лейбл используйте столбец G-25 спина. Прежде чем добавлять radiolabeled образца, центрифуга столбце 1 мин на 720 x g и removethe буфера, закупорить. Загрузите продукт radiolabeled реакции и центрифуги для 2 мин на 720 x g элюировать помечены зонд.
- Инкубируйте на 95 ° C за 5 мин до денатурировать зонда непосредственно перед гибридизации. Прохладный кратко на льду.
- Южный гибридизации
- Roll увлажненный мембраны в гибридизации трубку и добавить 25 мл свежеприготовленные гибридизации буфера: 0.5 M натрия фосфат буфера, pH 7.2, 7% додецилового сульфата натрия (SDS), 1% бычьим сывороточным альбумином (БСА). Инкубируйте на 65 ° C для 1-2 ч с вращением в печь гибридизации.
- Тщательно слейте раствор и добавить 25 мл буфера гибридизации плюс радиоизотопами зонд. Инкубируйте на 65 ° C для 16-18 ч с вращением.
- Слейте раствор в контейнер радиоактивных отходов. Выполните 5 моет 5 мин при комнатной температуре с фосфат-SDS стирки решение I: 40 мм натрия фосфат буфера, рН 7,2, 5% SDS, 0,5% BSA, 1 мм ЭДТА, рН 8.0.
- Выполнить 2 моет 15 мин при 65 ° C с фосфат-SDS стирки решение II: фосфат натрия буфера 40 мм, pH 7.2, 1% SDS, 1 мм ЭДТА, рН 8.0.
- Удаление мембраны из трубки гибридизации с помощью пинцета и место в ограничитель открыл пластиковый рукав. Обложка и подвергать изображений пластины. Попробуйте несколько различных воздействия раз до 4 d 4 h.
- При необходимости, полосы и повторно зонд пятно с помощью различных зонда примерно 3 - 4 раза. Раздеться, Проинкубируйте мембрану на 2 ч при температуре 65 ° C 25 мл 50% формамида, 2 x SSPE решения.
Примечание: 20 x SSPE Стоковый раствор: 3 M NaCl, 0,2 М NaH2PO4, 0,02 М ЭДТА, рН 7,4. - Слейте раствор в контейнер радиоактивных отходов. Выполните 2 моет 15 мин при температуре 65 ° C в фосфат-SDS мытья решения II.
- Проверьте мембраны с Гейгера и повторите зачистки протокол, если сигнал остается.
- Выполнение предварительно гибридизации и гибридизации, как описано выше.
2. Измерение мутации темпы и специфику штаммов S. cerevisiae
- Подготовить культура клетки
- Прививать единую колонию (здоровый дрожжи клетки занимает 2-3 дня при 30 ° C в форме надлежащего размера колоний) вырос на агаре YPD дополнена аденин 100 мг/мл 5 мл YPDA бульон с аденином 250 мг/мл (добавок аденин необходимо, только если штамм используется является аденин auxotroph). Растут в шейкер инкубатор или на вращателе при 30 ° C до тех пор, пока культура достигает насыщения (36-48 h для деформации без дефекта серьезный рост).
- Спин вниз клетки на ~ 2000 x g за 5 мин.
- Отменить супернатант и вымыть клетки с 5 мл стерильной воды.
- Ресуспензируйте клетки в 500 мл стерильной воды.
- Разбавление культуры клеток и обшивка
- -Выбор элементов управления разбавляют клетки в 96-луночных тарелку с коэффициентом 1,600,000 и пластины 100 мл каждой культуры на полную тарелку (COM).
- Чтобы выбрать для ura3 мутантов, тарелка 100 мл неразбавленного клеток на 5-фр пластину для штамма (WT) одичал тип. Разбавьте клетки соответственно, если уровень мутации штамма интерес, как ожидается, будет выше, чем в WT штамм.
- Чтобы выбрать для can1 мутантов, разбавленные клетки 5 раз и пластины 100 мл на canavanine содержащие пластину (CAN) для штамма WT. Коэффициент разрежения — 0,5.
- Инкубируйте пластины при 30 ° C до формы колонии дрождей. Для штаммов без роста дефект 2-3 дня обычно достаточно для COM и может пластины и 3-5 дней для тарелок 5-фр. Чтобы сравнить скорости мутации между разными штаммами, лучше выбрать тот же день роста для подсчета колоний. Храните пластины, готовы быть подсчитанным в холодной комнате (~ 4 ° C).
- Расчет ставки спонтанной мутации
- Количество видимых колоний на каждой пластине и принять к сведению колонии чисел
- Рассчитать уровень мутации, Дрейка формуле: μ = f ÷ ln (μNt). f — частота мутаций, определяется количество мутантов, деленное на количество клеток в окончательном культуре. NT-это количество клеток в культуре окончательный и μ является уровень мутации. Это уравнение может быть решена путем итеративный метод как метод Ньютона-Рафсона. Существуют другие методы оценки, включая Леа и Coulson тест, чтобы вычислить стоимость мутации15.
- Вычислите при условии нормального распределения журнала доверительный интервал utation ставок отдельных изолятов. 95% доверительный интервал наиболее часто используется.
- Анализ спектра мутаций
- Выбрать один колонии от каждого 5-фр или может пластины и обновить на YPDA плите.
- Выполнять колонии PCR усиливает URA3 или CAN1 гена и последовательности с целью выявления мутаций, с использованием следующих грунтовок: URA3-F (5'-CGCATATGTGGTGTTGAAGAA-3') и URA3-R (5'-TGTGAGTTTAGTATACATGCA-3') для обоих амплификации PCR и секвенирование гена 800 URA3 ВР; CAN1-F (5'-CTTCTACTCCGTCTGCTTTC-3') и CAN1-R (5'-CAGAGTTCTTCAGACTTC-3') для усиления и CAN1-AR (5'-TGAAATGTGAAGGCAGCGTT-3'), CAN1-9R (5'-CCTGCAACACCAGTGATA-3'), CAN1-10R (5'-GAGGATGTAACAGGGATGAAT-3') и (CAN1-BR 5'-CGGTGTATGACTTATGAGGG-3') для секвенирования 1800 bp CAN1 Репортер ген.
- Чтобы выполнить колонии PCR, Ресуспензируйте единую колонию в 5 мкл H2O и использовать 1 мкл в качестве шаблона. Добавить 1 мкл каждого из грунтовки (5 мкм бульон), 2 мкл (мг2 + плюс) 5 x KAPA2G буфера, 0.5 мкл dNTP (2,5 мм), 0.5 мкл из KAPA2G быстро ДНК-полимеразы, 12 мкл H2O.
- Для амплификации гена URA3 , запустите следующую программу ПЦР: 95 ° C за 5 мин; 5 циклов 95 ° c 10 s, 61 ° C 15 s, 72 ° C за 30 сек; 35 циклов 95 ° C 10 s, 61 ° C 15 s, 72 ° C за 30 сек; 72 ° C для 2 мин и удерживайте при 4 ° C. Для амплификации гена CAN1 , запустите следующую программу ПЦР: 95 ° C за 5 мин; 5 циклов 95 ° c 10 s, 61 ° C 15 s, 72 ° C для 45 s; 35 циклов 95 ° C 10 s, 61 ° C 15 s, 72 ° C для 45 s; 72 ° C для 2 мин и удерживайте при 4 ° C.
- Совместите результаты секвенирования последовательности ссылку, используя такие программы, как DNASTAR Seqman Pro или Sequencher для выявления мутаций.
- Сбор данных мутации мутации типа (рис. 4) или мутация местоположение (рис. 5). Вычислить конкретные мутации ставки по частоте мутаций (количество событий, наблюдается делится на количество мутантов виртуализированных) умножается на общий уровень мутации.
Результаты
Лечение геномной ДНК щелочью, следуют щелочного гель-электрофорез позволяет для полуколичественного определения фрагментацию ДНК из-за обилия стабильно включены ribonucleotides. Рисунок 2 показывает изображения гель дрожжей геномной ДНК относились с или без Кох5. M644L вариант Pol2, каталитическая субъединица Polε, снизил способность включать ribonucleotides, в то время как мутант M644G включает в себя более ribonucleotides чем WT полимеразы. Как подробно говорится в протоколе щелочного гель может далее зондируемой стренги конкретных Южная помарка анализа. С ведома расположения исследуемого геномной узла по отношению к его соседних происхождение, мы может выборочно зонд ribonucleotides, включены в зарождающейся ведущих или отставание пряди. Рисунок 3 показывает результаты Южная помарка зондирующего URA3 Репортер ген вставлен в два противоположных направления рядом начале стрельбы репликации происхождения, ARS30616. С помощью ведущих стренги специфичные зонды, мы наблюдаем, шаблон фрагментацию ДНК, вызванные щелочно расщепление на ribonucleotides в зарождающейся ведущих нити ДНК в штамм WT и штаммов, выражая варианты и полимеразы α, δ, ε, которые прослушивающем для Включение рибонуклеотид.
С помощью колебания эксперимента, мы можем измерить скорости мутации штаммов дрожжей, содержащие гены репортер. Рисунок 4 показывает скорости мутации на Ура3 и CAN1 локусов штаммов, выражая полимеразы WT или pol2-M644G -мутант с или без функциональных РНКазы H2. Отсутствие РНКазы H2 вызывает умеренное увеличение в общей скорости мутации в как фон. Однако при более внимательном изучении специфики мутации показывает, что в штаммов хватает РНКазы H2 (рис. 5), существует сильное увеличение частоты мутаций для коротких удалений, особенно в тандеме повторить последовательности. Рисунок 5A сравнивает мутации специфичность в WT и rnh201Δ штаммов и Рисунок 5B отображает мутационного воздействия дальнейшего повышенных рибонуклеотид включения в зарождающейся ведущих стренге дна, Поль ε.
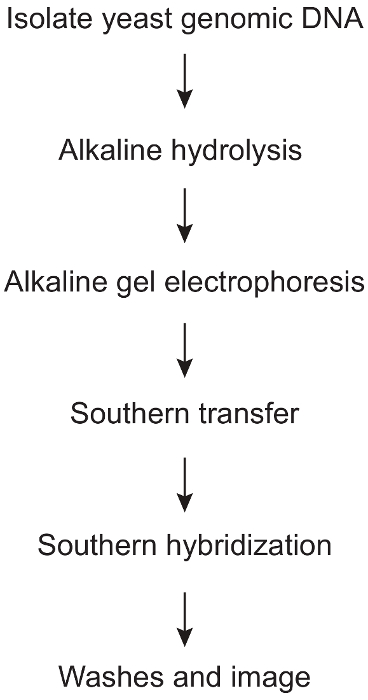
Рисунок 1: протокол этапы в strand конкретных обнаружения щелочно чувствительных участков, вызванные рибонуклеотид включения в геномной ДНК дрожжи. Обзор различных основных шагов в данной процедуре, начиная с дрожжи геномной ДНК изоляции и продолжает путем анализа окончательного Южная помарка. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 2: образ представителя результаты полученные щелочной-геля агарозы. Эта цифра была адаптирована из Ник McElhinny, С.а. и др. 5. дрожжи геномной ДНК от штаммов различных генотипов лечили KCl (нейтральный) или Кох (щелочной) и затем разделены на нейтральной или щелочной агарозном геле. «Вт» и «M644G» укажите статус Pol ε. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 3: Strand конкретных зондирование щелочной агарозном геле, Южная помарка. Эта цифра была адаптирована из Уильямс, J. S. et al. 13. (A) radiolabeled зонды отжига для зарождающейся ведущих стренге дна в пределах URA3 Репортер ген, вставленный в двух противоположных направлениях (или1 и OR2) недалеко от ARS306 . (B) представитель результаты Южный blotting с помощью зарождающейся ведущих стренги специфичные зондов. Отображаются зондирующего результаты для зарождающейся ведущих strand в штамма с WT-полимераз, или pol1-L868M, pol2-M644G или pol3-L612M вариант штаммы с или без РНКазы H2. POL1, POL2 и POL3 гены кодировать каталитического субблоков Pols α, ε и δ, соответственно. Варианты более разнородный рибонуклеотид включения5,12,17. (C) количественной оценки радиоактивных сигнала на долю общего числа в каждом Лейн (b). Значения выражаются в процентах от общего числа. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 4: показатели спонтанной мутации в штаммов дрожжей. Эта цифра была адаптирована из Ник McElhinny, С.а. и др. 5. репортер генов URA3 и CAN1 использовались в assay вперед мутации для измерения скорости мутации указанных штаммов. URA3 репортер находится в «ориентация 2» (OR2, рис. 3A). 95% доверительный интервал (ди) включен для каждого измерения. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 5: спектры мутации гена репортера URA3-OR2. Эта цифра была адаптирована из предыдущих публикаций5,18,19,20. Тип позиции и мутации, наблюдается в каждой независимой 5-фр устойчивостью колонии, выбранных в assay вперед мутации изображены в 804 bp URA3 кодирующая последовательность. Буквы указывают базы замен, открытые треугольников указывают один базовый удалений, закрытые треугольников указывают один базовый вставок и сплошной линии показывают короткие удалений. (A) мутация спектры в WT и rnh201Δ штаммов. Красные метки выше последовательности являются мутации, наблюдается в Вт, в то время как синие метки ниже последовательность в штамм rnh201Δ . (B) спектры мутации в pol2-M644G и rnh201Δ pol2-M644G штаммов. Красные метки выше последовательности являются мутации в pol2-M644G , а синий ниже последовательности для pol2-M644G rnh201Δ штаммов. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
Здесь мы описываем протоколы для двух наборов экспериментов, которые часто используются для полу количественно анализа включены во время репликации ДНК и мутагенных эффектов неустраненный ribonucleotides ribonucleotides. Хотя эти подходы включают модель эукариоты S. cerevisiae, эти методы можно легко адаптировать для других микробов и даже высших эукариот.
Зондирования для неустраненный ribonucleotides в ДНК с помощью щелочной агарозы в сочетании с Южный blotting предоставляет важную информацию относительно числа ribonucleotides настоящего и Стрэнд, в которую они были включены. Однако важно отметить, что щелочно чувствительность также может быть вызвано других повреждений ДНК, например abasic сайта. Еще одним средством обнаружения ribonucleotides в ДНК является лечение с очищенной РНКазы H2 фермента, как это было сделано в mammalian клетках21. Хотя наш подход предполагает зондирование для щелочно чувствительных участков в зарождающейся ведущих против отставание пряди на URA3 Репортер ген, используемые в наших экспериментах мутационного анализ, вполне возможно разработать зонды которые отжига в других местах в порядке чтобы получить важную информацию относительно рибонуклеотид плотность на других участках генома. Южная помарка частью процедуры также может использоваться в качестве ссылки для других универсальных ДНК зондирующего экспериментов.
Измерение скорости мутации, с помощью теста колебаний широко использовалась для анализа различных мутагенных события в микроорганизмов и таким образом не только анализ последствий мутагенных ribonucleotides в ДНК. Для этого эксперимента увеличивая количество независимых культур могут значительно повысить точность измерения. Независимых колоний можно получить путем мелирование штамм интерес на YPDA носитель или анализ независимых ползучих споровиков полученные тетраде рассечение пятно диплоидных дрожжей. В идеале, использование нескольких независимых гаплоидным дрожжей штаммов (например., от тетраде рассечение) рекомендуется для сведения к минимуму последствий колебаний напряжения деформации. Это особенно важно, когда наблюдается высокий уровень спонтанной мутации или когда сорт имеет рост дефекта. Дополнительный метод, который может использоваться для измерения спонтанной мутации стоимость накопления мутаций эксперимент22. В этом анализе мутация частота определяется для клеток после экстенсивного роста и может использоваться для вычисления скорости мутации. Муфта эксперимента накопления мутаций с глубоким последовательности технологии была продемонстрирована быть мощным инструментом для анализа скорости мутации и специфика через генома.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим всех нынешних и бывших членов Канкел лаборатории за их работу и обсуждения связанных с протоколом и повторно использованы данные, представленные здесь. Эта работа была поддержана проект Z01 ES065070 для T.A.K. из отдела интрамуральных исследования национальных институтов здравоохранения (НИЗ), Национальный институт окружающей среды медицинских наук (NIEHS).
Материалы
| Name | Company | Catalog Number | Comments |
| YPD media | 20 g dextrose, 20 g peptone, 10g yeast extract, in deionized H2O up to 1 L, add 20 g Bacto agar for solid media, autoclave. | ||
| COM plates | 1.3 g SC dropout mix, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar deionized H2O up to 1 L. Adjust pH to 5.8. Autoclave and 30 – 35 ml per plate. | ||
| CAN plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 1 L. Autoclave for 15 mins at 121 °C and cool down to 56 °C. Add 6 mL of filter-sterilized 1% canavanine sulfate solution. | ||
| 5-FOA plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 800 mL. Autoclave for 15 mins at 121 °C and cool down to 60 °C. Add 200 mL of filter-sterilized 0.5% 5-FOA solution. | ||
| L-Canavanine sulfate | US Biological | C1050 | |
| 5-FOA | US Biological | F5050 | |
| 20 mL glass culture tube | Any brand | ||
| Culture rotator in 30 °C incubator | Shaker incubator can be used instead | ||
| 96 well round bottom plates | Sterile, any brand | ||
| 3 mm glass beads | Fisher Scientific | 11-312A | Autoclave before use |
| 12-channel pipettes | Any brand | ||
| Ex Taq DNA Polymerase | TaKaRa | RR001 | |
| Epicentre MasterPure Yeast DNA Purification Kit | Epicenter | MPY80200 | |
| 3 M sodium acetate | Sigma-Aldrich | S7899 | |
| 100% ethanol | |||
| Qubit 2.0 Fluorometer | Invitrogen | Q32866 | Newer model available |
| dsDNA BR Assay kit | Invitrogen | Q32850 | |
| KOH | Sigma-Aldrich | 221473 | |
| EDTA | Sigma-Aldrich | E7889 | |
| Ficoll 400 | Dot Scientific Inc. | DSF10400 | |
| Bromocresol green | Eastman | 6356 | |
| Xylene cyanol FF | International Technologies Inc. | 72120 | |
| NaOH | Sigma-Aldrich | S8045 | |
| 1 M Tris-HCl (pH 8.0) | Teknova | T5080 | |
| SYBR Gold Nucleic Acid Gel Stain | Invitrogen | S11494 | |
| UV transilluminator | |||
| Amersham Nylon membrane Hybond-N+ | GE Healthcare | RPN303B | |
| 3 MM CHR Chromotography paper | Whatman | 3030-392 | |
| NaCl | Caledon | 7560-1 | |
| Stratalinker 1800 | Stratagene | ||
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| G-25 spin column | GE Healthcare | 27-5325-01 | |
| 1 M Sodium phosphate buffer (pH 7.2) | Sigma-Aldrich | NaH2PO4 (S9638); Na2HPO4 (S9390) | |
| SDS | Sigma-Aldrich | L4522 | |
| BSA | Sigma-Aldrich | A3059 | |
| Formamide | Sigma-Aldrich | 47671 | |
| Geiger counter |
Ссылки
- Joyce, C. M. Choosing the right sugar: how polymerases select a nucleotide substrate. Proceedings of the National Acaddemy of Sciences U S A. 94 (5), 1619-1622 (1997).
- Nick McElhinny, S. A., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proceedings of the National Acaddemy of Sciences U S A. 107 (11), 4949-4954 (2010).
- Sparks, J. L., et al. RNase H2-initiated ribonucleotide excision repair. Molecular Cell. 47 (6), 980-986 (2012).
- Li, Y., Breaker, R. R. Kinetics of RNA Degradation by Specific Base Catalysis of Transesterification Involving the 2'-Hydroxyl Group. Journal of the American Chemical Society. 121 (23), 5364-5372 (1999).
- Nick McElhinny, S. A., et al. Genome instability due to ribonucleotide incorporation into DNA. Nature Chemical Biology. 6 (10), 774-781 (2010).
- Williams, J. S., Kunkel, T. A. Ribonucleotides in DNA: origins, repair and consequences. DNA Repair (Amst). 19, 27-37 (2014).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nature Reviews Molecular Cell Biology. 17 (6), 350-363 (2016).
- Jones, M. E., Thomas, S. M., Rogers, A. Luria-Delbruck fluctuation experiments: design and analysis. Genetics. 136 (3), 1209-1216 (1994).
- Zheng, Q. A new practical guide to the Luria-Delbruck protocol. Mutat Research. 781, 7-13 (2015).
- Sambrook, J. Molecular Cloning: a Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2001).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The major roles of DNA polymerases epsilon and delta at the eukaryotic replication fork are evolutionarily conserved. PLoS Genetics. 7 (12), e1002407(2011).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides are signals for mismatch repair of leading-strand replication errors. Molecular Cell. 50 (3), 437-443 (2013).
- Williams, J. S., et al. Topoisomerase 1-mediated removal of ribonucleotides from nascent leading-strand DNA. Molecular Cell. 49 (5), 1010-1015 (2013).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nature Structural & Molecular Biology. 22 (3), 185-191 (2015).
- Foster, P. L. Methods for determining spontaneous mutation rates. Methods Enzymology. 409, 195-213 (2006).
- Williams, J. S., et al. Evidence that processing of ribonucleotides in DNA by topoisomerase 1 is leading-strand specific. Nature Structural & Molecular Biology. 22 (4), 291-297 (2015).
- Pavlov, Y. I., Shcherbakova, P. V., Kunkel, T. A. In vivo consequences of putative active site mutations in yeast DNA polymerases alpha, epsilon, delta, and zeta. Genetics. 159 (1), 47-64 (2001).
- Nick McElhinny, S. A., Kissling, G. E., Kunkel, T. A. Differential correction of lagging-strand replication errors made by DNA polymerases {alpha} and {delta}. Proceedings of the National Acaddemy of Sciences U S A. 107 (49), 21070-21075 (2010).
- Clark, A. B., Lujan, S. A., Kissling, G. E., Kunkel, T. A. Mismatch repair-independent tandem repeat sequence instability resulting from ribonucleotide incorporation by DNA polymerase epsilon. DNA Repair (Amst). 10 (5), 476-482 (2011).
- Lujan, S. A., et al. Mismatch repair balances leading and lagging strand DNA replication fidelity. PLoS Genetics. 8 (10), e1003016(2012).
- Reijns, M. A., et al. Enzymatic removal of ribonucleotides from DNA is essential for mammalian genome integrity and development. Cell. 149 (5), 1008-1022 (2012).
- Lujan, S. A., et al. Heterogeneous polymerase fidelity and mismatch repair bias genome variation and composition. Genome Research. 24 (11), 1751-1764 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены