Method Article
Estudando ribonucleótido incorporação: Deteção de vertente específica de ribonucleotídeos no genoma da levedura e mutagênese induzida pela ribonucleotídeo de medição
Neste Artigo
Resumo
Ribonucleotídeos estão entre os mais abundantes nucleotídeos não-canônicos incorporados do genoma durante a replicação do DNA nuclear eucariótica. Se não forem devidamente removidos, ribonucleotídeos podem causar mutagênese e dano do ADN. Aqui, apresentamos duas abordagens experimentais que são usadas para avaliar a abundância de incorporação de ribonucleotídeo DNA e seus efeitos mutagénicos.
Resumo
A presença de ribonucleotídeos no DNA nuclear tem demonstrada ser uma fonte de instabilidade genômica. A extensão da incorporação do ribonucleotídeo pode ser avaliada por hidrólise alcalina e electroforese do gel como o RNA é altamente suscetível à hidrólise em condições alcalinas. Isto, em combinação com a análise de Southern blot pode ser usado para determinar a localização e a vertente em que foram incorporados os ribonucleotídeos. No entanto, este procedimento só é semiquantitativo e pode não ser suficientemente sensível para detectar pequenas alterações no conteúdo do ribonucleotídeo, apesar de Southern blot vertente específica de sondagem melhora a sensibilidade. Como uma medida de uma das consequências mais marcantes de ribonucleotídeos no DNA biológicas, mutagênese espontânea pode ser analisado usando um ensaio de mutação para a frente. Usando genes repórter apropriado, raras mutações que resulta na perda de função podem ser selecionada e total e taxas de mutação específico podem ser medidas pela combinação de dados das experiências de flutuação com o sequenciamento de DNA do gene repórter. O ensaio de flutuação é aplicável para examinar uma ampla variedade de processos mutagênicos no fundo genético específico ou condições de crescimento.
Introdução
Durante a replicação do DNA eucariótica nuclear, ribonucleotídeos são incorporados ao genoma por todos os três principais replicases de DNA, DNA polimerase (Pols) α, ε e δ1,2. Reparo de excisão do RNase H2-dependente ribonucleótido (RER3) remove a maioria destes ribonucleotídeos incorporados.
Um ribonucleótido no DNA é suscetível à hidrólise, como 2' grupo hidroxila da fracção de açúcar pode atacar o fosfodiéster adjacente, liberando uma extremidade com um fosfato cíclico 2' - 3' e o outro com uma 5'-OH4. Condições alcalinas grandemente podem acelerar esta reação. Assim, a hidrólise de ribonucleotídeos incorporados durante a incubação em uma solução básica causa a fragmentação de DNA genômico, que pode ser visualizado por electroforese de agarose alcalina5. Este DNA pode ser transferido para uma membrana e analisado pela análise de Southern blot utilizando sondas de vertente específicas que permitem a identificação dos locais sensíveis ao alcaloide causada por ribonucleotídeos incorporados para o nascente líder - ou revestimento-strand DNA, respectivamente.
Em células de levedura, falta de atividade RNase H2, remoção de ribonucleotídeos pode ser iniciada quando topoisomerase eu (Top1) rouba o DNA incorporado ribonucleótido6,7. No entanto, quando Top1 fende do lado 3' o ribonucleotídeo, isso gera 5'-OH e 2' - 3' fosfato cíclica DNA extremidades que são refratárias a religation. A reparação ou transformação aberrante destas extremidades' sujas' pode provocar instabilidade genômica. Além disso, se a incisão ocorre dentro de uma sequência de DNA a repetição, o processo de reparação pode levar a mutações de exclusão. Isto é particularmente problemático para repetições em tandem, onde curto exclusões (de entre dois e cinco pares de bases) são comumente observados em células de RNase H2-deficiente. Os efeitos deletérios de Top1-dependente na ausência de levedura RNase H2 atividade são exacerbados em um mutante ε da DNA polimerase (pol2-M644G) promíscuo para incorporação ribonucleótido durante a síntese de vertente principal nascente.
Processamento de ribonucleotídeos no DNA leva a mutações espontâneas e este mutagênese pode ser detectada usando genes repórter apropriado e selecionando para a mudança fenotípica que acompanha. Um teste de flutuação ou experiência de Luria e Delbrück é um dos métodos mais comumente utilizados para medir taxas de mutação espontânea, usando o repórter selecionável genes8,9. Fermento, os genes URA3 e CAN1 podem ser usados como repórteres em um ensaio de mutação para a frente, o que permite a detecção de todos os tipos de mutação que resultam em perda da função do gene. A taxa de mutação espontânea é estimada como a mediana de observadas em múltiplas culturas paralelas, iniciadas a partir de colônias única sem mutações no gene repórter do alvo. Uma levedura RNase H2-deficiente estirpe como rnh201Δ tem uma taxa de mutação espontânea em geral moderadamente elevados em grande parte causada por uma elevada incidência de exclusões de 2-5 bp em tandem repeat sequências. Assim, para caracterizar totalmente os efeitos mutagênicos de ribonucleotídeos no genoma, taxas de mutação específica precisam ser determinado. Neste caso, os genes repórter URA3 ou CAN1 podem ser amplificados e sequenciados para determinar os tipos e locais das mutações, e taxas de mutação específica podem ser calculadas. Compilar as mutações identificadas em vários mutantes independentes de URA3 ou CAN1 então pode ser usado para gerar um espectro de mutação.
Protocolo
1. alcalina hidrólise e vertente específica de Southern Blot (Figura 1)
- Crescimento da célula e isolamento de DNA genômico
- Isole o DNA genômico de levedura usando um kit de purificação de DNA seguindo as instruções do fabricante do fermento. Use as culturas que estão na fase exponencial de crescimento entre (uma densidade óptica de 0.5) e 1. Resuspenda o pellet de DNA final em 35 mL de tampão TE (10 mM Tris-HCl, pH 7,5, ácido de 1 mM ácido etilenodiaminotetracético (EDTA)) 1x.
- Adicionar 1 mL de 5mg/mL RNase ao DNA e incube por 30 min a 37 ° C.
- Precipitar o DNA utilizando 0,1 volumes de acetato de sódio 3M (pH 5.2) e 2,5 volumes de etanol 100% por 20 min no gelo.
- Centrifugar a 16.000 x g por 20 min a 4 ° C. Remova o sobrenadante com uma pipeta.
- Lave o pellet duas vezes com 500 mL de etanol a 70%. Remova o sobrenadante com uma pipeta.
- Secar a pelota no banco e ressuspender em 35 ml de tampão TE 1x.
- Dose o DNA usando um fluorímetro (Tabela de materiais) com o kit de ensaio BR dsDNA.
- Precipitar a 5 mg de DNA de cada amostra utilizando 0,1 volumes de acetato de sódio 3M, volumes de pH 5.2 e 2.5 de 100% EtOH para um volume total de 200 mL (adicionar H2O, se necessário, adicional de 5 mg de DNA é necessário se o controle de gel neutro é necessário). Incube no gelo por 20 min.
- Centrifugar a 16.000 x g por 20 min a 4 ° C. Remova o sobrenadante com uma pipeta.
- Seque a pelota no banco. Resuspenda o DNA em 14 mL de H2O.
- Eletroforese de gel e hidrólise alcalina
- Adicione 6 mL de 1m KOH e incubar a 55 ° C por 2 h.
- Incube as amostras no gelo por 5 min.
- Adicionar 4 mL de 6x do amortecedor do carregamento de DNA alcalina: 300mm KOH, 6 mM EDTA, pH 8.0, polysucrose 18% (Tabela de materiais), 0,15% verde de bromocresol e 0,25% xileno cianol FF.
- Converter um gel alcalino que é composto de agarose a 1%, 50mm NaOH, 1 mM EDTA, pH 8.0. Use um pente que permite 24 mL de amostra de DNA para ser carregado por faixa.
- Funcione o gel à temperatura ambiente em tampão de eletroforese alcalina 1x: 50mm NaOH, 1 mM EDTA, pH 8.0. Incluem um marcador de tamanho apropriado do DNA.
- Spin para baixo os tubos brevemente e carregar o inteiro 24 mL de amostra para o gel de agarose alcalina. Electrophorese por 30 min em 30 V, seguido por 18-20 h em 10 V.
- Neutralize o gel para incubação 2 de 45 min à temperatura ambiente com agitação suave na neutralização Buffer i: 1 M Tris-HCl, 1,5 M de NaCl.
- Mancha o DNA embebendo o gel neutralizado em 200 mL de água contendo uma diluição de 1/10.000 da mancha SYBR Gold para 2 h à temperatura ambiente com agitação suave.
- Visualize o DNA usando um transiluminador UV. Alcaloide-sensibilidade aumentada provoca o aparecimento de pequenos fragmentos de DNA, indicando uma maior densidade de ribonucleotídeos não reparados no DNA5.
- Transferência do Sul
- Transferi o DNA do gel alcalino para a membrana de nylon positivamente carregado por ação capilar10. Um exemplo de como esta transferência pode ser configurada é fornecido no manual Sapeka e Russell está10.
- Mergulhe o gel alcalino para 15 min à temperatura ambiente em tampão alcalino transferência: 0,4 M de NaOH, 1 M de NaCl.
- Molhe a membrana de nylon (corte ao tamanho do gel) brevemente com água e em seguida, mergulhe por 5 min em tampão alcalino transferir.
- Configurar a plataforma de transferência invertendo-se 3 copos de vidro (100 mL) em um assadeira de vidro. Este prato deve ser ligeiramente maior que o tamanho do gel do agarose. Defina uma placa de vidro por cima deles.
- Drape 2 pedaços grandes de 3mm CHR cromatografia em papel (corte ao tamanho da largura do gel plus mais lados que podem armar sobre as bordas no reservatório tampão) sobre a placa de vidro.
- Despeje Alkaline Buffer de transferência sobre o papel para cromatografia e metade encher o prato de vidro. Suavizar as bolhas usando uma pipeta de vidro de 5 mL.
- Coloque o gel de agarose em cima, outra vez, suavizar as bolhas. Cerca todas as bordas do gel com parafilme. Despeje uma pequena quantidade de Buffer alcalino de transferir para o gel.
- Coloque a membrana de nylon previamente embebido em cima do gel. Suavizar as bolhas.
- Molhados 2 pedaços de papel cortado para o tamanho do gel do agarose. Coloque-os em cima da membrana e suavizar as bolhas.
- Coloque uma pilha de toalhas de papel (corte para um tamanho ligeiramente maior do que o gel de agarose) em cima do sanduíche de transferência. Cuidadosamente coloque uma placa de vidro em cima as toalhas de papel. Adicione um objeto ponderado para o topo (um livro com uma massa de 400 g funciona bem).
- Deixe a transferência prosseguir durante a noite em temperatura ambiente.
- Cuidadosamente, desmontar a pilha de transferência e mergulhe a membrana durante 15 minutos à temperatura de neutralização Buffer II: 0.5 M Tris-HCl, pH 7.2, 1 M de NaCl.
- Crosslink o DNA para a membrana de nylon carregado usando um agente reticulante UV. Coloque a membrana no cross-linker e usar a configuração de autocrosslink.
- Preparação do Sul da sonda
Nota: A abordagem descrita aqui para detectar sites sensíveis ao alcaloide no líder emergente-contra atraso-fio de uma cadeia de DNA é baseado em um protocolo previamente publicado11. Para a nossa experiência, a sondagem é realizada no gene repórter URA3 que tem sido inserido em uma das duas orientações (OR1 ou OR2) adjacentes à origem de replicação de ARS306 no cromossomo III (Figura 3A). A presença do alcaloide-sensíveis sites no DNA genômico de levedura é um indicador de incorporação ribonucleotídeo, permitindo o uso de ribonucleotídeos como os biomarcadores de DNA polimerase atividade12,13,14. Esta abordagem, usando uma sequência de DNA adjacente à eficiente ARS306 origem de replicação, de destino deve ser passível de outras localizações genômicas e genes-alvo também.- PCR-amplificação de uma base de 520 segmento de par do gene repórter URA3 usando o seguinte par de primer: URA3-F1 (5´-GCTACATATAAGGAACGTGCTGC) e URA3-R1 (5´-CTTTGTCGCTCTTCGCAATGTC).
- Realizar a reação de PCR usando 10-20 ng de DNA genômico de levedura como um modelo, 4 μL de cada primer (estoque de 10 μM), 10 μL de 10 x Ex Taq Buffer (Mg2 + plus), 8 μL da mistura do dNTP (2,5 mM cada), 0,5 μL de Ex Taq DNA polimerase (5 U/μL) e ajustar o volume final de 100 μL com H2O.
- Execute o seguinte programa PCR: 95 ° C por 5 min; 30 ciclos de 95 ° C por 1 min, a 55 ° C por 1 min, 72 ° C por 2 min; 72 ° C por 10 min e espera a 4 ° C.
- Purificar o produto do PCR usando um kit de purificação de PCR e eluir o DNA com 50 mL de H2O. Isso agora pode ser usado como o modelo na reação de radiolabeling.
- Use os seguintes primers para vertente específica radioativos: uso URA3-A (5´-CTCATCCTAGTCCTGTTGCTGCC) para a visualização de DNA que anneals a sonda A. Isso corresponde o nascente principal vertente DNA quando URA3 está em OR2 e o tnascent ficando strand DNA quando URA3 está em OR1. Usar URA3-B (5´-CAGTACCCTTAGTATATTCTCCAG) para a visualização de DNA nascente que anneals a sonda B. Isso corresponde o nascente principal vertente DNA quando URA3 estiver em OR1 e revestimento nascente strand DNA quando URA3 está em OR2 (Figura 3).
- Realizar a reação radiolabeling usando 10-20 ng amplificado URA3 fragmento como modelo de DNA, 2 μL da primeira demão (estoque de 10 μM), 5 μL de 10 x Ex Taq Buffer (Mg2 + plus), 4 μL de dNTPs 2,5 mM (menos dCTP), 5 μL (50 μCi) de um -32P-dCTP , 0,5 μL de ExTaq DNA polimerase (5 U/μL) e ajustar o volume final de 50 μL com H2O. executar o seguinte programa para radioativos: 94 ° C por 5 min; 25 ciclos de 94 ° C por 1 min, a 55 ° C por 30 s, 72 ° C por 1 min; 72 ° C por 5 min e espera a 4 ° C.
- Use uma coluna de rotação G-25 para remover marcador radioativo sem personalidade jurídica. Antes de adicionar a amostra radiolabeled, centrifugar a coluna por 1 min em 720 x g e removero tampão pipetando. Carrega o produto da reação radiolabeled e centrifugar por 2 min a 720 x g para eluir a sondas marcadas.
- Incube a 95 ° C por 5 min desnaturar a sonda imediatamente antes da hibridização. Cool brevemente no gelo.
- Hibridação do Sul
- Roll a membrana imersos em um tubo de hibridização e adicionar 25 mL de tampão de hibridização preparado: tampão de fosfato de sódio 0,5 M, pH 7,2, 7% Dodecil sulfato de sódio (SDS), 1% albumina de soro bovino (BSA). Incube a 65 ° C, durante 1-2 h com a rotação em um forno de hibridização.
- Cuidadosamente, decantar a solução e adicionar 25 mL de tampão de hibridização mais a sonda marcadas com radioisótopos. Incube a 65 ° C para 16-18 h com a rotação.
- Decantar a solução em um recipiente de resíduos radioactivo. Realizar 5 lavagens de 5 min à temperatura ambiente com tampão de fosfato de sódio fosfato-SDS lavagem solução i: 40 mM, pH 7,2, pH de 5% SDS, 0,5% BSA, 1 mM EDTA, 8.0.
- Executar 2 lavagens de 15 min a 65 ° C, com solução de lavagem fosfato-SDS II: 40 mM de tampão de fosfato de sódio, pH 7.2, 1% SDS, 1 mM EDTA, pH 8.0.
- Remover a membrana no tubo de hibridização usando uma pinça e coloque em um protetor de manga de plástico aberto. Cobrir e expor a uma placa de imagem. Tente um número de diferente tempos que variam de 4 h a 4D de exposição.
- Se necessário, strip e re-sonda blot usando uma sonda diferente aproximadamente 3 - 4 vezes. Para tira, incube a membrana para h 2 a 65 ° C, em 25 mL de uma formamida 50%, solução de x SSPE 2.
Nota: 20 x SSPE solução: 3 M NaCl, 0,2 M NaH2PO4, EDTA 0,02 M, pH 7,4. - Decantar a solução em um recipiente de resíduos radioactivo. Execute 2 lavagens de 15 min a 65 ° C em solução de lavagem fosfato-SDS II.
- Verifique se a membrana com um contador Geiger e repita o descascamento protocolo se sinal permanece.
- Realizar pré-hibridização e hibridação conforme descrito acima.
2. medir a taxa de mutação e especificidade em cepas de S. cerevisiae
- Preparar a cultura de células
- Inocular uma colônia única (colônias de 2-3 dias a 30 ° C, a forma adequada de porte do leva de células de levedura saudável) cultivada em ágar YPD suplementado com adenina de 100 mg/mL em 5 mL de caldo YPDA suplementado com adenina 250mg/mL (suplementação de adenina é necessário apenas se a tensão utilizada é um auxotroph de adenina). Cresce em uma incubadora shaker ou em rotacao a 30 ° C até que a cultura atinja a saturação (36-48 h para uma tensão sem um defeito grave de crescimento).
- Gire para baixo as células a ~ 2000 x g por 5 min.
- Desprezar o sobrenadante e lavar as células com 5 mL de água estéril.
- Ressuspender as células em 500 mL de água estéril.
- Diluição da cultura de pilha e chapeamento
- Para controles de não selecção, dilua as células em uma placa de 96 poços por um fator de 1.600.000 e placa 100 mL de cada cultura em um prato (COM) completa.
- Para selecionar para ura3 mutantes, placa 100 mL de células não diluídas em uma placa de 5-FOA para uma estirpe do selvagem-tipo (WT). Dilua as células em conformidade, se a taxa de mutação da estirpe de interesse deverá ser maior do que em uma cepa WT.
- Para selecionar para mutantes can1 , diluir as células 5-fold e placa 100 mL em uma placa contendo canavanine (CAN) para uma tensão WT. O factor de diluição é 0,5.
- Incube as placas a 30 ° C até formar colônias de levedura. Para cepas sem um defeito de crescimento, 2-3 dias é geralmente suficientes para COM e placas e 3-5 dias para placas 5-FOA. Para comparar as taxas de mutação entre estirpes diferentes, é melhor escolher o mesmo dia de crescimento para a contagem das colónias. Armazene as placas prontas a serem contados no quarto frio (~ 4 ° C).
- Cálculo da taxa de mutação espontânea
- Contagem das colónias visíveis em cada prato e tome nota dos números de colônia
- Calcular a taxa de mutação, usando a fórmula do Drake: μ = f ÷ ln (μNt). f é a frequência de mutação calculada pelo número de mutantes, dividido pelo número de células em cultura final. NT é o número de células em cultura final e μ é a taxa de mutação. Esta equação pode ser resolvida por um método iterativo, como o método de Newton-Raphson. Existem outros métodos de estimador, incluindo o teste de Lea e Coulson, para calcular a taxa de mutação15.
- Calcule o intervalo de confiança supondo log distribuição normal das taxas individuais isolados de utation. O intervalo de confiança de 95% é usado mais frequentemente.
- Análise de espectro de mutação
- Escolher uma única colônia de cada 5-FOA ou pode da placa e atualize-se sobre uma placa YPDA.
- Executar a colônia PCR para amplificar o gene URA3 ou CAN1 e sequência para detectar mutações usando os seguintes primers: URA3-F (5'-CGCATATGTGGTGTTGAAGAA-3') e URA3-R (5'-TGTGAGTTTAGTATACATGCA-3') para ambos os amplificação por PCR e sequenciamento do gene URA3 bp 800; CAN1-F (5'-CTTCTACTCCGTCTGCTTTC-3') e CAN1-R (5'-CAGAGTTCTTCAGACTTC-3') para amplificação e CAN1-AR (5'-TGAAATGTGAAGGCAGCGTT-3'), CAN1-9R (5'-CCTGCAACACCAGTGATA-3'), CAN1-10R (5'-GAGGATGTAACAGGGATGAAT-3') e (CAN1-BR 5'-CGGTGTATGACTTATGAGGG-3') para o sequenciamento do gene repórter 1800 bp CAN1 .
- Para realizar o PCR de colônia, resuspenda uma única colônia em 5 μL de H2O e 1 μL, usar como modelo. Adicionar 1 μL de cada um dos primers (estoque de 5 μM), 2 μL de tampão de x KAPA2G 5 (Mg2 + plus), 0,5 μL de dNTP (2,5 mM), 0,5 μL de KAPA2G rápido DNA polimerase, 12 μL de H2O.
- Para a amplificação do gene URA3 , execute o seguinte programa PCR: 95 ° C por 5 min; 5 ciclos de 95 ° C por 10 s, 61 ° C, por 15 s, 72 ° C por 30 s; 35 ciclos de 95 ° C por 10 s, 61 ° C, por 15 s, 72 ° C por 30 s; 72 ° C por 2 min e espera a 4 ° C. Para a amplificação do gene CAN1 , execute o seguinte programa PCR: 95 ° C por 5 min; 5 ciclos de 95 ° C por 10 s, 61 ° C, por 15 s, 72 ° C por 45 s; 35 ciclos de 95 ° C por 10 s, 61 ° C, por 15 s, 72 ° C por 45 s; 72 ° C por 2 min e espera a 4 ° C.
- Alinhe os resultados de sequenciamento para uma sequência de referência usando um programa como o DNASTAR Seqman Pro ou Sequencher para identificar as mutações.
- Compile os dados de mutações por tipo de mutação (Figura 4) ou localização de mutação (Figura 5). Calcule as taxas de mutação específica pela frequência de uma mutação (o número de eventos observados dividido pelo número de mutantes sequenciados) multiplicaram pela taxa de mutação global.
Resultados
Tratamento de DNA genômico com alcaloide seguido por eletroforese em gel alcalina permite detecção semi-quantitativa da fragmentação de DNA devido à abundância de ribonucleotídeos estàvel incorporados. A Figura 2 mostra as imagens de gel de levedura DNA genômico tratado com ou sem KOH5. A variante M644L de Pol2, a subunidade catalítica de Polε, reduziu a capacidade de incorporar ribonucleotídeos enquanto o mutante M644G incorpora ribonucleotídeos mais do que o polymerase WT. Conforme detalhado no protocolo, o gel alcalino pode ser ainda mais sondado por Southern blot vertente específica análise. Com o conhecimento da localização do site genômica sondado em relação a suas origens adjacentes, nós pode seletivamente sonda incorporados a nascente de ribonucleotídeos levando ou vertentes de retardamento. A Figura 3 mostra os resultados de Southern blot, sondando o gene repórter URA3 inserido em duas orientações opostas, perto de uma origem de replicação de acionamento do início, ARS30616. Usando o levando a sondas específicas de vertente, observamos que o padrão de fragmentação de DNA causadas por álcali-clivagem de ribonucleotídeos no DNA de vertente principal nascente em uma cepa WT e em cepas expressando as variantes de polymerases α, δ e ε que são promíscuas para incorporação de ribonucleotídeo.
Usando um experimento de flutuação, podemos medir as taxas de mutação de cepas de leveduras contendo genes de repórter. A Figura 4 mostra as taxas de mutação no locus CAN1 cepas expressando WT polimerase ou o mutante pol2-M644G com ou sem funcional RNase H2 e URA3. A falta de RNase H2 causa um aumento moderado em geral taxas de mutação em ambos os planos de fundo. No entanto, uma análise mais aprofundada da especificidade de mutação revela que em cepas faltando RNase H2 (Figura 5), há um forte aumento da taxa de mutação para exclusões curtas, particularmente em sequências de repetição em tandem. Figura 5A compara a especificidade de mutação em WT e rnh cepas201Δ e Figura 5B exibe o efeito mutacional do mais elevado ribonucleótido incorporação a cadeia de DNA principal nascente por Pol ε.
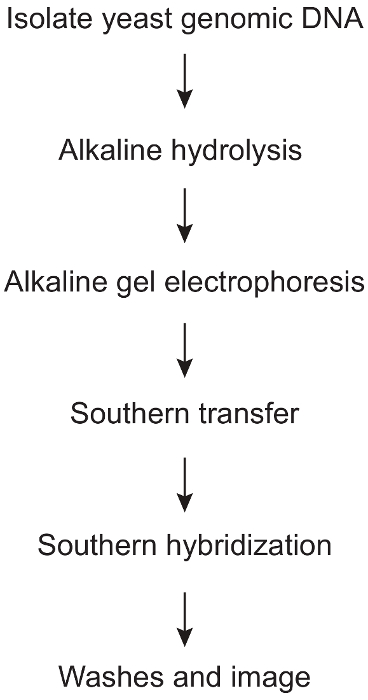
Figura 1: etapas envolvidas na vertente específica detecção sensível do alcaloide sites causada pela incorporação de ribonucleotídeo no DNA genômico de levedura do protocolo. Uma visão geral das várias etapas principais envolvidas nesse procedimento, começando com isolamento de DNA genômico de levedura e prosseguindo através da análise final Southern blot. Clique aqui para ver uma versão maior desta figura.

Figura 2: uma imagem representativa dos resultados obtidos por eletroforese em gel de agarose alcalina. Esta figura foi adaptada de Nick McElhinny, et al . S. A. 5. levedura DNA genômico de cepas de diferentes genótipos foi tratado com KCl (neutro) ou KOH (alcalina) e, em seguida, separados em um gel de agarose alcalina. "WT" e "M644G" indicam o status de Pol ε. clique aqui para ver uma versão maior desta figura.

Figura 3: Strand-específicos de um gel de agarose alcalina de probing Southern blot. Esta figura foi adaptada da Williams, et al . J. S. 13. (A) , as sondas radiolabeled recozem a nascente principal cadeia do DNA dentro do gene repórter URA3 que foi inserida em duas orientações opostas (OR1 e OR2) perto de ARS306 . (B) resultados representativos da mancha do Sul usando as nascentes principais vertente específica sondas. Exibido são os resultados de sondagem para a vertente principal nascente em uma cepa com as polimerases de DNA WT, ou o pol1-L868M, pol2-M644G ou o pol3-L612M variantes cepas com ou sem RNase H2. Os genes POL1, POL2 e POL3 codificam subunidades catalíticas de Pols α, ε e δ, respectivamente. As variantes são mais promíscuas para ribonucleótido incorporação5,12,17. (C) a quantificação do sinal radioativo por fração do total em cada faixa da (B). Valores são expressos em percentagem do total. Clique aqui para ver uma versão maior desta figura.

Figura 4: taxas de mutação espontânea em cepas de leveduras. Esta figura foi adaptada de Nick McElhinny, et al . S. A. 5. os genes repórter URA3 e CAN1 foram utilizados no ensaio de mutação para a frente para medir as taxas de mutação das estirpes indicadas. URA3 repórter está na "orientação 2" (OR2, Figura 3A). O intervalo de confiança de 95% (IC) é incluído para cada medição. Clique aqui para ver uma versão maior desta figura.

Figura 5: espectros de mutação para o gene repórter URA3-OR2. Esta figura foi adaptada de publicações anteriores5,18,19,20. O tipo de posição e mutação observado em cada colônia 5-FOA-resistente independente selecionada no ensaio de mutação para a frente são retratados no 804 bp a sequência de código URA3 . Letras indicam base substituições, triângulos abertos indicam exclusões de base única, fechados triângulos indicam inserções de base única, e linhas sólidas indicam curtas exclusões. (A) espectros de mutação em cepas WT e rnh201Δ . Etiquetas vermelhas acima a sequência são mutações observadas no WT enquanto rótulos azuis abaixo a sequência são aqueles observados com uma tensão de rnh201Δ . (B) espectros de mutação em cepas pol2-M644G e rnh201Δ pol2-M644G . Etiquetas vermelhas acima a sequência são mutações observadas em pol2-M644G enquanto azul abaixo a sequência para estirpes pol2-M644G rnh201Δ . Clique aqui para ver uma versão maior desta figura.
Discussão
Aqui, descrevemos os protocolos para dois conjuntos de experiências que são frequentemente usados para analisar semi-quantitativamente ribonucleotídeos incorporados durante a replicação do DNA e os efeitos mutagênicos de ribonucleotídeos não reparados. Embora essas abordagens envolvem a eucariota modelo S. cerevisiae, estas técnicas podem ser facilmente adaptadas para outros micróbios e eucariontes ainda maiores.
Sondagem para ribonucleotídeos não reparados no DNA utilizando eletroforese alcalina-agarose juntamente com mancha do Sul fornece informações importantes sobre o número de ribonucleotídeos presentes e a vertente na qual eles foram incorporados. No entanto, é importante notar que do alcaloide-a sensibilidade também pode ser causada por outras lesões de DNA, como um site da básica. Outro meio de detecção de ribonucleotídeos no DNA é pelo tratamento com enzima purificada de RNase H2, como foi feito em células de mamíferos,21. Embora nossa abordagem envolve sondagem para sites sensíveis ao alcaloide na nascente líder contra atraso vertentes no gene repórter da URA3 usada em nossos experimentos de análise mutacional, é possível projetar as sondas que recozem em outros locais, em ordem para obter informações importantes sobre a densidade ribonucleotídeo em outros locais do genoma. O borrão do Sul parte do procedimento também pode ser usado como referência para outras experiências sondagem genéricas de DNA.
Medição de taxas de mutação usando um teste de flutuação tem sido amplamente utilizada para a análise de vários eventos mutagênicos em microorganismos e, portanto, não se limita à análise das consequências mutagénicas de ribonucleotídeos no DNA. Para este experimento, aumentando o número de culturas independentes pode aumentar consideravelmente a precisão da medição. Colônias independentes podem ser obtidas por estrias uma estirpe de interesse no médio YPDA ou pela análise das colônias de esporos independentes obtidos por dissecação Tétrade de uma mancha de levedura diploide. Idealmente, o uso de várias cepas de leveduras haploides independente (ex., de dissecação Tétrade) é incentivado para minimizar o impacto da variação de tensão-à-tensão. Isto é particularmente importante quando a taxa de mutação espontânea é alta, ou quando a tensão tem um defeito de crescimento. Um método adicional que pode ser empregado para medir espontânea a taxa de mutação é uma acumulação de mutação experimentar22. Nesta análise, frequência de mutação é determinada para as células, após crescimento extensivo e pode ser usada para calcular a taxa de mutação. Acoplamento de um experimento de acumulação de mutação com a tecnologia de sequenciamento profundo tem demonstrado para ser uma poderosa ferramenta para analisar a taxa de mutação e especificidade através do genoma.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos a todos os antigos e atuais membros Kunkel laboratório para os seus trabalhos e discussões relacionadas ao protocolo e reutilizados dados aqui apresentados. Este trabalho foi financiado pelo projeto Z01 ES065070 para T.A.K. da divisão de pesquisa Intramural dos institutos nacionais de saúde (NIH), National Institute de Ciências de saúde ambiental (NIEHS).
Materiais
| Name | Company | Catalog Number | Comments |
| YPD media | 20 g dextrose, 20 g peptone, 10g yeast extract, in deionized H2O up to 1 L, add 20 g Bacto agar for solid media, autoclave. | ||
| COM plates | 1.3 g SC dropout mix, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar deionized H2O up to 1 L. Adjust pH to 5.8. Autoclave and 30 – 35 ml per plate. | ||
| CAN plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 1 L. Autoclave for 15 mins at 121 °C and cool down to 56 °C. Add 6 mL of filter-sterilized 1% canavanine sulfate solution. | ||
| 5-FOA plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 800 mL. Autoclave for 15 mins at 121 °C and cool down to 60 °C. Add 200 mL of filter-sterilized 0.5% 5-FOA solution. | ||
| L-Canavanine sulfate | US Biological | C1050 | |
| 5-FOA | US Biological | F5050 | |
| 20 mL glass culture tube | Any brand | ||
| Culture rotator in 30 °C incubator | Shaker incubator can be used instead | ||
| 96 well round bottom plates | Sterile, any brand | ||
| 3 mm glass beads | Fisher Scientific | 11-312A | Autoclave before use |
| 12-channel pipettes | Any brand | ||
| Ex Taq DNA Polymerase | TaKaRa | RR001 | |
| Epicentre MasterPure Yeast DNA Purification Kit | Epicenter | MPY80200 | |
| 3 M sodium acetate | Sigma-Aldrich | S7899 | |
| 100% ethanol | |||
| Qubit 2.0 Fluorometer | Invitrogen | Q32866 | Newer model available |
| dsDNA BR Assay kit | Invitrogen | Q32850 | |
| KOH | Sigma-Aldrich | 221473 | |
| EDTA | Sigma-Aldrich | E7889 | |
| Ficoll 400 | Dot Scientific Inc. | DSF10400 | |
| Bromocresol green | Eastman | 6356 | |
| Xylene cyanol FF | International Technologies Inc. | 72120 | |
| NaOH | Sigma-Aldrich | S8045 | |
| 1 M Tris-HCl (pH 8.0) | Teknova | T5080 | |
| SYBR Gold Nucleic Acid Gel Stain | Invitrogen | S11494 | |
| UV transilluminator | |||
| Amersham Nylon membrane Hybond-N+ | GE Healthcare | RPN303B | |
| 3 MM CHR Chromotography paper | Whatman | 3030-392 | |
| NaCl | Caledon | 7560-1 | |
| Stratalinker 1800 | Stratagene | ||
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| G-25 spin column | GE Healthcare | 27-5325-01 | |
| 1 M Sodium phosphate buffer (pH 7.2) | Sigma-Aldrich | NaH2PO4 (S9638); Na2HPO4 (S9390) | |
| SDS | Sigma-Aldrich | L4522 | |
| BSA | Sigma-Aldrich | A3059 | |
| Formamide | Sigma-Aldrich | 47671 | |
| Geiger counter |
Referências
- Joyce, C. M. Choosing the right sugar: how polymerases select a nucleotide substrate. Proceedings of the National Acaddemy of Sciences U S A. 94 (5), 1619-1622 (1997).
- Nick McElhinny, S. A., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proceedings of the National Acaddemy of Sciences U S A. 107 (11), 4949-4954 (2010).
- Sparks, J. L., et al. RNase H2-initiated ribonucleotide excision repair. Molecular Cell. 47 (6), 980-986 (2012).
- Li, Y., Breaker, R. R. Kinetics of RNA Degradation by Specific Base Catalysis of Transesterification Involving the 2'-Hydroxyl Group. Journal of the American Chemical Society. 121 (23), 5364-5372 (1999).
- Nick McElhinny, S. A., et al. Genome instability due to ribonucleotide incorporation into DNA. Nature Chemical Biology. 6 (10), 774-781 (2010).
- Williams, J. S., Kunkel, T. A. Ribonucleotides in DNA: origins, repair and consequences. DNA Repair (Amst). 19, 27-37 (2014).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nature Reviews Molecular Cell Biology. 17 (6), 350-363 (2016).
- Jones, M. E., Thomas, S. M., Rogers, A. Luria-Delbruck fluctuation experiments: design and analysis. Genetics. 136 (3), 1209-1216 (1994).
- Zheng, Q. A new practical guide to the Luria-Delbruck protocol. Mutat Research. 781, 7-13 (2015).
- Sambrook, J. Molecular Cloning: a Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2001).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The major roles of DNA polymerases epsilon and delta at the eukaryotic replication fork are evolutionarily conserved. PLoS Genetics. 7 (12), e1002407(2011).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides are signals for mismatch repair of leading-strand replication errors. Molecular Cell. 50 (3), 437-443 (2013).
- Williams, J. S., et al. Topoisomerase 1-mediated removal of ribonucleotides from nascent leading-strand DNA. Molecular Cell. 49 (5), 1010-1015 (2013).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nature Structural & Molecular Biology. 22 (3), 185-191 (2015).
- Foster, P. L. Methods for determining spontaneous mutation rates. Methods Enzymology. 409, 195-213 (2006).
- Williams, J. S., et al. Evidence that processing of ribonucleotides in DNA by topoisomerase 1 is leading-strand specific. Nature Structural & Molecular Biology. 22 (4), 291-297 (2015).
- Pavlov, Y. I., Shcherbakova, P. V., Kunkel, T. A. In vivo consequences of putative active site mutations in yeast DNA polymerases alpha, epsilon, delta, and zeta. Genetics. 159 (1), 47-64 (2001).
- Nick McElhinny, S. A., Kissling, G. E., Kunkel, T. A. Differential correction of lagging-strand replication errors made by DNA polymerases {alpha} and {delta}. Proceedings of the National Acaddemy of Sciences U S A. 107 (49), 21070-21075 (2010).
- Clark, A. B., Lujan, S. A., Kissling, G. E., Kunkel, T. A. Mismatch repair-independent tandem repeat sequence instability resulting from ribonucleotide incorporation by DNA polymerase epsilon. DNA Repair (Amst). 10 (5), 476-482 (2011).
- Lujan, S. A., et al. Mismatch repair balances leading and lagging strand DNA replication fidelity. PLoS Genetics. 8 (10), e1003016(2012).
- Reijns, M. A., et al. Enzymatic removal of ribonucleotides from DNA is essential for mammalian genome integrity and development. Cell. 149 (5), 1008-1022 (2012).
- Lujan, S. A., et al. Heterogeneous polymerase fidelity and mismatch repair bias genome variation and composition. Genome Research. 24 (11), 1751-1764 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados