Method Article
Studium Ribonukleotidreduktase Aufnahme: Strang-spezifischen Nachweis von Purine Hefegenoms und Messen Ribonukleotidreduktase induzierter Mutagenese
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Purine gehören zu den am häufigsten vorkommenden nicht-kanonischen Nukleotiden während Eukaryotische nukleare DNA-Replikation in das Genom integriert. Wenn nicht richtig entfernt, verursachen Purine DNA-Schäden und Mutagenese. Hier präsentieren wir Ihnen zwei experimentelle Ansätze, die verwendet werden, um die Fülle der Ribonukleotidreduktase Einbindung in DNA und seine mutagenen Wirkungen zu beurteilen.
Zusammenfassung
Das Vorhandensein der Purine in Kern-DNA hat sich gezeigt, eine Quelle der genomischen Instabilität sein. Das Ausmaß der Ribonukleotidreduktase Aufnahme beurteilt werden kann, durch alkalische Hydrolyse und gel-Elektrophorese als RNA Hydrolyse in alkalischen Bedingungen sehr anfällig ist. Dies, in Kombination mit südlicher Fleckanalyse lässt sich bestimmen Sie den Speicherort und Litze in die der Purine aufgenommen wurden. Aber dieses Verfahren ist nur semi-quantitativen und möglicherweise nicht empfindlich genug, um kleine Änderungen in Ribonukleotidreduktase Inhalte zu erkennen, obwohl das Strang-spezifische Südlicher Fleck sondieren die Empfindlichkeit verbessert. Als Maß für eines der auffälligsten biologischen Folgen der Purine in DNA kann spontane Mutagenese mit einem vorwärts Mutation Assay analysiert werden. Mit entsprechenden Reporter-Gene, können seltene Mutationen, die den Verlust der Funktion ausgewählt und allgemeine und spezifische Mutationsraten gemessen werden, indem Daten aus Fluktuation Experimente mit DNA-Sequenzierung des reportergens. Die Fluktuation-Assay ist für eine Vielzahl von mutagenen Prozessen in spezifischen genetischen Hintergrund oder Wachstumsbedingungen zu untersuchen.
Einleitung
Während der eukaryotischen nukleare DNA-Replikation fließen Purine in das Genom von allen drei großen DNA Replicases, DNA-Polymerasen (Pols) α, ε und δ1,2. RNase H2-abhängige Ribonukleotidreduktase Exzision Reparatur (RER3) wird den Großteil dieser eingebetteten Purine entfernt.
Ribonukleotidreduktase in DNA ist anfällig gegenüber Hydrolyse, 2'-Hydroxylgruppe des Zucker-Verbindungsgruppe die angrenzenden Phosphodiester-Anleihe, Freigabe von einem Ende mit einer zyklischen Phosphat 2' - 3' und das andere mit einer 5'-OH-4angreifen kann. Alkalische Bedingungen können diese Reaktion erheblich beschleunigt. So verursacht die Hydrolyse von eingebetteten Purine während der Inkubation in eine Basislösung Fragmentierung der genomischen DNA, die durch alkalische Agarose Elektrophorese5visualisiert werden kann. Diese DNA kann auf eine Membran übertragen und sondiert durch südlicher Fleckanalyse mit Strang-spezifischen Sonden, die es die Identifizierung von alkaliempfindlichen Websites durch Purine aufgenommen in der im Entstehen begriffenen führenden ermöglichen- oder -Verzögerungsstrang DNA verursacht, bzw..
In Hefezellen RNase H2 Aktivität fehlt kann Abbau der Purine eingeleitet werden, wenn Topoisomerase ich (Top1) die DNA im eingebetteten Ribonukleotidreduktase6,7 Nicks. Jedoch wenn Top1 auf 3' der die Ribonukleotidreduktase zerspaltet, generiert das 5'-OH und 2' - 3' zyklischen Phosphat DNA-enden, die refraktär gegenüber Religation sind. Reparatur oder abweichende Verarbeitung diese'schmutzigen 'enden können, genomische Instabilität führen. Darüber hinaus tritt der Schnitt innerhalb von einer wiederholte DNA-Sequenz, kann die Reparatur-Prozess zu Löschung Mutationen führen. Dies ist besonders problematisch für Tandem wiederholt, wo kurze Deletionen (von zwischen zwei und fünf Basenpaaren) sind häufig in RNase H2-defizienten Zellen beobachtet. Die Top1-abhängige schädlichen Wirkungen bei fehlender Hefe RNase H2 Aktivität werden in eine DNA-Polymerase ε Mutante (pol2-M644G) promiscuous Ribonukleotidreduktase Aufnahme während der im Entstehen begriffenen führenden Strang-Synthese verstärkt.
Verarbeitung der Purine in DNA führt zu spontanen Mutationen und diese Mutagenese kann durch die Verwendung geeigneter Reporter-Gene und Auswahl für die begleitenden phänotypische Veränderung erkannt werden. Eine Fluktuation Test oder Luria und Delbrück Experiment ist eine der am häufigsten verwendeten Methoden, um spontane Mutation mit wählbaren Reporter Gene8,9messen. In Hefe können die URA3 und CAN1 Gene als Reporter in einem vorwärts Mutation Assay verwendet werden, ermöglicht die Erkennung aller Arten der Mutation, die zum Verlust der Genfunktion. Die spontane Mutationsrate dürfte als der Median, die für mehrere parallele Kulturen begann aus einzelnen Kolonien ohne Mutationen in das Zielgen Reporter beobachtet. Eine Hefe RNase H2-defizienten Stamm wie rnh201Δ hat einen leicht erhöhten insgesamt spontane Mutationsrate, die weitgehend von einer erhöhten Inzidenz von 2-5 bp Löschungen im Tandem verursacht wird wiederholen Sequenzen. Also, um die mutagenen Wirkungen der Purine im Genom vollständig zu charakterisieren, spezifische Mutationsraten müssen ermittelt werden. In diesem Fall URA3 oder CAN1 Reporter-Gene können verstärkt und sequenziert, um festzustellen, die Typen und Standorte der Mutationen, und spezifische Mutationsraten errechnet werden. Kompilieren von Mutationen identifiziert in mehrere unabhängige URA3 oder CAN1 Mutanten kann dann verwendet werden, um eine Mutation-Spektrum zu erzeugen.
Protokoll
1. alkalische Hydrolyse und Strang-spezifische Südlicher Fleck (Abbildung 1)
- Zellwachstum und genomische DNA-Isolierung
- Isolieren Sie Hefe genomischer DNA mit einer Hefe DNA-Reinigung-Kit nach den Anweisungen des Herstellers. Verwenden Sie die Kulturen, die in der exponentiellen Wachstumsphase (eine optische Dichte von 0,5) bis 1 sind. Aufschwemmen der letzten DNA-Pellet in 35 mL 1 x TE-Puffer (10 mM Tris-HCl, pH 7,5, 1 mM Ethylenediaminetetraacetic Säure (EDTA)).
- Fügen Sie 1 mL von 5 mg/mL RNase A an die DNA und 30 min bei 37 ° c inkubieren
- Überstürzen sich die DNA mit 0,1 Volumen von 3 M Natriumacetat (pH 5,2) und 2,5 Volumina von 100 % Ethanol für 20 min auf Eis.
- Zentrifuge bei 16.000 x g für 20 min bei 4 ° C. Entfernen Sie den Überstand mit einer Pipette.
- Waschen Sie das Pellet zweimal mit 500 mL 70 % igem Ethanol. Entfernen Sie den Überstand mit einer Pipette.
- Trocknen Sie das Pellet auf Bank und Aufschwemmen in 35 ml 1 x TE-Puffer.
- Die DNA mit einem Fluorimeter (Table of Materials) mit DsDNA BR Assay Kit quantitate.
- Niederschlag von 5 mg von DNA von jeder Probe mit 0,1 Volumen von 3 M Natriumacetat, pH 5,2 und 2,5 Bände von 100 % EtOH für ein Gesamtvolumen von 200 mL (H2O hinzufügen, wenn nötig, zusätzliche 5 mg von DNA benötigt wird, wenn die neutralen Gel-Kontrolle notwendig ist). Inkubieren Sie für 20 min auf Eis.
- Zentrifuge bei 16.000 x g für 20 min bei 4 ° C. Entfernen Sie den Überstand mit einer Pipette.
- Trocknen Sie das Pellet auf Bank. Aufschwemmen der DNS in 14 mL H2O.
- Alkalische Hydrolyse und Gel-Elektrophorese
- 6 mL 1 M KOH hinzugeben und bei 55 ° C für 2 h inkubieren.
- Inkubieren Sie die Proben auf Eis für 5 min.
- Fügen Sie 4 mL 6 x Alkali DNA laden Puffer: 300 mM KOH, 6 mM EDTA, pH 8.0, 18 % Polysucrose (Table of Materials), 0,15 % Bromocresol grün und 0,25 % Xylol Cyanol FF.
- Werfen Sie eine alkalische Gel, die 1 % Agarose, 50 mM NaOH, 1 mM EDTA pH 8.0 besteht. Verwenden Sie einen Kamm, der für 24 mL DNA-Probe erlaubt, pro Bahn geladen werden.
- Führen Sie das Gel bei Raumtemperatur in 1 x Alkali Elektrophorese Puffer: 50 mM NaOH, 1 mM EDTA, pH 8.0. Enthalten Sie einen entsprechende DNA-Größe Marker.
- Spin-down die Rohre kurz und laden die gesamte 24 mL Probe in der alkalischen Agarosegel. Electrophorese für 30 min bei 30 V, gefolgt von 18-20 h bei 10 V.
- Das Gel für 2 Inkubationen von 45 min bei Raumtemperatur mit sanften Agitation in Neutralisation Puffer I: 1 M Tris-HCl, 1,5 M NaCl zu neutralisieren.
- Färben Sie die DNA durch Einweichen der neutralisierten Gels in 200 mL Wasser mit einer Verdünnung von 1/10.000 SYBR Gold Fleck für 2 h bei Raumtemperatur mit sanft schütteln.
- Visualisieren Sie die DNA mit Hilfe einer UV-Transilluminator. Erhöhter Alkali-Empfindlichkeit bewirkt, dass die Darstellung von kleineren DNA-Fragmenten, zeigt eine höhere Dichte von unrepariert Purine DNA-5.
- Südlichen transfer
- Übertragen Sie die DNA aus dem basischen Gel auf die positiv geladenen Nylon-Membran durch Kapillarwirkung10. In Sambrook und Russell es manuelle10kostenfrei ein Beispiel dafür, wie diese Übertragung einrichten kann.
- Einweichen der alkalische Gel für 15 min bei Raumtemperatur in alkalischen Puffer übertragen: 0,4 M NaOH, 1 M NaCl.
- Die Nylon-Membran (Schneiden auf die Größe des Gels) kurz mit Wasser nass, und dann einweichen für 5 min in alkalischen Puffer übertragen.
- Richten Sie die Transfer-Plattform durch invertieren 3 Glas Becher (100 mL) in ein Glas Auflaufform. Dieses Gericht sollte etwas größer als die Größe der Agarosegel. Legen Sie eine Glasplatte über Oberseite von ihnen.
- Drapieren Sie 2 große Stücke von 3 MM CHR Chromatographiepapier (Schneiden auf die Größe der Breite des Gel plus längeren Seiten, die über die Kanten in den Puffer Vorratsbehälter drapieren können) über die Glasplatte.
- Das Chromatographiepapier alkalischen Puffer übertragen übergießen und die Hälfte der Glasschale füllen. Glätten Sie die Bläschen mit einer 5-mL-Glas-Pipette.
- Legen Sie das Agarosegel auf, die Bläschen wieder glätten. Umgeben Sie alle Kanten des Gels mit Parafilm. Gießen Sie eine kleine Menge von alkalischen Puffer übertragen auf das Gel.
- Legen Sie die eingeweichter Nylon-Membran auf das Gel. Glätten Sie die Luftblasen.
- Nassen 2 Stück Papier schneiden auf die Größe der Agarose-Gel. Legen sie auf der Membran, und glätten Sie Bläschen.
- Legen Sie einen großen Stapel Papier Handtücher (Schnitt, etwas größer als das Agarosegel) auf den Transfer-Sandwich. Legen Sie vorsichtig eine Glasplatte auf das Küchenpapier. Fügen Sie eine gewichtete Objekt nach oben (ein Buch mit einer Masse von 400 g funktioniert gut).
- Lassen Sie die Übertragung über Nacht bei Raumtemperatur gehen.
- Sorgfältig die Transfer-Stack auseinander nehmen und genießen die Membran für 15 min bei Raumtemperatur in Neutralisation Puffer II: 0,5 M Tris-HCl, pH 7,2, 1 M NaCl.
- Crosslink die DNA der geladenen Nylon-Membran mit einer UV-Vernetzer. Legen Sie die Membran in der Vernetzer und verwenden Sie die Autocrosslink-Einstellung.
- Südlichen Sonde Vorbereitung
Hinweis: Zur Erkennung alkaliempfindlichen Websites in der im Entstehen begriffenen führenden-im Vergleich zu Verzögerungsstrang des DNA-Strangs basiert auf einem Protokoll der hier beschriebene Ansatz vorher11veröffentlicht. Für unser Experiment sondieren das URA3 Reportergen erfolgt auf die in einem der beiden Ausrichtungen (OR1 oder OR2) angrenzend an die ARS306 Replikation Herkunft auf Chromosom III (Abbildung 3A) eingefügt wurde. Die Anwesenheit von Alkali-empfindlichen Standorten in Hefe genomischer DNA ist ein Indikator für Ribonukleotidreduktase Gesellschaftsgründung, erlaubt die Verwendung von Purine als Biomarker der DNA-Polymerase-Aktivität12,13,14. Dieser Ansatz, mit einem Ziel DNA-Sequenz angrenzend an den effizienten ARS306 Ursprung der Replikation, sollte zu anderen genomischen Orten und Zielgene sowie zugänglich sein.- PCR-verstärken-520-Base paar Segment des reportergens URA3 unter Verwendung der folgenden Primerpaar: URA3-F1 (5´-GCTACATATAAGGAACGTGCTGC) und URA3-R1 (5´-CTTTGTCGCTCTTCGCAATGTC).
- Durchführung die PCR-Reaktion mit 10-20 ng Hefe genomic DNA als Vorlage, 4 μL jeder Grundierung (10 μM Lager), 10 μl 10 x Ex Taq Puffer (Mg2 + plus), 8 μl dNTP-Gemisch (2,5 mM), 0,5 μL des Ex-Taq DNA-Polymerase (5 U/μl) , und passen Sie das Endvolumen zu 100 μL mit H2O.
- Führen Sie das folgende PCR-Programm: 95 ° C für 5 min; 30 Zyklen von 95 ° C für 1 min, 55 ° C für 1 min, 72 ° C für 2 min; 72 ° C für 10 min und Halt bei 4 ° C.
- Reinigen Sie das PCR-Produkt mit einem PCR-Reinigung-Kit und eluieren der DNS mit 50 mL H2O. Hiermit kann jetzt als Vorlage in die radiolabeling Reaktion.
- Verwenden Sie die folgenden Primer für Strang-spezifische enzymatische: Verwendung URA3-A (5´-CTCATCCTAGTCCTGTTGCTGCC) zur Visualisierung von DNA, die Sonde A. glüht Dies entspricht der im Entstehen begriffenen führenden Strang DNA Wenn URA3 in OR2 und der rückständigen Strang DNA, wenn URA3 in OR1 ist Tnascent. Verwenden Sie URA3-B (5´-CAGTACCCTTAGTATATTCTCCAG) zur Visualisierung von entstehenden DNA, die Sonde B. glüht Dies entspricht der im Entstehen begriffenen führenden Strang DNA Wenn URA3 in OR1 und im Entstehen begriffene rückständigen Strang DNA Wenn URA3 OR2 wird (Abbildung 3).
- Führen Sie die radiolabeling Reaktion mit 10-20 ng verstärkt URA3 Fragment als Schablone DNA, 2 μL der Primer (10 μM bestand), 5 μL 10 x Ex Taq Puffer (Mg2 + plus), 4 μL von 2,5 mM dNTPs (abzüglich dCTP), 5 μL (50 μCi) der a -32P-dCTP , 0,5 μL der ExTaq DNA-Polymerase (5 U/μl) und die letzte Lautstärke zu 50 μL mit H2O. Run Folgendes für enzymatische Programm: 94 ° C für 5 min; 25 Zyklen von 94 ° C für 1 min, 55 ° C für 30 s, 72 ° C für 1 min; 72 ° C für 5 min und Halt bei 4 ° C.
- Verwenden Sie eine G-25 Spin-Spalte, um nicht inkorporierten radioaktiven Etikett zu entfernen. Vor Zugabe der radioaktiven Probe, Zentrifugieren der Spalte für 1 min bei 720 x g und Monta-Puffer durch pipettieren. Laden Sie die radioaktiven reaktionsproduktes und Zentrifuge für 2 min bei 720 X g, um die markierte Sonde eluieren.
- Inkubieren Sie bei 95 ° C für 5 min um die Sonde unmittelbar vor der Hybridisierung zu denaturieren. Kurz auf Eis abkühlen.
- Southern-Hybridisierung
- Die benetzte Membran in einer Hybridisierung Röhre Rollen und 25 mL frisch zubereitete Hybridisierung Puffer: 0,5 M-Natrium-Phosphat-Puffer, pH 7,2, 7 % Sodium Dodecyl Sulfat (SDS), 1 % Rinderserumalbumin (BSA). Bei 65 ° C für 1-2 h mit Drehung in eine Hybridisierung Ofen inkubieren.
- Vorsichtig Gießen Sie die Lösung und 25 mL Hybridisierung Puffer plus möglicher Sonde. Bei 65 ° C für 16-18 h mit Rotation inkubieren.
- Gießen Sie die Lösung in einen radioaktiven Abfallbehälter. Führen Sie 5 Wäschen von 5 min bei Raumtemperatur mit Phosphat-SDS waschen Lösung I: 40 mM Natrium-Phosphat-Puffer, pH 7,2, 5 % SDS, 0,5 % BSA, 1 mM EDTA, pH 8.0.
- Führen Sie 2 Wäschen von 15 min bei 65 ° C mit Phosphat-SDS waschen Lösung II: 40 mM-Natrium-Phosphat-Puffer, pH 7,2, 1 % SDS, 1 mM EDTA, pH 8.0.
- Entfernen Sie die Membran aus der Hybridisierung Tube mit einer Pinzette zu und legen Sie in einem geöffneten Kunststoffhülse Beschützer. Decken und setzen auf eine speicherfolie. Versuchen Sie eine Reihe von verschiedenen Belichtungszeiten von 4 h bis hin zu 4D.
- Falls erforderlich, Streifen und Re Sonde den Fleck mit einer anderen Sonde ca. 3 - 4 mal. Um zu Streifen, brüten Sie die Membran für 2 h bei 65 ° C in 25 mL einer 50 % Formamid, 2 X SSPE-Lösung.
Hinweis: 20 X SSPE-Stammlösung: 3 M NaCl, 0,2 M NaH2PO4, 0,02 M EDTA, pH 7.4. - Gießen Sie die Lösung in einen radioaktiven Abfallbehälter. Phosphat-SDS waschen Lösung II führen Sie 2 Wäschen von 15 min bei 65 ° C.
- Überprüfen Sie die Membran mit einem Geigerzähler und wiederholen Sie stripping Protokoll zu, wenn Signal bleibt.
- Führen Sie Pre-Hybridisierung und Hybridisierung, wie oben beschrieben.
2. Messung der Mutationsrate und Spezifität in S. Cerevisiae -Stämmen
- Vorbereiten der Zellkultur
- Impfen Sie eine einzige Kolonie (gesunder Hefe Zellen dauert ca. 2-3 Tage bei 30 ° C zu bilden, die richtige Größe Kolonien) gewachsen auf YPD Agar ergänzt mit 100 mg/mL Adenin in 5 mL YPDA Brühe ergänzt mit 250 mg/mL Adenin (die Ergänzung von Adenin ist nur notwendig wenn der verwendete Stamm ist ein Adenin-Auxotroph). Wachsen Sie in einem Shaker Inkubator oder auf ein Rotator bei 30 ° C, bis die Kultur Sättigung (36-48 h für eine Sorte ohne einen starken Wachstum defekt) erreicht.
- Spin-down der Zellen bei ~ 2000 X g für 5 Minuten.
- Den überstand verwerfen und die Zellen mit 5 mL sterilem Wasser zu waschen.
- Die Zellen in 500 mL sterilem Wasser aufschwemmen.
- Verdünnung der Zellkultur und Beschichtung
- Verdünnen Sie für nicht-Auswahl Steuerelemente die Zellen in einer 96-Well-Platte mit einem Faktor von 1.600.000 und Platte 100 mL einer jeden Kultur auf eine komplette Platte (COM).
- Wählen Sie für ura3 Mutanten, Platte 100 mL der unverdünnten Zellen auf einen 5-FOA Teller für ein Wildtyp (WT) Belastung. Verdünnen Sie die Zellen entsprechend, wenn die Mutationsrate der Belastung von Interesse wird voraussichtlich höher sein als in einem WT-Stamm.
- Can1 Mutanten auswählen, die Zellen 5-fach verdünnen und 100 mL auf einen Canavanine-haltigen Teller (CAN) für eine Belastung von WT-Platte. Der Verdünnungsfaktor ist 0,5.
- Inkubieren Sie die Platten bei 30 ° C bis Hefe Kolonien bilden. Für Stämme ohne Wachstum Mangel 2-3 Tage reicht in der Regel für COM und können Platten und 3-5 Tage für 5-FOA Platten. Um die Mutationsraten zwischen verschiedenen Stämmen zu vergleichen, ist es am besten, am selben Tag des Wachstums zu Kolonien zählen zu wählen. Speichern Sie die Platten bereit, in den kalten Raum (~ 4 ° C) gezählt werden.
- Berechnung der spontanen Mutationsrate
- Zählen Sie sichtbare Kolonien auf jeder Platte zu und machen Sie sich Notizen von Kolonie Zahlen
- Die Mutationsrate mit Drake Formel zu berechnen: μ = f ÷ ln (μNt). f ist die Mutation Frequenz berechnet, indem die Anzahl der Mutanten, geteilt durch die Anzahl der Zellen in der letzten Kultur. NT ist die Anzahl der Zellen in der letzten Kultur und μ ist die Mutationsrate. Diese Gleichung kann durch eine iterative Methode wie die Newton-Raphson Methode gelöst werden. Es gibt andere Schätzer Methoden, einschließlich des Lea und Coulson Tests, um die Mutation Rate15berechnen.
- Berechnen Sie das Konfidenzintervall vorausgesetzt Log Normalverteilung der Raten geht von einzelnen Isolate. Das 95 %-Konfidenzintervall wird am häufigsten verwendet.
- Analyse der Mutation Spektrum
- Wählen Sie eine einzige Kolonie von jeweils 5-FOA oder Platte können, und aktualisieren Sie auf einer YPDA Platte.
- Führen Sie Kolonie PCR URA3 oder CAN1 gen und Sequenz zu verstärken, um Mutationen, die unter Verwendung der folgenden Zündkapseln zu erkennen: URA3-F (5'-CGCATATGTGGTGTTGAAGAA-3 ") und URA3-R (5'-TGTGAGTTTAGTATACATGCA-3") für beide PCR-Amplifikation und Sequenzierung des Gens URA3 800 bp; CAN1-F (5'-CTTCTACTCCGTCTGCTTTC-3 ") und CAN1-R (5'-CAGAGTTCTTCAGACTTC-3") zur Verstärkung und CAN1-AR (5'-TGAAATGTGAAGGCAGCGTT-3 "), CAN1-9R (5'-CCTGCAACACCAGTGATA-3"), CAN1-10R (5'-GAGGATGTAACAGGGATGAAT-3 ") und CAN1-BR ( 5'-CGGTGTATGACTTATGAGGG-3 ") für die Sequenzierung des reportergens 1800 bp CAN1 .
- Um Kolonie PCR durchzuführen, eine einzige Kolonie in 5 μL von H2O aufschwemmen und 1 μl als Vorlage verwenden. Fügen Sie 1 μL jeder der Primer (5 μM bestand), 2 μL der 5 x KAPA2G-Puffer (Mg2 + plus), 0,5 μl dNTP (2,5 mM), 0,5 μL des KAPA2G schnell DNA-Polymerase, 12 μL von H2O.
- Für die Verstärkung des URA3 -Gens, führen Sie das folgende PCR-Programm: 95 ° C für 5 min; 5 Zyklen von 95 ° C für 10 s, 61 ° C für 15 s, 72 ° C für 30 s; 35 Zyklen von 95 ° C für 10 s, 61 ° C für 15 s, 72 ° C für 30 s; 72 ° C für 2 min und Halt bei 4 ° C. Für die Verstärkung des CAN1 Gens, führen Sie das folgende PCR-Programm: 95 ° C für 5 min; 5 Zyklen von 95 ° C für 10 s, 61 ° C für 15 s, 72 ° C für 45 s; 35 Zyklen von 95 ° C für 10 s, 61 ° C für 15 s, 72 ° C für 45 s; 72 ° C für 2 min und Halt bei 4 ° C.
- Richten Sie die Ergebnisse der Sequenzierung zu einer referenzsequenz mit einem Programm wie DNASTAR Seqman Pro oder Sequencher Mutationen zu identifizieren.
- Kompilieren Sie die Mutationen Daten durch Mutation Typ (Abbildung 4) oder Mutation Position (Abbildung 5). Berechnen Sie die Gesamtrate der Mutation die spezifische Mutation Preise durch die Häufigkeit einer Mutation (die Anzahl der beobachteten Ereignisse geteilt durch Anzahl der Mutanten, die sequenziert) multipliziert.
Ergebnisse
Behandlung von genomischer DNA mit Alkali, gefolgt von alkalischen Gelelektrophorese erlaubt semiquantitativen Nachweis von DNA-Fragmentierung wegen der Fülle an stabil eingebaute Purine. Abbildung 2 zeigt die Gelbilder der Hefe genomischer DNA mit oder ohne KOH5behandelt. Die M644L Variante des Pol2, die katalytische Untereinheit der Polε, hat die Möglichkeit, Purine zu integrieren, während die M644G mutierten mehr Purine als WT-Polymerase enthält reduziert. Gemäß dem Protokoll kann alkalische Gel Strang-spezifische Südlicher Fleck Analyse weiter sondiert werden. Mit dem Wissen über die Lage der sondierten genomische Website im Verhältnis zu seinen benachbarten Ursprung können wir selektiv Sonde Purine in der im Entstehen begriffenen integriert führende oder rückständigen Stränge. Abbildung 3 zeigt die Südlicher Fleck Ergebnisse sondieren das URA3 Reportergen in zwei entgegengesetzte Richtungen in der Nähe eine frühe Entlassung Replikation Ursprungs, ARS30616eingefügt. Durch den Einsatz führender Strang-spezifischen Sonden, beobachten wir das DNA-Fragmentierung Muster verursacht durch Alkali-Spaltung bei Purine in der im Entstehen begriffenen führenden Strang DNA in einem WT-Stamm und Stämme, die mit dem Ausdruck der Varianten der Polymerasen α, δ und ε, die für promiskuitiv sind Ribonukleotidreduktase Ver-
Verwenden eine Fluktuation Experiment, messen wir die Mutationsraten Hefestämme, die Reporter-Gene enthalten. Abbildung 4 zeigt die Mutationsraten bei URA3 und CAN1 Loci Stämme WT Polymerase oder den pol2-M644G -Mutanten mit oder ohne funktionale RNase H2 zum Ausdruck bringen. Das Fehlen von RNase H2 bewirkt eine moderate Erhöhung insgesamt Mutationsraten in beiden Hintergründe. Näherer Betrachtung der Mutation Besonderheit zeigt jedoch, dass in Stämmen RNase H2 fehlt (Abbildung 5), gibt es eine starke Zunahme der Mutationsrate für kurze Deletionen, besonders im Tandem repeat Sequenzen. Abbildung 5A vergleicht die Mutation Spezifität in WT und Rnh201Δ Stämme und Abbildung 5 b zeigt die mutagenen Wirkung der weiter erhöhten Ribonukleotidreduktase Einbeziehung in der im Entstehen begriffenen führenden DNA-Strang von Pol ε.
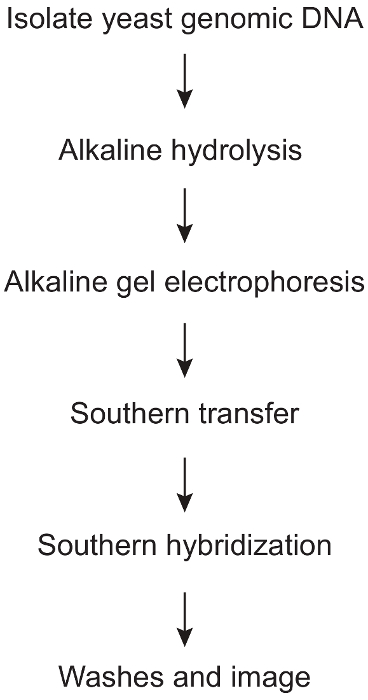
Abbildung 1: Protokoll Schritte bei der Strang-spezifischen Nachweis von alkaliempfindlichen Websites verursacht durch Ribonukleotidreduktase Einbindung in Hefe genomischer DNA. Eine Übersicht über die verschiedenen großen Schritte in diesem Verfahren, beginnend mit Hefe genomische DNA isoliert und durch die endgültige südlicher Fleckanalyse ausgehend. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: ein repräsentatives Bild der Ergebnisse durch alkalische Agarose-Gelelektrophorese erhalten. Diese Zahl wurde von Nick McElhinny, S. A. Et Al. angepasst 5. Hefe genomischer DNA aus Stämmen der verschiedenen Genotypen mit KCl (Neutral) oder KOH (alkalisch) behandelt wurde und dann getrennt auf einem neutralen oder alkalischen Agarosegel. "WT" und "M644G" zeigen den Status des Pol ε. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Strand-spezifische Sondierung von einer alkalischen Agarose-Gel durch Southern Blot. Diese Zahl wurde angepasst von Williams, J. S. Et Al. 13. (A) die radioaktiven Sonden Tempern, der im Entstehen begriffenen führenden DNA-Strang innerhalb der URA3 -Reporter-gen, die in der Nähe von ARS306 in zwei entgegengesetzte Richtungen (OR1 und OR2) eingefügt wurden. (B) repräsentative Ergebnisse der südlichen Beflecken mit der im Entstehen begriffenen führenden Strang-spezifischen Sonden. Angezeigt werden die bohrenden Ergebnisse für den im Entstehen begriffenen führenden Strang in einem Stamm mit WT DNA-Polymerasen oder pol1-L868M, pol2-M644G oder pol3-L612M Variante Stämme mit oder ohne RNase H2. Die POL1, POL2, und POL3 Gene kodieren katalytische Untereinheiten des Pols α, ε und δ, beziehungsweise. Die Varianten sind mehr promiscuous Ribonukleotidreduktase Aufnahme5,12,17. (C) Quantifizierung des radioaktiven Signals durch Bruch des gesamten in jeder Bahn aus (B). Werte werden als Prozentsatz der Summe angegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: spontane Mutationsraten in Hefestämme. Diese Zahl wurde von Nick McElhinny, S. A. Et Al. angepasst 5. URA3 und CAN1 Reporter-Gene wurden in den vorderen Mutation Assay verwendet, um die Mutationsraten der angegebenen Sorten messen. URA3 Reporter ist in "Lage 2" (OR2, Abbildung 3A). Das 95 %-Konfidenzintervall (CI) ist für jede Messung enthalten. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5: Mutation Spektren für das URA3-OR2 Reportergen. Diese Zahl wurde aus früheren Veröffentlichungen5,18,19,20angepasst. Die Position und Mutation Typ beobachtet in jeder unabhängige 5-FOA-resistenten Kolonie in der vorwärts-Mutation Assay ausgewählt werden dargestellt in der 804 bp URA3 kodierende Sequenz. Buchstaben bezeichnen Basis Substitutionen, offene Dreiecke zeigen einzelne base Löschungen, geschlossene Dreiecke zeigen einzelne base Einfügungen und durchgezogenen Linien zeigen kurze Deletionen. (A) Mutation Spektren in WT und rnh201Δ Stämme. Rote Etiketten über die Sequenz sind Mutationen in WT beobachtet, während blaue Etiketten unter der Sequenz in eine rnh201Δ Sorte beobachtet sind. (B) Mutation Spektren in pol2-M644G und pol2-M644G rnh201Δ Stämme. Rote Etiketten über die Sequenz sind Mutationen im pol2-M644G während blau unterhalb der Sequenz für pol2-M644G rnh201Δ -Stämme beobachtet. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Hier beschreiben wir die Protokolle für zwei Sätze von Experimenten, die häufig verwendet werden, um Purine aufgenommen während der DNA-Replikation und die mutagene Wirkung von unrepariert Purine Semi-quantitativ zu analysieren. Obwohl diese Ansätze die Modell Eukaryote S. Cerevisiaebeinhalten, können diese Techniken leicht zu anderen Mikroben und noch höheren Eukaryoten angepasst werden.
Sondierung für unrepariert Purine in DNA mit Hilfe von alkalischen Agarose Elektrophorese gepaart mit Southern blotting liefert wichtige Informationen über die Anzahl der Purine vorhanden und der Strang, in dem sie aufgenommen wurden. Es ist jedoch wichtig zu beachten, dass Alkali-Empfindlichkeit auch durch andere DNA-Läsionen, wie z. B. eine abasic Site verursacht werden kann. Ein weiteres Mittel zur Erkennung von Purine in der DNA ist durch die Behandlung mit gereinigtem RNase H2 Enzym wie in Säugerzellen21. Unser Ansatz beinhaltet, zwar sondieren für alkaliempfindlichen Fundplätze im Entstehen begriffenen führenden im Vergleich hinkt Stränge an das URA3 Reportergen in unseren Experimenten seiner Analyse verwendet ist es möglich, die Sonden zu entwerfen, die an anderen Orten in der Reihenfolge Tempern wichtige Informationen bezüglich Ribonukleotidreduktase Dichte an anderen genomischen Standorten zu gewinnen. Südlicher Fleck Teil des Verfahrens dient auch als Referenz für andere generische DNA bohrenden Experimente.
Messung von Mutationsraten mit einer Schwankung weithin für die Analyse der verschiedenen Mutagene Ereignisse in Mikroorganismen und somit beschränkt sich nicht auf Analyse der mutagenen Folgen der Purine in DNA. Für dieses Experiment kann die Zahl der unabhängigen Kulturen die Genauigkeit der Messung erheblich verbessern. Unabhängigen Kolonien erhalten Sie durch eine Belastung von Interesse auf YPDA Medium Streifen oder durch die Analyse der unabhängigen Spore Kolonien durch tetrade Dissektion des Fleckes diploiden Hefe gewonnen. Im Idealfall die Verwendung von mehreren unabhängigen haploiden Hefestämme (zB., aus tetrade Dissektion) wird angeregt, die Auswirkungen von Stamm zu Stamm Variation zu minimieren. Dies ist besonders wichtig, wenn die spontane Mutationsrate hoch ist oder wenn die Belastung ein Mangels Wachstum hat. Eine weitere Methode, die eingesetzt werden kann, um spontan zu messen ist die Mutationsrate eine Mutation Ansammlung experimentieren22. In dieser Analyse Mutation Häufigkeit richtet sich nach umfangreichen Wachstum für die Zellen und kann verwendet werden, um für die Mutationsrate berechnen. Kopplung von einer Mutation Ansammlung Experiment mit tiefen Sequenzierungstechnologie nachgewiesen wurde ein leistungsfähiges Werkzeug, um die Mutationsrate und Spezifität für das Genom zu analysieren sein.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir bedanken uns bei allen aktuelle und ehemalige Kunkel Lab Mitgliedern für ihre Arbeit und Diskussionen im Zusammenhang mit Protokoll und die hier vorgestellte Daten wiederverwendet. Diese Arbeit wurde von Projekt-Z01-ES065070, T.A.K. aus der Division des intramuralen Forschung von den National Institutes of Health (NIH), National Institute of Environmental Health Sciences (NIEHS) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| YPD media | 20 g dextrose, 20 g peptone, 10g yeast extract, in deionized H2O up to 1 L, add 20 g Bacto agar for solid media, autoclave. | ||
| COM plates | 1.3 g SC dropout mix, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar deionized H2O up to 1 L. Adjust pH to 5.8. Autoclave and 30 – 35 ml per plate. | ||
| CAN plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 1 L. Autoclave for 15 mins at 121 °C and cool down to 56 °C. Add 6 mL of filter-sterilized 1% canavanine sulfate solution. | ||
| 5-FOA plates | 1.3 g SC-Ura dropout powder, 1.7 g yeast nitrogen base without amino acids or (NH4)2SO4, 5 g (NH4)2SO4, 20 g dextrose, 20 g Bacto agar, 25 mg uracil and H2O up to 800 mL. Autoclave for 15 mins at 121 °C and cool down to 60 °C. Add 200 mL of filter-sterilized 0.5% 5-FOA solution. | ||
| L-Canavanine sulfate | US Biological | C1050 | |
| 5-FOA | US Biological | F5050 | |
| 20 mL glass culture tube | Any brand | ||
| Culture rotator in 30 °C incubator | Shaker incubator can be used instead | ||
| 96 well round bottom plates | Sterile, any brand | ||
| 3 mm glass beads | Fisher Scientific | 11-312A | Autoclave before use |
| 12-channel pipettes | Any brand | ||
| Ex Taq DNA Polymerase | TaKaRa | RR001 | |
| Epicentre MasterPure Yeast DNA Purification Kit | Epicenter | MPY80200 | |
| 3 M sodium acetate | Sigma-Aldrich | S7899 | |
| 100% ethanol | |||
| Qubit 2.0 Fluorometer | Invitrogen | Q32866 | Newer model available |
| dsDNA BR Assay kit | Invitrogen | Q32850 | |
| KOH | Sigma-Aldrich | 221473 | |
| EDTA | Sigma-Aldrich | E7889 | |
| Ficoll 400 | Dot Scientific Inc. | DSF10400 | |
| Bromocresol green | Eastman | 6356 | |
| Xylene cyanol FF | International Technologies Inc. | 72120 | |
| NaOH | Sigma-Aldrich | S8045 | |
| 1 M Tris-HCl (pH 8.0) | Teknova | T5080 | |
| SYBR Gold Nucleic Acid Gel Stain | Invitrogen | S11494 | |
| UV transilluminator | |||
| Amersham Nylon membrane Hybond-N+ | GE Healthcare | RPN303B | |
| 3 MM CHR Chromotography paper | Whatman | 3030-392 | |
| NaCl | Caledon | 7560-1 | |
| Stratalinker 1800 | Stratagene | ||
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| G-25 spin column | GE Healthcare | 27-5325-01 | |
| 1 M Sodium phosphate buffer (pH 7.2) | Sigma-Aldrich | NaH2PO4 (S9638); Na2HPO4 (S9390) | |
| SDS | Sigma-Aldrich | L4522 | |
| BSA | Sigma-Aldrich | A3059 | |
| Formamide | Sigma-Aldrich | 47671 | |
| Geiger counter |
Referenzen
- Joyce, C. M. Choosing the right sugar: how polymerases select a nucleotide substrate. Proceedings of the National Acaddemy of Sciences U S A. 94 (5), 1619-1622 (1997).
- Nick McElhinny, S. A., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proceedings of the National Acaddemy of Sciences U S A. 107 (11), 4949-4954 (2010).
- Sparks, J. L., et al. RNase H2-initiated ribonucleotide excision repair. Molecular Cell. 47 (6), 980-986 (2012).
- Li, Y., Breaker, R. R. Kinetics of RNA Degradation by Specific Base Catalysis of Transesterification Involving the 2'-Hydroxyl Group. Journal of the American Chemical Society. 121 (23), 5364-5372 (1999).
- Nick McElhinny, S. A., et al. Genome instability due to ribonucleotide incorporation into DNA. Nature Chemical Biology. 6 (10), 774-781 (2010).
- Williams, J. S., Kunkel, T. A. Ribonucleotides in DNA: origins, repair and consequences. DNA Repair (Amst). 19, 27-37 (2014).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nature Reviews Molecular Cell Biology. 17 (6), 350-363 (2016).
- Jones, M. E., Thomas, S. M., Rogers, A. Luria-Delbruck fluctuation experiments: design and analysis. Genetics. 136 (3), 1209-1216 (1994).
- Zheng, Q. A new practical guide to the Luria-Delbruck protocol. Mutat Research. 781, 7-13 (2015).
- Sambrook, J. Molecular Cloning: a Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2001).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The major roles of DNA polymerases epsilon and delta at the eukaryotic replication fork are evolutionarily conserved. PLoS Genetics. 7 (12), e1002407(2011).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides are signals for mismatch repair of leading-strand replication errors. Molecular Cell. 50 (3), 437-443 (2013).
- Williams, J. S., et al. Topoisomerase 1-mediated removal of ribonucleotides from nascent leading-strand DNA. Molecular Cell. 49 (5), 1010-1015 (2013).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nature Structural & Molecular Biology. 22 (3), 185-191 (2015).
- Foster, P. L. Methods for determining spontaneous mutation rates. Methods Enzymology. 409, 195-213 (2006).
- Williams, J. S., et al. Evidence that processing of ribonucleotides in DNA by topoisomerase 1 is leading-strand specific. Nature Structural & Molecular Biology. 22 (4), 291-297 (2015).
- Pavlov, Y. I., Shcherbakova, P. V., Kunkel, T. A. In vivo consequences of putative active site mutations in yeast DNA polymerases alpha, epsilon, delta, and zeta. Genetics. 159 (1), 47-64 (2001).
- Nick McElhinny, S. A., Kissling, G. E., Kunkel, T. A. Differential correction of lagging-strand replication errors made by DNA polymerases {alpha} and {delta}. Proceedings of the National Acaddemy of Sciences U S A. 107 (49), 21070-21075 (2010).
- Clark, A. B., Lujan, S. A., Kissling, G. E., Kunkel, T. A. Mismatch repair-independent tandem repeat sequence instability resulting from ribonucleotide incorporation by DNA polymerase epsilon. DNA Repair (Amst). 10 (5), 476-482 (2011).
- Lujan, S. A., et al. Mismatch repair balances leading and lagging strand DNA replication fidelity. PLoS Genetics. 8 (10), e1003016(2012).
- Reijns, M. A., et al. Enzymatic removal of ribonucleotides from DNA is essential for mammalian genome integrity and development. Cell. 149 (5), 1008-1022 (2012).
- Lujan, S. A., et al. Heterogeneous polymerase fidelity and mismatch repair bias genome variation and composition. Genome Research. 24 (11), 1751-1764 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten