Method Article
Метод ЧИП-экзо: Выявление белок-ДНК взаимодействий с Near пар оснований Precision
В этой статье
Резюме
Here, we present a protocol to achieve near base pair resolution of protein-DNA interactions. This is obtained by exonuclease treatment of DNA fragments selectively enriched by chromatin immunoprecipitation (ChIP-exo) followed by high throughput sequencing.
Аннотация
Иммунопреципитации хроматина (ЧИП) является незаменимым инструментом в области эпигенетике и регуляции генов, который изолирует специфические взаимодействия белок-ДНК. ЧИП в сочетании с высокой пропускной последовательности (чип-сл) обычно используется для определения геномных расположение белков, которые взаимодействуют с хроматином. Тем не менее, ЧИП-сл препятствует относительно низким разрешением отображения нескольких сотен пар оснований и высокого фонового сигнала. Метод ЧИП-экзо является усовершенствованной версией Обломок-SEQ, который существенно улучшает как разрешение и шума. Ключевое отличие методологии ЧИП-экзо является включение лямбда-экзонуклеазной пищеварение в технологическом процессе подготовки библиотеки для эффективного след левого и правого 5 'ДНК границы перекрещивающимися сайта белок-ДНК. Кристаллодержателя-экзо библиотеки затем подвергаются высокой пропускной последовательности. Полученные данные могут быть использованы для обеспечения уникальных и сверхвысокие понимание разрешения в функциональной организации Oе генома. Здесь мы опишем метод ЧИП-экзо, что мы оптимизировали и обтекаемый для систем млекопитающих и следующего поколения секвенирования посредством синтеза платформы.
Введение
Иммунопреципитации хроматина (ЧИП) является мощным методом для изучения механизмов регуляции генов путем селективного обогащения фрагментов ДНК, которые взаимодействуют с данным белком в живых клетках. Методы обнаружения фрагментов ДНК Обломок-обогащенная развивались , как технология улучшается, от обнаружения одного локуса (стандарт ЧИП-КПЦР) для гибридизации на олигонуклеотидных микрочипов (ЧИП-чип) до высокой пропускной последовательности (ЧИП-сл) 1. Хотя ЧИП-сл видел широкое применение, хроматина гетерогенность и неспецифических взаимодействий ДНК затрудняют качество данных, ведущий к ложным срабатываниям и неточного отображения. Чтобы обойти эти ограничения, д - р Франк Pugh разработал метод ЧИП-экзо 2. Характерной чертой чиповых-экзо является то, что она включает в себя 5 'к 3' экзонуклеазы, эффективно Footprinting связывания транскрипционных факторов местоположения. В результате методология ЧИП-экзо достигает более высокое разрешение, более широкий динамический диапазон detectioп, и нижний фоновый шум.
Хотя ЧИП-экзо является технически более сложным в освоении , чем Обломок-SEQ, она широко принята в качестве исследования стремятся получить уникальные ультра идеи высокого разрешения с использованием разнообразных биологических систем 3-8. Действительно, ЧИП-экзо был успешно применен для бактерий, дрожжей, мыши, крысы и клеточных систем человека. В качестве доказательства принципа, ЧИП-экзо первоначально использовался для определения точного связывания мотив для горсть дрожжей транскрипционных факторов 2. Этот метод был также использован в дрожжах , чтобы изучить организацию транскрипции предварительного комплекса инициации, и расшифровать subnucleosomal структуру различных гистонов 9,10. Совсем недавно мы использовали Обломок-экзо разрешить соседнюю TFIIB и Pol II связывания событий на человека промоутеров, и показал , что возникает широко распространена расходящимся транскрипции с особым местом инициации комплексов 11.
Рабочий процесс, представленный здесь оптимизирован и streamlined для млекопитающих Обломок-экзо (рисунок 1). Во- первых, живые клетки первичные или культуры тканей обрабатывают формальдегидом , чтобы сохранить в естественных условиях взаимодействия белок-ДНК посредством ковалентной сшивки. Клетки лизируют и хроматина стриженый ~ 100 - 500 фрагментов размером пар оснований. Микросхема выборочно обогащают для фрагментов ДНК сшивают с интересующего белка. На данный момент, Обломок-сл библиотеки обычно получают, что по своей сути ограничивает разрешение обнаружения до среднего размера фрагмента нескольких сотен пар оснований. Тем не менее, ЧИП-экзо преодолевает это ограничение путем обрезки левый и правый 5 'ДНК границы перекрещивающимися сайта белок-ДНК с лямбда-экзонуклеазы. библиотеки секвенирования затем построены из экзонуклеазной расщепленной ДНК, как описано ниже. Полученные в результате вложенные 5 'границы представляют собой след в естественных условиях взаимодействия белок-ДНК (рис 1, стадия 14), и обнаруживаются высокой пропускной последовательности. Altподжилки методология ЧИП-экзо более активное участие, чем Обломок-SEQ, переходы между большинством шагов требует простого мытья шарик, который сводит к минимуму потери пробы и экспериментальной изменчивости. Важно отметить, что так как ЧИП-экзо является усовершенствованной версией Обломок-, любой SEQ образца, который является успешным с микросхемой-SEQ должны также быть успешным с микросхемой-экзо.
Футпринтинга в естественных условиях взаимодействия белок-ДНК с результатами ЧИП-экзо в принципиально отличной от структуры данных Обломок-SEQ. Хотя общие Обломок-сл вызывающие абоненты могут быть применены к данным ЧИП-экзо, чтобы получить наиболее точные пиковые вызовы мы рекомендуем биоинформатики инструменты, специально разработанные с уникальной структурой данных ЧИП-экзо в виду. К ним относятся Genetrack, GEM, Мейс, Peakzilla и ExoProfiler 12-15.
протокол
Примечание: Двойной дистиллированная H 2 O или эквивалентный молекулярный класс рекомендуется для всех буферов и реакций смесей.
День 0: Подготовка материала и сотовый Урожай
1. Буфер Получение
- Готовят лизирующий Буферы 1 - 3 (таблицы 1 - 3) и 100 мкл полный ингибитор протеазы запас (CPI) в 50 мл каждого буфера непосредственно перед использованием. Подготовить запас CPI путем растворения одной таблетки в 1 мл молекулярного класса H 2 O.
- Приготовьте ChIP буферами (Таблицы 4 - 7). Добавьте 100 мкл CPI запаса до 50 мл блокирующего и Рипа буферов. Не добавляйте CPI в Трис и ЧИП элюирования буферов.
2. Отжиг адаптер Олигонуклеотиды
Примечание: Конкретные олигонуклеотидные последовательности могут быть найдены в разделе Дополнительная информация.
- Подготовить адаптер отжигом смесей (таблицы 8 - 9). Vortex смешивать и краткоспина трубки для сбора содержимого. Аликвоты по 100 мкл каждой смеси в 0,5 мл пробирки.
- Гибридизацию олигонуклеотида путем запуска программы (таблица 10) в Thermocycler.
Хранить отжигу олигонуклеотиды при -80 ° С.
3. В Vivo хроматина Сшивание с формальдегидом
Примечание: Для типичного ChIP эксперимента, исходный материал должен содержать примерно 50 миллионов клеток.
- Добавить свежий 37% метанол-свободного формальдегида маточного раствора в забуференном фосфатом физиологическом растворе (PBS), промывали клеточную культуру до конечной концентрации 1% (об / об), тщательно перемешать, и оставьте при комнатной температуре в течение 10 мин.
Примечание: Например, мы обычно добавляют 1,4 мл 37% свободного от метанола, формальдегида к клеткам в 50 мл PBS при комнатной температуре. Кроме того, избегайте формальдегида сшивающие в средствах массовой информации, так как белки в средствах массовой информации утолит некоторые из формальдегида. - Quench реакции сшивания путем добавления 2,5 М глицине для конечной концентрации 125 мМ. Тщательно перемешать.
- Передача клеток в 50 мл пробирку на льду. Спин клетки в течение 5 мин при 1000 х г при температуре 4 ° С. Слить супернатант.
- Добавить 1 мл охлажденного льдом 1X PBS к осадку клеток и ресуспендируют пипеткой. Переезд в 1,5 мл трубки.
- Спин клетки в течение 3 мин при 2000 х г при температуре 4 ° С. Отберите супернатант.
- Сразу Ледяная вспышка Клеточный осадок в 1,5 мл пробирки с жидким азотом. Хранить при температуре -80 ° С. Сшитые клетки стабильны до бесконечности при -80 ° С.
День 1: лизиса клеток, Ультразвуком и ЧИП
4. лизиса клеток
Примечание: Во всех секциях лизиса клеток, образцы должны храниться на льду или при температуре 4 ° С, чтобы свести к минимуму сшивок разворота.
- Кратко оттаивать сшитый осадок клеток. Тщательно ресуспендирования каждый осадок в 0,5 мл лизирующего буфера 1 и в сочетании с 4,5 мл лизирующего буфера 1 в полистирольной трубке 15 мл.
- Рок пробирки в течение 10 мин при 4 ° Cдля на качающейся платформе. Спин в течение 4 мин при 2000 х г при 4 ° С. Слить супернатант.
- Тщательно ресуспендирования каждый осадок в 0,5 мл лизирующего буфера 2 и добавляют 4,5 мл буфера для лизиса 2 раз ресуспендировали.
- Rock в течение 5 мин при 4 ° С на качающейся платформе. Спин в течение 5 мин при 2000 х г при 4 ° С. Слейте супернатант и осторожно нажмите избыток на бумажное полотенце.
- Тщательно ресуспендирования каждый осадок в 0,5 мл лизирующего буфера 3 и добавить 1 мл буфера для лизиса 3 раз ресуспендировали. Держите ядерные лизатов на льду и сразу же приступить к обработке ультразвуком.
5. Ультразвуком ядерной лизатов
Примечание: Во всех разделах ультразвуковой обработки, образцы должны храниться на льду, чтобы минимизировать сшивок разворот. Конкретная модель ультразвуковом используется совместно с протоколом ниже, можно найти в таблице материалов. Более подробная информация о конкретном использовании ультразвуковом и указаний в отношении других инструментов можно найти в разделе Дополнительная информация.
- Plасе резонансные адаптеры в 15 мл полистирольные пробирки, содержащие ядерные экстракты (металлический стержень не должен касаться стенки трубы).
- Соникатные ядерные лизаты в ледяной бане с холодной водой в течение 2 х 15 мин сессий с 30 сек ВКЛ / ВЫКЛ 30 сек при средней мощности. Только две разрушать ультразвуком 15 мл пробирки одновременно. Эти настройки работают в широком диапазоне клеточных линий и типов первичных клеток, но дальнейшая оптимизация может потребоваться, если хроматина механические ножницы неполна.
- Для проверки результатов обработки ультразвуком перенос 2 х 10 мкл образцов из лизата в 1,5 мл пробирки.
- Для обратного сшивок, объединить первые 10 мкл аликвоты 10 мкл ТЕ-РНКазы А и 0,2 мкл протеиназы K. инкубировать при 37 ° С в течение 30 мин. Добавить 4 мкл 6х красителя ДНК ксилола.
- Чтобы сохранить сшивок, объединить второй 10 мкл аликвоты с 2 мкл 6х красителя ДНК ксилола.
- Бегите оба образца на 1,5% агарозном геле при 140 В в течение приблизительно 30 - 45 мин до бромофенолового красителя в лестнице яs ¾ пути вниз геля.
- Если обработку ультразвуком успешно (большинство фрагментов ДНК срезанные до 100 - 500 пар оснований), передача лизаты обрабатывали ультразвуком в 2 мл пробирки, содержащие 150 мкл 10% Тритона Х-100. Vortex перемешать.
- Для того, чтобы осадить нерастворимый хроматина и твердые частицы, спин обрабатывают ультразвуком в ядерной лизата в течение 10 мин при 20000 х г при температуре 4 ° С.
- Передача супернатант в новую 2 мл пробирку. Немедленно приступить к микросхеме или хранения образцов при температуре -80 ° C до бесконечности. Обработанные ультразвуком экстракты не должны быть заморожены и разморожены более одного раза.
6. Сцепление антитела к Бисер
ПРИМЕЧАНИЕ: Никогда не вихревым или замораживания / оттаивания магнитных шариков, так как они будут разрушаться и увеличивать фоновый сигнал.
- После тщательного смешивания состава, аликвоты Y мкл шарик суспензии в 1,5 мл трубки, где Y = 1,1 х 2,5 х мкл (количество образцов CHIP). Связывание емкость для 2,5 мкл бисерной суспензии составляет до 13 мкг IgG. Вымойте Объединенные шарики 3x с 1 мл БлокировкаБуфер.
- Для более последовательного aliquotting шариков после стирок, ресуспендирования бусины в 10х первоначального объема суспензии (25 мкл / CHIP) блокирующим буфером. Алиготе 25 мкл на чип образца в 1,5 мл пробирки.
- Добавить 5 - 10 мкг антител к соответствующим аликвоты Ресуспендируют бусин. Принесите конечный объем до 250 мкл с блокирующего буфера. Инкубируйте образцы в течение 4 ч (в качестве альтернативы, в течение ночи в течение 16 часов) на вращающейся платформе при 4 ° С.
- Отберите супернатант. Для удаления несвязанного или избытка антитела, мыть шарики один раз с 1 мл блокирующего буфера.
- После аспирационных последней промывки немедленно добавить 50 миллионов клеток эквиваленты соницированного экстрактов (~ 1,6 мл), чтобы антитела: бусинки конъюгатов. Если экстракты обработанных ультразвуком не готовы, ресуспендирования бусины в 100 мкл блокирующего буфера, пока они не будут готовы. Крайне важно, чтобы никогда не позволить гранулы сухой.
7. иммунопреципитации хроматина (ЧИП)
- Для каждого образца Чипа, объединить 1,5 мл разрушать ультразвукомг экстрактов с антителами: Шарик конъюгаты в 1,5 мл или 2 мл пробирки.
- Выдержите трубки на ротатор мини-трубки в течение ночи (приблизительно 16 ч) со скоростью, установленной 9 при 4 ° С. Проверить после нескольких минут, чтобы обеспечить образцы не протекает и правильное смешивание.
День 2: ChIP Моет и на смоле ферментативных реакций
8. ChIP Моет
Примечание: Для того, чтобы свести к минимуму перекрестное загрязнение, кратко спина трубки между каждым мытьем. Для первого аспирацией, изменение советы между каждым образцом. В течение последующих промывок тот же наконечник может быть использован до тех пор, как он не касался бусинки. Дополнительная информация в разделе Указания по надлежащей ChIP промывки.
- Очень кратко спина трубки с помощью микроцентрифуге (~ 3 сек при 500 мкг) для сбора жидкости в шапках и разместить на магнитной стойке в течение 1 мин. В то время как все еще на магнитной стойке, экстракта аспирата.
- Добавить 0,75 мл RIPa буфера в каждую пробирку. Снимите трубки с магнитной стойкии инвертировать несколько раз перемешать. Заменить трубы на магнитной стойке и аспирата супернатант. Повторите 7x.
- Добавить 0,75 мл 10 мМ Трис HCl (рН 7,5), в каждую пробирку. Снимите трубки с магнитной стойки и инвертировать несколько раз перемешать. Заменить трубы на магнитной стойке и аспирата супернатант. Повторите 2 раза.
- После аспирационных последней промывки, переходите к полировать.
Примечание: После каждой реакции инкубации в разделах 9.3, 10.3, 11.3, 12.3, 13.3, 14.3 и 15.3, спиновые трубки кратко и место против магнитной стойке в течение 1 мин и аспирата супернатант. Промыть 2x с 0,75 мл RIPA буфером и 2 раза с 0,75 мл Трис-HCl (рН 7,5).
9. Полировка реакция
- Заполните Полировка расчеты основной смеси (таблица 11). Сделать Полировка смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу после последнего мытья ЧИП отсасывают, добавляют 50 мкл Полировка смеси для каждого образца смолы в то же время на магнитной стойке. Инкубируйте образцы в течение 30 мин при 3 мкгпри 30 ° С в термосмеситель.
- После аспирационных последней промывки, как описано в примечании после раздела 8.4, перейдите к A-хвостохранилище.
10. хвостохранилища реакция
- Заполните расчеты мастер - микс-хвостовых (таблица 12). Сделать хвостохранилища смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу же после того, как полирование секции, добавить 50 мкл хвостохранилища смеси для каждого образца смолы в то время как все еще на магнитной стойке. Инкубируйте образцы в течение 30 мин при 3 мкг при 37 ° С в термомиксер.
- После аспирационных последней промывки, как описано в примечании после раздела 8.4, перейдите к P7 лигирования адаптеров.
11. P7 Адаптер Лигирование реакции
- Заполните расчеты мастер Лигирование микс (Таблица 13). Сделать Лигирование смесь в 1,5 мл пробирку на льду. Пипетка перемешать.
- Сразу же после того, как раздел A-хвостохранилища, добавьте 48 мкл P7 Лигирование мастер-смеси и 2 мкл другого индекса Adapter тO каждый образец смолы в то же время на магнитной стойке. Инкубируйте образцы в течение 2 ч при 3 мкг при 25 ° С в термомиксер.
Примечание: Использование различных индексов для каждого образца позволит более образцов для секвенирования в клетке одного потока. - После аспирационных последней промывки, как описано в примечании после раздела 8.4, перейдите к Φ-29 Ник Ремонт.
12. Фи-29 Ник Ремонт Реакция
- Заполните Φ-29 вычисления основной смеси (таблица 14). Сделайте Φ-29 смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу же после раздела P7 лигирования, добавьте 50 мкл Φ-29 смеси для каждого образца смолы в то же время на магнитной стойке. Инкубируйте образцы в течение 20 мин при 3 мкг при 30 ° С в термосмеситель.
- После аспирационных последней промывки, как описано в примечании после раздела 8.4, перейдите к киназной реакции.
13. киназа реакции
- Заполните расчеты Master Mix киназа ( Таблица 15). Сделать киназа смешать в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу же после того, как Φ-29 секции, добавить 50 мкл киназной смешивать в каждый образец смолы в то время как все еще на магнитной стойке. Инкубируйте образцы в течение 20 мин при 3 мкг при 37 ° С в термомиксер.
- После аспирационных последней промывки, как описано в примечании после раздела 8.4, переходите к реакции Лямбда Экзонуклеазой.
14. Лямбда экзонуклеаза Реакция
- Наполните расчеты мастер смеси Лямбда экзонуклеазная (таблица 16). Сделать Лямбда экзонуклеаза смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу же после раздела киназного, добавьте 50 мкл Lambda экзонуклеазная перемешать до каждого образца смолы в то же время на магнитной стойке. Инкубируйте образцы в течение 30 мин при 3 мкг при 37 ° С в термомиксер.
- После аспирационных последней промывки , как описано в примечании после раздела 8.4, переходите к реакции RecJ ф.
15. RecJ еNuclease Реакция
- Заполните расчеты RecJ F мастер - смеси (таблица 17). Сделать RecJ F Перемешайте в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Сразу же после того, как секции Лямбда Экзонуклеазой, добавить 50 мкл RecJ F Перемешайте для каждого образца смолы в то время как все еще на магнитной стойке. Инкубируйте образцы в течение 30 мин при 3 мкг при 37 ° С в термомиксер.
- После аспирационных последней промывки, как описано в примечании после раздела 8.4, перейдите к чипу элюирования.
16. Элюирование и Crosslink Сторно
- Подготовка основной смеси, включающей: количество выборок х 1,1 х (200 мкл буфера для элюции ЧИП + 1 мкл 20 мг / мл протеиназы К) в 1,5 мл трубки. Добавить 200 мкл буфера для элюции ЧИП + протеиназы К основной смеси для каждого образца смолы.
- Инкубируйте образцы в течение ночи (приблизительно 16 ч) при 3 мкг при 65 ° С в термосмеситель с нагретой крышкой для предотвращения образования конденсата.
День 3: Извлечение ДНКиона и переходники Лигирование
17. Элюирование и Crosslink Реверсирование (продолжение)
- После инкубации в течение ночи при 65 ° С, кратко спина образцы для сбора конденсата. Место образцов на магнитной стойке в течение 1 мин.
- Передача 200 мкл супернатанта к новой 1,5 мл пробирку, содержащую 200 мкл ТЕ-буфера (рН 7,5).
18. Фенол Хлороформ изоамиловый спирт (PCIAA) Добыча
- Добавить 400 мкл фенола хлороформа и изоамилового спирта для каждого образца. Вихревой смешивать в течение 20 сек.
- Центрифуга в течение 10 мин при 20000 х г при комнатной температуре (RT). Аккуратно перенести 325 мкл верхнего водного слоя в свежую пробирку.
Примечание: Следите за тем, чтобы не передавать какие-либо из органического (нижний слой) в новую пробирку. - Добавьте 1/10 объема 3 М NaOAc (рН 5,5) и 1 мкл 20 мг / мл гликогена, к каждому образцу. Для нескольких образцов, подготовить мастер смеси.
- Добавьте 3 объемов охлаждаемого льдом 100% этанола к каждому образцу. вихревойсмешать. Выдержите в течение 15 мин при -80 ° С.
- Центрифуга в течение 15 мин при 20000 х г при 4 ° С. Супернатант осторожно декантируют, убедившись в том, чтобы не мешать гранул.
- Осторожно добавьте 500 мкл свежеприготовленные охлажденного льдом 70% этанола, убедившись в том, чтобы не мешать гранул. Центрифуга в течение 5 мин при 20000 х г при 4 ° С. Супернатант осторожно декантируют.
- Сухие гранулы в течение приблизительно 20 минут (или до полного высыхания) в вакууме скорость при 45 ° С.
ПРИМЕЧАНИЕ: Это точка паузы в протоколе. Сухие гранулы ДНК можно хранить при температуре -20 ° С. - Ресуспендируют гранул в 10 мкл DDH 2 O. пипеткой несколько раз над районом , где ДНК осаждали, даже несмотря на то, высушенный осадок не может быть видно.
- Перенесите 10 мкл каждого образца в свежую 0,3 мл ПЦР-пробирку или 8-стойку, в зависимости от количества проб.
19. P7 Primer Extension реакции
- Заполните P7 удлинения праймера расчеты основной смеси (таблица 18), Сделать Удлинитель смешать в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Добавляют 10 мкл смеси Extension (минус Φ-29 полимеразы), к каждому образцу 10 мкл. Пипетируйте перемешать.
- Выполнить образцы в Thermocycler с помощью программы (таблица 19) отжечь праймера с матрицей до 30 ° С "держать" шаг.
- Добавить 1 мкл Φ-29-полимеразы при 30 ° С "держать" шаг в программе. Пипетируйте перемешать. Резюме в оставшейся части программы (Таблица 19).
20. хвостохранилища реакция
- Заполните расчеты мастер - микс-хвостовых (Таблица 20). Сделать хвостохранилища смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Добавьте 10 мкл хвостохранилища смеси для каждого образца. Пипетируйте перемешать.
- В Thermocycler, инкубировать образцов в течение 30 мин при 37 ° С, а затем нагревать инактивированную в течение 20 мин при 75 ° С.
21. P5 Адаптер Лигирование реакции
- Фильл расчеты основной смеси Р5 адаптер Лигационные (таблица 21). Сделать Лигирование смесь в 1,5 мл пробирку на льду. Пипетируйте перемешать.
- Добавьте 20 мкл P5 лигирующей смеси для каждого образца. Пипетируйте перемешать.
- В Thermocycler, инкубировать образцов в течение 2 ч при 25 ° С.
22. Бусина Очистка
Примечание: Пожалуйста, смотрите раздел Материалы для деталей производителя на шарик очистить.
- Передача каждого образца 50 мкл на новый 1,5 мл трубки.
- Ресуспендируют очистить бусами на ротатор мини-трубки со скоростью, установленной 15 в течение 15 мин при комнатной температуре. Не позволяйте бусин отстояться перед пипеткой.
- Добавьте 60 мкл очистке шарики к образцу (1.2: 1 очистить шарики к образцу отношение имеет решающее значение для удаления ДНК, которая короче, чем 200 пар оснований). Пипетируйте перемешать в течение 20 сек.
- Ресуспендируют очистить бусами на ротатор мини-трубки со скоростью, установленной 15 в течение 3 мин при комнатной температуре. Кратко спина трубы.
- Место труб на магнитной стойке в течение 1 мин.Пипеткой прочь и отбросить супернатант.
ПРИМЕЧАНИЕ: НЕ используйте вакуум-аспирации в этом разделе, потому что он будет всасывать очистке бусы. - Добавьте 400 мкл свежеприготовленного RT 70% этанола каждого образца в то время как они находятся на магнитной стойке. Не смешивай. Отберите супернатант. Повторите 2 раза.
- Сушат очистке шарики в течение 10 мин при комнатной температуре. Цвет бисера будет потемнеть до светло-коричневого и очень маленькие трещины будут видны в осадке смолы, когда гранулы сухой.
- Для элюирования ДНК, добавляют 40 мкл 10 мМ Трис (рН 7,5). Тщательно пипеткой перемешать в течение 20 сек.
- Поместите образцы на магнитной стойке в течение 1 мин. Медленно пипеткой и переносят 36 мкл элюата непосредственно в 0,3 мл ПЦР-пробирки.
- Перейдем непосредственно к реакции или хранить элюатов ПЦР при -20 ° С.
День 4: ПЦР и гель анализ
23. ПЦР
- Заполните расчеты PCR Master Mix (таблица 22). Сделать ПЦР смесь. Пипеткой очень осторожно перемешать, чтобы избежатьпузыри.
- Добавьте 14 мкл ПЦР смесь к каждому образцу ДНК 36 мкл. Пипетируйте перемешать. Держите образцы на льду до готовности положить в Thermocycler.
- Для положительного контроля ПЦР, включают в заранее подготовленный библиотеку. Для отрицательного контроля ПЦР, включают в себя воду. Запуск образцов в Thermocycler с помощью программы ПЦР (табл. 23)
24. Гель Получение ДНК иссечение, и визуализация
- Тщательно очистить и промыть соответствующий размер геля коробку с деионизированной водой. Готовят 1,5% агарозном геле (содержащую 0,5 мг / мл этидий-бромида) с толстым, широким хорошо гребнем, который может содержать 60 мкл.
Примечание: этидий бромид (EtBr) является канцерогеном и должны быть обработаны с осторожностью. - Спин вниз ПЦР пробирки с образцами на короткое время для сбора конденсата.
- Добавьте 1/5 объема красителя ДНК ксилола 6х для комбинированного образца. Не используйте бромфенолового синего красителя в ДНК, поскольку она мигрирует в том же месте, что и ДНК образца.
- Загрузить весь образец Intо каждую лунку, предпочтительно с пустыми скважинами в между образцами.
Примечание: библиотеки с одинаковыми индексами не должен работать в том же самом геле. - Нагрузка 7 мкл 100 п.н. лестница с обеих сторон образцов. Запуск гель при 140 В в течение приблизительно 30-45 мин до бромфенол красителя в трапу пока ¾ путь вниз геля.
- Изображение и визуализировать гель на просвечивания при низкой установке УФ. Акцизный разделы агарозы, содержащие фрагменты ДНК 200 - 500 пар оснований. Будьте очень осторожны, чтобы избежать вырезания димера адаптер группа, которая работает на частоте 125 пар оснований.
- Поместите каждую вырезанную кусочек геля в 15 мл пробирку. Запись вес нетто каждого среза геля. Запишите этот вес непосредственно на трубе.
- Изображение, сохранять и аннотирования иссекают гель, чтобы подтвердить выбранный правильный диапазон размеров.
25. Гель Очистка
- Очищают ДНК из геля ломтиком вырезанной в соответствии с инструкциями изготовителя, со следующими изменениями.
- Растворите кусочек геля покачиваниемпри комнатной температуре на качающейся платформе. Это займет около 20 мин для растворения.
- Для получения высокоочищенного ДНК, мыть колонки с 0,5 мл буфера QG после растворенного геля прошедшего через колонку.
- После того, как буфер PE мыть, пусть колонки сидеть при комнатной температуре в течение 2 - 5 мин. Спин в течение 1 мин при 13000 х г при комнатной температуре.
- Для обеспечения возможности для реакции ПЦР смеси мастер-микс, Элюции образцов с 40 мкл EB буфера в свежую 1,5 мл трубки.
26. ДНК Количественное
- Подготовка образцов в соответствии с инструкциями изготовителя (таблица 24). Образцы Измерение в указанном приборе с оптическими трубками.
- После того, как библиотеки ЧИП-экзо количественно, представить для секвенирования на платформе , которая использует секвенирования посредством синтеза химии 16 , который совместим с адаптерами ДНК в этом протоколе.
Примечание: Как правило, 2 мкл образца достаточна для количественного определения, но более, может быть необходимо, если концентрация образца ниже. ДНК дает типичныйДиапазон LY от 50 до 200 нг. - После того, как количественному, хранить образцы при -20 ° С.
Результаты
Следующие цифры иллюстрируют репрезентативные результаты из протокола ЧИП-экзо, представленного здесь. В отличие от традиционных методик Обломок-сл с нескольких ферментативных стадий, ЧИП-экзо требует одиннадцать последовательно зависимых ферментативных реакций (рис 1). Таким образом, необходимо соблюдать осторожность при каждом шаге, чтобы гарантировать, что каждая компонента реакции добавляют к соответствующей главной реакционной смеси. Мы рекомендуем генерировать формульной таблицу, основываясь на реакции таблицах автоматически выполнять вычисления мастер реакционной смеси, печать результирующие таблицы, а затем проверять каждый отдельный элемент после того, как он будет добавлен в мастер-микс.
Мы используем целый ряд мер по контролю качества на протяжении всего протокола для обеспечения высокого качества результатов секвенирования (рисунок 2). Каждая реакция ЧИП содержит три основных компонента: 1) экстракт обрабатывают ультразвуком хроматина из клеток Интерее, 2) антитело, направленное против белка, представляющего интерес, и 3) ProteinG (или ProteinA) смолы для иммобилизации осажденных иммунных комплексов. Получение высококачественных экстрактов обрабатывают ультразвуком хроматина (фиг.2А) может быть довольно сложной задачей , так как условия обработки ультразвуком должны быть оптимизированы для каждого типа клеток и ультразвуковой обработки инструмента. Стоит времени , чтобы оптимизировать этот шаг проводить , поскольку самые высокие качества библиотеки начинают с в результате ультразвуковой обработки , что дает фрагменты ДНК в интервале от 100 - 500 пар оснований (рис 2А, "+" , полоса). Так как формальдегид сшивается лабильны и сам формальдегид имеет ограниченный срок годности, мы используем электрофоретического сдвига подвижности анализа (рис 2А, "-" полоса) , чтобы убедиться , что Озвученную экстракты содержат интактные сшивок белок-ДНК, очевидно , как супер-сдвиг ДНК-фрагменты. Для оценки фонового сигнала мы обычно выполняют "ложный" фишкой, что опускает антитела из реакции микросхеме. Высокое качество ЧИП-ехо подготовке библиотека будет иметь очень мало, если таковые имеются, фоновый сигнал в "притворной" фишкой ( "-") по отношению к конкретному ChIP антитела, в данном случае направлено против Pol II. Как видно на рисунке 2B, традиционные Обломок-сл библиотеки имеют значительно больше , чем фоновый сигнал Обломок-экзо. И, наконец, перед секвенирование, Обломок-экзо качества библиотеки оценивается на Bioanalyzer (рис 2C - D). Анализ точно измеряет распределение размера библиотеки и обнаруживает загрязняющие адаптер димеры (обозначается стрелкой), которые выполняются на 125 базисных пунктов. Если адаптер димеры присутствуют, то они снижают пропускную способность секвенирования. Поэтому, мы рекомендуем дополнительный шарик очистки, который будет эффективно удалять фрагментов ДНК менее 200 пар оснований.
ЧИП-экзо представляет собой мощный функциональный геномный метод, поскольку он является единственным методом, способным пространственно расходящиеся решения, инициируя, сделал паузу, и удлиняя РНК-полимеразы II Oна геному шкала (Рисунок 3) 11. Поскольку эти смежные связывающие события являются десятки пар оснований друг от друга, ЧИП-сл неспособен различить эти связывающие события с разрешающей способностью в несколько сотен пар оснований.

Рисунок 1: ЧИП-экзо схематичными. После того, как Чипа, адаптер Р7 лигируют с ультразвуковую обработку границ. Лямбда экзонуклеазная затем обрезает ДНК 5'-3 'до точки сшивок, таким образом Footprinting взаимодействие белок-ДНК. После элюирования и перекрещивающимися разворота, удлинения праймера синтезирует ДНК-дуплекс. И, наконец, Лигирование адаптера P5 обозначает левую и правую границы экзонуклеазы и полученную библиотеку подвергают высокой пропускной последовательности. Картирование концы 5'-тегов последовательностей на референсный геном разграничивает экзонуклеазной барьер и, следовательно, точное сайтом белок-ДНКсшивание. Рисунок редактировался Маном и Pugh 17. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
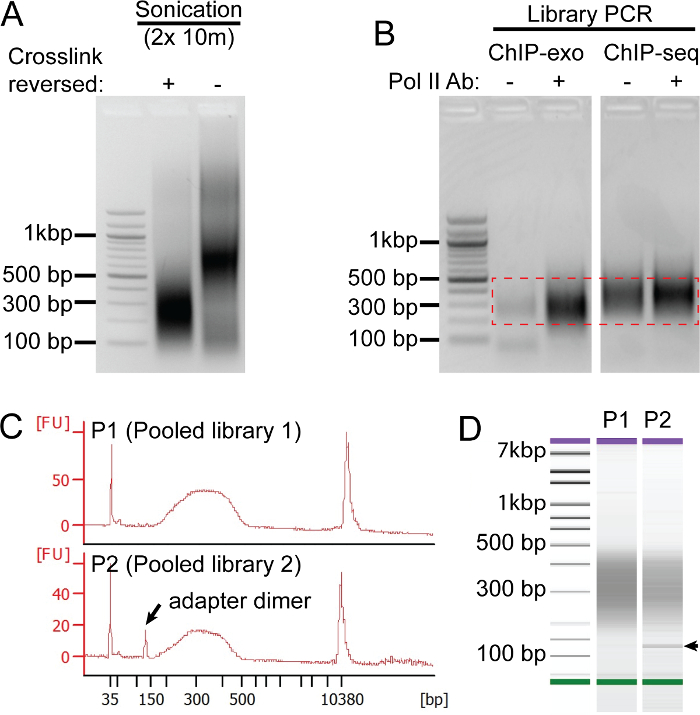
Рисунок 2: Контроль качества ЧИП-экзо. Панели переменного тока показывают репрезентативные результаты от несвязанных экспериментов. (А) Качество обработанной ультразвуком хроматина экстракта (из клеточной линии рака молочной железы HCC1806) оценивали с помощью электрофореза в агарозном геле на экстрактах с (+) и без (-) в пантографы наоборот. Малонарушенные сшивается будет вызывать комплексы белок-ДНК мигрировать медленнее. Таким образом, бег экстракт без сшивок реверсировать (-) позволяет за качество формальдегида сшивок быть оценены, которые имеют решающее значение для успешного микросхеме. (Б) Сравнение макете IP (-) ай Pol II (+) ЧИП-экзо и ЧИП-сл препараты библиотеки после 21 циклов ПЦР-амплификации. После ПЦР ДНК-фрагменты 200-500 п.о. вырезают (обозначается красной хэшированном коробкой) и очищали с использованием набора для экстракции геля. (Компакт - диск) В панели С, верхний след представляет собой идеальный след от объединенной библиотеки (Р1), а нижняя кривая показывает объединенном библиотеку (P2) , который содержит адаптер димеры. Панель D показывает график плотности ДНК, соответствующей библиотеки P1-2 следы от панели C (стрелка обозначает адаптер димера группу). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
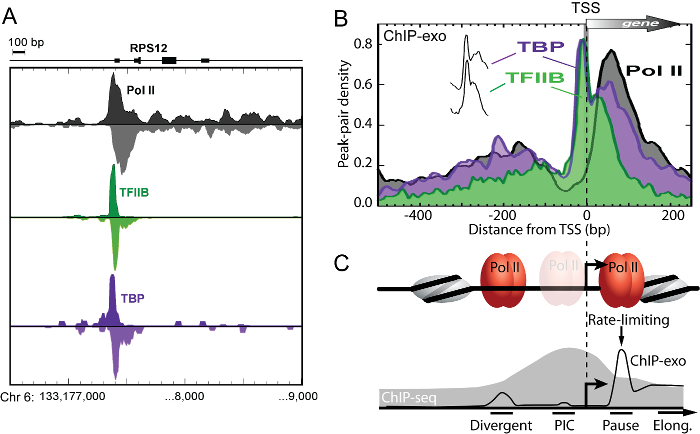
Рисунок 3: ЧИП-экзо Пространственно Решает Distinct двунаправленного инициации транскрипции Комплексы. (A) Smoothed распределение прядей-separatред ЧИП-экзо тег 5 'концы для Pol II, TFIIB и ТБФ в гене RPS12 человека в пролиферирующих клеток К562. (Б) Усредненные ЧИП-экзо узоры вокруг ближайшего RefSeq TSS. теги Пик-пара были выровнены по TSS гена-по-гена, Binned в неперекрывающихся интервалов 10BP по отношению к TSS, а затем среднее значение пиковая плотность пара по всей TFIIB занимаемого (п = 6,511) генов наносили на график, как процент от общего числа. Эти "шипы" ТБФ и TFIIB неразличимы (вертикально смещение в врезке). (C) Модель основана на данных панели B, иллюстрирующие различные инициации транскрипции комплексы решенные Обломок-экзо (черный след). Pol II заняли два отдельных разрешимые места, которые совпадали с участками расходящимся инициации транскрипции ( "расходящихся") и "Pause" сайтов. Это четкое пространственное разделение комплексов Pol II указывает на то, что расходящиеся стенограммы возникают из различных инициирующих комплексов. Подавляющее большинство Pol II сшитый або ут 50 б.п. ниже по потоку от TSS на сайте "Pause", где, как ожидается, чтобы сделать паузу после инициации транскрипции. Pol II был наиболее обедненный 20 - 60 п.н. выше УТП, где предварительно инициация сложные формы ( "ПИК"), что свидетельствует о том, что в среднем она вероятно тратит меньше времени, чем на приостановленных объектах. Это говорит о том, что в большинстве (но не обязательно все) случаи, когда-Поль II набирается, он быстро очищает промотор и предполагает Приостановленную-состояние примерно 30 - 50 пар оснований ниже по потоку от TSS, в соответствии с наблюдением, что выпуск Pol II пауза шаг, лимитирующей скорость в транскрипции. Эти смежные инициирующие комплексы являются неразрешимыми микросхемой-SEQ (на примере серой заполнения следа), поскольку его разрешение ограничено до нескольких сотен пар оснований. Рисунок редактировался Pugh и Venters 11. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
| реактив | Объем (мл) | [Final] |
| 1 М HEPES-KOH (рН 7,5) | 50 | 50 мМ |
| 5 М NaCl | 28 | 140 мМ |
| 0,5 М ЭДТА | 2 | 1 мМ |
| 100% Глицерин | 100 | 10% |
| 10% NP40 | 50 | 0,50% |
| 10% Triton X100 | 25 | 0,25% |
| DDH 2 O | Заполнить до 1 л |
Таблица 1. Рецепт буфера для лизиса 1. Фильтр с использованием фильтра 0,22 мкм. Хранить в 50 мл пробирки при 4 ° С. Добавьте 100 мкл CPI запас 50 мл буфера непосредственно перед использованием.
| реактив | Объем (мл) | [Final] |
| 1 М Трис-HCl (рН 8) | 10 | 10 мМ |
| 5 М NaCl | 40 | 200 мМ |
| 0,5 М ЭДТА | 2 | 1 мМ |
| 0,5 М EGTA | 1 | 0,5 мМ |
| DDh2 O | Заполнить до 1 л |
Таблица 2. Рецепт для Лизис буфера 2. Фильтр с использованием фильтра 0,22 мкм. Хранить в 50 мл пробирки при 4 ° С. Добавьте 100 мкл CPI запас 50 мл буфера непосредственно перед использованием.
| реактив | Объем (мл) | [Final] |
| 1 М Трис-HCl (рН 8) | 10 | 10 мМ |
| 5 М NaCl | 20 | 100 мМ |
| 0,5 М ЭДТА | 2 | 1 мМ |
| 0,5 М EGTA | 1 | 0,5 мМ |
| 10% деоксихолатом | 10 | 0,10% |
| N-лауроилсаркозин | 5 г | 0,5% (вес / объем) |
| DDH 2 O | Заполнить до 1 л |
Таблица 3. Рецепт буфера для лизиса 3. Фильтр с использованием фильтра 0,22 мкм. Хранить в 50 мл пробирки при 4 ° С. Добавьте 100 мкл CPI запас 50 мл буфера непосредственно перед использованием.
| реактив | Объем (мл) | [Final] |
| 10х PBS | 50 | 1x |
| Бычьим сывороточным альбумином | 2,5 г | 0,50% |
| DDH 2 O | Заполнить до 500 |
Таблица 4. Рецепт для блокировки Буфер. Фильтр с использованием фильтра 0,22 мкм. Хранить в 50 мл пробирки при 4 ° С. Добавьте 100 мкл CPI запас 50 мл буфера непосредственно перед использованием.
| реактив | Объем (мл) | [Final] |
| 1 М HEPES (рН 7,5) | 25 | 50 мМ |
| 0,5 М ЭДТА (рН 8) | 1 | 1 мМ |
| 10% деоксихолатом натрия | 35 | 0,70% |
| 10% NP40 | 50 | 1% |
| 1 M LiCl | 250 | 500 мМ |
| DDH 2 O | Заполнить до 500 |
Таблица 5. Рецепт для RIPA буфера. Фильтр с использованием фильтра 0,22 мкм. Хранить в 50 мл пробирки при 4 ° С. Добавьте 100 мкл CPI запас 50 мл буфера непосредственно перед использованием.
| реактив | Объем (мл) | [Final] |
| 1 М Трис-Cl (рН 7,5) | 2.5 | 50 мМ |
| 0,5 М ЭДТА | 1 | 10 мМ |
| 20% ДСН | 2.5 | 1% |
| DDH 2 O | Заполнить до 50 |
Таблица 6. Рецепт для ChIP элюции буфера. Фильтр с использованием фильтра 0,22 мкм. Хранить при комнатной температуре.
| реактив | Объем (мл) | [Final] |
| 1 М Трис-Cl (рН 7,5) | 0,5 | 10 мМ |
| DDH 2 O | Заполнить до 50 |
Таблица 7. Рецепт для ТЕ буфера. Фильтр с использованием фильтра 0,22 мкм. Хранить при температуре 4 ° С.
| Объем (мкл) | [Final] | |
| 100 мкМ ExA2-гХ | 75 | 15 мкМ |
| 100 мкМ ExA2-33 | 75 | 15 мкМ |
| 1 М Трис (рН 7,5) | 50 | 100 мМ |
| 5 М NaCl | 5 | 50 мМ |
| DDH 2 O | 295 | - |
| Общий объем | 500 |
Таблица 8. P7 адаптер Отжиг смеси.
| Объем (мкл) | [Final] | |
| 100 мкМ ExA1-58 | 75 | 15 мкМ |
| 100 мкМ ExA1-13 | 75 | 15 мкМ |
| 1 М Трис (рН 7,5) | 50 | 100 мМ |
| 5 М NaCl | 5 | 50 мМ |
| DDH 2 O | 295 | - |
| Общий объем | 500 |
Таблица 9. P5 адаптер Отжиг смеси.
| Temp (C) | Время |
| 95 | 5 мин |
| 72 | 5 мин |
| От 65 до 60 снижения рампы | 5 мин |
| От 55 до 50 снижения рампы | 3 мин |
| От 45 до 40 снижения рампы | 3 мин |
| 30 | 3 мин |
| 20 | 3 мин |
| 10 | 3 мин |
| 4 | навсегда |
Таблица 10. Адаптер Отжиг программы.
| 1x (мкл) | [Final] | |
| DDH 2 O | 39,8 | |
| 10x Реакционный буфер 2 | 5 | 1x |
| 100 мкМ АТФ | 0,5 | 1 мМ |
| 3 мМ дНТФ | 1.7 | 100 мкМ |
| 3 ед / мкл Т4-полимеразы | 1 | 3 U |
| 5 ед / мкл Кленова | 1 | 5 U |
| 10 ед / мкл Т4 полинуклеотидкиназы | 1 | 10 U |
| Общий объем реакционной смеси | 50 |
Таблица 11. Полировка мастер - микс.
| 1x (мкл) | [Final] | |
| DDH 2 O | 42,3 | |
| 10x Реакционный буфер 2 | 5 | 1x |
| 3 мМ дАТФ | 1.7 | 100 мкМ |
| 5 ед / мкл Кленова 3'-5 'Экзо минус | 1 | 5 U |
| Общий объем реакционной смеси | 50 |
Таблица 12. хвостохранилища мастер смеси.
| 1x (мкл) | [Final] | |
| DDH 2 O | 41 | |
| 100 мМ АТФ | 0,5 | 1 мМ |
| 10x Реакционный буфер 2 | 5 | 1x |
| 400 ед / мкл T4 ДНК Лигаза | 1.5 | 600 U |
| Объем смеси реакции | 48 | |
| 15 мМ адаптер Индекс | 2 | 30 пмоль |
| Общий объем реакционной смеси | 50 |
Таблица 13. P7 Адаптер Лигирование мастер - микс.
| 1x (мкл) | [Final] | |
| DDH 2 O | 41 | |
| 10x Φ-29 Буфер | 5 | 1x |
| 3 мМ дНТФ | 2.5 | 150 мкМ |
| 10 ед / мкл Φ-29-полимеразы | 1.5 | 15 U |
| Общий объем реакционной смеси | 50 |
Таблица 14. Φ-29 Ник мастер по ремонту микс.
| 1x (мкл) | [Final] | |
| DDH 2 O | 43,5 | |
| 100 мМ АТФ | 0,5 | 1 мМ |
| 10x Реакционный буфер 2 | 5 | 1x |
| 10 ед / мкл Т4 полинуклеотидкиназы | 1 | 10 U |
| Общий объем реакционной смеси | 50 |
Таблица 15. киназа мастер реакционной смеси.
| 1x (&# 956; л) | [Final] | |
| DDH 2 O | 43 | |
| 10x Лямбда буфера | 5 | 1x |
| 5 ед / мкл Лямбда экзонуклеазная | 2 | 10 U |
| Общий объем реакционной смеси | 50 |
Таблица 16. Лямбда экзонуклеаза Реакция мастер - микс.
| 1x (мкл) | [Final] | |
| DDH 2 O | 44 | |
| 10x Реакционный буфер 2 | 5 | 1x |
| 30 ед / мкл RecJ F экзонуклеазная | 1 | 30 U |
| Общая реакция volume | 50 |
Таблица 17. RecJ F Nuclease мастер реакционной смеси.
| 1x (мкл) | [Final] | |
| DDH 2 O | 6,45 | |
| 10x Φ-29 Буфер | 2 | 1x |
| 3 мМ дНТФ | 1.3 | 200 мкМ |
| 20 мкМ P7 Primer | 0,25 | 0,25 мкМ |
| Объем смеси реакции | 10 | |
| Осажденный этанол пробы | 10 | |
| Общий объем реакционной смеси | 20 |
Таблица 18. P7Удлинение праймера реакции мастер смесь.
| Temp (C) | Время |
| 95 | 5 мин |
| 65 | 5 мин |
| 30 | 2 мин |
| 30 | Удержание, пока не будет добавлено Φ-29 |
| 30 | 20 мин |
| 65 | 10 минут |
| 4 | навсегда |
Таблица 19. Программа P7 Primer Extension.
| 1x (мкл) | [Final] | |
| DDH 2 O | 5 | |
| 10x Реакция буфера 2 | 3 | 1x |
| 3 мМ дАТФ | 1 | 0,1 мМ |
| 5 ед / мкл Кленова 3 'к 5' экзо минус | 1 | 5 U |
| Объем смеси реакции | 10 | |
| Грунтовка расширенный образец | 20 | |
| Общий объем реакционной смеси | 30 |
Таблица 20. хвостохранилища мастер смеси.
| 1x (мкл) | [Final] | |
| DDH 2 O | 11.5 | |
| 10x T4 лигазы буфера | 5 | 1x |
| 15 мкМ ExA1-58 /13 адаптер | 2 | 30 пмоль |
| 400 ед / мкл ДНК-лигазы Т4 | 1.5 | 600 U |
| Объем смеси реакции | 20 | |
| A-белохвост образец | 30 | |
| Общий объем реакционной смеси | 50 |
Таблица 21. P5 Адаптер Лигирование мастер - микс.
| 1x (мкл) | [Final] | |
| 5x ПЦР буфера | 10 | 1x (2 мМ MgCl 2) |
| 10 мМ каждого дНТФ | 1 | 200 мкМ каждого |
| 20 мкМ P1.3 Primer | 1,25 | 0,5 мкМ |
| 20 мкМ P2.1 Primer | 1,25 | 0,5 мкМ |
| 2 ед / мкл Горячий старт полимеразной | 0,5 | 1 U |
| Объем смеси реакции | 14 | |
| Бусина элюировали образец | 36 | |
| Общий объем реакционной смеси | 50 |
Таблица 22. ПЦР.
| Temp (C) | Время | циклы |
| 98 | 30 сек | 1 |
| 98 | 10 сек | 15-21 (в зависимости от эффективности CHIP) |
| 52 | 30 сек | |
| 72 | 20 сек | |
| 72 | 2 мин | 1 |
| 4 | навсегда | Держать |
Таблица 23. Программа ПЦР.
| Стандартный Per ДНК (мкл) | За образец (мкл) | |
| 1: 200 разбавленный буфер смеси / краситель | 190 | 198 |
| стандарты ДНК | 10 | |
| библиотека ЧИП-экзо | 2 | |
| Общий объем | 200 | 200 |
Таблица 24. Количественное.
Обсуждение
Мы представляем функциональный геномный протокол для определения точного связывания расположение хроматина взаимодействующих белков непредвзято, общегеномного образом при близком разрешении пар оснований. Самым важным шагом для достижения вблизи базового разрешения отображения пара экзонуклеазная обработка ДНК Обломок-обогащается в то время как Иммунопреципитат остается на магнитной смолы. Якобы, белковые комплексы могут потенциально блокировать Footprinting в естественных условиях любого данного субъединиц (например, хроматина комплексов или нуклеосомнои коровой частицы). Тем не менее, как сообщалось ранее 10, так как формальдегид является неэффективным сшивающий, становится все менее вероятным , что несколько субъединиц комплекса будет сшивать ДНК и друг с другом в той же самой клетке в том же самом локусе. Таким образом, в естественных условиях футпринтинга отдельных субъединиц белкового комплекса, таких как индивидуальные гистонов субъединиц нуклеосомы, возможно с микросхемой-экзо.
т "> Наиболее заметные преимущества чиповых-экзо являются его разрешающая способность вблизи пар оснований и низкий фон. Ультра-высокое разрешение позволяет подробные структурные и пространственные идеи, которые будут сделаны на геном масштабе, которые в настоящее время не возможно с любым другим методом . с другой стороны, основным ограничением чиповых-экзо является то , что она является технически сложной молекулярно методологии биологии освоить. Кроме того, общие ограничения на этапе ChIP также применяются к чипу-экзо (например, коммерческой доступности антитела и специфичности , эпитоп доступность и сравнительно большое количество ячеек требуется). Распространенные ошибки включают плохое качество обработанной ультразвуком экстракты, с использованием класса антител не-фишку, а не держать образцы на льду в максимально возможной степени. Таким образом, условия обработки ультразвуком должны быть тщательно оптимизированы и каждый антитело подтверждено как описано выше 18 , чтобы избежать отрицательных последствий эти параметры могут иметь на экспериментальных результатах.Так далеко какГлубина секвенирование для целевого фактора транскрипции, мы, как правило, нацелены на около 20 миллионов однозначно выровнен читает. Так как Pol II и модификации гистонов более широко распространен, мы стремимся к 30 до 50 миллионов просмотров. Важно отметить, что, поскольку Обломок-экзо имеет значительно меньший фон, чем Обломок-SEQ, меньше чтений необходимы для достижения такой же глубины секвенирования.
Технология ЧИП-экзо в настоящее время широко применяется , несмотря на технические проблемы, так как надежные варианты по первоначальному протоколу продолжают публиковаться 17,19,20. В частности, один из вариантов , который может оказаться полезным для сложных скинуться белков называется Обломок-нексус, который использует один шаг перевязки , чтобы увеличить эффективность библиотечной подготовки 20. Таким образом, ЧИП-экзо это все чаще применяется и мощная методология для ультра-высокой разрешающей способностью отображения хроматина взаимодействующих белков в глобальном масштабе. Поскольку список коммерчески доступного ЧИП-класса муравейibodies продолжает расти, будущее применение методологии ЧИП-экзо будут направлены на картирование неизведанная генных регуляторных сетей, чтобы понять молекулярную схему ячейки в сверхвысоком разрешении. Кроме того, ЧИП-экзо, вероятно , будет доработана и адаптирована к занимаемой площади в естественных белок-РНК взаимодействий при близком разрешении пар оснований.
Раскрытие информации
The authors have nothing to disclose.
Благодарности
We thank the Venters Lab and members of the Molecular Physiology and Biophysics Department for helpful discussions. Special thanks to Frank Pugh for his guidance, mentoring, and many insightful discussions on the nuances of ChIP-exo while I was a post-doctoral fellow in his laboratory.
Материалы
| Name | Company | Catalog Number | Comments |
| 37% formaldehyde, methanol free, 10x10ml ampules | ThermoFisher Scientific | 28908 | Section 3 |
| Complete Protease Inhibitor cocktail (CPI) | Roche Life Science | 11873580001 | Sections 4, 8 |
| Bioruptor sonicator | Diagenode | UCD200 | Section 5 |
| 15 ml polystyrene tubes | BD Falcon | 352095 | Section 5 |
| MagSepharose Protein G Xtra beads | GE Healthcare | 28-9670-66 | Section 6 |
| DynaMag-1.5ml Side Magnetic Rack | Invitrogen | 12321D | Sections 6-17, 22 |
| Mini-Tube Rotator | Fisher Scientific | 05-450-127 | Sections 7, 22 |
| T4 DNA Polymerase | New England BioLabs | M0203 | Section 9 |
| DNA Polymerase I, Klenow | New England BioLabs | M0210 | Section 9 |
| 10x NEBuffer 2 (10x Reaction Buffer 2) | New England BioLabs | B7002 | Sections 9-11, 13, 15, 20 |
| Thermomixer C | Eppendorf | 5382 | Sections 9-16 |
| ATP | Roche Life Science | 010419979001 | Sections 9, 11, 13 |
| dNTPs | New England BioLabs | N0447 | Sections 9, 12, 19, 23 |
| T4 Polynucleotide Kinase | New England BioLabs | M0201 | Sections 9, 13 |
| dATP | New England BioLabs | N0440 | Sections 10, 20 |
| Klenow 3'-5' Exo Minus | New England BioLabs | M0212 | Sections 10, 20 |
| T4 DNA ligase | New England BioLabs | M0202 | Sections 11, 21 |
| Φ-29 DNA Polymerase | New England BioLabs | M0269 | Sections 12, 19 |
| 10x Φ-29 Buffer | New England BioLabs | B0269 | Sections 12, 19 |
| Lambda Exonuclease | New England BioLabs | M0262 | Section 14 |
| 10x Lambda Buffer | New England BioLabs | B0262 | Section 14 |
| RecJf Exonuclease | New England BioLabs | M0264 | Section 15 |
| Proteinase K | Roche Life Science | 03115828001 | Section 16 |
| Glycogen | Roche Life Science | 010901393001 | Section 18 |
| 10x T4 Ligase Buffer | New England BioLabs | B0202 | Section 21 |
| AMPure XP (clean up) beads | Beckman Coulter | A63881 | Section 22 |
| Q5 Hot Start DNA Polymerase | New England BioLabs | M0493 | Section 23 |
| 5x Q5 Buffer (5x PCR Buffer) | New England BioLabs | B9027 | Section 23 |
| Qubit Fluorometer | Invitrogen | Q33216 | Section 25 |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Section 25 |
| Optical Clear Qubit tubes | Invitrogen | Q32856 | Section 26 |
| Qubit dsDNA High Sensitivity Assay kit | Invitrogen | Q32851 | Section 26 |
Ссылки
- Venters, B. J., Pugh, B. F. How eukaryotic genes are transcribed. Crit Rev Biochem Mol Biol. 44, 117-141 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Chen, J., et al. Single-molecule dynamics of enhanceosome assembly in embryonic stem cells. Cell. 156, 1274-1285 (2014).
- Wales, S., Hashemi, S., Blais, A., McDermott, J. C. Global MEF2 target gene analysis in cardiac and skeletal muscle reveals novel regulation of DUSP6 by p38MAPK-MEF2 signaling. Nucleic acids research. 42, 11349-11362 (2014).
- Cho, S., et al. The architecture of ArgR-DNA complexes at the genome-scale in Escherichia coli. Nucleic Acids Res. 43, 3079-3088 (2015).
- Katainen, R., et al. CTCF/cohesin-binding sites are frequently mutated in cancer. Nat Genet. 47, 818-821 (2015).
- Murphy, M. W., et al. An ancient protein-DNA interaction underlying metazoan sex determination. Nat Struct Mol Biol. 22, 442-451 (2015).
- Zere, T. R., et al. Genomic Targets and Features of BarA-UvrY (-SirA) Signal Transduction Systems. PLoS One. 10, e0145035 (2015).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Rhee, H. S., Bataille, A. R., Zhang, L., Pugh, B. F. Subnucleosomal structures and nucleosome asymmetry across a genome. Cell. , 1377-1388 (2014).
- Pugh, B. F., Venters, B. J. Genomic Organization of Human Transcription Initiation Complexes. PLoS One. 11, e0149339 (2016).
- Albert, I., Wachi, S., Jiang, C., Pugh, B. F. GeneTrack--a genomic data processing and visualization framework. Bioinformatics. 24, 1305-1306 (2008).
- Guo, Y., Mahony, S., Gifford, D. K. High resolution genome wide binding event finding and motif discovery reveals transcription factor spatial binding constraints. PLoS computational biology. 8, e1002638 (2012).
- Bardet, A. F., et al. Identification of transcription factor binding sites from ChIP-seq data at high resolution. Bioinformatics. 29, 2705-2713 (2013).
- Wang, L., et al. MACE: model based analysis of ChIP-exo. Nucleic Acids Res. 42, e156 (2014).
- Bentley, D. R., et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456, 53-59 (2008).
- Rhee, H. S., Pugh, B. F. ChIP-exo method for identifying genomic location of DNA-binding proteins with near-single-nucleotide accuracy. Curr Protoc Mol Biol. Chapter 21, Unit 21.24 (2012).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22, 1813-1831 (2012).
- Serandour, A. A., Brown, G. D., Cohen, J. D., Carroll, J. S. Development of an Illumina-based ChIP-exonuclease method provides insight into FoxA1-DNA binding properties. Genome Biol. 14, R147 (2013).
- He, Q., Johnston, J., Zeitlinger, J. ChIP-nexus enables improved detection of in vivo transcription factor binding footprints. Nat Biotechnol. 33, 395-401 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены