Method Article
Il metodo ChIP-exo: Identificazione interazioni proteina-DNA con Vicino paia di basi di precisione
In questo articolo
Riepilogo
Here, we present a protocol to achieve near base pair resolution of protein-DNA interactions. This is obtained by exonuclease treatment of DNA fragments selectively enriched by chromatin immunoprecipitation (ChIP-exo) followed by high throughput sequencing.
Abstract
Immunoprecipitazione della cromatina (ChIP) è uno strumento indispensabile nel campo della epigenetica e regolazione genica che isola specifiche interazioni proteina-DNA. ChIP accoppiato ad alto rendimento sequenziamento (ChIP-seq) viene comunemente utilizzato per determinare la posizione genomica di proteine che interagiscono con cromatina. Tuttavia, Chip-Seq è ostacolata dalla risoluzione mappatura relativamente basso di diverse centinaia di paia di basi e di alta segnale di fondo. Il metodo ChIP-Exo è una versione raffinata di ChIP-Seq che migliora sostanzialmente al momento sia la risoluzione e rumore. La distinzione fondamentale della metodologia ChIP-Exo è l'incorporazione di lambda esonucleasi digestione nel flusso di lavoro di preparazione biblioteca all'impronta efficacemente sinistra e destra 5 confini 'del DNA del sito reticolazione proteina-DNA. Le librerie CHIP-exo vengono poi sottoposti ad alto rendimento sequenziamento. I dati risultanti può essere sfruttata per fornire intuizioni uniche e ultra-alta risoluzione nell'organizzazione funzionale of genoma. Qui, descriviamo il metodo ChIP-exo che abbiamo ottimizzato e semplificato per i sistemi di mammiferi e la piattaforma di sequenziamento per sintesi di nuova generazione.
Introduzione
Immunoprecipitazione della cromatina (ChIP) è un potente metodo per studiare i meccanismi di regolazione genica da selettivamente arricchente per frammenti di DNA che interagiscono con una certa proteina nelle cellule viventi. Metodi di rilevamento di frammenti di DNA chip arricchito si sono evoluti come la tecnologia migliora, dal rilevamento di un singolo locus (standard ChIP-qPCR) per ibridazione su microarray di oligonucleotidi (Chip-chip) a high-throughput sequencing (ChIP-Seq) 1. Anche se ChIP-Seq ha visto applicazione diffusa, l'eterogeneità della cromatina e le interazioni DNA aspecifiche hanno ostacolato la qualità dei dati che porta a falsi positivi e la mappatura imprecisa. Per aggirare queste limitazioni, il Dr. Frank Pugh ha sviluppato il metodo di ChIP-Exo 2. La caratteristica saliente di ChIP-Exo è che incorpora un 5 'a 3' esonucleasi, footprinting efficace fattore di trascrizione posizioni vincolanti. Come risultato, la metodologia ChIP-exo raggiunge risoluzione maggiore, una maggiore gamma dinamica di detection, e più basso rumore di fondo.
Anche se ChIP-exo è più tecnicamente impegnativo da padroneggiare rispetto ChIP-seq, esso viene ampiamente adottato come gli studi hanno lo scopo di acquisire conoscenze uniche ultra ad alta risoluzione utilizzando diversi sistemi biologici 3-8. Infatti, Chip-exo è stato applicato con successo a batteri, lieviti, topo, ratto, e sistemi di cellule umane. Come prova di principio, Chip-exo è stato originariamente utilizzato per identificare il motivo preciso vincolante per una manciata di trascrizione del lievito fattori 2. La tecnica è stata utilizzata anche nel lievito studiare l'organizzazione del complesso di pre-inizio della trascrizione, e per decifrare la struttura subnucleosomal di vari istoni 9,10. Più di recente, abbiamo sfruttato ChIP-exo per risolvere TFIIB adiacente e Pol II eventi a promotori umani vincolanti, e ha dimostrato che diffuso la trascrizione divergenti deriva da iniziazione distinti complessi 11.
Il flusso di lavoro qui presentata è ottimizzata e streamlined per mammiferi ChIP-Exo (Figura 1). In primo luogo, che vivono cellule di coltura primarie o tessuti sono trattati con formaldeide per preservare in vivo interazioni proteina-DNA attraverso una reticolazione covalente. Le cellule sono lisate e cromatina tosati a ~ 100 - 500 frammenti di dimensioni coppie di basi. ChIP poi arricchisce selettivamente per frammenti di DNA reticolati per la proteina di interesse. A questo punto, librerie ChIP-seq sono tipicamente preparati, che limita intrinsecamente la risoluzione di rilevamento per la dimensione media frammento di qualche centinaio di paia di basi. Tuttavia, ChIP-exo supera questa limitazione tagliando il diritto confini 5 'del DNA del sito reticolazione proteina-DNA con lambda esonucleasi e sinistro. librerie di sequenziamento sono poi costruiti da esonucleasi DNA digerito come di seguito dettagliato. I nidificate 5 'confini risultanti rappresentano un ingombro in vivo dell'interazione proteina-DNA (Figura 1, fase 14), e vengono rilevati da elevata produttività sequenziamento. although la metodologia ChIP-exo è più coinvolto di ChIP-seq, le transizioni tra la maggior parte delle operazioni richiede semplice lavaggio tallone, che riduce al minimo la perdita di campione e la variabilità sperimentale. È importante sottolineare che, dal momento che ChIP-Exo è una versione raffinata di ChIP-seq, qualsiasi campione che è successo con il circuito integrato-Seq dovrebbe anche avere successo con Chip-Exo.
Il footprinting in vivo di interazioni proteina-DNA con risultati ChIP-eso in una struttura dati fondamentalmente diversi dagli ChIP-seq. Anche se i chiamanti CHIP-ss incontrate possono essere applicate ai dati del chip-exo, per ottenere le chiamate di picco più precise si consiglia di strumenti bioinformatici specificamente progettati con la struttura unica di dati del chip-exo in mente. Questi includono Genetrack, GEM, MACE, Peakzilla, e ExoProfiler 12-15.
Protocollo
Nota: Double H 2 O distillata o grado equivalente molecolare è consigliato per tutti i buffer e le reazioni miscele.
Giorno 0: Preparazione del materiale e delle cellule Harvest
1. Preparazione Buffer
- Preparare Lysis Buffer 1 - 3 (tabelle 1 - 3) e aggiungere 100 ml completa inibitore della proteasi magazzino (CPI) a 50 ml di ogni buffer appena prima dell'uso. Preparare CPI magazzino sciogliendo una compressa in 1 ml di grado molecolare H 2 O.
- Preparare ChIP Buffer (Tabelle 4 - 7). Aggiungere 100 microlitri CPI stock da 50 ml di Blocco e buffer RIPA. Non aggiungere CPI di Tris e Chip eluizione buffer.
2. ricottura di oligonucleotidi Adapter
Nota: le sequenze oligonucleotidiche specifiche possono essere trovati nella sezione Informazioni aggiuntive.
- Preparare le miscele adattatore di ricottura (Tabelle 8 - 9). Vortex per miscelare e brevementeruotare i tubi per raccogliere contenuti. Un'aliquota di 100 ml di ogni mix in 0,5 ml tubi.
- Ibridare gli oligonucleotidi eseguendo il programma (Tabella 10) nel termociclatore.
Conservare ricotto oligonucleotidi a -80 ° C.
3. In Vivo cromatina reticolazione con formaldeide
NOTA: Per un tipico esperimento di chip, materiale di partenza dovrebbe contenere circa 50 milioni di cellule.
- Aggiungere fresca soluzione di formaldeide archivio esenti da metanolo 37% al tampone fosfato salino (PBS) lavato coltura cellulare ad una concentrazione finale di 1% (v / v), mescolare accuratamente, e lasciate riposare a temperatura ambiente per 10 min.
NOTA: per esempio, si aggiunge tipicamente 1,4 ml di formaldeide al 37% senza metanolo alle cellule in 50 ml di PBS a temperatura ambiente. Inoltre, evitare di formaldeide reticolazione in media da quando le proteine nei media spegnerà alcuni dei formaldeide. - Quench reticolazione reazione aggiungendo 2,5 M glicinae per una concentrazione finale di 125 mM. Mescolare accuratamente.
- Trasferire le cellule a tubo da 50 ml su ghiaccio. Spin cellule per 5 min a 1000 xga 4 ° C. Decantare il surnatante.
- Aggiungere 1 ml ghiacciata PBS 1x a pellet e risospendere pipettando. Trasferimento a 1,5 ml di tubo.
- Spin cellule per 3 min a 2000 xga 4 ° C. Aspirare il surnatante.
- Subito lampeggiare congelare pellet cellulare in 1,5 ml tubi con azoto liquido. Conservare a -80 ° C. cellule reticolati sono stabili a tempo indeterminato a -80 ° C.
Giorno 1: Cell Lysis, sonicazione, e Chip
4. cellulare Lysis
NOTA: Durante tutte le sezioni lisi delle cellule, i campioni devono essere tenuti in ghiaccio o a 4 ° C per ridurre al minimo l'inversione reticolazione.
- In breve disgelo reticolato pellet cellulare. Accuratamente risospendere ogni pellet in 0,5 ml di Lysis Buffer 1 e si combinano con 4,5 ml di Lysis Buffer 1 in una provetta di polistirene da 15 ml.
- Tubi di roccia per 10 minuti a 4 ° Cper su una piattaforma oscillante. Spin per 4 min a 2.000 xg a 4 ° C. Decantare il surnatante.
- Accuratamente risospendere ogni pellet in 0,5 ml di Lysis Buffer 2 e aggiungere 4,5 ml di Lysis Buffer 2 una volta risospeso.
- Oscillare per 5 minuti a 4 ° C su una piattaforma oscillante. Spin per 5 minuti a 2.000 xg a 4 ° C. Decantare il surnatante e delicatamente toccare in eccesso su carta assorbente.
- Accuratamente risospendere ogni pellet in 0,5 ml di Lysis Buffer 3 e aggiungere 1 ml di Lysis Buffer 3 una volta risospeso. Mantenere lisati nucleari sul ghiaccio e procedere immediatamente ad ultrasuoni.
5. sonicazione dei lisati nucleare
NOTA: Durante tutte le sezioni sonicazione, i campioni devono essere tenuti in ghiaccio per ridurre al minimo l'inversione reticolazione. Il modello specifico di sonicatore utilizzato in associazione con il protocollo di seguito si può trovare nella tabella dei materiali. Dettagli sul loro utilizzo sonicatore specifiche e linee guida per gli altri strumenti possono essere trovati nella sezione Informazioni aggiuntive.
- Pladattatori di risonanza asso in provette da 15 ml di polistirolo contenenti estratti nucleari (la barra metallica non deve toccare la parete del tubo).
- lisati nucleari Sonicare in un bagno di acqua ghiacciata per 2 x 15 sessioni minuti con 30 sec ON / OFF 30 sec a media potenza. sonicare Solo due provette da 15 ml alla volta. Queste impostazioni funzionano su una vasta gamma di linee di cellule e tipi di cellule primarie, ma un'ulteriore ottimizzazione può essere necessario se tosatura cromatina è incompleta.
- Per controllare i risultati sonicazione, il trasferimento di 2 x 10 campioni microlitri di lisato in provette da 1,5 ml.
- Per invertire legami crociati, unire la prima aliquota di 10 ml con 10 microlitri TE-RNasi A e 0,2 ml di proteinasi K. Incubare a 37 ° C per 30 min. Aggiungere 4 colorante xilene DNA microlitri 6x.
- Per conservare legami crociati, unire la seconda aliquota 10 microlitri con 2 ml 6x colorante xilene DNA.
- Eseguire entrambi i campioni su un gel di agarosio 1,5% a 140 V per circa 30 - 45 minuti fino a quando il colorante bromofenolo in scala is ¾ fino in fondo il gel.
- Se sonicazione era successo (la maggior parte dei frammenti di DNA tranciate a 100 - 500 bp), trasferimento sonicato lisati a 2 ml provette contenenti 150 ml di 10% Triton X-100. Vortex per miscelare.
- A pellet cromatina insolubile e detriti, di spin sonicato lisato nucleare per 10 minuti a 20.000 xg a 4 ° C.
- Trasferire il surnatante in una nuova provetta 2 ml. Procedere immediatamente a campioni di chip o conservare a -80 ° C a tempo indeterminato. estratti sonicato non devono essere congelati e scongelati più di una volta.
6. Giunto anticorpo per Beads
NOTA: Non vortice o di congelamento / scongelamento sfere magnetiche in quanto essi saranno in frantumi e aumentare segnale di fondo.
- Dopo aver accuratamente la miscelazione magazzino, un'aliquota Y ml tallone liquami in 1,5 ml tubo, dove Y = 1,1 x 2,5 x microlitri (numero di campioni di chip). capacità legante per 2,5 ml tallone impasto è fino a 13 mg IgG. Lavare perline pool 3x con 1 ml di bloccoBuffer.
- Per aliquotting più coerente delle perline dopo lavaggi, perline risospendere in 10x volume di liquami originale (25 ml / chip) con tampone di arresto. Aliquota 25 ml per campione ChIP in provette da 1,5 ml.
- Aggiungere 5 - 10 mg di anticorpo al corrispondente aliquota di perline risospendere. Portare il volume finale fino a 250 ml con tampone di arresto. Incubare i campioni per 4 ore (in alternativa, durante la notte per 16 ore) su una piattaforma rotante a 4 ° C.
- Aspirare il surnatante. Per rimuovere l'anticorpo non legato o in eccesso, lavare perline una volta con 1 ml di tampone di bloccaggio.
- Dopo aspirazione ultimo lavaggio, aggiungere immediatamente 50 milioni di equivalenti cellulari di estratti sonicati (~ 1,6 ml) di anticorpi: coniugati perline. Se gli estratti sonicati non sono pronti, perline risospendere in 100 microlitri tampone di bloccaggio fino a quando non sono pronti. E 'fondamentale per non lasciare mai le perle a secco.
7. Immunoprecipitazione della cromatina (ChIP)
- Per ogni campione di chip, combinare 1,5 ml di ultrasuonid estratti con anticorpi coniugati: tallone in 1,5 ml o 2 ml tubi.
- Incubare le provette su un rotatore mini-tubo durante la notte (circa 16 ore) alla impostazione della velocità di 9 a 4 ° C. Controllare dopo pochi minuti per garantire campioni non sono perdite e stanno mescolando in modo corretto.
2 ° giorno: Chip di lava e on-resina reazioni enzimatiche
8. lava ChIP
NOTA: Per ridurre al minimo la contaminazione incrociata, brevemente girare tubi tra ogni lavaggio. Per la prima aspirazione, cambiare punte tra ogni campione. Durante lavaggi successivi, la stessa punta può essere utilizzato fintanto che non toccare le perline. Vedere Informazioni aggiuntive Sezione indicazioni sul corretto lavaggio chip.
- Molto brevemente girare tubi usando una microcentrifuga (~ 3 sec a 500 xg) per raccogliere il liquido in tappi e posto sulla griglia magnetica per 1 min. Mentre ancora sulla cremagliera magnetica, estratto di aspirare.
- Aggiungere 0,75 ml RIPA tampone ad ogni provetta. Rimuovere i tubi di sostegno magneticoe capovolgere più volte per mescolare. Sostituire i tubi sulla cremagliera magnetica e aspirare il surnatante. Ripetere 7x.
- Aggiungere 0,75 ml 10 mM Tris HCl (pH 7.5) a ciascuna provetta. Rimuovere i tubi di sostegno magnetico e capovolgere più volte per mescolare. Sostituire i tubi sulla cremagliera magnetica e aspirare il surnatante. Ripetere 2x.
- Dopo aspirazione ultimo lavaggio, procedere alla lucidatura.
Nota: Dopo ogni reazione di incubazione nelle sezioni 9.3, 10.3, 11.3, 12.3, 13.3, 14.3, e 15.3, tubi di spin brevemente e posto contro sostegno magnetico per 1 min e aspirare il surnatante. Lavare 2x con 0,75 ml RIPA tampone e 2x con 0,75 ml di Tris HCl (pH 7,5).
9. lucidatura Reazione
- Compila lucidatura calcoli Master Mix (Tabella 11). Fai la lucidatura mix in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Subito dopo l'ultimo lavaggio chip è aspirato, aggiungere 50 ml di lucidatura mix per ogni campione di resina, mentre ancora sulla griglia magnetica. Incubare i campioni per 30 min a 3 xga 30 C in un Thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere alla A-tailing.
Reazione 10. A-tailing
- Compila calcoli Master Mix A-tailing (Tabella 12). Rendere A-tailing miscela in una provetta da 1,5 ml su ghiaccio. Pipetta per mescolare.
- Subito dopo la lucidatura sezione, aggiungere 50 ml di A-tailing mix per ogni campione di resina, mentre ancora sulla griglia magnetica. Incubare campioni per 30 min a 3 xg a 37 C in un thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere al P7 Adattatore legatura.
11. P7 Adattatore legatura di reazione
- Compila calcoli maestro legatura mix (Tabella 13). Fare legatura mescolare in 1,5 ml di tubo in ghiaccio. Pipetta per mescolare.
- Subito dopo A-tailing sezione, aggiungere 48 ml P7 legatura master mix e 2 ml di un diverso indice adattatore to ogni resina campione mentre ancora sulla griglia magnetica. Incubare i campioni per 2 ore a 3 XG a 25 C in un Thermomixer.
Nota: L'uso di diversi indici per ciascun campione consentirà più campioni da sequenziare in una singola cella di flusso. - Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere al Φ-29 Nick riparazione.
12. Phi-29 Nick riparazione Reazione
- Compila Φ-29 calcoli Master Mix (Tabella 14). Fare Φ-29 mix in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Subito dopo la sezione P7 legatura, aggiungere 50 ml di Φ-29 mix per ogni campione di resina, mentre ancora sulla griglia magnetica. Incubare i campioni per 20 minuti a 3 xg a 30 C in un thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere alla reazione di chinasi.
13. chinasi reazione
- Compila Kinase calcoli Master Mix ( Tabella 15). Fare chinasi mescolare in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Subito dopo la sezione Φ-29, aggiungere 50 ml di chinasi mescolano ad ogni resina campione mentre ancora sulla griglia magnetica. Incubare i campioni per 20 minuti a 3 xg a 37 C in un thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere alla reazione Lambda Exonuclease.
14. Lambda Exonuclease Reazione
- Compila Lambda esonucleasi calcoli Master Mix (Tabella 16). Fare Lambda Exonuclease mescolare in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Subito dopo la sezione chinasi, aggiungere 50 ml di Lambda Exonuclease mescolano ad ogni resina campione mentre ancora sulla griglia magnetica. Incubare campioni per 30 min a 3 xg a 37 C in un thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, procedere alla RecJ reazione f.
15. RecJ fReazione nucleasi
- Compila calcoli RecJ f Master Mix (Tabella 17). Fare RecJ f mix in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Subito dopo la sezione Lambda Exonuclease, aggiungere 50 ml di RecJ f mix per ogni campione di resina, mentre ancora sulla griglia magnetica. Incubare campioni per 30 min a 3 xg a 37 C in un thermomixer.
- Dopo aspirando ultimo lavaggio come descritto nella nota dopo la sezione 8.4, passare a chip eluizione.
16. eluizione e Crosslink inversione
- Preparare master mix: numero di campioni x 1,1 x (200 ml tampone di eluizione ChIP + 1 ml 20 mg / ml proteinasi K) in 1,5 ml tubo. Aggiungere 200 microlitri master mix ChIP tampone di eluizione + proteinasi K per ogni resina campione.
- Incubare campioni notte (circa 16 ore) a 3 xga 65 C in un thermomixer con un coperchio riscaldato per evitare la condensazione.
3 ° giorno: Estratto del DNAioni e adattatore di legatura
17. eluizione e Crosslink Reversal (continua)
- Dopo incubazione per una notte a 65 ° C, girare brevemente i campioni per la raccolta della condensa. I campioni sulla cremagliera magnetica per 1 min.
- Trasferire 200 microlitri surnatante di nuova provetta da 1,5 ml contenente 200 microlitri TE Buffer (pH 7,5).
18. fenolo cloroformio alcool isoamilico (PCIAA) Estrazione
- Aggiungere 400 microlitri fenolo cloroformio isoamilico alcol per ogni campione. Vortex per miscelare per 20 sec.
- Centrifugare per 10 min a 20.000 xg a temperatura ambiente (RT). Trasferire accuratamente 325 microlitri dello strato superiore acquosa in una nuova provetta.
Nota: Fare attenzione a non trasferire qualsiasi dell'organico (strato inferiore) nel nuovo tubo. - Aggiungere 1/10 volume di 3 M NaOAc (pH 5,5) e 1 ml di 20 mg / ml di glicogeno a ciascun campione. Per campioni multipli, preparare una master mix.
- Aggiungere 3 volumi di ghiaccio freddo 100% di etanolo per ogni campione. Vorticemescolare. Incubare per 15 minuti a -80 ° C.
- Centrifugare per 15 min a 20.000 xg a 4 ° C. decantare con attenzione il surnatante, facendo attenzione a non disturbare il pellet.
- Delicatamente aggiungere 500 ml appena fatte ghiacciata etanolo al 70%, facendo attenzione a non disturbare il pellet. Centrifugare per 5 min a 20.000 xg a 4 ° C. decantare attentamente surnatante.
- pellet secco per circa 20 minuti (o fino a secco) nel vuoto velocità di 45 ° C.
NOTA: Questo è un punto di pausa nel protocollo. pellet di DNA secchi possono essere conservati a -20 ° C. - Pellet Risospendere in 10 ml DDH 2 O. Dispensare più volte sopra la zona in cui, anche se il pellet secca non può essere visto il DNA pellet,.
- Trasferire 10 microlitri di ciascun campione in una nuova provetta 0,3 ml PCR o 8-cremagliera, a seconda del numero di campioni.
Reazione 19. P7 Primer Extension
- Compila P7 Primer Extension calcoli Master Mix (Tabella 18). Fare Extension Mix in 1,5 ml provetta in ghiaccio. Pipetta per mescolare.
- Aggiungere 10 ml di mix di estensione (meno Φ-29 polimerasi) per ogni campione 10 ml. Pipetta per mescolare.
- Campioni eseguito in termociclatore con il programma (Tabella 19) a ricottura primer per il modello fino a quando il 30 C "hold" passo.
- Aggiungere 1 ml Φ-29 polimerasi durante la 30 C "hold" passo nel programma. Pipetta per mescolare. Riprendere il resto del programma (Tabella 19).
Reazione 20. A-tailing
- Compila calcoli Master Mix A-tailing (Tabella 20). Rendere A-tailing miscela in una provetta da 1,5 ml su ghiaccio. Pipetta per mescolare.
- Aggiungere 10 ml di A-tailing mix per ogni campione. Pipetta per mescolare.
- Nel Termociclatore, incubare i campioni per 30 minuti a 37 ° C, e poi il calore inattivare per 20 minuti a 75 ° C.
21. P5 Adattatore legatura di reazione
- filL fuori P5 adattatore Ligation calcoli Master Mix (Tabella 21). Fare legatura mescolare in 1,5 ml di tubo in ghiaccio. Pipetta per mescolare.
- Aggiungere 20 ml di P5 legatura mescolano ad ogni campione. Pipetta per mescolare.
- Nel Termociclatore, incubare i campioni per 2 ore a 25 ° C.
22. Bead Pulizia
Nota: Si prega di consultare la sezione Materiali per i dettagli produttore sul tallone ripulire.
- Trasferire ciascun campione 50 microlitri di una nuova provetta da 1,5 ml.
- Risospendere ripulire perline su un rotatore mini-tubo alla regolazione della velocità del 15 per 15 minuti a temperatura ambiente. Non lasciate perline stabilirsi prima pipettaggio.
- Aggiungere 60 microlitri ripulire perline a campione (1.2: 1 ripulire perline per assaggiare il rapporto è fondamentale per rimuovere il DNA che è più breve di 200 paia di basi). Pipetta per mescolare per 20 sec.
- Risospendere ripulire perline su un rotatore mini-tubo alla regolazione della velocità del 15 per 3 minuti a temperatura ambiente. Brevemente girare tubi.
- Posizionare tubi sul sostegno magnetico per 1 min.Pipettare off e scartare il surnatante.
NOTA: NON utilizzare aspirazione a vuoto in questa sezione perché sarà aspirare le perle di clean-up. - Aggiungere 400 ml di appena fatto RT etanolo al 70% per ogni campione mentre sono sul sostegno magnetico. Non mescolare. Aspirare il surnatante. Ripetere 2x.
- Essiccare le perline di pulizia per 10 min a RT. Il colore delle perle diventerà scuro al marrone crepe e molto piccola luce saranno visibili nel pellet in resina quando perline sono asciutti.
- Per eluire il DNA, aggiungere 40 microlitri 10mM Tris (pH 7,5). Accuratamente pipetta per mescolare per 20 sec.
- Porre i campioni sul sostegno magnetico per 1 min. Lentamente pipettare e trasferire 36 ml eluato direttamente a 0,3 ml provette PCR.
- Procedere direttamente alla reazione o memorizzare eluati PCR a -20 ° C.
4 ° giorno: analisi PCR e Gel
23. PCR
- Compila calcoli PCR Master Mix (Tabella 22). Fai la PCR mix. Pipetta molto delicatamente per mescolare per evitarebolle.
- Aggiungere 14 ml di miscela PCR per ciascun campione di DNA 36 ml. Pipetta per mescolare. Mantenere i campioni in ghiaccio fino al momento di mettere in termociclatore.
- Per il controllo positivo di PCR, una biblioteca precedentemente preparato. Per il controllo negativo PCR, includere l'acqua. Campioni eseguito in termociclatore utilizzando programma PCR (Tabella 23).
24. Gel di Preparazione, DNA escissione, e visualizzazione
- Pulire e lavare una casella di gel di dimensioni adeguate con acqua deionizzata. Preparare un gel 1,5% (contenente 0,5 mg / ml di bromuro di etidio) con spessore, pettine largo-bene che può contenere 60 ml.
NOTA: il bromuro di etidio (EtBr) è un agente cancerogeno e deve essere maneggiato con cura. - Spin down tubi campione PCR brevemente per raccogliere la condensa.
- Aggiungere 1/5 volume di 6x tintura xilene DNA per campione combinato. Non utilizzare il blu bromofenolo in tinta DNA dal momento che migra nella stessa posizione come il campione di DNA.
- Caricare intero int campioneo ogni bene, preferibilmente con i pozzi vuoti in tra i campioni.
Nota: Le biblioteche con gli stessi indici non devono essere eseguiti nello stesso gel. - Carico 7 microlitri 100 bp scaletta su entrambi i lati dei campioni. Eseguire gel a 140 V per circa 30-45 minuti fino a quando bromofenolo colorante in scala è ¾ fino in fondo il gel.
- Immagine e visualizzare il gel su un transilluminatore a un livello basso UV. Asportare le sezioni di agarosio contenente frammenti di DNA 200-500 bp. Fate molta attenzione per evitare di tagliare fuori banda adattatore dimero che funziona a 125 bp.
- Mettere ogni fetta di gel asportato in un tubo da 15 ml. peso netto record di ogni fetta di gel. Scrivere questo peso direttamente sul tubo.
- Immagine, salvare e annotare il gel asportato per confermare la gamma di dimensioni corrette selezionato.
25. Gel Purificazione
- Purificare il DNA da gel fetta asportato in base alle istruzioni del fabbricante, con le seguenti modifiche.
- Sciogliere la fetta gel da dondoloa temperatura ambiente sul dondolo piattaforma. Ci vorranno circa 20 minuti per sciogliere.
- Per ottenere DNA altamente purificato, lavare colonne con 0,5 ml tampone QG gel dopo disciolto è passato attraverso colonna.
- Dopo il tampone di lavaggio PE, lasciate colonne siedono a temperatura ambiente per 2-5 min. Spin per 1 min a 13.000 xg a RT.
- Per lasciare spazio per master mix reazione PCR, campioni eluire con 40 microlitri EB tampone in una provetta da 1,5 ml.
26. DNA Quantificazione
- Preparare i campioni secondo le istruzioni del produttore (Tabella 24). Misura campioni in strumento specifico con tubi ottici.
- Una volta che le librerie ChIP-exo sono quantificati, presentare per il sequenziamento su una piattaforma che utilizza il sequenziamento per sintesi chimica 16 che è compatibile con gli adattatori di DNA in questo protocollo.
NOTA: Tipicamente, 2 microlitri di campione è sufficiente per la quantificazione, ma più può essere necessaria se la concentrazione del campione è inferiore. DNA produce tipicogamma ly tra 50 e 200 ng. - Dopo la quantificazione, conservare i campioni a -20 ° C.
Risultati
Le seguenti figure illustrano i risultati rappresentativi del protocollo ChIP-exo qui presentata. In contrasto con metodologie tradizionali chip ss con pochi passaggi enzimatici, Chip-exo richiede undici reazioni enzimatiche in sequenza dipendenti (Figura 1). Pertanto, occorre fare attenzione ad ogni passaggio per garantire che ciascun componente di reazione viene aggiunto al rispettivo master mix di reazione. Si consiglia di generare un foglio di calcolo formule in base alle tabelle di reazione per eseguire automaticamente i calcoli mix di master di reazione, la stampa delle tabelle conseguenti, e quindi il controllo via ogni voce dopo che è stato aggiunto al mix master.
Ci avvaliamo di una serie di misure di controllo della qualità in tutto il protocollo per garantire risultati di sequenziamento di alta qualità (Figura 2). Ogni reazione chip contiene tre componenti fondamentali: 1) sonicato estratto cromatina dalle cellule interest, 2) un anticorpo diretto contro la proteina di interesse, e 3) ProteinG (o Proteina) resina per immobilizzare gli immunocomplessi precipitati. Ottenere qualità sonicato estratti alti della cromatina (Figura 2A) può essere molto impegnativo dal momento che le condizioni di sonicazione deve essere ottimizzato per ogni tipo di cellula e strumento sonicazione. Vale la pena di tempo di ottimizzare questo passo spendere perché le librerie di altissima qualità iniziano con un risultato sonicazione che produce frammenti di DNA tra 100 - 500 bp (Figura 2A, "+" corsia). Dal legami crociati formaldeide sono labili e formaldeide per sé ha una shelf-life limitata, usiamo un test spostamento mobilità elettroforetica (Figura 2A, "-" corsia) per verificare che gli estratti sonicati contengono intatti legami crociati proteina-DNA, evidente come super-spostamento di i frammenti di DNA. Per valutare segnale di fondo che abitualmente eseguiamo un chip "finto" che omette di anticorpi dalla reazione chip. Una di alta qualità ChIP-exo preparazione biblioteca avrà molto poco, se del caso, il segnale di fondo nel chip "finto" ( "-") rispetto all'anticorpo ChIP specifico, in questo caso diretto contro Pol II. Come si vede nella figura 2B, librerie tradizionali chip seguenti sono sostanzialmente più segnale di fondo di ChIP-Exo. Infine, prima di qualità di biblioteca sequenziamento, Chip-Exo è valutata sulla Bioanalyzer (Figura 2C - D). Analisi misura con precisione la distribuzione delle dimensioni biblioteca e rileva contaminanti dimeri adattatore (indicati dalla freccia) che corrono a 125 bp. Se dimeri adattatori sono presenti, essi riducono la larghezza di banda di sequenziamento. Pertanto, si consiglia una pulizia tallone aggiuntivo che in modo efficiente rimuovere il DNA frammenti inferiore a 200 bp.
ChIP-Exo è una potente tecnica di genomica funzionale, perché è l'unico metodo in grado di risolvere spazialmente divergenti, avviando, in pausa, e allungamento RNA polimerasi II ona scala genome-wide (Figura 3) 11. Dal momento che questi eventi vincolanti adiacenti sono decine di coppie di basi a parte, Chip-Seq è in grado di distinguere questi eventi di legame con il potere di diverse centinaia di paia di basi risolvere.

Figura 1: Chip-exo schematica. Dopo Chip, l'adattatore P7 è legatura ai confini sonicazione. Lambda esonucleasi poi rifila DNA 5 'a 3' al punto di reticolazione, footprinting così l'interazione proteina-DNA. Dopo l'eluizione e reticolazione inversione, primer extension sintetizza il DNA duplex. Infine, la legatura dell'adattatore P5 segna i confini esonucleasi sinistro e destro e la libreria risultante viene sottoposta a high-throughput sequencing. Mappatura estremità 5 'del tag sequenza al genoma di riferimento delimita la barriera esonucleasi e quindi il luogo preciso della proteina-DNAreticolazione. Figura modificata da Rhee e Pugh 17. Clicca qui per vedere una versione più grande di questa figura.
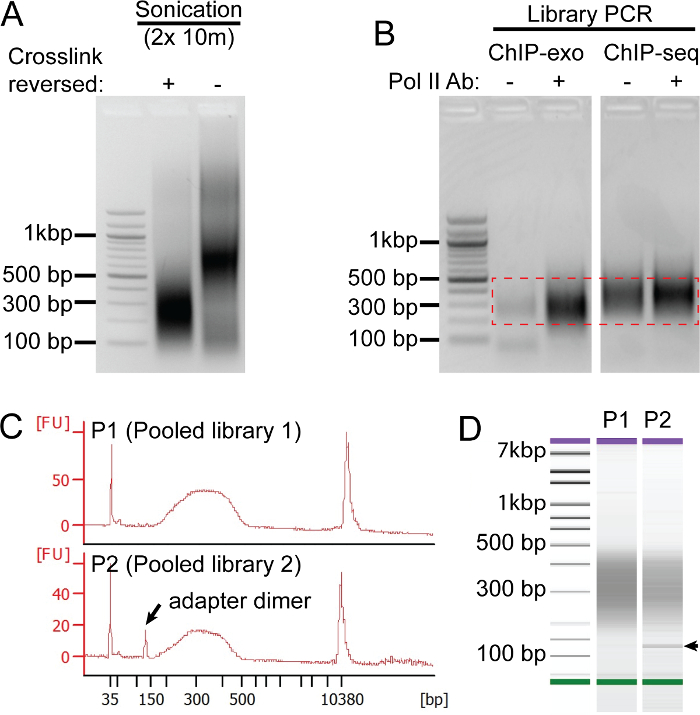
Figura 2: Controlli di Qualità ChIP-exo. Pannelli AC mostrano risultati rappresentativi da esperimenti indipendenti. (A) La qualità dell'estratto cromatina sonicato (dalla linea cellulare di cancro al seno umano HCC1806) è valutata mediante elettroforesi su gel di agarosio di estratti con (+) e senza (-) i legami crociati invertito. legami crociati intatti causerà complessi proteina-DNA a migrare più lento. Così, estratto di esecuzione senza reticolazioni invertito (-) consente la qualità dei legami crociati formaldeide da valutare, che sono critici per un chip successo. (B) Confronto di un IP finto (-) unND Pol II (+) ChIP-Exo e preparati libreria ChIP-Seq dopo 21 cicli di amplificazione PCR. Dopo la PCR, frammenti di DNA 200-500 bp vengono escisse (indicati con scatola hash rosso) e purificati utilizzando un kit di estrazione gel. (CD) Nel pannello C, la traccia superiore rappresenta un andamento ideale da una libreria pooled (P1), e la traccia inferiore mostra una libreria pooled (P2) che contiene dimeri adattatore. Pannello D mostra la trama densità di DNA corrispondenti alle tracce della biblioteca P1-2 dal pannello C (freccia indica adattatore banda dimero). Clicca qui per vedere una versione più grande di questa figura.
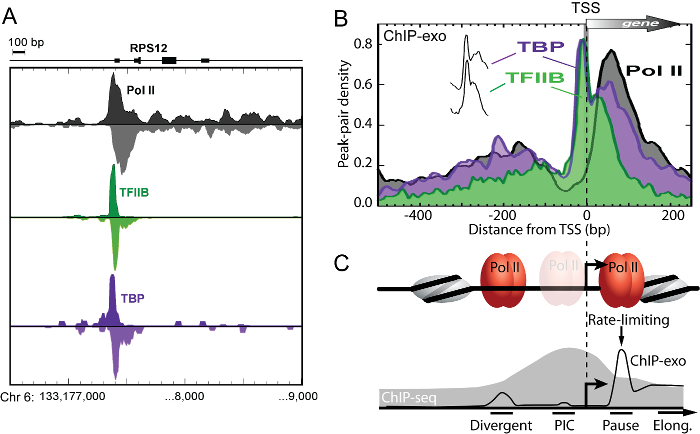
Figura 3: Chip-exo Risolve spazialmente distinto bidirezionale trascrizione iniziazione Complessi. (A) la distribuzione lisciato di Strand-separatEd ChIP-Exo tag 5 'finisce per Pol II, TFIIB, e TBP al gene RPS12 umano in proliferanti K562 cellule. (B) Averaged modelli ChIP-exo tutto il RefSeq più vicino TSS. Picco coppie tag sono stati allineati al TSS gene-by-gene categorizzata in non si sovrappongono gli intervalli di 10bp relativi al TSS, e quindi il valore medio di picco paio densità in tutti TFIIB-occupati (n = 6.511) geni sono stati tracciati come una percentuale del totale. I "picchi" di TBP e TFIIB sono indiscernibili (spostato verticalmente nel riquadro). (C) modello basato su dati panel B, illustrando distinte trascrizione complessi di iniziazione risolti da Chip-Exo (traccia nera). Pol II occupava due posizioni risolvibili separati che coincidono con i siti di trascrizione divergenti iniziazione ( "divergente") e siti di "pausa". Questa chiara separazione spaziale dei complessi Pol II indica che le trascrizioni divergenti derivano da distinti complessi di iniziazione. La stragrande maggioranza di Pol II ABO reticolato ut 50 bp a valle del TSS al sito "Pausa", in cui si prevede di pausa dopo l'inizio della trascrizione. Pol II è stato più impoverito 20 - 60 bp a monte del TSS dove il pre-iniziazione complesso ( "PIC") forme, indicando che in media è probabile che spende meno tempo lì che presso i siti in pausa. Questo suggerisce che nella maggior parte (ma non necessariamente tutti) i casi, una volta Pol II è reclutato, azzera velocemente il promotore e assume uno-stato di pausa di circa 30 - 50 bp a valle del TSS, coerente con l'osservazione che il rilascio Pol II pausa è un fattore limitante nella trascrizione. Questi complessi di iniziazione adiacenti sono irrisolvibile da Chip-Seq (illustrato da grigio traccia di riempimento) poiché la sua risoluzione è limitata a poche centinaia di paia di basi. Figura modificata da Pugh e Venters 11. Clicca qui per vedere una versione più grande di questa figura.
| Reagente | Volume (ml) | [Finale] |
| 1 M HEPES-KOH (pH 7,5) | 50 | 50 mm |
| 5 M NaCl | 28 | 140 mm |
| 0.5 M EDTA | 2 | 1 mM |
| 100% di glicerina | 100 | 10% |
| 10% NP40 | 50 | 0,50% |
| 10% Triton X100 | 25 | 0,25% |
| DDH 2 O | Riempire a 1 L |
Tabella 1. Ricetta per Lysis Buffer 1. Filtro utilizzando 0,22 micron filtro. Conservare in provette da 50 ml a 4 ° C. Aggiungere 100 microlitri CPI stock da 50 ml di tampone appena prima dell'uso.
| Reagente | Volume (ml) | [Finale] |
| 1 M Tris-HCl (pH 8) | 10 | 10 mM |
| 5 M NaCl | 40 | 200 mM |
| 0.5 M EDTA | 2 | 1 mM |
| 0,5 M EGTA | 1 | 0,5 mm |
| DDH2 O | Riempire a 1 L |
Tabella 2. Ricetta per Lysis Buffer 2. Filtro utilizzando 0,22 micron filtro. Conservare in provette da 50 ml a 4 ° C. Aggiungere 100 microlitri CPI stock da 50 ml di tampone appena prima dell'uso.
| Reagente | Volume (ml) | [Finale] |
| 1 M Tris-HCl (pH 8) | 10 | 10 mM |
| 5 M NaCl | 20 | 100 mM |
| 0.5 M EDTA | 2 | 1 mM |
| 0,5 M EGTA | 1 | 0,5 mm |
| 10% Desossicolato | 10 | 0,10% |
| N-lauroylsarcosine | 5 g | 0.5% (w / v) |
| DDH 2 O | Riempire a 1 L |
Tabella 3. Ricetta per Lysis Buffer 3. Filtro utilizzando 0,22 micron filtro. Conservare in provette da 50 ml a 4 ° C. Aggiungere 100 microlitri CPI stock da 50 ml di tampone appena prima dell'uso.
| Reagente | Volume (ml) | [Finale] |
| 10x PBS | 50 | 1x |
| Albumina sierica bovina | 2,5 g | 0,50% |
| DDH 2 O | Riempire a 500 |
Tabella 4. Ricetta per il blocco Buffer. Filtrare su filtro di 0,22 micron. Conservare in provette da 50 ml a 4 ° C. Aggiungere 100 microlitri CPI stock da 50 ml di tampone appena prima dell'uso.
| Reagente | Volume (ml) | [Finale] |
| 1 M HEPES (pH 7.5) | 25 | 50 mm |
| 0.5 M EDTA (pH 8) | 1 | 1 mM |
| 10% di sodio Desossicolato | 35 | 0,70% |
| 10% NP40 | 50 | 1% |
| 1 M LiCl | 250 | 500 mm |
| DDH 2 O | Riempire a 500 |
Tabella 5. Ricetta per RIPA buffer. Filtrare su filtro di 0,22 micron. Conservare in provette da 50 ml a 4 ° C. Aggiungere 100 microlitri CPI stock da 50 ml di tampone appena prima dell'uso.
| Reagente | Volume (ml) | [Finale] |
| 1 M Tris-Cl (pH 7.5) | 2.5 | 50 mm |
| 0.5 M EDTA | 1 | 10 mM |
| 20% SDS | 2.5 | 1% |
| DDH 2 O | Riempire fino 50 |
Tabella 6. Ricetta per Chip tampone di eluizione. Filtrare su filtro di 0,22 micron. Conservare a temperatura ambiente.
| Reagente | Volume (ml) | [Finale] |
| 1 M Tris-Cl (pH 7.5) | 0.5 | 10 mM |
| DDH 2 O | Riempire fino 50 |
Tabella 7. Ricetta per TE Buffer. Filtrare su filtro di 0,22 micron. Conservare a 4 ° C.
| Volume (ml) | [Finale] | |
| 100 micron ExA2-iX | 75 | 15 micron |
| 100 micron ExA2-33 | 75 | 15 micron |
| 1 M Tris (pH 7,5) | 50 | 100 mM |
| 5 M NaCl | 5 | 50 mm |
| DDH 2 O | 295 | - |
| Volume totale | 500 |
Tabella 8. P7 adattatore ricottura mix.
| Volume (ml) | [Finale] | |
| 100 micron ExA1-58 | 75 | 15 micron |
| 100 micron ExA1-13 | 75 | 15 micron |
| 1 M Tris (pH 7,5) | 50 | 100 mM |
| 5 M NaCl | 5 | 50 mm |
| DDH 2 O | 295 | - |
| Volume totale | 500 |
Tabella 9. P5 adattatore ricottura mix.
| Temp (C) | Tempo |
| 95 | 5 minuti |
| 72 | 5 minuti |
| 65-60 declino rampa | 5 minuti |
| 55-50 declino rampa | 3 min |
| 45-40 declino rampa | 3 min |
| 30 | 3 min |
| 20 | 3 min |
| 10 | 3 min |
| 4 | Per sempre |
Tabella 10. Adattatore di ricottura del programma.
| 1x (pl) | [Finale] | |
| DDH 2 O | 39.8 | |
| 10X Reaction Buffer 2 | 5 | 1x |
| 100 micron ATP | 0.5 | 1 mM |
| 3 dNTP mM | 1.7 | 100 pM |
| 3 U / ml polimerasi T4 | 1 | 3 U |
| 5 U / ml Klenow | 1 | 5 U |
| 10 U / ml T4 polinucleotide chinasi | 1 | 10 U |
| Volume di reazione totale | 50 |
Tabella master mix 11. lucidatura.
| 1x (ml) | [Finale] | |
| DDH 2 O | 42.3 | |
| 10X Reaction Buffer 2 | 5 | 1x |
| 3 mM dATP | 1.7 | 100 pM |
| 5 U / ml Klenow 3'-5 'Exo meno | 1 | 5 U |
| Volume di reazione totale | 50 |
Tabella 12. A-tailing master mix.
| 1x (ml) | [Finale] | |
| DDH 2 O | 41 | |
| 100 mM ATP | 0.5 | 1 mM |
| 10X Reaction Buffer 2 | 5 | 1x |
| 400 U / ml T4 DNA Ligase | 1.5 | 600 U |
| Volume di mix di reazione | 48 | |
| 15 Adattatore Indice mM | 2 | 30 picomoli |
| Volume di reazione totale | 50 |
Tabella 13. P7 Adattatore legatura master mix.
| 1x (ml) | [Finale] | |
| DDH 2 O | 41 | |
| 10x Φ-29 Buffer | 5 | 1x |
| 3 dNTP mM | 2.5 | 150 pM |
| 10 U / ml Φ-29 della polimerasi | 1.5 | 15 U |
| Volume di reazione totale | 50 |
Tabella 14. Φ-29 Nick Repair Master Mix.
| 1x (ml) | [Finale] | |
| DDH 2 O | 43.5 | |
| 100 mM ATP | 0.5 | 1 mM |
| 10X Reaction Buffer 2 | 5 | 1x |
| 10 U / ml T4 polinucleotide chinasi | 1 | 10 U |
| Volume di reazione totale | 50 |
Tabella 15. chinasi maestro mix di reazione.
| 1x (&# 956; l) | [Finale] | |
| DDH 2 O | 43 | |
| 10x Lambda Buffer | 5 | 1x |
| 5 U / ml Lambda esonucleasi | 2 | 10 U |
| Volume di reazione totale | 50 |
Tabella 16. Lambda Exonuclease reazione master mix.
| 1x (ml) | [Finale] | |
| DDH 2 O | 44 | |
| 10X Reaction Buffer 2 | 5 | 1x |
| 30 U / ml RecJ f esonucleasi | 1 | 30 U |
| Totale reazione volume | 50 |
Tabella 17. RecJ f nucleasi maestro mix di reazione.
| 1x (ml) | [Finale] | |
| DDH 2 O | 6.45 | |
| 10x Φ-29 Buffer | 2 | 1x |
| 3 mM dNTP | 1.3 | 200 pM |
| 20 micron P7 Primer | 0.25 | 0.25 micron |
| Volume di mix di reazione | 10 | |
| campione precipitato EtOH | 10 | |
| Volume di reazione totale | 20 |
Tabella 18. P7Primer Extension master mix di reazione.
| Temp (C) | Tempo |
| 95 | 5 minuti |
| 65 | 5 minuti |
| 30 | 2 min |
| 30 | Tenere fino a quando viene aggiunto Φ-29 |
| 30 | 20 min |
| 65 | 10 minuti |
| 4 | Per sempre |
Tabella 19. programma P7 Primer Extension.
| 1x (ml) | [Finale] | |
| DDH 2 O | 5 | |
| 10X Reaction Buffer 2 | 3 | 1x |
| 3 mM dATP | 1 | 0,1 mm |
| 5 U / ml Klenow 3 'a 5' meno Exo | 1 | 5 U |
| Volume di mix di reazione | 10 | |
| campione esteso Primer | 20 | |
| Volume di reazione totale | 30 |
Tabella 20. A-tailing master mix.
| 1x (ml) | [Finale] | |
| DDH 2 O | 11.5 | |
| 10x Ligase Buffer T4 | 5 | 1x |
| 15 micron ExA1-58 /13 adattatore | 2 | 30 picomoli |
| 400 U / ml T4 DNA Ligase | 1.5 | 600 U |
| Volume di mix di reazione | 20 | |
| Una coda di esempio | 30 | |
| Volume di reazione totale | 50 |
Tabella 21. P5 Adattatore legatura master mix.
| 1x (ml) | [Finale] | |
| 5x PCR Buffer | 10 | 1x (2 mM MgCl 2) |
| 10 mm di ogni dNTP | 1 | 200 pM ciascuno |
| 20 micron P1.3 Primer | 1.25 | 0,5 micron |
| 20 micron P2.1 Primer | 1.25 | 0,5 micron |
| 2 U / ml Hot Start polimerasi | 0.5 | 1 U |
| Volume di mix di reazione | 14 | |
| campione eluito Bead | 36 | |
| Volume di reazione totale | 50 |
Tabella 22. PCR.
| Temp (C) | Tempo | cicli |
| 98 | 30 sec | 1 |
| 98 | 10 sec | 15-21 (a seconda del rendimento ChIP) |
| 52 | 30 sec | |
| 72 | 20 sec | |
| 72 | 2 min | 1 |
| 4 | Per sempre | tenere |
Tabella 23. programma PCR.
| Per DNA standard (ml) | Per campione (ml) | |
| 1: 200 diluito tampone / colorante mix | 190 | 198 |
| norme di DNA | 10 | |
| libreria ChIP-exo | 2 | |
| Volume totale | 200 | 200 |
Tabella 24. quantificazione.
Discussione
Vi presentiamo un protocollo di genomica funzionale per determinare la posizione di rilegatura preciso per cromatina proteine interagenti in modo imparziale, genome-wide a risoluzione pressi coppia di basi. La fase più critica per ottenere vicino alla base risoluzione pair mappatura è il trattamento esonucleasi del DNA ChIP arricchiti mentre l'immunoprecipitato rimane sulla resina magnetica. Apparentemente, complessi proteici potrebbero potenzialmente bloccare footprinting in vivo di ogni subunità (ad esempio, i complessi di rimodellamento della cromatina o la particella nucleo nucleosoma). Tuttavia, come riportato in precedenza 10, poiché la formaldeide è un reticolante inefficiente, diventa sempre più improbabile che più subunità di un complesso sarebbero reticolare al DNA e l'altro nella stessa cella nello stesso locus. Così, in footprinting vivo di singole subunità di un complesso proteico, come ad esempio le singole subunità istoni di nucleosomi, è possibile con Chip-Exo.
t "> I vantaggi più importanti di ChIP-exo sono la sua risoluzione coppia vicino alla base e bassa sfondo. L'ultra-alta risoluzione permette intuizioni strutturali e spaziali dettagliate da effettuare su scala genomica che non sono attualmente possibile con qualsiasi altro metodo . d'altra parte, la limitazione principale di ChIP-exo è che si tratta di una metodologia di biologia molecolare tecnicamente difficile da padroneggiare. inoltre, le limitazioni generali del passo ChIP valgono anche per ChIP-eso (ad esempio, la disponibilità di anticorpi commerciale e specificità , epitopo accessibilità e relativamente grande numero di cellule necessarie). insidie comuni includono scarsa estratti sonicati qualità, utilizzando un anticorpo di grado non-chip, e non mantenendo campioni su ghiaccio per quanto possibile. così, condizioni sonicazione devono essere attentamente ottimizzati e ciascun anticorpo convalidato come precedentemente descritto 18 per evitare gli effetti dannosi questi parametri possono avere sul risultato sperimentale.Fino aprofondità di sequenziamento per un target fattore di trascrizione, di solito obiettivo per circa 20 milioni allineato in modo univoco letture. Dal momento che Pol II e modificazioni degli istoni sono distribuiti in modo più ampio, il nostro obiettivo per 30-50.000.000 legge. E 'importante notare che, poiché ChIP-Exo ha sostanzialmente meno sfondo di ChIP-seq, meno letture sono tenuti a raggiungere simili profondità di sequenziamento.
La tecnologia chip-exo viene ora ampiamente adottato nonostante le sfide tecniche, come robuste variazioni sul protocollo originale continuano ad essere pubblicati 17,19,20. In particolare, una variante che può risultare utile per difficile proteine chip è chiamato ChIP-nesso, che utilizza un unico passaggio legatura per aumentare l'efficienza del preparato libreria 20. In sintesi, ChIP-Exo è una metodologia sempre più utilizzata e potente per la mappatura ultra-alta risoluzione delle proteine della cromatina interagire su scala globale. Come l'elenco dei disponibili in commercio formica ChIP-gradeibodies continua a crescere, le future applicazioni della metodologia ChIP-exo saranno diretti a reti di regolazione genica inesplorato di mappatura per comprendere i circuiti molecolari della cellula in altissima risoluzione. Inoltre, ChIP-exo sarà probabilmente ulteriormente affinato e adattato alla presenza in vivo interazioni proteina-RNA al vicino risoluzione coppia di basi.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank the Venters Lab and members of the Molecular Physiology and Biophysics Department for helpful discussions. Special thanks to Frank Pugh for his guidance, mentoring, and many insightful discussions on the nuances of ChIP-exo while I was a post-doctoral fellow in his laboratory.
Materiali
| Name | Company | Catalog Number | Comments |
| 37% formaldehyde, methanol free, 10x10ml ampules | ThermoFisher Scientific | 28908 | Section 3 |
| Complete Protease Inhibitor cocktail (CPI) | Roche Life Science | 11873580001 | Sections 4, 8 |
| Bioruptor sonicator | Diagenode | UCD200 | Section 5 |
| 15 ml polystyrene tubes | BD Falcon | 352095 | Section 5 |
| MagSepharose Protein G Xtra beads | GE Healthcare | 28-9670-66 | Section 6 |
| DynaMag-1.5ml Side Magnetic Rack | Invitrogen | 12321D | Sections 6-17, 22 |
| Mini-Tube Rotator | Fisher Scientific | 05-450-127 | Sections 7, 22 |
| T4 DNA Polymerase | New England BioLabs | M0203 | Section 9 |
| DNA Polymerase I, Klenow | New England BioLabs | M0210 | Section 9 |
| 10x NEBuffer 2 (10x Reaction Buffer 2) | New England BioLabs | B7002 | Sections 9-11, 13, 15, 20 |
| Thermomixer C | Eppendorf | 5382 | Sections 9-16 |
| ATP | Roche Life Science | 010419979001 | Sections 9, 11, 13 |
| dNTPs | New England BioLabs | N0447 | Sections 9, 12, 19, 23 |
| T4 Polynucleotide Kinase | New England BioLabs | M0201 | Sections 9, 13 |
| dATP | New England BioLabs | N0440 | Sections 10, 20 |
| Klenow 3'-5' Exo Minus | New England BioLabs | M0212 | Sections 10, 20 |
| T4 DNA ligase | New England BioLabs | M0202 | Sections 11, 21 |
| Φ-29 DNA Polymerase | New England BioLabs | M0269 | Sections 12, 19 |
| 10x Φ-29 Buffer | New England BioLabs | B0269 | Sections 12, 19 |
| Lambda Exonuclease | New England BioLabs | M0262 | Section 14 |
| 10x Lambda Buffer | New England BioLabs | B0262 | Section 14 |
| RecJf Exonuclease | New England BioLabs | M0264 | Section 15 |
| Proteinase K | Roche Life Science | 03115828001 | Section 16 |
| Glycogen | Roche Life Science | 010901393001 | Section 18 |
| 10x T4 Ligase Buffer | New England BioLabs | B0202 | Section 21 |
| AMPure XP (clean up) beads | Beckman Coulter | A63881 | Section 22 |
| Q5 Hot Start DNA Polymerase | New England BioLabs | M0493 | Section 23 |
| 5x Q5 Buffer (5x PCR Buffer) | New England BioLabs | B9027 | Section 23 |
| Qubit Fluorometer | Invitrogen | Q33216 | Section 25 |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Section 25 |
| Optical Clear Qubit tubes | Invitrogen | Q32856 | Section 26 |
| Qubit dsDNA High Sensitivity Assay kit | Invitrogen | Q32851 | Section 26 |
Riferimenti
- Venters, B. J., Pugh, B. F. How eukaryotic genes are transcribed. Crit Rev Biochem Mol Biol. 44, 117-141 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Chen, J., et al. Single-molecule dynamics of enhanceosome assembly in embryonic stem cells. Cell. 156, 1274-1285 (2014).
- Wales, S., Hashemi, S., Blais, A., McDermott, J. C. Global MEF2 target gene analysis in cardiac and skeletal muscle reveals novel regulation of DUSP6 by p38MAPK-MEF2 signaling. Nucleic acids research. 42, 11349-11362 (2014).
- Cho, S., et al. The architecture of ArgR-DNA complexes at the genome-scale in Escherichia coli. Nucleic Acids Res. 43, 3079-3088 (2015).
- Katainen, R., et al. CTCF/cohesin-binding sites are frequently mutated in cancer. Nat Genet. 47, 818-821 (2015).
- Murphy, M. W., et al. An ancient protein-DNA interaction underlying metazoan sex determination. Nat Struct Mol Biol. 22, 442-451 (2015).
- Zere, T. R., et al. Genomic Targets and Features of BarA-UvrY (-SirA) Signal Transduction Systems. PLoS One. 10, e0145035 (2015).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Rhee, H. S., Bataille, A. R., Zhang, L., Pugh, B. F. Subnucleosomal structures and nucleosome asymmetry across a genome. Cell. , 1377-1388 (2014).
- Pugh, B. F., Venters, B. J. Genomic Organization of Human Transcription Initiation Complexes. PLoS One. 11, e0149339 (2016).
- Albert, I., Wachi, S., Jiang, C., Pugh, B. F. GeneTrack--a genomic data processing and visualization framework. Bioinformatics. 24, 1305-1306 (2008).
- Guo, Y., Mahony, S., Gifford, D. K. High resolution genome wide binding event finding and motif discovery reveals transcription factor spatial binding constraints. PLoS computational biology. 8, e1002638 (2012).
- Bardet, A. F., et al. Identification of transcription factor binding sites from ChIP-seq data at high resolution. Bioinformatics. 29, 2705-2713 (2013).
- Wang, L., et al. MACE: model based analysis of ChIP-exo. Nucleic Acids Res. 42, e156 (2014).
- Bentley, D. R., et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456, 53-59 (2008).
- Rhee, H. S., Pugh, B. F. ChIP-exo method for identifying genomic location of DNA-binding proteins with near-single-nucleotide accuracy. Curr Protoc Mol Biol. Chapter 21, Unit 21.24 (2012).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22, 1813-1831 (2012).
- Serandour, A. A., Brown, G. D., Cohen, J. D., Carroll, J. S. Development of an Illumina-based ChIP-exonuclease method provides insight into FoxA1-DNA binding properties. Genome Biol. 14, R147 (2013).
- He, Q., Johnston, J., Zeitlinger, J. ChIP-nexus enables improved detection of in vivo transcription factor binding footprints. Nat Biotechnol. 33, 395-401 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon