Method Article
ChIP-エキソ方法:近く塩基対の精度でタンパク質 - DNA相互作用を同定します
要約
Here, we present a protocol to achieve near base pair resolution of protein-DNA interactions. This is obtained by exonuclease treatment of DNA fragments selectively enriched by chromatin immunoprecipitation (ChIP-exo) followed by high throughput sequencing.
要約
クロマチン免疫沈降(チップ)エピジェネティクスと特定のタンパク質-DNA相互作用を分離する遺伝子調節の分野で不可欠なツールです。ハイスループットシークエンシング(チップ配列)に結合されたチップは、一般に、クロマチンと相互作用するタンパク質のゲノム位置を決定するために使用されます。しかしチップ-配列は数百塩基対の比較的低いマッピング分解能および高いバックグラウンドシグナルによって妨げられます。 ChIP-エキソ方法は、実質的に解像度とノイズの両方を改善するのChIP-seqのの洗練されたバージョンです。 ChIP-エキソ方法論の重要な違いは、効果的にタンパク質-DNA架橋部位の左右の5 'DNAボーダーフットプリントするためのライブラリ作成ワークフローのラムダエキソヌクレアーゼ消化の組み込みです。 ChIPのエキソライブラリーは、高スループット配列決定に供されます。得られたデータは、機能的な組織O中にユニークな超高解像度の洞察を提供するために活用することができゲノムF。ここで、我々は、哺乳動物系と次世代シーケンシング・バイ・合成プラットフォーム向けに最適化し、合理化しているのChIP-エキソ方法について説明します。
概要
クロマチン免疫沈降(チップ)が選択的に生きている細胞内の指定されたタンパク質と相互作用するDNA断片を濃縮することにより、遺伝子調節のメカニズムを研究するための強力な方法です。技術は、ハイスループットシークエンシング(チップ配列番号1)をオリゴヌクレオチドマイクロアレイ(チップオンチップ)のハイブリダイゼーションを単一遺伝子座(標準のChIP-定量PCR)の検出から、改善、チップ富化DNAフラグメントの検出方法が発展してきました。 ChIP-seqが幅広い応用、クロマチン異質と非特異的なDNA相互作用を見たが、偽陽性と不正確なマッピングにつながるデータ品質を妨げてきました。これらの制限を回避するために、博士はフランク・ピューは、チップエキソ方法2を開発しました。 ChIPのエキソの顕著な特徴は、それが効果的に位置転写因子結合をフットプリンティング、エキソヌクレアーゼ5 'から3'を組み込むことです。その結果、チップエキソ方法は、より高い解像度、detectioの大きなダイナミックレンジを達成しますnおよびより低いバックグラウンドノイズ。
ChIP-エキソは、より技術的に困難なのChIP-seqのよりマスターにではあるが研究は、多様な生物学的システム3-8を使用して、ユニークな超高解像度の洞察を得ることを目指しとして、それが広く採用されています。実際に、チップエキソ正常細菌、酵母、マウス、ラット、およびヒトの細胞系に適用されています。原理の証明としてチップエキソは当初、酵母転写因子2の一握りのための正確な結合モチーフを同定しました。技術はまた、転写開始前複合体の組織化を研究するために、種々のヒストン9,10のsubnucleosomal構造を解読するために、酵母中で使用しました。最近では、我々は人間のプロモーターで隣接TFIIBとポルII結合事象を解決するためのChIP-エキソを活用し、かつ広範な発散転写は11と錯体を形成する別個の開始から生じることを示しました。
ここで紹介するワークフローを最適化およびsされます哺乳類のChIP-エキソ( 図1)のためにtreamlined。まず、生きている一次または組織培養細胞は、共有架橋を介してインビボでタンパク質-DNA相互作用を維持するためにホルムアルデヒドで処理されます。細胞を溶解し、クロマチンは〜100に剪断された - 500塩基対のサイズの断片。チップはその後、選択的に目的のタンパク質に架橋DNA断片のために豊かにします。この時点でのChIP-seqのライブラリーは、典型的には、本質的に、数百塩基対の平均断片サイズに検出分解能を制限する、調製されます。しかし、のChIP-エキソはラムダエキソヌクレアーゼでタンパク質-DNA架橋部位の左右の5 'DNA境界をトリミングすることによって、この制限を克服します。以下に詳述するようにシークエンシングライブラリーは、その後、エキソヌクレアーゼ消化DNAから構成されています。得られたネストされた5 '境界は、タンパク質-DNA相互作用( 図1、ステップ14)とのインビボでのフットプリントを表し、ハイスループットシークエンシングによって検出されます。 AltキーChIP-エキソ方法論は、チップ-seqのよりも複雑であるハフ、ほとんどの工程間の遷移はサンプルのロスや実験的なばらつきを最小限に抑えるシンプルなビーズの洗浄が必要です。重要なことに、チップエキソは、チップのseq、またのChIP-エキソと成功するはずのChIP-seqのと成功している任意のサンプルの洗練されたバージョンがあるため。
ChIP-seqのとは根本的に別個のデータ構造でのChIP-エキソ結果とタンパク質-DNA相互作用のin vivoフットプリント。共通のChIP-seqの発信者が最も正確なピークのコールを取得するために、ChIPのエキソデータに適用することができるが、我々は、具体的に念頭に置いてのChIP-エキソデータのユニークな構造で設計されたバイオインフォマティクスツールをお勧めします。これらはGenetrack、GEM、MACE、Peakzilla、およびExoProfiler 12-15が含まれます。
プロトコル
注:ダブル蒸留H 2 Oまたは分子グレード同等物は、すべてのバッファとの反応ミックスをお勧めします。
0日目:材料の調製および細胞収穫
1.バッファの準備
- 3( 表1 - - 3)溶解バッファ1を準備し、使用直前に、各バッファの50ミリリットルに100μlの完全プロテアーゼ阻害剤の株式(CPI)を追加します。分子グレードのH 2 Oを1ミリリットル中に1錠を溶解させることによって、CPIの株式を準備します
- ( - 7表4)内蔵バッファを準備します。ブロッキングとRIPAバッファー50mlに100μlのCPIの株式を追加します。トリスおよびChIPの溶出バッファにCPIを追加しないでください。
アダプターオリゴヌクレオチドの2アニーリング
注:特定のオリゴヌクレオチド配列は、追加情報セクションに見出すことができます。
- アダプタアニーリングミックス( - 9表8)を準備します。ミックスする渦と簡単に内容物を収集するために、チューブをスピン。 0.5ミリリットルチューブに小分けし、各ミックスを100μl。
- サーマルサイクラーにプログラム( 表10)を実行することにより、オリゴヌクレオチドをハイブリダイズします。
ストアは、-80℃でオリゴヌクレオチドをアニール。
ホルムアルデヒドによるインビボクロマチン架橋3.
注:典型的なChIP実験のために、出発物質は、約50万個の細胞が含まれている必要があります。
- 緩衝化生理食塩水(PBS)をホスフェートに新鮮な37%メタノールフリーホルムアルデヒドストック溶液を加える(v / v)で、完全に混和1%の最終濃度まで細胞培養物を洗浄し、室温で10分間放置。
注:たとえば、私たちは、典型的には、室温PBS 50ml中の細胞に37%メタノールフリーホルムアルデヒドの1.4ミリリットルを追加します。メディア内のタンパク質は、ホルムアルデヒドの一部を急冷するので、また、メディアでのホルムアルデヒド架橋を避けます。 - 2.5 Mグリシンを添加することにより架橋反応をクエンチ125 mMのの最終濃度の電子。完全に混合。
- 氷上で50ミリリットルチューブに細胞を移します。 4℃で千×gで5分間、細胞をスピン。上清を除去。
- ピペッティングにより細胞ペレットを再懸濁に1ミリリットルの氷冷1×PBSを追加します。 1.5mlチューブに移します。
- 4℃で2000×gで3分間、細胞をスピン。上清を吸引。
- 直ちに液体窒素で1.5ミリリットルチューブに細胞ペレットを凍結点滅。 -80℃で保管してください。架橋された細胞を、-80℃で無期限に安定です。
1日目:細胞の溶解、超音波処理、チップ
4.細胞溶解
注:すべての細胞溶解セクションの間、サンプルは架橋反転を最小にするために氷上または4℃で保持しなければなりません。
- 簡単に言えば架橋細胞ペレットを解凍。徹底的に0.5ミリリットルの溶解緩衝液1中の各ペレットを再懸濁し、15ミリリットルポリスチレンチューブに4.5ミリリットルの溶解緩衝液1と組み合わせます。
- 4℃で10分間ロックチューブロッキングプラットフォーム上のため。 4℃で2000×gで4分間スピン。上清を除去。
- 徹底的に0.5ミリリットルの溶解緩衝液2に各ペレットを再懸濁し、4.5ミリリットル溶解バッファー2回再懸濁を追加します。
- ロッキングプラットフォーム上で4℃で5分間ロック。 4℃で2000×gで5分間スピン。上清を除去し、ペーパータオル上の過剰をタップします。
- 徹底的に0.5ミリリットルの溶解緩衝液3中の各ペレットを再懸濁し、1ミリリットル溶解バッファー3回再懸濁を追加します。氷の上に核溶解物を保管してください、すぐに超音波処理に進みます。
原子力溶解物の5.超音波処理
注:すべての超音波処理のセクションの間に、サンプルが架橋逆転を最小限に抑えるために氷上に維持しなければなりません。以下のプロトコルに関連して使用される超音波処理器の特定のモデルは、材料の表に見ることができます。特定の超音波処理装置の使用状況や他の楽器のためのガイドラインの詳細は、追加情報セクションに見出すことができます。
- PL核抽出物を含む15ミリリットルポリスチレンチューブでエース共鳴アダプタ(金属製のバーがチューブの壁に触れないようにしてください)。
- ミディアムパワーで/ 30秒OFF ON 30秒で2×15分間のセッションのための氷冷水浴中で超音波処理する核溶解物。一度に2 15ミリリットルのチューブを超音波処理します。これらの設定は、細胞株および初代細胞型の広い範囲で動作するが、クロマチン断片が不完全である場合、さらなる最適化が必要とされ得ます。
- 1.5ミリリットルチューブに溶解物から超音波処理の結果、転送2×10μlのサンプルを確認するには。
- 架橋を逆にするには、10μlのTE-RNase Aを、37℃で30分間プロテイナーゼKインキュベートの0.2μlの最初の10μlのアリコートを兼ね備えています。 4μlの6倍キシレンDNA色素を追加します。
- 架橋を維持するには、2μlの6×キシレンDNA染料と第二の10μlのアリコートを兼ね備えています。
- 私ははしごにブロモフェノール色素まで45分 - 約30、140 Vで1.5%アガロースゲル上で両方のサンプルを実行します。ゲルダウン道の3/4です。
- 超音波処理は、(ほとんどのDNA断片が100に剪断された - 500 bp)の成功した場合、転送は10%トリトンX-100の150μLを含む2ミリリットルチューブに溶解物を超音波処理しました。ミックスする渦。
- 不溶性クロマチンと破片をペレット化するために、スピンは4℃20,000×gで10分間の核溶解液を超音波処理しました。
- 新しい2 mlチューブに上清を移します。無期限に-80℃でのChIPまたはストアサンプルにすぐに進んでください。超音波処理した抽出物を凍結させ、複数回解凍するべきではありません。
6.カップリング抗体ビーズへ
注:彼らは粉々にし、バックグラウンドシグナルが増加しますので、絶対に渦または凍結/磁気ビーズを解凍します。
- 徹底的にY = 1.1×2.5μlのX(ChIPサンプルの数)を1.5 mlチューブへの株式、ビーズスラリーμlのアリコートYを混合した後。 2.5μlのビーズスラリーのための能力を結合すると、13μgのIgGに対する次第です。プールされたビーズをブロックする1ミリリットルで3回洗浄しバッファ。
- 洗浄の後、ビーズのより一貫した等分については、ブロッキング緩衝液で10倍、元のスラリーの体積(25μL/チップ)での再懸濁ビーズ。アリコート1.5ミリリットルチューブにチップサンプルあたり25μlの。
- 再懸濁ビーズのアリコートを対応する抗体を10μg - 5を追加します。ブロッキング緩衝液で250μlの最終容量を起動します。 4℃で一晩(16時間、代わりに)回転プラットフォーム上で4時間サンプルをインキュベートします。
- 上清を吸引。未結合または過剰な抗体を除去するために、ブロッキング緩衝液1ミリリットルで一回ビーズを洗浄します。
- ビーズ複合体:最後の洗浄を吸引した後、すぐに抗体に超音波処理抽出物(〜1.6ミリリットル)の5000万細胞同等物を追加します。超音波処理抽出物が準備されていない場合、彼らは準備が整うまで、ブロッキング緩衝液100μl中にビーズを再懸濁します。ビーズを乾燥させたことがないことが重要です。
7.クロマチン免疫沈降(チップ)
- 各チップのサンプルについて、超音波処理の1.5ミリリットルを組み合わせます抗体とD抽出物:1.5ミリリットルまたは2ミリリットルチューブ内のビーズ複合体。
- 4℃の9の速度設定で一晩(約16時間)ミニチューブローテーター上でチューブをインキュベートします。サンプルが漏れていないされ、適切に混合されていることを確認するために数分後に確認してください。
2日目:ChIPの洗浄およびオン樹脂の酵素反応
8. ChIPのウォッシュ
注:簡単に各洗浄の間にチューブをスピン、交差汚染を最小限にするために。最初の吸引のために、各サンプル間のヒントを変更します。その後の洗浄の間に、同チップは、ビーズを触れていない限り、使用することができます。適切なチップ洗浄の指示のための追加情報のセクションを参照してください。
- 非常に簡単にキャップ内の液体を収集し、1分間磁気ラック上に配置するために微量(500×gで2〜3秒)を使用してチューブをスピン。まだ磁気ラック上にある間に、吸引エキス。
- 各チューブに0.75ミリリットルのRIPAバッファーを追加します。磁気ラックからチューブを外しますそして、混合するために数回転倒。磁気ラックを吸引上清にチューブを交換してください。 7Xを繰り返します。
- 各チューブに0.75ミリリットルの10mMトリス塩酸(pH7.5)を追加します。磁気ラックからチューブを外し、混合するために数回転倒。磁気ラックを吸引上清にチューブを交換してください。 2回繰り返します。
- 最後の洗浄を吸引した後、研磨に進みます。
注意:1分と上清を吸引するための磁気ラックに対する各インキュベーションセクション9.3、10.3、11.3、12.3、13.3、14.3での反応、および15.3、スピンチューブ簡単と場所の後。 0.75ミリリットルのトリス塩酸(pH7.5)で0.75ミリリットルRIPA緩衝液および2倍で洗浄2回。
9.研磨反応
- マスターミックスの計算( 表11)を研磨記入。氷上で1.5mlチューブ内のミックスを研磨してください。ミックスするピペット。
- 最後のChIP洗浄液を吸引した直後は、しばらくまだ磁気ラック上の各サンプル樹脂に研磨ミックスの50μlを添加します。 3×gで30分間サンプルをインキュベートサーモミキサー中で30℃にて。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、テーリングに進みます。
10. A-テーリング反応
- A-テーリングマスターミックスの計算( 表12)を記入します。氷上の1.5mlチューブ内テーリングミックスを作ります。ミックスするピペット。
- まだ磁気ラック上にしながら、すぐにセクションを研磨した後、各サンプル樹脂に-テーリングミックス50μlのを追加します。サーモミキサー中で37℃で3×gで30分間サンプルをインキュベートします。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、P7アダプターライゲーションに進みます。
11. P7アダプターライゲーション反応
- ライゲーションマスターミックスの計算( 表13)を記入します。氷上で1.5mlチューブにライゲーションミックスを作ります。ミックスするピペット。
- すぐにA-テーリングセクションの後、48μlのP7のライゲーションマスターミックスと異なるアダプタインデックストンの2μlを添加しますまだ磁気ラック上にある間に、各サンプルの樹脂がo。サーモミキサーで25℃、3×gで2時間サンプルをインキュベートします。
注:各試料の異なるインデックスを使用することは、より多くのサンプルが単一のフローセルに配列決定することが可能になります。 - 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、Φ-29ニックの修復に進みます。
12.ピピ-29ニックの修復反応
- Φ-29マスターミックスの計算( 表14)を記入します。氷上の1.5mlチューブ内のΦ-29ミックスを作ります。ミックスするピペット。
- すぐにP7ライゲーションセクションの後、しばらくまだ磁気ラック上の各サンプル樹脂にΦ-29ミックス50μlのを追加します。サーモミキサー中で30℃にて3×gで20分間サンプルをインキュベートします。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、キナーゼ反応に進みます。
13.キナーゼ反応
- (キナーゼマスターミックスの計算に必要事項を記入してください表15)。キナーゼは、氷上で1.5ミリリットルチューブに混ぜてください。ミックスするピペット。
- すぐにΦ-29セクションの後、まだ磁気ラック上にしながら、各サンプルを樹脂に混ぜキナーゼの50μlを添加します。サーモミキサー中で37℃で3×gで20分間サンプルをインキュベートします。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、ラムダエキソヌクレアーゼ反応に進みます。
14.ラムダエキソヌクレアーゼ反応
- ラムダエキソヌクレアーゼマスターミックスの計算( 表16)を記入します。ラムダエキソヌクレアーゼは、氷上で1.5ミリリットルチューブに混ぜてください。ミックスするピペット。
- まだ磁気ラック上にしながら、直ちにキナーゼセクションの後、各サンプル樹脂にラムダエキソヌクレアーゼミックス50μlのを追加します。サーモミキサー中で37℃で3×gで30分間サンプルをインキュベートします。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、RecJ fが反応に進みます。
15. RecJ Fヌクレアーゼ反応
- マスターミックスの計算( 表17)f が RecJに必要事項を記入してください。氷上で1.5mlチューブでRecJ fが混在してください。ミックスするピペット。
- 磁気ラック上の静止しながら、すぐにラムダエキソヌクレアーゼセクションの後、各サンプル樹脂にRecJ fがミックス50μlのを追加します。サーモミキサー中で37℃で3×gで30分間サンプルをインキュベートします。
- 8.4節の後にノートに記載されているように最後の洗浄を吸引した後、ChIPの溶出に進みます。
16.溶出し、架橋逆転
- 1.5ミリリットルチューブ内のサンプル数のx 1.1×(200μlのChIPの溶出緩衝液+ 1μlの20 mg / mlでプロテイナーゼK):マスターミックスを調製します。各サンプル樹脂に200μlのChIPの溶出バッファー+プロテイナーゼKマスターミックスを追加します。
- 結露を防止するための加熱蓋付きサーモで65℃で一晩(約16時間)3×gでサンプルをインキュベートします。
3日目:DNA抽出イオンとアダプターライゲーション
17.溶出し、クロスリンク反転(継続)
- 65℃で一晩インキュベートした後、簡単に凝縮液を収集するために、サンプルをスピン。 1分間磁気ラック上のサンプルを配置します。
- 200μlのTE緩衝液(pH7.5)を含む新しい1.5ミリリットルチューブに200μlの上清を移します。
18.フェノールクロロホルムイソアミルアルコール(PCIAA)の抽出
- 各サンプルに400μlのフェノールクロロホルムイソアミルアルコールを追加します。渦は20秒間混合します。
- 室温(RT)で2万×gで10分間遠心します。慎重に新しいチューブに上層の水層の325μLを移します。
注:新しいチューブに有機(下層)のいずれかを転送しないように注意してください。 - 各試料に3 MのNaOAc(pHは5.5)と1μlの20 mg / mlとグリコーゲンの1/10容量を追加します。複数のサンプルについては、マスターミックスを準備します。
- 各サンプルに氷冷100%エタノールの3ボリュームを追加します。渦混合します。 -80℃で15分間インキュベートします。
- 4℃20,000×gで15分間遠心分離します。 慎重にペレットを乱さないように確認しながら、上清をデカントします。
- ゆっくりしたばかりのペレットを乱さないように確認しながら、氷冷70%エタノールを作った500μlを添加します。 4℃20,000×gで5分間遠心分離します。上清を注意深くデカントします。
- 45℃での高速真空中で約20分(または乾燥するまで)のための乾燥ペレット。
注:これは、プロトコルでの一時停止点です。乾燥したDNAペレットを-20℃で保存することができます。 - 繰り返しDNAが乾燥したペレットを見ることができなくても、ペレット化領域の上に10μlののddH 2 Oをピペットで再懸濁ペレット。
- サンプル数に応じて、新鮮な0.3ミリリットルのPCRチューブまたは8ラックに10μlの各サンプルを転送します。
19. P7プライマー伸長反応
- 記入P7プライマー伸長マスターミックスの計算( 表18)。 Extensionは氷上で1.5mlチューブに混ぜてください。ミックスするピペット。
- 各10μlのサンプルに拡張ミックス10μlの(マイナスΦ-29ポリメラーゼ)を追加します。ミックスするピペット。
- 30 Cは、ステップを「ホールド」するまで、テンプレートにアニーリングプライマーにプログラム( 表19)を使用してサーモ内のサンプルを実行します。
- プログラムのステップを「ホールド」30℃の間に1μlのΦ-29ポリメラーゼを追加します。ミックスするピペット。プログラム( 表19)の残りを再開します。
20. A-テーリング反応
- A-テーリングマスターミックスの計算( 表20)を記入します。氷上の1.5mlチューブ内テーリングミックスを作ります。ミックスするピペット。
- 各サンプルに、テーリングミックス10μlのを追加します。ミックスするピペット。
- サーモサイクラー中で、37℃で30分間、試料をインキュベートし、その後75℃で20分間熱不活性化。
21. P5アダプターライゲーション反応
- フィルリットルアウトP5アダプターライゲーションマスターミックスの計算( 表21)。氷上で1.5mlチューブにライゲーションミックスを作ります。ミックスするピペット。
- 各サンプルにP5ライゲーションミックスの20μlのを追加します。ミックスするピペット。
- サーモでは、25℃で2時間サンプルをインキュベートします。
22.ビーズのクリーンアップ
注:クリーンアップビーズ上のメーカーの詳細のための材料のセクションを参照してください。
- 新しい1.5 mlチューブに各50μlのサンプルを転送します。
- RTで15分間、15の速度設定でミニチューブローテーター上のビーズをクリーンアップ懸濁します。ビーズは、ピペッティングの前に和解させてはいけません。
- サンプリングするために60μlのクリーンアップビーズを追加します(1.2:比率をサンプリングするビーズをクリーンアップ1がより短い200塩基対であるDNAを除去することが重要です)。ピペットは、20秒間混合します。
- 室温で3分間、15の速度設定でミニチューブローテーター上のビーズをクリーンアップ懸濁します。簡単に言えば、チューブをスピン。
- 1分間磁気ラック上にチューブを入れます。ピペットでオフし、上清を捨てます。
注:それはクリーンアップビーズを吸い上げるますので、このセクションで真空吸引を使用しないでください。 - 彼らは、磁気ラック上にある間、各サンプルに新たに作製したRTの70%エタノール400μlを添加します。混ぜないで。上清を吸引。 2回繰り返します。
- RTで10分間クリーンアップビーズを乾燥させます。ビーズの色は茶色光に暗くなり、ビーズが乾燥しているときに非常に小さな亀裂が樹脂ペレットに表示されます。
- DNAを溶出するために、40μlの10mMのトリス(pHは7.5)を追加します。徹底的に20秒間混合するピペット。
- 1分間磁気ラック上のサンプルを配置します。ゆっくりピペットおよび0.3ミリリットルのPCRチューブに36μlの溶出液を直接転送します。
- -20℃でPCR反応またはストア溶出液に直接進みます。
4日目:PCRおよびゲル分析
23. PCR
- PCRマスターミックスの計算( 表22)を記入します。 PCRミックスを作成します。ピペットは非常に穏やかに回避するために混合します泡。
- 各36μlのDNAサンプルにPCRミックスの14μLを加えます。ミックスするピペット。サーマルサイクラーに入れて準備ができるまで氷上でサンプルを保管してください。
- PCRの陽性対照のために、先に調製したライブラリーが含まれます。 PCR陰性コントロールのために、水を含みます。 PCRプログラム( 表23)を使用してサーモ内のサンプルを実行します。
24.ゲルの調製、DNA切除、および可視化
- 完全に洗浄し、脱イオン水で適切なサイズのゲルボックスをすすぎます。 60μLを保持することができ、厚さ、幅の広いウェルコームで(0.5ミリグラム/ mlのエチジウムブロマイドを含む)を1.5%アガロースゲルを準備します。
注:エチジウムブロマイド(EtBrを)は、発がん性物質であり、注意して取り扱わなければなりません。 - 凝縮を収集するために簡単にPCRサンプルチューブをスピンダウン。
- 合わせたサンプルに6倍キシレンDNA色素の1/5容量を追加します。それがサンプルDNAと同じ場所に移行するため、DNA色素でブロモフェノールブルーを使用しないでください。
- 全サンプルのint型のロードO各ウェル、好ましくはサンプル間に空のウェルを有します。
注:同じインデックスを持つライブラリは、同じゲルで実行してはいけません。 - 負荷7μlのサンプルのいずれかの側の100bpラダー。ラダーでのブロモフェノール染料があるまで¾方法ゲルの下約30-45分間、140Vでゲルを実行します。
- 画像は、低UV設定でトランスイルミネーター上でゲルを視覚化。 500bpの - 200のDNA断片を含むアガロースのセクションを切除。 125塩基対で動作するアダプター二量体のバンドを切り出し避けるために非常に注意してください。
- 15ミリリットルチューブにそれぞれ切り出し、ゲルスライスを配置します。各ゲルスライスの正味重量を記録します。チューブに直接この重みを書きます。
- 画像は、保存、および選択された正しいサイズの範囲を確認するために、切り出したゲルに注釈を付けます。
25.ゲル精製
- 以下の変更を加えて、製造者の指示に従って切除したゲルスライスからDNAを精製します。
- 揺動によりゲルスライスを溶解RTでプラットフォームをロッキングオン。これは、溶解するために約20分かかります。
- 高度に精製されたDNAを得るために、カラムを通過した0.5ミリリットルのBuffer QG後の溶解ゲルでカラムを洗浄します。
- 5分 - のBuffer PEの洗浄後、カラムは2、室温で放置します。 RTで13,000×gで1分間スピン。
- 新しい1.5 mlチューブにPCR反応マスターミックス、40μlのEBバッファーで溶出サンプルのための部屋を許可するには。
26. DNA定量
- 製造元の指示( 表24)に記載のサンプルを準備します。光管を有する指定された楽器でサンプルを測定します。
- ChIP-エキソライブラリが定量化されると、このプロトコルでDNAアダプターと互換性のあるシークエンシング・バイ・合成化学16を使用してプラットフォーム上で配列決定のために提出します。
注:典型的には、試料の2μlを定量化するのに十分であるが、試料濃度が低い場合よりは、必要であり得ます。典型的なDNA収量50と200 ngの間光年の範囲。 - 定量化した後、-20℃で保存サンプル。
結果
次の図は、ここで紹介するのChIP-エキソプロトコルから代表的な結果を示しています。いくつかの酵素工程と、伝統的なのChIP-seqの方法論とは対照的に、ChIPのエキソは11順次依存酵素反応( 図1)が必要です。このように、注意が各反応成分が各反応マスターミックスに添加されることを確実にするために各ステップで注意しなければなりません。私たちは、自動的に反応マスターミックスの計算を実行するために、反応テーブルに基づいて、定型のスプレッドシートを生成する結果の表を印刷し、それがマスターミックスに追加された後、次に各項目をオフにチェックをお勧めします。
私たちは高品質の配列決定の結果( 図2)を確保するためのプロトコル全体の品質管理対策の数を採用しています。各チップの反応は、3つの基本的な構成要素が含まれています:1)intereの細胞からクロマチンの抽出物を超音波処理しましたST目的のタンパク質に対して向けられ、2)抗体、および3)タンパク質G(又はプロテイン)樹脂を沈殿させ、免疫複合体を固定化します。超音波処理条件は各細胞型および超音波処理機器用に最適化されなければならないので、高品質の超音波処理クロマチン抽出物( 図2A)を得ることは非常に困難な場合があります。 500bpの( 図2A、「+」レーン) -最高品質のライブラリは100間のDNA断片が得られる超音波処理結果で始まるので、このステップを最適化するために時間を費やす価値があります。ホルムアルデヒド架橋は不安定であり、ホルムアルデヒド自体が制限された貯蔵寿命を持って、我々は、ゲルシフトアッセイ( 図2A、 -レーン"")を使用しているためのスーパーシフト明らかなように、超音波処理抽出物は、無傷のタンパク質-DNA架橋が含まれていることを確認するために、 DNA断片。バックグラウンド信号を評価するために、私たちは日常のChIP反応から抗体を省略し、「モック」のChIPを行います。高品質のChIP-EXポルIIに対して向けられ、この場合の - ( "")は、特定のChIP抗体に比べて、Oライブラリの準備は「モック」のチップにはほとんど、もしあれば、バックグラウンド信号を持つことになります。 図2Bに見られるように、伝統的なのChIP-seqのライブラリは、チップエキソよりも実質的にバックグラウンド信号を有しています。最後に、シーケンシング、のChIP-エキソライブラリの質の前には、バイオアナライザー( - D図2C)で評価されます。分析は、正確にライブラリーサイズ分布を測定し、125塩基対で動作(矢印で示す)汚染アダプターダイマーを検出します。アダプターダイマーが存在する場合、それらは、配列決定の帯域幅を減少させます。したがって、我々は効率的にDNAが200塩基対未満を断片化除去する追加のビーズのクリーンアップをお勧めします。
ChIP-エキソは、それが空間的に発散解決できる唯一の方法であるため、強力な機能ゲノム技術で開始、一時停止、およびRNAポリメラーゼII Oを長くしますゲノムワイドスケール( 図3)11ナ。これらの隣接する結合事象離れ塩基対の数十あるために、チップ・配列は数百塩基対の分解能でこの結合事象を区別することができません。

図1: チップエキソ概略図。 ChIPの後、P7アダプタは、超音波処理の境界線に連結されています。ラムダエキソヌクレアーゼは、それによってタンパク質-DNA相互作用をフットプリンティング、架橋点に 'から3'のDNA 5をトリミング。溶出および架橋反転した後、プライマー伸長は、二重鎖DNAを合成します。最後に、P5アダプターのライゲーションは、左右のエキソヌクレアーゼ境界線をマークし、得られたライブラリーは、ハイスループット配列決定に供されます。参照ゲノムに配列タグの5 '末端をマッピングすることは、このようエキソヌクレアーゼバリアおよびタンパク質 - DNAの正確な部位を画定します架橋。図は、李承晩とピュー17から変更されました。 この図の拡大版をご覧になるにはこちらをクリックしてください。
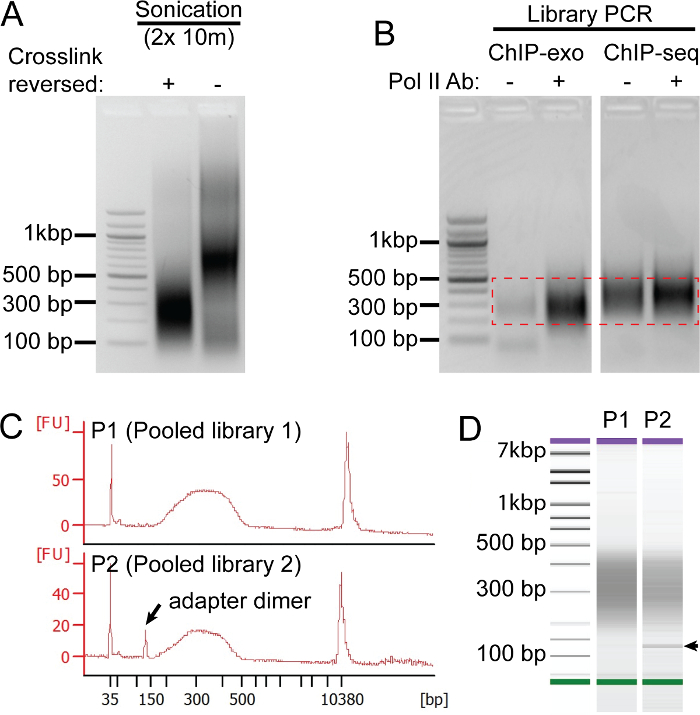
図2: チップエキソ品質を制御します。パネルA〜Cは無関係な実験からの代表的な結果を示しています。架橋が反転( - )(A)(ヒトHCC1806乳癌細胞株からの)超音波処理し、クロマチン抽出物の品質は、(+)とし、なし抽出物のアガロースゲル電気泳動によって評価されます。無傷の架橋は、タンパク質-DNA複合体が遅く移行する原因となります。このように、逆に架橋することなく実行されている抽出物は、( - )成功のChIPのために重要である評価されるホルムアルデヒド架橋の品質を可能にします。モックIPの(B)の比較( - )ANDポルII(+)のChIP-エキソおよびPCR増幅の21サイクル後のChIP-seqのライブラリの準備。 PCR後、DNA断片200〜500塩基対を切り出し(赤いハッシュされたボックスで示す)ゲル抽出キットを用いて精製します。 (CD)は 、パネルCでは、一番上のトレースは、プールされたライブラリー(P1)から理想的なトレースを表し、下のトレースは、アダプターダイマーが含まれているプールされたライブラリ(P2)を示しています。パネルDは、パネルC(矢印アダプターダイマーバンドを示す)からP1-2ライブラリトレースに相当するDNA濃度のプロットを示します。 この図の拡大版をご覧になるにはこちらをクリックしてください。
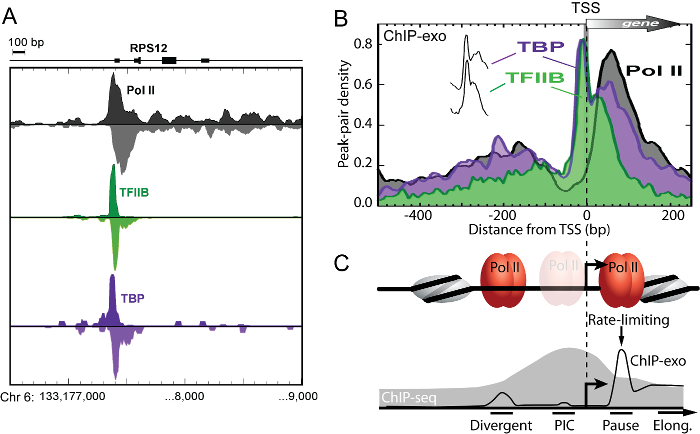
図3: チップエキソは、空間的に別個の双方向転写開始複合体を解決します。 (A)鎖-separatの平滑化分布EDのChIP-エキソタグ5 'ポルIIのために終了し、K562細胞増殖中のヒトRPS12遺伝子でTFIIB、及びTBP。 (B)は 、最も近いのRefSeq TSS周りのChIPエキソパターンを平均しました。すべてTFIIB-占有両端のピーク対タグがTSSに関連し、その後、平均ピーク対濃度値10BP間隔を非重複でビン化、TSS遺伝子バイ遺伝子に整列させた(N = 6511)の遺伝子は、以下のようにプロットしました合計のパーセント。 TBPとTFIIBの「スパイク」(縦に挿入図にオフセット)聞き取りにくいです。 (C)モデルは、チップエキソ(黒のトレース)によって解決異なる転写開始複合体を示し、パネルBのデータに基づきます。ポルIIは発散転写開始(「発散」)と「一時停止」のサイトのサイトと一致した二つの別々の解決可能な場所を占めていました。ポルII複合体のこの明確な空間的分離は、発散転写物が明確な開始複合体から生じることを示しています。大多数のPol II架橋ABO転写を開始した後に一時停止することが期待される「一時停止」サイト、でTSSの下流UT 50bpの。開始前複合体(「PIC」)の形態は、平均的にそれは可能性が一時停止したサイトでよりそこに少ない時間を費やしていることを示すTSSの上流60 bpの - ポルIIは、ほとんどの20を枯渇しました。ポルIIポーズリリースがあるという観察と一致して、TSSの下流50bpの - これは、ほとんどの(必ずしも全てではない)の場合にはポルIIが募集されたら、それは急速にプロモーターをクリアし、約30一時停止状態を想定していることを示唆しています転写における律速段階。その解像度は数百塩基対に限定されているので、これらの隣接開始複合体は、(灰色の塗りつぶしトレースで図示)のChIP-seqのことで解決不可能です。図は、ピューとVenters氏11から変更されました。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| 試薬 | 容量(ml) | [最後の] |
| 1 M HEPES-KOH(pH7.5)を | 50 | 50 mMの |
| 5 M NaClを | 28 | 140 mMの |
| 0.5 M EDTA | 2 | 1 mMの |
| 100%グリセロール | 100 | 10% |
| 10%NP40 | 50 | 0.50% |
| 10%トリトンX100 | 25 | 0.25% |
| ddH 2 O | 1 Lに記入 |
0.22μmのフィルターを用いて、 溶解緩衝液1.フィルタの表1のレシピ 。 4℃で50 mlチューブに保管してください。使用直前に緩衝液50mlに100μlのCPIの株式を追加します。
| 試薬 | 容量(ml) | [最後の] |
| 1 MのTris-HCl(pHは8) | 10 | 10 mMの |
| 5 M NaClを | 40 | 200 mMの |
| 0.5 M EDTA | 2 | 1 mMの |
| 0.5 M EGTA | 1 | 0.5 mMの |
| ddH2 O | 1 Lに記入 |
0.22μmのフィルターを用いて、 溶解緩衝2.フィルター表2.レシピ 。 4℃で50 mlチューブに保管してください。使用直前に緩衝液50mlに100μlのCPIの株式を追加します。
| 試薬 | 容量(ml) | [最後の] |
| 1 MのTris-HCl(pHは8) | 10 | 10 mMの |
| 5 M NaClを | 20 | 100 mMの |
| 0.5 M EDTA | 2 | 1 mMの |
| 0.5 M EGTA | 1 | 0.5 mMの |
| 10%デオキシコール酸 | 10 | 0.10% |
| Nラウロイルサルコシン | 5グラム | 0.5%(w / v)の |
| ddH 2 O | 1 Lに記入 |
0.22μmのフィルターを用いて、 溶解緩衝3.フィルタの表3レシピ 。 4℃で50 mlチューブに保管してください。使用直前に緩衝液50mlに100μlのCPIの株式を追加します。
| 試薬 | 容量(ml) | [最後の] |
| 10×PBS | 50 | 1倍 |
| ウシ血清アルブミン | 2.5グラム | 0.50% |
| ddH 2 O | 500に塗りつぶし |
ブロッキング表4.レシピバッファ。 0.22μmのフィルターを使用してフィルタリングします。 4℃で50 mlチューブに保管してください。使用直前に緩衝液50mlに100μlのCPIの株式を追加します。
| 試薬 | 容量(ml) | [最後の] |
| 1 M HEPES(pH7.5)を | 25 | 50 mMの |
| 0.5 M EDTA(pHは8) | 1 | 1 mMの |
| 10%デオキシコール酸ナトリウム | 35 | 0.70パーセント |
| 10%NP40 | 50 | 1% |
| 1 MのLiCl | 250 | 500 mMの |
| ddH 2 O | 500に塗りつぶし |
RIPAバッファー表5.レシピ。 0.22μmのフィルターを使用してフィルタリングします。 4℃で50 mlチューブに保管してください。使用直前に緩衝液50mlに100μlのCPIの株式を追加します。
| 試薬 | 容量(ml) | [最後の] |
| 1 Mトリス-Cl(pH7.5)を | 2.5 | 50 mMの |
| 0.5 M EDTA | 1 | 10 mMの |
| 20%SDS | 2.5 | 1% |
| ddH 2 O | 50に塗りつぶし |
ChIPの溶出バッファー表6.レシピ。 0.22μmのフィルターを使用してフィルタリングします。室温で保管してください。
| 試薬 | 容量(ml) | [最後の] |
| 1 Mトリス-Cl(pH7.5)を | 0.5 | 10 mMの |
| ddH 2 O | 50に塗りつぶし |
TEバッファー表7.レシピ。 0.22μmのフィルターを使用してフィルタリングします。 4℃で保管してください。
| 容量(μL) | [最後の] | |
| 100μMのExA2-IX | 75 | 15μM |
| 100μMのExA2-33 | 75 | 15μM |
| 1Mトリス(pH7.5)で | 50 | 100 mMの |
| 5 M NaClを | 5 | 50 mMの |
| ddH 2 O | 295 | - |
| 全容積 | 500 |
表8. P7アダプタアニーリングミックス。
| 容量(μL) | [最後の] | |
| 100μMのExA1-58 | 75 | 15μM |
| 100μMのExA1-13 | 75 | 15μM |
| 1Mトリス(pH7.5)で | 50 | 100 mMの |
| 5 M NaClを | 5 | 50 mMの |
| ddH 2 O | 295 | - |
| 全容積 | 500 |
表9. P5アダプタアニーリングミックス。
| 温度(℃) | 時間 |
| 95 | 5分 |
| 72 | 5分 |
| 65〜60ランプ減 | 5分 |
| 55〜50ランプ減 | 3分 |
| 45〜40ランプ減 | 3分 |
| 30 | 3分 |
| 20 | 3分 |
| 10 | 3分 |
| 4 | フォーエバー |
表10.アダプターアニーリングプログラム。
| 1倍(μL) | [最後の] | |
| ddH 2 O | 39.8 | |
| 10×反応緩衝液2 | 5 | 1倍 |
| 100μMのATP | 0.5 | 1 mMの |
| 3 mMののdNTP | 1.7 | 100μM |
| 3 U /μlのT4ポリメラーゼ | 1 | 3 U |
| 5 U /μlのクレノウ | 1 | 5 U |
| 10 U /μlのT4ポリヌクレオチドキナーゼ | 1 | 10 U |
| 全反応容量 | 50 |
表11.研磨マスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 42.3 | |
| 10×反応緩衝液2 | 5 | 1倍 |
| 3 mMのdATPを | 1.7 | 100μM |
| 5 U /μlのクレノウ3'-5 'エキソマイナス | 1 | 5 U |
| 全反応容量 | 50 |
表12. A-テーリングマスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 41 | |
| 100 mMのATP | 0.5 | 1 mMの |
| 10×反応緩衝液2 | 5 | 1倍 |
| 400 U /μlのT4 DNAライトディASE | 1.5 | 600 U |
| 反応混合物の体積 | 48 | |
| 15 mMのインデックスアダプタ | 2 | 30ピコモル |
| 全反応容量 | 50 |
表13. P7アダプターライゲーションマスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 41 | |
| 10倍Φ-29バッファ | 5 | 1倍 |
| 3 mMののdNTP | 2.5 | 150μM |
| 10 U /μlのΦ-29ポリメラーゼ | 1.5 | 15 U |
| 全反応容量 | 50 |
表14.Φ-29ニック修復マスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 43.5 | |
| 100 mMのATP | 0.5 | 1 mMの |
| 10×反応緩衝液2 | 5 | 1倍 |
| 10 U /μlのT4ポリヌクレオチドキナーゼ | 1 | 10 U |
| 全反応容量 | 50 |
表15.キナーゼ反応マスターミックス。
| 1倍(μリットル) | [最後の] | |
| ddH 2 O | 43 | |
| 10倍ラムダバッファ | 5 | 1倍 |
| 5 U /μlのラムダエキソヌクレアーゼ | 2 | 10 U |
| 全反応容量 | 50 |
表16.ラムダエキソヌクレアーゼ反応マスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 44 | |
| 10×反応緩衝液2 | 5 | 1倍 |
| 30エキソヌクレアーゼF U /μlのRecJ | 1 | 30 U |
| 全反応Volume | 50 |
表17. RecJ Fヌクレアーゼ反応マスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 6.45 | |
| 10倍Φ-29バッファ | 2 | 1倍 |
| 3 mMのdNTPを | 1.3 | 200μM |
| 20μMP7プライマー | 0.25 | 0.25μM |
| 反応混合物の体積 | 10 | |
| エタノール沈殿したサンプル | 10 | |
| 全反応容量 | 20 |
表18. P7プライマー伸長反応マスターミックス。
| 温度(℃) | 時間 |
| 95 | 5分 |
| 65 | 5分 |
| 30 | 2分 |
| 30 | Φ-29が追加されるまで保留 |
| 30 | 20分 |
| 65 | 10分 |
| 4 | フォーエバー |
表19. P7プライマー伸長プログラム。
| 1×(μL) | [最後の] | |
| ddH 2 O | 5 | |
| 10×反応緩衝液2 | 3 | 1倍 |
| 3 mMのdATPを | 1 | 0.1 mMの |
| 5 U /μlのクレノウ3 'から5'エキソマイナス | 1 | 5 U |
| 反応混合物の体積 | 10 | |
| プライマー拡張サンプル | 20 | |
| 全反応容量 | 30 |
表20. A-テーリングマスターミックス。
| 1×(μL) | [最後の] | |
| ddH 2 O | 11.5 | |
| 10×T4リガーゼバッファー | 5 | 1倍 |
| 15μMExA1-58 /13アダプター | 2 | 30ピコモル |
| 400 U /μlのT4 DNAリガーゼ | 1.5 | 600 U |
| 反応混合物の体積 | 20 | |
| 尾のサンプル | 30 | |
| 全反応容量 | 50 |
表21. P5アダプターライゲーションマスターミックス。
| 1×(μL) | [最後の] | |
| 5X PCRバッファー | 10 | 1×(2のMgCl 2) |
| 各dNTPの10mMの | 1 | 200μMの各 |
| 20μMP1.3プライマー | 1.25 | 0.5μM |
| 20μMP2.1プライマー | 1.25 | 0.5μM |
| 2 U /μlのホットスタートポリメラーゼ | 0.5 | 1 U |
| 反応混合物の体積 | 14 | |
| ビーズ溶出された試料 | 36 | |
| 全反応容量 | 50 |
表22. PCR。
| 温度(℃) | 時間 | サイクル |
| 98 | 30秒 | 1 |
| 98 | 10秒 | 15-21(ChIPの効率に応じて) |
| 52 | 30秒 | |
| 72 | 20秒 | |
| 72 | 2分 | 1 |
| 4 | フォーエバー | ホールド |
表23. PCRプログラム。
| あたりのDNAスタンダード(μL) | サンプルあたり(μL) | |
| 1:200に希釈し、バッファ/色素混合物 | 190 | 198 |
| DNA標準 | 10 | |
| ChIP-エキソライブラリ | 2 | |
| 全容積 | 200 | 200 |
表24.定量。
ディスカッション
我々は、クロマチンが近い塩基対の解像度で公平な、ゲノムワイドな方法で相互作用タンパク質の正確な結合位置を決定するために、機能ゲノムプロトコルを提示します。免疫沈降物は、磁性樹脂のままに近い塩基対のマッピング分解能を達成するための最も重要なステップは、チップに富むDNAのエキソヌクレアーゼ処理です。表向きは、タンパク質複合体は、潜在的に任意のサブユニット( 例えば、クロマチンリモデリング複合体またはヌクレオソームコア粒子)のin vivoフットプリントをブロックすることができます。ホルムアルデヒドは、非効率的な架橋剤であるので、以前に10、報告されているように、それは、複雑な複数のサブユニットは、同じ遺伝子座で同じ細胞中のDNAと相互に架橋するであろうことがますますにくくなります。したがって、そのようなヌクレオソームの個々のヒストンサブユニットなどのタンパク質複合体の個々のサブユニットのインビボフットプリンティング、チップエキソで可能です。
tは ">のChIP-エキソの最も顕著な利点は、その近くに塩基対の解像度と低バックグラウンドです。超高解像度は、他の方法で、現在は不可能なゲノムワイドなスケールで作られている詳細な構造と空間の洞察を可能にします一方、ChIPのエキソの主な制約は、習得する技術的に困難な分子生物学的方法論であるということである。また、チップステップの一般的な制限は、チップエキソ( 例えば 、市販の抗体の可用性および特異性に適用されます、エピトープの接近性、および必要な細胞の比較的多数)。一般的な落とし穴は、非チップグレード抗体を使用して、質の悪い超音波処理抽出物を含み、できるだけ氷上に試料を保持しない。これにより、超音波処理条件を注意深く最適化され、各されなければなりません以前は、これらのパラメータは、実験結果に有することができる有害な影響を回避するために、18に記載の抗体が検証します。の限り転写因子のターゲットのためのシーケンシングの深さは、我々は、典型的には、約20万人は一意に読み込む整列を目指します。ポルIIとヒストン修飾は、より広範に分布しているので、我々は30読み取る万ドルから50を目指しています。チップエキソチップ、配列よりも実質的に低いバックグラウンドを有しているため、より少ない読み出し、同様の配列決定の深さを達成するために必要とされることに留意することが重要です。
オリジナルのプロトコル上の堅牢なバリエーションが17,19,20を出版され続け、チップエキソ技術は、現在広く、その技術的な課題にもかかわらず、採用されています。具体的には、ChIPのタンパク質に困難のために有用であることを証明し得る一つのバリエーションは、ライブラリの準備20の効率を高めるために、単一の連結工程を使用したChIP-ネクサスと呼ばれています。要約すると、のChIP-エキソは、グローバル規模でのクロマチン相互作用するタンパク質の超高解像度のマッピングのため、ますます採用し、強力な方法があります。市販のChIPグレードアリのリストとしてibodiesは成長を続け、ChIPのエキソ方法論の将来のアプリケーションは、超高解像度での細胞の分子回路を理解するために、マッピング未知の遺伝子調節ネットワークに向けられるであろう。また、チップエキソは、おそらく、さらに洗練されたこと及び周辺の塩基対の分解能でのin vivoタンパク質-RNA相互作用をフットプリントするように適合されます。
開示事項
The authors have nothing to disclose.
謝辞
We thank the Venters Lab and members of the Molecular Physiology and Biophysics Department for helpful discussions. Special thanks to Frank Pugh for his guidance, mentoring, and many insightful discussions on the nuances of ChIP-exo while I was a post-doctoral fellow in his laboratory.
資料
| Name | Company | Catalog Number | Comments |
| 37% formaldehyde, methanol free, 10x10ml ampules | ThermoFisher Scientific | 28908 | Section 3 |
| Complete Protease Inhibitor cocktail (CPI) | Roche Life Science | 11873580001 | Sections 4, 8 |
| Bioruptor sonicator | Diagenode | UCD200 | Section 5 |
| 15 ml polystyrene tubes | BD Falcon | 352095 | Section 5 |
| MagSepharose Protein G Xtra beads | GE Healthcare | 28-9670-66 | Section 6 |
| DynaMag-1.5ml Side Magnetic Rack | Invitrogen | 12321D | Sections 6-17, 22 |
| Mini-Tube Rotator | Fisher Scientific | 05-450-127 | Sections 7, 22 |
| T4 DNA Polymerase | New England BioLabs | M0203 | Section 9 |
| DNA Polymerase I, Klenow | New England BioLabs | M0210 | Section 9 |
| 10x NEBuffer 2 (10x Reaction Buffer 2) | New England BioLabs | B7002 | Sections 9-11, 13, 15, 20 |
| Thermomixer C | Eppendorf | 5382 | Sections 9-16 |
| ATP | Roche Life Science | 010419979001 | Sections 9, 11, 13 |
| dNTPs | New England BioLabs | N0447 | Sections 9, 12, 19, 23 |
| T4 Polynucleotide Kinase | New England BioLabs | M0201 | Sections 9, 13 |
| dATP | New England BioLabs | N0440 | Sections 10, 20 |
| Klenow 3'-5' Exo Minus | New England BioLabs | M0212 | Sections 10, 20 |
| T4 DNA ligase | New England BioLabs | M0202 | Sections 11, 21 |
| Φ-29 DNA Polymerase | New England BioLabs | M0269 | Sections 12, 19 |
| 10x Φ-29 Buffer | New England BioLabs | B0269 | Sections 12, 19 |
| Lambda Exonuclease | New England BioLabs | M0262 | Section 14 |
| 10x Lambda Buffer | New England BioLabs | B0262 | Section 14 |
| RecJf Exonuclease | New England BioLabs | M0264 | Section 15 |
| Proteinase K | Roche Life Science | 03115828001 | Section 16 |
| Glycogen | Roche Life Science | 010901393001 | Section 18 |
| 10x T4 Ligase Buffer | New England BioLabs | B0202 | Section 21 |
| AMPure XP (clean up) beads | Beckman Coulter | A63881 | Section 22 |
| Q5 Hot Start DNA Polymerase | New England BioLabs | M0493 | Section 23 |
| 5x Q5 Buffer (5x PCR Buffer) | New England BioLabs | B9027 | Section 23 |
| Qubit Fluorometer | Invitrogen | Q33216 | Section 25 |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Section 25 |
| Optical Clear Qubit tubes | Invitrogen | Q32856 | Section 26 |
| Qubit dsDNA High Sensitivity Assay kit | Invitrogen | Q32851 | Section 26 |
参考文献
- Venters, B. J., Pugh, B. F. How eukaryotic genes are transcribed. Crit Rev Biochem Mol Biol. 44, 117-141 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Chen, J., et al. Single-molecule dynamics of enhanceosome assembly in embryonic stem cells. Cell. 156, 1274-1285 (2014).
- Wales, S., Hashemi, S., Blais, A., McDermott, J. C. Global MEF2 target gene analysis in cardiac and skeletal muscle reveals novel regulation of DUSP6 by p38MAPK-MEF2 signaling. Nucleic acids research. 42, 11349-11362 (2014).
- Cho, S., et al. The architecture of ArgR-DNA complexes at the genome-scale in Escherichia coli. Nucleic Acids Res. 43, 3079-3088 (2015).
- Katainen, R., et al. CTCF/cohesin-binding sites are frequently mutated in cancer. Nat Genet. 47, 818-821 (2015).
- Murphy, M. W., et al. An ancient protein-DNA interaction underlying metazoan sex determination. Nat Struct Mol Biol. 22, 442-451 (2015).
- Zere, T. R., et al. Genomic Targets and Features of BarA-UvrY (-SirA) Signal Transduction Systems. PLoS One. 10, e0145035 (2015).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Rhee, H. S., Bataille, A. R., Zhang, L., Pugh, B. F. Subnucleosomal structures and nucleosome asymmetry across a genome. Cell. , 1377-1388 (2014).
- Pugh, B. F., Venters, B. J. Genomic Organization of Human Transcription Initiation Complexes. PLoS One. 11, e0149339 (2016).
- Albert, I., Wachi, S., Jiang, C., Pugh, B. F. GeneTrack--a genomic data processing and visualization framework. Bioinformatics. 24, 1305-1306 (2008).
- Guo, Y., Mahony, S., Gifford, D. K. High resolution genome wide binding event finding and motif discovery reveals transcription factor spatial binding constraints. PLoS computational biology. 8, e1002638 (2012).
- Bardet, A. F., et al. Identification of transcription factor binding sites from ChIP-seq data at high resolution. Bioinformatics. 29, 2705-2713 (2013).
- Wang, L., et al. MACE: model based analysis of ChIP-exo. Nucleic Acids Res. 42, e156 (2014).
- Bentley, D. R., et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456, 53-59 (2008).
- Rhee, H. S., Pugh, B. F. ChIP-exo method for identifying genomic location of DNA-binding proteins with near-single-nucleotide accuracy. Curr Protoc Mol Biol. Chapter 21, Unit 21.24 (2012).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22, 1813-1831 (2012).
- Serandour, A. A., Brown, G. D., Cohen, J. D., Carroll, J. S. Development of an Illumina-based ChIP-exonuclease method provides insight into FoxA1-DNA binding properties. Genome Biol. 14, R147 (2013).
- He, Q., Johnston, J., Zeitlinger, J. ChIP-nexus enables improved detection of in vivo transcription factor binding footprints. Nat Biotechnol. 33, 395-401 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved