Method Article
Описание фактора транскрипции зависимая регуляция микроРНК Транскриптом
В этой статье
Резюме
Herein we propose a strategy to study the effect of a transcription factor of interest on the microRNA transcriptome using publically available data, computational resources and high throughput data from microRNA arrays after transfecting cells with small hairpin (sh)RNA targeting a transcription factor of interest.
Аннотация
В то время как регуляция транскрипции генов, кодирующих белок был хорошо изучен, очень мало известно о том, как факторы транскрипции, принимают участие в транскрипции некодирующих РНК, в частности микроРНК. Здесь мы предлагаем стратегию для изучения потенциальной роли фактора транскрипции в регуляции транскрипции микроРНК с использованием общедоступный данных, вычислительные ресурсы и высокие данные пропускной способности. Мы используем эпигенетическую подпись H3K4me3 для идентификации микроРНК промоторы и иммунопреципитации хроматина (CHIP) данные характерны чередования проекта ENCODE для выявления микроРНК промоутеров, которые обогащены сайтов связывания транскрипционных факторов. Трансфекцией клетки, представляющие интерес с shRNA ориентируетесь на транскрипционный фактор интереса и подвергая клетки к микроРНК массива, мы изучаем влияние этого фактора транскрипции на транскриптома микроРНК. В качестве иллюстративного примера мы используем наше исследование о влиянии STAT3 на микроРНК транскриптома хронического Lymphocytic лейкоз (ХЛЛ) клетки.
Введение
MicroRNAs являются эндогенными малые не кодирующие регуляторные РНК, которые, как правило, функционируют как негативные регуляторы экспрессии мРНК на пост-транскрипционном уровне. Приблизительно 1000 некодирующих от 20 до 25 нуклеотид длинные микроРНК обнаружены в геноме человека 1,2. MicroRNAs регулируют экспрессию генов посредством канонического спаривания оснований между последовательностью семенем микроРНК и ее комплементарной последовательностью соответствия семян, которая обычно расположена в нетранслируемой области 3 '(UTR) мишени мРНК. Все вместе, микроРНК регулируют более 30% протеина генов , кодирующих 3, но только очень мало известно о транскрипции с ДНК микроРНК. Было высказано предположение о том , что регулирование транскрипции микроРНК аналогична мРНК 4,5. В частности, подобно его активности в продвижении транскрипции генов , кодирующих белок, факторы транскрипции , как полагают , чтобы активировать транскрипцию микроРНК 6. Транскрипция фактор-мicroRNA взаимодействие было сообщено как фактор модулятора экспрессии гена 7, а также может формировать обратную связь и прямой подачей петли. Например, Yamakuchi и др. Сообщили , контур обратной связи , в котором р53 индуцирует экспрессию microRNA34a, что , в свою очередь , ингибирует трансляцию р53 репрессора SIRT и тем самым увеличивая активность p53 8.
В то время как конкретные примеры зависимого фактора транскрипции экспрессии микроРНК сообщалось, общепринятый метод, который дает информацию о том, как фактор транскрипции интерес регулирует экспрессию микроРНК-транскриптом отсутствует. Целью протокола предлагаемого в настоящем документе, чтобы обеспечить описание углубленную фактора транскрипции-зависимой регуляции микроРНК-транскриптома. Объединив общедоступный данные, биоинформатики инструменты и с использованием технологии микрочипов, исследователи, которые следуют за этот алгоритм будет иметь возможность захватить на геномной шкале, как любойфактор транскрипции, в любом типе клеток, представляющих интерес регулирует экспрессию микроРНК-транскриптом, а также изучить предполагаемый вклад фактора транскрипции мРНК в регуляции экспрессии микроРНК.
протокол
1. Определение фактора транскрипции сайтов связывания в промоторе микроРНК Гены Использование интеллектуального анализа данных подход
- Используйте Калифорнийский университет Санта-Круз (УСК) генома браузер для извлечения данных иммунопреципитация (ЧИП) секвенирования хроматина, сгенерированные в рамках проекта Энциклопедии элементов ДНК (ENCODE).
- Откройте браузер таблицы в геноме браузера УСК.
- Используйте следующие спецификации для извлечения таблицы: клады: (Млекопитающие), геном: (человека), Ассамблея: (Feb2009 (GRch37 / hg18)), группа: (регулирование), дорожки (TxnFactorChIP), таблица: (weEncoderesTFbsCo7steredv3), область : (Геном), формат вывода (все гены от выбранной таблицы).
- Сохранить вывод 1.1.2 как текстовый файл и в таблицу.
- Сортировка и фильтр для фактора транскрипции представляющего интерес (например, STAT2).
- Используйте список микроРНК промоутеров на основе H3K4me3 эпигенетических подписи имеющейся у Baeer и др. 9 . Скопируйте этот список в текстовый файл.
- Для того, чтобы соответствовать по координатам на геном человека, сопоставить данные из 1.1 и 1.2 (например, STAT3 связывания на предполагаемых микроРНК промоутеров) и определяют медианное аффинности связывания с использованием кода , написанного на C #, как указано в дополнительный файл 1 кодирования.
2. Используйте shRNA для понижающей регуляции экспрессии транскрипционного фактора интереса
- Пластина 1,5 х 10 6 клеток из 293 эмбриона человека линии клеток почки в 10 см пластины приблизительно 50% слияния (DMEM с 10% FBS).
- Трансфекции клеток от 293 эмбриона человека линии клеток почки с 5 мкг белка зеленой флуоресценции (GFP) лентивирусов, содержащего shRNA направленный к фактору транскрипции интересующего и с 5 мкг упаковочных векторов с использованием реагента для трансфекции для адгезивных клеток, в соответствии с протоколом производителя.
- В качестве контроля трансфекции клеток от 293 эмбриона человека линии клеток почки свскарабкался shRNA и упаковочные векторы в соответствии с протоколом производителя.
- Хранить смесь для трансфекции на клетках в течение 16 ч (при 37 ° С, СО 2 инкубатор), а затем замены среды до 10 мл свежей 10% DMEM среды с добавлением 10% FBS.
- Подождите 48 часов после трансфекции, центрифугирование клеточной культуры (300 XG, 5 мин) и собирают супернатант инфекционный. Фильтр супернатанта через фильтр в шприце с размером пор 0,45 мкм (25-мм поверхностно-активное вещество из ацетата целлюлозы без мембраны) для удаления плавающих клеток.
- Концентрат супернатант и собирают лентивирусов с использованием ultracentrifugal устройства фильтра с порогом 100 кДа. Спин при 950 мкг в течение 30 - 60 мин, пока объем был концентрат менее 250 мкл. Хранить концентрированный вирус при -80 ° С.
- Трансфекцию клетки с лентивирусов. Удалить замороженную лентивирус от -80 ° C морозильника и разморозить до комнатной температуры. Перенести 100 мкл вирусного супернатанта в свежую 1,5 мл пробирке.
- Бринг до объема в трубке до 1 мл со сниженной сывороточной средой. Добавить бромид hexadimethrine к 1 мл суспензии вируса в конечной концентрации 10 нг / мл, осторожно перемешать и дайте смеси отстояться в течение 5 мин.
- Центрифуга 5 × 10 6 клеток для каждой трансдукции и осторожно ресуспендируют осадок клеток в 0,5 мл среды , содержащей вирус. Пусть клетки остаются в инкубаторе в течение 4 - 24 ч, а затем добавляют 0,5 мл среды с 20% FBS до конечной концентрации 10% FBS.
- Подождите 48 до 72 ч и окрашивает клетки с пропидийиодидом (PI) и зеленого белка флуоресцентной (GFP) в соответствии с инструкциями изготовителя. Защищают клетки от света и использовать FACS сортировщик для измерения темпов GFP + / PI - клетки. Так как PI пятна только мертвые клетки, этот показатель является оценкой эффективности трансфекции в живых клетках.
- Сортировать положительную популяцию клеток к экспрессии GFP (GFP +) с помощью FACS сортировщик , как описано выше 10.
- Использование Westeгп иммунный блоттинг , как описано выше 10 , чтобы определить уровни фактора транскрипции интересующего до и после того, как инфицировать клетки интерес (например, ХЛЛ клетки) с назначенным shRNA.
3. Определение уровня экспрессии микроРНК Транскриптом в клетках, трансфицированных фактор транскрипции-shRNA
- Изолировать РНК с использованием коммерческого набора в соответствии с протоколом производителя.
- Добавьте РНК и гибридизуются его микроРНК микрочипов 11.
- Определить дифференциально выраженную микроРНК в клетках , трансфицированных фактор транскрипции-shRNA или с пустыми векторных элементов управления 5.
- Проверка результатов микрочипов для наиболее дифференциально экспрессируются микроРНК с использованием ПЦР в реальном времени 5.
4. Определить Промежуток между биоинформатики и подход shRNA в описании зависимых Транскриптом фактора транскрипции
- опргорностая ожидаемые и наблюдаемые соотношения генов микроРНК , которые укрывают сайтов связывания транскрипционных факторов в их промоутеров и были подавляются в транскрипции фактор-shRNA трансфицированных клеток, выполните следующие действия :
- Получить соотношение количества генов, которые укрывают фактор транскрипции интерес в их общей промотор / микроРНК из списка, генерируемого в 1.3. Этот список представляет собой ожидаемый коэффициент (например, гены микроРНК с STAT3 сайты связывания / всего генов микроРНК испытания = 0,25).
- Определить количество генов , которые были подавляются в фактор транскрипции-shRNA трансфицированных клеток из списка , генерируемого в 3.3 и имеет фактор транскрипции процентных связывающих сайтов из списка , генерируемого в 1.3. Это число / общее число генов подавляются является наблюдаемое отношение (например, гены микроРНК с STAT3 сайтов связывания / общее число подавляются микроРНК = 0,6).
- Используйте χ2 статистику для сравнения наблюдаемых и ожидаемых соотношений, которые были созданы выше, и определить, является ли список генов, которые были подавляются в фактор транскрипции-shRNA трансфицированных клеток обогащены сайтов связывания транскрипционных факторов в их промотор.
Результаты
STAT3 представляет собой фактор транскрипции , который обычно индуцирует транскрипцию генов , которые обладают противовоспалительными апоптическая и пролиферативные эффекты 12. Является ли STAT3 также влияют на некодирующие РНК транскриптом в настоящее время неизвестно. Во всех ХЛЛ клетках STAT3 является конститутивно фосфорилируется по остаткам серин 707 остатков 10,13. Фосфосерин STAT3 челноки к ядру, связывается с ДНК и активирует гены , как известно, активируется тирозина pSTAT3 в других типах клеток 10. Поскольку ХЛЛ характеризуется глобальной дерегуляции микроРНК сети 14, мы предположили , что присутствие серина pSTAT3 влияет на экспрессию микроРНК в клетках ХЛЛ.
Чтобы проверить эту гипотезу промоторы микроРНК, которые укрывают STAT3 сайты связывания должны были быть идентифицированы. Путем скрещивания данных , сгенерированных Бэра и др. 9 регионов с гистона modifi H2K4me3катион , который характеризуют промоторные сайты, сайты связывания STAT3 выявленных микросхемой-сл эксперимента 15, были идентифицированы предполагаемые сайты связывания STAT3. Используя этот подход , 160 предполагаемых промоутеров были обнаружены в почти 25% генов микроРНК исследованных (N = 200) , связывающими баллов в диапазоне от 100 (самый низкий балл) до 1000 (самый высокий балл) (таблица 1).
Впоследствии ХЛЛ клетки трансфицировали STAT3-shRNA или пустым вектором и с помощью микроРНК массива и определили 63 микроРНК , которые были вниз регулируется после трансфекции (рисунок 1) предполагая , что STAT3 способствует транскрипции этих микроРНК. Для 60% из 63 генов подавляются микроРНК (п = 38) Древесностружечные-сл данные подтвердили STAT3 связывание в мнимого промотора вверх по течению от места гена, что значительно больше, чем ожидалось случайно (р <0,0001). Девять микроРНК, которые были вниз регулируется после трансфекции,предполагая, что STAT3 негативно регулирует свои уровни транскрипции.
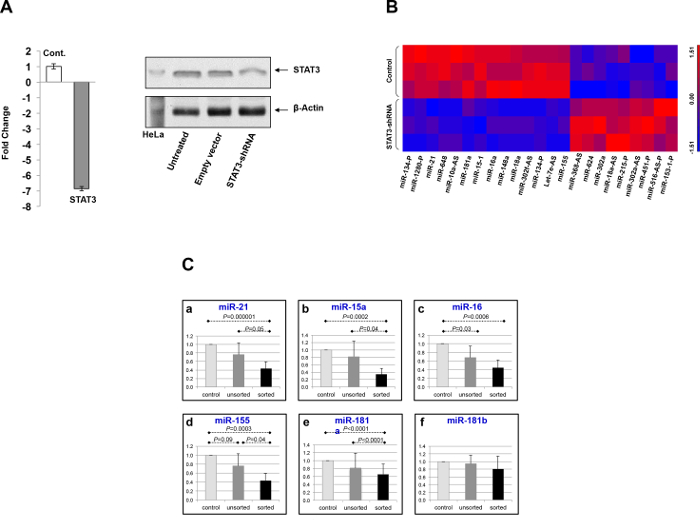
Рисунок 1. Трансфекция клеток ХЛЛ с STAT3-shRNA снижает уровни экспрессии белка STAT3 и мРНК STAT3. A:. После трансфекции ХЛЛ клеток с уровнями STAT3-shRNA мРНК STAT3 ( на левой панели, обнаруженной с помощью количественной ОТ -ПЦР) и уровни белка STAT3 (правая панель, обнаруженных западной иммуноблоттинга) значительно уменьшилось В: микроРНК массив ХЛЛ Показанные клетки 23 микроРНК, чья экспрессия существенно отличались между ХЛЛ клеток, трансфицированных STAT3-shRNA и ХЛЛ клеток, трансфицированных пустым вектором. Величина Р менее 0,01 считалось статистически значимым С:. Среднее значение предельного выражение , количественно с помощью RT-PCR 7 микроРНК , которые имели дифференциальную экспрессию после лечения STAT3-shRNA с использованиеммикроРНК массив. Столбики показывают стандартную ошибку среднего значения. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Дополнительное кодирование файлов 1. Пожалуйста , нажмите здесь , чтобы загрузить этот файл.
| Ген микро РНК | хромосома | Координаты старта Promoter | Конечные координаты Promoter | Медиана (диапазон) STAT3 счет связывания * |
| микроРНК-1205, микроРНК-1206, микроРНК-1207 | 8 q24.21 | 128961454 | 128962791 | 1000 (1000-1000) |
| микроРНК-1537 | 1 q42.3 | 236045425 | 236047415 | 1000 (1000-1000) |
| микроРНК-21 | 17 q23.1 | 57901872 | 57921277 | 1000 (112-1000) |
| микроРНК-3124 | 1 Q44 | 249115404 | 249123965 | 1000 (1000-1000) |
| микроРНК-451 | 17q11.2 | 27222251 | 27224114 | 1000 (1000-1000) |
| микроРНК-92b | 1 Q22 | 155162340 | 155168439 | 1000 (1000-1000) |
| микроРНК-3197 | 21 q22.2 | 42537544 | 42543023 | 943 (943-943) |
| микроРНК-646 | 20 q13.33 | 58712550 | 58715320 | 789 (789-789) |
| микроРНК-629 | 15 Q23 | 70383751 | 70394586 | 773 (661-885) |
| микроРНК-30e, микроРНК-30С-1 | 1 p34.2 | 41173077 | 41177703 | 759 (759-759) |
| микроРНК-3125 | 2 p24.3 | 12855381 | 12862915 | 756 (756-756) |
| микроРНК-3145 | 6 q23.3 | 138776942 | 138779365 | 743 (487-1000) |
| микроРНК-645 | 20 q13.13 | 49199911 | 49201187 | 743 (743-743) |
| микроРНК-1256 | 1 p36.12 | 21346830 | 21350211 | 725 (725-725) |
| микроРНК-619 | 12 q24.11 | 109248263 | 109253306 | 719 (719-719) |
| микроРНК-181а-2, микроРНК-181b-2 | 9 q33.3 | 127418928 | 127426139 | 710 (710-710) |
| микроРНК-29а, микроРНК-29b-1 | 7 q32.3 | 130583383 | 130597803 | 697 (482-1000) |
| микроРНК-202 | 10 q26.3 | 135069499 | 135077337 | 696 (393-1000) |
| микроРНК-3142, микроРНК-146а | 5 q34 | 159890882 | 159899475 | 671 (671-671) |
| микроРНК-548C | 12 q14.2 | 65000968 | 65011503 | 660 (660-660) |
| микроРНК-630 | 15 q24.1 | 72764289 | 72769197 | 627 (255-1000) |
| микроРНК-135b | 1 q32.1 | 205416952 | 205452990 | 622 (245-1000) |
| микроРНК-29c, микроРНК-29b-2 | 1 q32.2 | 207991044 | 208002382 | 608 (608-608) |
| микроРНК-1825 | 20 q11.21 | 30791020 | 30798310 | 604 (209-1000) |
| микроРНК-548h-1 | 14 q23.2 | 64578834 | 64581657 | 587 (174-1000) |
| микроРНК-612 | 11 q13.1 | 65183633 | 65198528 | 581 (157-1000) |
| микроРНК-148b | 12 q13.13 | 54717640 | 54721204 | 578 (578-578) |
| микроРНК-3174 | 15 q26.1 | 90543381 | 90549092 | 576 (152-1000) |
| let7a-3, let7b | 22 | 46480680 | 46481826 | 573 (146-1000) |
| микроРНК-1255a | 4 Q24 | 102263848 | 102272541 | 557 (557-557) |
Таблица 1. промоутеры мнимого микроРНК с сайтами связывания STAT3. Протокол предоставляет способ для идентификации связывания на предполагаемых промоутеров микроРНК с использованием интеллектуального анализа данных из опубликованных данных фактора транскрипции, В качестве примера, приведем здесь таблицу, изображающую связывание STAT3 с предполагаемыми микроРНК промоутеров. Связующий оценка дается на 0 до 1000 шкале от целых данных Seq генома ChIP , опубликованных в рамках проекта закодированного материала 15. Активаторы идентифицируются наличием H3K4me3 эпигенетическому подписи 9.
Обсуждение
Механизм, лежащий в основе РНК-полимеразы II-зависимую транскрипцию белок-кодирующих генов хорошо изучена. В то время как эти элементы составляют лишь 1% - 2% генома человека, данные из проекта ENCODE предполагают , что более 80% генома человека может подвергаться транскрипцию 17 и что регулирует транскрипцию некодирующих элементов ДНК остается в значительной степени неизвестны 6 ,
Несколько исследований, в которых указано , что Pol II также отвечает за транскрипцию некоторых не-белок-кодирующих генов , в том числе микроРНК 6, привело нас к разработке стратегии , которая может объединять данные из общедоступных ресурсов, вычислительных алгоритмов и в пробирке исследования , чтобы расшифровать потенциал функция фактора транскрипции интереса к регуляции транскрипции микроРНК. Стратегия предложено здесь включает в себя 2 важных шагов. Во-первых мы используем общедоступный данные для идентификации фактора транскрипции бinding в микроРНК промоутеров. С этой целью мы используем чип-Seq данные, опубликованные ENCODE консорциума для выявления связывания транскрипционных факторов и эпигенетические подпись, которая служит прообразом промотор как косвенный маркер микроРНК-промоутеров. Пересечение генетических координат из этих наборов данных дает грубую оценку, как часто транскрипционный фактор интереса связывается с микроРНК-промотора. Во-вторых, используя технологию shRNA, чтобы заглушить экспрессию фактора транскрипции, и подвергая клетки к микроРНК массива, можно исследовать функциональное значение фактора транскрипции на микроРНК-транскриптома.
Изменчивость в выражении микроРНК лишь частично объясняется зависимым фактором транскрипции регулирования. Стехиометрический изменчивость и другие клеточные или внеклеточные факторы играют важную роль и не смоделированы в предлагаемом алгоритме. Другие ограничения включают в себя следующее: анализ промотор, основанный на H3K4me3подпись была сделана на 939 аннотированных микроРНК. С тех пор были определены геномные расположение многих больше микроРНК. Однако, насколько нам известно, более полный список, который основан на обновленной базе данных еще не опубликованы. Из генов микроРНК 939, то H3K4me3, который служит прообразом участок промотора был идентифицирован в 781 генов микроРНК (83%). Следовательно, в то время как этот анализ явно основано на неполном наборе данных он захватывает значительную часть микроРНК-транскриптома.
Кроме того, эпигенетика подпись частично отпечатаны и в части соты. Поэтому порождаемость предполагаемых промоутеров, которые были определены с помощью эпигенетических маркеров может быть поставлена под сомнение. Поскольку H3K4me3 сохраняется независимо от транскрипции 18 обычно считается маркером импринтированных промоутеров. Поэтому аналитический алгоритм мы предлагаем здесь, могут пропустить клеточные специфические микроРНК промоторы, если эти промоторы были идентифицированы в другом сотовомтип. Наконец, любое заключение должно быть проверено и подтверждено эмпирически. В частности, связь между понижающей регуляции фактора транскрипции (с использованием shRNA подход) и экспрессию микроРНК (обозначаемого микроРНК массив) может предложить только прямое транскрипционный роль, которая должна быть подтверждена приемлемых анализов, таких как иммунопреципитации хроматина (CHIP) или электрофоретической подвижности методом сдвига (EMSA). Модификации метода, предложенного в изобретении, включают различные способы идентификации микроРНК промотор и различные способы нокдаун экспрессии фактора транскрипции, например, малых интерферирующих РНК вместо shRNA. Регуляция транскрипции микроРНК может существенно отличаться для микроРНК, которые находятся в пределах кодирующих белок генов (внутригенных микроРНК) и те, которые этого не делают (межгенные микроРНК). Поскольку внутригенных микроРНК обычно транскрибируется в сочетании с их генами 19 - хозяевах промотор обычно находится сразу upstreaм сайта начала транскрипции. Тем не менее для многих межгенных микроРНК сайта инициации транскрипции плохо аннотированных и инструментов прогнозирования, которые обычно используются для белок - кодирующих генов плохо работают 20.
Раскрытие информации
The authors declare no competing financial interests.
Благодарности
Это исследование было поддержано грантом Фонда ХЛЛ Global Research. Университет Техаса MD Anderson Cancer Center поддерживается частично Национальным институтом здоровья через онкологический центр поддержки Грант (P30CA16672).
Материалы
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies | 11668027 | |
| 0.45 µm syringe filter | Thermo Scientific (Nalgene) | 190-2545 | |
| Amicon ultracentrifugal filter device with threshold of 100 kDa | Merck Millipore | ||
| Polybrene | Merck Millipore | TR-1003-G | |
| TRIzol reagent | Life Tachnologies (Invitrogen) | 15596-026 | |
| 293 Cell line human | Sigma-Aldrich | 85120602 |
Ссылки
- Bentwich, I., et al. Identification of hundreds of conserved and nonconserved human microRNAs. Nat Genet. 37, 766-770 (2005).
- Hata, A. Functions of microRNAs in cardiovascular biology and disease. Annu Rev Physiol. 75, 69-93 (2013).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Piriyapongsa, J., Jordan, I. K., Conley, A. B., Ronan, T., Smalheiser, N. R. Transcription factor binding sites are highly enriched within microRNA precursor sequences. Biol Direct. 6, 61 (2011).
- Rozovski, U., et al. Signal transducer and activator of transcription (STAT)-3 regulates microRNA gene expression in chronic lymphocytic leukemia cells. Mol Cancer. 12, 50 (2013).
- Turner, M. J., Slack, F. J. Transcriptional control of microRNA expression in C. elegans: promoting better understanding. RNA Biol. 6, 49-53 (2009).
- Cui, Q., Yu, Z., Pan, Y., Purisima, E. O., Wang, E. MicroRNAs preferentially target the genes with high transcriptional regulation complexity. Biochem Biophys Res Commun. 352, 733-738 (2007).
- Yamakuchi, M., Lowenstein, C. J. MiR-34, SIRT1 and p53: the feedback loop. Cell Cycle. 8, 712-715 (2009).
- Baer, C., et al. Extensive promoter DNA hypermethylation and hypomethylation is associated with aberrant microRNA expression in chronic lymphocytic leukemia. Cancer Res. 72, 3775-3785 (2012).
- Hazan-Halevy, I., et al. STAT3 is constitutively phosphorylated on serine 727 residues, binds DNA, and activates transcription in CLL cells. Blood. 115, 2852-2863 (2010).
- Melo, S. A., et al. A TARBP2 mutation in human cancer impairs microRNA processing and DICER1 function. Nat Genet. 41, 365-370 (2009).
- Akira, S. Functional roles of STAT family proteins: lessons from knockout mice. Stem Cells. 17, 138-146 (1999).
- Frank, D. A., Mahajan, S., Ritz, J. B lymphocytes from patients with chronic lymphocytic leukemia contain signal transducer and activator of transcription (STAT) 1 and STAT3 constitutively phosphorylated on serine residues. J Clin Invest. 100, 3140-3148 (1997).
- Calin, G. A., et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 101, 11755-11760 (2004).
- Consortium, E. P. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS biology. 9, e1001046 (2011).
- Miranda, K. C., et al. A pattern-based method for the identification of MicroRNA binding sites and their corresponding heteroduplexes. Cell. 126, 1203-1217 (2006).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57-74 (2012).
- Lau, J. C., Hanel, M. L., Wevrick, R. Tissue-specific and imprinted epigenetic modifications of the human NDN gene. Nucl Acids Res. 32, 3376-3382 (2004).
- Corcoran, D. L., et al. Features of mammalian microRNA promoters emerge from polymerase II chromatin immunoprecipitation data. PLoS One. 4, e5279 (2009).
- Bhattacharyya, M., Feuerbach, L., Bhadra, T., Lengauer, T., Bandyopadhyay, S. MicroRNA transcription start site prediction with multi-objective feature selection. Stat Appl Genet Mol Biol. 11, (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены