Method Article
בתארו תקנה Dependent גורם שעתוק של transcriptome MicroRNA
In This Article
Summary
Herein we propose a strategy to study the effect of a transcription factor of interest on the microRNA transcriptome using publically available data, computational resources and high throughput data from microRNA arrays after transfecting cells with small hairpin (sh)RNA targeting a transcription factor of interest.
Abstract
בעוד תקנה שעתוק של גנים המקודדים חלבונים נחקרה בהרחבה, מעט מאוד ידוע על איך גורמי שעתוק המעורבים שעתוק של RNA ללא קידוד, במיוחד של מיקרו-רנ"א. כאן, אנו מציעים אסטרטגיה ללמוד את התפקיד הפוטנציאלי של גורם שעתוק בוויסות שעתוק של מיקרו RNA באמצעות נתונים זמינים לציבור, משאבים חישוביים ונתונים קצב העברת נתונים גבוה. אנו משתמשים חתימת אפיגנטיים H3K4me3 לזהות יזמי microRNA ו immunoprecipitation הכרומטין (שבב) נתוני -sequencing מפרויקט קידוד לזהות יזמי microRNA מעשירים עם גורם שעתוק אתרי קישור. על ידי transfecting תאים בעלי עניין עם shRNA מיקוד גורם שעתוק של עניין והכפפת התאים למערך microRNA, אנחנו לומדים את השפעת גורם שעתוק זה על transcriptome microRNA. כדוגמא המחשה נשתמש המחקר שלנו על השפעת STAT3 על transcriptome microRNA של ly הכרוניתלוקמיה mphocytic (CLL) תאים.
Introduction
מייקרו RNA הוא RNAs רגולטוריות קידוד הלא קטן אנדוגני כי בדרך כלל לתפקד כמו רגולטורים שליליים של ביטוי mRNA ברמת posttranscriptional. כ -1,000 ללא קידוד 20 עד 25 מיקרו-רנ"א ארוך נוקלאוטיד נמצאים בגנום האנושי 1,2. מיקרו RNA לווסת ביטוי גנים באמצעות זיווג בסיס הקנונית בין רצף זרע של microRNA ורצף המשחק זרע המשלים, הנמצא בדרך כלל על האזור מתורגמת '3 (UTR) של תעתיקי היעד. באופן קולקטיבי, מיקרו-רנ"א לווסת יותר מ -30% של הגנים המקודדים חלבונים 3, אבל רק מעט מאוד ידוע על שעתוק מ- DNA של מיקרו-רנ"א. הועלתה השערה כי הסדרת שעתוק microRNA דומה לזה של mRNA 4,5. בפרט, בדומה לפעילותה בקידום שעתוק של גנים המקודדים חלבונים, גורמי שעתוק נחשבים להפעיל שעתוק של מיקרו-רנ"א 6. תמלול גורם-מ 'גומלין icroRNA דווח כגורם אפנן של ביטוי גנים 7, והוא רשאי גם טופס היזון חוזר ולולאות-קדימה להאכיל. לדוגמה, Yamakuchi et al. דיווח לולאת משוב שבו p53 גורם הביטוי של microRNA34a, אשר בתורו מעכב תרגום של SIRT מדכא p53 ו- p53 ובכך להגדיל פעילות 8.
בעוד דוגמאות ספציפיות הביטוי תלויה גורם שעתוק של מיקרו-רנ"א דווחו, שיטה מקובלת המספק מידע על איך גורם שעתוק של עניין מסדיר את הביטוי של-transcriptome microRNA חסר. מטרת הפרוטוקול הציע בזאת היא לספק תיאור המעמיק של רגולציה גורמת תלויה שעתוק של-transcriptome microRNA. על ידי שילוב של נתונים זמינים לציבור, כלי ביואינפורמטיקה בטכנולוגיית microarray, חוקרים העוקבים אחר אלגוריתם זה יהיה מסוגל לתפוס בסולם גנומי איך כלגורם שעתוק לכל סוג תא של עניין מסדיר את הביטוי של transcriptome-microRNA וכדי לחקור תרומה משוערת של mRNA-גורם שעתוק ויסות ביטוי microRNA.
Protocol
1. זהה אתרי קישור גורם שעתוק ב האמרגן של גני MicroRNA שימוש בגישת כריית הנתונים
- השתמש דפדפן הגנום באוניברסיטת קליפורניה בסנטה קרוז (UCSC) כדי לחלץ immunoprecipitation הכרומטין הנתונים רצף (שבב) שנוצר כחלק האנציקלופדיה של אלמנט דנ"א הפרויקט (קידוד).
- פתח את דפדפן שולחן בדפדפן הגנום UCSC.
- השתמש במפרטים הבאים כדי לחלץ את השולחן: clade: (יונקים), הגנום: (אדם), הרכבה: (Feb2009 (GRch37 / hg18)), קבוצה: (תקנה), מסלול (TxnFactorChIP), שולחן: (weEncoderesTFbsCo7steredv3), באזור : (הגנום), תבנית פלט (כל הגנים משולחן שנבחר).
- שמור את הפלט של 1.1.2 כקובץ TXT ו- לתוך גיליון אלקטרוני.
- למיין ולסנן עבור גורם שעתוק של עניין (למשל, STAT2).
- השתמש ברשימת יזמי microRNA מבוסס על חתימת אפיגנטיקה H3K4me3 הזמינה ב Baeer et al. 9 . העתק רשימה זו לתוך קובץ .txt.
- כדי להתאים פי קואורדינטות על גנום האדם, למפות את הנתונים מ -1.1 ו -1.2 (למשל, STAT3 מחייבים על יזמי microRNA משוערים) ולקבוע זיקת מחייב החציוני באמצעות הקוד שנכתב ב- C חדה כפי שהמתואר משלים קידוד קובץ 1.
2. השתמשו shRNA כדי דאון-להסדיר את הביטוי של גורם שעתוק של ריבית
- פלייט 1.5 x 10 6 תאי 293 שורת תאי כליה עוברית אנושית בצלחת 10 סנטימטרים על כ 50% מפגש (DMEM עם 10% FBS).
- תאי Transfect מ 293 קו תא הכליה עוברי אנושי עם 5 מיקרוגרם של חלבון פלואורסצנטי הירוק (GFP) lentivirus המכיל shRNA מופנים אל גורם השעתוק של עניין עם 5 מיקרוגרם של וקטורי אריזה באמצעות מגיב transfection עבור תאים חסידים על פי הפרוטוקול של היצרן.
- כביקורת, transfect את התאים מן 293 שורת תאי כליה עוברית אנושית עםמקושקשות shRNA ואת וקטורי אריזה על פי הפרוטוקול של היצרן.
- שמור תערובת transfection על תאים עבור 16 שעות (ב 37 ° C, CO 2 באינקובטור), ולאחר מכן שנה בתקשורת עד 10 מ"ל התקשורת 10% טרי DMEM עם FBS 10%.
- המתן 48 transfection שעות שלאחר, בצנטריפוגה תרבית תאים (300 XG, 5 דק ') ולאסוף supernatant זיהומיות. סנן את supernatant דרך פילטר מזרק 0.45 מיקרומטר (קרום יצטט תאית חינם פעיל שטח 25 מ"מ) כדי להסיר את כל תאים צפו.
- לרכז את supernatant לאסוף את lentivirus באמצעות מכשיר מסנן ultracentrifugal עם סף של 100 kDa. ספין ב 950 XG עבור 30 - 60 דקות עד נפח כבר להתרכז פחות מ 250 μl. אחסן את הנגיף המרוכז ב -80 מעלות צלזיוס.
- Transfect התאים עם lentivirus. הסר lentivirus קפוא מהמקפיא -80 ° C ו להפשיר לטמפרטורת החדר. העברת 100 μl של supernatant ויראלי לצינור טרי 1.5 מיליליטר microfuge.
- בריןg את הנפח בצינור עד 1 מיליליטר עם מדיום בסרום מופחת. להוסיף ברומיד hexadimethrine עד 1 מ"ל ההשעיה וירוס עבור ריכוז סופי של 10 ng / ml, מערבבים בעדינות ונותנים לתערובת לעמוד במשך 5 דקות.
- צנטריפוגה 5 x 10 6 תאים עבור כל התמרה בעדינות resuspend התא גלולה ב 0.5 מ"ל של התקשורת המכיל וירוס. בואו התאים להישאר בחממה 4 - 24 שעות, ולאחר מכן להוסיף 0.5 מ"ל של מדיום עם FBS 20% לריכוז סופי של 10% FBS.
- מתן 48 עד 72 שעות ו להכתים את התאים עם יודיד propidium (PI) וחלבון פלורסנט ירוק (GFP) על פי הוראות היצרן. הגן על התאים מפני אור ולהשתמש סדרן FACS למדוד את שיעורי GFP + / PI - תאים. מאז כתמי PI תאים מתים בלבד, שיעור זה הוא אומדן של יעילות transfection בתאים חיים.
- מיינו את אוכלוסיית תאים חיובית ביטוי של GFP (+ GFP) על ידי סדרן FACS כפי שתואר לעיל 10.
- השתמש Westeסופג rn החיסונית כפי שתואר לעיל 10 כדי לקבוע את רמות פקטור שעתוק של עניין לפני ואחרי הדבקה של תאים בעלי עניין (למשל, תאים CLL) עם shRNA המיועד.
3. לקבוע את רמת הביטוי של transcriptome MicroRNA בתאים transfected עם תמלול Factor-shRNA
- לבודד RNA באמצעות ערכה מסחרית על פי הפרוטוקול של היצרן.
- לייבל RNA להכליא אותו microRNA microarray 11.
- קבע את microRNA לידי ביטוי באופן דיפרנציאלי בתאים transfected עם שעתוק גורם shRNA או עם פקדים וקטור ריק 5.
- אמת את תוצאות microarray עבור מיקרו RNA לידי ביטוי ביותר דיפרנציאלי באמצעות בזמן אמת 5 PCR.
4. לקבוע את החפיפה בין ביואינפורמטיקה וגישת shRNA בתיאור transcriptome Dependent גורם שעתוק
- כדי Detסמור היחסי הצפוי וצופה גני microRNA כי נמל גורם שעתוק מחייבים אתרי היזמים שלהם והיו downregulated בתאי transfected-shRNA גורם שעתוק, בצע את הפעולות הבאות:
- השג את היחס בין הגנים כי הנמל גורם שעתוק של עניין microRNA האמרגן / הכולל שלהם מהרשימה שנוצרה 1.3. רשימה זו היא היחס הצפוי (למשל, גני microRNA עם STAT3 אתרי קישור / גני microRNA הכוללים נבדקו = 0.25).
- לקבוע את מספר גנים שהיו downregulated בתאי transfected שעתוק הגורם-shRNA מהרשימה שנוצרה 3.3 ויש לו גורם שעתוק של אתרי קישור עניין מהרשימה שנוצרה 1.3. מספר / זה סה"כ גנים downregulated הוא היחס הנצפה (למשל, הגנים microRNA עם STAT3 אתרי הקישור / המספר הכולל של מיקרו-רנ"א downregulated = 0.6).
- השתמש χ2 סטטיסטיקה להשוות את היחסים ציינו וצפויים שנוצרו לעיל לקבוע אם הרשימה של גנים שהיו downregulated בתאי transfected שעתוק הגורם-shRNA מועשרת עם גורם שעתוק מחייבים אתרי האמרגן שלהם.
תוצאות
STAT3 הוא גורם שעתוק אשר בדרך כלל גורם שעתוק של גנים שיש להם אפופטוטיים אנטי ואפקטים שגשוג 12. אם STAT3 להשפיע גם על transcriptome RNA ללא קידוד לפי שעה לא ברור. בכל תאי CLL STAT3 עובר פוספורילציה constitutively על סרין 707 שאריות 10,13. Phosphoserine STAT3 הסעות לגרעין, נקשר ל- DNA, ומפעיל גנים הידועים בכך שהם מופעלים על ידי טירוזין pSTAT3 בסוגי תאים אחרים 10. בגלל CLL מאופיין הסרת הפיקוח הגלובלי של הרשת microRNA 14, שיערנו כי נוכחות של סרין pSTAT3 משפיע על הביטוי של מיקרו RNA בתאים CLL.
כדי לבחון זאת היפותזת היזמים של מיקרו-רנ"א כי הנמל STAT3 אתרי הקישור שהיה צורך לזהותם. באמצעות חציית נתונים שנוצרו על ידי בר ואח '. 9 של אזורים עם modifi היסטון H2K4me3קטיון מאפיינות אתרי אמרגן, עם STAT3 מחייב אתרים שזוהו על ידי ניסוי שבב seq 15, אתרי קישור משוערים STAT3 זוהו. בגישה זו 160 מקדמים משוערים אותרו קרוב ל -25% מן הגנים microRNA שנבדקו (N = 200) עם ציוני מחייב הנעים בין 100 (הציון הנמוך ביותר) 1,000 (הציון הגבוה ביותר) (טבלת 1).
תאים בהמשך CLL היו transfected עם STAT3-shRNA או עם וקטור ריק ושימוש מערך microRNA וזיהה 63 מיקרו-רנ"א שהוסדרו למטה בעקבות transfection (איור 1) דבר המצביע על כך STAT3 מקדם את שעתוק של מיקרו RNA אלה. עבור 60% של 63 גני microRNA downregulated (n = 38) נתוני שבב seq אשרו STAT3 מחייב אמרגן משוער במעלה זרם של מיקום הגן, באופן משמעותי יותר מצפוי באקראי (p <0.0001). תשעה רנ"א שהיו למטה מוסדר לאחר transfection,דבר המצביע על כך STAT3 לווסת את הרמות של שעתוק שלה לרעה.
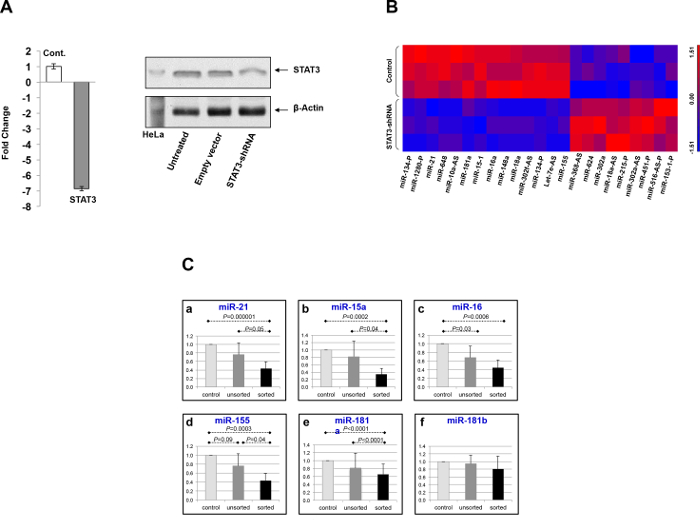
איור 1. Transfection של תאים CLL עם STAT3-shRNA מפחית את רמות הביטוי של חלבון STAT3 ו STAT3 mRNA. ת:. לאחר transfection של תאים CLL עם רמות STAT3-shRNA של STAT3 mRNA (פאנל משמאל, זוהה על ידי כמותי RT-PCR) ורמות של החלבון STAT3 (פאנל מימין, זוהה על ידי immunoblotting המערבי) ירד משמעותית ב: מערך microRNA של CLL תאים מתואר 23 מיקרו-רנ"א שביטויים שונה משמעותית בין תאי CLL טרנספקציה עם STAT3-shRNA ותאי CLL טרנספקציה עם וקטור ריק. ערך P של פחות מ 0.01 נחשב משמעותי מבחינה סטטיסטית C:. לכמת בהבעה המרושעת על ידי RT-PCR של 7 מיקרו-רנ"א שהיה ביטוי ההפרש לאחר הטיפול STAT3-shRNA באמצעותמערך microRNA. ברים מייצגים את שגיאת התקן של הממוצע. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
משלימה Coding קובץ 1. אנא לחץ כאן כדי להוריד את הקובץ.
| גן RNA Micro | כרומוזום | קואורדינטות התחלת יזם | קואורדינטות סוף היזם | חציון (טווח) ציון מחייב STAT3 * |
| miR-1205, miR-1206, miR-1207 | 8 q24.21 | 128961454 | 128962791 | 1000 (1000-1000) |
| miR-1537 | 1 q42.3 | 236045425 | 236047415 | 1000 (1000-1000) |
| miR-21 | 17 q23.1 | 57901872 | 57921277 | 1000 (112-1000) |
| miR-3124 | 1 q44 | 249115404 | 249123965 | 1000 (1000-1000) |
| miR-451 | 17q11.2 | 27222251 | 27224114 | 1000 (1000-1000) |
| miR-92b | 1 שאלה 22 | 155162340 | 155168439 | 1000 (1000-1000) |
| miR-3197 | 21 q22.2 | 42537544 | 42543023 | 943 (943-943) |
| miR-646 | 20 q13.33 | 58712550 | 58715320 | 789 (789-789) |
| miR-629 | 15 שאלה 23 | 70383751 | 70394586 | 773 (661-885) |
| miR-30e, miR-30C-1 | 1 p34.2 | 41173077 | 41177703 | 759 (759-759) |
| miR-3125 | 2 p24.3 | 12855381 | 12862915 | 756 (756-756) |
| miR-3145 | 6 q23.3 | 138776942 | 138779365 | 743 (487-1000) |
| miR-645 | 20 q13.13 | 49199911 | 49201187 | 743 (743-743) |
| miR-1256 | 1 p36.12 | 21346830 | 21350211 | 725 (725-725) |
| miR-619 | 12 q24.11 | 109248263 | 109253306 | 719 (719-719) |
| miR-181a-2, miR-181b-2 | 9 q33.3 | 127418928 | 127426139 | 710 (710-710) |
| miR-29a, miR-29b-1 | 7 q32.3 | 130583383 | 130597803 | 697 (482-1000) |
| miR-202 | 10 q26.3 | 135069499 | 135077337 | 696 (393-1000) |
| miR-3142, miR-146a | 5 q34 | 159890882 | 159899475 | 671 (671-671) |
| miR-548c | 12 q14.2 | 65000968 | 65011503 | 660 (660-660) |
| miR-630 | 15 q24.1 | 72764289 | 72769197 | 627 (255-1000) |
| miR-135b | 1 q32.1 | 205416952 | 205452990 | 622 (245-1000) |
| miR-29C, miR-29b-2 | 1 q32.2 | 207991044 | 208002382 | 608 (608-608) |
| miR-1825 | 20 q11.21 | 30791020 | 30798310 | 604 (209-1000) |
| miR-548h-1 | 14 q23.2 | 64578,834 | 64581657 | 587 (174-1000) |
| miR-612 | 11 q13.1 | 65183633 | 65198528 | 581 (157-1000) |
| miR-148b | 12 q13.13 | 54717640 | 54721204 | 578 (578-578) |
| miR-3174 | 15 q26.1 | 90543381 | 90549092 | 576 (152-1000) |
| let7a-3, let7b | 22 | 46480680 | 46481826 | 573 (146-1000) |
| miR-1255a | 4 שאלה 24 | 102263848 | 102272541 | 557 (557-557) |
טבלת 1. היזמים של משוערת microRNA עם אתרי קישור STAT3. הפרוטוקול מספק שיטה לזיהוי גורם שעתוק מחייב על יזמים משוערים של מייקרו RNA באמצעות כריית נתונים של נתונים שפורסמו. כדוגמא, אנו מציגים כאן שולחן המתאר את הכריכה של STAT3 יזמי microRNA משוערים. הציון המחייב ניתן סולם של 0 עד 1,000 מנתונים seq שבב הגנום כולו מתפרסמים במסגרת פרויקט ENCODE 15. היזמים מזוהים על ידי הנוכחות של חתימת H3K4me3 אפיגנטיים 9.
Discussion
המנגנון שבבסיס RNA פולימראז II- השעתוק התלוי של גני המקודדים חלבונים, נחקר בהרחבה. בעוד אלמנטים אלה מהווים רק 1% - 2% של הגנום האנושי, ראיות מפרויקט הקידוד מראים כי למעלה מ -80% של הגנום האנושי עשויים לעבור שעתוק 17 ומה מסדיר את התעתיק של רצפי דנ"א ללא קידוד נותר ברובה לא ידוע 6 .
מספר מחקרים, אשר הצביעו על כך Pol II הוא גם אחראי על שעתוק של גנים מסוימים שאינם המקודדים חלבונים כולל 6 מיקרו-רנ"א, הוביל אותנו לפתח אסטרטגיה המשלבים נתונים ממקורות זמינים בפומבי, אלגוריתמים חישוביים וגם במחקרים במבחנה לפענח את הפוטנציאל פונקציה של גורם שעתוק של עניין בוויסות שעתוק של מיקרו-רנ"א. האסטרטגיה הציעה בזאת כולל 2 שלבים קריטיים. ראשית אנו משתמשים בנתונים זמינים בפומבי לזהות b גורם שעתוקinding ב יזמי microRNA. לשם כך אנו משתמשים צ'יפ-seq נתונים שפרסמו קונסורציום הקידוד לזהות גורם שעתוק וחתימה אפיגנטיים מחייב שמאפיין אמרגן כסמן עקיף-יזמי microRNA. חציית הקואורדינטות הגנטיות ממערכות נתונים אלה מספקת הערכה גסה של כמה תכוף גורם שעתוק של עניין נקשר אמרגן microRNA. שנית באמצעות טכנולוגית shRNA להשתיק את הביטוי של גורם שעתוק וההכפפה לתאי מערך microRNA, אפשר לחקור את המשמעות התפקודית של גורם שעתוק על-transcriptome microRNA.
ההשתנות בביטוי microRNA מוסברות באופן חלקי בלבד על ידי רגולציה תלויה גורם שעתוק. השתנות stoichiometric וגורמים הסלולר או תאיים אחרים ממלאים תפקיד חשוב ואינם הדמיה של האלגוריתם המוצע. מגבלות אחרות כוללות את הפעולות הבאות: ניתוח האמרגן המבוסס על H3K4me3החתימה נעשתה על 939 מיקרו-רנ"א המבואר. מאז המיקומים הגנומי של מיקרו-רנ"א רבים יותר זוהו. עם זאת למיטב ידיעתנו רשימה מקיפה יותר המבוססת על מסד נתונים עדכני לא פורסם עדיין. של 939 גני microRNA, את H3K4me3 המאפיין באזור האמרגן זוהה 781 גני microRNA (83%). לפיכך, בעוד ניתוח זה מבוסס בבירור על בסיס נתונים שלמים היא לוכדת על אחוז ניכר של-transcriptome microRNA.
יתר על כן, חתימת אפיגנטיקה היא בחלקה מוטבעת ובחלקה תא ספציפי. לכן, generability של יזמים משוערים שהוגדרו על ידי סמנים אפיגנטיקה עשוי להיחקר. בגלל H3K4me3 נמשכת עצמאית של שעתוק 18 זה נחשב בדרך כלל כסמן של יזמים מוטבעים. האלגוריתם אנליטית אנו מציעים במסמך זה עשוי ולכן לפספס יזמי microRNA תא ספציפי אם היזמים הללו זוהו בכל cell- שונהסוּג. לבסוף, כל מסקנה צריכה להיבדק ואשרה באופן אמפירי. בעיקר, את הקשר בין רגולציה כלפי מטה של גורם שיעתוק (שימוש בגישה shRNA) וביטוי microRNA (מזוהה על ידי מערך microRNA) יכול רק להציע תפקיד תעתיק ישיר כי חייבת להיות מאושרת על ידי מבחני מקובל כגון immunoprecipitation הכרומטין (שבב) או ניידות electrophoretic assay משמרת (Emsa). שינויים בשיטה הציעה בזאת לכלול דרכים שונות של זיהוי אמרגן microRNA ודרכים שונות של דריסת הביטוי של גורם שעתוק, למשל, RNA התערבות קטן במקום shRNA. הרגולציה תעתיק של מיקרו RNA עשוי להיות שונה באופן מהותי עבור מיקרו RNA הנמצאים בתוך הגנים המקודדים חלבונים (מיקרו-רנ"א intragenic) ואלה לא (מיקרו-רנ"א-גניים). בגלל מיקרו-רנ"א intragenic משועתקים בדרך כלל בשילוב עם גנים המארח שלהם 19 האמרגן נמצא בדרך כלל מיד upstreaמ 'באתר תחילת השעתוק. אולם עבור מייקרו-רנ"א-גניים רבים באתר תחילת השעתוק הוא מבואר גרוע, וכלי חיזוי שאינם בשימוש נפוץ עבור גנים מקודדים חלבונים לבצע גרוע 20.
Disclosures
The authors declare no competing financial interests.
Acknowledgements
מחקר זה נתמך על ידי מענק מטעם קרן המחקר גלובל CLL. האוניברסיטה של מרכז טקסס MD Anderson Cancer נתמך בחלקו על ידי המכון הלאומי לבריאות בארה"ב באמצעות מענק תמיכה במרכז לחקר הסרטן (P30CA16672).
Materials
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies | 11668027 | |
| 0.45 µm syringe filter | Thermo Scientific (Nalgene) | 190-2545 | |
| Amicon ultracentrifugal filter device with threshold of 100 kDa | Merck Millipore | ||
| Polybrene | Merck Millipore | TR-1003-G | |
| TRIzol reagent | Life Tachnologies (Invitrogen) | 15596-026 | |
| 293 Cell line human | Sigma-Aldrich | 85120602 |
References
- Bentwich, I., et al. Identification of hundreds of conserved and nonconserved human microRNAs. Nat Genet. 37, 766-770 (2005).
- Hata, A. Functions of microRNAs in cardiovascular biology and disease. Annu Rev Physiol. 75, 69-93 (2013).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Piriyapongsa, J., Jordan, I. K., Conley, A. B., Ronan, T., Smalheiser, N. R. Transcription factor binding sites are highly enriched within microRNA precursor sequences. Biol Direct. 6, 61 (2011).
- Rozovski, U., et al. Signal transducer and activator of transcription (STAT)-3 regulates microRNA gene expression in chronic lymphocytic leukemia cells. Mol Cancer. 12, 50 (2013).
- Turner, M. J., Slack, F. J. Transcriptional control of microRNA expression in C. elegans: promoting better understanding. RNA Biol. 6, 49-53 (2009).
- Cui, Q., Yu, Z., Pan, Y., Purisima, E. O., Wang, E. MicroRNAs preferentially target the genes with high transcriptional regulation complexity. Biochem Biophys Res Commun. 352, 733-738 (2007).
- Yamakuchi, M., Lowenstein, C. J. MiR-34, SIRT1 and p53: the feedback loop. Cell Cycle. 8, 712-715 (2009).
- Baer, C., et al. Extensive promoter DNA hypermethylation and hypomethylation is associated with aberrant microRNA expression in chronic lymphocytic leukemia. Cancer Res. 72, 3775-3785 (2012).
- Hazan-Halevy, I., et al. STAT3 is constitutively phosphorylated on serine 727 residues, binds DNA, and activates transcription in CLL cells. Blood. 115, 2852-2863 (2010).
- Melo, S. A., et al. A TARBP2 mutation in human cancer impairs microRNA processing and DICER1 function. Nat Genet. 41, 365-370 (2009).
- Akira, S. Functional roles of STAT family proteins: lessons from knockout mice. Stem Cells. 17, 138-146 (1999).
- Frank, D. A., Mahajan, S., Ritz, J. B lymphocytes from patients with chronic lymphocytic leukemia contain signal transducer and activator of transcription (STAT) 1 and STAT3 constitutively phosphorylated on serine residues. J Clin Invest. 100, 3140-3148 (1997).
- Calin, G. A., et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 101, 11755-11760 (2004).
- Consortium, E. P. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS biology. 9, e1001046 (2011).
- Miranda, K. C., et al. A pattern-based method for the identification of MicroRNA binding sites and their corresponding heteroduplexes. Cell. 126, 1203-1217 (2006).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57-74 (2012).
- Lau, J. C., Hanel, M. L., Wevrick, R. Tissue-specific and imprinted epigenetic modifications of the human NDN gene. Nucl Acids Res. 32, 3376-3382 (2004).
- Corcoran, D. L., et al. Features of mammalian microRNA promoters emerge from polymerase II chromatin immunoprecipitation data. PLoS One. 4, e5279 (2009).
- Bhattacharyya, M., Feuerbach, L., Bhadra, T., Lengauer, T., Bandyopadhyay, S. MicroRNA transcription start site prediction with multi-objective feature selection. Stat Appl Genet Mol Biol. 11, (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved