Method Article
Descrivendo un regolamento Dipendente Fattore di trascrizione del MicroRNA trascrittoma
In questo articolo
Riepilogo
Herein we propose a strategy to study the effect of a transcription factor of interest on the microRNA transcriptome using publically available data, computational resources and high throughput data from microRNA arrays after transfecting cells with small hairpin (sh)RNA targeting a transcription factor of interest.
Abstract
Mentre la regolazione della trascrizione dei geni codificanti proteine è stato ampiamente studiato, poco si sa su come fattori di trascrizione sono coinvolti nella trascrizione di RNA non codificanti, in particolare dei microRNA. Qui, proponiamo una strategia per studiare il ruolo potenziale del fattore di trascrizione nella regolazione della trascrizione dei microRNA utilizzando i dati pubblicamente disponibili, risorse di calcolo e di dati ad alta produttività. Usiamo il H3K4me3 firma epigenetica per identificare i promotori di microRNA e immunoprecipitazione della cromatina (chip) di dati -sequencing dal progetto ENCODE per identificare i promotori microRNA che si arricchiscono con siti di legame fattore di trascrizione. Transfettando cellule di interesse con shRNA mira un fattore di trascrizione di interesse e sottoporre le cellule a matrice microRNA, si studia l'effetto di questo fattore di trascrizione sul trascrittoma microRNA. A titolo di esempio illustrativo usiamo il nostro studio sull'effetto di STAT3 sul trascrittoma microRNA di mente croniciLeucemia mphocytic (CLL) cellule.
Introduzione
I microRNA sono endogeni piccoli RNA non codificanti normativi che di solito funzionano come regolatori negativi di espressione dell'mRNA a livello post-trascrizionale. Circa 1000 non codificante 20 a 25 nucleotidi lunghe microRNA si trovano nel genoma umano 1,2. I microRNA regolano l'espressione genica attraverso l'accoppiamento di base canonica tra la sequenza seme del microRNA e il suo complementare sequenza partita seme, che è comunemente trova in regione 3 'non tradotta (UTR) del mRNA bersaglio. Collettivamente, microRNA regolano oltre il 30% dei geni codificanti proteine 3, ma solo poco si sa circa la trascrizione dal DNA dei microRNA. È stato suggerito che la regolazione dei microRNA trascrizione è simile a quella di mRNA 4,5. In particolare, simile alla sua attività di promozione trascrizione dei geni codificanti proteine, fattori di trascrizione sono pensati per attivare la trascrizione dei microRNA 6. Fattore di trascrizione-microRNA gioco è stato segnalato come un fattore di modulazione dell'espressione genica 7, e può anche formare feed-back e feed-forward loop. Ad esempio, Yamakuchi et al. Riportato un anello di retroazione in cui p53 induce l'espressione di microRNA34a, che a sua volta inibisce traduzione della SIRT p53 repressore e aumentando così l'attività p53 8.
Considerando che sono stati riportati esempi specifici di fattore di trascrizione espressione dei microRNA dipendente, un metodo accettato che fornisce informazioni su come un fattore di trascrizione di interesse regola l'espressione del microRNA-trascrittoma è carente. Lo scopo del protocollo suggerito qui è quello di fornire una descrizione approfondita della trascrizione Regolazione fattore-dipendente del microRNA-transcriptome. Combinando i dati pubblicamente disponibili, strumenti di bioinformatica e utilizzando la tecnologia microarray, i ricercatori che seguono questo algoritmo sarebbe in grado di catturare su scala genomica come qualsiasifattore di trascrizione in qualsiasi tipo di cellula di interesse regola l'espressione del microRNA-trascrittoma e per esplorare un contributo putativo del fattore di trascrizione-mRNA nella regolazione dell'espressione di microRNA.
Protocollo
1. Identificare fattore di trascrizione siti di legame nel promotore di geni microRNA Utilizzando Approach Data Mining
- Utilizzare il browser genoma Università della California a Santa Cruz (UCSC) per estrarre cromatina immunoprecipitazione dati (chip) di sequenziamento generati come parte della Encyclopedia of DNA elemento del progetto (ENCODE).
- Aprire il browser tavolo nel browser genoma UCSC.
- Utilizzare le seguenti specifiche per estrarre la tabella: Clade: (mammiferi), genoma: (umano), Montaggio: (Feb2009 (GRch37 / hg18)), gruppo: (regolamento), brano (TxnFactorChIP), tabella: (weEncoderesTFbsCo7steredv3), regione : (Genoma), il formato di output (tutti i geni da tabella selezionata).
- Salvare l'output da 1.1.2 come un file .txt e in un foglio elettronico.
- Ordinamento e filtro per il fattore di trascrizione di interesse (ad esempio, STAT2).
- Utilizzare l'elenco dei promotori microRNA basate su H3K4me3 epigenetica firma disponibile presso Baeer et al. 9 . Copia questo elenco in un file .txt.
- Per abbinare secondo coordinate sul genoma umano, mappare i dati da 1.1 e 1.2 (ad esempio, vincolante STAT3 su promotori microRNA putativi) e determinare il legame mediana affinità con il codice scritto in C diesis come indicato nel supplementare di file 1 codifica.
2. Utilizzare shRNA di down-regolare l'espressione di un fattore di trascrizione di interesse
- Piastra 1,5 x 10 6 cellule provenienti da 293 linea di cellule renali di embrioni umani a 10 cm piastra a circa il 50% di confluenza (DMEM con 10% FBS).
- cellule trasfezione da 293 linea di cellule renali embrionali umane con 5 mg di verde lentivirus proteina fluorescente (GFP) contenente shRNA diretti al fattore di trascrizione di interesse e con 5 mg di vettori di confezionamento con il reagente di trasfezione per cellule aderenti secondo il protocollo del produttore.
- Come controllo, trasfezione le cellule a partire da 293 linea di cellule renali embrionali umane constrapazzate shRNA ei vettori imballaggio secondo il protocollo del produttore.
- Mantenere mix trasfezione su cellule per 16 ore (a 37 ° C, CO 2 incubatore), poi cambiare supporto a 10 ml fresca 10% DMEM media con 10% FBS.
- Attendere 48 ore dopo la trasfezione, centrifugare la coltura cellulare (300 xg, 5 min) e raccogliere il surnatante infettive. Filtrare il surnatante con un filtro a siringa da 0,45 micron (25-mm tensioattivo libero di cellulosa membrana in acetato) per rimuovere eventuali cellule galleggianti.
- Concentrare il surnatante e raccogliere il lentivirus utilizzando un dispositivo di filtro ultracentrifugo con soglia di 100 kDa. Spin a 950 xg per 30 - 60 min fino a quando il volume è stato concentrato a meno di 250 microlitri. Conservare il virus concentrato a -80 ° C.
- Trasfezione delle cellule con il lentivirus. Rimuovere lentivirus congelate da -80 ° C freezer e scongelare a temperatura ambiente. Trasferire 100 ml di surnatante virale per un fresco 1,5 ml provetta da microcentrifuga.
- Bring il volume nel tubo di 1 ml con ridotta mezzo siero. Aggiungere hexadimethrine bromuro di 1 ml di sospensione di virus per la concentrazione finale di 10 ng / ml, mescolare delicatamente e lasciare riposare l'impasto per 5 min.
- Centrifugare 5 x 10 6 cellule per la trasduzione e delicatamente risospendere il pellet cellulare in 0,5 ml di supporti contenenti virus. Lasciare le cellule rimangono nell'incubatrice: 4 - 24 ore, quindi aggiungere 0,5 ml di terreno con 20% FBS ad una concentrazione finale del 10% FBS.
- Attendere 48 a 72 ore e macchiare le cellule con ioduro di propidio (PI) e la proteina fluorescente verde (GFP) in base alle istruzioni del produttore. Proteggere le cellule dalla luce e usare un sorter FACS per misurare i tassi di GFP + / PI - cellule. Dal momento che le macchie PI solo cellule morte, questo tasso è una stima di efficienza di trasfezione nelle cellule viventi.
- Ordina la popolazione di cellule positive per l'espressione della GFP (GFP +) da FACS sorter come descritto in precedenza 10.
- Usa Western blotting immunitario come precedentemente descritto 10 per determinare i livelli di un fattore di trascrizione di interesse prima e dopo infettare le cellule di interesse (per esempio, cellule CLL) con designato shRNA.
3. Determinare il livello di espressione dei microRNA trascrittoma in cellule trasfettate con Fattore di trascrizione-shRNA
- Isolare RNA utilizzando un kit commerciale secondo il protocollo del produttore.
- Etichettare il RNA e ibridare al microRNA microarray 11.
- Determinare il microRNA differenzialmente espressi in cellule trasfettate con fattore di trascrizione-shRNA o con comandi vuoto vettore 5.
- Convalidare i risultati di microarray per i microRNA più differenzialmente espressi nel formato Real-time PCR 5.
4. Determinare la sovrapposizione tra la bioinformatica e l'approccio shRNA nel descrivere il trascrittoma Dependent fattore di trascrizione
- per detErmellino i rapporti attesi e osservati di geni microRNA che ospitano fattore di trascrizione siti nel loro promotori vincolanti e sono stati inibiti nella trascrizione fattore-shRNA cellule trasfettate, effettuare le seguenti operazioni:
- Ottenere il rapporto di geni che ospitano il fattore di trascrizione di interesse per il loro promoter / microRNA totale dalla lista generata in 1.3. Questo elenco è il rapporto atteso (ad esempio, i geni microRNA con siti di legame STAT3 / totale geni microRNA testati = 0.25).
- Determinare il numero di geni che sono stati diminuito l'in cellule trasfettate fattore di trascrizione-shRNA dalla lista generata in 3.3 ed è fattore di trascrizione del legame di interesse siti dalla lista generata in 1.3. Questo / Numero totale di geni ha diminuito l'numero è il rapporto osservato (ad esempio, i geni microRNA con STAT3 siti di legame / numero totale dei microRNA ha diminuito l'= 0.6).
- Utilizzare χ2 statistiche per confrontare i rapporti osservati e attesi che sono stati generati al di sopra e determinare se l'elenco dei geni che sono stati downregulated in cellule trasfettate fattore di trascrizione-shRNA sono arricchiti con siti di legame fattore di trascrizione nel loro promotore.
Risultati
STAT3 è un fattore di trascrizione che induce tipicamente la trascrizione di geni che sono anti apoptotico ed effetti proliferativi 12. Sia STAT3 riguardano anche la non codificante RNA trascrittoma è attualmente sconosciuto. In tutte le cellule CLL STAT3 è costitutivamente fosforilata in serina 707 residui 10,13. Fosfoserina STAT3 navette per il nucleo, si lega al DNA, e attiva i geni noti per essere attivato da tirosina pSTAT3 in altri tipi di cellule 10. Poiché CLL è caratterizzato dalla deregolamentazione globale della rete microRNA 14, abbiamo ipotizzato che la presenza di serina pSTAT3 influenza l'espressione dei microRNA in cellule CLL.
Per verificare questa ipotesi promotori di microRNA che ospitano STAT3 siti di legame dovevano essere identificati. Incrociando i dati generati da Baer et al. 9 delle regioni con l'istone modifi H2K4me3cazione che caratterizzano i siti promotore, con STAT3 di legame siti individuati dagli esperimenti ChIP-ss 15, sono stati identificati putativi siti di legame STAT3. Usando questo approccio a 160 promotori putativi sono stati rilevati in quasi il 25% dei geni microRNA esaminati (N = 200) con punteggi vincolanti che vanno da 100 (il punteggio più basso) a 1.000 (il punteggio più alto) (Tabella 1).
Successivamente le cellule sono state trasfettate con CLL STAT3-shRNA o con un vettore vuoto e con un array microRNA ed identificati 63 microRNAs che sono state regolate fino a seguito della trasfezione (Figura 1) suggerendo che STAT3 promuove la trascrizione di questi microRNA. Per il 60% dei 63 geni microRNA ha diminuito l'(n = 38) dei dati ChIP-Seq confermati STAT3 vincolante in un putative promotore a monte del luogo gene, un numero significativamente maggiore di quanto previsto dal caso (p <0,0001). Nove microRNA che sono stati down-regolato dopo la trasfezione,suggerendo che STAT3 regolano negativamente i livelli di trascrizione.
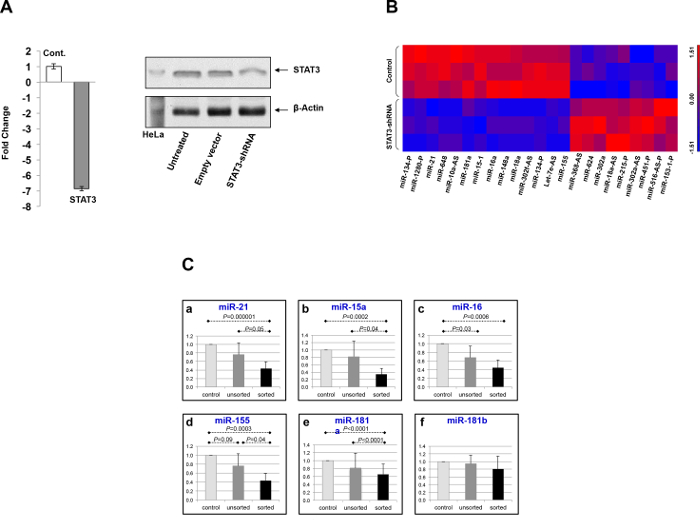
Figura 1. Trasfezione di cellule CLL con STAT3-shRNA riduce i livelli di espressione di STAT3 proteine e STAT3 mRNA. A:. Dopo la trasfezione di cellule CLL con livelli di STAT3-shRNA di STAT3 mRNA (pannello sinistro, rilevato da RT-PCR quantitativa) e livelli della proteina STAT3 (pannello di destra, rilevati da immunoblotting occidentale) significativamente diminuita B: microRNA array di CLL cellule raffigurati 23 microRNA cui espressione differivano significativamente tra le cellule CLL trasfettate con STAT3-shRNA e cellule CLL trasfettate con un vettore vuoto. Valore di p inferiore a 0,01 è stato considerato statisticamente significativo C:. L'espressione media quantificato mediante RT-PCR di 7 microRNA che avevano espressione differenziale dopo il trattamento STAT3-shRNA utilizzando ilarray di microRNA. Barre rappresentano l'errore standard della media. Cliccate qui per vedere una versione più grande di questa figura.
Supplemental file di codifica 1. Clicca qui per scaricare il file.
| Gene Micro RNA | Cromosoma | Coordinate di inizio Promotore | Coordinate finali Promotore | Mediana (range) STAT3 punteggio vincolante * |
| miR-1205, miR-1206, miR-1207 | 8 q24.21 | 128961454 | 128962791 | 1000 (1000-1000) |
| miR-1537 | 1 q42.3 | 236045425 | 236047415 | 1000 (1000-1000) |
| miR-21 | 17 q23.1 | 57901872 | 57921277 | 1000 (112-1000) |
| miR-3124 | 1 Q44 | 249115404 | 249123965 | 1000 (1000-1000) |
| miR-451 | 17q11.2 | 27222251 | 27224114 | 1000 (1000-1000) |
| miR-92b | 1 Q22 | 155162340 | 155168439 | 1000 (1000-1000) |
| miR-3197 | 21 q22.2 | 42537544 | 42543023 | 943 (943-943) |
| miR-646 | 20 q13.33 | 58712550 | 58715320 | 789 (789-789) |
| miR-629 | 15 Q23 | 70383751 | 70394586 | 773 (661-885) |
| miR-30e, miR-30c-1 | 1 p34.2 | 41173077 | 41177703 | 759 (759-759) |
| miR-3125 | 2 p24.3 | 12855381 | 12862915 | 756 (756-756) |
| miR-3145 | 6 q23.3 | 138776942 | 138779365 | 743 (487-1000) |
| miR-645 | 20 q13.13 | 49199911 | 49201187 | 743 (743-743) |
| miR-1256 | 1 p36.12 | 21346830 | 21350211 | 725 (725-725) |
| miR-619 | 12 q24.11 | 109248263 | 109253306 | 719 (719-719) |
| miR-181 A-2, miR-181b-2 | 9 q33.3 | 127418928 | 127426139 | 710 (710-710) |
| miR-29a, miR-29b-1 | 7 q32.3 | 130583383 | 130597803 | 697 (482-1000) |
| miR-202 | 10 q26.3 | 135069499 | 135077337 | 696 (393-1000) |
| miR-3142, miR-146a | 5 Q34 | 159890882 | 159899475 | 671 (671-671) |
| miR-548C | 12 q14.2 | 65000968 | 65011503 | 660 (660-660) |
| miR-630 | 15 q24.1 | 72764289 | 72769197 | 627 (255-1000) |
| miR-135b | 1 q32.1 | 205416952 | 205452990 | 622 (245-1000) |
| miR-29c, miR-29b-2 | 1 q32.2 | 207991044 | 208002382 | 608 (608-608) |
| miR-1825 | 20 q11.21 | 30791020 | 30798310 | 604 (209-1000) |
| miR-548h-1 | 14 q23.2 | 64578.834 | 64581657 | 587 (174-1000) |
| miR-612 | 11 q13.1 | 65183633 | 65198528 | 581 (157-1000) |
| miR-148b | 12 q13.13 | 54717640 | 54721204 | 578 (578-578) |
| miR-3174 | 15 q26.1 | 90543381 | 90549092 | 576 (152-1000) |
| let7a-3, let7b | 22 | 46480680 | 46481826 | 573 (146-1000) |
| miR-1255a | 4 Q24 | 102263848 | 102272541 | 557 (557-557) |
Tabella 1. I promotori di putativo microRNA con siti di legame STAT3. Il protocollo prevede un metodo per identificare fattore di trascrizione vincolante per i promotori putativi di microRNA che utilizzano il data mining dei dati pubblicati. A titolo di esempio, vi presentiamo qui una tabella che descrive il legame di STAT3 di promotori microRNA putativi. Il punteggio di legame è dato ad una scala da 0 a 1.000 dal genoma chip di dati ss integrali pubblicati come parte del progetto ENCODE 15. I promotori sono identificati dalla presenza del H3K4me3 firma epigenetica 9.
Discussione
Il meccanismo alla base della RNA polimerasi II trascrizione dipendente dei geni codificanti proteine è stata ampiamente studiata. Mentre questi elementi costituiscono solo l'1% - 2% del genoma umano, la prova del progetto ENCODE suggeriscono che oltre l'80% del genoma umano può subire trascrizione 17 e quello che regola la trascrizione degli elementi di DNA non codificanti rimane in gran parte sconosciuta 6 .
Diversi studi, che ha indicato che Pol II è anche responsabile per la trascrizione di alcuni geni non codificanti proteine, tra cui microRNA 6, ci ha portato a sviluppare una strategia che combina i dati da fonti pubblicamente disponibili, algoritmi di calcolo e in vitro di decifrare il potenziale funzione di un fattore di trascrizione di interesse nella regolazione della trascrizione dei microRNA. La strategia suggerita qui comprende 2 passaggi critici. In primo luogo usiamo i dati pubblicamente disponibili per identificare i fattori di trascrizione Binding in promotori di microRNA. A tal fine utilizziamo i dati Chip-Seq pubblicati dal consorzio ENCODE per identificare vincolante fattore di trascrizione e la firma epigenetica che caratterizza promotore come un indicatore indiretto di microRNA-promotori. Attraversando le coordinate genetiche da questi set di dati fornisce una stima lorda di quanto frequente un fattore di trascrizione di interesse si lega al microRNA-promotore. In secondo luogo, utilizzando la tecnologia shRNA mettere a tacere l'espressione di un fattore di trascrizione e sottoponendo le cellule di microRNA-array, è possibile esplorare il significato funzionale di un fattore di trascrizione sul microRNA-trascrittoma.
La variabilità di espressione dei microRNA è solo parzialmente spiegata dal fattore di trascrizione regolazione dipendente. variabilità stechiometrico e altri fattori cellulari o extracellulari svolgono un ruolo importante e non sono simulate nella algoritmo proposto. Altre limitazioni sono i seguenti: L'analisi promoter basata sulla H3K4me3firma è stato fatto su 939 microRNA annotati. Da allora sono state individuate le posizioni genomiche di molte altre microRNA. Tuttavia, per quanto a nostra conoscenza una lista più completa che si basa su un database aggiornato non è stato ancora pubblicato. Dei 939 geni microRNA, la H3K4me3 che caratterizza la regione del promotore è stata identificata in 781 geni microRNA (83%). Quindi, mentre questa analisi è chiaramente basato su un insieme di dati incompleta cattura una frazione significativa del microRNA-transcriptome.
Inoltre, epigenetica firma è in parte impressa e in parte-specifica delle cellule. Pertanto, la generability di promotori putativi che sono stati definiti da epigenetica marcatori può essere messa in discussione. Perché H3K4me3 persiste indipendente trascrizione 18 è generalmente considerato un marker di promotori impresso. L'algoritmo di analisi che proponiamo qui può quindi mancare promotori microRNA specifico per cellulari se questi promotori sono stati identificati in un cellulo diversoDigitare. Infine, ogni conclusione deve essere testato e confermato empiricamente. In particolare, l'associazione tra la regolamentazione verso il basso di un fattore di trascrizione (utilizzando l'approccio shRNA) e l'espressione di microRNA (identificato da array di microRNA) può solo suggerire un ruolo trascrizionale diretta che dovrebbe essere confermata mediante saggi accettabili, come immunoprecipitazione della cromatina (chip) o mobilità elettroforetica saggio spostamento (EMSA). Le modifiche al metodo suggerito qui sono diversi modi di identificare microRNA promotore e diversi modi di abbattere l'espressione di un fattore di trascrizione, per esempio, piccoli RNA interferenti anziché shRNA. La regolazione della trascrizione dei microRNA può essere sostanzialmente diverso per microRNA che risiedono all'interno di geni codificanti proteine (microRNA intragenica) e quelli che non lo fanno (microRNA intergenic). Perché microRNA intragenica sono comunemente trascritte in collaborazione con i loro geni ospitanti 19 il promotore di solito si trova immediatamente upstream il sito di inizio della trascrizione. Tuttavia per molti microRNA intergenic il sito di inizio della trascrizione è poco annotata, e strumenti di previsione che vengono comunemente utilizzati per geni che codificano proteine scarso rendimento 20.
Divulgazioni
The authors declare no competing financial interests.
Riconoscimenti
Questo studio è stato sostenuto da una sovvenzione della Fondazione Global Research LLC. L'Università del Texas MD Anderson Cancer Center è supportato in parte dal National Institutes of Health attraverso un Cancer Center Support Grant (P30CA16672).
Materiali
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies | 11668027 | |
| 0.45 µm syringe filter | Thermo Scientific (Nalgene) | 190-2545 | |
| Amicon ultracentrifugal filter device with threshold of 100 kDa | Merck Millipore | ||
| Polybrene | Merck Millipore | TR-1003-G | |
| TRIzol reagent | Life Tachnologies (Invitrogen) | 15596-026 | |
| 293 Cell line human | Sigma-Aldrich | 85120602 |
Riferimenti
- Bentwich, I., et al. Identification of hundreds of conserved and nonconserved human microRNAs. Nat Genet. 37, 766-770 (2005).
- Hata, A. Functions of microRNAs in cardiovascular biology and disease. Annu Rev Physiol. 75, 69-93 (2013).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Piriyapongsa, J., Jordan, I. K., Conley, A. B., Ronan, T., Smalheiser, N. R. Transcription factor binding sites are highly enriched within microRNA precursor sequences. Biol Direct. 6, 61 (2011).
- Rozovski, U., et al. Signal transducer and activator of transcription (STAT)-3 regulates microRNA gene expression in chronic lymphocytic leukemia cells. Mol Cancer. 12, 50 (2013).
- Turner, M. J., Slack, F. J. Transcriptional control of microRNA expression in C. elegans: promoting better understanding. RNA Biol. 6, 49-53 (2009).
- Cui, Q., Yu, Z., Pan, Y., Purisima, E. O., Wang, E. MicroRNAs preferentially target the genes with high transcriptional regulation complexity. Biochem Biophys Res Commun. 352, 733-738 (2007).
- Yamakuchi, M., Lowenstein, C. J. MiR-34, SIRT1 and p53: the feedback loop. Cell Cycle. 8, 712-715 (2009).
- Baer, C., et al. Extensive promoter DNA hypermethylation and hypomethylation is associated with aberrant microRNA expression in chronic lymphocytic leukemia. Cancer Res. 72, 3775-3785 (2012).
- Hazan-Halevy, I., et al. STAT3 is constitutively phosphorylated on serine 727 residues, binds DNA, and activates transcription in CLL cells. Blood. 115, 2852-2863 (2010).
- Melo, S. A., et al. A TARBP2 mutation in human cancer impairs microRNA processing and DICER1 function. Nat Genet. 41, 365-370 (2009).
- Akira, S. Functional roles of STAT family proteins: lessons from knockout mice. Stem Cells. 17, 138-146 (1999).
- Frank, D. A., Mahajan, S., Ritz, J. B lymphocytes from patients with chronic lymphocytic leukemia contain signal transducer and activator of transcription (STAT) 1 and STAT3 constitutively phosphorylated on serine residues. J Clin Invest. 100, 3140-3148 (1997).
- Calin, G. A., et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 101, 11755-11760 (2004).
- Consortium, E. P. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS biology. 9, e1001046 (2011).
- Miranda, K. C., et al. A pattern-based method for the identification of MicroRNA binding sites and their corresponding heteroduplexes. Cell. 126, 1203-1217 (2006).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57-74 (2012).
- Lau, J. C., Hanel, M. L., Wevrick, R. Tissue-specific and imprinted epigenetic modifications of the human NDN gene. Nucl Acids Res. 32, 3376-3382 (2004).
- Corcoran, D. L., et al. Features of mammalian microRNA promoters emerge from polymerase II chromatin immunoprecipitation data. PLoS One. 4, e5279 (2009).
- Bhattacharyya, M., Feuerbach, L., Bhadra, T., Lengauer, T., Bandyopadhyay, S. MicroRNA transcription start site prediction with multi-objective feature selection. Stat Appl Genet Mol Biol. 11, (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon