Method Article
Descripción de una regulación dependiente de factor de transcripción del transcriptoma MicroARN
En este artículo
Resumen
Herein we propose a strategy to study the effect of a transcription factor of interest on the microRNA transcriptome using publically available data, computational resources and high throughput data from microRNA arrays after transfecting cells with small hairpin (sh)RNA targeting a transcription factor of interest.
Resumen
Mientras que la regulación de la transcripción de genes que codifican proteínas se estudió extensamente, poco se conoce sobre cómo los factores de transcripción están implicados en la transcripción de los ARN no codificantes, específicamente de microARN. A continuación, se propone una estrategia para estudiar el posible papel del factor de transcripción en la regulación de la transcripción de microRNAs utilizando los datos públicamente disponibles, los recursos computacionales y de datos de alto rendimiento. Utilizamos la firma epigenética H3K4me3 para identificar promotores de microARN y la cromatina immunoprecipitation (CHIP) -sequencing datos del proyecto ENCODE para identificar promotores de microARN que se enriquecen con los sitios de unión del factor de transcripción. Mediante la transfección de células de interés con shRNA dirigidas a un factor de transcripción de interés y someter las células a la matriz microARN, se estudia el efecto de este factor de transcripción en el transcriptoma de microARN. A modo de ejemplo ilustrativo usamos nuestro estudio sobre el efecto de STAT3 en el transcriptoma de microARN Ly crónicaleucemia mphocytic (LLC) de células.
Introducción
Los microARN son ARN reguladoras no codificantes pequeñas endógenos que normalmente funcionan como reguladores negativos de la expresión de ARNm a nivel postranscripcional. Aproximadamente 1000 no codificante de 20 a 25 nucleótidos microRNAs largos se encuentran en el genoma humano 1,2. MicroARNs regulan la expresión génica a través de apareamiento de bases entre la secuencia canónica semilla de la microRNA y su secuencia complementaria partido de semillas, que comúnmente se encuentra en la región no traducida 3 '(UTR) de los ARNm diana. Colectivamente, microRNAs regulan más de 30% de los genes de codificación de proteínas 3, pero sólo se conoce poco acerca de la transcripción a partir del ADN de microARN. Se ha sugerido que la regulación de la transcripción microRNA es similar a la de mRNA 4,5. En particular, similar a su actividad en la promoción de la transcripción de genes de codificación de proteínas, se cree que los factores de transcripción para activar la transcripción de microRNAs 6. Factor de Transcripción-microRNA interacción ha sido reportado como un factor modulador de la expresión génica 7, y también pueden formar feed-back y bucles de alimentación hacia adelante. Por ejemplo, Yamakuchi et al. Informó de un bucle de retroalimentación en el que p53 induce la expresión de microRNA34a, que a su vez inhibe la traducción de la SIRT p53 represor y aumentando de este modo la actividad de p53 8.
Considerando que se han reportado ejemplos específicos de expresión dependiente de factor de transcripción de los microARN, un método aceptado que proporciona información sobre cómo un factor de transcripción de interés regula la expresión del microARN-transcriptoma es insuficiente. El propósito del protocolo sugerido en este documento es proporcionar una descripción en profundidad de la transcripción de la regulación dependiente del factor de la microRNA-transcriptoma. Al combinar los datos públicamente disponibles, herramientas bioinformáticas y el uso de la tecnología de microarrays, los investigadores que siguen este algoritmo sería capaz de capturar en una escala genómica cómo cualquierfactor de transcripción en cualquier tipo de célula de interés regula la expresión de la microRNA-transcriptoma y para explorar una contribución putativa del factor de transcripción-mRNA en la regulación de la expresión de microARN.
Protocolo
1. Identificar factor de transcripción sitios de unión en el promotor de los genes de microARN Usando enfoque de minería de datos
- Utilice el genoma navegador de la Universidad de California en Santa Cruz (UCSC) para extraer los datos de la cromatina immunoprecipitation (CHIP) de secuenciación generados como parte del proyecto Enciclopedia de elementos de ADN (ENCODE).
- Abra el explorador de tablas en la UCSC genoma navegador.
- Utilice las siguientes especificaciones para extraer la tabla: Clade: (mamíferos), genoma: (Ser humano), Asamblea: (Feb2009 (GRch37 / hg18)), grupo: (regulación), pista (TxnFactorChIP), tabla: (weEncoderesTFbsCo7steredv3), región : (Genoma), formato de salida (todos los genes de la tabla seleccionada).
- Guardar la salida de 1.1.2 como un archivo .txt y en una hoja de cálculo.
- Ordenar y filtrar para el factor de transcripción de interés (por ejemplo, STAT2).
- Utilice la lista de promotores de microARN basado en H3K4me3 epigenética firma disponible en Baeer et al. 9 . Copia esta lista en un archivo .txt.
- Para hacer coincidir de acuerdo con coordenadas en el genoma humano, asignar los datos a partir de 1,1 y 1,2 (por ejemplo, STAT3 de unión en los promotores de microARN putativo) y determinar la afinidad de unión mediana utilizando el código escrito en C agudo como se indica en suplementario archivo 1 de codificación.
2. Uso shRNA para regular a la baja la expresión de un factor de transcripción de interés
- Plate 1,5 x 10 6 células de la línea celular 293 de riñón embrionario humano en 10 cm de placas a aproximadamente 50% de confluencia (DMEM con 10% FBS).
- transfectar células de la línea celular 293 de riñón embrionario humano con 5 g de lentivirus proteína verde fluorescente (GFP) que contiene shRNA dirigidos al factor de transcripción de interés y con 5 g de vectores de empaquetamiento usando el reactivo de transfección de células adherentes de acuerdo con el protocolo del fabricante.
- Como control, transfectar las células de la línea celular 293 de riñón embrionario humano conrevueltos shRNA y los vectores de envasado de acuerdo con el protocolo del fabricante.
- Mantenga mezcla de transfección en las células durante 16 horas (a 37 ° C, incubadora de CO 2), a continuación, cambiar el medio a 10 ml fresco 10% de medio DMEM con 10% de FBS.
- Espere 48 horas después de la transfección, se centrifuga el cultivo celular (300 xg, 5 min) y recoger el sobrenadante infeccioso. Se filtra el sobrenadante a través de un filtro de jeringa de 0,45 m (25 mm tensioactivo de celulosa libre de membrana de acetato) para eliminar cualquier célula flotante.
- Se concentra el sobrenadante y recoger el lentivirus utilizando un dispositivo de filtro de ultracentrifugación con umbral de 100 kDa. Giran a 950 xg durante 30 - 60 min hasta que el volumen ha sido concentrado a menos de 250 l. Almacenar el virus concentrado a -80 ° C.
- Transfectar las células con el lentivirus. Eliminar lentivirus congelado de -80 ° C congelador y descongelar a temperatura ambiente. Transferir 100 l de sobrenadante viral a un nuevo tubo de microcentrífuga de 1,5 ml.
- Bring hasta el volumen en el tubo a 1 ml con medio con suero reducido. Añadir bromuro de hexadimetrina a 1 ml de suspensión de virus para la concentración final de 10 ng / ml, mezclar suavemente y se deja reposar la mezcla durante 5 min.
- Centrifugar 5 x 10 6 células por cada transducción y suavemente volver a suspender el sedimento celular en 0,5 ml de medio que contiene virus. Deje que las células permanecen en la incubadora 4 - 24 horas, a continuación, añadir 0,5 ml de medio con FBS al 20% a una concentración final de 10% de SFB.
- Espere 48 a 72 hr y teñir las células con yoduro de propidio (PI) y la proteína fluorescente verde (GFP) de acuerdo con las instrucciones del fabricante. Proteger a las células de la luz y utilizar un clasificador FACS para medir las tasas de GFP + / PI - células. Dado que las manchas de IP única célula muerta, esta tasa es una estimación de la eficacia de transfección en células vivas.
- Clasificar la población de células positivas para la expresión de GFP (GFP +) por FACS clasificador como se describió anteriormente 10.
- uso Westeblotting inmune rn como se describió previamente 10 para determinar los niveles de un factor de transcripción de interés antes y después de la infección de las células de interés (por ejemplo, células LLC) con shRNA designado.
3. Determinar el nivel de expresión de microARN transcriptoma en células transfectadas con factor de transcripción-shRNA
- Aislar el ARN utilizando un kit comercial de acuerdo con el protocolo del fabricante.
- Etiquetar el ARN y se hibridan a microARN microarrays 11.
- Determinar el microRNA expresado diferencialmente en las células transfectadas con factor de transcripción-shRNA o con los controles de vector vacío 5.
- Validar los resultados de microarrays para los microRNAs más expresados diferencialmente utilizando PCR en tiempo real 5.
4. Determinar la superposición entre la Bioinformática y el Enfoque shRNA Al describir el transcriptoma dependiente factor de transcripción
- para detERMINE las proporciones esperadas y observadas de los genes de microARN que albergan factor de transcripción sitios de unión en sus promotores y fueron regulados a la baja en el factor de transcripción shRNA células transfectadas, haga lo siguiente:
- Obtener la relación de los genes que albergan el factor de transcripción de interés en su promotor / micro ARN total a partir de la lista generada en 1.3. Esta lista es la proporción esperada (por ejemplo, los genes de microARN con los sitios de unión de STAT3 / total de genes de microARN probados = 0,25).
- Determinar el número de genes que son regulados negativamente en las células transfectadas factor de transcripción shRNA de la lista generada en 3.3 y tiene factor de transcripción de sitios de unión de interés de la lista generada en 1.3. Este número / número total de genes regulados negativamente es la proporción observada (por ejemplo, los genes microRNA con sitios de unión de STAT3 / número total de microRNAs downregulated = 0,6).
- Utilice χ2 estadísticas para comparar las proporciones observadas y esperadas que se generaron por encima y determinar si la lista de genes que se downregulated en las células transfectadas factor de transcripción shRNA están enriquecidas con factor de transcripción sitios de unión en su promotor.
Resultados
STAT3 es un factor de transcripción que normalmente induce la transcripción de genes que tienen propiedades anti efectos proliferativos 12 apoptótica y. Ya sea STAT3 también afecta a la ARN no codificante transcriptoma es actualmente desconocido. En todas las células de CLL STAT3 es constitutivamente fosforilada en serina 707 residuos de 10,13. Fosfoserina STAT3 servicio de enlace con el núcleo, se une al ADN, y activa los genes conocidos para ser activado por tirosina pSTAT3 en otros tipos de células 10. Debido CLL se caracteriza por la desregulación global de la red microRNA 14, la hipótesis de que la presencia de serina pSTAT3 afecta a la expresión de microRNAs en las células de LLC.
Para probar esta hipótesis promotores de microRNAs que albergan sitios de unión de STAT3 tenían que ser identificados. Al cruzar los datos generados por Baer et al. 9 de las regiones con la histona modifi H2K4me3catión que caracterizan a los sitios de promotor, con STAT3 sitios identificados por el experimento chip-ss 15 de unión, se identificaron sitios de unión putativos STAT3. Usando este enfoque se detectaron 160 promotores putativos en casi el 25% de los genes de microARN examinados (N = 200) con las puntuaciones de unión que van desde 100 (la puntuación más baja) a 1.000 (la puntuación más alta) (Tabla 1).
Posteriormente las células de CLL fueron transfectadas con STAT3-shRNA o con un vector vacío y el uso de matriz de microARN y se identificaron 63 microRNAs que son regulados hacia abajo después de la transfección (Figura 1) lo que sugiere que STAT3 promueve la transcripción de estos microRNAs. Para el 60% de los 63 genes regulados negativamente microARN (n = 38) de datos de chip-ss confirmados STAT3 unión en un supuesto promotor aguas arriba de la localización de genes, un número significativamente mayor de lo esperado por azar (p <0,0001). Nueve microRNAs que se redujeron reguladas después de la transfección,lo que sugiere que STAT3 regulan negativamente sus niveles de transcripción.
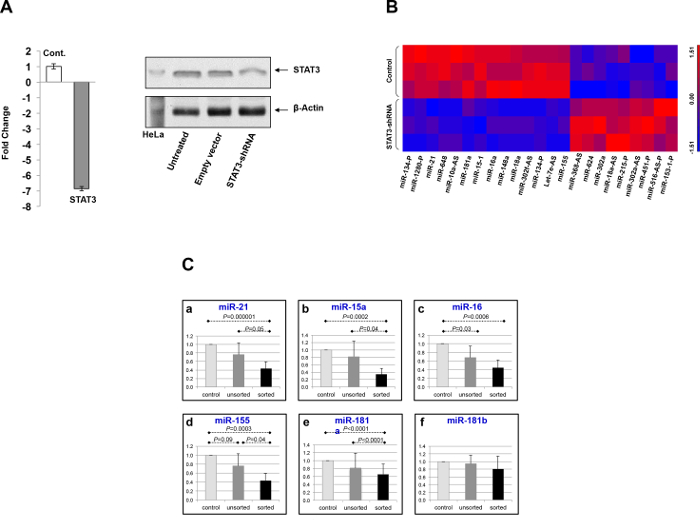
Figura 1. La transfección de las células de LLC con STAT3-shRNA reduce los niveles de expresión de la proteína STAT3 y mRNA STAT3. A:. Después de la transfección de las células de LLC con niveles de STAT3-shRNA de STAT3 ARNm (panel izquierdo, detectado por RT-PCR cuantitativa) y los niveles de la proteína STAT3 (panel de la derecha, que se detectó por inmunotransferencia Western) disminuyó significativamente B: matriz de micro ARN de la LLC células representan 23 microRNAs cuya expresión difiere significativamente entre las células de CLL transfectadas con STAT3-shRNA y células de CLL transfectadas con un vector vacío. Valor de p menor de 0,01 fue considerado estadísticamente significativo C:. La expresión medio cuantificado por RT-PCR de 7 microRNAs que tenían expresión diferencial después del tratamiento STAT3-shRNA usando elmatriz de microARN. Las barras representan el error estándar de la media. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Archivo Codificación suplementario 1. Haga clic aquí para descargar este archivo.
| Gen de ARN micro | Cromosoma | Las coordenadas de inicio promotor | Coordenadas finales promotor | La mediana (rango) de puntuación de unión de STAT3 * |
| MIR-1205, miR-1206, el miR-1207 | 8 q24.21 | 128961454 | 128962791 | 1000 (1000-1000) |
| MIR-1537 | 1 q42.3 | 236045425 | 236047415 | 1000 (1000-1000) |
| miR-21 | 17 q23.1 | 57901872 | 57921277 | 1000 (112-1000) |
| MIR-3124 | 1 q44 | 249115404 | 249123965 | 1000 (1000-1000) |
| miR-451 | 17q11.2 | 27222251 | 27224114 | 1000 (1000-1000) |
| miR-92b | 1 q22 | 155162340 | 155168439 | 1000 (1000-1000) |
| MIR-3197 | 21 q22.2 | 42537544 | 42543023 | 943 (943-943) |
| miR-646 | 20 q13.33 | 58712550 | 58715320 | 789 (789 a 789) |
| miR-629 | 15 q23 | 70383751 | 70394586 | 773 (661 a 885) |
| miR-30e, miR-30c-1 | 1 p34.2 | 41173077 | 41177703 | 759 (759 a 759) |
| MIR-3125 | 2 p24.3 | 12855381 | 12862915 | 756 (756-756) |
| MIR-3145 | 6 q23.3 | 138776942 | 138779365 | 743 (487-1000) |
| miR-645 | 20 q13.13 | 49199911 | 49201187 | 743 (743 a 743) |
| MIR-1256 | 1 p36.12 | 21346830 | 21350211 | 725 (725 a 725) |
| miR-619 | 12 q24.11 | 109248263 | 109253306 | 719 (719-719) |
| miR-181 A-2, miR-181b-2 | 9 q33.3 | 127418928 | 127426139 | 710 (710-710) |
| miR-29a, 29b-miR-1 | 7 q32.3 | 130583383 | 130597803 | 697 (482-1000) |
| miR-202 | 10 q26.3 | 135069499 | 135077337 | 696 (393-1000) |
| MIR-3142, miR-146a | 5 q34 | 159890882 | 159899475 | 671 (671 a 671) |
| miR-548C | 12 Q14.2 | 65000968 | 65011503 | 660 (660 a 660) |
| miR-630 | 15 q24.1 | 72764289 | 72769197 | 627 (255-1000) |
| miR-135b | 1 q32.1 | 205416952 | 205452990 | 622 (245-1000) |
| miR-29c, miR-29b-2 | 1 q32.2 | 207991044 | 208002382 | 608 (608 a 608) |
| MIR-1825 | 20 q11.21 | 30791020 | 30798310 | 604 (209-1000) |
| miR-548h-1 | 14 q23.2 | 64578.834 | 64581657 | 587 (174-1000) |
| miR-612 | 11 q13.1 | 65183633 | 65198528 | 581 (157-1000) |
| miR-148b | 12 q13.13 | 54717640 | 54721204 | 578 (578-578) |
| MIR-3174 | 15 q26.1 | 90543381 | 90549092 | 576 (152-1000) |
| let7a-3, let7b | 22 | 46480680 | 46481826 | 573 (146-1000) |
| miR-1255a | 4 Q24 | 102263848 | 102272541 | 557 (557 a 557) |
Tabla 1. promotores putativos de microRNA con sitios de unión de STAT3. El protocolo proporciona un método para identificar la unión de los promotores putativos de microRNAs utilizando la minería de datos de los datos publicados factor de transcripción. A modo de ejemplo, presentamos aquí una tabla que representa la unión de STAT3 a los promotores putativos de microARN. La puntuación de unión se da en una escala del genoma de datos seq chip enteros publicados como parte del proyecto ENCODE 15 0 a 1.000. Los promotores se identifican por la presencia de la firma epigenética H3K4me3 9.
Discusión
El mecanismo subyacente a la polimerasa de transcripción de ARN II- dependiente de proteínas de codificación de genes ha sido ampliamente estudiado. Si bien estos elementos constituyen sólo el 1% - 2% del genoma humano, la evidencia del proyecto ENCODE sugieren que más del 80% del genoma humano puede someterse a transcripción 17 y lo regula la transcripción de los elementos de ADN no codificantes sigue siendo en gran parte desconocida 6 .
Varios estudios, que indicaban que Pol II también es responsable de la transcripción de algunos genes no codificantes de proteínas que incluyen microRNAs 6, nos ha llevado a desarrollar una estrategia que combinan datos de recursos disponibles públicamente, algoritmos computacionales y estudios in vitro para descifrar el potencial función de un factor de transcripción de interés en la regulación de la transcripción de microARN. La estrategia sugerida en este documento incluye 2 pasos críticos. En primer lugar se utilizan los datos disponibles públicamente para identificar factores de transcripción bncontrar en promotores de microARN. A tal fin se utilizan los datos de Chip-Seq publicados por el consorcio ENCODE para identificar unión al factor de transcripción y la firma epigenética que tipifica como promotor de un marcador indirecto de microARN-promotores. Cruzando las coordenadas genéticos a partir de estos conjuntos de datos proporciona una estimación bruta de la frecuencia con un factor de transcripción de interés se une a microARN-promotor. En segundo lugar mediante el uso de tecnología de shRNA para silenciar la expresión de un factor de transcripción y sometiendo las células a microRNA-array, es posible explorar el significado funcional de un factor de transcripción en el microRNA-transcriptoma.
La variabilidad en la expresión de microARN se explica sólo en parte por la regulación dependiente de factor de transcripción. variabilidad estequiométrico y otros factores celulares o extracelulares juegan un papel importante y no se simulan en el algoritmo propuesto. Otras limitaciones incluyen los siguientes: El análisis promotor basado en la H3K4me3la firma se realizó en 939 microARNs anotados. Desde entonces se han identificado los lugares del genoma de muchos más microARN. No obstante a lo mejor de nuestro conocimiento una lista más completa que se basa en una base de datos actualizada no se ha publicado todavía. De los 939 genes de microARN, el H3K4me3 que tipifica la región promotora se identificó en 781 genes microARN (83%). Por lo tanto, mientras que este análisis se basa claramente en un conjunto de datos incompleto que captura una fracción significativa de la microRNA-transcriptoma.
Por otra parte, la epigenética firma se imprime en parte y en parte de células específicas. Por lo tanto, la generabilidad de promotores putativos que fueron definidos por los marcadores epigenéticos pueden ponerse en duda. Debido H3K4me3 persiste independiente de la transcripción 18 en general se considera un marcador de promotores impresos. Por lo tanto, el algoritmo de análisis que proponemos en este documento puede pasar por alto los promotores de microARN específicos de la célula si estos promotores fueron identificados por células en una diferentetipo. Por último, cualquier conclusión debe ser probado y confirmado empíricamente. Lo más notable es la asociación entre la regulación a la baja de un factor de transcripción (utilizando el enfoque shRNA) y la expresión de microARN (identificada por el conjunto de microARN) sólo puede sugerir un papel directo transcripcional que debe ser confirmado mediante ensayos aceptables tales como inmunoprecipitación de la cromatina (CHIP) o la movilidad electroforética cambio de ensayo (EMSA). Las modificaciones en el método sugerido en este documento incluyen diferentes formas de identificar promotor microARN y diferentes formas de derribar la expresión de un factor de transcripción, por ejemplo, pequeños ARN de interferencia en lugar de shRNA. La regulación de la transcripción de microARN puede ser sustancialmente diferente para los microRNAs que residen dentro de la codificación de los genes de proteínas (microRNAs intragenic) y los que no lo hacen (microRNAs intergénicas). Debido a que los microRNAs intragenic comúnmente se transcriben junto con sus genes del huésped 19 el promotor se encuentra generalmente inmediatamente upstream el sitio de inicio de la transcripción. Sin embargo, para muchos microRNAs intergénicas del sitio de inicio de la transcripción está mal anotado, y herramientas de predicción que se utilizan comúnmente para la proteína de los genes que codifican un mal desempeño 20.
Divulgaciones
The authors declare no competing financial interests.
Agradecimientos
Este estudio fue apoyado por una beca de la Fundación de Investigación Global de la LLC. La Universidad de Texas MD Anderson Cancer Center está apoyado en parte por los Institutos Nacionales de Salud a través de un Centro de Grant la ayuda del cáncer (P30CA16672).
Materiales
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies | 11668027 | |
| 0.45 µm syringe filter | Thermo Scientific (Nalgene) | 190-2545 | |
| Amicon ultracentrifugal filter device with threshold of 100 kDa | Merck Millipore | ||
| Polybrene | Merck Millipore | TR-1003-G | |
| TRIzol reagent | Life Tachnologies (Invitrogen) | 15596-026 | |
| 293 Cell line human | Sigma-Aldrich | 85120602 |
Referencias
- Bentwich, I., et al. Identification of hundreds of conserved and nonconserved human microRNAs. Nat Genet. 37, 766-770 (2005).
- Hata, A. Functions of microRNAs in cardiovascular biology and disease. Annu Rev Physiol. 75, 69-93 (2013).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Piriyapongsa, J., Jordan, I. K., Conley, A. B., Ronan, T., Smalheiser, N. R. Transcription factor binding sites are highly enriched within microRNA precursor sequences. Biol Direct. 6, 61 (2011).
- Rozovski, U., et al. Signal transducer and activator of transcription (STAT)-3 regulates microRNA gene expression in chronic lymphocytic leukemia cells. Mol Cancer. 12, 50 (2013).
- Turner, M. J., Slack, F. J. Transcriptional control of microRNA expression in C. elegans: promoting better understanding. RNA Biol. 6, 49-53 (2009).
- Cui, Q., Yu, Z., Pan, Y., Purisima, E. O., Wang, E. MicroRNAs preferentially target the genes with high transcriptional regulation complexity. Biochem Biophys Res Commun. 352, 733-738 (2007).
- Yamakuchi, M., Lowenstein, C. J. MiR-34, SIRT1 and p53: the feedback loop. Cell Cycle. 8, 712-715 (2009).
- Baer, C., et al. Extensive promoter DNA hypermethylation and hypomethylation is associated with aberrant microRNA expression in chronic lymphocytic leukemia. Cancer Res. 72, 3775-3785 (2012).
- Hazan-Halevy, I., et al. STAT3 is constitutively phosphorylated on serine 727 residues, binds DNA, and activates transcription in CLL cells. Blood. 115, 2852-2863 (2010).
- Melo, S. A., et al. A TARBP2 mutation in human cancer impairs microRNA processing and DICER1 function. Nat Genet. 41, 365-370 (2009).
- Akira, S. Functional roles of STAT family proteins: lessons from knockout mice. Stem Cells. 17, 138-146 (1999).
- Frank, D. A., Mahajan, S., Ritz, J. B lymphocytes from patients with chronic lymphocytic leukemia contain signal transducer and activator of transcription (STAT) 1 and STAT3 constitutively phosphorylated on serine residues. J Clin Invest. 100, 3140-3148 (1997).
- Calin, G. A., et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A. 101, 11755-11760 (2004).
- Consortium, E. P. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS biology. 9, e1001046 (2011).
- Miranda, K. C., et al. A pattern-based method for the identification of MicroRNA binding sites and their corresponding heteroduplexes. Cell. 126, 1203-1217 (2006).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57-74 (2012).
- Lau, J. C., Hanel, M. L., Wevrick, R. Tissue-specific and imprinted epigenetic modifications of the human NDN gene. Nucl Acids Res. 32, 3376-3382 (2004).
- Corcoran, D. L., et al. Features of mammalian microRNA promoters emerge from polymerase II chromatin immunoprecipitation data. PLoS One. 4, e5279 (2009).
- Bhattacharyya, M., Feuerbach, L., Bhadra, T., Lengauer, T., Bandyopadhyay, S. MicroRNA transcription start site prediction with multi-objective feature selection. Stat Appl Genet Mol Biol. 11, (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados