Method Article
Эффективное использование Иммунопреципитация хроматина Ограничение количество биомассы
В этой статье
Резюме
Мы описываем надежного метода иммунопреципитации хроматина с использованием первичных Т-клеток. Метод основан на стандартных подходов, но использует конкретный набор условий и реагентов, которые повышают эффективность для ограниченного количества клеток. Важно отметить, что подробное описание фазовый анализ Данные представлены.
Аннотация
Иммунопреципитации хроматина (чип) является широко используемый метод определения взаимодействия различных белков с ДНК в хроматин живых клеток. Примеры включают последовательности конкретных ДНК-связывающий факторы транскрипции, гистонов и их различные состояния модификации, ферменты, такие как РНК-полимеразы и вспомогательные факторы, а также компоненты репарации ДНК. Несмотря на свою вездесущность, есть отсутствие соответствующий современным требованиям, подробные методологии и скамейки подготовке материалов и для точного анализа позволяет количественным показателям взаимодействия. Из-за этого недостатка информации, а также потому, что, как и любой иммунопреципитации условия должны быть повторно оптимизирован для новых наборов экспериментальных условий, чип анализа является восприимчивым к неточной или плохо количественные результаты.
Наш протокол, в конечном счете, полученных из семенной работы по транскрипционный фактор: 1,2 взаимодействий ДНК, но включает в себя ряд улучшений сенсибилизацииВиты и воспроизводимость для трудных для получения типов клеток. Протокол успешно используется 3,4, как с помощью количественной ПЦР количественно ДНК обогащения, или с использованием полуколичественного варианта ниже протокола.
Этот количественный анализ ПЦР усиленный материал выполняется вычислительно, и представляет собой ограничивающий фактор в анализе. Важные элементы управления и другие соображения включают в себя использование изотипу антител, а также оценки контрольной области геномной ДНК, таких как межгенной области предсказал не быть связанными белка в исследовании (или ожидается не показать изменения под экспериментальных условий). Кроме того, стандартная кривая исходного материала для каждого образца чип используется для получения абсолютных уровнях обогащения экспериментального материала. Использование стандартных кривых помогает принимать во внимание различия между наборов праймеров, независимо от того, насколько тщательно они предназначены, а также эффективностью отличаютсяences во всем диапазоне концентраций шаблон для одного набора праймеров. Наш протокол отличается от других, которые доступны в 5-8, что мы широко охватывали спустя, фазовый анализ.
протокол
1. Выделение мышью селезенки Наивные CD4 Т-клеток
- Жертвоприношение мышь гуманным образом согласуется с Институциональные уходу и использованию животных комитета (IACUC) протоколов. Проанализируйте селезенки и поместить его в чашку Петри, содержащую 10 мл DMEM с 10% FBS.
- Раздавить селезенки использованием матового концами двух предметных стекол, чтобы освободить спленоцитам. Передача клеточной суспензии в 15 мл коническую трубку.
- Собирают клетки центрифугированием при 200 х г (~ 1200 оборотов в минуту на клинической центрифуге с типичным диаметром ротора) в течение 5 мин при 4 ° C.
- Ресуспендируют клеток в 2 мл ACK буфера для лизиса эритроцитов, 1 мин при комнатной температуре (RT). Остановить реакцию добавлением 8 мл DMEM с FBS.
- Собирают клетки центрифугированием при 200 х г в течение 5 мин при 4 ° С.
- Ресуспендируют клеток в 5 мл DMEM содержащей FBS и проходят через 70 мкм сетчатый фильтр (BD Сокол, Cat # 352350). Граф клеток и перейтик изоляции наивных CD4 Т-клеток с использованием набора для выделения CD4 (Miltenyi, Cat # 130-095-248), следуя инструкциям производителя. Сбор наивным Т-клеток CD4 центрифугированием при 200 х г в течение 5 мин при 4 ° С. Ресуспендируют клеток в 10 мл среды DMEM с FBS.
2. Подготовка хроматина
- Добавить 37% формальдегида в конечной концентрации 1% к суспензии клеток в DMEM и осторожно встряхнуть при комнатной температуре (например, при помощи Nutator) в течение 15 мин к сшиванию ДНК-белковых комплексов.
- Стоп сшивающего добавлением 1 М глицина в конечной концентрации 125 мМ. Продолжайте рок в течение 5 мин при комнатной температуре.
- Собирают клетки центрифугированием при 200 х г в течение 5 мин при 4 ° С.
- Вымойте клетки ресуспендированием в 5 мл ледяной PBS содержащих ингибиторы протеазы. Стирать в ледяной PBS содержащих ингибиторы протеазы 3 раза общего и собирать клетки центрифугированием при 4 ° С.
- Ресуспендируют клеток в 1 млледяной лизиса клеток буфер, содержащий ингибиторы протеазы. Инкубируют на льду в течение 15 мин. Эффективность лизиса клеток может быть определена путем ресуспендирования небольшую аликвоту клеток в 0,4%-ного раствора трипанового синего (Sigma, Cat # T8154) и наблюдение с помощью микроскопа.
- Собирают ядер центрифугированием при 200g (обычно ~ 1200 оборотов в минуту) в течение 5 мин при 4 ° C. Осторожно удалите супернатант.
- Ресуспендируют ядер в 500 мкл буфера для лизиса ядерный содержащего ингибиторы протеаз. Инкубируют на льду в течение 15 мин.
- Разрушать ультразвуком клеток с использованием Sonicator Misonix 3000, размер зонда 1,6 мм: Выходной уровень 4, 15 сек взрыв, 4 раза на льду. Каждый образец должен быть охлажден на льду в течение 1-2 мин, после обработки ультразвуком еще раз, чтобы избежать перегрева образцов. Перегрев может вызвать изменение поперечных связей.
- Центрифуга ультразвуком хроматина при 16000 х г (~ 13200 оборотов в минуту в микроцентрифуге) в течение 5 мин при 4 ° C.
- Возьмите 20 мкл аликвоты ясно supernatant, добавляют краситель ДНК загрузки и проверки ультразвуком ДНК с помощью электрофореза на 2% агарозном геле (идеальный размер ультразвуком ДНК для большинства применений составляет 200-500 п.н.).
- Определение концентрации ДНК с помощью УФ спектрофотометра. Стриженый хроматина может быть использована немедленно создать хроматина реакции иммунопреципитацией или хранили при -80 ° C. Обычно мы получаем 7,5-10 мкг ДНК из 2-3 X 10 6 очищенных CD4 Т-клеток на мышь.
3. Хроматина Иммунопреципитация (Все шаги должны осуществляться при температуре 0-4 ° C)
- Развести ультразвуком хроматина с ДНК концентрации 5-10 мкг / мл (общий объем = 1 мл) на чипе буфером для разведения с ингибиторами протеазы.
- Сохранить 100 мкл (10%) в качестве входных данных. Хранить на льду.
- Алиготе 450 мкл каждого из двух 1,7 мл пробирок микроцентрифужную как изотипического контроля (мышь или кролика IgG) и интерес антитело. Если несколько антител того же изотипа использовании одного контр изотипаПР будет достаточно для выполнения такого анализа. Вам понадобится несколько элементов управления изотипом если антитела другого источника животного или изотипом используются.
- Добавить 2-5 мкг антитела, в зависимости от специфичности антител использовали или изотипа контроль к соответствующей трубы.
- Rock трубы использовании Nutator в течение ночи при 4 ° С, чтобы позволить формирование хроматин-антитело.
- Добавить 25 мкл белка G магнитных шариков (1:1 суспензии взвешенных бусы) к вышеуказанной смеси и позволить, чтобы качаться в течение 2 ч при 4 ° С. Бусы из наших поставщиков может быть использован непосредственно.
- Поместите микроцентрифужную труб на магнитную стойку и позволяют собирать бусы на намагниченной стороной.
- Осторожно снимите решение стремлением не нарушая бисером.
- Добавить 1 мл промывочного раствора низкой соли и позволяют осторожно встряхнуть течение 5 мин на Nutator. Соберите бусин с помощью магнитного подставке, и снимите промывочного раствора. Повторите еще раз.
- Добавить 1 мл промывочного раствора высокой соли и позволяют качать течение 5 мин на Nutator. Соберите валиками магнитного подставке, и снимите промывочного раствора. Повторите еще раз.
- Добавить 1 мл промывочного раствора лития хлорида и позволяют качать течение 5 мин на Nutator. Соберите валиками магнитного подставке, и снимите промывочного раствора. Повторите еще раз. Использование LiCl улучшает эффективное удаление неспецифического хроматина взаимодействия с шариками.
- Добавьте 1 мл раствора ТЕ и позволяют качать течение 5 мин на Nutator. Соберите валиками магнитного подставке, и снимите промывочного раствора.
- Элюции ДНК из гранул путем добавления 250 мкл буфера для элюции. Rock в течение 15 мин при комнатной температуре. Отбирают пипеткой элюата и сохраните этот материал в новой трубки микроцентрифужную 1,7 мл. Повторите еще раз, и объединить оба элюирований. Откажитесь от бисера.
- Чтобы изменить сшивок добавляют 5 М NaCl до конечной концентрации 0,3 М и 1 мкл РНКазы А (20 м г / мл) в элюированной ДНК. Добавить 400 мкл буфера для элюции с входами сохранен на шаге 3.2 до объема 500 мкл. К входам добавить NaCl до 0,3 М, 1 мкл РНКазы А, 10 мкл 0,5 М ЭДТА, 20 мкл 1 М Трис-HCl, рН 6,5 и 1 мкл протеиназы К (20 мг / мл).
- Инкубировать пробирки в течение ночи при 65 ° С в сухом нагревательный блок. Чтобы избежать испарения образцов, печати или разместить груз на трубы, чтобы удержать их от открытия. Гибридизации печи также может быть использован.
- Пусть трубы охлаждаться до комнатной температуры. Добавляют 1 мл 100%-ного этанола и инкубируют 2 ч, в течение ночи при -80 ° С для осаждения ДНК.
- Центрифуга труб при 16000 х г в течение 15 минут, чтобы осадить ДНК. Промойте осадок ДНК один раз 70% этанолом и воздушно-сухих гранул. Ресуспендируют осадок ДНК в 100 мкл дистиллированной воды автоклаве.
- Очищают ДНК с использованием набора QIAquick центрифужных колонок и элюировали в общем объеме 50 мкл буфера для элюции. Эта ДНК готов к использованию для ПЦР.
- Соберите все образцы ДНК из чипа и соответствующего входных отсчетов. Кроме того, собирать всех реагентов для ПЦР и праймеров для целевого региона, а также управления региона. Используйте "горячего старта" Taq ДНК-полимеразы. Мы будем использовать SYBR Green основе анализа для количественной амплификации ДНК в этой процедуре, но количественной ПЦР Mastermix наборы могут быть использованы, если это необходимо.
- Сделать серийные разбавления вход ДНК для получения стандартной кривой в анализе количественной ПЦР: например, 10%, 1%, 0,1% и 0,01% в элюирующего буфера (от спин QIAquick столбцов на этапе 3.18). Диапазон концентрации и приращение этой стандартный набор шаблонов кривой может меняться в зависимости от ожидаемого уровня чип обогащения, и т.д. Распределить 10 мкл входных выборок ДНК в соответствующие лунки 96-луночного планшета (Genemate, кошка # T-3182-1 ), в двух экземплярах.
- Внесите 10 мл ChIP ДНК из изотипом контроля, а также специфических антител в соответствующихскважин, в трех экземплярах.
- Сделать "мастер смеси", содержащей либо мишень праймеров или контроль праймеры (0,1 мкМ конечную концентрацию каждого праймера) и обойтись 10 мкл в соответствующие лунки. Например, в шаблоне ниже обойтись мастер-микс содержащие целевые праймеров в колодцы отмечены зеленым и обойтись мастер-микс с управлением праймеров в колодцы отмечены красным.
- Крышка ПЦР планшет с оптической пленкой герметизации и центрифугируют при 500 х г в клинической центрифуге (~ 1200 оборотов в минуту) в течение 1 мин при комнатной температуре, чтобы собрать все в нижней части скважины.
- Начало реакции ПЦР. Мы будем использовать LightCycler 480 II (Roche) операционной LightCycler 480SW 1.5 программного обеспечения для запуска кПЦР в этом примере.
Преинкубация: 1 цикл: 95 ° C / 5 мин.
Усиление: 40-45 циклов: 94 ° C / 5 сек, 60 ° С / 5 сек, 72 ° C/10 сек (температура отжига должна быть на 2-3 ° С ниже, чем температура плавления температуре праймеров).
Кривая плавления: 1 цикл.
Охлаждение: 1 цикл.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| 10% Входное | 10% Входное | Изотипом | Конкретный | |||||||||
| B | 1% Входной | 1% Входной | Изотипом | Конкретный | ||||||||
| C | 0,1% Входной | 0,1% Входной | Изотипом | Конкретный | ||||||||
| Ре | 0,01% Входная | 0,01% Входная | ||||||||||
| Ми | 10% Входное | 10% Входное | Изотипом | Конкретный | ||||||||
| Фа | 1% Входной | 1% Входной | Изотипом | Конкретный | ||||||||
| Соль | 0,1% Входной | 0,1% Входной | Изотипом | Конкретный | ||||||||
| H | 0,01% Входная | 0,01% Входная |
Зеленый: Используйте целевые праймеров региона.
Красный: используйте праймеры управления региона.
5. Анализ
- Программа кПЦР программного обеспечения для количественного определения абсолютного количества ДНК. Важно отметить, что использование стандартной кривой для интерполяции ДНК количествах в конкретных неизвестных и изотипа образцов позволяет оценить и соответствующие качества и эффективности всех наборов праймеров в диапазоне концентраций найдены в образцах. Кроме того, она также обеспечивает морэлектронной надежный метод для преобразования Cq (C Т или С р) до конечного значения относительного раза результаты, чем usingΔΔCq обогащения и связанных с ними методов.
- К образцу редактор и отметить лунках, содержащих входные выборки из различных разведениях (10%, 1%, 0,1% и 0,01%) и их стандартные значения кривой. Также маркировать скважин с неизвестными образцами чип, точно так, как пластины ПЦР выложено. В эквивалентной программного обеспечения, используемого в других машинах кПЦР, назначать множеств, соответствующих реакций для каждой пары праймеров, стандартные значения кривой и экспериментальной и контрольной пробы изотипом соответственно.
- В программном обеспечении LightCycler, перейдите на подмножества редактор и отметьте подмножества включая скважины с входных отсчетов, а также неизвестных образцов чипа. Неизвестные образцы будут количественно с использованием стандартной кривой, полученной с известными концентрациями входных выборок для анализа. В эквивалентной программного обеспечения, используемого в других машинах кПЦР назначают подмножества соотвonding для всех реакций для каждой пары праймеров.
- После ПЦР-амплификации завершения выполнения анализа с "АБС Quant/2nd Производные Max" и выберите подмножество для анализа и нажмите ОК. В эквивалентной программного обеспечения, используемого в других машинах кПЦР, конвертировать Cq значение количество ДНК в каждом подмножестве.
- Нажмите кнопку "Рассчитать", которая сгенерирует стандартную кривую с использованием известных концентраций входных отсчетов и будет отображать абсолютное количество ДНК, присутствующей в неизвестных образцов.
- Если качество фрагментированной ДНК хорошо, и если праймеры специфически связываются с целевой области, ПЦР эффективность должна быть близкой к 2.
- Выполнить анализ кривых плавления с "ТМ Calling" за тот же подмножество, если таковые имеются. Единственный пик указывает, что только один конкретный продукт амплифицировали. Амплификация специфической ДНК также может быть проверена путем электрофореза ПЦР-продукта вместе с лэддер ДНК через гель агарозы.
- Экспорт данных в виде вкладки-текстового файла с разделителями, который может быть открыт в Microsoft Excel для дальнейшего анализа.
- С помощью Microsoft Excel, разделите количество ДНК для специфических антител с контрольным изотипом для целевых праймеров. Повторите этот шаг для праймеров управления региона. Если несколько антител того же изотипа используются для различных иммунопреципитации, нормализуют каждую из этих со значениями из того же изотипа управления. Кроме того, если несколько целевых пар праймеров использовании количества ДНК из одного набора пары праймеров управления должна быть использована для нормализации каждой цели (фиг. 1 и 2). Выход значения представляют собой антитела иммунопреципитация кратное обогащение в каждой точке относительно неспецифического иммунопреципитации фон антителом.
- Разделите кратное обогащение (до изотипом контроль) для целевых праймеров с кратное обогащение (до изотипический контроль) для праймеров контрольной области для получения относительно обогащениянесвязанные области управления (рис. 1). Важно отметить, отметим, что из-за генерации стандартных кривых с общей ДНК ввод для каждого образца, каждая из вышеуказанных значений иммунопреципитации интерполированных из стандартных кривых уже нормализованы к и выражали как доля вход на машине программного обеспечения.
- Если жесткость буферов, используемых для иммунопреципитации или стиральные слишком высока, то возможно, что надежный усиления из образцов иммунопреципитации с использованием специфических антител будет наблюдаться, в то время как усиление от контроля изотипа иммунопреципитации образцы не будут соблюдены. В таком случае, лучше, чтобы вычесть сумму Изотип чип управления с конкретным антителом чип для целевой области и несвязанный регионе. Относительное обогащение может быть рассчитана путем деления разности для целевого региона разница для несвязанных области (рис. 3).
Результаты
Иммунопреципитация хроматина (чип) Протокол, представленные здесь, позволяет обеспечить учет различий, если таковые имеются, в количестве ДНК, используемые в ПЦР за счет использования пара праймеров, которая усиливает несвязанные области генома, таким образом, выступающей в качестве "управления загрузкой". В примере, показанном на рисунке 3, мы использовали кодирующей области гена мыши ACTB как область несвязанный нашим интерес белка и NFAT связывания фактора транскрипции участок на мышь Il2 промотора целевой области. Кроме того, область несколько тысяч пар нуклеотидов выше или ниже целевой области известно, не имеют связывания представляющего интерес белка могут быть выбраны для этой цели. При проектировании пары праймеров для целевого региона, идеальный размер ПЦР-продукта составляет приблизительно 100-200 пар оснований. Важно также, чтобы убедиться, что вычислительно эти праймеры не связывают больше нигде в геноме.
Относительное обогащение белка, связанного с изменениемразличны областей генома можно сравнить, если тот же набор управляющих изотипа и специфические антитела выбраны. Обогащение различных белков на том же регионе мишень будет зависеть от специфичности и аффинности антитела использованы, а также количество конкретного белка, связанного с ДНК. Например, относительное обогащение в случае чипы выполнены для гистонов обычно возвращают выше обогащение значение по сравнению с фактором транскрипции, которые могут связываться с ДНК в ответ на различные клеточные сигналы. Если специфичность антитела хорошо, обычно мы наблюдаем 2-10 кратное обогащение по области свободного элемента управления (фиг. 3 и 4).
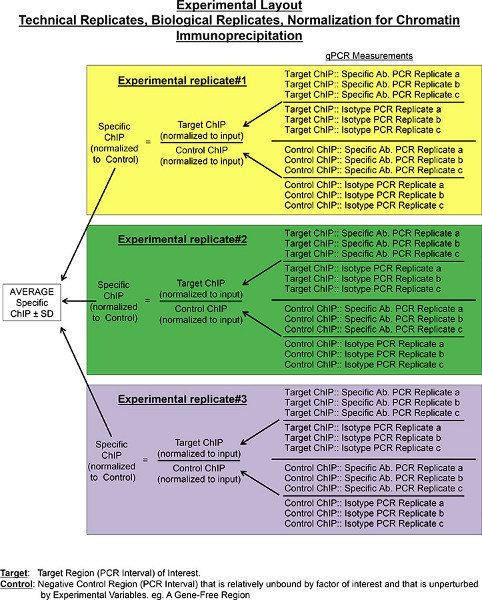
Рисунок 1. Экспериментальная схема описания расчетов для получения Relatив обогащение чип для специфических антител в сайте-мишени. Количественная ПЦР используют для получения ДНК количествах в чип образцы иммунопреципитации использованием специфического антитела и его изотипа управления. Количественная ПЦР выполняется с использованием праймеров охватывающих последовательности-мишени и несвязанного управления области генома. Для каждого чипа образца, ПЦР выполняется в трех экземплярах, обозначенное в техническом повторяет, В и С, а также относительное обогащение рассчитывается для каждой повторности, как описано в протоколе. Расчеты средний технический выходных значений выполняются. Отклонение также могут быть проанализированы на этом шаге, а в идеале должно быть низким. Технических расчетов ошибки, однако, не включены в окончательный формула для вычисления ошибки между биологической репликации. Скорее, измеренную биологическую изменение самой своей природе содержит технические вариации. Три экспериментальных повторяет (в коробках желтого цвета, зеленый и фиолетовый) используются на чип эксперимент, чтобы получить А.В.erage сигнал чипа и его стандартное отклонение (SD). Нажмите здесь, чтобы увеличить рисунок .

Рисунок 2. Альтернативный метод расчета чип обогащения. В случае, когда количество ДНК иммунопреципитации из изотипов антител является крайне низкой и количественной ПЦР amplication плохой, это количество ДНК можно вычесть из количества ДНК иммунопреципитации с использованием конкретного антитела, с получением раза обогащения для каждого технического репликации. Такой метод расчета должен быть использован во всех трех экспериментальных репликации, чтобы получить среднее сигнал чип и его стандартное отклонение (SD), как описано на фигуре 1.

Рисунок 3. Снимок Microsoft Excel таблицы показывает формула позволяет получить относительное обогащение. Данные из трех экспериментальных повторы (в коробке желтого цвета, зеленый и фиолетовый) с тремя техническими повторяет каждый был использован для расчета кратное обогащение. Использования расчета протокол, описанный на рисунке 1 изображен на эту таблицу. Нажмите здесь, чтобы увеличить рисунок .

Рисунок 4. Относительное обогащение NFAT транскрипционного фактора на Ил-2промоутера. Обычные CD4 Т-клеток (Tcon) и регулирующий CD4 Т-клеток (Tregs) были выделены из Foxp3EGFP мышей. Небольшая часть Tcon клетки стимулировали PMA и иономицина течение 30 мин. ChIP проводили, как описано в протоколе исследования. Данные, представленные здесь, от трех экспериментальных повторяется с тремя техническими повторяет каждый. Относительное обогащение транскрипционных факторов и NFATc1 NFATc2 на мышь IL2 промоутером в этих типах клеток изображен как раз над обогащением несвязанные области 3.
Обсуждение
Протокол выше, обеспечивает надежный метод точного количественного ДНК обогащения из первичных лимфоцитов использованием чипа. Одна из главных причин устойчивости в данном протоколе, является включение биологических повторяет. Приведенные выше протокол использует трех повторов, обогащение, для которых вычисляется независимо. Выходы затем усредняются, чтобы обеспечить степень обогащения и стандартные отклонения рассчитаны, чтобы обеспечить меру изменчивости. Для каждого образца, три технических воспроизводит также выполняются устранить изменения пробами, ПЦР-амплификации и т.д. количество вариаций в техническом повторяется, как правило, значительно меньше, чем наблюдаемое различных биологических образцах. Вторая причина устойчивости является включение нескольких технического контроля, в том числе по изотипу контрольными антителами, использование стандартной кривой ДНК ввод, и использование управления ампликона, соответствующего неспецифической геномной области.
Ограничения описанного выше способа аналогичны тем, которые встречаются обычно с использованием чипа. Способ имеет несколько бедных пространственное разрешение, ограничивающим фактором является сдвиг фрагментов ДНК с помощью ультразвука. Таким образом, разрешающая способность метода только около 500 пар оснований ДНК. Эксперименты, которые требуют более высокого разрешения могут воспользоваться микрококковой нуклеазой 8 или экзонуклеазную пищеварения восстановленных фрагментов (чип-экзо) 9,10. Кроме того, чип препаратов описано выше, соединенный с высокой пропускной последовательности восстановленных фрагментов (ChIPseq, чип-чип или след-SEQ) 11,12 вместо количественной ПЦР, производит выборку по-разному стриженый обогащенный фрагменты, пересечение которых может быть приняты как добросовестный сайта взаимодействия. Тем не менее, метод, представленный здесь обеспечивает надежный способ тщательно и аккуратно количественного ДНК совместно с факторами транскрипции, нуклеосомой компонентов, MODманьяков гистонов и других белков в определенных местах, представляющих интерес.
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Эта работа была поддержана грантами NIH CA141009 и GM39067. Мы благодарим Е. Р. Парнелл и Yarrington замечания по письменной части.
Материалы
| Name | Company | Catalog Number | Comments |
| Formaldehyde | Sigma | F-8775 | Store at RT |
| Phosphate Buffered Saline | Hyclone | SH30256.01 | Store at 4 °C |
| Protease Inhibitor tablets | Roche | 04693116001 | Store at 4 °C |
| Protein G magnetic beads | Active Motif | 101945 | Store at 4 °C |
| RNase A (20 mg/ml) | EMD Millipore | 556746 | Store at -20 °C |
| Proteinase K (20 mg/ml) | Roche | 03115879001 | Dissolve in 50 mM Tris-HCl, 10 mM CaCl2, pH 8.0 |
| Platinum Taq DNA Polymerase | Invitrogen | 10966-034 | Store at -20 °C |
| SYBR Green I | Invitrogen | S7567 | Store at -20 °C |
| 1 M Glycine | Store at RT | ||
| Cell lysis buffer (5 mM Pipes, pH 8.0; 85 mM KCl; 0.5% NP-40) | Store at 4 °C | ||
| Nuclear lysis buffer (50 mM Tris, pH 8.1; 10 mM EDTA; 1% SDS) | Store at RT | ||
| ChIP dilution buffer (0.01% SDS; 1.1% Triton X-100; 1.2 mM EDTA; 16.7 mM Tris pH 8.1; 190 mM NaCl) | Store at 4 °C | ||
| Low salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 150 mM NaCl) | Store at 4 °C | ||
| High salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 600 mM NaCl) | Store at 4 °C | ||
| LiCl wash buffer (0.25 M LiCl; 1% NP-40; 1% Sodium Deoxycholate, 1 mM EDTA; 10 mM Tris pH 8.0) | Store at 4 °C | ||
| TE buffer (10 mM Tris, pH 7.4, 1 mM EDTA) | Store at 4 °C | ||
| Elution buffer (1% SDS; 0.1 M NaHCO3) | Prepare fresh | ||
| 5 M NaCl | Store at RT | ||
| 0.5 M EDTA | Store at RT | ||
| 1 M Tris-HCl, pH 6.5 | Store at RT | ||
| Table of Specific Reagents | |||
| Clay Adams Brand Nutator | Becton Dickinson | Model: 421105 | |
| Magnetic Stand | Promega | Z5342 | |
| Qiaquick PCR Purification Kit | Qiagen | 28106 | |
| Masonix Sonicator 3000 | QSonica | Model: S3000 | |
| UV Spectrophotometer | NanoDrop Technologies | ND-1000 | |
| Heating Block | VWR | 13259-030 | |
| Rotator | VWR | 80085-692 | |
| Refrigerated bench top centrifuge | Beckman Coulter | Model: Allegra X-12R | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Table of Equipment | |||
Ссылки
- Weinmann, A. S., Bartley, S. M., Zhang, T., Zhang, M. Q., Farnham, P. J. Use of chromatin immunoprecipitation to clone novel E2F target promoters. Molecular and Cellular Biology. 21, 6820-6832 (2001).
- Weinmann, A. S., Farnham, P. J. Identification of unknown target genes of human transcription factors using chromatin immunoprecipitation. Methods. 26, 37-47 (2002).
- Li, Q., et al. Constitutive nuclear localization of NFAT in Foxp3+ regulatory T cells independent of calcineurin activity. J. Immunol. 188, 4268-4277 (2012).
- Shakya, A., Kang, J., Chumley, J., Williams, M. A., Tantin, D. Oct1 is a switchable, bipotential stabilizer of repressed and inducible transcriptional states. The Journal of Biological Chemistry. 286, 450-459 (2011).
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Lubelsky, Y., Macalpine, H. K., Macalpine, D. M. Genome-wide localization of replication factors. Methods. 57, 187-195 (2012).
- O'Neill, L. P., VerMilyea, M. D., Turner, B. M. Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations. Nature Genetics. 38, 835-841 (2006).
- Sikes, M. L., et al. A streamlined method for rapid and sensitive chromatin immunoprecipitation. Journal of Immunological Methods. 344, 58-63 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nature Biotechnology. 26, 1351-1359 (2008).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены