Method Article
Effiziente Chromatin Immunopräzipitation mit limitierende Mengen an Biomasse
In diesem Artikel
Zusammenfassung
Wir beschreiben eine robuste Methode zur Chromatinimmunpräzipitation mit primären T-Zellen. Die Methode basiert auf Standard-Ansätzen gegründet, nutzt aber einen bestimmten Satz von Bedingungen und Reagenzien, die Effizienz für begrenzte Mengen von Zellen einer zu verbessern. Wichtig ist, dass auf eine detaillierte Beschreibung des Datenanalysephase dargestellt.
Zusammenfassung
Chromatin Immunopräzipitation (ChIP) ist eine weit verbreitete Methode zur Bestimmung der Wechselwirkungen verschiedener Proteine mit DNA in Chromatin lebender Zellen. Beispiele umfassen sequenzspezifische DNA-bindende Transkriptionsfaktoren, Histone und ihre verschiedenen Modifikationen Zustände, Enzyme, wie RNA-Polymerasen und zugehörige Faktoren und DNA-Reparatur-Komponenten. Trotz seiner Allgegenwart, gibt es einen Mangel an up-to-date, detaillierte Methoden sowohl für Bank Vorbereitung des Materials und für die genaue Analyse ermöglicht quantitative Metriken der Interaktion. Durch diesen Mangel an Informationen, und auch, weil, wie jeder Immunpräzipitation Bedingungen müssen für neue Sätze von experimentellen Bedingungen neu optimiert werden kann, ist der Chip-Assay anfällig für fehlerhafte oder schlecht quantitative Ergebnisse.
Unser Protokoll wird schließlich vom bahnbrechenden Arbeiten auf Transkriptionsfaktor abgeleitet: DNA-Wechselwirkungen 1,2, sondern beinhaltet eine Reihe von Verbesserungen an Sensibilisierungvity und Reproduzierbarkeit für schwer zu erhalten Zelltypen. Das Protokoll wurde erfolgreich eingesetzt, 3,4, beide unter Verwendung von DNA-qPCR Bereicherung quantifizieren, oder mit Hilfe eines semi-quantitative Variante des unter Protokoll.
Diese quantitative Analyse der PCR-amplifizierten Materials rechnerisch durchgeführt und stellt einen begrenzenden Faktor in dem Assay. Wichtige Kontrollen und andere Überlegungen schließen die Verwendung eines Isotyp-Antikörper, sowie Auswertung einer Kontrolle Region von genomischer DNA, wie ein Zwischengenregion vorhergesagt nicht durch das Protein unter Studie (oder erwartet gebunden werden keine Änderungen unter zeigen die experimentellen Bedingungen). Darüber hinaus wird eine Standardkurve von Vormaterial für jeden Span Probe zur Ableitung absoluten Werte der Anreicherung im experimentellen Material. Verwendung von Standard-Kurven hilft zur Berücksichtigung von Unterschieden zwischen Primer-Sets nehmen, unabhängig davon, wie vorsichtig sie sind entworfen, und auch Effizienz unterscheidenschiede über den Bereich der Template-Konzentrationen für eine Primer-Set. Unser Protokoll ist anders als die anderen, die verfügbar sind 5-8 in die wir ausgiebig decken die später Analysephase.
Protokoll
1. Isolation von Maus Milz Naive CD4 T-Zellen
- Aufopferung der Maus auf humane Weise im Einklang mit Institutional Animal Care und Use Committee (IACUC) Protokolle. Präparieren Sie die Milz und legen Sie es in Petrischale mit 10 ml DMEM mit 10% FBS.
- Crush die Milz mit mattierten Enden von zwei Glasplatten, die Splenozyten freizugeben. Übertragen Sie die Zellsuspension in einem 15 ml konischen Röhrchen.
- Sammeln der Zellen durch Zentrifugation bei 200 xg (~ 1.200 UpM einer klinischen Zentrifuge mit einer typischen Rotordurchmesser) für 5 min bei 4 ° C.
- Die Zellen in 2 ml ACK-Puffer zur Lyse roter Blutkörperchen, 1 min bei Raumtemperatur (RT). Stoppen der Reaktion durch Zugabe von 8 ml DMEM mit FBS.
- Sammeln der Zellen durch Zentrifugation bei 200 × g für 5 min bei 4 ° C.
- Die Zellen in 5 ml DMEM mit FBS und durch ein 70 um-Sieb (BD Falcon, Cat # 352350). Zählen Sie die Zellen und gehendie Isolation von naiven CD4-T-Zellen mit einem CD4 Isolation Kit (Miltenyi, Cat # 130-095-248) mit den Anweisungen des Herstellers. Sammeln naiven CD4 T-Zellen durch Zentrifugation bei 200 × g für 5 min bei 4 ° C. Die Zellen in 10 ml DMEM mit FBS.
2. Herstellung von Chromatin
- In 37% Formaldehyd in einer Endkonzentration von 1% zu der Zellsuspension in DMEM und sanft bei Raumtemperatur (z. B. mit einem Nutator) schaukeln für 15 Minuten zu vernetzen DNA-Protein-Komplexe.
- Stoppen Vernetzung durch Zugabe von 1 M Glycin bis zu einer Endkonzentration von 125 mM. Weiterhin für 5 min bei RT rocken.
- Sammeln der Zellen durch Zentrifugation bei 200 × g für 5 min bei 4 ° C.
- Die Zellen werden durch Resuspendieren in 5 ml eiskaltem PBS enthaltend Proteaseinhibitoren. Waschen in eiskaltem PBS enthaltend Proteasehemmer 3 mal insgesamt und sammeln die Zellen durch Zentrifugation bei 4 ° C.
- Die Zellen in 1 mleiskaltem Lyse-Puffer mit Protease-Inhibitoren. Inkubieren auf Eis für 15 min. Die Effizienz der Lyse kann durch Resuspendieren Ein kleines Aliquot von Zellen in 0,4% Trypan-Blau-Lösung (Sigma, Cat # T8154) und die Beobachtung mit einem Mikroskop bestimmt werden.
- Sammeln der Kerne durch Zentrifugation bei 200 xg (typischerweise ~ 1.200 rpm) für 5 min bei 4 ° C. Vorsichtig den Überstand verwerfen.
- Resuspendieren die Kerne in 500 ul der nuklearen Lysepuffer Proteaseinhibitoren. Inkubieren auf Eis für 15 min.
- Mit Ultraschall die Zellen unter Verwendung eines Misonix Sonicator 3.000, Sonde Größe 1,6 mm: Ausgangspegel 4, 15 sec Burst, 4 mal auf Eis. Jede Probe muss auf Eis für 1-2 min vor Beschallung es wieder eine Überhitzung zu vermeiden Proben gekühlt. Überhitzung kann bewirken, dass die Umkehrung der Querverweisen.
- Zentrifuge die beschallte Chromatin bei 16.000 xg (13.200 rpm ~ in einer Mikrozentrifuge) für 5 min bei 4 ° C.
- Nehmen Sie einen 20 ul Aliquot klar Supernatant, fügen DNA Ladefarbstoff und überprüfen Sie die beschallte DNA durch Elektrophorese durch ein 2% Agarosegel (ideale Größe der beschallten DNA für die meisten Anwendungen ist 200-500 bp).
- Bestimmen DNA-Konzentration mit einem UV-Spektralphotometer. Sheared Chromatin kann sofort verwendet werden, um die Einrichtung Chromatinimmunpräzipitation Reaktion oder bei -80 ° C. Typischerweise erhalten wir 7,5-10 ug DNA 2-3 X 10 6 gereinigten CD4 T-Zellen pro Maus.
3. Chromatin Immunopräzipitation (Alle Schritte müssen bei 0-4 ° C durchgeführt werden)
- Verdünnen Sie beschallten Chromatin zu einer DNA-Konzentration von 5-10 g / ml (Gesamtvolumen = 1 ml) in ChIP Verdünnungspuffers mit Protease-Inhibitoren.
- Sparen Sie 100 ul (10%) als Eingabe. Lagern auf Eis.
- Aliquot 450 ul jeweils in zwei 1,7 ml Mikrozentrifugenröhrchen als Isotypenkontrolle (Maus oder Kaninchen-IgG) und Antikörper von Interesse bezeichnet. Wenn mehrere Antikörper des gleichen Isotyps verwendet werden, eine einzige Isotyp control ausreichen werden, um diese Analyse durchzuführen. Sie müssen mehrere Isotypkontrollen wenn Antikörper eines anderen tierischen Ursprungs oder Isotyp verwendet werden.
- In 2-5 ug Antikörper, abhängig von der Spezifität der verwendeten Antikörper oder Isotyp-Kontrolle, um die jeweiligen Rohre.
- Schaukeln Sie die Rohre mit einem Nutator Nacht bei 4 ° C, um die Bildung von Chromatin-Antikörper-Komplexe zu ermöglichen.
- In 25 ul Protein G magnetischen Kügelchen (1:1 Slurry suspendiert Perlen) zu der obigen Mischung und lassen Sie sie für mindestens 2 Stunden bei 4 ° C rocken Perlen aus unserer Lieferanten können direkt verwendet werden.
- Legen Sie die Mikrozentrifugenröhrchen auf einem magnetischen Standfuß und lassen die Perlen auf der magnetisierten Seite zu sammeln.
- Entfernen Sie vorsichtig die Lösung durch Aspiration, ohne die Perlen.
- 1 ml salzarme Waschlösung und lassen Sie sanft für 5 min auf einem Nutator rocken. Sammeln Sie die Perlen mit einem Magnetfuß und entfernen Sie die Waschlösung. Wiederholen einmal.
- 1 ml der hohen Salz-Waschlösung und lassen Sie sie für 5 min auf einem Nutator rocken. Sammeln Sie die Perlen mit den magnetischen Standfuß und entfernen Sie die Waschlösung. Wiederholen einmal.
- 1 ml Lithiumchlorid Waschlösung und lassen Sie für 5 min auf einem Nutator rocken. Sammeln Sie die Perlen mit den magnetischen Standfuß und entfernen Sie die Waschlösung. Wiederholen einmal. Die Verwendung von LiCl verbessert die effektive Entfernung von unspezifisch Chromatin-Wechselwirkungen mit den Beads.
- 1 ml TE-Lösung und lassen Sie für 5 min auf einem Nutator rocken. Sammeln Sie die Perlen mit den magnetischen Standfuß und entfernen Sie die Waschlösung.
- Eluieren der DNA von den Beads durch Zugabe von 250 ul Elutionspuffer. Schaukeln für 15 min bei Raumtemperatur. Pipette aus dem Eluat und speichern dieses Material in einem neuen 1,7 ml Mikrozentrifugenröhrchen. Wiederholen Sie noch einmal und kombinieren beide Elutionen. Entsorgen Sie die Perlen.
- Zur Umkehr des Vernetzungen werden 5 M NaCl auf eine Endkonzentration von 0,3 M und 1 ul RNase A (20 m g / ml) zu der eluierten DNA. In 400 ul Elutionspuffer zu den Eingängen in Schritt 3.2, gespeichert, um das Volumen 500 ul zu machen. Um die Eingänge hinzuzufügen NaCl bis 0,3 M, 1 ul RNase A, 10 ul 0,5 M EDTA, 20 ul 1 M Tris-HCl pH 6,5 und 1 ul Proteinase K (20 mg / ml).
- Die Röhrchen über Nacht bei 65 ° C in einem trockenen Heizblock. Zum Verdampfen von Proben Dichtung zu vermeiden oder zu platzieren ein Gewicht auf den Rohren, um sie von Öffnung zu halten. Hybridisierungsofen können ebenfalls verwendet werden.
- Lassen Sie die Rohre auf RT abkühlen. 1 ml 100% Ethanol und Inkubation 2 h-Nacht bei -80 ° C zur Ausfällung von DNA.
- Zentrifugieren bei 16.000 g für 15 min zur Pelletierung der DNA. Waschen Sie das DNA-Pellet einmal mit 70% Ethanol und Luft trocknen des Pellets. Resuspendieren des DNA-Pellet in 100 ul autoklavierte destilliertem Wasser.
- Entschlacken DNA mit dem QIAquick Spin Columns und eluieren in einem Gesamtvolumen von 50 ul Elutionspuffer. Diese DNA ist einsatzbereit für die PCR.
- Sammeln Sie alle DNA-Proben von Chip und entsprechenden Eingang Proben. Außerdem sammeln Sie alle PCR-Reagenzien und Primer für die Zielregion sowie eine Kontrolle Region. Verwenden Sie einen "Warmstart" Taq DNA Polymerase. Wir werden eine SYBR Green-basierte Assays zur DNA-Amplifikation in diesem Verfahren zu quantifizieren, aber qPCR Mastermix Kits können verwendet werden, wenn es erforderlich ist.
- Einen seriellen Verdünnung der eingesetzten DNA, um eine Standardkurve in der qPCR Analyse zu erzeugen: zum Beispiel 10%, 1%, 0,1% und 0,01% in Elutionspuffer (von QIAquick Spin-Säulen in Schritt 3.18). Der Konzentrationsbereich und Zuwachs dieser Standardkurve Template-Set auf den erwarteten Niveaus von ChIP Anreicherung kann variieren, Abgabemotor etc. 10 ul Eingang DNA-Proben in entsprechenden Vertiefungen einer 96-Well-Platte (Genemate, cat # T-3182-1 ), in zweifacher Ausfertigung.
- Dosiernadel 10 ml ChIP DNA von Isotyp-Kontrolle sowie spezifische Antikörper in jeweiligeBrunnen, in dreifacher Ausfertigung.
- Machen Sie eine "Master-Mix", die entweder Ziel Primer oder Kontroll-Primer (0,1 pM Endkonzentration von jedem Primer) und verzichten 10 ul in entsprechende Vertiefungen. Zum Beispiel wird in der Vorlage unter verzichten Mastermix enthaltenden gerichtete Primer in Vertiefungen grün markiert verzichten Mastermix mit Kontroll-Primer in Vertiefungen rot markiert.
- Decken Sie die PCR-Platte mit optischem Kunststoff Versiegelungsfilm und Zentrifuge bei 500 xg in einer klinischen Zentrifuge (~ 1.200 rpm) für 1 min bei RT, alles auf dem Boden des Bohrlochs zu sammeln.
- Starten Sie die PCR-Reaktion. Wir verwenden die LightCycler 480 II (Roche) Betriebssystem LightCycler 480SW 1.5 Software, um die qPCR in diesem Beispiel auszuführen.
Pre-Inkubation: 1 Zyklus: 95 ° C / 5 min.
Amplifikation: 40-45 Zyklen: 94 ° C / 5 sec, 60 ° C / 5 sec, 72 ° C/10 sec (Annealing-Temperatur um 2-3 ° C geringer als der Schmelzpunkt temperature der Primer).
Schmelzkurve: 1 Zyklus.
Kühlung: 1 Zyklus.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| A | 10% Eingangs- | 10% Eingangs- | Isotyp | Spezifische | ||||||||
| B | 1% Eingang | 1% Eingang | Isotyp | Spezifische | ||||||||
| C | 0,1% Eingang | 0,1% Eingang | Isotyp | Spezifische | ||||||||
| D | 0,01% Eingang | 0,01% Eingang | ||||||||||
| E | 10% Eingangs- | 10% Eingangs- | Isotyp | Spezifische | ||||||||
| F | 1% Eingang | 1% Eingang | Isotyp | Spezifische | ||||||||
| G | 0,1% Eingang | 0,1% Eingang | Isotyp | Spezifische | ||||||||
| H | 0,01% Eingang | 0,01% Eingang |
Grün: Verwenden Zielregion Primer.
Rot: Verwenden Steuerbereich Primer.
5. Analyse
- Programm der qPCR-Software für die Quantifizierung der absolute DNA-Menge. Wichtig ist, ermöglicht die Verwendung einer Standardkurve interpoliert DNA Mengen in den unbekannten Proben bestimmten Isotyp und die Auswertung und Anpassung der Qualität und Effizienz aller Primer setzt über einen Bereich von Konzentrationen in den Proben gefunden. Darüber hinaus ist es auch ein more zuverlässige Methode, um die Cq (C t oder C p)-Werte bis zur endgültigen relativen fache Anreicherung Ergebnisse als usingΔΔCq und verwandten Methoden zu konvertieren.
- Gehen Sie auf die Sample-Editor und markieren Sie die Vertiefungen mit Eingang Proben von verschiedenen Verdünnungen (10%, 1%, 0,1% und 0,01%) mit ihren Standardkurve Werte. Auch bezeichnen die Brunnen mit unbekannten Chip-Proben, genau so, wie die PCR-Platte gelegt wird. In gleicher Software auch in anderen qPCR Maschinen bezeichnen Teilmengen entsprechend Reaktionen für jedes Primerpaar Standardkurve Wert und experimentelle und Isotypenkontrolle Probe entsprechend.
- In der LightCycler-Software, um den Subset-Editor gehen und markieren Sie die Teilmengen einschließlich der Wells mit Eingang Proben sowie die unbekannten Chip-Proben. Die unbekannten Proben quantifiziert mittels Standardkurve aus den bekannten Konzentrationen des Eingangs Proben für die Analyse erzeugt werden. In gleicher Software auch in anderen qPCR Maschinen verwendet, bezeichnen Teilmengen entspr.onding allen Reaktionen für jeden Primer-Paaren.
- Nach der PCR-Amplifikation abgeschlossen ist, führen Sie die Analyse mit "Abs Quant/2nd Derivative Max" und wählen Sie die Teilmenge zur Analyse und drücken Sie OK. In gleicher Software auch in anderen qPCR Maschinen, konvertieren Cq Wert auf die DNA-Menge in jeder Teilmenge.
- Drücken Sie auf "Berechnen", die in der Standard-Kurve mit den bekannten Konzentrationen des Eingangs Proben erzeugen und die absolute Menge der DNA in den unbekannten Proben anzuzeigen.
- Wenn die Qualität der DNA geschert ist gut, und wenn die Primer spezifisch an die Zielregion, sollten PCR-Effizienz in der Nähe von 2 sein.
- Führen Sie eine Schmelzkurvenanalyse mit "Tm Calling" für die gleiche Teilmenge, falls verfügbar. Einen einzelnen Peak zeigt an, dass nur ein bestimmter amplifiziert. Amplifikation spezifischer DNA kann auch durch Elektrophorese des PCR-Produkts mit DNA-Leiter auf einem Agarosegel getestet werden.
- Exportieren von Daten als Tab-Textdatei, die in Microsoft Excel zur weiteren Analyse kann geöffnet werden.
- Mit Microsoft Excel, teilen Sie die DNA-Menge für den spezifischen Antikörpers mit dem Isotypenkontrolle für gezielte Primer. Wiederholen Sie diesen Schritt für die Kontrolle Region Primer. Wenn mehrere Antikörper des gleichen Isotyps für verschiedene Immunpräzipitationen verwendet werden, zu normalisieren jeder von diesen mit den Werten aus dem gleichen Isotyp-Kontrolle. Außerdem, wenn mehrere gezielte Primer-Paare verwendet werden, sollte die DNA-Mengen von einer Steuerung Primerpaar Set zur Normalisierung jedes Targets (1 und 2) verwendet werden. Die Ausgangswerte stellen die spezifischen Antikörper Immunpräzipitation fache Anreicherung an jeder Stelle in Bezug auf nicht-spezifische Antikörper Hintergrund Immunpräzipitation.
- Teilen Sie die fache Anreicherung (die Kontrolle Isotyp) zur gezielten Primer mit dem fache Anreicherung (die Kontrolle Isotyp) zur Steuerung Region Primer zu einer Anreicherung in Bezug auf eine zu bekommenKontrolle ungebundenen Region (Abbildung 1). Wichtig ist zu beachten, daß wegen der Erzeugung von Standardkurven mit insgesamt eingesetzten DNA für jede Probe, wobei jede der genannten Immunpräzipitation vom Standard-Kurven interpoliert werden bereits normalisiert, ausgedrückt als der Anteil der Gesamtmenge an von der Maschine Software.
- Wenn die Stringenz der Puffer für die Immunpräzipitation oder Waschen verwendet wird, zu hoch ist, ist es möglich, dass robuste Amplifikation von Proben mit spezifischen Antikörpern immunpräzipitiert beobachtet wird, werden während Amplifikation von Isotyp-Kontrolle immunpräzipitiert Proben nicht beobachtet werden. In einem solchen Szenario ist es besser, die Menge an Isotypkontrollreagenzien Chip von spezifischen Antikörper-Chip sowohl für die Zielregion und der ungebundenen Region zu subtrahieren. Relative Anreicherung kann, indem die Differenz zur Zielregion von Differenz für ungebundene Region (Abbildung 3) berechnet werden.
Ergebnisse
Das Chromatin Immunopräzipitation (ChIP)-Protokoll hier vorgestellten Steuerungen für Unterschiede, wenn überhaupt, in der Höhe von DNA in der PCR durch die Verwendung eines Primer-Paar, das ein ungebundenes Region Genom verstärkt eingesetzt und dienen somit als "Ladekontrolle". In dem Beispiel in 3 gezeigt ist, haben wir die kodierende Region des Maus istB Gen als eine Region in unseren ungebundenes Protein von Interesse und des Transkriptionsfaktors NFAT Bindungsstelle auf Maus Il2 Promotor als Zielbereich verwendet. Alternativ kann ein Bereich mehrere Kilobasen stromaufwärts oder stromabwärts von der Zielregion bekanntlich keine Bindung des Proteins von Interesse haben für diesen Zweck gewählt werden. Bei der Gestaltung einer Primer-Paar für die Zielregion, ist die ideale Größe des PCR-Produkts etwa 100-200 bp. Es ist auch wichtig, um sicherzustellen, dass diese rechnerisch Primer nicht anderswo im Genom binden.
Relative Anreicherung eines Proteins gebunden different Regionen des Genoms kann verglichen werden, wenn der gleiche Satz von Isotyp-Kontrolle und Antikörpern ausgewählt werden kann. Anreicherung von verschiedenen Proteinen auf das gleiche Ziel Region über die Spezifität und Affinität des Antikörpers verwendet werden, sowie die Menge des spezifischen Proteins an die DNA gebunden abhängen. Zum Beispiel wird in Bezug Anreicherung im Falle von Chips für Histone durchgeführt i. d. R. eine höhere Anreicherung Wert, wenn ein Transkriptionsfaktor, der an DNA in Reaktion auf verschiedene zelluläre Signale binden verglichen. Wenn die Spezifität des Antikörpers ist gut, wir in der Regel beobachten eine 2-10 fache Anreicherung über die Region ungebundenes Steuerelement (Abbildungen 3 und 4).
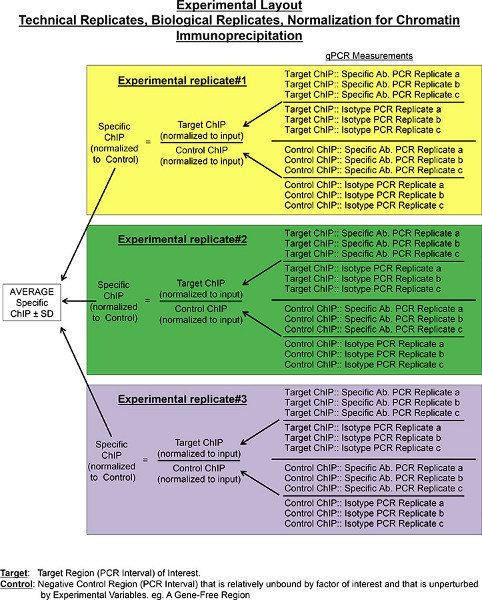
Abbildung 1. Experimentelle Layout beschreiben Berechnungen Relat erhaltenIVE Anreicherung Chip für spezifische Antikörper an der Zielstelle. Quantitative PCR verwendet wird, um DNA-Mengen in der Chip-Proben zu erhalten immunpräzipitiert Verwendung eines spezifischen Antikörpers und seine Isotyp-Kontrolle. Quantitative PCR durchgeführt unter Verwendung der Primer überspannt Zielsequenz und ungebundenes Steuerelement Region Genom. Für jeden Chip Probe wird die PCR in dreifacher Ausführung durchgeführt, angegeben als technische a, b und c repliziert und relative Anreicherung für jede Wiederholung berechnet, wie in dem Protokoll erläutert. Berechnungen für durchschnittlichen technischen Ausgangswerte werden durchgeführt. Veränderung kann auch bei diesem Schritt analysiert werden, und idealerweise sollte gering sein. Die technischen Fehler Berechnungen sind jedoch nicht in der endgültigen Formel für die Berechnung des Fehlers zwischen biologischen Replikate enthalten. Vielmehr ist die gemessene biologische Variation selbst auch von Natur aus enthält die technischen Unterschiede. Drei Experimentwiederholungen (in Kisten gelb, grün und lila) werden pro Chip Experiment verwendet, um einen av erhaltenschnittlich ChIP-Signal und dessen Standardabweichung (SD). Klicke hier, um eine größere Abbildung anzuzeigen .

Abbildung 2. Alternative Verfahren zur Berechnung von Chip-Anreicherung. In Fällen, in denen die Menge an DNA von der Isotyp-Antikörper immunpräzipitiert ist extrem niedrig und qPCR amplication schlecht ist, diese Menge an DNA aus der DNA-Menge abgezogen immunpräzipitiert Verwendung eines spezifischen Antikörpers, um den Falz zu erhalten Bereicherung für jeden technischen replizieren. Dieses Verfahren der Berechnung ist in allen drei experimentellen verwendet werden repliziert, um eine durchschnittliche Chipsignal und dessen Standardabweichung (SD), wie in Abbildung 1 beschrieben erhalten.

Abbildung 3. Snapshot von Microsoft Excel-Tabelle zeigt Berechnungen relative Anreicherung zu erhalten. Daten von drei experimentellen Wiederholungen (in Kisten gelb, grün und lila) mit drei technischen Replikate verwendet wurde, um fache Anreicherung berechnen. Die Nutzung der Berechnung Protokoll in Abbildung 1 beschrieben wird, in dieser Tabelle dargestellt. Klicke hier, um eine größere Abbildung anzuzeigen .

Abbildung 4. Relative Anreicherung von NFAT Transkriptionsfaktoren an der IL2Promotors. Konventionelle CD4 T-Zellen (Tcon) und regulatorische CD4 T-Zellen (Tregs) wurden von Foxp3EGFP Mäusen isoliert. Ein kleiner Bruchteil der Zellen mit PMA Tcon und Ionomycin wurde für 30 min stimuliert. Chip wurde durchgeführt wie im experimentellen Protokoll erläutert. Die hier gezeigten Daten ist von drei experimentellen wiederholt mit drei technischen Replikate. Relative Anreicherung von Transkriptionsfaktoren NFATc2 NFATc1 und auf der Maus IL2-Promotor in diesen Zelltypen als fache Anreicherung über einem ungebundenen Region 3 dargestellt.
Diskussion
Das Protokoll über eine robuste Methode zur Quantifizierung von DNA genau Anreicherung von primären Lymphozyten mit ChIP. Ein wichtiger Grund für Robustheit in diesem Protokoll ist die Aufnahme der biologischen Replikate. Die obige Protokoll verwendet drei Wiederholungen, die Anreicherung, für die unabhängig berechnet. Die Ausgänge werden dann gemittelt, um einen Grad der Anreicherung und Standardabweichungen berechnet, um ein Maß der Variabilität bereitzustellen. Für jede Probe repliziert drei technischen auch durchgeführt werden, um Variationen bei der Probenhandhabung, PCR-Amplifikation, etc. Die Menge der Veränderung der technischen wiederholt typischerweise viel geringer als die zwischen verschiedenen biologischen Proben beobachtet beseitigen. Ein zweiter Grund für Robustheit ist die Einbeziehung der verschiedenen technischen Überwachung einschließlich Isotyp-Kontroll-Antikörper, die Verwendung eines Standard-Eingabe DNA-Kurve, und die Verwendung einer Steuerung entsprechend einem Amplikon unspezifische genomischen Region.
Einschränkungen des obigen Verfahrens sind ähnlich zu denen in der Regel mit ChIP angetroffen. Das Verfahren leidet unter etwas schlechter räumlicher Auflösung, wobei der limitierende Faktor die Scherung von DNA-Fragmenten durch Beschallung. Daher ist das Auflösungsvermögen des Verfahrens nur etwa 500 bp der DNA. Experimente, die eine höhere Auflösung erfordern, können Gebrauch machen von Mikrokokkus Nucleaseverdau 8 oder Exonucleaseverdau der gewonnenen Fragmente (ChIP-exo) 9,10. Darüber hinaus beschrieben ChIP Vorbereitungen oben gekoppelt Hochdurchsatz-Sequenzierung der gewonnenen Fragmente (ChIPseq, ChIP-seq oder Chip-SEQ) 11,12 anstelle von qPCR, produziert eine Auswahl von unterschiedlich geschert angereichert Fragmente, deren Schnittpunkt kann genommen als bona fide Ort der Interaktion. Dennoch bietet die hier vorgestellte Methode eine robuste Möglichkeit der sorgfältig und genau zu quantifizieren DNA Verbindung mit Transkriptionsfaktoren, Nukleosomen Komponenten, modified Histone und andere Proteine an spezifischen Stellen des Interesses.
Offenlegungen
Kein Interessenkonflikt erklärt.
Danksagungen
Diese Arbeit wurde vom NIH Zuschüsse CA141009 und GM39067 unterstützt. Wir danken E. Parnell und R. Yarrington für Kommentare zu den schriftlichen Teil.
Materialien
| Name | Company | Catalog Number | Comments |
| Formaldehyde | Sigma | F-8775 | Store at RT |
| Phosphate Buffered Saline | Hyclone | SH30256.01 | Store at 4 °C |
| Protease Inhibitor tablets | Roche | 04693116001 | Store at 4 °C |
| Protein G magnetic beads | Active Motif | 101945 | Store at 4 °C |
| RNase A (20 mg/ml) | EMD Millipore | 556746 | Store at -20 °C |
| Proteinase K (20 mg/ml) | Roche | 03115879001 | Dissolve in 50 mM Tris-HCl, 10 mM CaCl2, pH 8.0 |
| Platinum Taq DNA Polymerase | Invitrogen | 10966-034 | Store at -20 °C |
| SYBR Green I | Invitrogen | S7567 | Store at -20 °C |
| 1 M Glycine | Store at RT | ||
| Cell lysis buffer (5 mM Pipes, pH 8.0; 85 mM KCl; 0.5% NP-40) | Store at 4 °C | ||
| Nuclear lysis buffer (50 mM Tris, pH 8.1; 10 mM EDTA; 1% SDS) | Store at RT | ||
| ChIP dilution buffer (0.01% SDS; 1.1% Triton X-100; 1.2 mM EDTA; 16.7 mM Tris pH 8.1; 190 mM NaCl) | Store at 4 °C | ||
| Low salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 150 mM NaCl) | Store at 4 °C | ||
| High salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 600 mM NaCl) | Store at 4 °C | ||
| LiCl wash buffer (0.25 M LiCl; 1% NP-40; 1% Sodium Deoxycholate, 1 mM EDTA; 10 mM Tris pH 8.0) | Store at 4 °C | ||
| TE buffer (10 mM Tris, pH 7.4, 1 mM EDTA) | Store at 4 °C | ||
| Elution buffer (1% SDS; 0.1 M NaHCO3) | Prepare fresh | ||
| 5 M NaCl | Store at RT | ||
| 0.5 M EDTA | Store at RT | ||
| 1 M Tris-HCl, pH 6.5 | Store at RT | ||
| Table of Specific Reagents | |||
| Clay Adams Brand Nutator | Becton Dickinson | Model: 421105 | |
| Magnetic Stand | Promega | Z5342 | |
| Qiaquick PCR Purification Kit | Qiagen | 28106 | |
| Masonix Sonicator 3000 | QSonica | Model: S3000 | |
| UV Spectrophotometer | NanoDrop Technologies | ND-1000 | |
| Heating Block | VWR | 13259-030 | |
| Rotator | VWR | 80085-692 | |
| Refrigerated bench top centrifuge | Beckman Coulter | Model: Allegra X-12R | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Table of Equipment | |||
Referenzen
- Weinmann, A. S., Bartley, S. M., Zhang, T., Zhang, M. Q., Farnham, P. J. Use of chromatin immunoprecipitation to clone novel E2F target promoters. Molecular and Cellular Biology. 21, 6820-6832 (2001).
- Weinmann, A. S., Farnham, P. J. Identification of unknown target genes of human transcription factors using chromatin immunoprecipitation. Methods. 26, 37-47 (2002).
- Li, Q., et al. Constitutive nuclear localization of NFAT in Foxp3+ regulatory T cells independent of calcineurin activity. J. Immunol. 188, 4268-4277 (2012).
- Shakya, A., Kang, J., Chumley, J., Williams, M. A., Tantin, D. Oct1 is a switchable, bipotential stabilizer of repressed and inducible transcriptional states. The Journal of Biological Chemistry. 286, 450-459 (2011).
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Lubelsky, Y., Macalpine, H. K., Macalpine, D. M. Genome-wide localization of replication factors. Methods. 57, 187-195 (2012).
- O'Neill, L. P., VerMilyea, M. D., Turner, B. M. Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations. Nature Genetics. 38, 835-841 (2006).
- Sikes, M. L., et al. A streamlined method for rapid and sensitive chromatin immunoprecipitation. Journal of Immunological Methods. 344, 58-63 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nature Biotechnology. 26, 1351-1359 (2008).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten