Method Article
Efficiente immunoprecipitazione della cromatina utilizzando Limitare quantità di biomassa
In questo articolo
Riepilogo
Descriviamo un metodo robusto per immunoprecipitazione della cromatina utilizzando cellule T primarie. Il metodo si basa su approcci standard, ma utilizza uno specifico insieme di condizioni e reagenti che migliorano l'efficienza per una limitata quantità di cellule. Importante, una descrizione dettagliata della fase di analisi dei dati è presentato.
Abstract
Immunoprecipitazione della cromatina (ChIP) è un metodo ampiamente utilizzato per determinare le interazioni delle diverse proteine con il DNA in cromatina delle cellule viventi. Gli esempi includono sequenza-specifici di DNA binding fattori di trascrizione, istoni ed i loro diversi stati di modifica, enzimi come la RNA polimerasi e fattori accessori e componenti di riparazione del DNA. Nonostante la sua ubiquità, vi è una mancanza di up-to-date, metodologie dettagliate per entrambi preparazione banco di materiale e per l'analisi accurata permettendo metriche quantitative di interazione. A causa di questa mancanza di informazioni, e anche perché, come ogni immunoprecipitazione, le condizioni devono essere ri-ottimizzato per una nuova serie di condizioni sperimentali, il saggio di ChIP è suscettibile di risultati inesatti o mal quantitativa.
Il nostro protocollo deriva in ultima analisi dal lavoro seminale sul fattore di trascrizione: interazioni DNA 1,2, ma incorpora una serie di miglioramenti per sensibività e riproducibilità per difficili da ottenere i tipi di cellule. Il protocollo è stato usato con successo 3,4, sia utilizzando qPCR per quantificare arricchimento DNA, oppure utilizzando una variante semi-quantitativa del sottostante protocollo.
Questa analisi quantitativa di materiale amplificato per PCR viene eseguita computazionalmente, e rappresenta un fattore limitante nel saggio. Controlli importanti e altre considerazioni includono l'uso di un anticorpo isotipo-abbinato, così come la valutazione di una regione di controllo del DNA genomico, come ad esempio una regione intergenica predetto di non essere vincolata dalla proteina in studio (o anticipato di non mostrare i cambiamenti in le condizioni sperimentali). Inoltre, una curva standard di materiali di input per ogni campione ChIP è usato per derivare i livelli assoluti di arricchimento nel materiale sperimentale. Utilizzo di curve standard aiuta a tenere conto delle differenze tra le serie di primer, indipendentemente da come sono progettati con cura, e anche l'efficienza differirerenze in tutto il range di concentrazioni modello per un singolo set primer. Il nostro protocollo è diverso dagli altri che sono disponibili in 5-8 che coprono estensivamente la, fase successiva analisi.
Protocollo
1. Isolamento del mouse CD4 naïve splenica cellule T
- Sacrifica il mouse in maniera umana coerente con Institutional Animal Care e utilizzo Comitato protocolli (IACUC). Sezionare la milza e posizionarlo nella capsula di Petri contenente 10 ml di DMEM con 10% FBS.
- Schiacciare la milza con estremità glassati di due vetrini per liberare le splenociti. Trasferire la sospensione cellulare in un tubo da 15 ml.
- Raccogliere le cellule per centrifugazione a 200 xg (~ 1.200 rpm per una centrifuga clinica con un diametro tipico rotore) per 5 min a 4 ° C.
- Risospendere le cellule in 2 ml di tampone di lisi ACK globuli rossi, 1 min a temperatura ambiente (RT). Arrestare la reazione aggiungendo 8 ml di DMEM con FBS.
- Raccogliere le cellule per centrifugazione a 200 xg per 5 minuti a 4 ° C.
- Risospendere le cellule in 5 ml di DMEM contenente FBS e passare attraverso un filtro a maglia di 70 micron (BD Falcon, Cat # 352350). Contare le cellule e procedereper l'isolamento di cellule CD4 T naive CD4 cellule utilizzando un kit di isolamento (Miltenyi, Cat # 130-095-248) utilizzando le istruzioni del produttore. Raccogliere le cellule T CD4 naive per centrifugazione a 200 xg per 5 minuti a 4 ° C. Risospendere le cellule in 10 ml di DMEM con FBS.
2. Preparazione della cromatina
- Aggiungere 37% di formaldeide ad una concentrazione finale di 1% alla sospensione cellulare in DMEM e agitare delicatamente a TA (esempio usando un nutator) per 15 min di cross-link DNA: complessi proteici.
- Arrestare reticolazione aggiungendo 1 M glicina ad una concentrazione finale di 125 mM. Continuare al rock per 5 minuti a temperatura ambiente.
- Raccogliere le cellule per centrifugazione a 200 xg per 5 minuti a 4 ° C.
- Lavare le cellule da risospendere in 5 ml di ghiacciata PBS contenenti inibitori della proteasi. Lavare in PBS freddo inibitori della proteasi contenenti totale 3 volte e raccogliere le cellule per centrifugazione a 4 ° C.
- Risospendere le cellule in 1 mltampone ghiacciato lisi cellulare contenente inibitori di proteasi. Incubare in ghiaccio per 15 min. L'efficienza di lisi cellulare può essere determinato da risospendere una piccola aliquota di cellule in soluzione 0,4% trypan blue (Sigma, Cat. # T8154) e osservando con un microscopio.
- Raccogliere i nuclei mediante centrifugazione a 200 xg (tipicamente ~ 1.200 rpm) per 5 min a 4 ° C. Scartare con attenzione il surnatante.
- Risospendere i nuclei in 500 microlitri di tampone di lisi contenente inibitori delle proteasi nucleare. Incubare in ghiaccio per 15 min.
- Sonicare le cellule utilizzando un Sonicator Misonix 3000, sonda formato 1.6 mm: Livello di uscita 4, 15 sec scoppio, 4 volte su ghiaccio. Ogni campione deve essere raffreddato in ghiaccio per 1-2 min prima sonicating nuovamente per evitare il surriscaldamento dei campioni. Il surriscaldamento può causare l'inversione dei collegamenti trasversali.
- Centrifugare la cromatina sonicato a 16.000 g (circa 13.200 rpm in una microcentrifuga) per 5 minuti a 4 ° C.
- Prendete un 20 microlitri aliquota di chiara supernatant, aggiungere DNA colorante carico e controllare il DNA sonicato tramite elettroforesi su gel di agarosio al 2% (dimensione ideale del DNA sonicato per la maggior parte delle applicazioni è 200-500 bp).
- Determinare la concentrazione di DNA utilizzando uno spettrofotometro UV. Cromatina tosata può essere utilizzato immediatamente per impostare reazione cromatina immunoprecipitazione o conservato a -80 ° C. Tipicamente si ottiene 7,5-10 DNA mcg 2-3 x 10 6 cellule T CD4 purificate per topo.
3. Immunoprecipitazione della cromatina (Tutte le operazioni devono essere effettuate a 0-4 ° C)
- Diluire cromatina sonicato ad una concentrazione di DNA ug / ml 5-10 (volume totale = 1 ml) in tampone di diluizione chip con inibitori della proteasi.
- Salvare 100 microlitri (10%) come input. Conservare in ghiaccio.
- Aliquota 450 ml ciascuno in due 1,7 ml provette per microcentrifuga con etichetta come controllo isotipico (mouse o coniglio IgG) e anticorpi di interesse. Se vengono utilizzate più anticorpi dello stesso isotipo, un singolo contr isotipoolo sarà sufficiente effettuare questa analisi. Avrete bisogno di più controlli isotipo se vengono utilizzati gli anticorpi di origine animale o isotipo diverso.
- Aggiungere 2-5 mg di anticorpo specifico, a seconda della specificità dell'anticorpo utilizzato o controllo isotipico alle rispettive provette.
- Roccia i tubi usando una nutator notte a 4 ° C per permettere la formazione di complessi cromatina-anticorpo.
- Aggiungere 25 ml di proteina G biglie magnetiche (01:01 slurry di perline in sospensione) alla miscela sopra e permettere al rock per almeno 2 ore a 4 ° C. Perline da nostro fornitore possono essere utilizzati direttamente.
- Posizionare i tubi microcentrifuga su un supporto magnetico e consentono di raccogliere le perline sul lato magnetizzato.
- Rimuovere con cura la soluzione per aspirazione senza disturbare le perline.
- Aggiungere 1 ml di soluzione a basso sale lavare e far oscillare delicatamente per 5 minuti su una nutator. Raccogliere le perle utilizzando un supporto magnetico e rimuovere la soluzione di lavaggio. Ripetere una volta.
- Aggiungere 1 ml di soluzione di alto sale lavare e consentire al rock per 5 minuti su una nutator. Raccogliere le perline utilizzando il supporto magnetico e rimuovere la soluzione di lavaggio. Ripetere una volta.
- Aggiungere 1 ml di soluzione di lavaggio cloruro di litio e consentire al rock per 5 minuti su una nutator. Raccogliere le perline utilizzando il supporto magnetico e rimuovere la soluzione di lavaggio. Ripetere una volta. L'uso di LiCl migliora l'effettiva rimozione di interazioni cromatina non specifiche con le perline.
- Aggiungere 1 ml di soluzione di TE e consentire al rock per 5 minuti su una nutator. Raccogliere le perline utilizzando il supporto magnetico e rimuovere la soluzione di lavaggio.
- Eluire il DNA dalle perline aggiungendo 250 microlitri di tampone di eluizione. Roccia per 15 minuti a temperatura ambiente. Pipettare off l'eluato e salvare questo materiale in un nuovo 1,7 ml microcentrifuga. Ripetere ancora una volta e di combinare entrambi i eluizioni. Scartare le perline.
- Per invertire i legami incrociati aggiungono 5 M NaCl ad una concentrazione finale di 0,3 M e 1 ml di RNase A (20 m g / ml) per il DNA eluito. Aggiungere 400 microlitri di tampone di eluizione agli ingressi salvate nel passo 3.2, per fare un volume 500 microlitri. Agli ingressi aggiungere NaCl allo 0,3 M, 1 ml di RNasi A, 10 ml di 0,5 M EDTA, 20 ml di 1 M Tris-HCl pH 6.5 e 1 ml di proteinasi K (20 mg / ml).
- Incubare le provette notte a 65 ° C in un blocco di riscaldamento a secco. Per evitare l'evaporazione di campioni, sigillo o posizionare un peso sui tubi per impedire loro di apertura. Un forno di ibridazione può anche essere usato.
- Lasciate raffreddare le provette a temperatura ambiente. Aggiungere 1 ml di etanolo al 100% e incubare 2 hr-notte a -80 ° C al DNA precipitato.
- Centrifugare le provette a 16.000 xg per 15 min a pellet il DNA. Lavare il pellet di DNA una volta che con il 70% di etanolo e asciugare il pellet. Risospendere il pellet di DNA in 100 ml di acqua distillata autoclavato.
- Purificare DNA usando Qiaquick spin column ed eluire in volume totale di 50 microlitri di tampone di eluizione. Questo DNA è pronto all'uso per la PCR.
- Raccogliere tutti i campioni di DNA da campioni di chip di ingresso e corrispondenti. Inoltre, raccogliere tutti i reagenti PCR e primer per regione mirata e una regione di controllo. Utilizzare un "hotstart" Taq DNA polimerasi. Useremo un saggio di verde a base di SYBR per quantificare l'amplificazione del DNA in questa procedura, ma i kit qPCR MasterMix può essere utilizzato se necessario.
- Effettuare una diluizione seriale di DNA input per generare una curva standard nell'analisi qPCR: per esempio 10%, 1%, 0,1% e 0,01% in tampone di eluizione (dalle colonne di spin QIAquick a passo 3.18). L'intervallo di concentrazione e di incremento di questo modello di set di curva standard possono variare in base ai livelli attesi di arricchimento di chip, ecc Dispensare 10 ml di campioni di DNA di ingresso nella rispettivi pozzetti di una piastra a 96 pozzetti (Genemate, cat # T-3182-1 ), in duplice copia.
- Pipettare 10 ml DNA chip dal controllo isotipico, così come anticorpo specifico nella rispettivapozzi, in triplice copia.
- Fare un "master mix" che contiene sia primer bersaglio o primer di controllo (0.1 mM concentrazione finale di ciascun primer) ed erogare 10 microlitri in rispettivi pozzetti. Per esempio, nel modello di seguito dispensare Master Mix-primer contenente mirate nei pozzi marcato verde e dispensare master mix con primer di controllo in pozzetti contrassegnato rosso.
- Coprire la piastra PCR con ottica film plastico e centrifugare a 500 xg in una centrifuga clinica (~ 1.200 rpm) per 1 min a RT per raccogliere tutto al fondo del pozzo.
- Avviare la reazione di PCR. Useremo il LightCycler 480 II (Roche) operativo LightCycler 480SW 1.5 del software per eseguire il qPCR in questo esempio.
Pre-incubazione: 1 ciclo: 95 ° C / 5 min.
Amplificazione: 40-45 cicli: 94 ° C / 5 sec, 60 ° C / 5 sec, 72 ° C/10 sec (ricottura temperatura dovrebbe essere di 2-3 ° C inferiore alla temperatur fusionee dei primer).
Curva di fusione: 1 ciclo.
Raffreddamento: 1 ciclo.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| A | 10% Input | 10% Input | Isotipo | Specifico | ||||||||
| B | 1% Input | 1% Input | Isotipo | Specifico | ||||||||
| C | 0,1% Input | 0,1% Input | Isotipo | Specifico | ||||||||
| D | 0,01% Input | 0,01% Input | ||||||||||
| E | 10% Input | 10% Input | Isotipo | Specifico | ||||||||
| F | 1% Input | 1% Input | Isotipo | Specifico | ||||||||
| G | 0,1% Input | 0,1% Input | Isotipo | Specifico | ||||||||
| H | 0,01% Input | 0,01% Input |
Verde: Utilizzare regione primer mirati.
Rosso: Utilizzare il controllo regione primer.
5. Analisi
- Programmare il software qPCR per la quantificazione della quantità assoluta di DNA. Importante, l'uso di una curva standard per interpolare i valori di DNA nei campioni specifici e isotipo sconosciuti consente la valutazione e la congruenza della qualità e l'efficienza di tutti Primer tramonta oltre l'intervallo di concentrazioni riscontrate nei campioni. Inoltre, fornisce anche una more affidabile metodo per convertire i valori Cq (C t o C p) ai risultati di arricchimento volte rispetto finali di usingΔΔCq e relativi metodi.
- Andare all'editor campione e segnare i pozzetti contenenti campioni di ingresso di varie diluizioni (10%, 1%, 0,1% e 0,01%) con i loro valori della curva standard. Etichettare anche i pozzetti con campioni di chip sconosciuti, esattamente come la piastra PCR è disposto. Nel software equivalente utilizzato in altre macchine qPCR, designare sottoinsiemi, corrispondenti alle reazioni per ogni coppia di primer, valore curva standard e sperimentali e isotipo campione di controllo di conseguenza.
- Nel software LightCycler, andare all'editor sottoinsieme e segnare i sottoinsiemi tra cui i pozzetti con i campioni di ingresso, nonché i campioni di chip sconosciuti. I campioni sconosciuti saranno quantificati utilizzando la curva standard generata dalle concentrazioni note di campioni di ingresso per l'analisi. Nel software equivalente utilizzato in altre macchine qPCR, designato sottoinsiemi corrisponding a tutte le reazioni per ogni coppie di primer.
- Dopo l'amplificazione PCR è completa, eseguire l'analisi con "Abs Quant/2nd Derivata Max" e selezionare il sottoinsieme di analisi e premere il tasto OK. Nel software equivalente utilizzato in altre macchine qPCR, convertire il valore Cq alla quantità di DNA in ciascuna sezione.
- Premere il tasto "Calcola" che genererà la curva standard utilizzando le concentrazioni note di campioni di ingresso e visualizza la quantità assoluta di DNA presente nei campioni sconosciuti.
- Se la qualità del DNA tranciata è buona, e se i primer si legano specificamente alla regione mirata, efficienza della PCR dovrebbe essere vicino a 2.
- Effettuare una analisi della curva di melting con "Tm Calling" per lo stesso sottoinsieme, se disponibile. Un picco singolo indica che solo un prodotto specifico è stato amplificato. L'amplificazione di DNA specifica può essere provato elettroforesi il prodotto di PCR insieme con scaletta DNA attraverso un gel di agarosio.
- Esportare i dati come una scheda-file di testo delimitato, che può essere aperto in Microsoft Excel per ulteriori analisi.
- Utilizzo di Microsoft Excel, dividere la quantità di DNA per l'anticorpo specifico con il controllo isotipico per fondi mirati. Ripetere questo passaggio per la Regione primer di controllo. Se più anticorpi dello stesso isotipo sono utilizzati per diverse immunoprecipitazione, normalizzare ciascuna di queste con i valori dello stesso isotipo di controllo. Inoltre, se si utilizzano più coppie di primer mirati, le quantità di DNA da un controllo singolo innesco pair set dovrebbero essere utilizzati per la normalizzazione di ciascun target (figure 1 e 2). I valori di uscita rappresentano lo specifico anticorpo immunoprecipitazione arricchimento piega in ogni posizione rispetto al non-specifico fondo immunoprecipitazione anticorpo.
- Dividere l'arricchimento piega (a isotipo di controllo) per i primer mirate con l'arricchimento piega (a isotipo di controllo) per il controllo regione primer per ottenere l'arricchimento rispetto ad unaregione non legato di controllo (Figura 1). È importante sottolineare che, notare che a causa della generazione di curve standard con il DNA totale di ingresso per ogni campione, ciascuno dei valori di cui sopra immunoprecipitazione interpolati dalle curve standard sono già normalizzato a, ed espressa come, la frazione del totale di ingresso dal software della macchina.
- Se la severità delle buffer utilizzati per immunoprecipitazione o lavaggio è troppo alta, è possibile che l'amplificazione robusta da campioni immunoprecipitati con anticorpi specifici sarà osservato, mentre l'amplificazione dal controllo isotipico immunoprecipitato non saranno osservati campioni. In un tale scenario, è meglio sottrarre la quantità di chip di controllo isotipo da ChIP anticorpo specifico sia per la regione di destinazione e la regione non legato. Arricchimento relativa può essere calcolata dividendo la differenza per la regione di destinazione per differenza per regione non associato (Figura 3).
Risultati
L'immunoprecipitazione della cromatina (ChIP) protocollo presentato qui i controlli per le differenze, se del caso, nella quantità di DNA utilizzati in PCR mediante l'utilizzo di una coppia di primer che amplifica una regione legata di genoma, quindi servire come un "controllo del carico". Nell'esempio illustrato nella figura 3, abbiamo utilizzato la regione codificante del gene ACTB topo come una regione non legato alla nostra proteina di interesse e del sito di legame NFAT fattore di trascrizione su topo Il2 promotore come regione bersaglio. In alternativa, una regione diversi kilobases monte oa valle della regione di destinazione nota per avere alcun legame della proteina di interesse possono essere scelti per questo scopo. Quando si progetta una coppia di primer per la regione di destinazione, la dimensione ideale del prodotto di PCR è di circa 100-200 bp. E 'anche importante verificare computazionalmente che questi primer non vincolano in nessun'altra parte del genoma.
Arricchimento relativo di una proteina legata alla differenti regioni del genoma possono essere confrontati se lo stesso insieme di isotipo di controllo e di anticorpi specifici sono scelti. Arricchimento delle varie proteine sulla stessa regione bersaglio dipende dalla specificità ed affinità dell'anticorpo utilizzato così come la quantità di proteina specifico legato al DNA. Per esempio, l'arricchimento relativo nel caso di chip eseguite per istoni restituirà in genere un arricchimento valore più elevato rispetto ad un fattore di trascrizione che può legarsi al DNA in risposta a vari segnali cellulari. Se la specificità dell'anticorpo è buona, di solito osserviamo una piega arricchimento 2-10 sulla regione di controllo non associato (figure 3 e 4).
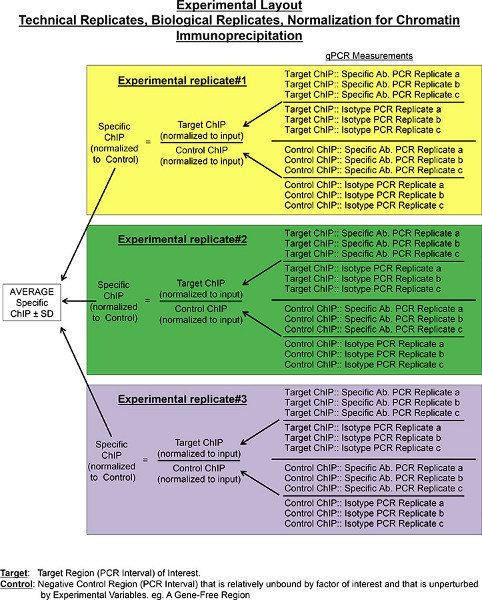
Figura 1. Layout Sperimentale descrivendo i calcoli per ottenere relaive arricchimento di chip per l'anticorpo specifico al sito di destinazione. PCR quantitativa viene utilizzata per ottenere quantità di DNA nei campioni di chip immunoprecipitata utilizzando un anticorpo specifico e il suo controllo isotipico. PCR quantitativa viene effettuata utilizzando primer che abbracciano sequenza bersaglio e regione di controllo non associato genoma. Per ogni campione di chip, PCR viene eseguita in triplice copia, indicati come tecnico replica a, b, c, e relativo arricchimento viene calcolato per ogni replica, come spiegato nel protocollo. Vengono eseguiti i calcoli per i valori di output tecnici medi. Varianza può anche essere analizzato in questa fase, e idealmente dovrebbe essere basso. I calcoli errore tecnico però non sono inclusi nella formula finale per il calcolo di errore tra biologico replicati. Piuttosto, la variazione biologica misura in sé anche contiene intrinsecamente la variante tecnico. Tre sperimentale replica (in scatole di colore giallo, verde e viola) sono utilizzati per esperimenti di ChIP per ottenere un avsegnale ChIP erage e la sua deviazione standard (SD). Clicca qui per ingrandire la figura .

Figura 2. Metodi alternativi di calcolo di arricchimento chip. Nei casi in cui la quantità di DNA immunoprecipitata dal isotipo è estremamente basso e qPCR amplificazione è scarsa, questa quantità di DNA possono essere sottratti dalla quantità di DNA immunoprecipitati utilizzando un anticorpo specifico per ottenere la piega arricchimento per ogni replicare tecnica. Tale metodo di calcolo deve essere utilizzato in tutte e tre repliche sperimentali di ottenere un segnale medio del truciolo e la sua deviazione standard (SD), come descritto in Figura 1.

Figura 3. Istantanea di Microsoft Excel foglio di calcolo che mostra i calcoli per ottenere relativo arricchimento. Dati da tre repliche sperimentali (in scatole di colore giallo, verde e viola) con tre tecniche replicati ciascuno è stato utilizzato per calcolare l'arricchimento piega. Utilizzo del protocollo di calcolo descritto nella figura 1 è rappresentato in questo foglio di calcolo. Clicca qui per ingrandire la figura .

Figura 4. Arricchimento relativo di fattore di trascrizione NFAT in IL2promotore. CD4 convenzionali cellule T (TCON) e CD4 cellule T regolatorie (Treg) sono stati isolati da topi Foxp3EGFP. Una piccola frazione di cellule TCON è stata stimolata con PMA e ionomicina per 30 min. ChIP è stata eseguita come descritto nel protocollo sperimentale. I dati qui presentati sono da tre repliche sperimentali con tre tecniche replicati ciascuno. Arricchimento relativo di fattori di trascrizione NFATc1 e NFATc2 sul mouse IL2 promotore in questi tipi di cellule è raffigurato come arricchimento volte nel corso di una regione non legato 3.
Discussione
Il protocollo di cui sopra fornisce un metodo robusto di quantificare con precisione l'arricchimento del DNA da linfociti primari che utilizzano chip. Un importante motivo per la robustezza in questo protocollo è l'inserimento di biologico replica. Il protocollo precedente utilizza tre repliche, l'arricchimento per il quale viene calcolato in modo indipendente. Le uscite vengono quindi mediati per fornire un grado di arricchimento e deviazioni standard calcolate per fornire una misura della variabilità. Per ogni campione, tre repliche tecnico vengono eseguiti anche per eliminare variazione nella manipolazione dei campioni, amplificazione PCR, ecc La quantità di variazione nelle ripetizioni tecniche è in genere molto inferiore a quello osservato tra diversi campioni biologici. Una seconda ragione per robustezza è l'inclusione di più controlli tecnici, compresi anticorpi isotipo-matched controllo, l'uso di una curva standard di DNA di ingresso, e l'uso di un amplicone di controllo corrispondente ad una regione genomica non specifico.
Jove_content Limiti del metodo di cui sopra sono simili a quelle incontrate in genere utilizzando chip. Il metodo soffre alquanto scarsa risoluzione spaziale, il fattore limitante essendo la tranciatura di frammenti di DNA mediante sonicazione. Pertanto, il potere risolutivo del metodo è solo di circa 500 bp di DNA. Gli esperimenti che richiedono la risoluzione più alta possibile fare uso di nucleasi micrococcica digestione 8, o exonuclease digestione dei frammenti recuperati (ChIP-exo) 9,10. Inoltre, i preparativi ChIP sopra descritti accoppiato a high-throughput sequencing dei frammenti recuperati (ChIPseq, ChIP-seq o ChIP-Seq) 11,12 al posto di qPCR, produce un campionamento di diverso tranciati frammenti arricchiti, l'intersezione dei quali possono essere preso come un luogo in buona fede di interazione. Tuttavia, il metodo qui presentato fornisce un modo affidabile di cura e precisione quantificando associazione DNA con fattori di trascrizione, componenti nucleosomi, modficati istoni e di altre proteine in siti specifici di interesse.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni NIH CA141009 e GM39067. Ringraziamo E. Parnell e R. Yarrington per i commenti sulla parte scritta.
Materiali
| Name | Company | Catalog Number | Comments |
| Formaldehyde | Sigma | F-8775 | Store at RT |
| Phosphate Buffered Saline | Hyclone | SH30256.01 | Store at 4 °C |
| Protease Inhibitor tablets | Roche | 04693116001 | Store at 4 °C |
| Protein G magnetic beads | Active Motif | 101945 | Store at 4 °C |
| RNase A (20 mg/ml) | EMD Millipore | 556746 | Store at -20 °C |
| Proteinase K (20 mg/ml) | Roche | 03115879001 | Dissolve in 50 mM Tris-HCl, 10 mM CaCl2, pH 8.0 |
| Platinum Taq DNA Polymerase | Invitrogen | 10966-034 | Store at -20 °C |
| SYBR Green I | Invitrogen | S7567 | Store at -20 °C |
| 1 M Glycine | Store at RT | ||
| Cell lysis buffer (5 mM Pipes, pH 8.0; 85 mM KCl; 0.5% NP-40) | Store at 4 °C | ||
| Nuclear lysis buffer (50 mM Tris, pH 8.1; 10 mM EDTA; 1% SDS) | Store at RT | ||
| ChIP dilution buffer (0.01% SDS; 1.1% Triton X-100; 1.2 mM EDTA; 16.7 mM Tris pH 8.1; 190 mM NaCl) | Store at 4 °C | ||
| Low salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 150 mM NaCl) | Store at 4 °C | ||
| High salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 600 mM NaCl) | Store at 4 °C | ||
| LiCl wash buffer (0.25 M LiCl; 1% NP-40; 1% Sodium Deoxycholate, 1 mM EDTA; 10 mM Tris pH 8.0) | Store at 4 °C | ||
| TE buffer (10 mM Tris, pH 7.4, 1 mM EDTA) | Store at 4 °C | ||
| Elution buffer (1% SDS; 0.1 M NaHCO3) | Prepare fresh | ||
| 5 M NaCl | Store at RT | ||
| 0.5 M EDTA | Store at RT | ||
| 1 M Tris-HCl, pH 6.5 | Store at RT | ||
| Table of Specific Reagents | |||
| Clay Adams Brand Nutator | Becton Dickinson | Model: 421105 | |
| Magnetic Stand | Promega | Z5342 | |
| Qiaquick PCR Purification Kit | Qiagen | 28106 | |
| Masonix Sonicator 3000 | QSonica | Model: S3000 | |
| UV Spectrophotometer | NanoDrop Technologies | ND-1000 | |
| Heating Block | VWR | 13259-030 | |
| Rotator | VWR | 80085-692 | |
| Refrigerated bench top centrifuge | Beckman Coulter | Model: Allegra X-12R | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Table of Equipment | |||
Riferimenti
- Weinmann, A. S., Bartley, S. M., Zhang, T., Zhang, M. Q., Farnham, P. J. Use of chromatin immunoprecipitation to clone novel E2F target promoters. Molecular and Cellular Biology. 21, 6820-6832 (2001).
- Weinmann, A. S., Farnham, P. J. Identification of unknown target genes of human transcription factors using chromatin immunoprecipitation. Methods. 26, 37-47 (2002).
- Li, Q., et al. Constitutive nuclear localization of NFAT in Foxp3+ regulatory T cells independent of calcineurin activity. J. Immunol. 188, 4268-4277 (2012).
- Shakya, A., Kang, J., Chumley, J., Williams, M. A., Tantin, D. Oct1 is a switchable, bipotential stabilizer of repressed and inducible transcriptional states. The Journal of Biological Chemistry. 286, 450-459 (2011).
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Lubelsky, Y., Macalpine, H. K., Macalpine, D. M. Genome-wide localization of replication factors. Methods. 57, 187-195 (2012).
- O'Neill, L. P., VerMilyea, M. D., Turner, B. M. Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations. Nature Genetics. 38, 835-841 (2006).
- Sikes, M. L., et al. A streamlined method for rapid and sensitive chromatin immunoprecipitation. Journal of Immunological Methods. 344, 58-63 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nature Biotechnology. 26, 1351-1359 (2008).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon