Method Article
Imunoprecipitação da Cromatina eficiente usando Limitando quantidades de biomassa
Neste Artigo
Resumo
Nós descrevemos um método robusto para cromatina imunoprecipitação utilizando células T primárias. O método baseia-se em métodos normais, mas utiliza um conjunto específico de condições e reagentes que melhoram a eficiência de uma quantidade limitada de células. Importante, é apresentada uma descrição detalhada da fase de análise de dados.
Resumo
Cromatina imunoprecipitação (CHIP) é um método amplamente utilizado para determinar as interações entre diferentes proteínas com o DNA na cromatina de células vivas. Exemplos incluem sequências específicas de DNA de ligação fatores de transcrição, histonas e seus diferentes estados de modificação, enzimas como RNA polimerases e fatores auxiliares e componentes de reparação do ADN. Apesar de sua onipresença, há uma falta de up-to-date, metodologias detalhadas para ambos banco preparação do material e para a análise precisa permitindo métricas quantitativas de interação. Devido a esta falta de informação, e também porque, como qualquer imunoprecipitação, as condições devem ser re-otimizado para novos conjuntos de condições experimentais, o ensaio chip é suscetível a resultados imprecisos ou mal quantitativa.
Nosso protocolo é em última análise, derivada do trabalho seminal no fator de transcrição: interações DNA 1,2, mas incorpora uma série de melhorias para sensibilizantesvidade e reprodutibilidade para os tipos de células de difícil obtenção. O protocolo foi utilizado com sucesso 3,4, ambos utilizando qPCR para quantificar o enriquecimento do ADN, ou uma variante usando semi-quantitativa do protocolo abaixo.
Esta análise quantitativa de material amplificado por PCR é realizada computacionalmente, e representa um factor limitante no ensaio. Controlos e outras considerações importantes incluem a utilização de um isotipo do anticorpo, bem como a avaliação de uma região do ADN genómico de controlo, tal como uma região intergénica não predito para ser ligado pela proteína sob estudo (ou antecipados não apresentam alterações ao abrigo as condições experimentais). Além disso, uma curva padrão de material de entrada para cada amostra de chip é usada para determinar os níveis absolutos de enriquecimento do material experimental. Uso de curvas padrão ajuda a levar em conta as diferenças entre conjuntos de primers, independentemente de como eles são projetados com cuidado, e também a eficiência diferemcias em toda a gama de concentrações de modelo para um único conjunto de primers. Nosso protocolo é diferente de outros que estão disponíveis 5-8 em que extensivamente cobrir a, fase posterior análise.
Protocolo
1. Isolamento de mouse Esplenopatias Naïve células T CD4
- Sacrificar o mouse em uma forma humana consistente com o uso do Comité (IACUC) protocolos institucionais Cuidados e Animal. Dissecar o baço e colocá-lo em uma placa de Petri contendo 10 ml de DMEM com 10% de FBS.
- Esmagar o baço usando fosco extremidades de duas lâminas de vidro para liberar os esplenócitos. Transferência da suspensão de células num tubo cónico de 15 ml.
- Recolher as células por centrifugação a 200 xg (~ 1200 rpm durante uma centrífuga clínica típica, com um diâmetro do rotor) durante 5 min a 4 ° C.
- Ressuspender as células em 2 ml de tampão ACK para lisar as células vermelhas do sangue, 1 min, à temperatura ambiente (RT). Parar a reacção pela adição de 8 ml de DMEM com FBS.
- Recolher as células por centrifugação a 200 xg durante 5 min a 4 ° C.
- Ressuspender as células em 5 ml de DMEM contendo FBS e passar através de um filtro de malha de 70 (BD Falcon, Cat # 352350). Contar as células e procederpara o isolamento de células T CD4 + naive, utilizando um kit de isolamento de CD4 (Miltenyi, Cat # 130-095-248), utilizando as instruções do fabricante. Recolher as células T CD4 + naive por centrifugação a 200 xg durante 5 min a 4 ° C. Ressuspender as células em 10 ml de DMEM com FBS.
2. Preparação da cromatina
- Adicionar 37% de formaldeído até uma concentração final de 1% para a suspensão de células em DMEM e agitar suavemente à temperatura ambiente (por exemplo, usando um Nutator) durante 15 minutos para reticular ADN: proteína.
- Parar de reticulação por adição de 1 M de glicina para uma concentração final de 125 mM. Continuar a agitar durante 5 min à TA.
- Recolher as células por centrifugação a 200 xg durante 5 min a 4 ° C.
- Lave as células por ressuspensão em 5 ml de inibidores de protease de gelados contendo PBS. Lavar com inibidores de PBS gelado contendo protease 3 vezes no total e recolher as células por centrifugação a 4 ° C.
- Ressuspender as células em 1 mltampão de lise celular gelado contendo inibidores de protease. Incubar no gelo por 15 min. A eficiência da lise das células pode ser determinada por ressuspensão uma pequena alíquota de células em solução de 0,4% de azul de tripano (Sigma, Cat # T8154) e observando-se com um microscópio.
- Recolher os núcleos por centrifugação a 200 xg (tipicamente ~ 1200 rpm) durante 5 min a 4 ° C. Descartar cuidadosamente o sobrenadante.
- Ressuspender o núcleos em 500 ul de tampão de lise nuclear contendo inibidores de protease. Incubar no gelo por 15 min.
- Sonicate as células usando um Sonicator Misonix 3000, a sonda tamanho 1,6 milímetros: Nível de saída 4, 15 seg explosão, quatro vezes no gelo. Cada amostra deve ser resfriado em gelo por 1-2 minutos antes sonicando-lo novamente para evitar o superaquecimento das amostras. O sobreaquecimento pode provocar a reversão das ligações transversais.
- Centrifugar a cromatina sonicado a 16.000 xg (~ 13.200 rpm numa microcentrífuga) durante 5 min a 4 ° C.
- Dê uma alíquota de 20 mL de supern claroatant, adicionar corante de carga de ADN e verificar o DNA sonicado por electroforese através de um gel de agarose a 2% (tamanho ideal do DNA sonicado para a maioria das aplicações é de 200-500 pb).
- Determinar a concentração de ADN, utilizando um espectrofotómetro de UV. Cromatina cortado pode ser usado imediatamente para configurar reação de imunoprecipitação da cromatina ou armazenadas a -80 ° C. Normalmente obtemos 7,5-10 DNA mg 2-3 x 10 6 células T CD4 purificadas por mouse.
3. Cromatina imunoprecipitação (todos os passos devem ser realizados a 0-4 ° C)
- Diluir cromatina sonicado a uma concentração de ADN de ug / ml 5-10 (volume total = 1 ml) em tampão de diluição de chip com os inibidores da protease.
- Guardar 100 ul (10%) como entrada. Guarde no gelo.
- Alíquota de 450 mL em cada dois tubos de 1,7 ml de microcentrífuga rotulados como controle de isotipo (rato ou coelho IgG) e anticorpos de interesse. Se forem usados vários anticorpos do mesmo isotipo, um único isotipo control serão suficientes para realizar esta análise. Você vai precisar de vários controles isotipo se os anticorpos de uma fonte animal diferente ou isotipo são usados.
- Adicionar 2-5 ug de um anticorpo específico, dependendo da especificidade do anticorpo utilizado, ou controlo de isotipo para os respectivos tubos.
- Balançar os tubos utilizando uma Nutator a noite a 4 ° C para permitir a formação de complexos anticorpo-cromatina.
- Adicionar 25 ul de proteína G esferas magnéticas (01:01 suspensão dos grânulos em suspensão) à mistura acima e deixa-se agitar durante pelo menos 2 horas a 4 ° C. Beads do nosso fornecedor pode ser usado diretamente.
- Colocar os tubos de microcentrífuga com um suporte magnético e permitem que as esferas se recolher no lado magnetizado.
- Remova cuidadosamente a solução por aspiração, sem perturbar as contas.
- Adicionar 1 ml de solução de lavagem baixo sal e deixe balançar suavemente durante 5 min em um Nutator. Recolher os grânulos utilizando um suporte magnético e remover a solução de lavagem. Repetir uma vez.
- Adicionar 1 ml de solução de lavagem de alto sal e deixa-se agitar durante 5 minutos num Nutator. Recolher os grânulos utilizando o suporte magnético e remover a solução de lavagem. Repetir uma vez.
- Adicionar 1 ml de solução de lavagem de cloreto de lítio e deixa-se agitar durante 5 minutos num Nutator. Recolher os grânulos utilizando o suporte magnético e remover a solução de lavagem. Repetir uma vez. A utilização de LiCl melhora a remoção eficaz de cromatina interacções não específicas com os grânulos.
- Adicionar 1 ml de solução TE e deixar agitar durante 5 min num Nutator. Recolher os grânulos utilizando o suporte magnético e remover a solução de lavagem.
- Eluir o ADN a partir das pérolas por adição de 250 ul de tampão de eluição. Rocha por 15 min em temperatura ambiente. Pipeta fora o fluido e guardar este material em um novo microtubo de 1,7 ml. Repita mais uma vez e combinar ambos os eluições. Descarte as contas.
- Para inverter as ligações cruzadas adicionar 5 M de NaCl a uma concentração final de 0,3 M e 1 ml de RNase A (20 m g / ml) para o DNA eluído. Adicionar 400 ul de tampão de eluição, para as entradas de salvas no passo 3.2, até perfazer o volume de 500 uL. Para adicionar as entradas de NaCl a 0,3 M, 1 ml de RNase A, 10 ul de EDTA 0,5 M, 20 ul de 1 M de Tris-HCl a pH 6,5 e 1 jil de proteinase K (20 mg / ml).
- Incubar os tubos durante a noite a 65 ° C num bloco de aquecimento a seco. Para evitar a evaporação de amostras, um selo ou colocar um peso sobre os tubos para mantê-los a partir da abertura. Um forno de hibridação podem também ser usados.
- Deixe os tubos legais para RT. Adicionar 1 ml de etanol a 100% e incubar 2 horas durante a noite, à temperatura de -80 ° C para precipitar o ADN.
- Centrifugar os tubos a 16.000 xg durante 15 min para sedimentar o ADN. Lava-se a pelete de ADN uma vez com etanol a 70% e secar ao ar o sedimento. Ressuspender o sedimento de DNA em 100 ul de água destilada autoclavada.
- Purifica-se o ADN utilizando colunas QIAquick e elui no volume total de 50 ul de tampão de eluição. Este ADN está pronto para utilização para a PCR.
- Recolha todas as amostras de DNA de amostras de entrada chip e correspondente. Além disso, a recolha de todos os reagentes e os iniciadores de PCR para a região alvo, bem como uma região de controlo. Usar um "HotStart" Taq DNA polimerase. Vamos utilizar um ensaio à base de SYBR Green para quantificar a amplificação do ADN, neste procedimento, mas qPCR mastermix kits podem ser usadas se necessário.
- Fazer uma diluição em série de ADN de entrada para gerar uma curva-padrão na análise de qPCR: por exemplo, 10%, 1%, 0,1% e 0,01% em tampão de eluição (a partir das colunas QIAquick na etapa 3.18). A gama de concentrações e de incremento deste conjunto de molde da curva padrão pode variar com base nos níveis esperados de enriquecimento de chip, etc Dispensar 10 pi de amostras de ADN de entrada nos respectivos poços de uma placa de 96 poços (Genemate, cat # T-3182-1 ), em duplicata.
- Pipetar 10 ml de DNA de ChIP de controlo de isotipo, bem como um anticorpo específico no respectivopoços, em triplicado.
- Faça uma "master mix", contendo tanto primers alvo ou primers de controle (0,1 mM concentração final de cada primer) e dispensar 10 ml em respectivos poços. Por exemplo, no modelo abaixo dispensar mistura mestre contendo iniciadores específicas em poços de cor verde e dispensar mistura principal com os iniciadores de controlo nos poços marcados a vermelho.
- Cobrir a placa com película de PCR de vedação de plástico óptico e centrifugar a 500 xg numa centrifugadora clínica (~ 1200 rpm) durante 1 min à temperatura ambiente para recolher tudo na parte inferior do poço.
- Iniciar a reacção da PCR. Usaremos o LightCycler 480 II (Roche) operando 480SW 1.5 software LightCycler para executar o qPCR neste exemplo.
Pré-incubação: um ciclo: 95 ° C / 5 min.
Amplificação: 40-45 ciclos de: 94 ° C / 5 seg, 60 ° C / 5 seg, 72 ° C/10 seg (a temperatura de recozimento deve ser de 2-3 ° C a menos do que a temperatur fusãoe um dos iniciadores).
Curva de fusão: 1 ciclo.
Refrigeração: 1 ciclo.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| A | 10% de entrada | 10% de entrada | Isotype | Específico | ||||||||
| B | 1% de Entrada | 1% de Entrada | Isotype | Específico | ||||||||
| C | 0,1% de Entrada | 0,1% de Entrada | Isotype | Específico | ||||||||
| D | 0,01% de entrada | 0,01% de entrada | ||||||||||
| E | 10% de entrada | 10% de entrada | Isotype | Específico | ||||||||
| F | 1% de Entrada | 1% de Entrada | Isotype | Específico | ||||||||
| G | 0,1% de Entrada | 0,1% de Entrada | Isotype | Específico | ||||||||
| H | 0,01% de entrada | 0,01% de entrada |
Verde: Use primers região de destino.
Vermelho: Use o controle de primers região.
5. Análise
- Programa do software qPCR para a quantificação das quantidades absolutas do ADN. Mais importante, a utilização de uma curva padrão para interpolar quantidades de ADN nas amostras específicas de isotipo e desconhecidos permite a avaliação e combinando da qualidade e eficiência de todo o iniciador se ajusta sobre a gama de concentrações encontradas nas amostras. Além disso, também proporciona uma more método fiável para converter os valores Cq (C ou C t p) para enriquecimento relativo resulta de dobragem finais que usingΔΔCq e métodos relacionados.
- Ir para o editor de amostra e marcam os poços que contêm as amostras de entrada de várias diluições (10%, 1%, 0,1% e 0,01%), com os valores da curva padrão. Rotular também os poços com amostras de chip desconhecidos, exatamente como a placa de PCR é colocado para fora. Em software equivalente utilizado em outras máquinas qPCR, designar subconjuntos correspondentes às reações para cada par de primers, o valor de curva padrão e amostra de controlo experimental e isotipo conformidade.
- No software LightCycler, ir para o editor subconjunto e marcar os subconjuntos, incluindo os poços com amostras de entrada, bem como as amostras de aparas de desconhecidos. As amostras desconhecidas será quantificado utilizando a curva padrão gerada a partir das concentrações conhecidas de amostras de entrada para a análise. Em software equivalente utilizado em outras máquinas qPCR, designado subconjuntos corresponding para todas as reacções para cada um dos pares de iniciadores.
- Após a amplificação por PCR é completa, realizar a análise com "Abs Quant/2nd Derivative Max" e selecionar o subconjunto para análise e pressione OK. No software equivalente utilizado noutras máquinas de qPCR, converter o valor Cq à quantidade de ADN em cada um dos subconjuntos.
- Pressione "Calcular" que irá gerar a curva padrão utilizando concentrações conhecidas de amostras de entrada e mostrará que a quantidade absoluta de ADN presente nas amostras desconhecidas.
- Se a qualidade do DNA cortado é bom, e se os iniciadores ligar-se especificamente à região alvo, a eficiência da PCR deveria ser próximo de 2.
- Realizar uma análise de curva de fusão com "Tm Calling" para o mesmo subconjunto, se disponível. Um único pico indica que apenas um produto específico foi amplificado. A amplificação de DNA específica pode ser também testada por electroforese do produto de PCR, juntamente com a escada de DNA através de um gel de agarose.
- Exportar dados como um guia-arquivo de texto delimitado, que pode ser aberto no Microsoft Excel para análise posterior.
- Usando o Microsoft Excel, dividir a quantidade de DNA para o anticorpo específico com o controle de isotipo de primers específicos. Repita este passo para os primers da região de controle. Se forem usados vários anticorpos do mesmo isotipo para diferentes imunoprecipitações, normalizar cada uma delas com os valores do mesmo controlo de isotipo. Além disso, se os pares de iniciadores múltiplos orientados são utilizadas, as quantidades de ADN a partir de um único conjunto de pares iniciadores de controlo deve ser utilizada para a normalização de cada alvo (Figuras 1 e 2). Os valores de saída representa o anticorpo imunoprecipitação enriquecimento vezes em cada local específico em relação ao fundo de imunoprecipitação anticorpo não específico.
- Divida o enriquecimento vezes (para isotipo controle) para primers alvo, com o enriquecimento de vezes (para isotipo controle) para controle de primers região para obter enriquecimento relativo a umaregião não acoplado controlo (Figura 1). Importante, notar que por causa da geração de curvas padrão com o ADN total de entrada para cada amostra, cada um dos valores de imunoprecipitação acima interpolada a partir das curvas padrão já estão normalizados para, e expressa como a fracção do total de entrada de pelo software da máquina.
- Se o rigor dos tampões utilizados para imunoprecipitação ou lavagem é demasiado alta, é possível que a amplificação a partir de amostras robusto imunoprecipitadas com anticorpos específicos pode ser observada, enquanto que a amplificação a partir de amostras de controlo de isotipo imunoprecipitadas não serão observadas. Em tal cenário, é melhor para subtrair a quantidade de chip de controle Isotype de chip anticorpo específico, tanto para a região alvo ea região não ligado. Enriquecimento relativa pode ser calculada dividindo a diferença para a região alvo, por diferença para a região não ligado (Figura 3).
Resultados
A cromatina imunoprecipitação (CHIP) protocolo apresentado aqui controles para as diferenças, se houver, na quantidade de DNA utilizado na PCR através da utilização de um par de primers que amplifica uma região do genoma não ligado, servindo, assim, como um "controle de carga". No exemplo mostrado na Figura 3, utilizou-se a região de codificação do gene de rato ACTB como uma região não acoplada para a proteína de interesse e o local de ligação ao factor de transcrição NFAT no rato Il2 promotora como a região alvo. Alternativamente, uma região várias quilobases a montante ou a jusante da região de destino conhecida por ter nenhuma ligação da proteína de interesse podem ser escolhidos para essa finalidade. Ao projetar um par de primers para a região de destino, o tamanho ideal do produto da PCR é de aproximadamente 100-200 pb. Também é importante para verificar a computacionalmente que estes primers não se ligam a qualquer outro lugar no genoma.
Enriquecimento relativo de uma proteína ligada a different regiões do genoma podem ser comparadas, se o mesmo conjunto de controlo de isotipo e os anticorpos específicos são escolhidos. Enriquecimento de várias proteínas na mesma região-alvo vai depender da especificidade e afinidade do anticorpo utilizado, bem como a quantidade de proteína ligada a ADN específica. Por exemplo, o enriquecimento relativo, no caso de batatas fritas realizadas para histonas tipicamente devolver um valor de enriquecimento mais elevado quando comparado com um factor de transcrição que se pode ligar ao ADN em resposta a vários sinais celulares. Se a especificidade do anticorpo é bom, que tipicamente observar um enriquecimento de 2-10 vezes em relação à região de controlo não acoplado (Figuras 3 e 4).
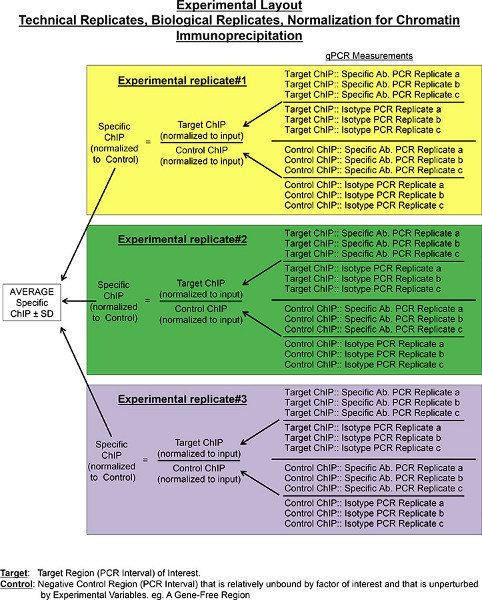
Figura 1. Delineamento experimental descrevendo cálculos para obter relative enriquecimento de chip para anticorpo específico no sítio alvo. PCR quantitativa é usado para obter quantidades de DNA em amostras de aparas de imunoprecipitados utilizando um anticorpo específico e o seu controlo de isotipo. PCR quantitativo é realizada utilizando primers que abrangem seqüência alvo e região de genoma controlo independente. Para cada amostra de chip, a PCR é realizada em triplicado, indicado como técnica replica a, b e c, e enriquecimento relativo é calculado para cada repetição, tal como explicado no protocolo. Cálculos para valores de saída técnicos médios são executadas. Variância também pode ser analisada a este passo, e, idealmente, deve ser baixa. Os cálculos de erro técnico, contudo, não estão incluídos na fórmula final para o cálculo do erro entre biológica repetições. Em vez disso, a própria variação biológica medido também inerentemente contém a variação técnica. Três experimental repetições (em caixas de cor amarela, verde e roxo) são usados por experiência chip para obter uma avsinal ChIP tura e seu desvio padrão (SD). Clique aqui para ver a figura maior .

Figura 2. Outro método de cálculo de enriquecimento chip. Nos casos em que a quantidade de ADN imunoprecipitados a partir do anticorpo de isotipo é extremamente baixo e qPCR amplificação é pobre, essa quantidade de ADN pode ser subtraída da quantidade de ADN imunoprecipitado utilizando um anticorpo específico para obter a dobra enriquecimento para todos os replicados técnica. Este método de cálculo deve ser utilizado em todos os três réplicas experimentais para obter um sinal médio chip e o seu desvio padrão (SD), como descrito na Figura 1.

Figura 3. Snapshot de planilha do Microsoft Excel mostrando os cálculos para obter enriquecimento relativo. Dados de três repetições experimentais (em caixas de cor amarela, verde e roxo) com três repetições técnica foi utilizada para calcular o enriquecimento vezes. Utilização do protocolo de cálculo descrito na Figura 1 está representado nesta planilha. Clique aqui para ver a figura maior .

Figura 4. O enriquecimento relativo de factor de transcrição NFAT para o IL2promotor. Convencionais células T CD4 (TCON) e CD4 células T reguladoras (Tregs) foram isolados de camundongos Foxp3EGFP. Uma pequena fração de células tcon foi estimulado com PMA e Ionomicina por 30 min. Chip foi realizada como explicado no protocolo experimental. Os dados aqui apresentados é de três repetições experimentais, com três repetições de cada técnica. O enriquecimento relativo dos factores de transcrição e NFATc1 NFATc2 no rato IL2 promotor nestes tipos de células é descrito como o enriquecimento de dobragem sobre uma região não ligada 3.
Discussão
O protocolo acima fornece um método robusto de quantificar com precisão o enriquecimento do ADN a partir dos linfócitos primários usando chip. Uma das principais razões para a robustez deste protocolo é a inclusão da diversidade biológica repetições. O protocolo acima usa três repetições, o enriquecimento para os quais é calculado de forma independente. As saídas são então calculadas para fornecer um grau de enriquecimento e desvios padrão calculados para proporcionar uma medida de variabilidade. Para cada amostra, três repetições técnica são também realizados para eliminar variações na manipulação da amostra, amplificação por PCR, etc A quantidade de variação nas repetições técnicas é tipicamente muito menor do que a observada entre as diferentes amostras biológicas. Uma segunda razão para a robustez é a inclusão de múltiplos controles técnicos, incluindo anticorpos isotipo de controlo, o uso de uma curva padrão, o ADN de entrada e à utilização de um produto de amplificação de controlo correspondente a uma região genómica não específica.
Jove_content As limitações do método acima são semelhantes aos que se encontram geralmente usando o chip. O método sofre um pouco pobre resolução espacial, o fator limitante é o corte de fragmentos de DNA por sonicação. Portanto, o poder de resolução do método é apenas cerca de 500 pb de DNA. Experimentos que requerem maior resolução pode fazer uso de Micrococo nuclease digestão 8 ou exonuclease digestão dos fragmentos recuperados (CHIP-exo) 9,10. Além disso, as preparações ChIP descrito acima acoplada a sequenciação de alto rendimento de fragmentos recuperados (ChIPseq, Chip-seq ou chip-SEQ), em substituição de 11,12 qPCR, produz uma amostragem dos fragmentos cortados enriquecidas diferente, a intersecção das quais pode ser tomado como um site fidedigno de interacção. No entanto, o método aqui apresentado fornece uma maneira robusta de cuidado e precisão quantificar DNA associação com fatores de transcrição, componentes nucleossomos, modificaçãoified histonas e outras proteínas em locais específicos de interesse.
Divulgações
Não há conflito de interesse declarados.
Agradecimentos
Este trabalho foi financiado pelo NIH concede CA141009 e GM39067. Agradecemos E. Parnell e R. Yarrington para comentários sobre a parte escrita.
Materiais
| Name | Company | Catalog Number | Comments |
| Formaldehyde | Sigma | F-8775 | Store at RT |
| Phosphate Buffered Saline | Hyclone | SH30256.01 | Store at 4 °C |
| Protease Inhibitor tablets | Roche | 04693116001 | Store at 4 °C |
| Protein G magnetic beads | Active Motif | 101945 | Store at 4 °C |
| RNase A (20 mg/ml) | EMD Millipore | 556746 | Store at -20 °C |
| Proteinase K (20 mg/ml) | Roche | 03115879001 | Dissolve in 50 mM Tris-HCl, 10 mM CaCl2, pH 8.0 |
| Platinum Taq DNA Polymerase | Invitrogen | 10966-034 | Store at -20 °C |
| SYBR Green I | Invitrogen | S7567 | Store at -20 °C |
| 1 M Glycine | Store at RT | ||
| Cell lysis buffer (5 mM Pipes, pH 8.0; 85 mM KCl; 0.5% NP-40) | Store at 4 °C | ||
| Nuclear lysis buffer (50 mM Tris, pH 8.1; 10 mM EDTA; 1% SDS) | Store at RT | ||
| ChIP dilution buffer (0.01% SDS; 1.1% Triton X-100; 1.2 mM EDTA; 16.7 mM Tris pH 8.1; 190 mM NaCl) | Store at 4 °C | ||
| Low salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 150 mM NaCl) | Store at 4 °C | ||
| High salt wash buffer (0.1% SDS; 1% Triton X-100; 2 mM EDTA; 20 mM Tris pH 8.1; 600 mM NaCl) | Store at 4 °C | ||
| LiCl wash buffer (0.25 M LiCl; 1% NP-40; 1% Sodium Deoxycholate, 1 mM EDTA; 10 mM Tris pH 8.0) | Store at 4 °C | ||
| TE buffer (10 mM Tris, pH 7.4, 1 mM EDTA) | Store at 4 °C | ||
| Elution buffer (1% SDS; 0.1 M NaHCO3) | Prepare fresh | ||
| 5 M NaCl | Store at RT | ||
| 0.5 M EDTA | Store at RT | ||
| 1 M Tris-HCl, pH 6.5 | Store at RT | ||
| Table of Specific Reagents | |||
| Clay Adams Brand Nutator | Becton Dickinson | Model: 421105 | |
| Magnetic Stand | Promega | Z5342 | |
| Qiaquick PCR Purification Kit | Qiagen | 28106 | |
| Masonix Sonicator 3000 | QSonica | Model: S3000 | |
| UV Spectrophotometer | NanoDrop Technologies | ND-1000 | |
| Heating Block | VWR | 13259-030 | |
| Rotator | VWR | 80085-692 | |
| Refrigerated bench top centrifuge | Beckman Coulter | Model: Allegra X-12R | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Table of Equipment | |||
Referências
- Weinmann, A. S., Bartley, S. M., Zhang, T., Zhang, M. Q., Farnham, P. J. Use of chromatin immunoprecipitation to clone novel E2F target promoters. Molecular and Cellular Biology. 21, 6820-6832 (2001).
- Weinmann, A. S., Farnham, P. J. Identification of unknown target genes of human transcription factors using chromatin immunoprecipitation. Methods. 26, 37-47 (2002).
- Li, Q., et al. Constitutive nuclear localization of NFAT in Foxp3+ regulatory T cells independent of calcineurin activity. J. Immunol. 188, 4268-4277 (2012).
- Shakya, A., Kang, J., Chumley, J., Williams, M. A., Tantin, D. Oct1 is a switchable, bipotential stabilizer of repressed and inducible transcriptional states. The Journal of Biological Chemistry. 286, 450-459 (2011).
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Lubelsky, Y., Macalpine, H. K., Macalpine, D. M. Genome-wide localization of replication factors. Methods. 57, 187-195 (2012).
- O'Neill, L. P., VerMilyea, M. D., Turner, B. M. Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations. Nature Genetics. 38, 835-841 (2006).
- Sikes, M. L., et al. A streamlined method for rapid and sensitive chromatin immunoprecipitation. Journal of Immunological Methods. 344, 58-63 (2009).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147, 1408-1419 (2011).
- Rhee, H. S., Pugh, B. F. Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature. 483, 295-301 (2012).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nature Biotechnology. 26, 1351-1359 (2008).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados
Usamos cookies para melhorar sua experiência em nosso site.
Ao continuar usando nosso site ou clicando em 'continuar', você concorda em aceitar nossos cookies.
