Method Article
Determinando a estabilidade sérica da enzima adenosina desaminase 1 humana
Neste Artigo
Resumo
Neste artigo, detalhamos métodos para caracterizar a capacidade de uma enzima de reter a função quando incubada a 37 ° C no soro humano, uma propriedade farmacológica conhecida como estabilidade sérica. Essa capacidade pode ser um fator chave na previsão do perfil farmacocinético de uma enzima e sua adequação para uso terapêutico.
Resumo
O conceito de estabilidade enzimática é normalmente usado para se referir à termoestabilidade de uma enzima - sua capacidade de reter estrutura e atividade à medida que a temperatura aumenta. Para uma enzima terapêutica, outras medidas de estabilidade também podem ser críticas, particularmente sua capacidade de reter a função no soro humano a 37 ° C, que chamamos de estabilidade sérica. Aqui, descrevemos um ensaio in vitro para avaliar a estabilidade sérica da enzima Homo sapiens adenosina desaminase I (HsADA1) do tipo selvagem usando um procedimento de microplaca baseado em absorbância. Especificamente, este manuscrito descreve a preparação de tampões e reagentes, um método que organiza a coincubação de HsADA1 no soro, um método para analisar as amostras de teste usando um leitor de microplacas e uma análise de acompanhamento para determinar a fração de atividade que uma enzima HsADA1 retém no soro em função do tempo. Discutimos ainda considerações para adaptar este protocolo a outras enzimas, usando um exemplo de uma enzima Homo sapiens quinurenase, para ajudar na adaptação do protocolo a outras enzimas onde a estabilidade sérica é de interesse.
Introdução
O método a seguir permite que um usuário avalie quantitativamente a capacidade de uma enzima de reter sua atividade quando exposta a condições que imitam o que ela encontrará após a injeção intravenosa. O método in vitro imita essas condições in vivo e consiste na incubação da enzima em soro humano agrupado a 37 °C e análises temporais da retenção da atividade enzimática. Referimo-nos à capacidade de uma enzima de reter atividade nessas condições como sua estabilidade sérica, e o método de análise da atividade enzimática aproveita as diferenças na absorbância entre o substrato de uma enzima e o produto resultante. O conceito de estabilidade sérica não é apenas específico da enzima e também tem sido aplicado a várias outras modalidades de tratamento. Por exemplo, a estabilidade sérica de aptâmeros de RNA direcionados às proteínas spike COVID foi previamente avaliada monitorando sua degradação pós-incubação com soro fetal de bezerro1. Os peptídeos antibacterianos também foram avaliados quanto à sua capacidade de suprimir o crescimento bacteriano pós-incubaçãocom soro humano 2.
HsADA1 é uma enzima que catalisa a conversão de adenosina ou 2-desoxiadenosina em inosina ou 2-desoxiinosina, respectivamente3. A adenosina tem um pico de absorção de 260 nm, enquanto a inosina absorve mais forte a 250 nm4. Essa mudança nos picos de absorção pode ser detectada em um leitor de microplacas por uma diminuição na intensidade de absorção a 260 nm quando HsADA1 é adicionado à adenosina. A enzima HsADA1 tem implicações importantes no corpo humano, e sua deficiência causa uma grave imunodeficiência combinada (ADA-SCID)5. O homólogo bovino de HsADA1, BtADA1, pode ser utilizado como uma terapêutica de reposição enzimática para o tratamento de ADA-SCID, e mostramos anteriormente que o HsADA1 selvagem perde sua atividade quando incubado com soro humano combinado, potencialmente dificultando seu uso como terapêutico6. Portanto, selecionamos a enzima HsADA1 do tipo selvagem para demonstrar um procedimento para determinar a estabilidade sérica de uma enzima. Um método de purificação detalhado para HsADA1 foi descrito anteriormente6.
No protocolo a seguir (conforme detalhado na Figura 1), demonstramos como co-incubar HsADA1 do tipo selvagem em soro humano combinado a 37 ° C. Durante esse período, as amostras de teste são coletadas em pontos de tempo definidos e congeladas rapidamente para análise futura. Uma vez que todas as amostras tenham sido coletadas, um ensaio de microplaca é executado em que cada amostra é combinada com o substrato, com a diminuição da absorbância resultante sendo uma correlação para a atividade retida da enzima. Resultados representativos que ilustram a estabilidade sérica de HsADA1 são mostrados e, como essa métrica pode ser relevante para determinar o valor terapêutico potencial de outras enzimas, também discutimos considerações para adaptar este protocolo a uma enzima Homo sapiens quinureninase modificada (HsKYNase) e quaisquer enzimas em geral. A HsKYNase é uma enzima envolvida no metabolismo do triptofano e é capaz de degradar os subprodutos do triptofano quinurenina e hidroxi-quinurenina (OH-Kyn) em ácido antranílico e ácido hidroxi-antranílico (OH-AA), respectivamente. A modulação do metabolismo do triptofano mediada por enzimas pode ser de relevância terapêutica7.
Protocolo
1. Incubação de soro
- Prepare um estoque de 10x de HsADA1 em 1x PBS pH 7,4 (1x PBS) a uma concentração final de 10 μM. Descongele uma alíquota de 15 mL de soro humano agrupado e pré-aqueça a 37 ° C. Pré-aqueça uma alíquota de 50 mL de 1x PBS a 37 °C.
- Preparar as misturas de incubação enzima-soro adicionando 100 μL do stock 10x HsADA1 a 900 μL de soro humano agrupado num tubo de microcentrífuga de baixa ligação, denominado mistura enzima + soro. Em um tubo de microcentrífuga de baixa ligação separado, adicione 100 μL do estoque 10x HsADA1 a 900 μL de 1x PBS para comparação com um controle sem soro, conhecido como mistura de enzima + 1x PBS. A concentração final de HsADA1 em cada mistura será de 1 μM.
- Prepare um controle adicional em um tubo de microcentrífuga de baixa ligação consistindo de 100 μL de 1x PBS misturado com 900 μL de soro humano agrupado. Isso servirá como controle não enzimático para a mistura enzima + soro. Além disso, faça um controle composto por 1 mL de 1x PBS. Isso servirá como controle não enzimático para a mistura enzimática + 1x PBS.
- Alíquota de 100 μL de cada mistura da etapa 1.2 em tubos separados de baixa ligação e diluir 1:1 com 100 μL de glicerol a 30% (v/v) em 1x PBS. Congelar rapidamente com azoto líquido e conservar a -80 °C. Essas amostras serão o ponto de tempo do Dia 0 e a concentração de HsADA1 nelas será de 500 nM. Repetir este passo para as misturas de controlo não enzimáticas a partir do ponto 1.3. Selar todas as enzimas e misturas de controlo com película transparente e colocá-las numa incubadora a 37 °C.
- Após 24 h, alíquota de 100 μL de cada amostra incubada e diluir 1:1 com 100 μL de glicerol a 30% (v/v) em 1x PBS. Congelar rapidamente com azoto líquido e conservar a -80 °C. Essas amostras serão o ponto de tempo do Dia 1. Voltar a colocar todas as misturas enzimáticas e de controlo na incubadora a 37 °C. Repita esta etapa em 72 h e 120 h.
NOTA: As amostras alíquotas não precisam necessariamente ser congeladas rapidamente. Alternativamente, o usuário pode realizar o ensaio baseado em microplacas imediatamente após a coleta de cada amostra, embora curvas padrão frescas e estoques de substrato de adenosina devam ser preparados cada vez que uma amostra é testada.
2. Ensaio baseado em microplacas
- Preparar o leitor de microplacas estabelecendo um protocolo de leitura cinética (figura 2) para medir a absorvância a 260 nm com o intervalo de leitura mais curto durante 30 minutos e pré-aquecer o leitor até um ponto de regulação de 37 °C.
- Descongele todas as amostras coletadas no gelo. Diluir cada amostra descongelada 1:10 usando 1x PBS pré-aquecido a 37 °C como diluente em tubos de microcentrífuga de baixa ligação. A nova concentração de HsADA1 nessas amostras será de 50 nM.
- Faça uma diluição de 10x em 1x PBS da amostra de controle de soro não enzimático também. Além disso, prepare um estoque de adenosina 5 mM adicionando 66,8 mg de adenosina a 50 mL de 1x PBS. Prepare uma diluição de 250 μM de adenosina adicionando 400 μL do estoque de adenosina a 7.600 μL de 1x PBS e pré-aqueça a 37 ° C em uma incubadora.
- Em uma microplaca compatível com UV de 96 poços, adicione 160 μL da diluição de adenosina de 250 μM e 40 μL de cada amostra de proteína diluída em triplicado para um volume total de 200 μL. Isso produzirá uma concentração final de adenosina de 200 μM e uma concentração enzimática final de 10 nM.
- Repetir a diluição para as amostras de controlo adicionando 40 μl do soro diluído ou 1x controlos PBS e 160 μl da diluição de adenosina de 250 μM em triplicata. Isso também produzirá uma concentração final de adenosina de 200 μM, mas sem enzima.
NOTA: Pode ser benéfico adicionar todas as enzimas diluídas e amostras de controle individualmente à microplaca primeiro e, em seguida, adicionar o substrato de adenosina com uma micropipeta de 12 canais para reduzir o tempo total de carregamento. Além disso, placas transparentes aos raios UV devem ser usadas nesta etapa para evitar que a própria placa confunda o sinal de absorbância medido. - Medir a absorvância de cada alvéolo a 260 nm durante 30 min a 37 °C. Uma vez feito isso, exporte os dados para uma planilha para análise posterior.
3. Análise dos dados do leitor de microplacas
- Determine a inclinação da região linear dos dados de absorbância em função do tempo para os primeiros minutos dos dados da microplaca. Para as amostras diluídas em soro, isso correspondeu aos primeiros ~ 120 s. Para as amostras diluídas em PBS 1x, isso correspondeu aos primeiros ~ 270 s. Esta inclinação dos valores de absorbância decrescente terá um valor numérico negativo e terá unidades de variação em unidades de absorbância por segundo (ΔA260/s).
- Subtrair a inclinação do controlo negativo que consiste em soro humano agrupado da inclinação dos dados enzimática + soro. Execute a mesma operação nas misturas de enzima + 1x PBS subtraindo a inclinação do controle 1x PBS.
- Normalize todas as inclinações ajustadas para a inclinação original em t = 0 h para obter a fração de atividade restante usando a Equação 1.
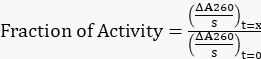 Equação 1
Equação 1 - Plote a fração de atividade restante versus o tempo.
Resultados
As figuras mostram os resultados do ensaio executado quando conduzido com HsADA1 do tipo selvagem. A Figura 3A, B ilustra as curvas de declínio da absorbância a 260 nm das amostras provenientes das misturas 1x PBS/soro-enzima para HsADA1 de tipo selvagem após a adição de adenosina. Essa absorbância em declínio em função dos dados de tempo é o que o usuário pode esperar após a conclusão bem-sucedida do ensaio baseado em microplacas e é semelhante aos dados de absorbância que surgiriam após a adição de quantidades suficientes de HsADA1 funcional à adenosina. A Figura 3C,D ilustra as curvas de absorbância dos controles negativos testados, que não contêm enzima após a adição de adenosina. Em comparação com as amostras de enzimas reais, a mudança de absorbância desses controles negativos é quase insignificante. A partir da Figura 3, pode-se observar que as inclinações das curvas enzimáticas mudam em função do tempo de incubação. Por exemplo, a inclinação do ponto de tempo do Dia 0 é drasticamente mais íngreme do que o ponto de tempo do Dia 5.
A Figura 4 ilustra a fração de atividade retida pelo HsADA1 do tipo selvagem em função do tempo quando incubado em 1x PBS ou soro humano agrupado a 37 ° C. Especificamente, as inclinações de todas as curvas de declínio de absorbância na Figura 3A, B são normalizadas para as inclinações do Dia 0 de seus respectivos grupos. Como pode ser visto na Figura 4, há uma divergência na atividade retida para HsADA1 do tipo selvagem em 1x PBS versus soro humano agrupado. Especificamente, a enzima perde sua atividade mais rapidamente no soro humano combinado, com a enzima retendo aproximadamente 10% de sua atividade original no Dia 5. Uma perda de atividade ainda é detectável quando HsADA1 é incubado em 1x PBS, embora a enzima retenha aproximadamente 75% de sua atividade. Isso é congruente com os resultados de Jennings et al., também relatando uma diminuição na atividade após a incubação com soro a 37 ° C em quase 40% após 48 h6. Lu et al. também mostraram que a atividade da HsADA1 diminui nos fluidos corporais pleural, peritoneal e cefalorraquidiano à temperatura ambiente (25 °C)8.
Para demonstrar a flexibilidade desse protocolo, nós o usamos para testar a estabilidade sérica de outra enzima de origem humana, uma variante de quinureninase humana modificada anteriormente apelidada de HsKYNase66-W102H-T333N7. HsKYNase66_W102H-T333N catalisa eficientemente a conversão de OH-Kyn em OH-AA, semelhante à enzima HsKYNase do tipo selvagem, embora seja na verdade um mutante de reversão de dois aminoácidos de uma variante de quinureninase humana projetada com especificidade de substrato alterada, ou seja, que degrada eficientemente a quinurenina em ácido antranílico7. OH-Kyn absorve fortemente a 379 nm, enquanto OH-AA não, resultando em declínio mensurável da absorbância após a adição de nossa variante HsKYNase projetada ao OH-kyn 9,10. A Figura 5A,B ilustra as curvas de declínio da absorbância a 379 nm das amostras provenientes das misturas 1x PBS/soro-enzima para HsKYNase após a adição de OH-Kyn. A Figura 5C,D ilustra as curvas de absorbância dos controles testados que não contêm enzimas. Como visto com HsADA1, há uma clara diferença nas inclinações das curvas de absorbância entre as amostras. Isso é especialmente evidente na Figura 5A, pois a inclinação do ponto de tempo de 0 h é muito mais íngreme do que os pontos de tempo de 3 h e 24 h. A Figura 6 ilustra a fração correspondente de atividade retida para a HsKYNase testada em função do tempo. Como HsADA1, HsKYNase66-W102H-T333N perde atividade no soro mais rapidamente do que em PBS (~ 85% de atividade perdida versus ~ 40% ao longo de 24 h). Embora a estabilidade sérica dessa variante enzimática não tenha sido testada anteriormente, ela segue a tendência geral de outras variantes da HsKYNase, que também perderam a maior parte de sua atividade em 24 horas no soro a 37 ° C7.
De um modo geral, as enzimas podem perder atividade ao longo do tempo devido a uma série de fatores, incluindo pH, concentração de sal, temperaturae muito mais. Portanto, um resultado que não acompanhe essa tendência pode ser considerado anormal. Por exemplo, se a atividade enzimática aumentar drasticamente ao longo do tempo, isso pode indicar que os resultados são inválidos. Isso pressupõe que as condições enzimáticas não sejam alteradas, e estamos falando estritamente no sentido do tempo.

Figura 1: Fluxograma detalhando as etapas do protocolo para HsADA1. A figura mostra as etapas seguidas neste protocolo. Clique aqui para ver uma versão maior desta figura.

Figura 2: Configurações de leitura cinética do leitor de microplacas. Um protocolo de leitura cinética foi criado para medir a absorbância a 260 nm por 30 min a 37 °C com o intervalo mínimo. A figura mostra as configurações escolhidas para configurar este protocolo. Clique aqui para ver uma versão maior desta figura.

Figura 3: Curvas de absorbância versus tempo para HsADA1 após a adição de 200 μM de adenosina. (A) Curvas de absorbância versus tempo para HsADA1 incubado com soro após a adição de 200 μM de adenosina. (B) 1x HsADA1 incubado com PBS. (C) Controle soro negativo. (D) 1x controle negativo PBS (n = 3). Clique aqui para ver uma versão maior desta figura.
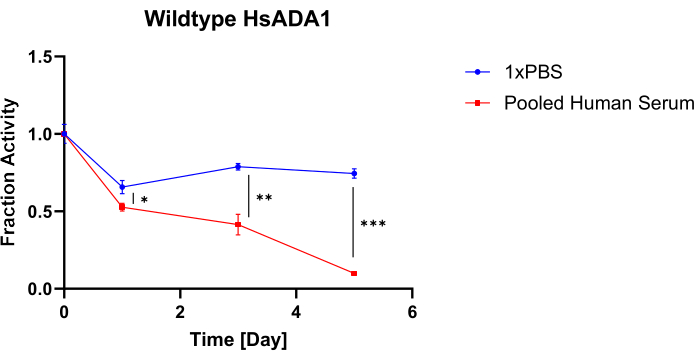
Figura 4: Retenção da atividade de HsADA1. A atividade é avaliada em 1x PBS versus soro humano combinado a 37 °C. A fração da atividade original retida para HsADA1 incubada em 1x PBS versus soro humano combinado ao longo de 5 dias foi determinada (barras = média ± DP, n = 3). Um teste t não pareado bicaudal foi usado *p<0,05, **p < 0,01 e *** p < 0,001. Clique aqui para ver uma versão maior desta figura.
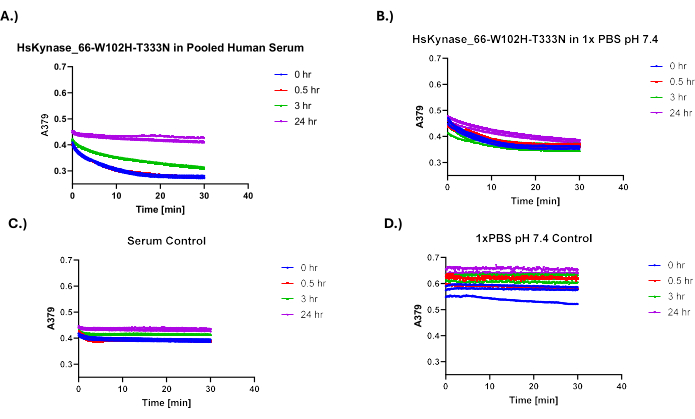
Figura 5: Curvas de absorbância versus tempo para HsKYNase66-W102H-T333N após a adição de 400 μM OH-Kyn. (A) Curvas de absorbância versus tempo para HsKYNase incubada com soro após a adição de 400 μM OH-Kyn. (B) 1x HsKYNase incubada com PBS. (C) Controle soro negativo. (D) 1x controle negativo PBS (n=3). Clique aqui para ver uma versão maior desta figura.
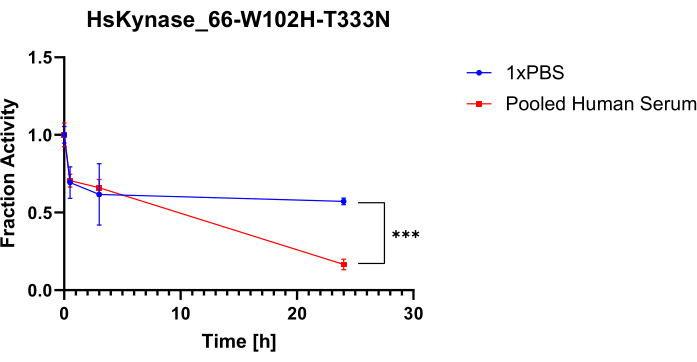
Figura 6: Retenção de atividade de HsKYNase66-W102H-T333N. A atividade é avaliada em 1x PBS versus soro humano combinado a 37 °C. A fração da atividade original retida para HsKYNase66-W102H-T333N incubada em 1x PBS versus soro humano agrupado ao longo de 24 h foi determinada (barras = média ± DP, n = 3). Um teste t não pareado bicaudal foi usado; p < 0,001. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Curva padrão de adenosina e inosina. O gráfico foi desenhado para 0-200 μM. Adenosina ou inosina foi adicionada a 1x PBS nas concentrações observadas e, em seguida, a absorbância a 260 nm foi medida. (n=3). Clique aqui para baixar este arquivo.
Figura suplementar 2: Curva padrão de OH-Kyn. O gráfico foi desenhado para 0-400 μM. O OH-Kyn foi adicionado a 1x PBS nas concentrações observadas e, em seguida, a absorbância a 379 nm foi medida. (n=3). Clique aqui para baixar este arquivo.
Figura suplementar 3: Curvas de absorbância versus tempo a 260 nm com concentrações crescentes de HsADA1 após a adição de 200 μM de adenosina. (A) Curvas de reação para concentrações variáveis de HsADA1. (B) Mudança de dobra na inclinação da região linear das curvas de absorbância em relação a um controle PBS 1x (barras = média ± DP, n = 3). Um teste t não pareado bicaudal foi usado **p < 0,01 e ****p < 0,0001. Clique aqui para baixar este arquivo.
Figura suplementar 4: Curvas de absorbância versus tempo a 379 nm com concentrações crescentes de HsKYNase projetada após a adição de 400 μM de OH-Kyn. (A) Curvas de reação para concentrações variáveis de HsKYNase projetada. (B) Mudança de dobra na inclinação da região linear das curvas de absorbância em relação a um controle PBS 1x (barras = média ± DP, n = 3). O teste t não pareado bicaudal foi usado ****p < 0,0001. Clique aqui para baixar este arquivo.
Figura 5 suplementar: Curvas de absorbância versus tempo a 379 nm usando enzima desativada por calor em 400 μM de OH-Kyn. A enzima HsKYNase desativada por calor foi adicionada a 400 μM de OH-Kyn em 1x PBS e, em seguida, a absorbância a 379 nm foi medida. (n=3). Clique aqui para baixar este arquivo.
Discussão
Este protocolo usa a mudança de absorbância à medida que o substrato é convertido no produto para medir a atividade de uma enzima. Como tal, o substrato e o produto devem ter perfis espectrais distintos. Este é o caso da adenosina e da inosina, ambas com perfis espectrais distintos e coeficientes de extinção entre 260-265 nm 6,8,12,13. Este ensaio é inspirado em vários trabalhos anteriores. Kalackar, por exemplo, utilizou a mudança na absorbância a 265 nm como um correlato direto para a atividade da adenosina desaminase14. Gracia et al. e Lu et al. empregaram uma abordagem enraizada na lei de Beer-Lambert, que relaciona a velocidade inicial com a diferença nos coeficientes de extinção entre adenosina e inosina 8,13. Na mesma linha, OH-Kyn e OH-AA também têm perfis espectrais distintos, pois OH-Kyn absorve fortemente a 379 nm, enquanto OH-AA não 9,10. O espectro de absorbância dos metabólitos pode ser facilmente verificado usando uma varredura de espectro completo em muitos leitores de microplacas, o que, por sua vez, informaria ao usuário se é possível rastrear o progresso de uma reação enzimática. Como afirmado anteriormente, Lu et al. analisaram anteriormente a estabilidade nativa de HsADA1 em diferentes fluidos corporais, e Jennings et al. empregaram um método muito semelhante para avaliar a estabilidade sérica de HsADA1 recombinante e uma enzima mutante 6,8. Este método, como o de Jennings et al., é único porque podemos usar enzimas expressas recombinantemente em oposição às derivadas nativamente. Ao fazer isso, esse método pode ser extrapolado para proteínas mutantes não encontradas nativamente em organismos vivos.
Para combinações de produto/substrato que não podem ser medidas usando um ensaio de absorbância baseado em placa, este protocolo pode ser modificado para incorporar vários outros métodos analíticos alternativos que rastreiam a degradação do substrato ou a formação do produto para ensaios cinéticos, como cromatografia líquida de alta eficiência (HPLC), que, por exemplo, foi usada anteriormente para avaliar a cinética da adenosina desaminase e da quinureninase15, 16,17. Apenas o método para determinar o substrato restante ou o produto criado após um certo tempo (se não for determinado continuamente) deve variar. A escolha do ensaio dependerá, obviamente, da reação catalisada pela enzima de interesse, e uma consideração importante nessa escolha também será a compatibilidade de um determinado método analítico com soro humano diluído. De igual importância também pode ser o tempo total necessário para analisar todas as amostras de interesse, e um dos principais benefícios da utilização do ensaio de microplacas é a capacidade de analisar facilmente as taxas de degradação em até 96 amostras por vez.
Os parâmetros do ensaio, como a enzima na placa e as concentrações de substrato, precisarão ser ajustados para enzimas individuais de interesse. Para todos os ensaios aqui realizados, foi escolhida uma concentração de substrato acima de Km em uma ordem de grandeza maior (1,15-1,26 OOM). O tipo selvagem HsADA1 tem um Km de ~ 14 μM para adenosina, e a concentração final de adenosina na placa usada foi de 200 μM3. Da mesma forma, a variante HsKYNase projetada testada tem um Km ~ 22 μM para OH-Kyn, e a concentração final de OH-Kyn na placa usada foi de 400 μM7. Um requisito importante para as concentrações de substrato de interesse para o ensaio de microplaca é que o substrato obedeça à lei de Beer-Lambert dentro da faixa dada, de modo que uma diminuição da absorbância possa ser linearmente correlacionada com a degradação do substrato (Figura Suplementar 1 e Figura Suplementar 2). Essa correlação linear elimina a necessidade de converter a mudança de absorbância em concentração de substrato, pois realizar essa conversão não alteraria a fração de atividade restante.
Quanto à concentração da enzima, o principal critério a considerar é que haja degradação suficiente do substrato em relação ao fundo. Tanto para HsADA1 quanto para HsKYNase projetada, usamos valores da literatura publicados anteriormente para gerar resultados representativos 6,7. Como mostramos aqui, o uso de tempos mais curtos com concentrações enzimáticas mais altas pode ser viável, assim como o uso de tempos mais longos e concentrações enzimáticas mais baixas. Em geral, um período de tempo mais longo/menor concentração enzimática pode dar resultados mais reprodutíveis, a menos que o próprio substrato seja propenso à degradação. Nesse sentido, tanto a quinurenina quanto seus derivados, incluindo OH-Kyn, se degradam de forma relativamente rápida à temperatura ambiente, resultando em um amarelecimento perceptível da mistura de substrato18. Para outras enzimas, um experimento simples de escalonamento de concentração pode ser conduzido para identificar concentrações com as curvas de reação e separação de fundo mais ideais. A realização desse escalonamento de concentração em HsADA1 mostra que, dentro de uma faixa de 1 a 10 nM, a maior separação de fundo é alcançada (Figura Suplementar 3). A realização deste mesmo experimento de escalonamento de concentração com HsKYNase projetada mostra uma tendência semelhante com a maior separação de fundo em 2 μM (Figura Suplementar 4). No entanto, ambos os ensaios (HsADA1 versus HsKYNase) não possuem a mesma sensibilidade. Para HsADA1, uma concentração enzimática na placa um décimo daquela usada no protocolo ainda é distinguível do fundo em níveis estatisticamente significativos (Figura Suplementar 3). O mesmo não vale para o HsKYNase, o que significa que pode ser mais difícil avaliar quantitativamente quanta atividade o HsKYNase retém depois de perder mais de 90% de sua atividade. Isso representa uma limitação potencial do método do leitor de microplacas.
Além disso, uma observação que fizemos para a HsKYNase foi que, após o carregamento da placa, havia uma lacuna imediata entre as leituras de absorbância entre a enzima + 1x amostras de PBS e o controle 1x PBS (Figura 5B, D). Essa lacuna estava presente em todos os momentos. Como também observamos esse fenômeno ao testar a enzima desativada pelo calor (aquecida a 95 ° C por 10 min), concluímos que não era resultado da degradação do substrato mediada por enzimas (Figura Suplementar 5). Embora tenhamos prosseguido com os valores originais de absorbância de controle, estudos futuros poderiam usar controles enzimáticos desativados para eliminar essa discrepância. No entanto, o custo de aquisição ou produção da enzima de interesse deve ser levado em consideração. Tanto para HsKYNase quanto para HsADA1, houve alguma variabilidade nos valores de absorbância inicial das amostras de teste, com algumas começando em um valor de absorbância mais baixo do que seus controles associados (Figura 3 e Figura 5). Isso pode ser parcialmente atribuído ao tempo de atraso na adição de substrato a todas as amostras de proteína, seguido pelo carregamento no leitor de placas. No entanto, como a concentração de substrato usada é muito maior do que os valores de Km das enzimas usadas, não se espera que isso leve a efeitos dependentes da concentração de substrato nas encostas observadas.
Na seção de protocolo e resultados representativos, as amostras são aliquotadas nos pontos de tempo indicados, com o tempo de amostragem variando com base no perfil de atividade de uma enzima. Este esquema de amostragem dependerá do perfil de atividade de uma enzima de interesse no soro. Se uma enzima perde atividade rapidamente, então um intervalo de amostragem menor será desejado, como é o caso do mutante HsKYNase_66-W102H-T333N testado, e o oposto é verdadeiro para enzimas com maior estabilidade sérica. Como tal, é provável que seja benéfico realizar um teste deste protocolo em que as alíquotas são amostradas em intervalos pequenos (1 h, 2 h, etc.) e grandes (24 h, 48 h, etc.) para pesquisar como é o perfil de estabilidade sérica para uma nova enzima de interesse. É claro que, à medida que se torna mais robusto, a estabilidade sérica se tornará menos relevante em comparação com a depuração renal ou outros mecanismos de depuração de proteínas in vivo .
Como as quinureninases diferem de HsADA1, modificamos vários parâmetros de ensaio, ou seja, concentração de enzima durante a incubação em soro ou 1x PBS, intervalo de amostragem, concentração de enzima de ensaio de microplaca, concentração de substrato de ensaio de microplaca e, é claro, o próprio substrato. As alterações são: o estoque enzimático 10x estava na concentração de 100 μM, o que, após diluir com soro/PBS 1x, resultou na concentração de 10 μM. O intervalo de amostragem no método de incubação do soro foi ajustado porque a HsKYNase perde atividade mais rapidamente no soro do que na HsADA1. Portanto, o método incluiu amostragem em pontos de tempo de 0,5 h, 3 h e 24 h. Além disso, as amostras de HsKYNase não foram congeladas rapidamente, sendo imediatamente analisadas usando o ensaio de microplaca. O leitor de placas foi configurado para ler a 379 nm para monitorar o progresso da reação enzimática. Outra diferença importante no ensaio baseado em microplacas ao estudar a enzima HsKYNase é a preparação do substrato. Um estoque de 2 mM de OH-Kyn foi feito dissolvendo 4,5 mg de OH-Kyn em 10 mL de 1x PBS. Ao mesmo tempo, 6 mg de fosfato de piridoxal (PLP), o cofator HsKYNase, foram dissolvidos em 1 mL de 1x PBS para fazer um estoque de 100x de PLP. Uma diluição de 500 μM de OH-Kyn foi preparada adicionando 2,5 mL de estoque de OH-Kyn a 7,5 mL de 1x PBS, bem como 100 μL do estoque de 100x PLP. Para executar o ensaio de microplaca, 80 μL da diluição de 500 μM OH-Kyn e 20 μL das amostras de proteína aliquotada ou controles negativos foram adicionados em triplicata a uma microplaca compatível com UV de 96 poços. Isso produziu uma concentração final de OH-Kyn de 400 μM e uma concentração final de enzima de 2 μM, após a qual a absorbância a 379 nm foi medida por 30 min a 37 ° C. Ao contrário da HsADA1, que foi diluída antes de ser analisada no leitor de placas, a HsKYNase não foi diluída.
Embora este protocolo seja relativamente fácil, um desafio potencial pode estar na preparação dos estoques originais de enzimas. Todas as enzimas utilizadas na seção de resultados representativos foram expressas em E. coli e purificadas por cromatografia de afinidade à base de níquel seguida de cromatografia de exclusão de tamanho (SEC). Várias outras fontes descreveram habilmente alternativas ao nosso método de purificação, abrangendo organismos hospedeiros e processamento a jusante19,20. Este ensaio tenta fundamentalmente determinar a capacidade de uma enzima de reter sua atividade no soro humano agrupado. Uma suposição em nossa metodologia é que a perda de atividade de uma enzima é permanente, ou seja, a remoção de uma enzima desativada do soro não restaurará sua atividade. Isso é significativo porque, neste protocolo, diluímos ainda mais as misturas enzimáticas com glicerol e 1x PBS antes do congelamento instantâneo e após o descongelamento. Se essa perda de atividade for reversível após a remoção do soro, essas diluições adicionais impedirão que o ensaio capture esse fenômeno. Se isso for motivo de preocupação para o usuário, então 1.) soro humano combinado pode ser usado como diluente em vez de 1x PBS na preparação de todas as soluções de substrato e amostra de teste, e 2.) O congelamento instantâneo e a adição de glicerol podem ser ignorados e as amostras podem ser processadas imediatamente para manter a enzima em seu ambiente original. Por fim, embora este ensaio possa ser usado para investigar os efeitos específicos do soro na atividade enzimática, nenhuma conclusão pode ser feita sobre o mecanismo específico por trás de tais efeitos. Se essa perda de atividade é causada por desdobramento enzimático, agregação, perda de cofator ou clivagem por proteases séricas precisaria de mais investigação. Qualificações semelhantes podem ser ditas da enzima incubada 1x PBS, pois conclusões mecanicistas não podem ser derivadas deste ensaio.
No contexto do desenvolvimento de produtos biológicos proteicos como terapêutica, a capacidade de reter a atividade no soro é uma métrica importante21. Uma enzima terapêutica que perde sua atividade muito rapidamente teria que ser dosada repetidamente para obter benefício clínico. Este trabalho fornece aos usuários um ensaio in vitro para rastrear quantitativamente as enzimas para melhorar a estabilidade do soro. Se combinadas com esforços de evolução direcionados, enzimas com estabilidade sérica superior podem ser rastreadas e selecionadas, dando maior potencial de traduzibilidade. Além disso, prevemos que este protocolo também possa funcionar com sistemas mais avançados, por exemplo, para medir a estabilidade do soro onde a enzima é encapsulada. Das et al. aplicaram um ensaio para várias enzimas diferentes embaladas em partículas semelhantes a vírus e testaram sua estabilidade em diferentes condições de desnaturação, como solventes orgânicos22.
Divulgações
JB e MRJ são inventores de patentes ou pedidos de patentes relacionados às enzimas adenosina desaminase e/ou quinureninase. Todos os outros autores não têm conflitos de interesse a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo National Institutes of Health [1DP2CA280622-01] e financiamento da Biolocity. Agradecemos à Dra. Maria Jennings e Andrea Fox por fornecerem os vetores de expressão HsADA1 e HsKYNase.
Materiais
| Name | Company | Catalog Number | Comments |
| Adenosine | Sigma Aldrich | A9251-25G | 25 g |
| BioTek Synergy HT Microplate Reader | |||
| Eppendorf LoBind Microcentrifuge Tubes: Protein | Fisher Scientific | 13-698-795 | 2 mL |
| Glycerol | Fisher Scientific | G33-4 | 4 L |
| HsKYNase66-W102H-T333N | In-house | ||
| Human Serum, Pooled | MP Biomedicals | 92931149 | 100 mL |
| Hydroxy-kynurenine | Cayman Chemicals | 27778 | |
| Inosine | TCI | I0037 | 25 g |
| PBS, 1x pH 7.4+/- 0.1 | Corning | 21-040-CM | |
| Pyridoxal 5-phosphate monohydrate, 99% | Thermo Scientifc | 228170010 | 1 g |
| UV-STAR MICROPLATE, 96 WELL, COC, F-BOTTOM | Greiner Bio | 655801 | |
| Wildtype Human Adenosine Deaminase 1 | In-house |
Referências
- Valero, J., et al. A serum-stable RNA aptamer specific for SARS-COV-2 neutralizes viral entry. Proc Natl Acad Sci U S A. 118 (50), e2112942118 (2021).
- Iannuzo, N., et al. High-throughput screening identifies synthetic peptides with antibacterial activity against mycobacterium abscessus and serum stability. ACS Omega. 7 (27), 23967-23977 (2022).
- Ma, M. T., Jennings, M. R., Blazeck, J., Lieberman, R. L. Catalytically active holo homo sapiens adenosine deaminase i adopts a closed conformation. Acta Crystallograph Sect D. 78 (1), 91-103 (2022).
- Li, W., et al. Determination of 4 nucleosides via one reference compound in chinese cordyceps by hplc-uv at equal absorption wavelength. Natural Prod Comm. 18 (3), 1934578X231161410 (2023).
- Whitmore, K. V., Gaspar, H. B. Adenosine deaminase deficiency - more than just an immunodeficiency. Front Immunol. 7, 314 (2016).
- Jennings, M. R., et al. Optimized expression and purification of a human adenosine deaminase in E. coli and characterization of its asp8asn variant. Prot Express Purificat. 213, 106362 (2024).
- Blazeck, J., et al. Bypassing evolutionary dead ends and switching the rate-limiting step of a human immunotherapeutic enzyme. Nat Catalysis. 5 (10), 952-967 (2022).
- Lu, J., Grenache, D. G. Development of a rapid, microplate-based kinetic assay for measuring adenosine deaminase activity in body fluids. Clinica Chimica Acta. 413 (19), 1637-1640 (2012).
- Maciel, L. G., Dos Anjos, J. V., Soares, T. A. Fast and low-cost evaluation of hydroxykynurenine activity. MethodsX. 7, 100982 (2020).
- Bokman, A. H., Schweigert, B. S. 3-hydroxyanthranilic acid metabolism. Iv. Spectrophotometric evidence for the formation of an intermediate. Arch Biochem Biophys. 33 (2), 270-276 (1951).
- Tanford, C. . Advances in protein chemistry. 23, 121-282 (1968).
- Tritsch, G. L. Validity of the continuous spectrophotometric assay of kalckar for adenosine deaminase activity. Anal Biochem. 129 (1), 207-209 (1983).
- Gracia, E., et al. The catalytic site structural gate of adenosine deaminase allosterically modulates ligand binding to adenosine receptors. FASEB J. 27 (3), 1048-1061 (2013).
- Kalckar, H. M. Differential spectrophotometry of purine compounds by means of specific enzymes: Iii. Studies of the enzymes of purine metabolism. J Bio Chem. 167 (2), 461-475 (1947).
- Hartwick, R., Jeffries, A., Krstulovic, A., Brown, P. R. An optimized assay for adenosine deaminase using reverse phase high pressure liquid chromatography. J Chromatographic Sci. 16 (9), 427-435 (1978).
- Paul, M. K., Grover, V., Mukhopadhyay, A. K. Merits of hplc-based method over spectrophotometric method for assessing the kinetics and inhibition of mammalian adenosine deaminase. J Chromatography B. 822 (1), 146-153 (2005).
- Ubbink, J. B., Vermaak, W. J. H., Bissbort, S. H. High-performance liquid chromatographic assay of human lymphocyte kynureninase activity levels. J Chromatography B: Biomed Sci Appl. 566 (2), 369-375 (1991).
- Tsentalovich, Y. P., Snytnikova, O. A., Forbes, M. D. E., Chernyak, E. I., Morozov, S. V. Photochemical and thermal reactivity of kynurenine. Exp Eye Res. 83 (6), 1439-1445 (2006).
- Demain, A. L., Vaishnav, P. Production of recombinant proteins by microbes and higher organisms. Biotechnol Adv. 27 (3), 297-306 (2009).
- Gräslund, S., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Faber, M. S., Whitehead, T. A. Data-driven engineering of protein therapeutics. Curr Opin Biotechnol. 60, 104-110 (2019).
- Das, S., Zhao, L., Elofson, K., Finn, M. G. Enzyme stabilization by virus-like particles. Biochemistry. 59 (31), 2870-2881 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados