Method Article
Purificação de afinidade de uma proteína marcada com 6X-His usando um sistema de cromatografia líquida de proteína rápida
Neste Artigo
Resumo
Este artigo fornece um procedimento para a purificação de afinidade de uma proteína recombinante humana, a endonuclease 1 (FEN1), que foi marcada com uma marca de 6X-histidina. O protocolo envolve a utilização de duas colunas distintas de íons metálicos imobilizados para a purificação da proteína marcada.
Resumo
A caracterização funcional das proteínas requer que elas sejam expressas e purificadas em quantidades substanciais com alta pureza para realizar ensaios bioquímicos. O sistema de Cromatografia Líquida de Proteína Rápida (FPLC) permite a separação de alta resolução de misturas complexas de proteínas. Ao ajustar vários parâmetros no FPLC, como selecionar a matriz de purificação apropriada, regular a temperatura da amostra de proteína e gerenciar a taxa de fluxo da amostra na matriz e a taxa de eluição, é possível garantir a estabilidade e a funcionalidade da proteína. Neste protocolo, demonstraremos a versatilidade do sistema FPLC para purificar a proteína 6X-His-tagged flap endonuclease 1 (FEN1), produzida em culturas bacterianas. Para melhorar a eficiência da purificação de proteínas, vamos nos concentrar em várias considerações, incluindo empacotamento e preparação adequados da coluna, injeção de amostra usando um loop de amostra, taxa de fluxo de aplicação da amostra na coluna e parâmetros de eluição da amostra. Finalmente, o cromatograma será analisado para identificar frações contendo altos rendimentos de proteína e considerações para armazenamento adequado de proteína recombinante a longo prazo. A otimização dos métodos de purificação de proteínas é crucial para melhorar a precisão e a confiabilidade da análise de proteínas.
Introdução
Inúmeras estratégias estão disponíveis para compreender a biologia celular. Uma abordagem envolve uma estratégia de cima para baixo, em que mutações genéticas são introduzidas em um gene, seguidas pela avaliação das mudanças fenotípicas resultantes em um organismo modelo. Por outro lado, uma abordagem reducionista envolve a elucidação inicial dos mecanismos moleculares e funções enzimáticas de uma determinada proteína, acompanhada pela caracterização de suas interações com outros componentes celulares. Posteriormente, avalia-se o impacto dessa proteína em uma via biológica. Embora cada abordagem de pesquisa possua suas vantagens e limitações inerentes, alcançar uma compreensão abrangente de uma via biológica requer investigações interdisciplinares.
Com o DNA sendo o projeto genético da vida, entender os mecanismos de duplicação de DNA e manutenção do genoma tem sido uma área de interesse ativo por mais de sete décadas. Estudos no campo da replicação do DNA produziram dados abundantes sobre as estruturas e funções individuais de numerosas proteínas de replicação. Essas investigações, que englobam aspectos mecanísticos e ensaios de atividade bioquímica, foram viabilizadas por meio da purificação dessas proteínas, possibilitando seu exame meticuloso em meio in vitro . Consequentemente, a purificação de proteínas surge como uma técnica indispensável e onipresente na maioria dos esforços de pesquisa voltados para desvendar insights mecanicistas sobre a replicação do DNA.
Este artigo apresenta uma metodologia para isolar uma proteína de replicação de DNA marcada com 6X-histidina, que foi superexpressa em células bacterianas. A proteína de interesse é a endonuclease 1 do retalho humano (FEN1), uma nuclease específica da estrutura que desempenha um papel fundamental na replicação da fita atrasada e também é um participante crítico nas vias de reparo do DNA, como o reparo por excisão de base (BER) 1 , 2 , 3 . A função primária do FEN1 é clivar a base de uma estrutura de retalho deslocada de 5', um intermediário que surge durante a replicação do DNA ou BER. Inicialmente, investigações bioquímicas avaliando a atividade enzimática do FEN1 sugeriram um mecanismo de "rastreamento", no qual a nuclease reconheceria a extremidade 5'-fosfato livre de uma estrutura de retalho e, em seguida, seguiria ao longo do retalho até sua base antes de clivá-lo4. Pesquisas subsequentes revelaram que o FEN1 opera por meio de um mecanismo de "rosqueamento", no qual primeiro se liga à base do retalho e, em seguida, enfia a extremidade 5 'livre através de seu sítio ativo antes da clivagem (Figura 1)5. A capacidade de superexpressar e isolar o FEN1 recombinante facilitou esses avanços, permitindo que os pesquisadores o empregassem em investigações bioquímicas e estruturais.
A cromatografia de afinidade é um método de separação comumente usado para purificar o DNA. Esta técnica usa a afinidade de ligação reversível de proteínas-alvo em relação a ligantes imobilizados em uma resina para capturar especificamente a proteína de interesse. Uma das bioafinidades mais amplamente utilizadas é a interação robusta entre o aminoácido, histidina e íons metálicos, como níquel e cobalto e, portanto, pode ser capturada em uma resina carregada com Ni2+ ou Co2+.
A sequência de DNA que codifica uma sequência de 6-9 resíduos de histidina (His) é freqüentemente incorporada à construção do plasmídeo que codifica a proteína de interesse (no terminal N ou no terminal C), marcando a proteína com uma etiqueta 6X-His ou uma marca poli-His. A proteína marcada com His pode então ser facilmente purificada por cromatografia de afinidade de metal imobilizado (IMAC), um subtipo de cromatografia de afinidade em que os íons metálicos na resina capturam proteínas com uma etiqueta de afinidade, que pode ser eluída posteriormente usando agentes de eluição apropriados. Íons de metais de transição, como Ni2+ e Co2+ , podem ser imobilizados em matrizes de agarose ou sílica gel derivadas de grupos N,N,N'-tris-(carboximetil)-etilenodiamina ou ácido nitrilotriacético (NTA)6.
Os ligantes metálicos são conhecidos por serem robustos contra a degradação por fatores físicos, químicos e biológicos e possuem essa vantagem sobre outros tipos de ligantes 6,7. Além disso, a marca His é uma marca relativamente pequena e não afeta significativamente a estrutura ou função da proteína7. No entanto, em um sistema de expressão bacteriana, muitas proteínas expressas cromossomicamente têm afinidade por íons metálicos e podem co-purificar com a proteína alvo. Níquel e cobalto são os íons metálicos típicos usados em matrizes IMAC. A resina Ni-NTA e a resina à base de cobalto TALON são comumente usadas para a purificação de proteínas marcadas com His.
Ni-NTA versus TALON
Os respectivos íons metálicos de Ni-NTA e TALON são imobilizados na resina por meio de ligantes NTA. Acredita-se que o Ni-NTA tenha uma capacidade de ligação mais alta, ligando-se a até 100 mg / mL de proteína. Isso pode resultar em um maior rendimento de proteína, com a ressalva de que as proteínas contaminantes podem ser co-purificadas. Em contraste, a resina tem uma maior especificidade de ligação para proteínas marcadas com His e pode ser capaz de produzir frações de maior pureza. Neste estudo, pretendemos comparar a eficiência de purificação de ambas as resinas usando o sistema automatizado de cromatografia líquida de proteína rápida referenciado, o sistema NGC (ver Tabela de Materiais).
Buffers e compatibilidade
Os tampões são necessários durante a purificação da proteína para lise celular, preparação da amostra, equilíbrio da resina e eluição da proteína capturada da resina. Os tampões Tris, MOPS e HEPES até uma concentração de 100 mM são os tampões compatíveis conhecidos para a resina Ni-NTA. Os tampões geralmente incluem agentes redutores para evitar a oxidação de proteínas e agregação de proteínas. No entanto, acima de um limite, os agentes redutores podem retirar a resina de íons metálicos. A concentração recomendada de agentes redutores, como beta-mercaptoetanol (BME) ou ditiotreitol (DTT), é inferior a 1 mM para as resinas à base de níquel e cobalto acima mencionadas.
Os tampões para a purificação de hFEN1 são tampões Tris contendo NaCl, BME, fluoreto de fenilmetilsulfonil (PMSF), EDTA e glicerol. O NaCl mantém a proteína na forma solúvel e interrompe as interações moleculares, como a ligação ao DNA. O BME reduz as proteínas oxidadas e, assim, evita a agregação de proteínas. PMSF) é um inibidor de protease que previne a degradação mediada por protease da proteína-alvo. O EDTA elimina os cátions divalentes da amostra, impedindo seu acesso a nucleases e proteases. O glicerol aumenta a estabilidade da proteína na forma aquosa. Além disso, o tampão de lise contém comprimidos inibidores de protease completos para garantir a máxima proteção da proteína-alvo contra a degradação de proteases durante a lise celular. Os tampões de equilíbrio e eluição contêm imidazol, com o tampão de eluição contendo quantidades maiores para o imidazol deslocar a proteína ligada da resina durante a eluição.
Sistema de cromatografia de próxima geração (NGC)
Este sistema automatizado de cromatografia de média pressão projetado para cromatografia líquida de proteína de fluxo rápido (FPLC) usa duas bombas para bombear simultaneamente dois tampões diferentes e é capaz de injetar uma ampla gama de volumes de amostra de 250 μL a 100 mL. O loop de amostra (conhecido como Dynaloop neste sistema) possibilita injetar volumes de amostra maiores. O sistema pode ser operado usando o software Chromlab, que facilita a criação de métodos personalizados, a manipulação de execuções de purificação e a análise de picos de UV e frações de proteínas.
Protocolo
1. Preparação da amostra
- Para purificar FEN1 recombinante, expresse a construção (pET-FCH-FEN1) em células BL21 (DE3) conforme descrito anteriormente por Ononye et al.8.
- Inocular o meio LB (Tabela 1) com 1% de cultura durante a noite e fazer crescer as células a 37 °C até que a DO atinja 0,6.
- Induza com 0,4 M de isopropil-beta-D-tiogalactosídeo (IPTG) e cultive a cultura por mais 3 h.
- Colha as células centrifugando a cultura a 5.000 × g durante 15 min a 4 °C. Armazene o pellet em um freezer a -80°C.
- No dia da purificação, remova o pellet celular do freezer a -80 ° C, deixe-o descongelar no gelo e ressuspenda o pellet em 100 mL de tampão de lise (Tabela 1).
NOTA: O lisado celular deve sempre ser colocado no gelo durante o manuseio para evitar proteólise. Depois de descongelado, todo o processo de purificação deve ser concluído no mesmo dia. O lisado celular não deve ser armazenado na geladeira durante a noite.
- Transferir a suspensão para um copo de 250 ml e sonicar no gelo com 30 s ON e 30 s OFF a 35% de amplitude durante 15 min.
NOTA: Consulte as configurações do fabricante para identificar a amplitude. - Transferir a amostra sonicada para um tubo de centrifugação e centrifugar a 27 000 × g durante 30 min a 4 °C para obter um lisado límpido. Colocar o lisado de células em gelo no frigorífico a 4 °C até à injecção da amostra.
2. Preparando colunas de afinidade
- Encaixe as colunas (uma para a resina Ni-NTA e outra para a resina à base de cobalto) em um suporte de fixação de forma que as colunas sejam mantidas o mais retas possível, evitando qualquer inclinação das colunas. Certifique-se de que a parte inferior das colunas esteja conectada com uma rolha e adicione 1-2 mL de buffer inicial na parte inferior de cada coluna para evitar bolhas de ar durante o empacotamento da coluna.
- Agite os frascos de resina suavemente até que a resina esteja totalmente ressuspensa. Abra o topo das colunas e despeje 10 mL de cada pasta de forma contínua para evitar a inclusão de bolhas de ar. Despeje a resina contra uma espátula de metal ou haste de vidro presa na borda da coluna para evitar bolhas de ar na coluna.
3. Empacotamento de colunas usando um adaptador de fluxo
- Desconecte a rolha da coluna e conecte a parte inferior a uma extremidade da tubulação. Conecte a outra extremidade da tubulação à porta UV do sistema FPLC.
- Certifique-se de que a caixa de suporte da cama foi puxada para dentro do corpo do adaptador de fluxo e deslize o corpo do adaptador de fluxo para o topo da coluna (Figura 2A). Com a trava do came na posição 1, abaixe o adaptador de fluxo tomando cuidado para não inseri-lo no buffer (Figura 2B). Coloque a trava do came na posição 2 (Figura 2B). Conecte a tubulação ao sistema e inicie o fluxo do buffer de partida por 1-2 min para livrar a tubulação de ar e evitar bolhas de ar durante a embalagem.
- Coloque a trava de volta na posição 1 e abaixe o adaptador de fluxo no buffer em um movimento suave, evitando a inclusão de bolhas de ar, e coloque a trava de volta na posição 2. Isso permite que algum buffer entre no adaptador de fluxo e remova quaisquer bolhas restantes.
- Coloque a trava na posição 3 e aperte a base do came e o anel de travamento (Figura 2B).
- Inicie o fluxo de tampão a 10 mL / min para embalar a coluna. Continue esta etapa por 3 min ou até que a resina esteja bem embalada.
4. Preparando o loop de amostra do sistema FPLC
- Conecte a tubulação da parte superior do sample loop ao Loop E no sistema FPLC (Figura 3).
- Conecte a tubulação da parte inferior do sample loop ao Waste 2 no sistema FPLC.
- Insira uma extremidade da tubulação na porta da coluna do sistema FPLC e coloque a outra extremidade no frasco de resíduos.
- Insira a tubulação da bomba A em água bidestilada filtrada (ddH2O).
- Clique duas vezes nas configurações da bomba usando o software (Figura 4A, rotulado como 2) e selecione o loop de injeção da bomba do sistema (Figura 4C).
- Clique nas configurações de vazão (Figura 4A, rotulada como 1) e altere a vazão para 10 mL/min. Insira 0% na caixa %B (Figura 4B).
- Inicie o fluxo para lavar o sample loop com água, o que empurrará a vedação deslizante para baixo.
- Assim que a vedação deslizante atingir a parte inferior do sample loop, pare o fluxo. Comute a bomba A de ddH2O para o buffer inicial. Conecte a tubulação da parte superior do sample loop ao Waste 2 e da parte inferior ao Loop E. Deixe a tubulação da porta da coluna no waste.
- Inicie o fluxo a 10 mL/min. Assim que a vedação deslizante empurrada para cima pelo buffer de partida de entrada atingir o topo do sample loop, pare o fluxo. O loop de amostra agora está pronto para injeção de amostra.
5. Injeção de amostra
- Clique duas vezes nas configurações da bomba e selecione Loop de carga manual (Figura 4C).
- Desaparafuse o tubo do Loop E e coloque-o no recipiente de resíduos.
- Conecte a tubulação da porta da coluna à tubulação na parte superior do adaptador de fluxo.
- Extrair a amostra com uma seringa de 30 ml; Em seguida, conecte a seringa ao adaptador da porta de injeção.
- Enrosque a seringa na porta de injeção e injete a amostra, certificando-se de que ela seja visivelmente injetada no laço da amostra. Armazenar 100 μL da amostra ( lisado de entrada do rótulo) num tubo de microcentrífuga a -20 °C para análise SDS-PAGE
NOTA: Dependendo do volume total da amostra, podem ser necessárias 2-3 injeções. - Conecte a porta de injeção com a tampa da coluna.
- Conecte a tubulação da parte inferior do sample loop de volta à porta Loop E.
- Coloque a tubulação da bomba B no tampão de eluição.
6. Criação e análise de métodos usando o software vinculado ao sistema FPLC
- Navegue até a guia Página Inicial e selecione Novo método.
- Selecione a guia Configurações do método na coluna esquerda, navegue até o menu suspenso Tipo de coluna e selecione Personalizado (Figura 5).
- Insira o volume da coluna (5 mL).
- Insira a taxa de fluxo necessária para a execução (1 mL/min).
- Navegue até Seleção de unidade e selecione VC (volumes de coluna) como a unidade base do método.
- Certifique-se de que a Entrada A esteja definida como Buffer A e a Entrada B esteja definida como Buffer B (a configuração padrão).
- Selecione a guia Estrutura de tópicos do método no menu do lado esquerdo. Adicione as etapas da purificação na seguinte ordem: equilíbrio, aplicação de amostra, lavagem da coluna e eluição.
- Em equilíbrio, defina o volume de equilíbrio para 10 CV. Certifique-se de que a configuração do buffer seja mantida em 0% B.
- Na guia aplicativo de exemplo , defina o volume de exemplo observando o loop de exemplo. A posição da vedação deslizante indica o volume da amostra.
- Sob a lavagem da coluna, defina o volume de lavagem para 5 CV. Desabilite a coleta de frações para esta etapa; Colete a lavagem como um todo em um béquer durante a corrida, em vez de coletar como frações.
- Para eluição, defina o volume do gradiente para 14 CV a partir de 0% Buffer B a 100% Buffer B.
- Salve o método e inicie a execução.
- Certifique-se de que o coletor de frações comece na posição da primeira fração.
- Insira o nome da execução e clique em Iniciar.
- Durante a fase de equilíbrio, monitore a curva UV em tempo real. Certifique-se de que todas as conexões estejam seguras e sem vazamentos; Confirme um circuito completo procurando o buffer que flui para o recipiente de resíduos. Se a curva UV não se estabilizar durante os últimos minutos de equilíbrio, estenda a etapa de equilíbrio até que a estabilização seja alcançada (Figura 6) .
- No momento da aplicação da amostra, troque os tubos que vão para o recipiente de resíduos por um béquer limpo. Durante esta etapa, colete as proteínas não ligadas que saem dos tubos de resíduos como o fluxo (rotule como FT).
- Procure um aumento acentuado na curva UV que provavelmente atinja a leitura máxima possível de 3.000 maU (Figura 6).
NOTA: Uma curva típica se estabiliza em 3.000 maU durante a maior parte da etapa de aplicação da amostra. - Durante a lavagem da coluna, à medida que as proteínas residuais não ligadas estão sendo lavadas com apenas as proteínas ligadas permanecendo na resina, procure uma queda acentuada na curva UV (Figura 6). Se a curva UV não tiver caído até o final da etapa de lavagem, estenda a etapa clicando em manter a etapa até que o UV tenha caído o suficiente. No início desta etapa, transfira os tubos de resíduos do béquer FT para outro béquer limpo rotulado como Lavagem.
- Quando a etapa de eluição começar, certifique-se de que o coletor de frações se mova para a posição e comece a coletar frações. Visualize a eluição da proteína procurando picos na curva UV (Figura 6).
- Após a conclusão da execução, navegue até a guia Início e selecione Abrir Execução. Abra a execução para visualizar o cromatograma e selecione integração de pico no menu superior para marcar os picos obtidos durante a execução.
- Selecione a guia Frações para visualizar as frações que contêm proteína eluída.
7. Análise da PÁGINA SDS
- Misture 15 μL de cada fração contendo proteína com 5 μL de tampão de carga de amostra SDS (Tabela 1). Aquecer as amostras a 95 °C durante 5 min.
- Carregue as amostras em géis pré-moldados e execute a 160 V por 45 min junto com o marcador de proteína prestained.
- Manchar os géis na solução de coloração azul brilhante de Coomassie por 30 min (Tabela 1).
- Lave o gel em ddH2O (repita 3x).
- Desinstale o gel em solução de descoloração por 1 h (Tabela 1).
- Observe as bandas de proteínas; detectar FEN1 na marca de 42 kDa.
- Frações de piscina contendo FEN1 e concentrar a piscina usando um filtro centrífugo de 10 kDa. Alíquota e conservar no congelador a -80 °C.
Resultados
Os lisados celulares BL21 (DE3) que expressam hFEN1 foram passados através de resinas Ni-NTA e TALON balanceadas. A resina Ni-NTA é carregada com íons Ni2+ e tem uma alta capacidade de ligação. Os resultados mostram que a resina Ni-NTA produz uma quantidade maior de FEN1 em comparação com a resina TALON (Figura 7). A resina Ni-NTA também é conhecida por se ligar de forma não específica a outras proteínas expressas cromossomalmente. O lisado celular passou pela resina à base de cobalto, foi purificado com alta pureza, mas com menor rendimento, em comparação com o Ni-NTA. Como muitas outras proteínas de ligação ao DNA, a FEN1 foi posteriormente passada através da resina de exclusão de tamanho para remover as impurezas. hFEN1 pode ser identificado na marca de 43 kDa.
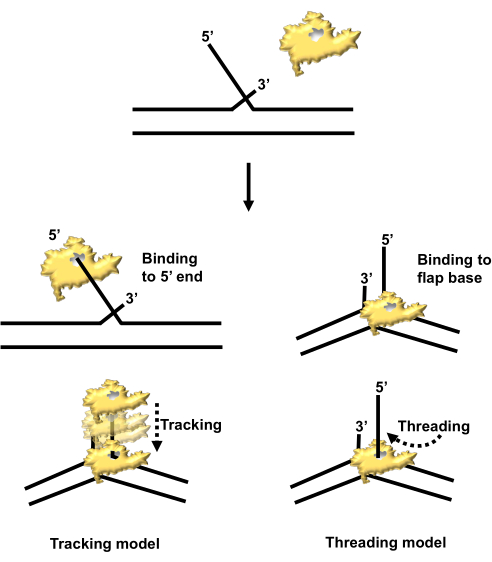
Figura 1: Mecanismo de clivagem do retalho por hFEN1: rastreamento versus rosqueamento. Inicialmente, pensava-se que o FEN1 (representado em amarelo) se ligava à extremidade 5' da estrutura do retalho e seguia através dela até a base do retalho (esquerda). Estudos posteriores revelaram que o FEN1 primeiro se liga à base do retalho e enfia o retalho de 5' através de seu sítio ativo (direita). Abreviatura: hFEN1 = endonuclease 1 do retalho humano. Clique aqui para ver uma versão maior desta figura.

Figura 2: Adaptador de fluxo. (A) Componentes do adaptador de fluxo usado para o processo de empacotamento e purificação da coluna; (B) as três posições da trava do came usadas para apertar/afrouxar o O-ring e fixar o adaptador no lugar. A imagem ampliada mostra o estado do O-ring (solto/apertado) nas posições 1,2 e 3 de cima para baixo. As imagens foram tiradas com permissão da 9. Clique aqui para ver uma versão maior desta figura.

Figura 3: Módulo de injeção do sistema de cromatografia líquida de proteína rápida. As várias portas do módulo de injeção conectam o sistema de cromatografia líquida de proteína rápida à coluna, loop de amostra e resíduos. A imagem foi tirada com permissão de 10. Clique aqui para ver uma versão maior desta figura.

Figura 4: Controle do sistema. (A) Guia de controle do sistema na qual as configurações de fluxo podem ser manipuladas e o caminho do fluxo pode ser visto; (B) janela de configurações de taxa de fluxo; (C) janela de configurações da bomba. Clique aqui para ver uma versão maior desta figura.
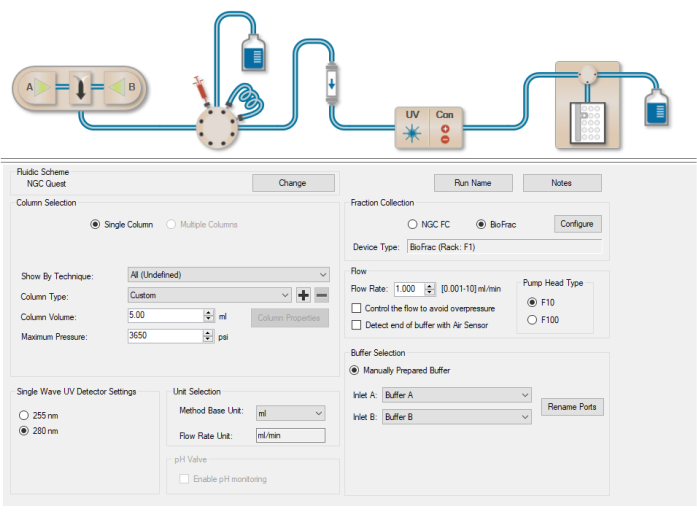
Figura 5: Janela de configurações do método. Uma janela para inserir detalhes de purificação, como tipo de coluna, volume de coluna, unidade base do método e taxa de fluxo para a execução. Clique aqui para ver uma versão maior desta figura.

Figura 6: Esquema da curva UV durante o FPLC: A curva UV sobe e desce de acordo com a fase de purificação. No início do equilíbrio, a curva UV pode ficar distorcida devido à presença de bolhas de ar ou contaminantes na tubulação. À medida que o equilíbrio continua, a curva deve se estabilizar como uma linha plana; Durante a injeção da amostra, à medida que as proteínas não ligadas à resina fluem, a curva UV aumenta até que um limite máximo seja atingido. A etapa de lavagem, que libera as proteínas residuais não ligadas, mostra uma diminuição gradual nas medições de UV. Finalmente, durante a fase de eluição, quando a proteína alvo é eluída, um pico é visto na curva UV. Clique aqui para ver uma versão maior desta figura.

Figura 7: Imagem em gel SDS PAGE. Pista 1-Marcador de peso molecular; Entrada Ni-NTA de pista 2-1% (lisado celular); Fluxo de pista 3-1% da resina Ni-NTA; Lane 4-hFEN1 purificado a partir de resina Ni-NTA; Entrada de 5-1% de pista (lisado celular); Lane 6-1% de fluxo da resina à base de cobalto; Lane 7-hFEN1 purificado a partir de resina à base de cobalto; a seta preta representa FEN1 a 42,5 kDa. Abreviaturas: SDS-PAGE = dodecil sulfato de sódio; MW = peso molecular; hFEN1 = endonuclease 1 do retalho humano; NTA = ácido nitrilotriacético. Clique aqui para ver uma versão maior desta figura.
| Buffers e soluções | Componentes |
| Tampão de lise | 50 mM Tris-HCl [pH 8,0] |
| 500 mM NaCl | |
| 1 mM de beta-mercaptoetanol (BME) | |
| 1 mM de fluoreto de fenil-metilsulfonilo (PMSF) | |
| 1 mM EDTA | |
| 1 comprimido de inibidor de protease completo | |
| 10 mM de imidazol | |
| 10% de glicerol | |
| Equilibração/Tampão de lavagem | 50 mM Tris-HCl [pH 8,0] |
| 500 mM NaCl | |
| 1 mM BME | |
| 1 mM PMSF | |
| 10 mM de imidazol | |
| 10% de glicerol | |
| Tampão de eluição | 50 mM Tris-HCl [pH 8,0] |
| 500 mM NaCl | |
| 1 mM BME | |
| 1 mM PMSF | |
| 1 M imidazol | |
| 10% de glicerol | |
| Buffer de carregamento de amostra SDS | Tampão de amostra Laemmli de 900 μL 4x |
| 100 μL de EMB | |
| Solução de coloração azul brilhante Coomassie | 50% ddH2O |
| 40% de metanol | |
| 10% de ácido acético | |
| 0,1% Azul brilhante Coomassie R-250 | |
| Solução de descoloração | 50% ddH2O |
| 40% de metanol | |
| 10% de ácido acético |
Tabela 1: Composição do tampão.
Discussão
A cromatografia de afinidade é uma técnica amplamente utilizada para purificar proteínas de ligação ao DNA. A cromatografia de afinidade de metal imobilizado (IMAC) é um tipo específico de cromatografia de afinidade que usa íons metálicos para capturar os resíduos de histidina de uma sequência de peptídeos. É por isso que a "etiqueta 6X-His" ou "etiqueta poli-His" é anexada ao terminal N ou ao terminal C das proteínas a serem purificadas. O níquel e o cobalto são os íons metálicos mais comumente usados e variam em sua compatibilidade com reagentes como BME e TDT normalmente usados na purificação. Embora as purificações tenham sido realizadas em concentrações de 1 mM BME ou DTT, o uso desses reagentes ainda pode não ser confiável. A incompatibilidade pode ser observada durante o equilíbrio. Se o tampão que flui através da resina parecer marrom, isso indica oxidação dos íons metálicos e pode retirar os íons metálicos da resina. Isso pode reduzir drasticamente a capacidade de ligação da resina.
Para garantir alta pureza e rendimento de proteína, as interações de afinidade entre íons metálicos e a marca His devem ser favorecidas em relação a outras possíveis interações baseadas em carga. Isso garantirá que toda a proteína seja eluída de uma só vez, em vez de eluída em diferentes fases da eluição. Para conseguir isso, a proteína deve ter uma carga muito baixa durante a purificação. Se os valores de pH dos tampões estiverem mais próximos do ponto isoelétrico da proteína alvo, a carga da proteína pode ser mantida baixa. Como o ponto isoelétrico do FEN1 é de aproximadamente 8,8, nossos tampões são mantidos em pH 8.
Acredita-se que a resina Ni-NTA, carregada com íons Ni, tenha uma maior capacidade de ligação a proteínas e produza uma quantidade maior de proteína em comparação com a resina à base de cobalto TALON. Isso também significa que há um maior potencial de ligação de proteínas não específicas e, portanto, menor pureza. A menor capacidade de ligação do TALON é complementada por maior ligação específica e pelo rendimento de proteína de alta pureza. Isso é consistente com nossos resultados mostrando um maior rendimento de proteína purificada de Ni-NTA em comparação com o TALON.
Independentemente da resina, a pureza e o rendimento são significativamente afetados pelo empacotamento da coluna. O empacotamento eficiente pode ser obtido usando o adaptador de fluxo conectado ao sistema de cromatografia automatizado NGC9. Em taxas de fluxo altas o suficiente, o adaptador de fluxo pode embalar efetivamente a resina e evitar o acúmulo de buffer na coluna. O adaptador de fluxo junto com o NGC permite a comparação precisa de ambas as resinas devido à regulação da taxa de fluxo, pressão e gradiente de eluição, bem como à coleta eficiente de frações de proteínas.
Por meio do software Chromlab, as condições de execução podem ser monitoradas em tempo real10. A taxa de fluxo pode ser alterada a qualquer momento durante a execução. Durante a aplicação da amostra, a pressão do sistema pode exceder o limite (600 psi). Nesse caso, diminuir a vazão durante a etapa de aplicação da amostra pode reduzir a pressão. O software também possui uma função de retenção de passo através da qual qualquer passo pode ser estendido conforme necessário. Esta função é particularmente útil durante as etapas de lavagem e eluição da coluna. Durante a lavagem da coluna, espera-se que o gráfico UV diminua e quase atinja 0 - isso significa que a maioria das proteínas não ligadas foi lavada da resina. Caso a linha UV continue a subir ou não desça, a etapa de lavagem pode ser estendida até que o gráfico esperado seja visto. Da mesma forma, durante a eluição, a eluição da proteína alvo pode ser visualizada através de picos no gráfico UV. Se, inesperadamente, nenhum pico for visível, a etapa de eluição pode ser estendida até que os picos previstos sejam observados.
Dicas e truques
Durante a etapa de equilíbrio, o tampão deve ser injetado na coluna e o tampão de resíduos deve ser continuamente liberado no frasco de resíduos; Isso garante que todas as conexões sejam firmes e à prova de vazamentos. Em caso de vazamento de qualquer local do NGC, todas as portas ao redor da área devem ser verificadas, pois a origem do vazamento pode ser diferente de onde o vazamento é percebido. Se o fluxo foi iniciado, mas nenhum buffer está entrando na coluna, é provável que o ar esteja preso dentro da tubulação. Limpe o sistema antes de continuar.
Durante a injeção de amostra, se a contrapressão do sistema estiver resistindo à injeção, a tubulação deve ser desparafusada do Loop E e colocada no frasco de resíduos durante a injeção. É importante aparafusá-lo de volta no Loop E antes de iniciar o programa. Caso o buffer de equilíbrio ou amostra seja acumulado na coluna durante a execução, a execução deve ser pausada e o adaptador de fluxo removido. A tubulação deve ser desparafusada da parte inferior da coluna e o tampão ou amostra deve fluir. Se a amostra fluir, certifique-se de coletá-la como o fluxo.
Se as frações executadas no gel SDS PAGE mostrarem altos níveis de proteínas contaminantes, as frações podem ser executadas em ambos os lados dos picos vistos no cromatograma. Freqüentemente, para proteínas de baixa expressão, as frações puras não apresentam picos significativos no UV.
Compreender as diferenças entre as propriedades da resina à base de níquel e da resina à base de cobalto permite a seleção da resina mais adequada para as futuras aplicações da proteína purificada. Por exemplo, espera-se que as proteínas usadas em ensaios bioquímicos in vitro tenham a mais alta pureza possível, uma situação em que a resina à base de cobalto é adequada. No entanto, se a proteína for usada para a produção de anticorpos, é necessário um rendimento significativo de proteína purificada para o processo. A resina Ni-NTA poderia servir como uma opção melhor neste caso. Este artigo teve como objetivo trazer à tona as diferenças nessas resinas através da purificação de FEN1.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Este trabalho foi financiado por doações da National Science Foundation (1929346) e da American Cancer Society (RSG-21-028-01). Também gostaríamos de agradecer aos membros do laboratório Balakrishnan pelas discussões úteis.
Materiais
| Name | Company | Catalog Number | Comments |
| 4x Laemmli sample buffer | BioRad | 1610747 | |
| Acetic acid | Merck | UN2789 | |
| Beta-mercaptoethanol (BME) | Sigma | M-6250 | |
| Chromlab software version 6.1.27.0 | BioRad | operates the NGC system | |
| Complete MINI protease inhibitor tablet | Roche | 11836153001 | |
| Coomassie Brilliant Blue R | Sigma | B0149 | |
| Dithiothreitol (DTT) | Dot Scientific | DSD11000 | |
| Econo-Column glass | BioRad | 7371512 | |
| Ethylene diamine tetraacetic acid (EDTA) | Dot Scientific | DSE57020 | |
| Flow adaptor | BioRad | 7380014 | |
| Glycerol | Dot Scientific | DSG22020 | |
| Imidazole | Dot Scientific | DSI52000 | |
| Methanol | Fisher Scientific | A412 | |
| Mini PROTEAN TGX gels | BioRad | 4561084 | |
| NGC Chromatography System | BioRad | automated liquid chromatography system | |
| Ni-NTA Agarose | Qiagen | 1018244 | |
| Phenyl-methyl-sulfonyl fluoride | Dot Scientific | DSP20270 | |
| PreScission Plus Protein Dual Color Standards | BioRad | 1610374 | |
| Sodium chloride | Dot Scientific | DSS23020 | |
| TALON metal affinity resin | Takara | 635502 | |
| Tris Base | DST60040 |
Referências
- Balakrishnan, L., Bambara, R. A. Flap endonuclease 1. Annu Rev Biochem. 82, 119-138 (2013).
- Asagoshi, K. FEN1 functions in long patch base excision repair under conditions of oxidative stress in vertebrate cells. Mol Cancer Res. 8 (2), 204-215 (2010).
- Lyamichev, V., Brow, M. A., Dahlberg, J. E. Structure-specific endonucleolytic cleavage of nucleic acids by eubacterial DNA polymerases. Science. 260 (5109), 778-783 (1993).
- Bornarth, C. J., Ranalli, T. A., Henricksen, L. A., Wahl, A. F., Bambara, R. A. Effect of flap modifications on human FEN1 cleavage. Biochemistry. 38 (40), 13347-13354 (1999).
- Xu, Y., Potapova, O., Leschziner, A. E., Grindley, N. D., Joyce, C. M. Contacts between the 5' nuclease of DNA polymerase I and its DNA substrate. J Biol Chem. 276 (32), 30167-30177 (2001).
- Wen-Hui, K., Kuo, K., Chase, H. A. Exploiting the interactions between poly-histidine fusion tagsand immobilized metal ions. Biotechnol Lett. 33 (6), 1075-1084 (2011).
- Charlton, A., Zachariou, M. Immobilized metal ion affinity chromatography of native proteins. Methods Mol Biol. 421, 25-35 (2008).
- Ononye, O. E., Njeri, C. W., Balakrishnan, L. Analysis of DNA processing enzyme FEN1 and its regulation by protein lysine acetylation. Methods Mol Biol. 1983, 207-224 (2019).
- Econo-column flow adaptor instruction manual. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/M7380014.pdf (2015)
- NGC Chromatography systems and ChromLab software instrument guide version 3.3. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/10026252.pdf (2015)
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados